É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Seqüenciamento da microflora bacteriana no sangue periférico: a nossa experiência com pacientes infectados pelo HIV
Neste Artigo
Resumo
Nossa experiência vai mostrar como realizar uma análise de seqüenciamento de translocação de espécies bacterianas no sangue periférico de pacientes HIV positivos.
Resumo
O trato gastrointestinal saudável é fisiologicamente colonizada por uma grande variedade de micróbios comensais que influenciam o desenvolvimento do sistema 1,2 humoral e celular imune da mucosa.
Microbiota é protegido do sistema imunológico através de uma forte barreira mucosa. Infecções e antibióticos são conhecidos por alterar tanto a barreira do trato gastrointestinal normal ea composição das bactérias residentes, o que pode resultar em possíveis alterações imunológicas 3.
HIV causa uma brecha na barreira gastrointestinal com insuficiência progressiva da imunidade da mucosa e vazamento para a circulação sistêmica de bioprodutos bacteriana, como lipopolissacarídeos e fragmentos de DNA de bactérias, que contribuem para a ativação imune sistêmica 4-7. Translocação microbiana está implicado em HIV / AIDS imunopatogênese e resposta à terapia 4,8.
Nosso objetivo foi caracterizar a composição de bactérias translocação no sangue periférico de pacientes infectados pelo HIV. A perseguir o nosso objectivo é criar uma reação de PCR para o gene 16S panbacteric ribosomial seguido por uma análise de seqüenciamento.
Resumidamente, o sangue total de ambos os sujeitos infectados pelo HIV e saudável é usado. Dado que os indivíduos saudáveis apresentam homeostase intestinal normal não translocação de microflora é esperado nesses pacientes. Após a colheita de sangue total por punção venosa e separação do plasma, o DNA é extraído do plasma e usado para executar uma ampla gama reação de PCR para o gene 16S panbacteric ribosomial 9. Análises de purificação seguinte produto PCR, clonagem e sequenciamento são executadas.
Protocolo
Manipulação de amostras de sangue contaminado pelo HIV requer algumas recomendações importantes.
Todas as amostras de sangue devem ser transportados em robusta contentores estanques. É preciso ter cuidado ao coletar a amostra para evitar a contaminação do exterior do container e de qualquer documentação que acompanha a amostra.
Todas as pessoas de processamento de sangue infectados devem usar luvas. As luvas devem ser mudadas e as mãos lavadas após a conclusão do processamento das amostras.
Processamento de amostras de sangue infectado pelo HIV deve ser feito em uma capa de gabinete classe II de risco biológico.
Mecânicos auxiliares de pipetagem deve ser usado.
Uso de agulhas ou outros instrumentos cortantes (incluindo por exemplo, pipetas de vidro ou tubos capilares) deve ser limitada a situações em que não há alternativa.
Superfícies de laboratório devem ser descontaminados com um desinfectante químico apropriado após um derramamento de sangue e quando as atividades são concluídas.
Materiais contaminados utilizados devem ser descontaminados antes de reutilizar ou deve ser eliminado de forma correcta por via de resíduos hospitalares.
Cada incidente sobre a exposição ocupacional a sangue ou fluidos potencialmente infectantes (ou seja, aqueles que requerem precauções universais) deve ser tratada como uma emergência médica, as intervenções devem ser iniciadas imediatamente para ser eficaz.
O protocolo exige cinco dias para sua conclusão. O prazo é detalhado na Figura 1.
Espécies bacterianas são identificados usando os métodos descritos na Figura 2.
1. Coleta de Amostras
- 9 ml de sangue total é desenhada em tubos contendo EDTA.
- Os tubos são centrifugados a 2000 rpm por 10 minutos a RT para obter plasma.
- O plasma é coletado em um 2 ml estéril Eppendorf-tubo.
2. Extração de DNA de amostras de plasma
- Desinfectar adequadamente capô, pipetas e materiais necessários para o experimento, a fim de garantir a esterilidade.
- Posição de todos os materiais sob luzes UV por pelo menos 30 minutos.
- Limpe as luvas com um desinfectante.
- Molhe um papel toalha poucos em etanol e colocá-los sob o capô. Cada vez que uma dica é descartado limpe a pipeta sobre as toalhas de papel molhado.
DNA é extraído usando um kit comercial, seguindo as instruções do fabricante (Easy-DNA Kit, Invitrogen, Carlsbad CA, EUA).
- 350 uL de plasma são colocados em um tubo eppendorf estéril de 2ml.
- 350 mL de água ultrapura filtrados são usados como controle negativo.
- Adicionar 10 mL de lisozima (1mg/ml) com as amostras.
- Incubar por 30 minutos a 37 ° C.
- Adicione 500 ml de solução de Lise e misture delicadamente com as amostras.
- Incubar durante 7 minutos a 65 ° C.
- Adicionar 900 mL de clorofórmio para as amostras.
- Vortex vigorosamente até que as amostras são uniformemente viscosa.
- Adicione 200 ml de solução-precipitação e vortex vigorosamente.
- Centrifugar as amostras a 10.500 rpm por 10 minutos a RT para separar as fases e formam a interface.
- Transferir a fase superior aquosa para um tubo de microcentrífuga fresco contendo 1 ml de etanol 100%.
- Centrifugar as amostras a 10.500 rpm por 10 minutos a 4 ° C.
- Remover o etanol.
- Adicionar 1 ml de etanol 70%.
- Centrifugar amostras na velocidade máxima por 10 minutos a 4 ° C.
- Remover o etanol. O etanol residual deve ser removida com uma pipettator.
- Ressuspender o sedimento em 50 mL de água ultrapura.
- Leia concentração de DNA com um espectrofotômetro.
3. Gene 16S rRNA PCR
Amplificação por PCR é realizado como descrito anteriormente 9.
- Amplificar DNA em uma mistura de reação de 100 L com 10 mL de tampão PCR 10X, 5 mL de 25 mM MgCl2, 5 mL de 2 mM dNTPs total, 1 mL de 50 mM de primer RW01 (AACTGGAGGAAGGTGGGGAT), 1 mL de 50 mM de primer DG74 (AGGAGGTGATCCAACCGCA), 34 mL de H20 e 0,5 mL de Taq polimerase (AmpliTaq Gold, Applied Biosystem, Foster City, CA, EUA).
- Transfira a mistura da PCR em filtros para evitar possível contaminação por Taq polimerase bacteriana.
- Filtros de centrifugação (microcontrolador, Millipore, Billerica, MA, EUA) por 30 minutos a 500 rcf a 4 ° C.
- Mix alíquota do PCR de acordo com o número de amostras que precisam ser amplificados (controles positivos e negativos de PCR, amostras de água e extraído).
- Use as seguintes condições termociclador: 94 ° C por 10 minutos, 40 vezes por 1 minuto cada vez que a 94 ° C, 55 ° C e 72 ° C e 10 min a 72 ° C.
- Visualize o produto da PCR em um gel de agarose 2%. Use 100 bp DNA ladder. PCR tamanho do produto é de aproximadamente 360 pb (Figura 3).
4. PCR Purification produto
Só PCR amostras positivas devem ser purificada. Purificação é realizada utilizandoinstruções de um fabricante de kit de seguir comercial (PureLink PCR MICROKIT, Invitrogen, Carlsbad CA, EUA).
- Adicionar 4 volumes de tampão de ligação contendo isopropanol por um volume de produto da PCR.
- Transferência de produto de PCR com tampão de ligação a uma coluna.
- Centrifugar por 1 minuto a 10,000 rcf na RT.
- Lavar coluna com tampão de lavagem contendo etanol.
- Centrifugar por 1 minuto a 10,000 rcf na RT.
- Centrifugar a velocidade máxima durante 1 minuto a RT para secar a membrana de sílica e remover qualquer tampão de lavagem residual com etanol.
- Adicionar 10 mL de água ultrapura.
- Incubar por 1 minuto à temperatura ambiente.
- Centrifugar a velocidade máxima por 2 minutos em temperatura ambiente para coletar o DNA purificado.
5. Clonagem
Lisogenia Caldo (LB) Preparação da placa
- Dissolver 25 gr de mistura LB em aproximadamente 800 ml de água.
- Trazer o volume final para 1 L.
- Adicionar 15 gr de Bactoagar.
- Autoclave por 20 minutos.
- Após autoclavagem, vigorosamente redemoinho da solução no frasco para misturar agar derretido.
- Arrefecer a solução à 50 ° C.
- Adicionar Ampicilina (50 mg / ml) e agite até que se dissolva.
- Preparar placas a uma profundidade de aproximadamente 3mm.
- Deixar as placas em temperatura ambiente.
- Spread X-Gal (40mg/ml) e IPTG (100 mM) em cada placa LB e incubar a 37 ° C até que esteja pronto para uso.
Transformação de células competentes
Transformação é feita usando instruções de um fabricante de kit de seguir comercial (Topo TA cloning kit, Invitrogen, Carlsbad CA, EUA).
- Prepare a mistura de reacção clonagem (1-4 mL do produto da PCR fresco, 1 mL de solução salina, 1 mL do vetor e água se necessário).
- Mistura de reacção gentilmente e incubar por 5-10 minutos em temperatura ambiente.
- Coloque a reação no gelo.
- Adicione 2 mL da reação de clonagem em um frasco de células competentes e misture delicadamente.
- Incubar no gelo por 30 minutos.
- De choque térmico as células por 30 segundos a 42 ° C.
- Transferir imediatamente o tubo em gelo.
- Adicionar 250 mL de meio.
- Tampe o tubo e agitar tighly o tubo horizontalmente a 37 ° C por 1 hora.
- Spread 50 mL de cada transformação em uma placa pré-aquecida seletivo.
- Incubar durante a 37 ° C.
Após a incubação, colônias brancas e azuis se desenvolvem em placas. Colônias brancas são positivos para PCR de inserção de produtos e colônias azuis são negativas para a PCR de inserção do produto.
6. Análise de seqüenciamento
Reação de sequenciamento
- A mistura é feita com estes reagentes: 2,5 mL de DNA, uma cartilha mL (5pmol / mL) 1,1 mL Big Dye (Applied Biosystem, Foster City, CA, EUA).
- Condições térmicas cycler: 96 ° C por 1 minuto, 25 vezes para 96 ° C por 10 segundos, 55 ° C por 15 segundos e 60 ° C por 4 min.
Purificação coluna
- Para cada reação, prepare uma coluna MicroSpin (Qiagen, Milan, Itália).
- Inverter coluna e vórtice de mistura de resina.
- Pressão fora da parte inferior da coluna, afrouxe a tampa ¼ de volta, e coloque em uma coluna de tubo de microcentrífuga.
- Centrífuga 3200 rpm por 1 minuto.
- Transferência de coluna para um tubo de microcentrífuga limpo.
- Pipeta cuidadosamente a reação de seqüenciamento toda PCR para o centro da coluna.
- Centrífuga 3200 rpm por 1 minuto.
- DNA purificado vai eluite no tubo (20 l de água ultrapura).
Análise de seqüenciamento
- Carregar a amostra purificada (20 l) em 96 com vários poços da placa.
- Carregue a placa para o seqüenciador. Sequenciadores de DNA automatizados gerar um cromatograma de quatro cores mostrando os resultados da execução do seqüenciamento.
- De entrada a seqüência de nucleotídeos como uma consulta nos bancos de dados seqüência de público. A pesquisa é realizada nas bases de dados NCBI e servidores, com a ferramenta de pesquisa local Alinhamento Básico (BLAST).
- Considere as bactérias apenas com homologias 98-100%.
7. Resultados representante
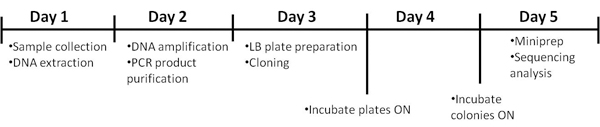
Figura 1. Timeline do procedimento.
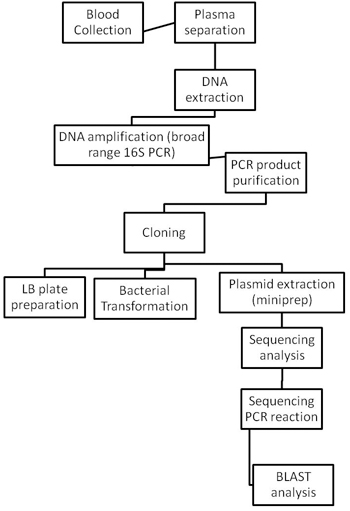
Figura 2. Fluxogramas dos processo de identificação bacteriana inteira.
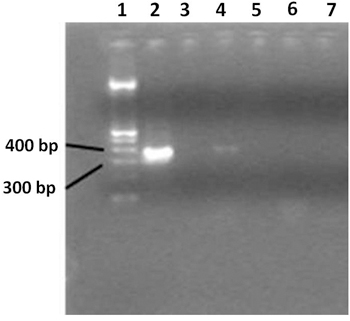
Figura 3. Gel agarose 2% mostrando 16S rRNA gene ampla gama de produtos PCR. Raia 1 contém uma escada de DNA 100bp, pista 2 contém o controle de PCR positiva, pista 3 mostra o controle PCR negativo. Faixa 4 mostra as amostras de um paciente HIV-positivo, e lane 5 contém água. Faixa 6 shows amostras de um indivíduo saudável e uma reação de PCR negativo; lane 7 contém água. Somente os pacientes HIV positivos exibe uma amplificação PCR positivo. Água ultrapura utilizada como controle negativo durante a etapa de extração, indica que nenhuma contaminação ocorreu.
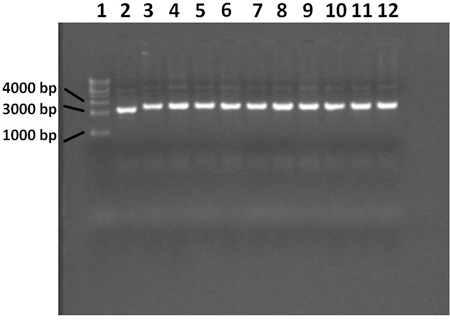
Figura 4. Gel 0,7% agarose mostrando plasmídeo extraído com o procedimento miniprep. Raia 1 contém um 1 escada de DNA kilobase, pista 2 contém a colônia de controle azul, faixas de 3 a 12 contêm colônias brancas. O plasmídeo da colônia azul não contém a bula do produto da PCR. Todas as 10 colônias brancas contêm plasmídeo com a inserção correta.

Figura 5. Mostra um exemplo de análise de seqüenciamento de bactérias no plasma de um indivíduo HIV-positivo. Nossos resultados mostram que a translocação microbiana em HIV doença envolve uma flora polimicrobic, que não é visto em HIV-negativos assuntos, sugerindo falha substancial de imunidade intestinal no controle da translocação bacteriana.
Access restricted. Please log in or start a trial to view this content.
Discussão
Vimos por este meio mostrar um protocolo de PCR / seqüenciamento para caracterizar a translocação de bactérias no sangue periférico de indivíduos infectados pelo HIV.
Resultados mais confiáveis são obtidos quando se utiliza plasma coletadas em tubos contendo EDTA. Após coleta de sangue, plasma deve ser separado por centrifugação dentro de 03/02 horas, para evitar hemólise e degradação fragmento de DNA. As amostras são armazenadas primeiro em -20 ° C por aproxima...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Agradecimentos
Somos gratos a Gianni Scimone para assistência excelente, com tomada de vídeo.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número Catálogo | Comentários (opcional) |
|---|---|---|---|
| Easty DNA-Kit | Invitrogen | K 180001 | |
| Clorofórmio | Sigma-Aldrich | C2432 | |
| Etanol | Sigma-Aldrich | E7203 | |
| UltraPure Waer | Invitrogen | 10977049 | |
| Lisozima | Fluka | 62970 | 10 mcg / mL em água destilada |
| AmpliTaq Gold | Applied Biosystem | 26478701 | |
| Microcontrolador 100 | Millipore | 42413 | |
| PCR | Invitrogen | ||
| Agarose | Eppendorf | C1343 | |
| Escada de DNA | Invitrogen | 15628019 | |
| Purelink PCR micro kit | Invitrogen | K310050 | |
| LB Agar em pó, | Invitrogen | 22700025 | |
| Bactoagar | Invitrogen | ||
| Ampicilina | Invitrogen | 11593027 | 10 mg / mL em água destilada |
| X-gal | Invitrogen | 15520034 | 40mg/ml em DMF |
| IPTG | Invitrogen | 15529019 | 100mM em água destilada |
| Topo TA cloning kit | Invitrogen | K450002 | |
| Purelink rápida kit Miniprep Plasmid | Invitrogen | K210010 | |
| Big dye | Applied Biosystem | 4337455 | |
| DyeEx 2,0 spin-kit | Qiagen | 63204 |
Referências
- Hooper, L. V., Macpherson, A. J. Immune adaptations that maintain homeostasis with the intestinal microbiota. Nat Rev Immunol. 10, 159-169 (2010).
- Macpherson, A. J., Harris, N. L. Interactions between commensal intestinal bacteria and the immune system. Nat Rev Immunol. 4, 478-485 (2004).
- Kanauchi, O., Mitsuyama, K., Araki, Y., Andoh, A. Modification of intestinal flora in the treatment of inflammatory bowel disease. Curr Pharm Des. 9, 333-346 (2003).
- Brenchley, J. M. Microbial translocation is a cause of systemic immune activation in chronic HIV infection. Nat Med. 12, 1365-1371 (2006).
- Brenchley, J. M., Price, D. A., Douek, D. C. HIV disease: fallout from a mucosal catastrophe. Nat Immunol. 7, 235-239 (2006).
- Jiang, W. Plasma levels of bacterial DNA correlate with immune activation and the magnitude of immune restoration in persons with antiretroviral-treated HIV infection. J Infect Dis. 199, 1177-1185 (2009).
- Marchetti, G. Microbial translocation is associated with sustained failure in CD4+ T-cell reconstitution in HIV-infected patients on long-term highly active antiretroviral therapy. AIDS. 22, 2035-2038 (2008).
- Marchetti, G. Role of Microbial Translocation and Immune Hyperactivation in Disease Progression of HIV+ Patients with Preserved CD4 Count in the Absence of ART. The 17th Conference on Reteroviruses and Opportunistic Infections (CROI), San Francisco, CA, USA, , (2010).
- Greisen, K., Loeffelholz, M., Purohit, A., Leong, D. PCR primers and probes for the 16S rRNA gene of most species of pathogenic bacteria, including bacteria found in cerebrospinal fluid. J Clin Microbiol. 32, 335-351 (1994).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados