Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Site spécifique génie chromosome bactérien: ΦC31 intégrase médiation Cassette Exchange (IMCE)
Dans cet article
Résumé
Une méthode rapide et efficace d'intégrer l'ADN étranger dans des souches d'intérêt accepteurs pré-faites, dites souches piste d'atterrissage, est décrite. Procédé permettant d'intégration spécifique de site d'une cassette d'ADN dans le locus piste d'ingénierie d'une souche donnée, par conjugaison et l'expression de l'intégrase ΦC31.
Résumé
Le chromosome bactérien peut être utilisé pour maintenir de façon stable l'ADN étranger dans la plage méga-base 1. Intégration dans le chromosome contourne questions comme la réplication du plasmide, le plasmide de la stabilité, incompatibilité plasmidique, et le plasmide de la variance du nombre de copies. Cette méthode utilise l'intégrase spécifique de site à partir du phage Streptomyces (Φ) C31 2,3. L'intégrase catalyse une recombinaison ΦC31 direct entre deux sites d'ADN spécifiques: attB et attP (34 et 39 pb, respectivement) 4. Cette recombinaison est stable et ne pas revenir 5. Un "piste" (LP) de séquence constitué par un gène de résistance à la spectinomycine-, aadA (SpR), et le E. coli ß-glucuronidase gène (uidA) flanqué par des sites attP a été intégré dans les chromosomes de Sinorhizobium meliloti, Ochrobactrum anthropi et Agrobacterium tumefaciens dans une région intergénique, l'ampC locus, et le locus tetA, respectivement. S. meliloti est utilisé dans ce protocole. Vecteurs donateurs mobilisables contenant des sites attB flanquant un stuffer protéine fluorescente rouge (RFP) gène et un gène de résistance aux antibiotiques ont également été construits. Dans cet exemple la gentamicine pJH110 plasmide de résistance est utilisé. Le gène DP 6 peuvent être remplacés par une construction souhaité à l'aide Sph I et Pst I. Sinon, une construction synthétique flanqué par des sites attB peut être sous-cloné dans un vecteur mobilisable comme pK19mob 7. L'expression du gène de l'intégrase ΦC31 (cloné à partir d'pHS62 8) est entraîné par le promoteur lac, sur une large gamme d'hôtes mobilisable plasmide pRK7813 9.
Un protocole d'accouplement tetraparental est utilisé pour transférer la cassette des bailleurs de fonds dans la souche LP remplaçant ainsi les marqueurs dans la séquence LP avec la cassette des bailleurs de fonds. Ces cellules sont trans-intégrants. Trans-intégrants sont formées avec un rendement typique de 0,5%. Trans-intégrants se trouvent généralement dans les premières colonies 500-1000 dépistées par la sensibilité aux antibiotiques ou bleu-blanc de dépistage utilisant le 5-bromo-4-chloro-3-indolyl-beta-D-acide glucuronique (X-gluc). Ce protocole contient les procédures d'accouplement et de sélection pour la création et l'isolement de trans-intégrants.
Protocole
1. La production de la culture
- Préparer stériles milieux liquides: TY 10 (5 g / l de tryptone, extrait de levure 3 g / l, 0,44 g / l de chlorure de calcium dihydraté), et LB 11 (10 g / l de tryptone, extrait de levure 5 g / l à 5 g / le chlorure de sodium, pH 7).
- Inoculer à partir d'une seule colonie: SmUW227 (S. meliloti LP-souche: la construction sera décrite ailleurs, la souche de détails de construction sont disponibles sur demande) () dans 5 ml de TY médias avec 50 pg / ml de spectinomycine. Ensemencer les souches suivantes dans 5 ml de milieu liquide LB, additionnel avec l'antibiotique donné: E. coli DH5a 12 contenant pJC2, l'expression de l'intégrase plasmide, avec 10 pg / ml de tetracycline; E. coli MT616 13, le mobilisateur, avec 10 pg / ml chloramphénicol, et E. coli DH5a contenant pJH110, la cassette plasmide donneur, avec 5 pg / ml de gentamicine.
- Incuber E. Les souches de E. nuit à 37 ° C avec une agitation constante. Incuber S. souche meliloti à 30 ° C pendant deux jours avec agitation. Il est possible d'obtenir un S. la culture meliloti nuit avec un plus grand inoculum (1:500 sous-culture d'une culture saturée).
2. Préparation de la culture et de mixage
- Laver 1,5 ml de culture pour chaque souche par la collecte du culot cellulaire par centrifugation à 17 000 xg pendant 30 secondes et la remise en suspension dans 1 ml stérile 0,85% de NaCl; répétition une fois.
- Resuspendre le culot dans 100 ul de solution stérile de NaCl à 0,85%.
- Individuellement repérer 10 ul de la culture lavée pour chaque souche sur une plaque de gélose TY plaine. Ces taches sont des taches de contrôle.
- Ajouter 40 ul de chaque souche à l'exclusion du E. coli DH5a 12 contenant pJC2 dans un tube stérile, mélange, et la tache 120 pi de ce mélange sur une plaque de gélose TY plaine. Cet endroit est le contrôle non-négatif de l'intégrase.
- Ajouter 40 ul de chaque souche dans un t stérileube, mélange, et la tache 160 ul de ce mélange sur une plaque de gélose TY plaine. Cet endroit est le lieu d'accouplement IMCE.
- Permettez-taches à sécher dans un hotte à flux laminaire.
- Plaques d'étanchéité et incuber à 30 ° C pendant la nuit.
3. Isolement de Trans-intégrants
- Taches de contrôle Streak et place IMCE sur des plaques d'agar TY complété avec 200 ug / ml de streptomycine (SmUW227 est basé sur Rm1021 qui porte une mutation conférant une résistance de haut niveau à la streptomycine 14) et 30 pg / ml de gentamicine.
- Pour le calcul de l'efficacité IMCE, resuspendre environ ¼ de la tache d'accouplement IMCE dans 500 ul stérile à 0,85% de NaCl. Assurez-10-dilutions à 10 -7. Dilutions 10 -2 plaques par 10 -5 sur gélose TY complétées avec 200 ug / ml de streptomycine et 30 ug / ml de gentamicine, de sélectionner pour trans-intégrants. Dilutions plaques 10 -3 through 10 -7 sur gélose TY complétées avec 200 ug / ml de streptomycine, de sélectionner pour les deux trans-intégrants et les bénéficiaires potentiels, à savoir les bénéficiaires au total.
- Pour l'isolement de sans marqueurs trans-intégrants resuspendre environ un quart de la tache d'accouplement IMCE dans 500 pi de solution stérile de NaCl à 0,85%. Assurez-10-dilutions à 10 -6. Dilutions plaques 10 -4 à 10 -6 sur quatre des plaques d'agar TY complétées avec 200 ug / ml de streptomycine et 200 pg / ml X-gluc.
- Incuber les plaques à 30 ° C pendant 3 jours.
- Voir DP trans-intégrants sous la lumière verte (525 nm) et à travers un filtre rouge (> 610 nm) 15.
- Calculer l'efficacité IMCE pour cent en UFC de trans-intégrants par rapport à la UFC de bénéficiaires au total. Environ la moitié des trans-intégrants aura subi un échange de cassette vrai ce qui les rend blanche et à la spectinomycine sensible.
- Pour trouver les sans marqueurs trans-intégrants (wici le vecteur donneur ne contient pas de Gm r comme dans pJH110) écran pour une colonie blanche (une période d'incubation prolongée, 1-2 jours supplémentaires à la température ambiante, permettra d'améliorer la différenciation des colonies par X-gluc, cela est nécessaire dans S. meliloti SmUW227), confirmer la sensibilité à la spectinomycine de la colonie par le dépistage sur une plaque de gélose TY sans antibiotiques et une plaque de gélose TY contenant 100 ug / ml de spectinomycine.
4. Les résultats représentatifs
Après trois jours d'incubation sur TY complétée avec la streptomycine et la gentamicine les stries de contrôle ne devrait pas avoir de croissance. La série IMCE devrait avoir une croissance confluente sur la strie la tête et de nombreuses colonies sur la strie seconde, comme on le voit dans la figure 2. L'efficacité de la trans-intégration, exprimée en pourcentage de la trans-intégrants pour les bénéficiaires au total, devrait être de l'ordre de 0,5%. Environ la moitié des trans-intégrants sera spectinomycine sensibles et blancattestant qu'ils ont subi un échange de cassette vrai. Trans-intégrants contenant la cassette testeur donateurs rfp de pJH110 devrait afficher discernable fluorescence DP lorsqu'on les examine sous la lumière verte (525 nm) et à travers un filtre rouge (> 610 nm) 15.
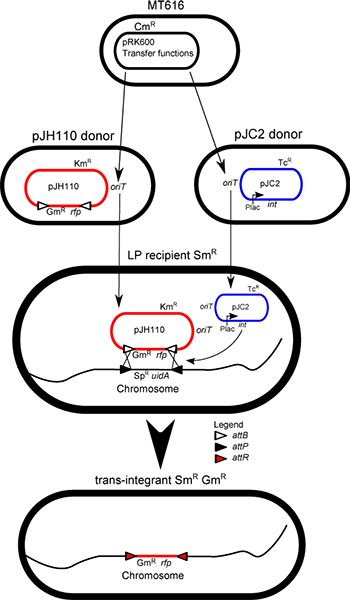
. Figure 1 illustre un mélange Conjugaison: Aidé par l'expression des gènes de transfert de pRK600, tous les plasmides sont transférés au hasard de la cellule à cellule. Ce transfert se traduit par la création de trans-intégrants dans le mélange, grâce à l'acquisition de la LP-souche de ces deux plasmides requis pour IMCE, l'intégrase (int) pJC2 plasmide auxiliaire et le pJH110 plasmide donneur. La cassette des bailleurs de fonds du donneur non-réplication plasmidique (pJH110) sont échangées via ΦC31 activité de l'intégrase avec les marqueurs de la LP-cassette sur le chromosome, ce qui entraîne la perte de la LP-marqueurs (Spr et uidA) et le maintien de la cassette des bailleurs de fonds (DP et Gm r) dans le trans-résultante intégrante.
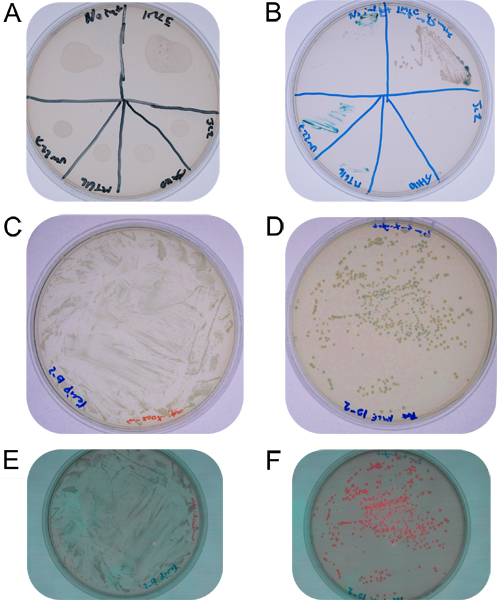
Figure 2. A: B plaque à godets accouplement sur une plaque non-sélective TY montrant mélanges de cellules séchées à la surface de la gélose:. Points d'accouplement striées de la streptomycine-gentamicine-X-gluc plaque, en haut à gauche dans le sens horaire à partir aucun contrôle de l'intégrase, IMCE en S. meliloti, E. coli DH5a contenant le contrôle pJC2, E. coli contenant DH5a pJH110 commande, E. coli MT616 de contrôle, et S. . meliloti UW227 de contrôle C: 10 -2 dilution de la remise en suspension place d'accouplement sur TY streptomycine-X-gluc agar D:. 10 -2 dilution de la remise en suspension place d'accouplement sur les TY-streptomycine gentamicine-X-gluc agar (colonies bleues sont recombinants simples, colonies blanches ont fait l'objet vrai. échange cassette) E: 10 -2 dilution de la remise en suspension place d'accouplement sur TY streptomycine-X-gluc agar montrant l'absence de fluorescence F:. 10 -2 dilution de la remise en suspension place d'accouplement sur les TY-streptomycine gentamicine-X-gluc agar montrant deux niveaux de fluorescence (brillantes colonies correspondent à des colonies bleues et ont une expression plus élevée rfp probablement à cause de promoteur lecture de la séquence si vecteur, où les colonies ayant subi vrai-cassette d'échange contiennent des DP avec seulement son promoteur immédiate sans lecture par le biais du promoteur lac dans le vecteur, qui est absent.).
Discussion
La technique IMCE permet l'intégration efficace d'une cassette d'ADN flanqué attB unique dans le LP-lieu d'une souche auparavant conçu. Une fois la construction désirée est cloné en place de la DP pour créer la cassette des bailleurs de fonds, la technique ne nécessite pas de purification ultérieure de l'ADN et de la transformation, ce qui rend très robuste. Il est essentiel que les contrôles de croissance appropriés sont inclus, pour être certain de la résistance aux ...
Déclarations de divulgation
Pas de conflits d'intérêt déclarés.
Remerciements
Pour Margaret Smith CM d'avoir bien voulu fournir le clone de l'intégrase
Le soutien financier de:
Génome Canada / Génome Prairie
La découverte du CRSNG et des subventions de projets stratégiques
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Entreprise | Numéro de catalogue | Commentaires |
| Streptomycine | Bioshop Canada Inc | STP101 | |
| Spectinomycine | Bioshop Canada Inc | SPE201 | |
| La gentamicine | Bioshop Canada Inc | GTA202 | |
| Choramphénicol | Bioshop Canada Inc | CLR201 | |
| Tétracycline | Bioshop Canada Inc | TET701 | |
| Kanamycine | Bioshop Canada Inc | KAN201 | |
| Bactériologiques agar de qualité | Bioshop Canada Inc | AGR001 | |
| Tryptone | Bioshop Canada Inc | TRP402 | |
| Extrait de levure | Bioshop Canada Inc | YEX401 | |
| Chlorure de sodium | Bioshop Canada Inc | SOD001 | |
| Chlorure de calcium | Bioshop Canada Inc | CCL444 | |
| X-gluc | Or Biotechnology Inc | G1281C1 | |
| E. coli MT616 souche | Disponible sur demande | Également utilisé en dehors de notre laboratoire | |
| E. coli souche pJC2 | Dans la maison, disponibles sur demande | ||
| E. coli souche pJH110 | Dans la maison, disponible en request | ||
| SmUW227 souche | Dans la maison, disponibles sur demande |
Références
- Itaya, M., Tsuge, K., Koizumi, M., Fujita, K. Combining two genomes in one cell: Stable cloning of the Synechocystis PCC6803 genome in the Bacillus subtilis 168 genome. Proc. Natl. Acad. Sci. U.S.A. 102, 15971-15976 (2005).
- Kushtoss, S., Rao, R. N. Analysis of the Integration Function of the Streptomycete Bacteriophage FC31. J. Mol. Biol. 222, 897-908 (1991).
- Brown, W. R., Lee, N. C., Xu, Z., Smith, M. C. Serine recombinases as tools for genome engineering. Methods. 53, 372-379 (2011).
- Groth, A. C., Olivares, E. C., Thyagarajan, B., Calos, M. P. A phage integrase directs efficient site-specific integration in human cells. Proc. Natl. Acad. Sci. U.S.A. 97, 5995-6000 (2000).
- Rowley, P. A., Smith, M. C., Younger, E. A motif in the C-terminal domain of FC31 integrase controls the directionality of recombination. Nuc. Acid. Res. 36, 3879-3891 (2008).
- Campbell, R. E. A monomeric red fluorescent protein. Proc. Natl. Acad. Sci. U.S.A. 99, 7877-7882 (2002).
- Schafer, A. Small mobilizable multi-purpose cloning vectors derived from the Escherichia coli plasmids pK18 and pK19: selection of defined deletions in the chromosome of Corynebacterium glutumicum. Gene. 145, 69-73 (1994).
- Thorpe, H. M., Smith, M. C. In vitro site-specific integration of bacteriophage DNA catalyzed by a recombinase of the resolvase/invertase family. Proc. Natl. Acad. Sci. 95, 5505-5510 (1998).
- Jones, J. D., Gutterson, N. An efficient mobilizable cosmid vector, pRK7813, and its use in a rapid method for marker exchange in Pseudomonas fluorescens strain HV37a. Gene. 61, 299-306 (1987).
- Beringer, J. E. R Factor transfer in Rhizobium leguminosarum. J. Gen. Microbiol. 84, 188-198 (1974).
- Lennox, E. S. Transduction of linked genetic characters of the host by bacteriophage P1. Virology. 1, 190-206 (1955).
- Hanahan, D. Studies on transformation of Escherichia coli with plasmids. J. Mol. Biol. 166, 557-580 (1983).
- Charles, T. C., Finan, T. M. Genetic map of Rhizobium meliloti megaplasmid pRmeSU47b. J. Bacteriol. 172, 2469-2476 (1990).
- Leigh, J. A., Signer, E. R., Walker, G. C. Exopolysaccharide-deficient mutants of Rhizobium meliloti that form ineffective nodules. Proc. Natl. Acad. Sci. U.S.A. 82, 6231-6235 (1985).
- Heil, J. R., Nordeste, R. F., Charles, T. C. The fluorescence theatre: a cost-effective device using theatre gels for fluorescent protein and dye screening. Can. J. Microbiol. 57, 339-342 (2011).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proc. Natl. Acad. Sci. U.S.A. 97, 6640-6645 (2000).
- Lesic, B., Rahme, L. G. Use of the lambda Red recombinase system to rapidly generate mutants in Pseudomonas aeruginosa. BMC Mol. Biol.. 9, 20-20 (2008).
- Choi, K. -. H., Schweizer, H. P. mini-Tn7 insertion in bacteria with single attTn7 sites: example Pseudomonas aeruginosa. Nat. Protocols. 1, 153-161 (2006).
- Thomason, L. C., Calendar, R., Ow, D. W. Gene insertion and replacement in Schizosaccharomyces pombe mediated by the Streptomyces bacteriophage FC31 site-specific recombination system. Molecular Genetics and Genomics. 265, 1031-1038 (2001).
- Katzen, F. Gateway recombinational cloning: a biological operating system. Expert Opin. Drug Discovery. , 571-586 (2007).
- Charles, T. C., Doty, S. L., Nester, E. W. Construction of Agrobacterium strains by electroporation of genomic DNA and its utility in analysis of chromosomal virulence mutations. Appl. Environ. Microbiol. 60, 4192-4194 (1994).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon