Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Site-spezifische bakterielle Chromosom Engineering: ΦC31 Integrase vermittelte Cassette Exchange (IMCE)
In diesem Artikel
Zusammenfassung
Eine schnelle und effiziente Methode, um Fremd-DNA von Interesse in vorgefertigte Akzeptor Stämme, sogenannte Landing-Pad-Stämme, die Integration beschrieben wird. Das Verfahren erlaubt eine ortsspezifische Integration eines DNA-Kassette in die gentechnisch Landeplatz Ort einer gegebenen Dehnung, durch Konjugation und Expression des ΦC31 Integrase.
Zusammenfassung
Die Bakterien-Chromosom kann verwendet werden, um stabil zu halten Fremd-DNA in der Mega-Basen-Bereich 1 werden. Integration in das Chromosom umgeht Themen wie Plasmidreplikation, der Stabilität von Plasmiden, Plasmid Inkompatibilität und Plasmid-Kopienzahl Varianz. Diese Methode verwendet die ortsspezifische Integrase aus dem Streptomyces Phagen (Φ) C31 2,3. Die Integrase katalysiert ΦC31 eine direkte Rekombination zwischen zwei spezifischen DNA-sites: attB und attP (34 und 39 bp) 4. Diese Rekombination ist stabil und nicht zurückkehren 5. Ein "Landeplatz" (LP)-Sequenz bestehend aus einer Spectinomycin-Resistenz-Gen, aadA (SPR) und der E. coli ß-Glucuronidase-Gen (uidA) von attP flankiert in die Chromosomen der Sinorhizobium meliloti, Ochrobactrum anthropi und Agrobacterium tumefaciens in einem Zwischengenregion integriert wurde, bin daspC Locus und die tetA Locus, jeweils. S. meliloti ist in diesem Protokoll verwendet. Mobilisierbaren Vektoren, die Donor attB flankieren eine Stuffer rot fluoreszierendes Protein (RFP)-Gen und ein Antibiotika-Resistenz-Gen wurde ebenfalls konstruiert. In diesem Beispiel wird Gentamicin resistente Plasmid pJH110 verwendet wird. Der RFP-Gen 6 kann mit einem gewünschten Konstrukt mit Sph I und Pst I ersetzt werden Alternativ kann ein synthetisches Konstrukt von attB-Stellen flankiert sein können subkloniert in einen mobilisierbaren Vektor wie pK19mob 7. Die Expression des Integrase-Gens ΦC31 (kloniert von pHS62 8) durch den lac-Promotor angetrieben wird, auf einem mobilisierbaren Plasmids mit breitem Wirtsbereich pRK7813 9.
Ein tetraparental passenden Protokoll wird verwendet, um den Spender Kassette in der LP-Stamm und ersetzt die Marker in der LP-Sequenz mit der Donor-Kassette zu übertragen. Diese Zellen sind trans-Integranten. Trans-Integranten werden mit einem typischen Wirkungsgrad von 0,5% gebildet. Trans-Integranten werden typischerweise innerhalb der ersten 500-1,000 Kolonien durch Antibiotika-Empfindlichkeit oder blau-Weiß-Screening unter Verwendung von 5-Brom-4-chlor-3-indolyl-beta-D-Glucuronsäure (X-gluc) gescreent vorhanden. Dieses Protokoll enthält die Paarungs-und Auswahlverfahren für das Erstellen und Isolieren von trans-Integranten.
Protokoll
1. Die Produktion von Kultur
- Planen sterilen flüssigen Medien: TY 10 (5 g / l Trypton, 3 g / l Hefeextrakt, 0,44 g / l Calciumchlorid-Dihydrat), und LB 11 (10 g / l Trypton, 5 g / l Hefeextrakt, 5 g / Natriumchlorid, pH 7).
- Impfen von einer einzigen Kolonie: SmUW227 (S. meliloti LP-Stamm: Bau wird an anderer Stelle beschrieben werden, Stamm Bau Details auf Anfrage) () in 5 ml TY-Medium mit 50 pg / ml Spectinomycin. Beimpfen die folgenden Stämme in 5 ml LB flüssige Medien, in Ergänzung mit dem Antibiotikum gegeben: E. coli DH5 12 mit pJC2, die Integrase-Expressionsplasmid, mit 10 ug / ml Tetracyclin, E. coli MT616 13, die Mobilizer, mit 10 ug / ml Chloramphenicol, und E. coli DH5 mit pJH110, der Spender Kassette Plasmid, mit 5 pg / ml Gentamicin.
- Inkubieren E. coli-Stämme über Nacht bei 37 ° C unter ständigem Schütteln. Inkubieren S. meliloti-Stamm bei 30 ° C für zwei Tage unter Schütteln. Es ist möglich, einen S. zu erhalten meliloti Kultur über Nacht mit einem größeren Inokulum (1:500 Subkultur einer gesättigten Kultur).
2. Kultur Vorbereitung und Mixing
- Waschen 1,5 ml der Kultur für jeden Stamm durch das Sammeln des Zellpellets durch Zentrifugation bei 17.000 × g für 30 Sekunden und Resuspendieren in 1 ml steriler 0,85% NaCl; Wiederholung einmal.
- Pellet in 100 ul sterilem 0,85% NaCl.
- Individuell vor Ort 10 ul gewaschene Kultur für jeden Stamm auf einer Ebene TY-Agar-Platte. Diese Punkte sind Steuer-Spots.
- In 40 ul von jedem Stamm ohne den E. coli DH5 12 mit pJC2 in einem sterilen Röhrchen, mischen, und vor Ort 120 ul dieser Mischung auf einer Ebene TY-Agar-Platte. Dieser Spot ist der No-Integrase negative Kontrolle.
- In 40 ul jedes Stammes in einem sterilen tube, mischen, und vor Ort 160 ul dieser Mischung auf einer Ebene TY-Agar-Platte. Dieser Spot ist der Punkt IMCE Paarung.
- Willkommen Spots in einer Sterilbank trocknen.
- Seal Platten und inkubieren bei 30 ° C über Nacht.
3. Isolierung von Trans-Integranten
- Streak-Steuerung und Flecken IMCE Spot auf TY-Agar-Platten mit 200 ug / ml Streptomycin (SmUW227 basiert auf Rm1021 Basis, die eine Mutation verleiht High-Level-Resistenz gegen Streptomycin 14 trägt) und 30 mu g / ml Gentamicin ergänzt.
- Für die Berechnung der IMCE Effizienz, resuspendieren ca. ¼ der IMCE Paarung vor Ort in 500 ul steriler 0,85% NaCl. Machen Sie 10-fache serielle Verdünnungen bis 10 -7. Platten Verdünnungen 10 -2 bis 10 -5 auf TY-Agar-Platten mit 200 ug / ml Streptomycin und 30 mu g / ml Gentamicin, ergänzt um für trans-Integranten wählen. Platten Verdünnungen 10 -3 through 10 -7 auf TY-Agar-Platten mit 200 ug / ml Streptomycin, ergänzt um für beide trans-Integranten und potentiellen Empfängern, dh insgesamt Empfänger auszuwählen.
- Für die Isolierung von trans-markerlosen Integranten resuspendieren etwa ein Viertel der IMCE Paarung vor Ort in 500 ul sterilem 0,85% NaCl. Make 10-fache serielle Verdünnungen bis 10 -6. Platten Verdünnungen 10 -4 bis 10 -6 auf vier TY Agarplatten mit 200 ug / ml Streptomycin und 200 ug / ml X-Gluc ergänzt.
- Die Platten bei 30 ° C für 3 Tage.
- Zeige RFP trans-Integranten unter grünem Licht (525 nm) und durch einen roten Filter (> 610 nm) 15.
- Berechnen Sie die prozentuale IMCE Effizienz als CFU von trans-Integranten der CFU von insgesamt Empfängern verglichen. Etwa die Hälfte der trans-Integranten wird wahr Kassette Austausch erfahren haben was sie weiß und Spectinomycin und Kleinschreibung.
- Um die markerlosen trans-Integranten (w findenhier die Donor-Vektor nicht enthalten Gm r wie in pJH110)-Bildschirm für eine weiße Kolonie (ein erweiterter Inkubationszeit 1-2 zusätzliche Tage bei Raumtemperatur, wird die Differenzierung der Kolonien über X-gluc zu verbessern, ist dies notwendig, S. meliloti SmUW227), bestätigen der Kolonie Spectinomycin Empfindlichkeit durch Screening auf einer TY-Agarplatte ohne Antibiotika und einem TY-Agar-Platte mit 100 pg / ml Spectinomycin.
4. Repräsentative Ergebnisse
Nach drei Tagen Inkubation auf TY mit Streptomycin und Gentamicin die Kontrolle Streifen sollte kein Wachstum. Die Streifen sollten IMCE konfluenten Wachstums auf dem Kopf Streifen und viele Kolonien auf dem zweiten Streifen, wie in 2 zu sehen. Die Effizienz des Transports Integration, als der Prozentsatz von trans-Integranten zu insgesamt Empfänger exprimiert, sollte im Bereich von 0,5%. Etwa die Hälfte der trans-Integranten wird Spectinomycin empfindliche und weißzeigen sie wahre Kassette Austausch unterzogen. Trans-Integranten, die die Tester RFP Spender Kassette aus pJH110 anzeigen soll erkennbar RFP Fluoreszenz, wenn sie unter grünem Licht (525 nm) und durch einen roten Filter (> 610 nm) 15 angesehen.
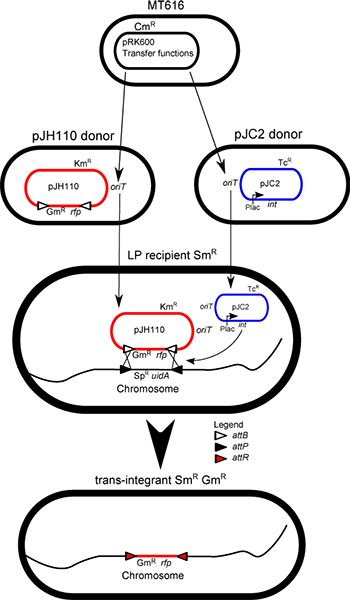
. 1 Konjugieren Mischung Darstellung: Mit Hilfe der Expression der Gene aus pRK600 Übertragung, werden alle Plasmide zufällig von Zelle zu Zelle übertragen. Diese Übertragung führt zur Erstellung von trans-Integranten in der Mischung, durch die LP-Belastung der Übernahme der beiden Plasmide für IMCE, die Integrase (int) Helferplasmid pJC2 und das Donorplasmid pJH110 erforderlich. Der Spender Kassette aus dem nicht-replizierenden Donorplasmid (pJH110) über ΦC31 Integrase-Aktivität mit den Markern der LP-Kassette auf dem Chromosom ausgetauscht, was zu einem Verlust der LP-Marker (Spr und uidA) und die Aufrechterhaltung des Spenders Kassette (RFP und Gm r) in der resultierenden trans-Integranten.
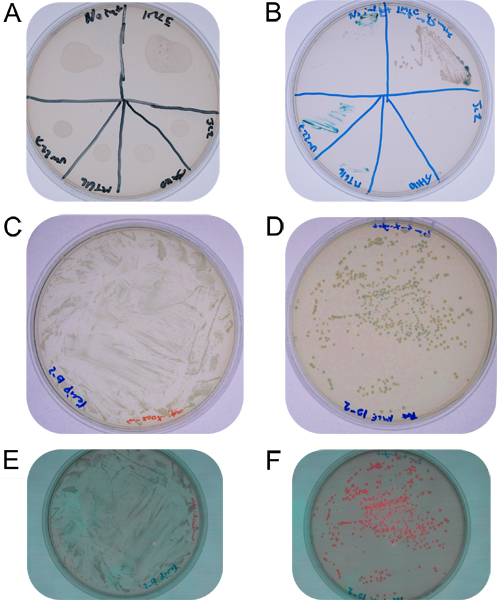
Abbildung 2. A:. Mating Spot Platte auf einem nicht-selektiven TY Schild mit getrockneten Zell-Mischungen auf der Agar-Oberfläche B: Mating Flecken auf Streptomycin-Gentamicin-X-Gluc Platte ausgestrichen, von links oben keine Integrase-Regler im Uhrzeigersinn, IMCE in S. meliloti, E. coli DH5 mit pJC2 Kontrolle, E. coli DH5a mit pJH110 Kontrolle, E. coli MT616 Kontrolle, und S. . meliloti UW227 Kontrolle C: 10 -2 Verdünnung der Paarung Spot Resuspension auf TY Streptomycin-X-Gluc Agar D:. 10 -2 Verdünnung der Paarung Spot Resuspension auf TY-Streptomycin Gentamicin-X-Gluc-Agar (blaue Kolonien sind einzelne Rekombinanten, weiße Kolonien unterzogen wurden wahr. Kassettenaustausch) E: 10 -2 Verdünnung der Paarung Spot Resuspension auf TY Streptomycin-X-Gluc-Agar zeigt fehlende Fluoreszenz F:. 10 -2 Verdünnung der Paarung Spot Resuspension auf TY-Streptomycin Gentamicin-X-Gluc-Agar zeigt zwei Ebenen der Fluoreszenz (heller Kolonien, blaue Kolonien entsprechen und eine höhere Expression rfp vermutlich auf Lese-Promotor wenn von Vektorsequenz, wo Kolonien viele Veränderungen und-Kassettenaustausch enthalten RFP mit nur seinen unmittelbaren Promotor ohne Durchlese vom lac-Promotor in mit der Vektor ist die nicht vorhanden.).
Access restricted. Please log in or start a trial to view this content.
Diskussion
Die IMCE Technik ermöglicht die effiziente Integration von einem einzigen attB flankiert DNA-Kassette in die LP-Locus eines zuvor entwickelten Stamm. Sobald das gewünschte Konstrukt anstelle der RFP, den Spender Kassette erstellen geklont wird, kommt die Technik nicht erforderlich, anschließende DNA-Reinigung und Transformation, so dass es sehr robust. Es ist wichtig, dass geeignete Wachstumsbedingungen Steuerelemente enthalten sind, sicher zu sein, die Resistenz gegen Antibiotika aufgrund der Erzeu...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Um Margaret CM Smith für die freundliche Bereitstellung des Integrase-Klon
Drittmittelförderung von:
Genome Kanada / Genome Prairie
NSERC Discovery and Strategic Project Zuschüsse
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | Kommentare |
| Streptomycin | Bioshop Kanada Inc. | STP101 | |
| Spectinomycin | Bioshop Kanada Inc. | SPE201 | |
| Gentamicin | Bioshop Kanada Inc. | GTA202 | |
| Chloramphenicol | Bioshop Kanada Inc. | CLR201 | |
| Tetracycline | Bioshop Kanada Inc. | TET701 | |
| Kanamycin | Bioshop Kanada Inc. | KAN201 | |
| Bakteriologische Qualität Agar | Bioshop Kanada Inc. | AGR001 | |
| Trypton | Bioshop Kanada Inc. | TRP402 | |
| Hefeextrakt | Bioshop Kanada Inc. | YEX401 | |
| Kochsalz | Bioshop Kanada Inc. | SOD001 | |
| Kalziumchlorid | Bioshop Kanada Inc. | CCL444 | |
| X-gluc | Gold-Biotechnology Inc. | G1281C1 | |
| E. coli-Stamm MT616 | Auf Anfrage erhältlich | Auch außerhalb unseres Labors verwendet | |
| E. coli-Stamm pJC2 | Im Haus, auf Anfrage erhältlich | ||
| E. coli-Stamm pJH110 | Im Haus, verfügbar durch request | ||
| SmUW227 Belastung | Im Haus, auf Anfrage erhältlich |
Referenzen
- Itaya, M., Tsuge, K., Koizumi, M., Fujita, K. Combining two genomes in one cell: Stable cloning of the Synechocystis PCC6803 genome in the Bacillus subtilis 168 genome. Proc. Natl. Acad. Sci. U.S.A. 102, 15971-15976 (2005).
- Kushtoss, S., Rao, R. N. Analysis of the Integration Function of the Streptomycete Bacteriophage FC31. J. Mol. Biol. 222, 897-908 (1991).
- Brown, W. R., Lee, N. C., Xu, Z., Smith, M. C. Serine recombinases as tools for genome engineering. Methods. 53, 372-379 (2011).
- Groth, A. C., Olivares, E. C., Thyagarajan, B., Calos, M. P. A phage integrase directs efficient site-specific integration in human cells. Proc. Natl. Acad. Sci. U.S.A. 97, 5995-6000 (2000).
- Rowley, P. A., Smith, M. C., Younger, E. A motif in the C-terminal domain of FC31 integrase controls the directionality of recombination. Nuc. Acid. Res. 36, 3879-3891 (2008).
- Campbell, R. E. A monomeric red fluorescent protein. Proc. Natl. Acad. Sci. U.S.A. 99, 7877-7882 (2002).
- Schafer, A. Small mobilizable multi-purpose cloning vectors derived from the Escherichia coli plasmids pK18 and pK19: selection of defined deletions in the chromosome of Corynebacterium glutumicum. Gene. 145, 69-73 (1994).
- Thorpe, H. M., Smith, M. C. In vitro site-specific integration of bacteriophage DNA catalyzed by a recombinase of the resolvase/invertase family. Proc. Natl. Acad. Sci. 95, 5505-5510 (1998).
- Jones, J. D., Gutterson, N. An efficient mobilizable cosmid vector, pRK7813, and its use in a rapid method for marker exchange in Pseudomonas fluorescens strain HV37a. Gene. 61, 299-306 (1987).
- Beringer, J. E. R Factor transfer in Rhizobium leguminosarum. J. Gen. Microbiol. 84, 188-198 (1974).
- Lennox, E. S. Transduction of linked genetic characters of the host by bacteriophage P1. Virology. 1, 190-206 (1955).
- Hanahan, D. Studies on transformation of Escherichia coli with plasmids. J. Mol. Biol. 166, 557-580 (1983).
- Charles, T. C., Finan, T. M. Genetic map of Rhizobium meliloti megaplasmid pRmeSU47b. J. Bacteriol. 172, 2469-2476 (1990).
- Leigh, J. A., Signer, E. R., Walker, G. C. Exopolysaccharide-deficient mutants of Rhizobium meliloti that form ineffective nodules. Proc. Natl. Acad. Sci. U.S.A. 82, 6231-6235 (1985).
- Heil, J. R., Nordeste, R. F., Charles, T. C. The fluorescence theatre: a cost-effective device using theatre gels for fluorescent protein and dye screening. Can. J. Microbiol. 57, 339-342 (2011).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proc. Natl. Acad. Sci. U.S.A. 97, 6640-6645 (2000).
- Lesic, B., Rahme, L. G. Use of the lambda Red recombinase system to rapidly generate mutants in Pseudomonas aeruginosa. BMC Mol. Biol.. 9, 20-20 (2008).
- Choi, K. -H., Schweizer, H. P. mini-Tn7 insertion in bacteria with single attTn7 sites: example Pseudomonas aeruginosa. Nat. Protocols. 1, 153-161 (2006).
- Thomason, L. C., Calendar, R., Ow, D. W. Gene insertion and replacement in Schizosaccharomyces pombe mediated by the Streptomyces bacteriophage FC31 site-specific recombination system. Molecular Genetics and Genomics. 265, 1031-1038 (2001).
- Katzen, F. Gateway recombinational cloning: a biological operating system. Expert Opin. Drug Discovery. , 571-586 (2007).
- Charles, T. C., Doty, S. L., Nester, E. W. Construction of Agrobacterium strains by electroporation of genomic DNA and its utility in analysis of chromosomal virulence mutations. Appl. Environ. Microbiol. 60, 4192-4194 (1994).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten