Method Article
Surveillance spatiale Ségrégation en surface Colonizing populations microbiennes
Dans cet article
Résumé
Le rôle de l'assortiment (ségrégation spatiale) dans les scénarios d'évolution peut être examinée à l'aide des systèmes microbiens simples en laboratoire qui permettent le réglage de la distribution spatiale contrôlée. En modifiant la densité cellulaire fondateur, différents niveaux d' assortiment peuvent être visualisées en utilisant des souches bactériennes marquées par fluorescence dans les biofilms de colonies de Bacillus subtilis.
Résumé
Microbes provide an intriguing system to study social interaction among individuals within a population. The short generation times and relatively simple genetic modification procedures of microbes facilitate the development of the sociomicrobiology field. To assess the fitness of certain microbial species, selected strains or their genetically modified derivatives within one population, can be fluorescently labelled and tracked using microscopy adapted with appropriate fluorescence filters. Expanding colonies of diverse microbial species on agar media can be used to monitor the spatial distribution of cells producing distinctive fluorescent proteins.
Here, we present a detailed protocol for the use of green- and red-fluorescent protein producing bacterial strains to follow spatial arrangement during surface colonization, including flagellum-driven community movement (swarming), exopolysaccharide- and hydrophobin-dependent growth mediated spreading (sliding), and complex colony biofilm formation. Non-domesticated isolates of the Gram-positive bacterium, Bacillus subtilis can be utilized to scrutinize certain surface spreading traits and their effect on two-dimensional distribution on the agar-solidified medium. By altering the number of cells used to initiate colony biofilms, the assortment levels can be varied on a continuous scale. Time-lapse fluorescent microscopy can be used to witness the interaction between different phenotypes and genotypes at a certain assortment level and to determine the relative success of either.
Introduction
Dans les dernières décennies, les microbes ont été reconnus comme des communautés sociales associées aux divers écosystèmes sur terre 1,2. Contrairement aux cultures planctoniques utilisées dans la pratique générale du laboratoire, les microbes dans l'environnement montrent un large éventail de structures communautaires spatiales en fonction du contexte écologique. Les systèmes microbiens simples peuvent être utilisés pour comprendre l'importance des structures spatiales sur l'évolution des interactions sociales 3,4. Publications dans les 2-3 dernières années à l' aide des deux systèmes modèles eucaryotes et procaryotes ont mis en évidence l'impact des structures spatiales sur la stabilité de la coopération au sein des populations microbiennes 5-8. En outre, obliger les interactions entre les microbes, par exemple métabolique alimentation croisée, pourraient également modifier la distribution spatiale des partenaires d' interaction 9-11. L'influence de la structure spatiale dans ces études est principalement examinée à l'aide de surface attachée cellules sessiles habitant la sorte-disant biofilms ou dans des colonies en croissance sur la surface d'un milieu gélosé. La dérive génétique résultant en grande variété spatiale peut être observée dans les colonies microbiennes où l' épuisement des nutriments au bord d'un résultat d'expansion de la division cellulaire à médiation en série de goulets d'étranglement génétique qui provoque une forte probabilité de fixation locale pour certains lignages clonales 12. La dérive génétique peut donc être utilisé pour examiner le rôle de la ségrégation spatiale dans les colonies microbiennes.
Dans l'environnement, les biofilms sont des communautés multispécifiques entourées par la matrice polymère auto-produit 13. La structure du biofilm, la fonction et la stabilité dépendent d'un réseau complexe d'interactions sociales où les signaux d'échange de bactéries, des composants et des ressources matrice, ou en compétition pour l' espace et les nutriments en utilisant des toxines et des antibiotiques. Bacillus subtilis est un vivant dans le sol et la bactérie racine colonisant qui développe très organisé communautés biofilm 14. Par analogie avec l'socialeinsectes, B. cellules subtilis emploient une division de la stratégie de la main - d'œuvre, le développement de sous - populations de producteurs de la matrice extracellulaire et anthropophages, cellules mobiles, les spores dormantes et d' autres types de cellules 15,16. Le processus de différenciation est dynamique et peut être modifiée par les conditions environnementales 17,18.
Stratégies de colonisation en surface par les bactéries peuvent être facilement manipulées dans des conditions de laboratoire en modifiant la concentration d'agar dans le milieu de croissance. À des niveaux de gélose faible (0,2-0,3%), les bactéries hébergeant flagelles actives sont capables de nager, tandis que la gélose semi-solide (0,7-1% d' agar) facilite flagelle communautaire entraîné la propagation, appelé grouillant 19-21. En l'absence de flagelles, certaines souches bactériennes sont capables de se déplacer sur un milieu semi-solide par glissement, à savoir l' expansion de la population dépendante de la croissance facilitée par la matrice de exopolysaccharide et d' autres composés sécrétés hydrophobine 22-24. Enfin, les bactéries qui sont capable de la forme de développement de biofilm colonies architecturalement complexes sur un milieu agar dur (1,2-2% de ) 14,17,25. Bien que ces traits sont examinés dans le laboratoire en ajustant précisément les conditions, dans les habitats naturels de ces stratégies épandage en surface pourrait transit progressivement de l' un à l' autre en fonction des conditions environnementales 26. Alors que seule la motilité à base de cellules est essentielle lors de l' initiation du développement d' un biofilm à l'interface air-liquide dans des bactéries Gram-positives et 27 séronégatifs, biofilms complexes de colonies de B. subtilis ne sont pas affectés par la suppression de la motilité des flagelles 28. Cependant, l' organisation spatiale lors du développement de B. subtilis biofilm de colonies dépend de la densité de l'inoculum bactérien utilisé pour initier le biofilm 8.
Ici, nous utilisons B. subtilis montrent que la ségrégation spatiale pendant la colonisation de la surface dépend du mécanisme de motili au niveau des populationsty (c. -à fourmillement ou de glissement) et le développement des colonies de biofilm dépend de la densité cellulaire fondatrice. Nous présentons un outil de microscopie à fluorescence qui peut être appliqué pour surveiller en permanence la croissance microbienne du biofilm, la colonisation de surface et de l'assortiment à l'échelle macro. En outre, une méthode de quantification est présenté pour déterminer l'abondance de la déformation relative de la population.
Protocole
1. Préparation de la culture des médias, des plaques d'agar et biofilms semi-solide, pré-cultures
- Préparation moyen pour Essaimage et coulissante
- Dissoudre 2 g de Lenox Broth (LB) et 0,7 g d'agar-agar dans 100 ml d'eau permutée et autoclave pendant 20 min à 120 ° C. Utilisez de petits volumes (50-200 ml) pour améliorer la reproductibilité entre les expériences.
- Immédiatement après la stérilisation, fermer le bouchon de la bouteille moyenne pour réduire l'évaporation et le placer dans un incubateur à 55 ° C pendant au moins 2 h.
- Après que la température moyenne a tempéré à 55 ° C, verser 20 ml de gélose de milieu LB à 90 mm de diamètre polystyrène boîte de Pétri stérile sous une hotte de laboratoire. Pour les expériences time-lapse, verser 5 ml milieu gélose LB par 35 mm de diamètre en polystyrène boîte de Pétri.
- Fermez la boîte de Pétri immédiatement après la coulée, empiler pas plus de 4 plaques au-dessus de l'autre et de laisser le milieu de gélose solidifier pendant au moins 1 h.
- 2xSG Medium Préparation pour Colony biofilms
- Dissoudre 1,6 g de bouillon nutritif, 0,2 g de KCl, 0,05 g de MgSO 4 7H 2 O, et 1,5 g d'agar-agar dans 100 ml d' eau permutée et autoclave pendant 20 min à 120 ° C. Utilisez de petits volumes (50-200 ml) pour améliorer la reproductibilité entre les expériences.
- Immédiatement après la stérilisation, fermer le bouchon de la bouteille moyenne pour réduire l'évaporation et de placer la bouteille dans un incubateur à 55 ° C pendant au moins 2 h.
- Après que la température moyenne est auto-ajustée à 55 ° C, ajouter 0,1 ml stérilisé par filtration 1M Ca (NO 3) 2 solution, 0,1 ml stérilisé par filtration mM MnCl2 solution 100, 0,1 ml stérilisé par filtration mM de FeSO4 solution 1 et 0,5 ml une solution de glucose à 20% stérile.
- Dans une hotte stérile de laboratoire, versez 20 ml de milieu agar 2x SG par 90 mm de diamètre en polystyrène boîte de Pétri. Pour les expériences time-lapse, verser 5 ml milieu gélose LB par 35 mm de diamètre en polystyrène boîte de Pétri.
- Close la boîte de Pétri immédiatement après la coulée, empiler la plaque sur le dessus de l'autre, mais pas plus de 4 plaques, et que le milieu de gélose se solidifier pendant au moins 1 h.
- Préparation des cultures de démarrage
NOTE: Le B. subtilis 168, NCIB 3610 souches dérivées utilisées dans les procédés décrits ci - dessous constitutivement produire des protéines vert ou au rouge fluorescence et ont été décrits avant 8,27. Souches sont conservées en routine dans le congélateur à -80 ° C.- Inoculer cultures starter de C stocks de -80 ° dans 3 ml de milieu LB et incuber pendant la nuit (16-18 h) à 37 ° C à l'horizontale agitation (225 rpm). Ne pas incuber la culture de plus de 18 heures aussi sauvage isole de B. subtilis sont essentiellement sujettes à l' agrégation et à former un biofilm dans le tube à essai.
2. Co-inoculation de fluorescence labelisée souches bactériennes pour la surface d'épandage
- Séchage des semi-solides plaques d'agar pour Essaim etGlissement de B. subtilis.
- des plaques d'agar à sec pour l'essaimage et le glissement pendant 20 minutes avant l'inoculation. Des plaques sèches non couvertes dans une hotte à flux laminaire (voir figure 1).
NOTE: fourmillement bactérienne et le glissement dépend de la sécheresse du milieu de gélose semi-solide. Séchage insuffisant permet l'accumulation d'eau sur le milieu de gélose résultant en natation du flagelle médiée. Résultats prolongées de temps de séchage en l'absence de l'essaimage.
- des plaques d'agar à sec pour l'essaimage et le glissement pendant 20 minutes avant l'inoculation. Des plaques sèches non couvertes dans une hotte à flux laminaire (voir figure 1).
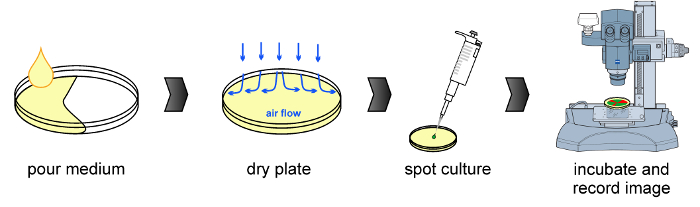
Figure 1:.. Flux de travail expérimental La procédure commune est représentée sur la figure, y compris la préparation du milieu de culture, le séchage de la détection plaque, inoculation et la microscopie à fluorescence (de gauche à droite) S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
- Co-inoculationdes cultures bactériennes pour Essaimage et coulissantes
- Déterminer les densités optiques des cultures de départ durant la nuit à 600 nm et mélanger des souches au vert et au rouge-fluorescentes normalisées densité protéine produisant de B. subtilis NCIB 3610 ou son dérivé Δ hag dans un tube de 1,5 ml de réaction. Par exemple, mélanger 100 pi de la souche 1 avec (100 * [DO 600 de la culture pendant la nuit de la souche 1] / [DO 600 de la culture pendant la nuit de la souche 2]) ul de la souche 2. Modérément vortex (3 sec à vitesse max) pour une répartition homogène.
NOTE: B. subtilis NCIB 3610 souches sont inoculées à observer l' essaimage, tandis que leurs dérivés Δ hag sont utilisés pour glisser. - Point 2 pl de culture mixte sur le milieu d'une plaque pré-séchée (voir la figure 1) et sécher en outre la plaque pendant 10 minutes après l' inoculation.
- Incuber les plaques à 37 ° C en position verticale pour permettre l'excès d'humidité se condense sur le couvercle et non sur la surface de la gélose.
NOTE: Le temps d' incubation pour B. subtilis essaimage est typiquement comprise entre 8-16 h. En règle générale, le bord de l'essaim atteint le côté de la 90 mm boîte de Pétri en 8 h. Sliding est un processus plus lent et nécessite au moins 16-42 heures d'incubation. Au bout de 36 heures, le front de glissement atteint le côté de la boîte de Petri de 90 mm. - Pour les expériences time-lapse, placez le diamètre de 35 mm des boîtes de Petri dans une chambre d'incubation de stade préchauffé réglé à 37 ° C. Assurez-vous que le couvercle de la boîte de Petri reste retirée pendant toute la durée de l'expérience. Fixer le couvercle de l'incubateur d'étape à 40 ° C pour contourner la formation de buée sur la partie supérieure de l'incubateur.
- Déterminer les densités optiques des cultures de départ durant la nuit à 600 nm et mélanger des souches au vert et au rouge-fluorescentes normalisées densité protéine produisant de B. subtilis NCIB 3610 ou son dérivé Δ hag dans un tube de 1,5 ml de réaction. Par exemple, mélanger 100 pi de la souche 1 avec (100 * [DO 600 de la culture pendant la nuit de la souche 1] / [DO 600 de la culture pendant la nuit de la souche 2]) ul de la souche 2. Modérément vortex (3 sec à vitesse max) pour une répartition homogène.
3. Co-inoculation par fluorescence Labelled Souches bactériennes avec différentes densités cellulaires initiales
- Séchage des plaques de gélose pour la formation Colony biofilms de B. subtilis.
- Sécher les plaques pour le développement des colonies de biofilm sans couverture dans un flux laminairehotte pendant 15 minutes avant l'inoculation.
REMARQUE: Les résultats de séchage insuffisant à une augmentation de l' humidité et de la natation ou l' essaimage peut être possible 29. Le séchage trop longs résultats dans les petites colonies de biofilms.
- Sécher les plaques pour le développement des colonies de biofilm sans couverture dans un flux laminairehotte pendant 15 minutes avant l'inoculation.
- Préparation de 10 fois dilué Cultures de démarrage pour Colony biofilms
- Mélanger 100 pi de protéine vert- et rouge fluorescent produisant des cultures de démarrage durant la nuit de B. subtilis 168 dans un tube réactionnel de 1,5 ml et légèrement au vortex pendant une distribution homogène. Préparer une série de dilutions de 10 fois dans du milieu LB.
- Spot 2 pi de non-dilué ou 10 1, 10 2, 10 3, 10 4 cultures mixtes dilué sur la plaque contenant du milieu biofilm induisant.
REMARQUE: 6 à 9 colonies de biofilms peuvent être engagées sur un seul 90 mm boîte de Petri en prenant en compte que les colonies sont séparées à égale distance les uns des autres. - Incuber les plaques à 30 ° C en position verticale pour permettre l'excès d'humidité se condense sur le couvercle etnon pas sur la surface de la gélose.
NOTE: Le temps d'incubation de B. subtilis biofilm est compris entre 1 et 3 jours. En général, le biofilm de colonies de B. subtilis atteint sa taille moyenne et de la structure complexe en 2 jours. - Pour les expériences time-lapse, placez un seul inoculum au milieu d'un diamètre boîte de Pétri de 35 mm et placez le plat dans une chambre d'incubation de l'étape préchauffé fixée à 30 ° C. Faire en sorte que la partie supérieure de la boîte de Petri reste retirée pendant toute la durée de l'expérience. Fixer le couvercle de l'incubateur de l'étape à 35 ° C pour contourner la formation de buée sur la partie supérieure de l'incubateur.
4. Détection de microscopie à fluorescence Labelled Souches
- Description de l'équipement pour l'imagerie.
- Pour détecter la colonisation de surface et le signal de fluorescence, utiliser un zoom microscope motorisé fluorescence stéréo (voir liste détaillée dans le tableau Matériaux) équipé d'un objectif 0.5X Planapo, deux LEDsources de lumière froide (un pour la détection par fluorescence et un pour la lumière visible), des jeux de filtres pour la GFP (excitation à 470/40 nm et émission à 525/50 nm) et mRFP (excitation à 572/25 nm et émission à 629 / 62 nm) et une caméra monochrome à haute résolution.
- Effectuer l'acquisition et le traitement d'image avec un logiciel approprié disponible pour le microscope à zoom stéréo y compris multicanal et modules time-lapse. Pour l'expérience de laps de temps, utiliser un incubateur d'étape de chauffe standard monté sur le microscope à zoom stéréo avec un adaptateur.
- Imagerie de Essaim et expansion coulissante
- Utiliser le grossissement le plus faible pour capter la plus grande surface possible de la plaque de 90 mm. Définir l'origine de l'inoculation (milieu de l'assiette de 90 mm de Pétri) au coin du champ visible pour le suivi radial d'expansion bactérienne et fluorescence.
- Ajuster le temps d'exposition optimal en fonction de l'intensité du signal de fluorescence.
NOTE: Pour expre constitutivementgènes de fluorescence SSED dans B. subtilis, au vert et au rouge fluorescence avec 1,5 et 3 sec temps d'exposition peuvent être utilisés, respectivement. En outre, 10 msec temps d'exposition est appropriée pour la lumière visible.
- Utiliser le grossissement qui permet la détection de toute la colonie du biofilm et ajuster la colonie au milieu du champ de vision.
NOTE: En ce qui concerne l'essaimage et les expansions de glissement, les temps d'exposition optimal pour détecter les signaux de fluorescence dans les colonies de biofilm dépend du niveau des gènes codant des protéines fluorescentes d'expression. Pour les résultats représentatifs ci-dessous, au vert et au rouge, la fluorescence a été détectée en utilisant des intervalles de 1 à 3 secondes d'exposition, respectivement. - Pour l'imagerie time-lapse, obtenir des images à certains intervalles en utilisant des temps d'exposition constants.
- Enregistrez les images enregistrées fluorescence stéréomicroscope dans un format de fichier qui est reconnu par le logiciel ImageJ pour l'analyse quantitative des données.
5. Datune analyse
- Pour analyser la surface occupée par chaque souche fluorescente différemment marqué, ouvrez le fichier d'intérêt dans le logiciel ImageJ élargi avec un plugin BioVoxxel.
- Quand une fenêtre appelée "Bio-formats Options d'importation" apparaît là où seules les options «Ouvrez toutes les séries" et "Autoscale" sont sélectionnés, ouvrez le fichier en cliquant sur "OK".
REMARQUE: Les fichiers sont affichés sous la forme d'un empilement de trois images, une pour chaque canal utilisé pour enregistrer une image au microscope (vert-, rouge-fluorescence et lumineux champ des images). - Séparer la pile en images de canaux individuels en sélectionnant "Image" - "Stacks" - "Pile à Images" dans le panneau de contrôle de ImageJ.
NOTE: Les images apparaissent et sont numérotés 1/3 (canal vert), 2/3 (canal rouge) et 3/3 (champ lumineux). Ici, l'image lumineuse-champ est exclu de l'analyse.
- Quand une fenêtre appelée "Bio-formats Options d'importation" apparaît là où seules les options «Ouvrez toutes les séries" et "Autoscale" sont sélectionnés, ouvrez le fichier en cliquant sur "OK".
- Pour analyser les images, transformer chacune en une image 8 bits en sélectionnant"Image" - "Type" - "8 bits".
- Pour déterminer la zone occupée en pixel 2, réinitialiser l'échelle des images à l' aide "Analyser" - "Set Scale". Quand une fenêtre apparaît avec différentes options d'échelle, remettre la balance en sélectionnant "Cliquez pour supprimer Scale". Cochez l'option "Global" pour supprimer l'échelle de toutes les images ouvertes.
- Pour supprimer l'arrière-plan, tracer une zone ovale (région d'intérêt, ROI) en dehors de la zone fluorescente en utilisant l'outil "Oval" dans le panneau de commande ImageJ.
- Pour veiller à ce que la taille de l'ovale de fond est le même pour toutes les images analysées, ajoutez-le au gestionnaire de ROI via le [t] caractère du clavier. Une fenêtre du gestionnaire de ROI arrive où l'arrière-plan ROI ovale peut être enregistré via "Plus" - options "Enregistrer".
- Si l'arrière-plan ROI ovale est visible sur l'image, mesurer l'intensité de la zone en choisissant "Analyser" - "Mesure".
NOTE: Les résultats de Afenêtre apparaît, entre autres, l'intensité de fluorescence moyenne est affichée dans la colonne intitulée "Mean". - Soustraire la valeur de l'intensité moyenne de fluorescence de fond de l'image en décochant l'arrière-plan ROI ovale, en cliquant sur "Process" - "Math" - "Soustraire" et l'insertion de la valeur mesurée.
- Appliquer un seuil à l'image via le "Image" - "Ajuster" - option "Seuil". Sélectionnez la méthode Otsu et noir et blanc (B & W). Cochez l'option "d'arrière-plan foncé" et d'employer le seuil en cliquant sur "Appliquer".
NOTE: L'image change à une image binaire où la zone au-dessus du seuil est représenté en blanc et que en dessous du seuil est en noir. - Sélectionnez tout au-dessus du seuil via le "Analyser" - option "Analyser les particules". Dans la fenêtre avec les paramètres, conservez les options par défaut et de garder les "Afficher les résultats" et "Summarize "options cochées. Cliquez sur" OK "pour afficher le résumé dans la fenêtre des résultats et l'affichage de la zone occupée dans la colonne intitulée" Secteur total ".
Résultats
Systèmes de laboratoire des populations bactériennes fournissent une approche attrayante pour explorer les questions écologiques ou d'évolution. Ici, trois modes de B. de colonisation de surface subtilis ont été utilisés pour examiner l'aspect de l' assortiment de la population, à savoir la séparation des génétiquement identiques, mais par fluorescence différentes souches marquées. Essaimage, qui est une personne à charge le mouvement de B. surface collective flagelle subtilis, résultats dans une population très mixte. Dans ces colonies grouillantes, les bactéries et les serre-rouge fluorescent zones colonisées ont été chevauchent (voir figure 2A). La colonisation rapide de la surface peut être suivie dans le temps (vidéo Figure 1). Au cours de l' essaimage de B. subtilis, une mince couche de cellules se développe à partir du centre d'inoculation après quelques heures d'incubation (voir la figure 2B).
4752 / 54752fig2.jpg "/>
Figure 2: Essaimage expansion de B. . subtilis La colonie grouillante contient des souches vert et au rouge fluorescent qui ont été mélangés 1: 1 avant l' inoculation. (A) Après 15 h, le vert- et rouge-fluorescence (GFP et RFP, respectivement) ont été détectés avec des filtres de fluorescence appropriés. (B) Images de couche mince de fourmillement B. subtilis sont présentés à des moments choisis extraits de la vidéo Figure 1. Barre d'échelle = 5 mm. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
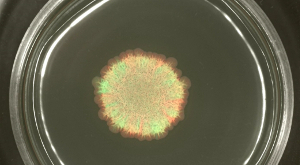
Cependant, quand B. souches subtilis, qui font défaut flagelles fonctionnels , mais sont capables de se propager à l'aide de produits exopolysaccharide, hydrophobine et surfactine, ont été repérés sur un milieu de gélose semi-solide, les souches marquées différemment ont été séparés dans certains defsecteurs de l' INED (voir la figure 3A). Le développement de la colonie de glissement peut être enregistré dans le temps (voir la figure 3B ou vidéo Figure 2).

Figure 3: Coulissante colonie de B. . La colonie subtilis contient des souches vert et au rouge fluorescent qui ont été mélangés 1: 1 avant l' inoculation. (A) Après 24 h, le vert- et rouge-fluorescence (GFP et RFP, respectivement) ont été détectés avec des filtres de fluorescence appropriés. (B) Images de la B. subtilis disque coulissantes sont présentés à des points de temps choisis extraits de la vidéo Figure 2. Barre d'échelle = 5 mm. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
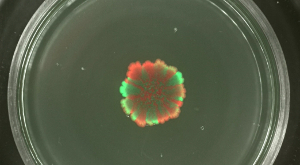
Alors que les niveaux de fourmillement et sli assortimentding colonies en expansion ne pouvaient pas être modifiées, la séparation spatiale des souches fluorescentes marquées différemment dans le biofilm de colonies peut être influencée par les densités cellulaires de départ. Quand un biofilm de colonies de B. subtilis a été lancé avec une densité cellulaire élevée des populations mixtes, les souches vert et au rouge fluorescent ont montré peu ou pas de variété spatiale (voir la figure 4). Au contraire, lorsque la densité cellulaire pour initier la biolfilm était faible, les secteurs au vert et au rouge fluorescence claire n'a pu être détecté par microscopie à fluorescence. Le niveau de l' assortiment est clairement dépendant du niveau de la population d' initiation de biofilm de dilution. Vidéo figures 3 et 4 présentent l'expansion des colonies pour la dilution la plus élevée et la plus basse des souches inoculées.

Figure 4: niveau Assortiment dans les biofilms de colonies de B. subtilis à. différentes densités cellulaires initiales Les biofilms de colonies de souches vert- et rouge-fluorescence sont présentés après 2 jours qui ont été inoculés avec différentes densités cellulaires initiales (de haut en bas: non dilué à 10 5 fois dilué initier des cultures, respectivement). Barre d' échelle = 5 mm. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Le rapport des tensions au vert et au rouge fluorescent peut être encore quantifié à l'aide du logiciel ImageJ qui permet la caractérisation quantitative de la structure de la population et de la compétitivité des souches utilisées pour les expériences.
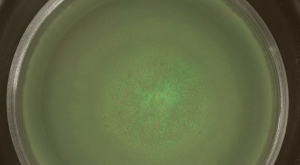
Vidéo Figure 1: Time lapse images de l' essaimage B. subtilis initié avec 1: 1. mélange de souches vert et au rouge fluorescent (clic pour télécharger.) La vidéo montre un cours de temps de 10 heures. Barre d' échelle = 7 mm.

Vidéo Figure 2: Temps images lapse de B. coulissant subtilis initié avec 1: 1. mélange de souches vert et au rouge fluorescent (clic pour télécharger.) La vidéo montre un cours de temps de 24 heures. Barre d'échelle = 5 mm.
Vidéo Figure 3: Temps images de déchéance de B. biofilms subtilis colonie initiées avec 1: 1 mélange de vert-. Et les souches fluorescentes de couleur rouge à des densités cellulaires élevées (clic droit pour télécharger.) La vidéo montre un cours de temps de 48 heures. Barre d'échelle = 5 mm.
Vidéo Figure 4: Temps images de déchéance de B. biofilm de colonies subtilis amorcées avec 1: 1 mélange de souches vert et au rouge fluorescent , à des densités cellulaires faibles. (Clic droit pour télécharger.) La vidéo montre un cours de temps de 48 heures. Barre d'échelle = 5 mm.
Discussion
La disponibilité d'une boîte à outils pour les bactéries fluorescentes facilite non seulement l'étude de l' expression génique hétérogène 30,31 et la localisation des protéines 32, mais aussi l'analyse de la répartition spatiale des souches au sein d' une population 8. Des marqueurs fluorescents avec suffisamment différentes longueurs d'onde d'excitation et d'émission permettent de localiser distinctement deux souches qui sont autrement impossibles à distinguer les uns des autres lorsqu'ils sont mélangés. Le protocole décrit peut être utilisé pour l' observation de la dynamique des populations dans les cultures mixtes, par exemple des expériences de compétition ou de synergie entre les souches ou espèces. La capacité de déterminer les abondances relatives des marquées par fluorescence souches dans une population mixte ne se limite pas à la surface grouillante ci-joint, de glissement, ou des colonies de biofilms, mais peut également être utilisé pour d'autres systèmes de biofilm multicellulaires, y compris submergée, le débit d'interface cellulaire ou air-support biofilms 27,33-35.
_content "> Alors que la technique présentée est un outil puissant pour détecter la distribution spatiale des souches et des expériences de compétition de conception, il permet également suivant l' expression du gène hétérogénéité dans les colonies en expansion. Les conditions de culture décrites ici sont valables pour B. subtilis et les paramètres exacts d'expansion sur agar médias peuvent nécessiter l' optimisation pour d' autres espèces ou souches 20. Placer les échantillons dans une chambre d'incubation alors que l' imagerie permet à l'expérimentateur de suivre la dynamique des populations dans le temps, bien que l' attention devrait être accordée au niveau d'humidité dans la chambre pendant l'incubation.Les techniques décrites ici requièrent également la modification génétique des souches bactériennes étudiées de telle sorte que les souches expriment des marqueurs fluorescents qui peuvent être distingués les uns des autres. En outre, en plus d'avoir une excitation distincte et des spectres d'émission, il est recommandé que les deux marqueurs fluorescents choisis ont quant mêmerendements um (ie rapport des photons absorbés qui sont émis) et sont exprimés dans un niveau comparable. En outre, les changements d'intensité relative dans le temps peuvent être mesurés et normalisée à un point de temps d'une expérience précoce. L'augmentation ou la diminution relative peuvent être ensuite comparés entre les différents fluorophores avec différents rendements quantiques. Pour que le système expérimental présenté, les différentes protéines et vert- fluorescentes de couleur rouge ont été testés préalablement 36,37 pour sélectionner les paires fluorescentes optimales qui peuvent être détectés chez B. subtilis. La durée d'exposition optimale doit être déterminée pour chaque protéine fluorescente et l'échantillon. Certaines densités cellulaires ou plusieurs couches de cellules pourraient être nécessaires pour détecter le signal de manière efficace au sein de la population. Certaines protéines fluorescentes peuvent avoir de faibles intensités dans les cellules bactériennes en raison de l'expression inefficace et / ou la traduction de la protéine et le rendement quantique donc faible. Ces marqueurs fluorescents inefficaces pourrait reduce la sensibilité du système et de prolonger le temps nécessaire pour détecter les souches bactériennes, entraînant éventuellement une cytotoxicité par la lumière d'excitation. Les intensités de fluorescence peuvent être modifiés en conséquence en modifiant le promoteur utilisé pour exprimer le gène rapporteur codant fluorescent. Un niveau d'expression qui est trop élevée pourrait donner lieu à une surproduction inutile de la protéine fluorescente conduisant à des coûts de mise en forme néfastes pour la bactérie. Lors de la réalisation des expériences de compétition, on doit considérer le coût de la production notamment de la protéine fluorescente dans les cellules. Des expériences de contrôle, où les marqueurs fluorescents sont échangés entre les souches concouru ou lorsque deux souches isogéniques ne différant que par leurs marqueurs fluorescents sont en compétition les uns contre les autres, sont toujours nécessaires pour déterminer un biais vers un marqueur. Les durées de vie des protéines fluorescentes dans les cellules pourraient également affecter l'intensité mesurée. En outre, l'auto-fluorescence de certaines espèces bactérienness pourrait nécessiter l'utilisation de différents marqueurs fluorescents autres que celles décrites ici.
Pour déterminer précisément la distribution spatiale et l'abondance des souches bactériennes distinctes, le signal de fond provenant de la première protéine fluorescente en utilisant le filtre de fluorescence pour le deuxième marqueur fluorescent et vice versa doit être testé individuellement sur des échantillons de monocultures (contenant des bactéries produisant un seul marqueur ). Ceci permet à la soustraction de chevauchement des intensités de signal fluorescent. Surtout, comme le stéréomicroscope enregistre le signal de fluorescence au-dessus de la colonie en expansion, le protocole présenté est commode pour déterminer l'arrangement spatial en deux dimensions. L'architecture de la population bactérienne en expansion peut entraîner des niveaux variables de fluorescence (c. -à -rides structures ressemblant peuvent contenir d' autres cellules présentant des intensités plus élevées de fluorescence locale). Par conséquent, l'analyse décrit des images determines la répartition spatiale, mais pas l'abondance des souches dans un certain emplacement. Protocoles précédents ont décrit la préparation des échantillons pour l' essaimage 20 ou l' imagerie de fluorescence de la dynamique de la population dans les colonies bactériennes 38, mais notre protocole combine ces techniques. D' autres techniques de microscopie qui permettent l'observation de trois résolution dimensions de la structure de la population (par exemple confocale à balayage laser de microscopie 39,40 ou microscopie illumination structurée 41) peuvent être appliqués pour les échantillons avec des complexités structurales accrues. Ces techniques supplémentaires prennent également en charge la détection unique cellule sur la base des souches 31 qui ne sont pas disponibles en utilisant stéréomicroscopes.
Déclarations de divulgation
Les auteurs déclarent qu'ils ont aucun intérêt financier concurrents.
Remerciements
Ce travail a été financé par la subvention KO4741 / 3-1 de la Deutsche Forschungsgemeinschaft (DFG). En outre, le laboratoire de Á.TK a été soutenue par une Marie Sklodowska Curie intégration de carrière subvention (PheHetBacBiofilm) et accorder KO4741 / 2-1 du DFG. TH, AD, RG-M,. Et EM ont été pris en charge par l'école internationale de recherche Max Planck, la fondation Alexander von Humboldt, Consejo Nacional de Ciencia y Tecnología-allemand d'échanges universitaires (DAAD CONACyT-), et des bourses CMHD, respectivement.
matériels
| Name | Company | Catalog Number | Comments |
| Lennox Broth (LB) | Carl Roth GmbH | X964 | |
| Agar-agar, Kobe I | Carl Roth GmbH | 5210 | |
| Petri dish (90 mm diameter) | any | NA | Use Petri dishes without ventillation cams |
| Petri dish (35 mm diameter) | any | NA | Use Petri dishes without ventillation cams |
| Difco Nutrient Broth | BD Europe | 234000 | |
| KCl | any | NA | |
| MgSO,4 7H2O | any | NA | |
| Ca(NO3)2 4H2O | any | NA | |
| MnCl24H2O | any | NA | |
| FeSO4 | any | NA | |
| D-Glucose | any | NA | |
| Fluorescence AxioZoom V16 time-lapse microscope | Carl Zeiss Microscopy GmbH | see bellow detailed description | |
| AxioZoom V16 Microscope body | Carl Zeiss Microscopy GmbH | 435080 9030 000 | |
| Phototube Z 100:0 for Axio Zoom V16 | Carl Zeiss Microscopy GmbH | 435180 9020 000 | without eyepieces |
| Fluar Illuminator Z mot Fluorescence intermediate tube for Axio Zoom.V16 | Carl Zeiss Microscopy GmbH | 435180 9060 000 | |
| Controller EMS 3 | Carl Zeiss Microscopy GmbH | 435610 9010 000 | |
| System Control Panel SYCOP 3 | Carl Zeiss Microscopy GmbH | 435611 9010 000 | |
| Reflector module Z | Carl Zeiss Microscopy GmbH | 435180 9160 000 | For Fluar Illuminator Z mot on Axio Zoom.V16 and SYCOP 3 |
| Filter set 38 HE eGFP shift free (E) | Carl Zeiss Microscopy GmbH | 489038 9901 000 | EX BP 470/40, BS FT 495, EM BP 525/50 |
| Filter set 63 HE mRFP shift free (E) | Carl Zeiss Microscopy GmbH | 489063 0000 000 | EX BP 572/25, BS FT 590, EM BP 629/62 |
| Mount S | Carl Zeiss Microscopy GmbH | 435402 0000 000 | |
| Objektive PlanApo Z 0,5x/0,125 FWD 114 mm | Carl Zeiss Microscopy GmbH | 435280 9050 000 | 164 mm parfocal length; M62x0.75 thread at front |
| Coarse/fine drive with profile column | Carl Zeiss Microscopy GmbH | 435400 0000 000 | 490 mm, 10 kg load capacity, compatible with stand bases 300/450 |
| Stand base 450 | Carl Zeiss Microscopy GmbH | 435430 9902 000 | |
| Cold-light source Zeiss CL 9000 LED CAN | Carl Zeiss Microscopy GmbH | 435700 9000 000 | |
| CAN-bus cable | Carl Zeiss Microscopy GmbH | 457411 9011 000 | 2.5 m length |
| Slit-ring illuminator | Carl Zeiss Microscopy GmbH | 417075 9010 000 | d = 66 mm |
| Flexible light guide 1500 | Carl Zeiss Microscopy GmbH | 417063 9901 000 | 8/1,000 mm |
| Illumination Adapter for light guide | Carl Zeiss Microscopy GmbH | 000000 1370 927 | |
| Lightguide HXP with liquid fill | Carl Zeiss Microscopy GmbH | 000000 0482 760 | ø3 mm x 2,000 mm |
| Camera Adapter 60N-C | Carl Zeiss Microscopy GmbH | 426113 0000 000 | 2/3" 0.63X |
| High Resolution Microscopy Camera AxioCam MRm Rev. 3 FireWire | Carl Zeiss Microscopy GmbH | 426509 9901 000 | |
| AxioCam FireWire Trigger Cable Set | Carl Zeiss Microscopy GmbH | 426506 0002 000 | for direct shutter synchronization |
| ZEN pro 2012 | Carl Zeiss Microscopy GmbH | 410135 1002 120 | Blue edition, requires min. Windows 7 64-bit |
| ZEN Module Time Lapse | Carl Zeiss Microscopy GmbH | 410136 1031 110 | |
| Standard Heating Stage Top Incubator | Tokai Hit | INUL-MS1-F1 | |
| Zeiss Stereo Microscope Base Adapter | Tokai Hit | MS-V12 | |
| Softwares | |||
| ImageJ | National Institute of Health, Bethesda, MD, USA | v 1.49m | |
| BioVoxxel plugin | BioVoxxel | http://www.biovoxxel.de/development/ |
Références
- Nadell, C. D., Xavier, J. B., Foster, K. R. The sociobiology of biofilms. FEMS Microbiol Rev. 33 (1), 206-224 (2009).
- West, S. A., Griffin, A. S., Gardner, A., Diggle, S. P. Social evolution theory for microorganisms. Nat Rev Microbiol. 4 (8), 597-607 (2006).
- Kovács, &. #. 1. 9. 3. ;. T. Impact of spatial distribution on the development of mutualism in microbes. Front Microbiol. 5, 649 (2014).
- Martin, M., Hölscher, T., Dragoš, A., Cooper, V. S., Kovács, &. #. 1. 9. 3. ;. T. Laboratory evolution of microbial interactions in bacterial biofilms. J Bacteriol. , (2016).
- Drescher, K., Nadell, C. D., Stone, H. A., Wingreen, N. S., Bassler, B. L. Solutions to the public goods dilemma in bacterial biofilms. Curr Biol. 24 (1), 50-55 (2014).
- Momeni, B., Waite, A. J., Shou, W. Spatial self-organization favors heterotypic cooperation over cheating. Elife. 2, e00960 (2013).
- van Dyken, J. D., Müller, M. J., Mack, K. M., Desai, M. M. Spatial population expansion promotes the evolution of cooperation in an experimental Prisoner's Dilemma. Curr Biol. 23 (10), 919-923 (2013).
- van Gestel, J., Weissing, F. J., Kuipers, O. P., Kovács, &. #. 1. 9. 3. ;. T. Density of founder cells affects spatial pattern formation and cooperation in Bacillus subtilis biofilms. ISME J. 8 (10), 2069-2079 (2014).
- Momeni, B., Brileya, K. A., Fields, M. W., Shou, W. Strong inter-population cooperation leads to partner intermixing in microbial communities. Elife. 2, e00230 (2013).
- Müller, M. J., Neugeboren, B. I., Nelson, D. R., Murray, A. W. Genetic drift opposes mutualism during spatial population expansion. Proc Natl Acad Sci U S A. 111 (3), 1037-1042 (2014).
- Pande, S., et al. Privatization of cooperative benefits stabilizes mutualistic cross-feeding interactions in spatially structured environments. ISME J. 10, 1413-1423 (2016).
- Hallatschek, O., Hersen, P., Ramanathan, S., Nelson, D. R. Genetic drift at expanding frontiers promotes gene segregation. Proc Natl Acad Sci U S A. 104 (50), 19926-19930 (2007).
- Flemming, H. C., Wingender, J. The biofilm matrix. Nat Rev Microbiol. 8 (9), 623-633 (2010).
- Vlamakis, H., Chai, Y., Beauregard, P., Losick, R., Kolter, R. Sticking together: building a biofilm the Bacillus subtilis way. Nat Rev Microbiol. 11 (3), 157-168 (2013).
- Lopez, D., Kolter, R. Extracellular signals that define distinct and coexisting cell fates in Bacillus subtilis. FEMS Microbiol Rev. 34 (2), 134-149 (2010).
- Lopez, D., Vlamakis, H., Kolter, R. Generation of multiple cell types in Bacillus subtilis. FEMS Microbiol Rev. 33 (1), 152-163 (2009).
- Mhatre, E., Monterrosa, R. G., Kovács, &. #. 1. 9. 3. ;. T. From environmental signals to regulators: Modulation of biofilm development in Gram-positive bacteria. J Basic Microbiol. , (2014).
- Zhang, W., et al. Nutrient depletion in Bacillus subtilis biofilms triggers matrix production. New J Physics. 16, 015028 (2014).
- Kearns, D. B. A field guide to bacterial swarming motility. Nat Rev Microbiol. 8 (9), 634-644 (2010).
- Morales-Soto, N., et al. Preparation, imaging, and quantification of bacterial surface motility assays. J Vis Exp. (98), (2015).
- Angelini, T. E., Roper, M., Kolter, R., Weitz, D. A., Brenner, M. P. Bacillus subtilis spreads by surfing on waves of surfactant. Proc Natl Acad Sci U S A. 106 (43), 18109-18113 (2009).
- Grau, R. R., et al. A Duo of Potassium-Responsive Histidine Kinases Govern the Multicellular Destiny of Bacillus subtilis. MBio. 6 (4), e00581 (2015).
- Park, S. Y., Pontes, M. H., Groisman, E. A. Flagella-independent surface motility in Salmonella enterica serovar Typhimurium. Proc Natl Acad Sci U S A. 112 (6), 1850-1855 (2015).
- van Gestel, J., Vlamakis, H., Kolter, R. From cell differentiation to cell collectives: Bacillus subtilis uses division of labor to migrate. PLoS Biol. 13 (4), e1002141 (2015).
- Abee, T., Kovács, &. #. 1. 9. 3. ;. T., Kuipers, O. P., van der Veen, S. Biofilm formation and dispersal in Gram-positive bacteria. Curr Opin Biotechnol. 22 (2), 172-179 (2011).
- Kovács, &. #. 1. 9. 3. ;. T. Bacterial differentiation via gradual activation of global regulators. Curr Genet. 62 (1), 125-128 (2016).
- Hölscher, T., et al. Motility, chemotaxis and aerotaxis contribute to competitiveness during bacterial pellicle biofilm development. J Mol Biol. 427 (23), 3695-3708 (2015).
- Seminara, A., et al. Osmotic spreading of Bacillus subtilis biofilms driven by an extracellular matrix. Proc Natl Acad Sci U S A. 109 (4), 1116-1121 (2012).
- Patrick, J. E., Kearns, D. B. Laboratory strains of Bacillus subtilis do not exhibit swarming motility. J Bacteriol. 191 (22), 7129-7133 (2009).
- de Jong, I. G., Beilharz, K., Kuipers, O. P., Veening, J. W. Live Cell Imaging of Bacillus subtilis and Streptococcus pneumoniae using Automated Time-lapse Microscopy. J Vis Exp. (53), e3145 (2011).
- Garcia-Betancur, J. C., Yepes, A., Schneider, J., Lopez, D. Single-cell analysis of Bacillus subtilis biofilms using fluorescence microscopy and flow cytometry. J Vis Exp. (60), (2012).
- Turnbull, L., et al. Super-resolution imaging of the cytokinetic Z ring in live bacteria using fast 3D-structured illumination microscopy (f3D-SIM). J Vis Exp. (91), e51469 (2014).
- Barken, K. B., et al. Roles of type IV pili, flagellum-mediated motility and extracellular DNA in the formation of mature multicellular structures in Pseudomonas aeruginosa biofilms. Environ Microbiol. 10 (9), 2331-2343 (2008).
- Oliveira, N. M., et al. Biofilm formation as a response to ecological competition. PLoS Biol. 13 (7), e1002191 (2015).
- Wang, X., et al. Probing phenotypic growth in expanding Bacillus subtilis biofilms. Appl Microbiol Biotechnol. 100, 4607-4615 (2016).
- Detert Oude Weme, R. G., et al. Single cell FRET analysis for the identification of optimal FRET-pairs in Bacillus subtilis using a prototype MEM-FLIM system. PLoS One. 10 (4), e0123239 (2015).
- Overkamp, W., et al. Benchmarking various green fluorescent protein variants in Bacillus subtilis, Streptococcus pneumoniae, and Lactococcus lactis for live cell imaging. Appl Environ Microbiol. 79 (20), 6481-6490 (2013).
- Stannek, L., Egelkamp, R., Gunka, K., Commichau, F. M. Monitoring intraspecies competition in a bacterial cell population by cocultivation of fluorescently labelled strains. J Vis Exp. (83), e51196 (2014).
- Bridier, A., Briandet, R. Contribution of confocal laser scanning microscopy in deciphering biofilm tridimensional structure and reactivity. Methods Mol Biol. 1147, 255-266 (2014).
- Khajotia, S. S., Smart, K. H., Pilula, M., Thompson, D. M. Concurrent quantification of cellular and extracellular components of biofilms. J Vis Exp. (82), e50639 (2013).
- Neu, T. R., Lawrence, J. R. Innovative techniques, sensors, and approaches for imaging biofilms at different scales. Trends Microbiol. 23 (4), 233-242 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon