Method Article
Monitoramento Espacial Segregação na superfície Colonizar microbianas Populações
Neste Artigo
Resumo
O papel da variedade (segregação espacial) em cenários evolutivas podem ser examinadas utilizando sistemas microbianos simples no laboratório que permitem o ajuste da distribuição espacial controlada. Ao modificar a densidade de células fundador, vários níveis de sortimento pode ser visualizada usando cepas de bactérias fluorescente etiquetado nos biofilmes de colónias de Bacillus subtilis.
Resumo
Microbes provide an intriguing system to study social interaction among individuals within a population. The short generation times and relatively simple genetic modification procedures of microbes facilitate the development of the sociomicrobiology field. To assess the fitness of certain microbial species, selected strains or their genetically modified derivatives within one population, can be fluorescently labelled and tracked using microscopy adapted with appropriate fluorescence filters. Expanding colonies of diverse microbial species on agar media can be used to monitor the spatial distribution of cells producing distinctive fluorescent proteins.
Here, we present a detailed protocol for the use of green- and red-fluorescent protein producing bacterial strains to follow spatial arrangement during surface colonization, including flagellum-driven community movement (swarming), exopolysaccharide- and hydrophobin-dependent growth mediated spreading (sliding), and complex colony biofilm formation. Non-domesticated isolates of the Gram-positive bacterium, Bacillus subtilis can be utilized to scrutinize certain surface spreading traits and their effect on two-dimensional distribution on the agar-solidified medium. By altering the number of cells used to initiate colony biofilms, the assortment levels can be varied on a continuous scale. Time-lapse fluorescent microscopy can be used to witness the interaction between different phenotypes and genotypes at a certain assortment level and to determine the relative success of either.
Introdução
Nas últimas décadas, os micróbios foram reconhecidas como comunidades sociais associados com vários ecossistemas da Terra 1,2. Em contraste com as culturas planctônicas utilizados na prática laboratorial geral, os micróbios no ambiente mostram uma grande variedade de estruturas espaciais da comunidade, dependendo da configuração ecológico. Sistemas microbianos simples podem ser utilizados para compreender a consequência de estruturas espaciais sobre a evolução das interacções sociais 3,4. Publicações nos últimos 2-3 anos usando ambos os sistemas modelo eucarióticos e procarióticos destacou o impacto das estruturas espaciais sobre a estabilidade da cooperação no seio das populações microbianas 5-8. Além disso, obrigam as interacções entre os micróbios, por exemplo, alimentação cruzada metabólica, podem também alterar a distribuição espacial dos parceiros interactuantes 9-11. A influência da estrutura espacial nesses estudos é principalmente examinada utilizando células sésseis anexado superfície que habitam o modobiofilmes -chamado ou em colónias em crescimento na superfície de um meio de agar. A deriva genética resultando em alta variedade espacial pode ser observado em colônias microbianas onde o esgotamento de nutrientes na borda de um resultado de expansão de divisão celular mediadas em série de gargalos genéticos que faz com alta probabilidade de fixação local para certos linages clonais 12. A deriva genética pode ser, portanto, utilizados para examinar o papel da segregação espacial em colônias microbianas.
No ambiente, os biofilmes são comunidades multiespecíficas rodeados por matriz polimérica auto-produzido 13. Estrutura de biofilme, função e estabilidade dependem de uma complexa rede de interações sociais onde os sinais de câmbio bactérias, componentes e recursos da matriz, ou competir por espaço e nutrientes por meio de toxinas e antibióticos. Bacillus subtilis é um solo de habitação e bactéria colonizadora raiz que desenvolve altamente organizada comunidades de biofilme 14. Em analogia com o social,insectos, B. células subtilis empregar uma divisão da estratégia de trabalho, o desenvolvimento de subpopulações de produtores da matriz extracelular e canibais, células móveis, esporos dormentes e outros tipos de células 15,16. O processo de diferenciação é dinâmico e pode ser alterada por condições ambientais 17,18.
Estratégias de colonização por bactérias da superfície pode ser facilmente manipulados sob condições de laboratório, modificando a concentração de agar no meio de crescimento. Em níveis de agar baixas (0,2-0,3%), as bactérias que abrigam flagelos ativa são capazes de nadar, enquanto agar semi-sólido (0,7-1% de agar) facilita comunidade dirigida flagelo se espalhando, chamado de enxame 19-21. Na ausência de flagelo, certas estirpes bacterianas são capazes de se mover ao longo do meio semi-sólido por via de deslizamento, isto é, expansão da população dependente crescimento facilitada pela matriz exopolissacarídeo e outros compostos de hidrofobina secretadas 22-24. Por fim, as bactérias que são capable da forma de desenvolvimento de biofilme colônias arquitetonicamente complexos em meio agar rígido (1,2-2%) 14,17,25. Enquanto essas características são examinadas em laboratório, ajustando com precisão as condições, em habitats naturais dessas estratégias de divulgação de superfície pode trânsito gradualmente de um para outro, dependendo das condições ambientais 26. Enquanto motilidade única base da célula é crítico durante o início do desenvolvimento do biofilme na interface ar-líquido em bactérias tanto Gram-positivas e -negativas 27, biofilmes complexos de colónias de B. subtilis não são afectadas pela eliminação de motilidade flagelar 28. No entanto, a organização espacial durante o desenvolvimento do B. subtilis biofilmes de colónias depende da densidade do inoculo bacteriano utilizado para iniciar o biofilme 8.
Aqui, usamos B. subtilis para mostrar que a segregação espacial durante a colonização da superfície depende do mecanismo de motili nível da populaçãoTy (ou seja enxame ou deslizante), e o desenvolvimento de colónias de biofilme depende da densidade da célula fundadora. Apresenta-se uma ferramenta de microscopia fluorescente que pode ser aplicado para monitorizar continuamente o crescimento microbiano biofilme, a colonização da superfície e variedade na escala macro. Além disso, um método de quantificação é apresentada para determinar a abundância relativa estirpe na população.
Protocolo
1. Preparação de Meios de Cultura, semi sólidos placas de ágar e biofilme, Pré-culturas
- Preparação meio para pulular e deslizando
- Dissolve-se 2 g de Lenox Broth (LB) e 0,7 g de agar-agar em 100 ml de água com iões permutados e autoclave durante 20 min a 120 ° C. Use pequenos volumes (50-200 ml) para melhorar a reprodutibilidade entre os experimentos.
- Imediatamente após a esterilização, fechar a tampa do recipiente de forma a reduzir a evaporação e colocar num incubador a 55 ° C durante pelo menos 2 h.
- Após a temperatura do meio foi temperada para 55 ° C, verter 20 ml de agar de meio LB em um diâmetro de 90 mm numa caixa de Petri de poliestireno sob um capuz estéril laboratório. Para experiências de lapso de tempo, despeje 5 ml agar meio LB por 35 mm de diâmetro de poliestireno placa de Petri.
- Fechar a placa de Petri imediatamente após o vazamento, a pilha não há mais do que 4 placas em cima uns dos outros e que o meio de agar solidificar durante pelo menos 1 h.
- 2xSG Médio Preparação Colony biofilmes
- Dissolve-se 1,6 g de Caldo Nutriente, 0,2 g de KCl, 0,05 g de MgSO 4 7H 2 O, e 1,5 g de agar-agar em 100 ml de água com iões permutados e autoclave durante 20 min a 120 ° C. Use pequenos volumes (50-200 ml) para melhorar a reprodutibilidade entre os experimentos.
- Imediatamente após a esterilização, fechar a tampa do recipiente de forma a reduzir a evaporação e colocar a garrafa em uma incubadora a 55 ° C durante pelo menos 2 h.
- Após a temperatura do meio tem auto-ajustado para 55 ° C, adiciona-se 0,1 ml esterilizado por filtração a 1 M de Ca (NO3) 2 solução, 0,1 ml esterilizado por filtração mM de MnCl2 solução de 100, 0,1 ml esterilizado por filtração um FeSO 4 mM de solução, e 0,5 ml solução estéril de glicose a 20%.
- Em uma capa estéril laboratório, despeje 20 ml de meio agar 2x SG por 90 mm de diâmetro de poliestireno placa de Petri. Para experiências de lapso de tempo, despeje 5 ml agar meio LB por 35 mm de diâmetro de poliestireno placa de Petri.
- ClosÊ O prato Petri imediatamente após o vazamento, a placa de empilhar umas sobre as outras, mas não mais do que 4 placas, e deixar o meio de ágar, para solidificar durante pelo menos 1 h.
- Preparação de culturas de arranque
NOTA: O B. subtilis 168, NCIB 3610 estirpes derivadas utilizadas nos métodos descritos abaixo constitutivamente produzem proteínas com efeito de estufa ou vermelho-de fluorescência foram descritas antes e 8,27. As estirpes rotineiramente são armazenados no congelador -80 ° C.- Inocular culturas de arranque a partir de estoques de -80 ° C em 3 ml de meio LB e incubar durante a noite (16-18 horas) a 37 ° C com agitação horizontal (225 rpm). Não incubar a cultura mais de 18 horas como selvagens isolados de B. subtilis são principalmente propenso a agregar e formar um biofilme no tubo de ensaio.
2. Co-inoculação de fluorescência etiquetado estirpes bacterianas para a superfície de espalhamento
- Secagem de placas de ágar semi-sólidas para pululando eDeslizante de B. subtilis.
- placas de agar secas para enxame e deslizando durante 20 min antes da inoculação. Secar as placas a descoberto numa câmara de fluxo laminar (ver Figura 1).
NOTA: swarming bacteriana e deslizando depende da secura do meio agar semi-sólido. secagem insuficiente permite a acumulação de água no meio de agar resultando na natação mediada pelo flagelo. Prolongou o tempo de secagem resulta em falta de enxameação.
- placas de agar secas para enxame e deslizando durante 20 min antes da inoculação. Secar as placas a descoberto numa câmara de fluxo laminar (ver Figura 1).
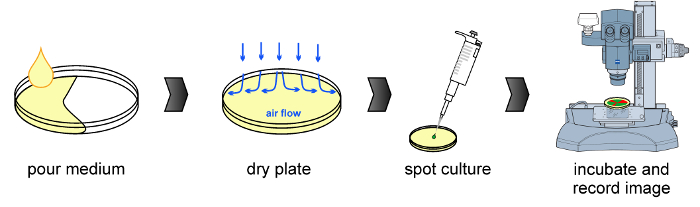
Figura 1:.. Fluxo de trabalho Experimental O procedimento comum é representado na figura, incluindo a preparação do meio de cultura, secando a detecção microscopia de placa, inoculação e fluorescência (da esquerda para a direita) Por favor clique aqui para ver uma versão maior desta figura.
- Co-inoculaçãode culturas bacterianas para pulular e deslizando
- Determinar as densidades ópticas das culturas de arranque durante a noite a 600 nm e misture densidade cepas produtoras de proteínas com efeito de estufa e vermelho fluorescentes normalizadas de B. subtilis NCIB 3610 ou um seu derivado Δ hag em um tubo de 1,5 ml de reacção. Por exemplo, misturar 100 ml de tensão 1 com (100 * [OD 600 da cultura durante a noite de tensão 1] / [OD 600 da cultura durante a noite de tensão 2]) ul da estirpe 2. Ligeiramente vortex (3 seg em velocidade máxima) para distribuição homogénea.
NOTA: B. subtilis NCIB 3610 cepas são inoculados observar swarming, enquanto seus derivados Δ hag são utilizados para correr. - Spot 2 ul de cultura mista em meio de uma placa pré-secou-se (ver Figura 1) e secar ainda mais a placa durante 10 min após a inoculação.
- Incubar as placas a 37 ° C em posição vertical para permitir que o excesso de humidade se condense sobre a tampa e não sobre a superfície do ágar.
NOTA: O tempo de incubação para B. subtilis swarming é tipicamente entre 8-16 horas. Geralmente, a extremidade do enxame atinge o lado do prato de Petri 90 milímetros em 8 h. Deslizante é um processo mais lento e requer, pelo menos, 16-42 horas de incubação. Após 36 h, a frente deslizante alcance o lado da placa de Petri de 90 milímetros. - Para experiências de lapso de tempo, coloque os 35 mm de diâmetro placas de Petri numa câmara de incubação de estágio pré-aquecido fixada em 37 ° C. Certifique-se de que a tampa da placa de Petri removido permanece durante toda a duração da experiência. Coloque a tampa da incubadora fase a 40 ° C para contornar a formação de humidade na parte superior da incubadora.
- Determinar as densidades ópticas das culturas de arranque durante a noite a 600 nm e misture densidade cepas produtoras de proteínas com efeito de estufa e vermelho fluorescentes normalizadas de B. subtilis NCIB 3610 ou um seu derivado Δ hag em um tubo de 1,5 ml de reacção. Por exemplo, misturar 100 ml de tensão 1 com (100 * [OD 600 da cultura durante a noite de tensão 1] / [OD 600 da cultura durante a noite de tensão 2]) ul da estirpe 2. Ligeiramente vortex (3 seg em velocidade máxima) para distribuição homogénea.
3. Co-inoculação de fluorescência Labelled bacterianas As estirpes com diferentes densidades celulares iniciais
- A secagem de placas de agar para Formação de Colónias de biofilme de B. subtilis.
- Seque as placas para o desenvolvimento colónia biofilme sem cobertura em um fluxo laminarcapuz durante 15 min antes da inoculação.
NOTA: Faltam secagem resulta em aumento da umidade e natação ou enxame pode ser possível 29. A secagem demasiado longos resulta em pequenas colónias de biofilme.
- Seque as placas para o desenvolvimento colónia biofilme sem cobertura em um fluxo laminarcapuz durante 15 min antes da inoculação.
- Preparação de 10 vezes fermentos diluída Colony biofilmes
- Misture 100 ml de proteína de estufa e vermelho fluorescente que produzem culturas de arranque durante a noite de B. subtilis 168 num tubo de reacção de 1,5 ml e levemente vórtice para distribuição homogénea. Preparam-se séries de diluição de 10 vezes em meio LB.
- Spot 2 ul de não-diluído ou 10 1, 10 2, 10 3, 10 4 culturas mistas diluídas na placa contendo meio de indução de biofilme.
NOTA: 6 a 9 colónias biofilme pode ser iniciado em uma única placa de Petri de 90 milímetros, tendo em conta que as colónias são separadas a uma distância igual uma da outra. - Incubar as placas a 30 ° C em posição vertical para permitir que o excesso de humidade para condensar-se na tampa enão sobre a superfície do ágar.
NOTA: O tempo de incubação para B. subtilis biofilme é entre 1 a 3 dias. Geralmente, o biofilme colónia de B. subtilis atinge o seu tamanho médio e estrutura complexa em 2 dias. - Para as experiências de lapso de tempo, colocar um único inoculo no meio de uma placa de Petri de diâmetro de 35 milímetros e colocar o prato numa câmara de incubação fase pré-aquecido fixada em 30 ° C. Certifique-se de que a parte superior da placa de Petri removido permanece durante toda a duração da experiência. Coloque a tampa da incubadora fase a 35 ° C para contornar a formação de humidade na parte superior da incubadora.
4. Detecção de microscopia de fluorescência de Labelled Estirpes
- Descrição do Equipamento for Imaging.
- Para detectar a colonização de superfície e sinal de fluorescência, use um microscópio com zoom motorizado de fluorescência estéreo (ver lista detalhada na Tabela Materiais) equipado com um 0,5X PlanApo Objetivo, dois LEDfontes de luz fria (um para detecção de fluorescência e um para a luz visível), conjuntos de filtros para GFP (excitação a 470/40 nm e emissão a 525/50 nm) e MRFP (excitação a 572/25 nm e emissão a 629 / 62 nm), e uma câmara monocromática alta resolução.
- Execute aquisição e processamento de imagem com o software apropriado disponível para o zoom microscópio estéreo incluindo multicanal e módulos de lapso de tempo. Para o experimento de lapso de tempo, use uma incubadora de estágio de aquecimento padrão montado no microscópio zoom estéreo com um adaptador.
- Imagiologia de pululando e deslizante Expansão
- Usar a menor ampliação para capturar a maior área possível da placa de 90 mm. Definir a origem de inoculação (no meio da placa de Petri de 90 milímetros) para o canto do campo visível para o controlo bacteriano expansão radial e de fluorescência.
- Ajustar o tempo de exposição óptimo, dependendo da intensidade do sinal de fluorescência.
NOTA: Para expre constitutivamentegenes de fluorescência em sseD B. subtilis, verde- e vermelho-de fluorescência com o tempo de exposição de 1,5 e 3 seg pode ser utilizado, respectivamente. Além disso, 10 o tempo de exposição mseg é apropriado para a luz visível.
- Utilizar a ampliação que permite a detecção de toda a colónia de biofilme e ajustar a colónia no meio do campo de vista.
NOTA: Como para enxame e expansões de deslizamento, os tempos de exposição óptima para detectar os sinais de fluorescência nas colónias biofilme depende do nível dos genes codificadores de proteínas fluorescentes expressão. Para os resultados representativos abaixo, verde- e vermelho-fluorescência foi detectada usando intervalos de 1 e 3 de exposição seg, respectivamente. - Para geração de imagens de lapso de tempo, obter imagens em determinados intervalos usando tempos de exposição constantes.
- Guardar as imagens gravadas de fluorescência estereomicroscópio em um formato de arquivo que seja reconhecido pelo software ImageJ para análise quantitativa dos dados.
5. Datuma Análise
- Para analisar a área ocupada por cada cepa fluorescente diferente rotulados, abra o arquivo de interesse em software ImageJ expandida com um plugin BioVoxxel.
- Quando uma janela chamada "bio-formatos opções de importação" aparece onde apenas as opções "Abra todas as séries" e "Autoscale" são selecionados, abra o arquivo, clicando em "OK".
NOTA: Os arquivos são exibidos como uma pilha de três imagens, uma para cada canal utilizado para gravar uma imagem no microscópio (estufa, vermelho-fluorescência e de campo brilhante imagens). - Separe a pilha em imagens de canais individuais, selecionando "Imagem" - "Pilhas" - "pilha para imagens" no painel de controle ImageJ.
NOTA: As imagens aparecem e são contados como 1/3 (canal verde), 2/3 (canal vermelho) e 3/3 (de campo brilhante). Aqui, a imagem de campo brilhante é excluído da análise.
- Quando uma janela chamada "bio-formatos opções de importação" aparece onde apenas as opções "Abra todas as séries" e "Autoscale" são selecionados, abra o arquivo, clicando em "OK".
- Para analisar as imagens, para transformar cada um em uma imagem de 8 bits, selecionando"Imagem" - "Tipo" - "8-bit".
- Para determinar a área ocupada em pixels 2, repor a escala das imagens usando "Analisar" - "Set Scale". Quando aparece uma janela com opções de escala diferentes, repor a escala, selecionando "clique para remover Scale". Marque a opção "Global" para remover a escala para todas as imagens abertas.
- Para remover o fundo, desenhar uma área oval (região de interesse, ROI) fora da área fluorescente utilizando a ferramenta "Oval" no painel de controle do ImageJ.
- Para assegurar que o tamanho da oval do fundo é a mesma para todas as imagens analisadas, adicioná-lo ao gerente ROI através do [t] carácter do teclado. A janela do Gerenciador de ROI surge onde o ROI oval fundo podem ser guardados através de "mais" - opções de "salvar".
- Se o fundo ROI oval é visível na imagem, medir a intensidade da área, escolhendo "Analisar" - "Measure".
Nota: Os resultados Ajanela onde entre outros a intensidade de fluorescência média é exibido na coluna denominada "Média". - Subtrair o valor da intensidade média de fluorescência de fundo da imagem desselecionando o fundo ROI oval, clicando "Processo" - "matemática" - "subtrair" e inserir o valor medido.
- Aplicar um limite para a imagem através da "Imagem" - "Ajuste" - opção "Threshold". Selecione o método de Otsu e preto e branco (B & W). Marque a opção "background Dark" e empregar o limite, clicando em "Aplicar".
NOTA: As alterações de imagem para uma imagem binária onde a área acima do limiar é mostrado em branco e que abaixo do limiar é mostrado em preto. - Selecionar tudo acima do limiar através do "Analisar" - opção "Analisar Particles". Na janela com as configurações, mantenha as opções padrão e manter os "Mostrar resultados" e "Summarize "opções marcadas. Clique em" OK "para exibir o resumo na janela de resultados ea exibição da área ocupada, na coluna denominada" Área Total ".
Resultados
Laboratório de Sistemas de populações bacterianas fornecer uma abordagem atraente para explorar questões ecológicas ou evolutivas. Aqui, três modos de colonização superfície de B. subtilis foram usadas para analisar o aparecimento de diversas formas população, isto é, a segregação de geneticamente idênticas, mas diferentes estirpes fluorescentemente marcado. Swarming, que é um movimento de superfície coletiva dependente flagelo do B. subtilis, resulta em uma população altamente misto. Nessas colónias que pululam, as bactérias com efeito de estufa e vermelho fluorescentes áreas colonizadas foram sobrepostos (ver Figura 2A). A colonização superfície rápida pode ser seguida em tempo (Vídeo Figura 1). Durante enxame de B subtilis, uma camada fina de células expande do centro da inoculação, após algumas horas de incubação (ver Figura 2B).
4752 / 54752fig2.jpg "/>
Figura 2: Enxameando expansão de B. . subtilis A colônia swarming contém estirpes de efeito estufa e vermelho-fluorescente que foram misturadas 1: 1 antes da inoculação. (A) Após 15 h, a estufa e vermelho-de fluorescência (GFP e RFP, respectivamente) foram detectados com filtros de fluorescência adequados. (B) Imagens de fina camada de swarming B. subtilis são mostrados em pontos de tempo selecionado extraídos de Vídeo Figura 1. Barra de escala = 5 mm. Por favor clique aqui para ver uma versão maior desta figura.
No entanto, quando B. subtilis, estirpes que faltam flagelos funcional, mas que são capazes de se espalhar com a ajuda de exopolissacárido produzido, hidrofobina e surfactina, foram vistos em meio de agar semi-sólido, as estirpes diferentemente marcadas foram separadas em certa defsectores INED (ver Figura 3A). O desenvolvimento da colónia de deslizamento pode ser gravada no tempo (ver Figura 3B ou vídeo Figura 2).

Figura 3: Deslizamento colônia de B. . subtilis A colônia contém estirpes de efeito estufa e vermelho-fluorescente que foram misturadas 1: 1 antes da inoculação. (A) Após 24 h, a estufa e vermelho-de fluorescência (GFP e RFP, respectivamente) foram detectados com filtros de fluorescência adequados. (B) Imagens do B. subtilis disco deslizantes são mostrados em pontos de tempo selecionados extraídos de Vídeo Figura 2. Barra de escala = 5 mm. Por favor clique aqui para ver uma versão maior desta figura.
Embora os níveis de sortimento de swarming e SLIDing colónias em expansão não pode ser modificado, a segregação espacial de estirpes fluorescentes marcadas de forma diferente no biofilme colónia pode ser influenciada pelas densidades celulares de partida. Quando uma colónia de biofilme B. subtilis foi iniciado com elevada densidade celular das populações mistas, as estirpes de estufa e vermelho fluorescentes mostraram uma pequena ou nenhuma variedade espacial (ver Figura 4). Pelo contrário, quando a densidade de células para iniciar a biolfilm foi baixa, sectores estufa e vermelho-clara fluorescência podia ser detectado por microscopia de fluorescência. O nível variedade foi claramente dependente do nível de diluição da população iniciar biofilme. Vídeo Figura 3 e 4 apresentam a expansão colónia para a diluição mais baixa e mais alta das estirpes inoculadas.

Figura 4: Nível de Variedade em biofilmes de colônias de B. subtilis a. várias densidades celulares iniciais Os biofilmes de colónias de estirpes de estufa e vermelho-de fluorescência são apresentados após 2 dias que foram inoculados com diferentes densidades celulares iniciais (de cima para baixo: não diluído a 10 5 vezes diluído iniciar culturas, respectivamente). Barra de escala = 5 mm. Por favor clique aqui para ver uma versão maior desta figura.
A proporção de estirpes de estufa e vermelho fluorescentes pode ser ainda quantificado utilizando o software ImageJ que permite a caracterização quantitativa da estrutura da população e a competitividade das estirpes utilizadas para as experiências.
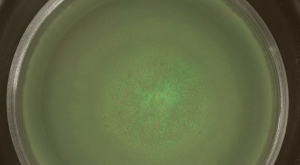
Vídeo Figura 1: Time lapse images de swarming B. subtilis iniciado com 1: 1. mistura de estirpes de efeito estufa e vermelho fluorescente (Clique direito a download.) O vídeo mostra um curso de tempo de 10 horas. Barra de escala = 7 mm.

Vídeo Figura 2: imagens lapso de tempo de correr B. subtilis iniciado com 1: 1. mistura de estirpes de efeito estufa e vermelho fluorescente (Clique direito a download.) O vídeo mostra um curso de tempo de 24 horas. Barra de escala = 5 mm.
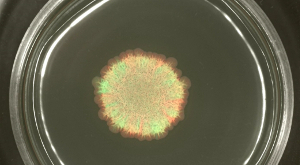
Vídeo Figura 3: imagens lapso de tempo B. biofilmes subtilis colônia iniciado com 1: 1 mistura de verde-. E estirpes vermelho-fluorescente em altas densidades celulares (Clique direito a download.) O vídeo mostra um curso de tempo de 48 horas. Barra de escala = 5 mm.
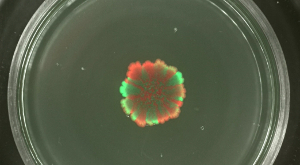
Vídeo Figura 4: imagens lapso de tempo B. biofilmes subtilis colónias iniciada com 1: 1 mistura de estirpes de estufa e vermelho fluorescentes a densidades celulares baixas. (Clique direito a download.) O vídeo mostra um curso de tempo de 48 horas. Barra de escala = 5 mm.
Discussão
A disponibilidade de uma caixa de ferramentas fluorescente para as bactérias, não só facilita o estudo da expressão do gene e proteína heterogénea 30,31 localização 32, mas também a análise da distribuição espacial das estirpes dentro de uma população 8. marcadores fluorescentes com suficientemente diferentes comprimentos de onda de excitação e emissão permitem localizar distintamente duas estirpes que de outra forma não se distinguem uns dos outros quando misturado. O protocolo descrito pode ser utilizado para observar a dinâmica populacional em culturas mistas, por exemplo, experimentos de competição ou sinergismo entre estirpes ou espécies. A capacidade para determinar as abundâncias relativas estirpes de marcação fluorescente numa população mista não se restringe à superfície enxame ligados, deslizante, ou colónias de biofilme, mas também podem ser utilizados para outros sistemas de biofilme multicelulares, incluindo submerso, fluxo de interface celular ou meio de ar- biofilmes 27,33-35.
_content "> Enquanto a técnica apresentada é uma ferramenta poderosa para detectar a distribuição espacial das estirpes e experiências de competição de criação, como também permite a expressão do gene seguinte heterogeneidade em colónias em expansão. As condições de cultura aqui descritas aplicam-se para B. subtilis e os parâmetros exactos de expansão sobre meio de agar podem requerer optimização para outras espécies ou estirpes de 20. a colocação das amostras em uma câmara de incubação, enquanto permite que o experimentador imagiologia para seguir a dinâmica das populações no tempo, embora a atenção deve ser dada para o nível de humidade no interior da câmara durante a incubação.As técnicas descritas aqui também requerem a modificação genética das estirpes bacterianas examinados de modo a que as estirpes expressam marcadores fluorescentes que podem ser distinguidas umas das outras. Além disso, além de ter excitação e espectros de emissão distintos, recomenda-se que os dois marcadores fluorescentes escolhidos têm quant semelhanterendimentos da UM (ie proporção de fotões absorvidos que são emitidos) e são expressos em um nível comparável. Além disso, as alterações de intensidade relativa no tempo pode ser medida e normalizada para um ponto de tempo precoce de uma experiência. O aumento relativo ou diminuição pode ser então comparadas entre os diferentes fluoróforos com diferentes eficiência quântica. Para o sistema experimental apresentado, as proteínas e verde- fluorescentes vermelho-diferentes foram testadas previamente 36,37 para seleccionar para os pares fluorescentes mais optimizados que possam ser detectados em B. subtilis. O tempo de exposição ideal deve ser determinada para cada proteína fluorescente e amostra. Certos densidades celulares ou várias camadas de células pode ser necessário para detectar o sinal de forma eficiente dentro da população. Certas proteínas fluorescentes pode ter baixas intensidades das células bacterianas devido à expressão ineficiente e / ou a tradução da proteína e, assim, baixo rendimento quântico. Tais marcadores fluorescentes ineficientes poderia reduce a sensibilidade do sistema e aumentar o tempo necessário para detectar as estirpes bacterianas, possivelmente resultando na citotoxicidade pela luz de excitação. As intensidades fluorescentes pode ser consequentemente modificada através da alteração do promotor utilizado para expressar o gene que codifica repórter fluorescente. Um nível de expressão que é demasiado elevada poderia resultar em excesso desnecessário da proteína fluorescente que conduz a custos de fitness prejudiciais para a bactéria. Ao realizar experimentos de competição, deve-se considerar o custo de particular a produção da proteína fluorescente nas células. As experiências de controlo, onde os marcadores fluorescentes são trocados entre as linhagens competiram ou onde duas estirpes isogénicas que diferem apenas em seus marcadores fluorescentes são competiram entre si, são sempre necessários para determinar qualquer viés em direção a um marcador. Os tempos de vida das proteínas fluorescentes dentro das células também pode afectar a intensidade medida. Além disso, a autofluorescência de determinada espécie bacterianas pode exigir o uso de diferentes outros do que o descrito aqui marcadores fluorescentes.
Para determinar com precisão a distribuição espacial e a abundância das estirpes bacterianas distintas, o sinal de fundo proveniente da primeira proteína fluorescente durante a utilização do filtro de fluorescência para o segundo marcador fluorescente e vice-versa, deve ser individualmente testado em amostras de monoculturas (contendo as bactérias que produzem apenas um marcador ). Isto permite que a subtracção de sobreposição de intensidades de sinal fluorescente. É importante notar, como o estereomicroscópio regista o sinal de fluorescência a partir de cima da colónia expansão, o protocolo apresentado é conveniente para determinar o arranjo espacial em duas dimensões. A arquitectura da população bacteriana expansão pode resultar em níveis variáveis de fluorescência (isto é, rugas estruturas semelhantes podem conter mais células que exibem elevadas intensidades de fluorescência locais). Portanto, a análise descrita das imagens de determines a distribuição espacial, mas não a abundância das estirpes dentro de um determinado local. Protocolos anteriores descreveram a preparação de amostras para swarming 20 ou fluorescência de imagem da dinâmica da população nas colônias bacterianas 38, mas nosso protocolo combina estas técnicas. Outras técnicas de microscopia que permitem a observação de três resolução dimensional da estrutura da população (por exemplo, laser confocal microscopia de varrimento ou microscopia 39,40 iluminação estruturada 41) pode ser aplicado a amostras com complexidades estruturais aumentados. Estas técnicas adicionais também suportam a detecção de uma única célula base das estirpes 31 que não está disponível usando oculares.
Divulgações
Os autores declaram que não têm interesses financeiros concorrentes.
Agradecimentos
Este trabalho foi financiado pela concessão KO4741 / 3-1 da Deutsche Forschungsgemeinschaft (DFG). Além disso, o laboratório de Á.TK foi apoiado por uma bolsa de integração na carreira Marie Skłodowska Curie (PheHetBacBiofilm) e concede KO4741 / 2-1 da DFG. TH, AD, RG-M., E EM foram apoiados pela International Max Planck Research School, Fundação Alexander von Humboldt, Consejo Nacional de Ciencia y Tecnología-Serviço Alemão de Intercâmbio Acadêmico (DAAD CONACyT-), e bolsas de estudo JSMC, respectivamente.
Materiais
| Name | Company | Catalog Number | Comments |
| Lennox Broth (LB) | Carl Roth GmbH | X964 | |
| Agar-agar, Kobe I | Carl Roth GmbH | 5210 | |
| Petri dish (90 mm diameter) | any | NA | Use Petri dishes without ventillation cams |
| Petri dish (35 mm diameter) | any | NA | Use Petri dishes without ventillation cams |
| Difco Nutrient Broth | BD Europe | 234000 | |
| KCl | any | NA | |
| MgSO,4 7H2O | any | NA | |
| Ca(NO3)2 4H2O | any | NA | |
| MnCl24H2O | any | NA | |
| FeSO4 | any | NA | |
| D-Glucose | any | NA | |
| Fluorescence AxioZoom V16 time-lapse microscope | Carl Zeiss Microscopy GmbH | see bellow detailed description | |
| AxioZoom V16 Microscope body | Carl Zeiss Microscopy GmbH | 435080 9030 000 | |
| Phototube Z 100:0 for Axio Zoom V16 | Carl Zeiss Microscopy GmbH | 435180 9020 000 | without eyepieces |
| Fluar Illuminator Z mot Fluorescence intermediate tube for Axio Zoom.V16 | Carl Zeiss Microscopy GmbH | 435180 9060 000 | |
| Controller EMS 3 | Carl Zeiss Microscopy GmbH | 435610 9010 000 | |
| System Control Panel SYCOP 3 | Carl Zeiss Microscopy GmbH | 435611 9010 000 | |
| Reflector module Z | Carl Zeiss Microscopy GmbH | 435180 9160 000 | For Fluar Illuminator Z mot on Axio Zoom.V16 and SYCOP 3 |
| Filter set 38 HE eGFP shift free (E) | Carl Zeiss Microscopy GmbH | 489038 9901 000 | EX BP 470/40, BS FT 495, EM BP 525/50 |
| Filter set 63 HE mRFP shift free (E) | Carl Zeiss Microscopy GmbH | 489063 0000 000 | EX BP 572/25, BS FT 590, EM BP 629/62 |
| Mount S | Carl Zeiss Microscopy GmbH | 435402 0000 000 | |
| Objektive PlanApo Z 0,5x/0,125 FWD 114 mm | Carl Zeiss Microscopy GmbH | 435280 9050 000 | 164 mm parfocal length; M62x0.75 thread at front |
| Coarse/fine drive with profile column | Carl Zeiss Microscopy GmbH | 435400 0000 000 | 490 mm, 10 kg load capacity, compatible with stand bases 300/450 |
| Stand base 450 | Carl Zeiss Microscopy GmbH | 435430 9902 000 | |
| Cold-light source Zeiss CL 9000 LED CAN | Carl Zeiss Microscopy GmbH | 435700 9000 000 | |
| CAN-bus cable | Carl Zeiss Microscopy GmbH | 457411 9011 000 | 2.5 m length |
| Slit-ring illuminator | Carl Zeiss Microscopy GmbH | 417075 9010 000 | d = 66 mm |
| Flexible light guide 1500 | Carl Zeiss Microscopy GmbH | 417063 9901 000 | 8/1,000 mm |
| Illumination Adapter for light guide | Carl Zeiss Microscopy GmbH | 000000 1370 927 | |
| Lightguide HXP with liquid fill | Carl Zeiss Microscopy GmbH | 000000 0482 760 | ø3 mm x 2,000 mm |
| Camera Adapter 60N-C | Carl Zeiss Microscopy GmbH | 426113 0000 000 | 2/3" 0.63X |
| High Resolution Microscopy Camera AxioCam MRm Rev. 3 FireWire | Carl Zeiss Microscopy GmbH | 426509 9901 000 | |
| AxioCam FireWire Trigger Cable Set | Carl Zeiss Microscopy GmbH | 426506 0002 000 | for direct shutter synchronization |
| ZEN pro 2012 | Carl Zeiss Microscopy GmbH | 410135 1002 120 | Blue edition, requires min. Windows 7 64-bit |
| ZEN Module Time Lapse | Carl Zeiss Microscopy GmbH | 410136 1031 110 | |
| Standard Heating Stage Top Incubator | Tokai Hit | INUL-MS1-F1 | |
| Zeiss Stereo Microscope Base Adapter | Tokai Hit | MS-V12 | |
| Softwares | |||
| ImageJ | National Institute of Health, Bethesda, MD, USA | v 1.49m | |
| BioVoxxel plugin | BioVoxxel | http://www.biovoxxel.de/development/ |
Referências
- Nadell, C. D., Xavier, J. B., Foster, K. R. The sociobiology of biofilms. FEMS Microbiol Rev. 33 (1), 206-224 (2009).
- West, S. A., Griffin, A. S., Gardner, A., Diggle, S. P. Social evolution theory for microorganisms. Nat Rev Microbiol. 4 (8), 597-607 (2006).
- Kovács, &. #. 1. 9. 3. ;. T. Impact of spatial distribution on the development of mutualism in microbes. Front Microbiol. 5, 649 (2014).
- Martin, M., Hölscher, T., Dragoš, A., Cooper, V. S., Kovács, &. #. 1. 9. 3. ;. T. Laboratory evolution of microbial interactions in bacterial biofilms. J Bacteriol. , (2016).
- Drescher, K., Nadell, C. D., Stone, H. A., Wingreen, N. S., Bassler, B. L. Solutions to the public goods dilemma in bacterial biofilms. Curr Biol. 24 (1), 50-55 (2014).
- Momeni, B., Waite, A. J., Shou, W. Spatial self-organization favors heterotypic cooperation over cheating. Elife. 2, e00960 (2013).
- van Dyken, J. D., Müller, M. J., Mack, K. M., Desai, M. M. Spatial population expansion promotes the evolution of cooperation in an experimental Prisoner's Dilemma. Curr Biol. 23 (10), 919-923 (2013).
- van Gestel, J., Weissing, F. J., Kuipers, O. P., Kovács, &. #. 1. 9. 3. ;. T. Density of founder cells affects spatial pattern formation and cooperation in Bacillus subtilis biofilms. ISME J. 8 (10), 2069-2079 (2014).
- Momeni, B., Brileya, K. A., Fields, M. W., Shou, W. Strong inter-population cooperation leads to partner intermixing in microbial communities. Elife. 2, e00230 (2013).
- Müller, M. J., Neugeboren, B. I., Nelson, D. R., Murray, A. W. Genetic drift opposes mutualism during spatial population expansion. Proc Natl Acad Sci U S A. 111 (3), 1037-1042 (2014).
- Pande, S., et al. Privatization of cooperative benefits stabilizes mutualistic cross-feeding interactions in spatially structured environments. ISME J. 10, 1413-1423 (2016).
- Hallatschek, O., Hersen, P., Ramanathan, S., Nelson, D. R. Genetic drift at expanding frontiers promotes gene segregation. Proc Natl Acad Sci U S A. 104 (50), 19926-19930 (2007).
- Flemming, H. C., Wingender, J. The biofilm matrix. Nat Rev Microbiol. 8 (9), 623-633 (2010).
- Vlamakis, H., Chai, Y., Beauregard, P., Losick, R., Kolter, R. Sticking together: building a biofilm the Bacillus subtilis way. Nat Rev Microbiol. 11 (3), 157-168 (2013).
- Lopez, D., Kolter, R. Extracellular signals that define distinct and coexisting cell fates in Bacillus subtilis. FEMS Microbiol Rev. 34 (2), 134-149 (2010).
- Lopez, D., Vlamakis, H., Kolter, R. Generation of multiple cell types in Bacillus subtilis. FEMS Microbiol Rev. 33 (1), 152-163 (2009).
- Mhatre, E., Monterrosa, R. G., Kovács, &. #. 1. 9. 3. ;. T. From environmental signals to regulators: Modulation of biofilm development in Gram-positive bacteria. J Basic Microbiol. , (2014).
- Zhang, W., et al. Nutrient depletion in Bacillus subtilis biofilms triggers matrix production. New J Physics. 16, 015028 (2014).
- Kearns, D. B. A field guide to bacterial swarming motility. Nat Rev Microbiol. 8 (9), 634-644 (2010).
- Morales-Soto, N., et al. Preparation, imaging, and quantification of bacterial surface motility assays. J Vis Exp. (98), (2015).
- Angelini, T. E., Roper, M., Kolter, R., Weitz, D. A., Brenner, M. P. Bacillus subtilis spreads by surfing on waves of surfactant. Proc Natl Acad Sci U S A. 106 (43), 18109-18113 (2009).
- Grau, R. R., et al. A Duo of Potassium-Responsive Histidine Kinases Govern the Multicellular Destiny of Bacillus subtilis. MBio. 6 (4), e00581 (2015).
- Park, S. Y., Pontes, M. H., Groisman, E. A. Flagella-independent surface motility in Salmonella enterica serovar Typhimurium. Proc Natl Acad Sci U S A. 112 (6), 1850-1855 (2015).
- van Gestel, J., Vlamakis, H., Kolter, R. From cell differentiation to cell collectives: Bacillus subtilis uses division of labor to migrate. PLoS Biol. 13 (4), e1002141 (2015).
- Abee, T., Kovács, &. #. 1. 9. 3. ;. T., Kuipers, O. P., van der Veen, S. Biofilm formation and dispersal in Gram-positive bacteria. Curr Opin Biotechnol. 22 (2), 172-179 (2011).
- Kovács, &. #. 1. 9. 3. ;. T. Bacterial differentiation via gradual activation of global regulators. Curr Genet. 62 (1), 125-128 (2016).
- Hölscher, T., et al. Motility, chemotaxis and aerotaxis contribute to competitiveness during bacterial pellicle biofilm development. J Mol Biol. 427 (23), 3695-3708 (2015).
- Seminara, A., et al. Osmotic spreading of Bacillus subtilis biofilms driven by an extracellular matrix. Proc Natl Acad Sci U S A. 109 (4), 1116-1121 (2012).
- Patrick, J. E., Kearns, D. B. Laboratory strains of Bacillus subtilis do not exhibit swarming motility. J Bacteriol. 191 (22), 7129-7133 (2009).
- de Jong, I. G., Beilharz, K., Kuipers, O. P., Veening, J. W. Live Cell Imaging of Bacillus subtilis and Streptococcus pneumoniae using Automated Time-lapse Microscopy. J Vis Exp. (53), e3145 (2011).
- Garcia-Betancur, J. C., Yepes, A., Schneider, J., Lopez, D. Single-cell analysis of Bacillus subtilis biofilms using fluorescence microscopy and flow cytometry. J Vis Exp. (60), (2012).
- Turnbull, L., et al. Super-resolution imaging of the cytokinetic Z ring in live bacteria using fast 3D-structured illumination microscopy (f3D-SIM). J Vis Exp. (91), e51469 (2014).
- Barken, K. B., et al. Roles of type IV pili, flagellum-mediated motility and extracellular DNA in the formation of mature multicellular structures in Pseudomonas aeruginosa biofilms. Environ Microbiol. 10 (9), 2331-2343 (2008).
- Oliveira, N. M., et al. Biofilm formation as a response to ecological competition. PLoS Biol. 13 (7), e1002191 (2015).
- Wang, X., et al. Probing phenotypic growth in expanding Bacillus subtilis biofilms. Appl Microbiol Biotechnol. 100, 4607-4615 (2016).
- Detert Oude Weme, R. G., et al. Single cell FRET analysis for the identification of optimal FRET-pairs in Bacillus subtilis using a prototype MEM-FLIM system. PLoS One. 10 (4), e0123239 (2015).
- Overkamp, W., et al. Benchmarking various green fluorescent protein variants in Bacillus subtilis, Streptococcus pneumoniae, and Lactococcus lactis for live cell imaging. Appl Environ Microbiol. 79 (20), 6481-6490 (2013).
- Stannek, L., Egelkamp, R., Gunka, K., Commichau, F. M. Monitoring intraspecies competition in a bacterial cell population by cocultivation of fluorescently labelled strains. J Vis Exp. (83), e51196 (2014).
- Bridier, A., Briandet, R. Contribution of confocal laser scanning microscopy in deciphering biofilm tridimensional structure and reactivity. Methods Mol Biol. 1147, 255-266 (2014).
- Khajotia, S. S., Smart, K. H., Pilula, M., Thompson, D. M. Concurrent quantification of cellular and extracellular components of biofilms. J Vis Exp. (82), e50639 (2013).
- Neu, T. R., Lawrence, J. R. Innovative techniques, sensors, and approaches for imaging biofilms at different scales. Trends Microbiol. 23 (4), 233-242 (2015).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados