Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Identification et isolement des Oligopotent et des progéniteurs myéloïdes Lineage-commis de moelle osseuse de souris
Dans cet article
Résumé
Nous démontrons comment identifier et isoler les 6 sous-ensembles de progéniteurs myéloïdes de murins de la moelle osseuse à l’aide d’une combinaison de magnétique et de la fluorescence de tri (MACS et FACS). Ce protocole peut être utilisé pour les essais de culture in vitro (cultures de méthylcellulose ou liquide), des expériences in vivo transfert adoptif et analyses ARN/protéines.
Résumé
Les progéniteurs myéloïdes qui donnent les neutrophiles, les monocytes et les cellules dendritiques (CD) peuvent être identifiés en et isolées de la moelle osseuse de souris pour les analyses hématologiques et immunologiques. Par exemple, les études sur les propriétés moléculaires et cellulaires des populations progéniteur myéloïde peuvent révéler les mécanismes sous-jacents de transformation leucémique, ou démontrer comment le système immunitaire réagit à l’exposition de l’agent pathogène. Auparavant les stratégies cytométrie en flux décrits pour l’identification des progéniteurs myéloïdes ont permis des avancées significatives dans de nombreux domaines, mais les fractions qu'ils identifient sont très hétérogènes. Les stratégies de blocage plus couramment utilisés définissent des fractions de la moelle osseuse qui sont enrichies pour les populations souhaitées, mais contiennent également un grand nombre de progéniteurs « contaminer ». Nos études récentes ont résolu une grande partie de cette hétérogénéité et le protocole que nous présentons ici permet l’isolement des 6 sous-populations d’oligopotent et progéniteurs myéloïdes lineage-commis de 2 décrit précédemment les fractions de la moelle osseuse. Le protocole décrit 3 stades : des cellules d’isolement 1) de la moelle osseuse, 2) enrichissement de progéniteurs hématopoïétiques par cellule magnétique activé tri (appauvrissement de la lignée de MACS) et 3) identification de sous-ensembles progéniteur myéloïde par cytométrie en flux (y compris fluorescence-lancée de cellules tri, FACS, si vous le souhaitez). Cette approche permet la quantification de l’ancêtre et l’isolement pour une variété d’applications in vitro et in vivo et a déjà donné lieu à nouveau aperçu de voies et les mécanismes des neutrophiles, monocytes et différenciation de DC.
Introduction
Monocytes, les neutrophiles et les cellules dendritiques (CD) sont des cellules myéloïdes qui découlent des progéniteurs hématopoïétiques, principalement dans la moelle osseuse, par un processus appelé myelopoiesis. Progéniteurs myéloïdes communs (CMPs) sont susceptibles de produire des cellules myéloïdes, ainsi que mégacaryocytes et les érythrocytes, mais les cellules lymphoïdes pas. Progéniteurs de granulocyte-monocyte (BPF), qui sont dérivées du CMPs, produisent des granulocytes et des monocytes, mais ont perdu mégacaryocyte et érythrocytaire potentiel. Monocytes et classique et plasmacytoides DCs (CDC/PDC) semblent également découler de progéniteurs communs appelées progéniteurs de monocyte-DC (AMD), qui sont produites par le CMPs. graduel restriction du potentiel de la lignée aboutit à lineage-commis cellules souches : progéniteurs de granulocyte, progéniteurs des monocytes et progéniteurs des cellules dendritiques (Figure 1).
Weissman et ses collaborateurs ont que CMPs sont retrouvent dans le Lin– c-Kit+ Sca-1– CD34 (LKS–)+ FcγRlo fraction de moelle osseuse de souris, les BPF est exposées dans le CD34 LKS– + FcγR Salut fraction1. Toutefois, ces fractions « CMP » et « GMP » sont très hétérogènes. Par exemple, la fraction « GMP » contient également des progéniteurs de la lignée-commis de granulocytes et les monocytes progéniteurs1,2. Les AMD auraient séparément CX3CR1+ Flt3+ CD115+ progéniteurs qui expriment également CD34 et FcγR3,4. Les AMD donnent lieu à cDC/pDC-production commune DC progéniteurs (CDP), qui ont été signalés à exprimer des niveaux inférieurs de c-Kit (CD117) et ne sont pas inclus dans la fraction LKS– 5.
On pensait auparavant que les monocytes se posent via une voie unique (COP/MOP-GMP-MDP-monocyte). Compatible avec ce modèle, monocyte-commis progéniteurs produites par les bonnes pratiques de fabrication (nommé progéniteurs de monocyte, MPs)2 et les AMD (nommés progéniteurs communs de monocyte, cMoPs)6 semblent être les mêmes cellules sur la base de l’expression du marqueur de surface partagée . Cependant, nous avons récemment démontré que les monocytes sont produites indépendamment par PRIG et AMD et étaient capables de distinguer entre les députés et cMoPs unicellulaires RNA séquençage7.
Nous avons récemment modifié la Weissman « CMP » et la stratégie de blocage « GMP » afin d’identifier 6 sous-fractions de moelle osseuse de souris C57BL/6J contenant différent oligopotent et sous-ensembles progéniteur myéloïde lineage-commis. Nous avons pour la première fois que la coloration pour Ly6C et CD115 permet l’isolement des oligopotent PRIG, ainsi que des progéniteurs de granulocytes (GPs) et des progéniteurs de monocyte (MPs et cMoPs, qui nous est actuellement impossible de séparer) de la fraction « GMP »2 (LKS - CD34+ FcγRSalut porte ; La figure 1). Par la suite, nous avons démontré que les AMD se trouvent principalement dans la fraction « CMP » (CD34 LKS– + FcγRlo gate), qui contient aussi Flt3+ CD115lo et Flt3– sous-ensembles7 (Figure 1 ). Le CMP-Flt3+ CD115lo fraction donne les BPF et les AMD sur transfert adoptif. Le sous-ensemble de CMP-Flt3– contient des progéniteurs qui semblent être des intermédiaires entre les cellules de CMP-Flt3+ CD115lo et BPF. Contrairement aux AMD, le CMP-Flt3+ CD115lo et COP/MOP-Flt3– fractions possèdent également mégacaryocyte et érythrocytaire potentiel.
Il est important de noter, toutefois, que l'on ignore actuellement si les fractions « CMP » contiennent des cellules souches qui sont vraiment oligopotent (par exemple, les cellules individuelles au sein de la COP/MOP-Flt3+ CD115lo fraction qui possèdent des neutrophiles, monocytes, DC, mégacaryocyte et érythrocytaire potentiel), ou alternativement, constituent un mélange de géniteurs ayant un potentiel de lignée plus restreint. Colonie formant des dosages (cultures de méthylcellulose) a révélé des cellules avec des granulocytes (neutrophiles), érythrocytes, monocyte et mégacaryocyte potentiel (cellules GEMM) dans le « CMP », CMP-Flt3+ CD115lo et fractions de CMP-Flt3– 1 ,7, mais ne permettent pas l’évaluation du potentiel de DC. En revanche, formant des dosages des colonies a démontré l’existence d’oligopotent BPF (progéniteurs neutrophile et monocytes potentiels) dans le « GMP » fraction1,2, et cela est pris en charge par ces dernières monocellulaires d’analyse transcriptomique8. Il n'est pas actuellement connu, cependant, si ces BPF oligopotent produire aussi d’autres granulocytes (éosinophiles, basophiles et mastocytes).
Après ces études, nous démontrons maintenant comment 7 surface marqueurs (c-Kit, Sca-1, CD34, FcγR, Flt3, Ly6C et CD115) peut être utilisé pour identifier et isoler les 6 sous-ensembles d’oligopotent et commis à la lignée des progéniteurs myéloïdes. Le protocole décrit ici peut être appliqué pour les essais de culture in vitro (cultures de méthylcellulose ou liquide), expériences de transfert adoptif in vivo chez la souris et l’analyse moléculaire (séquençage de RNA en vrac et unicellulaires, Western Blot, etc.).
Le protocole se compose de 3 étapes : 1) préparation d’une suspension de cellule de la moelle osseuse des cellules, 2) programme d’enrichissement des progéniteurs hématopoïétiques (tri cellulaire magnétique activé) et 3) identification et isolement si vous le souhaitez, de sous-ensembles de progéniteurs de flux cytométrie en flux (en utilisant un analyseur ou un trieur, selon le cas). La première étape est l’isolement des cellules de moelle osseuse des fémurs et tibias de souris euthanasiés et ressemble aux autres protocoles décrits antérieurement9. Ensuite, l’échantillon est enrichi pour des cellules souches et progénitrices utilisant un cocktail d’anticorps dirigés contre des marqueurs de surface cellulaire des érythrocytes, polynucléaires neutrophiles, monocytes, lymphocytes, etc., pour épuiser les cellules différenciées. Ce n’est pas obligatoire, mais fortement recommandé pour optimiser la détection des sous-ensembles progénitrices et de réduire la quantité d’anticorps nécessaires à l’identification de l’ancêtre et le temps requis pour la cytométrie en flux. Le protocole de l’appauvrissement de lignage ci-dessous décrit Magnetic-Activated cellule Tri (MACS) en utilisant un Kit souris lignée cellulaire épuisement (qui contient les anticorps biotinylés contre CD5, CD45R (B220), CD11b, Gr-1 (Ly6G/C), 7-4 et Ter-119, plus anti-biotine microbilles) et un séparateur magnétique automatisé. L’étape finale consiste à déterminer (et le tri, si vous le souhaitez) des sous-ensembles progénitrices par cytométrie en flux. Le panel d’anticorps décrits ci-dessous (voir également tableau 1) a été conçu pour être utilisé dans un cytomètre en flux (analyseur ou trieur) avec les 4 lasers (405 nm, 488 nm, 561 nm, 640 nm).
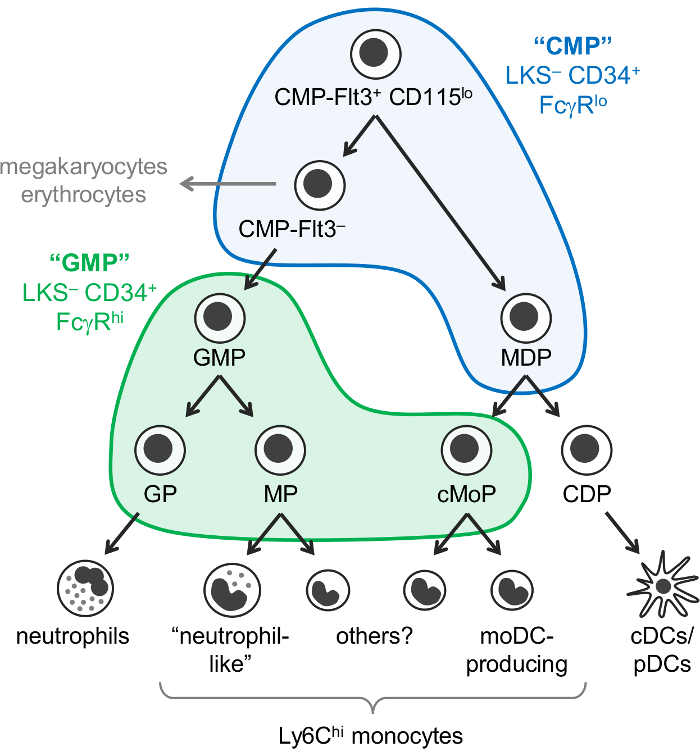
Figure 1 : progéniteurs DC, les monocytes et les neutrophiles et les voies de différenciation. Le modèle récemment révisé de myelopoiesis7 est illustré, avec les portes de Weissman pour « CMPs » (bleus) et les « Bonnes pratiques de fabrication » (vert)1 superposées. Ce chiffre a été modifié par Yáñez al 20177. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Protocole
Toutes les méthodes décrites ici ont été approuvées par l’animalier institutionnel et utilisation Comité (IACUC) du centre médical Cedars-Sinai.
1. isolement de la moelle osseuse de souris et de la préparation d’une Suspension de cellules du même
- Euthanasier la souris conformément aux lignes directrices.
- Placez la souris euthanasiée sur son dos et vaporisez-le avec 70 % d’éthanol (EtOH). Pratiquer une incision de petits (3-5 mm) dans chaque membre postérieur à la cheville niveau en utilisant les tranchants, ciseaux pointus ou tirer la peau vers le haut vers le corps pour retirer des jambes et exposer les muscles et les os.
- Retirez les muscles (quadriceps, ischio-jambiers, etc.) les os en les taillant avec des ciseaux, suivre la direction de l’OS. Veillez à ne pas endommager les os.
- Disloquer les fémurs de la hanche et couper le tissu conjonctif et musculaire pour détacher les pieds, en veillant à ne pas couper dans l’OS.
- Retirez les pieds en disloquant les chevilles des ossifiés, mettre les os dans un tube de PBS stérile ou de médias et les transportent sur la glace à la salle de culture de tissus.
Remarque : Pour d’autres applications (p. ex., dérivant des macrophages, des cellules de la moelle épinière total), il est possible de laisser les os sur la glace pendant un certain temps, mais pour l’isolement de l’ancêtre, il est important de procéder aussi rapidement que possible afin d’éviter une diminution de marqueurs de surface importantes (surtout CD115). - Dans une armoire de biosécurité, supprimer n’importe quel tissus mous restants des os en les frottant avec papier de soie.
Remarque : Le protocole doit être effectué dans une armoire de sécurité biologique si un échantillon stérile est nécessaire pour les essais de culture ou in vivo de cellules après triage des FACS. Si les cellules stériles ne sont pas nécessaires, l’ensemble du processus est possible sur le banc de laboratoire. - Plongez brièvement les os chez 70 % EtOH à désinfecter les eux et puis dans du PBS stérile pour les laver.
- Séparer les fémurs des tibias et des fibules en sectionnant le genou et jeter les fibules.
- Couper les extrémités de l’OS avec des ciseaux pointus.
- Récolter la moelle osseuse avec du PBS stérile froide ainsi jusqu'à ce que les os apparaissent en blancs :
- Saisissez chaque os avec une pince et insérer une aiguille 26G reliée à une seringue de 10 mL contenant du PBS stérile froide dans l’axe de l’os à une extrémité.
- Tenez l’os au-dessus d’un tube à centrifuger 50 mL et rincer 2-5 mL de PBS à travers l’os à la récolte de la moelle.
- Récolter la moelle de tous les 4 OS par souris (2 fémurs et 2 tibias).
- Pipetez doucement de haut en bas avec une pipette de 1 mL à désagréger la moelle épinière pour former une suspension cellulaire.
- Filtrer la suspension de la moelle osseuse à travers un morceau de 30 mailles de nylon μm pour éliminer les morceaux d’os et agrégats, générant ainsi une suspension monocellulaire de cellules.
- Centrifuger la suspension monocellulaire à 280 x g pendant 5 min et remettre le culot cellulaire dans un tampon stérile coloration (PBS + 0,5 % BF + 2 mM EDTA), à l’aide de 5 mL coloration tampon par souris (par exemple, 10 mL pour les cellules regroupées à partir d’et le fémur et le tibia de 2 souris).
- Prendre un 10 μL aliquote de l’échantillon, mélangez-le avec 10 μL bleu Trypan et charge 10 μL de l’échantillon de marqués au bleu de Trypan qui en résulte sur un hémocytomètre. Compter les cellules à l’aide d’un microscope optique.
Remarque : Si les cellules sont trop concentrés pour le comptage fiable, diluer l’échantillon avant d’ajouter le bleu Trypan.
2. progénitrices enrichissement par cellule magnétique activé tri (MACS)
- Centrifuger la suspension de toute cellule unique (ou autant de cellules qu’est tenus d’obtenir le rendement désiré) à 280 x g pendant 5 min à 4° C et remettre le culot dans 40 μL de tampon (PBS + 0,5 % BF + 2 mM EDTA) par 107 cellules de coloration.
Remarque : Il est également possible d’utiliser le tampon de MACS du fabricant de kit de déplétion lineage (voir Table des matières) comme une alternative au tampon de coloration. - Effectuer l’épuisement de la lignée conformément aux instructions fournies avec le kit de l’appauvrissement de lignage. Les instructions suivantes concernent le kit vu dans la Table des matières. Si un kit différent est utilisé, suivre les instructions du fabricant et passez à l’étape 2.3.
- Ajouter 10 μL biotine-anticorps cocktail par 107 cellules, mix de pipetage et incuber pendant 10 min à 4 ° C.
- Ajouter 30 μL de tampon/MACS de coloration de la mémoire tampon par 107 cellules et mélanger.
- Ajouter 20 μL anti-biotine microbeads par 107 cellules. Bien mélanger et laisser incuber une supplémentaire de 15 min à 4 ° C.
NOTE : Vortex les microbilles avant de les ajouter aux cellules de s’assurer qu’ils sont complètement dispersées. - Ajouter 1 mL de tampon tampon/MACS par 107 cellules de coloration, centrifuger les cellules à 280 x g pendant 5 min et remettre le culot cellulaire dans 500 μL coloration tampon/MACS tampon pour jusqu'à 108 cellules. Pour plus de 108 cellules, intensifier le volume de tampon tampon/MACS de coloration en conséquence.
- Préparer le séparateur magnétique automatique selon les instructions du fabricant.
- Placer le tube contenant les cellules marquées dans le séparateur et choisir le programme de sélection négative.
- Recueillir la fraction négative, qui contient les cellules de (Lin–) lignée négatif progénitrices enrichi. Jeter la fraction positive, qui contient les cellules de lignée positive marquée par magnétisme.
- Centrifuger le Lin– cellules à 280 x g pendant 5 min et Resuspendre le culot dans 2 mL de tampon tampon/MACS par souris (par exemple, 4 mL si la moelle osseuse a été regroupée à partir de 2 souris) de coloration.
Remarque : Le culot doit maintenant être blanc au lieu de rouge parce que les érythrocytes ont été épuisés. - Prendre un 10 μL aliquote des cellules Lin– , mélangez-le avec 10 μL bleu Trypan et charge 10 μL de l’échantillon de marqués au bleu de Trypan qui en résulte sur un hémocytomètre. Compter les cellules à l’aide d’un microscope optique.
Remarque : Si les cellules sont trop concentrés pour le comptage fiable, diluer l’échantillon avant d’ajouter le bleu Trypan.
3. myéloïdes progénitrices Identification et isolement par cellule activée par Fluorescence triant (FACS)
- Pour les tubes de microcentrifuge (1,5 ou 2 mL) étiquette 9 : 1) non souillées enregistré cellules, 2-8) unique de cellules colorées avec des anticorps conjugués fluorophore contre FcγR (CD16/32), c-Kit, Sca-1, CD34, Flt3 (CD135), Ly6C et CD115 pour compensation de sélection et de couleur de tension et 9) échantillons de cellules à colorer avec les 7 anticorps pour l’identification de souches et de tri.
- Distribuer 100 000 cellules à chacun des tubes de contrôle (tubes 1-8) et toutes les cellules restantes (ou moins si vous le souhaitez) dans le tube d’échantillon (tube 9).
Remarque : Si le volume de l’échantillon est supérieur à 1,5 à 2 mL, il peut être nécessaire pour distribuer les cellules de l’échantillon dans un tube de 15 mL, centrifuger eux et Resuspendre le culot (étape 3.3 ci-dessous) et ensuite transférer les cellules dans un tube de microcentrifugeuse pour les étapes ultérieures de coloration. - Centrifuger les cellules à 1 500 g pendant 5 min et remettre en suspension les pellets de tube de contrôle dans 100 μl tampon (PBS + 0,5 % BF + 2 mM EDTA) et le culot de tube d’échantillon dans 100 μl de coloration coloration tampon par 5 x 106 cellules (par exemple, 200 μl à 1 x 107 cellules).
- Ajouter 2 μg/mL anti-CD16/CD32-APC-Cy7 FcγR tache unique tube et le tube à essais. Vortex doucement pour mélanger et incuber pendant 10 min à 4 ° C.
Remarque : Il est important de colorer les cellules de l’échantillon avec l’anticorps FcγR (CD16/32), avant d’ajouter les autres anticorps pour empêcher la concurrence en raison de la liaison non spécifique induite par le Fc des anticorps. Pas Incuber les cellules avec un FcγR réactif de blocage. - Ajouter les autres anticorps pour les tubes de coloration unique correspondants (1 anticorps) et le tube à essais (tous les 6 anticorps) : 10 μg/mL c-Kit-Pacific Blue 2 μg/mL Sca-1-PE-Cy7, 25 μg/mL CD34-FITC, 10 μg/mL Flt3-APC, 2 µg/mL Ly6C-PerCP-Cy5.5 et 4 μg/mL CD115-PE. Vortex doucement et laisser incuber pendant 15 min à 4 ° C.
- Ajouter 900 μL coloration buffer pour les tubes de contrôle et 1 mL coloration par 107 cellules dans le tube échantillon. Centrifuger les cellules à 1 500 g pendant 5 min et Resuspendre le culot cellulaire en souillant la mémoire tampon : 200 μL / tube témoin et 500 μL / 25 x 106 cellules dans le tube à essais. Transférer les cellules resuspendues dans tubes de 5 mL FACS.
Remarque : Si le volume de l’échantillon est supérieur à 1,5 à 2 mL, il peut être nécessaire de transférer les cellules dans un tube de 15 mL pour la centrifugation. - Préparer le cytomètre en flux (analyseur ou trieur) selon les instructions du fabricant.
- Exécutez le non colorées et unique teinté de cellules témoins à travers le cytomètre de mettre en place les tensions et les compensations de couleur.
- Exécuter les cellules de l’échantillon, porte les sous-ensembles de progéniteurs comme indiqué ci-dessous et résumées dans la Figure 2et si vous le souhaitez, effectuer des FACS tri pour isoler les sous-ensembles de progéniteurs pertinentes.
Remarque : Il est généralement nécessaire d’acquérir environ 200 000 cellules afin d’obtenir au moins 1 000 événements dans chaque porte de géniteur.- Créer un terrain en dot de toutes les cellules, affichant FSC-A (axe x) et SSC-A (axe y) et porte pour exclure les débris et les cellules mortes = > « live » de cellules.
- Créer un terrain en dot des cellules « en direct », affiche un FSC-H (axe x) et FSC-W (axe y) et porte pour exclure les doublets = > porte de vivre/singulet (FSC).
- Créer un terrain de dot de vivre/singulet cellules (FSC), affiche un SSC-H (axe x) et SSC-W (axe y) et porte pour exclure les doublets = > live/singulet (FSC) / porte de singulet (SSC).
- Créer un terrain de dot de vivre/singulet (FSC) / cellules singulet (SSC), affiche un Sca-1 (axe x) et c-Kit (axe y) et porte pour choisir c-Kit+ Sca-1– cellules = > cellules LKS– .
- Créer un terrain en dot des cellules LKS– , affichant CD34 (axe x) et FcγR (axe y) et porte pour sélectionner CD34+ FcγRlo cellules = > « CMPs » et CD34+ FcγRSalut cellules = > « Bonnes pratiques de fabrication ».
Remarque : Il est important de porte le « CMPs » et les « Bonnes pratiques de fabrication » aussi précisément que possible. Il est utile d’utiliser un terrain de densité Pseudo-couleur ou un tracé d’isolignes pour déclenchement précis. - Créer un terrain de dot de « CMPs », affichant CD115 (axe x) et Flt3 (axe y) et porte pour sélectionner :
Flt3+ CD115lo cellules = > CMP-Flt3+ CD115lo cellules,
Flt3– CD115lo cellules = > CMP-Flt3– cellules,
Flt3+ CD115Salut cellules = > AMD. - Créer un terrain de dot de « Bonnes pratiques de fabrication », affichant Ly6C (axe x) et FcγR (axe y) et porte pour sélectionner Ly6C–cellules = > « Cellules GMP-Ly6C– » et Ly6C+ cellules = > « Cellules GMP-Ly6C+ ».
- Créer un terrain de dot de « Cellules de GMP-Ly6C– », affichant CD115 (axe x) et Flt3 (axe y) et porte pour sélectionner :
Flt3– CD115lo cellules = > BPF. - Créer un terrain de dot de « Cellules de GMP-Ly6C+ », affichant CD115 (axe x) et Flt3 (axe y) et porte pour sélectionner :
Flt3– CD115lo cellules = > GPs,
Flt3– CD115Salut cellules = > MPs + cMoPs.
Résultats
En utilisant le protocole décrit ci-dessus, il est possible d’obtenir 100 millions de cellules (y compris les globules rouges, ou 50 millions de cellules nucléées) des fémurs et tibias (2 cuisses) de souris C57BL/6J (6-8 semaines vieux, hommes ou femmes). 1-2 millions Lin– cellules peuvent être isolées par souris par épuisement de MACS des cellules Lin+ .
Chacun des sous-ensembles progéniteur my...
Discussion
La Weissman gating stratégie pour souris progéniteur myéloïde identification1 a été l’étalon-or pour les immunologistes et hématologues depuis près de 20 ans, mais il semble maintenant que les portes « CMP » et « GMP » sont très hétérogènes et plus précis stratégies de blocage sont nécessaires. Le protocole que nous avons décrite ici permet l’identification d’oligopotent et de sous-ensembles de lignée-commis chez les souris C57BL/6J pour une quantification plus pré...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce protocole a été développé à l’aide de fonds du Conseil de gouverneurs Regenerative Medicine Institut au Cedars-Sinai Medical Center (HSG), une carrière à bourse d’immunologie de l’Association américaine des immunologistes (à AY et HSG) et une bourse de chercheur de l’American Society of Hematology (à AY). Nous remercions le Flow Cytometry Core au Cedars-Sinai Medical Center d’assistance avec le tri des FACS.
matériels
| Name | Company | Catalog Number | Comments |
| Mouse: Wild-type C57BL/6J (CD45.2) | The Jackson Laboratories | Cat#JAX:000664 | |
| Lineage Cell Depletion Kit, mouse | Miltenyi Biotec | Cat#130-090-858 | |
| Rat anti-mouse CD34 (clone RAM34) FITC | BD Biosciences | Cat#553733 | |
| Rat anti-mouse CD16/CD32 (FcγR; clone 93) APC-Cy7 | BioLegend | Cat#101327 | |
| Rat anti-mouse Ly6A/E (Sca-1; clone 108113) PE-Cy7 | BioLegend | Cat#108114 | |
| Rat anti-mouse CD117 (c-Kit; clone 2B8) Pacific Blue | BioLegend | Cat#105820 | |
| Rat anti-mouse Ly6C (clone HK1.4) PerCP-Cy5.5 | BioLegend | Cat#128012 | |
| Rat anti-mouse CD115 (clone AFS98) PE | BioLegend | Cat#135506 | |
| Rat anti-mouse CD135 (Flt3; clone A2F10.1) APC | BD Biosciences | Cat#560718 | |
| CountBright Absolute Counting Beads | Thermo Fisher Scientific | Cat#C36950 | |
| AutoMACS Separator | Miltenyi Biotec | N/A | Use the "deplete" program |
| BD LSRFortessa | BD Biosciences | N/A | 5 lasers, 15 colors |
| BD FACS Aria III cell sorter | BD Biosciences | N/A | 5 lasers, 13 colors |
| FlowJo | FlowJo, LLC | https://www.flowjo.com | For further analysis of the .fcs files |
Références
- Akashi, K., Traver, D., Miyamoto, T., Weissman, I. L. A clonogenic common myeloid progenitor that gives rise to all myeloid lineages. Nature. 404 (6774), 193-197 (2000).
- Yáñez, A., Ng, M. Y., Hassanzadeh-Kiabi, N., Goodridge, H. S. IRF8 acts in lineage-committed rather than oligopotent progenitors to control neutrophil vs monocyte production. Blood. 125 (9), 1452-1459 (2015).
- Auffray, C., et al. CX3CR1+ CD115+ CD135+ common macrophage/DC precursors and the role of CX3CR1 in their response to inflammation. Journal of Experimental Medicine. 206 (3), 595-606 (2009).
- Fogg, D. K., et al. A clonogenic bone marrow progenitor specific for macrophages and dendritic cells. Science. 311 (5757), 83-87 (2006).
- Onai, N., et al. Identification of clonogenic common Flt3+M-CSFR+ plasmacytoid and conventional dendritic cell progenitors in mouse bone marrow. Nature Immunology. 8 (11), 1207-1216 (2007).
- Hettinger, J., et al. Origin of monocytes and macrophages in a committed progenitor. Nature Immunology. 14 (8), 821-830 (2013).
- Yáñez, A., et al. Granulocyte-monocyte progenitors and monocyte-dendritic cell progenitors independently produce functionally distinct monocytes. Immunity. 47 (5), 890-902 (2017).
- Olsson, A., et al. Single-cell analysis of mixed-lineage states leading to a binary cell fate choice. Nature. 537 (7622), 698-702 (2016).
- Amend, S. R., Valkenburg, K. C., Pienta, K. J. Murine hind limb long bone dissection and bone marrow isolation. Journal of Visualized Experiments. (110), (2016).
- Yáñez, A., Goodridge, H. S. Interferon regulatory factor 8 and the regulation of neutrophil, monocyte, and dendritic cell production. Current Opinion in Hematology. 23 (1), 11-17 (2016).
- Manz, M. G., Miyamoto, T., Akashi, K., Weissman, I. L. Prospective isolation of human clonogenic common myeloid progenitors. Proceedings of the National Academy of Sciences of the United States of America. 99 (18), 11872-11877 (2002).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon