Method Article
Microbiopsy absorbant d’échantillonnage et d’Extraction de l’ARN pour mini-Invasive, simultané de sang et analyse de la peau
Dans cet article
Résumé
Dans cet article, nous montrons comment la technique de microbiopsy absorbant est effectuée et comment l’échantillon peut être utilisé pour l’extraction de l’ARN pour l’échantillonnage simple et simultanée de la peau et le sang de façon mini-invasive.
Résumé
Biopsie cutanée classique limite la recherche clinique avec des domaines cosmétiquement sensibles ou des applications pédiatriques en raison de son caractère invasif. Nous décrivons ici le protocole afin d’utiliser un dispositif absorbant à base de microneedle, microbiopsy absorbant, pour mini-invasive d’échantillonnage de mélange de sang et de peau. Notre objectif est de contribuer à faciliter des progrès rapides dans la recherche clinique, la mise en place des biomarqueurs de la maladie de la peau et réduire les risques pour les participants à la recherche clinique. Contrairement aux techniques de biopsie de peau classique, le microbiopsy absorbant peut être effectuée en quelques secondes et ne nécessite pas de formation intensive grâce à son design simple. Dans ce rapport, nous décrivons l’utilisationdes microbiopsy absorbant, y compris le chargement et la demande, le bénévole. Ensuite, nous montrons comment isoler l’ARN de l’échantillon absorbée. Enfin, nous montrent l’utilisation de la transcription inverse quantitative PCR (RT-qPCR) pour quantifier les niveaux d’expression de mRNA de sang (CD3E et CD19) et peau (KRT14 et TYR). Les méthodes que nous décrivons utilisent les kits de plateau et réactifs. Ce protocole offre une approche mini-invasive pour l’échantillonnage simultané de la peau et le sang au sein de la même matrice microbiopsy absorbant. Nous avons identifié les comités d’éthique humaine, des cliniciens et des bénévoles d’appuyer cette approche de la recherche en dermatologie.
Introduction
Biopsie cutanée est une des techniques plus essentiels en dermatologie pour peau d’échantillonnage et diagnostic ultérieur des maladies de la peau au moyen d’évaluation histopathologique. La technique de biopsie consiste à un professionnel de la santé à l’aide d’une biopsie lame ou poinçon à retirer la lésion suspecte sur la peau du patient pour l’examen1. Bien que la technique soit efficace, il est très envahissante et limite la recherche clinique comme le point de terminaison fait habituellement appel à des techniques de biologie moléculaire2,3. Analyse moléculaire des maladies de la peau a le potentiel de fournir des informations biologiques très spécifiques qu’analyse histopathologique ne saurait, facilitant ainsi la drogue découverte et maladie diagnostic4,5. En outre, la demande d’échantillon de techniques moléculaires plus est relativement faible et peut entraîner une réduction de l’utilisation des animaux et permettre un plus grand nombre de répétitions. Par conséquent, il est clairement nécessaire pour une autre technique qui permet l’analyse moléculaire en recherche clinique et diminue les risques pour les participants.
Pour répondre à ce besoin dans le domaine, notre groupe a développé une plate-forme diagnostique microneedle roman, microbiopsy absorbant, ce qui permet la collecte d’une quantité infime de peau mélangée avec du sang dans une manière simple et peu invasive6. L’objectif de cette publication est de décrire la microbiopsy absorbant comme un outil d’échantillonnage pour faciliter l’analyse moléculaire grâce à l’extraction de l’ARN en recherche clinique.
Nous avons décrit précédemment, la première version de microbiopsy, microbiopsy de peau, qui se compose d’un micro aiguille faite d’un dessin de trois couches de tôle pour extraire les petits morceaux de tissus de peau7. La nouveauté de cet appareil vient de multiples points de contact de la micro aiguille qui permet d’extraction efficace tissu3. En revanche, une biopsie à l’emporte-pièce circulaire peau fournit qu’un seul point de contact et déchire tout simplement la peau sans capturer n’importe quel échantillon dans certains cas. Basé sur le microbiopsy de la peau, nous avons développé récemment le microbiopsy absorbant qui a tant de sang et de peau d’échantillonnage des capacités. Le dispositif s’est avéré être réalisable pour utilisation dans les régions pauvres en ressources dans une récente étude épidémiologique6.
Grâce à sa conception simple, le microbiopsy absorbant peut être effectuée en quelques secondes et ne nécessite pas de formation approfondie. En outre, anesthésique local n’est pas nécessaire, et le site d’application ne provoque pas de cicatrices. Le présent protocole permet aux chercheurs ou professionnels de la santé sans échantillonnage pertinente de formation pour obtenir des données pour l’analyse moléculaire ciblée de la peau. Nous prévoyons microprélèvement dispositifs pour devenir la routine dans la recherche sur la peau à l’avenir.
Bien que le microbiopsy a été signalée dans d’autres études de la maladie de peau qui implique des techniques de diagnostic moléculaire6,8,9,10, tels que les virus du papillome humain détection de l’ADN, ce protocole est le premier à démontrer les détails des techniques d’extraction et de traitement échantillon pour le microbiopsy absorbant. En outre, c’est le premier rapport décrivant le profil d’expression de gène relative de la peau et des cellules sanguines dans les échantillons de microbiopsy.
Protocole
L’étude a été approuvée par métro South Human Research Ethics Committee et l’Université du Queensland Human Research Ethics Committee (HREC-13-QPAH-551 et UQ2013001551) et le Comité d’éthique humaine (200607) University of South Australia.
1. absorbant Microbiopsy Fabrication
- Laser de couper des pièces micro aiguille de 50 µm qualité médicale et acier inoxydable couche absorbante (papier filtre), respectivement (Figure 1).
Remarque : Autres pièces, y compris le piston et le boîtier, de l’appareil sont fabriqués avec des techniques de prototypage rapide, telles que l’impression 3D. - Faire tremper toutes les pièces, à l’exception de la couche absorbante, à 70 % d’éthanol (EtOH) pendant 5 min et air sécher pendant 30 min par la suite.
- Assembler le trois couches de microneedle, avec la couche absorbante au milieu (Figure 1 a).
- Insérez la micro aiguille assemblé dans la fente du piston.
- Mettre sur le ressort et introduire le piston dans le boîtier (Figure 1C). Éviter le micro aiguille toucher quoi que ce soit dans la procédure d’assemblage pour assurer la qualité de l’aiguille.
- Sceller le microbiopsy absorbant assemblé dans un emballage pour la stérilisation par irradiation-γ avant d’utiliser.
2. l’échantillonnage avec la Microbiopsy absorbant
- Porter des gants jetables et spray mains et outils, tels que pinces, avec 70 % EtOH.
- Désinfectez la zone d’application avec un imbibé d’alcool.
- Supprimer la microbiopsy de l’emballage stérilisé de rayonnement γ sans toucher le micro aiguille dans le processus.
- Charger l’appareil en tirant sur le piston pour comprimer le ressort jusqu'à ce qu’il y a un « clic » sonore et les écluses de piston en place.
- Viser le dispositif à la peau à échantillonner, veiller à ce que la zone d’échantillonnage est dans une position fixe.
- Assurez-vous que le périphérique est approximativement perpendiculaire à la peau, puis appliquez ≥ 1 kg de force à l’appareil sur la peau.
Remarque : Application d’une force vers le bas dans la peau est nécessaire pour assurer une pénétration suffisante de la microneedle. - Appuyez sur le déclencheur et de maintenir le dispositif en place pour un minimum de 10 s pour l’échantillon de sang être absorbés.
Remarque : Si l’appareil est libéré avant 10 s, il est moins échantillon capturé.
3. Extraction de l’ARN
Remarque : Les procédures d’extraction de RNA ont été modifiés du protocole des constructeurs pour l’isolation optimale de l’ARN d’échantillon microbiopsied. Tous les réactifs et la colonne utilisée dans l’extraction de l’ARN sont inclus dans le kit, sauf indication contraire.
- Sortez le piston et absorbant micro aiguille de l’affaire par les mains doucement. Veiller à ce que la pointe de la micro aiguille n’entre pas en contact avec quoi que ce soit au moment du retrait.
- Retirer le micro aiguille absorbant le piston. Agripper les deux trous sur le micro aiguille avec une paire de pince stérile et tirer.
- Mettre le micro aiguille intact dans un tube de microcentrifuge de 2 mL (pas de kit d’isolation de la RNA ; voir Table des matières) préremplie avec 50 µL de tampon d’Extraction, avec le côté de l’aiguille vers le bas, vers le bas. Pour minimiser la perte de l’échantillon, laisser le tube sur la glace sèche pendant 5 min avant de traiter l’échantillon.
- Vortex brièvement (3 à 5 s) à supprimer certains des matériaux prélevés de la pointe.
- Incuber le micro aiguille dans le tube sur un balancier pendant 30 min à 42 ° C.
Remarque : La solution tampon est absorbée dans la couche absorbante immédiatement. - Placer la partie supérieure du niveau micro aiguille (le côté opposé de l’aiguille) avec le bord supérieur du tube à centrifuger 2 mL à l’aide d’une pince stérile.
- Fermer le couvercle du tube de telle sorte que la partie supérieure de la micro aiguille est maintenue en place entre le capuchon et tube.
- Tourner le tube à 16 000 x g ou à vitesse maximale pendant 30 s pour enlever les matériaux prélevés de la couche absorbante de l’appareil.
- Retirez délicatement le micro aiguille avec une pince sans tremper dans la solution tampon. Maintenir le tube et jeter le microneedle.
- Centrifuger les échantillons à 3 000 x g pendant 2 min. Pipette le surnageant contenant l’ARN extrait soigneusement dans un nouveau tube de microcentrifuge.
- Ajouter 250 µL de tampon de conditionnement sur la membrane de filtre de colonne de purification et incuber pendant 5 min à température ambiante.
- Centrifuger la colonne dans le tube de prélèvement à 16 000 x g ou à la vitesse maximale pendant 1 minute.
- Ajouter 50 µL de 70 % EtOH dans l’échantillon prélevé d’étape 3,9. Bien mélanger en doucement pipetage de haut en bas.
- Transférer le mélange (~ 100 µL) dans la colonne de purification préconditionnés.
- Centrifuger pendant 2 min à 100 g x immédiatement, suivie d’une centrifugation à 16 000 x g pendant 30 s pour supprimer les intermédiaires.
- Ajouter 100 µL de tampon de lavage 1 (W1) dans la colonne de purification et centrifuger pendant 1 min à 8 000 x g.
- Préparer la solution de DNase en ajoutant 5 µL de DNase j’ai Solution-mère à 35 µL de tampon de RDD dans une micro-centrifugeuse distincte de tube pour chaque échantillon. Mélanger en retournant délicatement.
Remarque : Le traitement de la DNase doit être effectuée sur la colonne de purification, au lieu de dans la plaque PCR par la suite, en raison de la quantité de l’échantillon. - Ajouter 40 µL du mélange de l’incubation de DNase directement dans la membrane de colonne de purification. Incuber à température ambiante pendant 15 minutes.
- Ajouter 40 µL de W1 dans la membrane de colonne de purification. Centrifuger à 8 000 x g pendant 15 s.
- Pipetter 100 µL de tampon de lavage 2 (W2) dans la colonne de purification après le traitement de la DNase et centrifuger pendant 1 min à 8 000 x g.
- Ajouter un autre à 100 µL de W2 dans la colonne de purification et centrifuger pendant 2 min à 16 000 x g. cocher de la colonne de purification pour n’importe quel tampon de lavage résiduelle. Re-centrifuger à 16 000 x g pendant 1 min, si n’importe quel tampon de lavage reste.
- Transférer la colonne de purification dans un nouveau tube de microcentrifugation 0,5 mL fourni dans le kit.
- Distribuer 11 µL de RNase eau exempte (non fournie dans le kit d’isolation d’ARN), au lieu de tampon d’élution (EB), directement sur la membrane de la colonne de purification. Appuyer doucement sur l’embout de la pipette à la surface de la membrane pendant la distribution de l’eau exempte de RNase pour assurer une absorption maximale d’eau exempte de RNase dans la membrane. Incuber à température ambiante pendant 2 min.
- Centrifuger la colonne pendant 1 min à 1 000 x g pour distribuer l’eau exempte de RNase dans la colonne, puis centrifuger pendant 16 000 x g à 1 min à éluer l’ARN.
Remarque : La quantification de RNA n’est pas recommandée en raison de la faible quantité de l’échantillon. - Arrêter le point #1 : stocker l’ensemble de l’échantillon RNA à-80 ° C si la synthèse de cDNA n’est pas exécutée immédiatement.
Remarque : Évitez congélation si pas nécessaire compte tenu de la faible concentration de l’échantillon de RNA.
4. synthèse de cDNA
- Décongeler l’échantillon congelé sur la glace (à partir de 3,25 étape).
- Synthèse de cDNA de l’ARN total avec kit de synthèse de cDNA conformément à la procédure ci-dessous.
- Préparer le mélange de la réaction : 11 µL d’ARN total, 4 µL de 5 x TransAmp tampon, 1 µL de la transcriptase inverse, 4 µL d’eau exempte de RNase/DNase. Mélanger doucement de pipetage.
- Exécuter la transcription renversée sur un appareil cycleur thermique : 25 ° C pendant 10 min, 42 ° C pendant 15 min, suivie d’une étape d’inactivation de final de la transcriptase inverse à 85 ° C pendant 5 min.
- Arrêter le point #2 : conserver l’échantillon inverse transcrit à-80 ° C jusqu'à l’utilisation.
5. qPCR réaction et analyse de données
Remarque : Les amorces pour qPCR réactions ont été conçus pour enjamber les frontières intron pour éviter l’amplification de l’ADN génomique à l’aide de NCBI Primer BLAST (www.ncbi.nlm.nih.gov/tools/primer-blast/). Le gène de référence utilisé dans cette étude est RPLP0 (voir la section « Discussion » pour plus d’informations).
- Décongeler l’échantillon congelé sur la glace (à partir de 4.5 étape).
- Effectuer qPCR avec le kit de mélange maître qPCR conformément à la procédure ci-dessous.
- Préparer le mélange principal contenant les éléments suivants par réaction : 5 µL du mélange de réaction x qPCR 2, 0,4 µL d’apprêt avant de 10 µM, 0,4 µL d’apprêt inverse de 10 µM, 2,2 µL d’eau exempte de DNase. Mélanger doucement de pipetage. Mettre en place les échantillons en géométrie.
- Distribuer 8 µL du mélange maître dans chaque puits d’une plaque PCR 384 puits premier, puis 2 µL du cDNA de chaque échantillon (un total de 10 µL / puits).
Remarque : Aucun contrôle de modèle (NTC), aucun contrôle de la transcriptase inverse (-RT) figurent parmi les témoins négatifs. - Sceller la plaque avec film adhésif et centrifuger brièvement.
- Exécuter la réaction d’amplification sur un thermocycleur en temps réel : 95 ° C pendant 2 min (activation de la polymérase), suivie de 40 cycles de 95 ° C pendant 5 s (dénaturation), 60 ° C pendant 10 s (recuit) et 72 ° C pendant 10 s (extension).
- Calculer la concentration en ARN d’échantillons par interpolation d’une courbe standard, préparée à partir de modèles avec des concentrations connues.
- Créer une Nouvelle expérience dans le logiciel.
- Cliquez sur définir et définir des normes à mettre en place une dilution en série de plaques.
- Sélectionnez et agencez les puits pour les échantillons et étalons. Cliquez sur appliquer.
- Cliquez sur analyser dans l’onglet résultats afin d’évaluer la courbe d’étalonnage. Confirmer la pente, la valeur de R2 , erreur et efficacité de l’amplification respectent les critères expérimentaux.
- Vérifier les quantités des échantillons inconnus visuellement sur la courbe d’étalonnage selon leurs valeurs de Ct.
Remarque : La construction de la courbe d’étalonnage, y compris le choix du facteur de dilution, s’effectue selon des protocoles publiés11,12,13.
- Effectuer des analyse d’expression génique de l’ARNm en utilisant la méthode ΔCt (Ct est normalisée à un gène de référence).
- Soustraire la valeur de Ct du gène cible de la valeur de Ct du gène de référence pour obtenir la valeur de ΔCt.
- Moyenne des valeurs ΔCt de répétitions techniques.
- Tracer et d’analyser l’expression des gènes avec statistiques logiciel11,14 (voir Table des matières).
Remarque : Par ailleurs, l’analyse de l’expression génique s’effectue selon protocoles publiés13.
Résultats
Nous avons précédemment rapporté que le microneedle de le microbiopsy peau pénètre la peau environ 500 µm profond7. La conception de micro aiguille de microbiopsy peau est très semblable à celui sur microbiopsy absorbant (Figure 2 a). Alors que le microbiopsy absorbant consistait en une couche absorbante au milieu pour l’absorption de sang, la peau microbiopsy contenait un canal pour capturer mécanique des tissus cutanés. L’utilisation de couche absorbante a également conduit à une légère différence dans la dimension micro aiguille (absorbant : 1,50 x 0,50 x 0,21 mm vs la peau : 1,50 x 0,50 x 0,15 mm).
Figure 2 b montre les sites demande 5 min après absorbant et microbiopsies de peau ont été appliqués sur le bras gauche volar d’homme volontaire. En raison de la similitude entre les deux dessins de microneedle, les sites d’application depuis les deux appareils étaient comparables, avec érythème mineur. Les deux sites d’application n’étaient pas visibles après 48 h avec aucune cicatrice gauche derrière. Ceci appuie l’hypothèse que cet appareil minimalement invasif a le potentiel pour aider plusieurs sites d’application d’écran ou à exécuter sur une base régulière.
Figure 2C montre une image représentative de la couche absorbante de l’absorbant microbiopsy après avoir appliqué au bras d’un homme de volar bénévoles. Comme le montre la figure, quelques petits morceaux de peau ont été capturés près du bout de la microneedle, et peu de sang a été absorbée par le papier filtre. Cela indique que l’appareil pénètre la peau et capturé la peau et le sang simultanément au sein de la même matrice microbiopsy absorbant. Figure 2d montre un échantillonnage après la peau microbiopsy, l’ancienne génération de microbiopsy, à titre de comparaison. Le canal de la microbiopsy de peau capturé un petit morceau de peau, mais la quantité de sang sur le micro aiguille était faible par rapport à la microbiopsy absorbant. Les deux sites d’application n’étaient pas visibles sous 48 h. Dans l’expérience, la microbiopsy absorbant a été appliqué avec un temps de retenue de 10 s après l’application, tandis que la peau microbiopsy est sorti immédiatement après l’application en raison de la différence de conception.
Comme le montre la Figure 3 a, deux groupes d’absorbant microbiopsy ont participé à cette expérience basée sur le protocole d’application : « Libération immédiate » et tenue 10-s'. Pour le groupe de la « Libération immédiate », l’appareil a été utilisée avec le protocole de microbiopsy de peau original même, avec le dispositif étant supprimé du site demande immédiatement après l’application. Pour la tenue de 10-s ' groupe, le dispositif a été maintenu en place après l’application pour 10 s pour améliorer la collecte de l’échantillon. Les deux groupes de microbiopsy absorbant ont été mis en place pour démontrer comment l’approche d’application pourrait affecter la quantité d’échantillon. Le temps de 10 s retenue a été choisi comme un délai raisonnable sur le plan clinique pour démontrer que la durée d’application affecte la quantité d’échantillon récupérable.
La quantité d’ARN récupéré des deux groupes microbiopsy absorbants ont été 0.33 ± 0,39 ng « Libération immédiate » et 1,43 ± 0,88 ng pour tenue 10-s' (Figure 3 a, n = 20), ce qui suggère une augmentation de 4 fois et la durée de maintien supplémentaire. Ceci indique qu’appliquer le dispositif absorbant et la durée de maintien de 10 s a entraîné plusieurs ARN extrait. La différence peut être due à la présence de l’échantillon sanguin accru dans la couche absorbante (Figure 3C). En effet, le groupe « Libération immédiate » (Figure 3 b) n’a pas pu recueillir une quantité semblable de sang avec la couche absorbante par rapport à la tenue de 10-s ' groupe (Figure 3C). Il est à noter également que les microbiopsies plus immédiatement libérés étaient à proximité de l’axe des abscisses, suggérant qu’ils étaient des événements négatifs ou affichent une très faible quantité d’ARN. Par conséquent, le résultat validé l’hypothèse selon laquelle le temps de retenue aurait un impact sur les performances de l’appareil que peut prendre un temps pour le sang être absorbée par la couche absorbante.
Étant donné que l’appareil a été conçu pour le sang et la peau d’échantillonnage, qPCR a été utilisé pour détecter l’expression de la peau et le sang des biomarqueurs pour les deux appareils pour comparaison. Nous avons utilisé la tyrosinase, TYR, comme biomarqueur pour des mélanocytes et KRT14 comme biomarqueur pour des kératinocytes de l’épiderme viable. Échantillons de peau microbiopsy faisaient parties de l’expérience à titre de comparaison. Tel qu’illustré à la Figure 4, bien que la peau et microbiopsies absorbants ont été appliqués différemment en raison de la différence dans la conception, l’expression niveaux pour tous les deux la peau biomarqueurs TYR et KRT14 étaient comparables pour les deux appareils tel qu’indiqué par le données. Biomarqueurs de globules blancs (CD3E, lymphocytes T et CD19, cellules B) se sont révélés plus répandus dans les échantillons de microbiopsy absorbant que dans des échantillons de peau microbiopsy. Ce résultat suggère que la microbiopsy absorbant performants dans la collecte de sang mais toujours maintenu la capacité pour capturer la peau par rapport à la microbiopsy de la peau (n = 5).
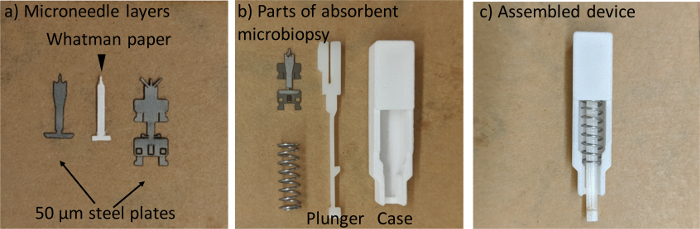
Figure 1. La fabrication des absorbant microbiopsy. la micro aiguille trois couches. (b) toutes les pièces de l’appareil. (c) la microbiopsy absorbant assemblé. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
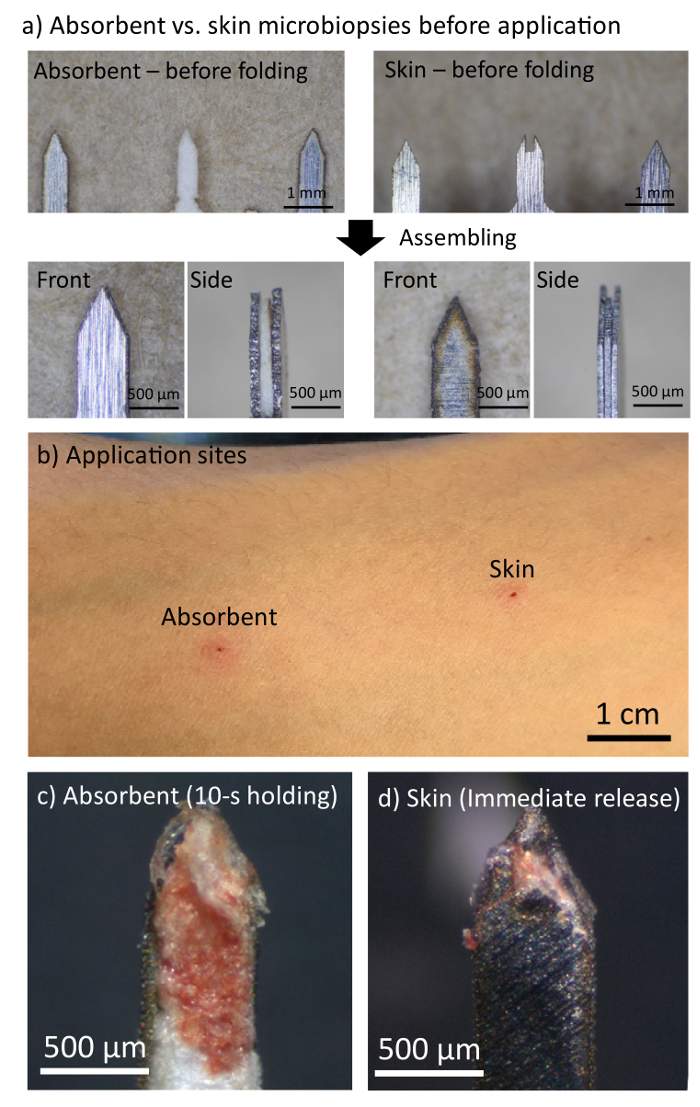
Figure 2. Le microbiopsy absorbant a été capable de capturer des échantillons de peau et le sang en même temps sans laisser une cicatrice sur le bras gauche volar des hommes bénévoles. une comparaison entre les micro-aiguilles de microbiopsies absorbante et de la peau. (b) l’application sites laissés par les microbiopsies absorbant et peau 5 min après l’application. (c, d) Micro-aiguilles de microbiopsies absorbante et de la peau après l’application. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
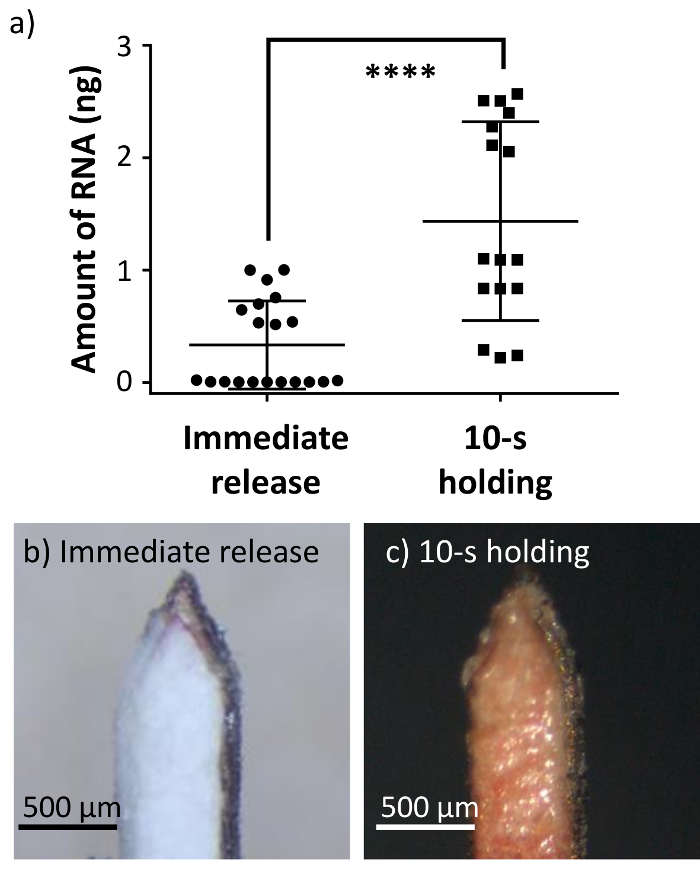
Figure 3. L’absorbant microbiopsy capturé plus de sang et d’échantillon de RNA lorsqu’il est appliqué pour 10-s. (a) le temps de 10 s après l’application retenue correspondait à un montant plus élevé de l’ARN que libérer l’appareil immédiatement (n = 20). Barres d’erreur représentent S.D. moyen (***p< 0,0001). (b, c) Photos représentatives de l’absorbant microbiopsies après application avec « Libération immédiate » et tenue 10-s' approches. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
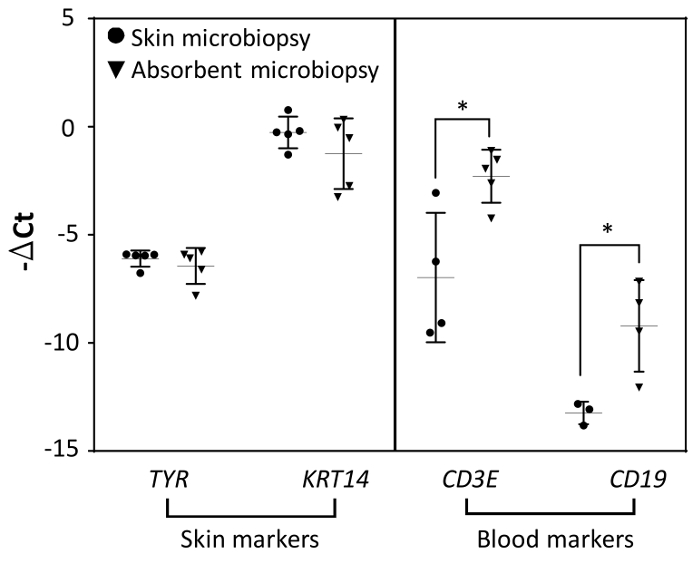
Figure 4. La comparaison des ARNm de l’expression entre la peau et microbiopsies absorbant (n = 5). L’expression des gènes a été normalisée avec gène de référence RPLP0. Barres d’erreur représentent S.D. de la moyenne (*p< 0,05). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Discussion
Ces résultats démontrent que la microbiopsy absorbante peut servir comme outil d’échantillonnage simple et peu invasive du mélange de sang et la peau pour la caractérisation moléculaire. Application du dispositif conformément à notre protocole est indispensable à l’obtention des résultats fiables, comme en témoigne la différence de montant de RNA récupérée à l’aide de protocoles d’application différentes (Figure 3). Une fois que l’échantillon est extrait, l’échantillon suivant traitement pas pour l’extraction de l’ARN est très similaire aux protocoles établis15,16. En outre, depuis les étapes initiales d’extraction d’ARN qui sont modifiés pour la microbiopsy absorbant, un autre changement clé est l’utilisation d’eau exempte de RNase pour améliorer les résultats pour les applications en aval. En outre, il est à noter que le gène de référence utilisé dans cette étude est RPLP0. RPLP0, dont la fonction est bien connue pour les différentes cellules et tissus types17, a été signalé comme un gène de référence approprié pour une utilisation dans les cancers cutanés non mélanocytaires et lésions précancéreuses18.
Une des limites principales de l’appareil est la suppression de la microbiopsy de l’appareil, ce qui prend du temps et augmente potentiellement le risque de perte d’échantillon, en particulier pour les échantillons sensibles comme RNA. Néanmoins, le problème peut être surmonté en refroidissement préalable de tous les outils de traitement stérile, tels que les tubes de microcentrifuge 2 mL, sur la glace sèche.
L’utilisation de la microbiopsy absorbant est simple et ne nécessite pas de formation intensive. Biopsie conventionnelle n’est pas nécessaire que microbiopsy permet la caractérisation moléculaire avec des techniques de diagnostic moléculaire établi, comme la RT-qPCR. Pour quantifier et démontrer la capacité de prélèvement de microbiopsy absorbant, littérature qui implique l’extraction de l’ARN du tissu de la peau humaine a été étudiée. Une peau de mm typique 3.0 ou 4.0 punch biopsie, trois études ont signalé les montants de RNA extraction variait de 50 à 200 ng par mg de tissu de peau19,20,21. En comparant avec les 1,43 ng d’ARN qui a été récupéré de l’absorbant microbiopsy en moyenne (Figure 3), le poids des tissus cutanés échantillonnée devrait se situer de 0,29 à 115 µg basé sur le même rapport de RNA-à-tissu provenant d’études de peau punch biopsie.
Ce protocole n’est pas sans écueils potentiels, bien que certains problèmes peuvent être facilement surmontés. Par exemple, les données d’extraction ARN a suggéré une variation considérable même avec le temps de 10 s retenue (Figure 3). Pour résoudre le problème, une solution consiste à optimiser le protocole d’application. Paramètres tels que la force appliquent sur la peau et temps de maintien doit être testé et optimisé pour réduire les variations dans l’extraction de l’échantillon. L’autre problème potentiel est l’élimination des microbiopsy de l’appareil, qui peut-être nuire à l’intégrité de l’échantillon et la récupération. Bien que le retrait de la microbiopsy pour l’extraction de l’ARN est une approche efficace, l’ensemble de la procédure est fastidieuse, et l’échantillon pourrait être exposé à la contamination dans le processus. Par conséquent, la modification du protocole de traitement échantillon est hautement souhaitée afin de garantir l’intégrité de l’échantillon et de prévenir la perte de l’échantillon.
Une fois que les deux questions ci-dessus sont abordées, il est prévu que l’appareil deviendra un outil standard pour la recherche clinique. Il est important de noter que l’appareil capte échantillon de peau et le sang en même temps et cela devrait prendre en considération lors de l’analyse des données d’expression de gène. En conclusion, ce protocole décrit les détails de l’exécution de microbiopsy absorbant comme un outil simple et peu invasif pour peau combinée et prélèvements sanguins et l’échantillon suivant de traitement pour le profil d’expression génique relatif.
Déclarations de divulgation
Aucun conflit d’intérêts ne déclarés.
Remerciements
Nous tenons à remercier NHMRC bourses APP1109749 et APP1111216, University of Queensland Centennial Scholarship et bourse de recherche postdoctorale International.
matériels
| Name | Company | Catalog Number | Comments |
| Absorbent microbiopsy fabrication and sampling | |||
| Absorbent Microbiopsy | Trajan Scientific and Medical | N/A | https://www.trajanscimed.com/ |
| Whatman filter paper, Grade 1 | Sigma Aldrich | WHA1001325 | N/A |
| RNA extraction | |||
| PicoPure RNA Isolation Kit | ThermoFisher | KIT0214 | Including all buffer soltuions described in protocol |
| UltraPure DNase/RNase-Free Distilled Water | ThermoFisher | 10977015 | Improving RNA quality in RNA elution step |
| 2.0 mL Microcentrifuge Tube, Sterile | Thomas Scientific | 1226S74 | N/A |
| Microcentrifuge 5415R | Eppendorf | Z605212 | N/A |
| cDNA synthesis | |||
| SensiFAST cDNA Synthesis Kit | Bioline | BIO-65053 | N/A |
| CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855196 | N/A |
| qPCR reaction and data analysis | |||
| SensiFAST SYBR Lo-ROX Kit | Bioline | BIO-94005 | Including reagents described in qPCR reaction steps |
| MicroAmp Optical Adhesive Film | ThermoFisher Scinentific | 4311971 | N/A |
| MicroAmp Optical 384-Well Reaction Plate | ThermoFisher Scinentific | 4343370 | N/A |
| QuantStudio 6 Flex Real-Time PCR System | ThermoFisher Scinentific | 4485694 | N/A |
| GraphPad Prism (v6.04) | GraphPad | N/A; Windows version | Plotting and statistical analysis in qPCR data analysis steps |
| PCR primers | |||
| RPLP0 F | Sigma Aldrich | N/A | ATC AAC GGG TAC AAA CGA GTC |
| RPLP0 R | Sigma Aldrich | N/A | CAG ATG GAT CAG CCA AGA AGG |
| TYR F | Sigma Aldrich | N/A | TCA GCA CCC CAC AAA TCC TAA |
| TYR R | Sigma Aldrich | N/A | AAT CGG CTA CAG ACA ATC TGC |
| KRT14 F | Sigma Aldrich | N/A | CCT CCT CCC AGT TCT CCT |
| KRT14 R | Sigma Aldrich | N/A | ACA CCA CCT TGC CAT CG |
| CD3E F | Sigma Aldrich | N/A | CAA AGG GGA CAA AAC AAG GAG |
| CD3E R | Sigma Aldrich | N/A | GTT CTC CAG AGG GTC AGA TG |
| CD19 F | Sigma Aldrich | N/A | TTC TGC CTG TGT TCC CTT G |
| CD19 R | Sigma Aldrich | N/A | GCG TCA CTT TGA AGA ATC TCC T |
Références
- Wang, C. Y., Maibach, H. I. Why minimally invasive skin sampling techniques? A bright scientific future. Cutaneous and Ocular Toxicology. 30, 1-6 (2011).
- Kirstein, O. D., et al. Minimally invasive microbiopsies: a novel sampling method for identifying asymptomatic, potentially infectious carriers of Leishmania donovani. International journal for parasitology. 47, 609-616 (2017).
- Lin, L. L. The skin microbiopsy. , The University of Queensland. (2015).
- Dhingra, N., et al. Molecular profiling of contact dermatitis skin identifies allergen-dependent differences in immune response. Journal of Allergy and Clinical Immunology. 134, 362-372 (2014).
- Chapman, P. B., et al. Improved Survival with Vemurafenib in Melanoma with BRAF V600E Mutation. New England Journal of Medicine. 364, 2507-2516 (2011).
- Kirstein, O. D., et al. Minimally invasive microbiopsies: a novel sampling method for identifying asymptomatic, potentially infectious carriers of Leishmania donovani. International journal for parasitology. 47, 609-616 (2017).
- Lin, L. L., et al. Microbiopsy engineered for minimally invasive and suture-free sub-millimetre skin sampling. F1000Research. 2, (2013).
- Tom, L. N., et al. Skin microbiopsy for HPV DNA detection in cutaneous warts. Journal of the European Academy of Dermatology and Venereology. 30, e216-e217 (2016).
- Sobarun, P., et al. Microbiopsy Biomarker Profiling in a Superficial Melanoma Resembling a Pigmented Basal Cell Carcinoma. JAMA Dermatology. 153, 334(2017).
- Banan, P., Lin, L. L., Lambie, D., Prow, T., Soyer, H. P. Effects of Ex Vivo Skin Microbiopsy on Histopathologic Diagnosis in Melanocytic Skin Lesions. JAMA Dermatology. 149, 1107(2013).
- GraphPad, GraphPad - FAQ 1753 - Prism 3 -- Calculating "Unknown" Concentrations using a Standard Curve. , Available from: https://www.graphpad.com/support/faq/prism-3-calculating-unknown-concentrations-using-a-standard-curve/ (2018).
- Kubista, M., et al. The real-time polymerase chain reaction. Molecular Aspects of Medicine. 27, 95-125 (2006).
- Goni, R., García, P., Foissac, S. The qPCR data statistical analysis. , 1-9 (2009).
- GraphPad Statistics Guide. , (1995).
- Goldstein, N. B., et al. Isolating RNA from precursor and mature melanocytes from human vitiligo and normal skin using laser capture microdissection. Experimental dermatology. 25, 805-811 (2016).
- Shearstone, J. R., Allaire, N. E., Campos-Rivera, J., Rao, S., Perrin, S. Accurate and precise transcriptional profiles from 50 pg of total RNA or 100 flow-sorted primary lymphocytes. Genomics. 88, 111-121 (2006).
- Bär, M., Bär, D., Lehmann, B. Selection and Validation of Candidate Housekeeping Genes for Studies of Human Keratinocytes-Review and Recommendations. Journal of Investigative Dermatology. 129, 535-537 (2009).
- Hoang, V. L. T., et al. RNA-seq reveals more consistent reference genes for gene expression studies in human non-melanoma skin cancers. PeerJ. 5, e3631(2017).
- Döbbeling, U. Simultaneous RNA and DNA Extraction from Biopsy Material, Culture Cells, Plants, and Bacteria. Nucleic Acid Protocols Handbook, The. , Humana Press. 53-56 (2000).
- Bruning, O., et al. RNA isolation for transcriptomics of human and mouse small skin biopsies. BMC Research Notes. 4, 438(2011).
- Berglund, S. R., et al. Optimized Methodology for Sequential Extraction of RNA and Protein from Small Human Skin Biopsies. Journal of Investigative Dermatology. 127, 349-353 (2007).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon