Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Multiplexage et transfection d'ADN à haut débit à l'aide de la technologie de nanodispensation acoustique
Dans cet article
Résumé
Ce protocole décrit la transfection plasmique à haut débit des cellules de mammifères dans une plaque de 384 puits utilisant la technologie acoustique d'éjection de gouttelettes. La distribution et le multiplexage d'ADN, longs et sujets aux erreurs, mais aussi la distribution de réactifs transfection, sont pilotés par un logiciel et exécutés par un dispositif de nanodistributeur. Les cellules sont ensuite ensecées dans ces puits préremplis.
Résumé
La transfection cellulaire, indispensable pour de nombreuses études biologiques, nécessite le contrôle de nombreux paramètres pour une réalisation précise et réussie. Le plus souvent effectué à faible débit, il est d'ailleurs long et sujet aux erreurs, d'autant plus lorsque le multiplexage de plusieurs plasmides. Nous avons développé une méthode facile, rapide et précise pour effectuer la transfection cellulaire dans une mise en page de plaque de 384 puits en utilisant la technologie acoustique d'éjection de gouttelettes (ADE). Le dispositif nanodistributeur utilisé dans cette étude est basé sur cette technologie et permet une livraison précise de nanovolume à grande vitesse à partir d'une plaque de puits source à une plaque de destination. Il peut distribuer et multiplex ADN et réactif de transfection selon une feuille de calcul préconçue. Ici, nous présentons un protocole optimal pour effectuer la transfection plasmide à haut débit à base d'ADE qui permet d'atteindre une efficacité allant jusqu'à 90% et une cotransfection de près de 100% dans les expériences de cotransfection. Nous étendons le travail initial en proposant une macro basée sur une feuille de calcul conviviale, capable de gérer jusqu'à quatre plasmides/puits à partir d'une bibliothèque contenant jusqu'à 1 536 plasmides différents, et une application de guide de pipetage à base de tablettes. La macro conçoit le modèle (s) nécessaire de la plaque source et génère les fichiers prêts à l'emploi pour l'application nanodispenser et tablette. Le protocole de transfection en quatre étapes implique i) un diluant se passer d'un manipulateur liquide classique, ii) la distribution plasmide et le multiplexage, iii) un réactif de transfection distribué par le nanodispenser, et iv) le placage cellulaire sur les puits préremplis. Le contrôle logiciel décrit du multiplexage et de la transfection du plasmide ADE permet même aux non-spécialistes sur le terrain d'effectuer une transfection cellulaire fiable d'une manière rapide et sûre. Cette méthode permet d'identifier rapidement les paramètres optimaux pour un type de cellule donné et peut être transposée à des approches manuelles et à échelle supérieure. Le protocole facilite les applications, telles que la protéine ORFeome humaine (ensemble de cadres de lecture ouverts [ORFs] dans un génome) expression ou CRISPR-Cas9-basé validation de la fonction génique, dans les stratégies de dépistage non pooled.
Introduction
La méthode présentée ici décrit en détail comment effectuer le multiplexage et la transfection du plasmide de l'ADN dans les cellules de mammifères à haut débit à l'aide d'un nanodistributeur liquide à base acoustique dans une plaque de 384 puits, même pour les non-spécialistes dans le domaine. Cette méthode1 récemment publiée permet d'exécuter jusqu'à 384 multiplesx d'ADN plasmide indépendants et des conditions de transfection dans une expérience, en moins de 1 h. Les expériences simples ou de cotransfection ont été couronnées de succès, atteignant près de 100 % cotransfection au sein de la population de cellules transfectées. Ce protocole facilite la transfection parce que la plupart des étapes fastidieuses, longues et sujettes aux erreurs sont maintenant pilotées par logiciel (voir la figure 1 pour un aperçu général). D'autres efforts ont été faits pour développer des outils dédiés pour améliorer la facilité d'utilisation tout en évitant les erreurs humaines au cours du processus global et pour promouvoir la transfection réussie, même pour les non-spécialistes dans le domaine. Le protocole décrit comprend une feuille de calcul macro « conviviale » que nous avons développée afin de gérer 384 conditions de transfection indépendantes avec des possibilités de multiplexage allant jusqu'à quatre plasmides dans chaque puits. La macro génère automatiquement des modèles de la plaque source (s) pour charger le volume plasmide de l'ADN prévu à partir de solutions de stock de départ et les fichiers nécessaires pour conduire le logiciel nanodispenser sur la conception expérimentale qui a été entré. Comme la distribution manuelle de l'ADN dans une plaque source de 384 puits est fastidieuse et sujette aux erreurs, nous avons également développé une application dédiée à la tablette pour guider l'utilisateur tout en distribuant une solution d'ADN selon le modèle.
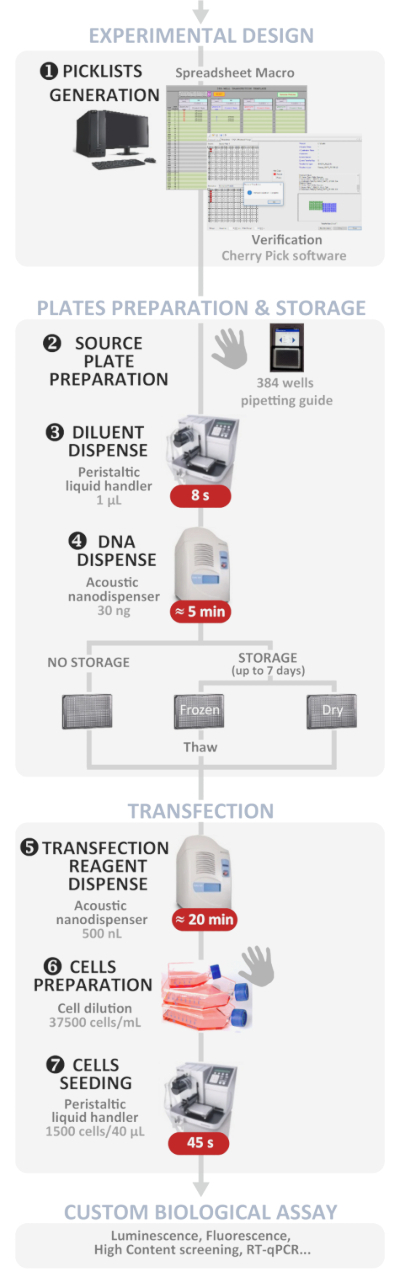
Figure 1 : Flux de travail expérimental. Représentation schématique du protocole de transfection inverse automatisé automatisé optimal (de la conception expérimentale à l'analyse biologique personnalisée). Les étapes manuelles sont indiquées par le symbole de la main et le temps approximatif de chaque étape est écrit dans une boîte rouge. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
Beaucoup d'expériences cellulaires commencent par la transfection d'ADN plasmique, et même si de nombreux réactifs dédiés ont été et sont encore en cours de développement pour améliorer l'efficacité de la transfection et / ou faciliter la procédure, beaucoup reste à faire2,3 , 4. la transfection de cellules plasmiques d'ADN implique plusieurs étapes pour atteindre l'efficacité élevée, telle qu'une prise complexe initiale, l'évasion endosomal, et le transport cytoplasmique au noyau5,6. En plus de la précipitation de calcium ou des techniques physiques telles que l'électroporation ou la microinjection à l'aide d'appareils dédiés7, les méthodes chimiques modernes se sont concentrées sur l'amélioration de la livraison des cellules d'ADN tout en abaissant la cytoxicité cellulaire8, 9. L'utilisation de lipides ou de polymères cationiques formant des complexes liposome-like et, plus récemment, des systèmes de chimie polymère nonliposomal a rendu la transfection plus facile et plus efficace10. Malgré ces développements, la transfection cellulaire nécessite toujours des compétences spécifiques à effectuer avec précision que la plupart de ces protocoles de transfection physique ou chimique exigent des scientifiques de préparer manuellement chaque condition de réaction de transfection de l'ADN, donc altérer le débit. Pour contourner ce problème, des protocoles de transfection inverse ont été développés à l'aide de réactifs chimiques de transfection11,12,13, permettant à l'utilisateur de tester ou de combiner plusieurs plasmides d'une manière plus rapide. Dans ces protocoles, des complexes d'acide nucléique avec des réactifs de transfection sont formés avant d'ensemencer les cellules sur les complexes. Cependant, ces protocoles inverses sont encore limités par la manipulation manuelle des solutions d'ADN et par la combinaison de chacune des conditions indépendantes. Bien qu'il soit possible de les exécuter dans un format de plaque de 96 puits, la préparation de l'ADN et les dispenses seront fastidieuses, et il y aura probablement des erreurs. Lorsque différentes quantités de plusieurs plasmides d'ADN sont nécessaires et multiplexées les unes avec les autres, la transfection cellulaire devient encore plus difficile à réaliser et plus de temps, et les erreurs humaines deviennent tout à fait inévitables. L'extension jusqu'au format de plaque de 384 puits dans une approche de transfection inverse, en dépit de quelques conditions de transfection multiplexed d'ADN, devient un défi impossible en raison des raisons suivantes. i) Les quantités d'ADN, de réactif de transfection ou de mélange de réaction à gérer sont inférieures à 1 L pour chaque puits. ii) Le multiplexage des plasmides pour 384 conditions indépendantes devient extrêmement compliqué. La livraison dans chacun des 384 puits est également iii) très longue et iv) sujette aux erreurs. En effet, la distribution de la bonne solution dans les puits attendus est difficile à gérer car les faibles volumes déjà distribués ne permettent pas de surveiller visuellement entre les puits vides et déjà remplis. v) Enfin, il y a un risque élevé de séchage du mélange par évaporation avant l'ajout des cellules en raison du temps nécessaire pour effectuer les étapes de distribution nécessaires. En résumé, le facteur limitant pour mettre en place des tests de transfection de plasmide d'ADN à haut débit semble être la miniaturisation de l'analyse, ce qui implique un multiplexage et une gestion à faible volume qui ne peuvent plus être manipulés manuellement, mais qui ne sont pas non plus réalisables dans un manière fiable par les manipulateurs de liquides péristatiques classiques.
Comme preuve de difficulté à automatiser de tels essais et à obtenir un débit élevé, seules quelques tentatives d'automatisation de la transfection ont été publiées jusqu'à présent : un format de plaque de 96 puits à l'aide d'un dispositif commercial de manutention des liquides et des précipitations de phosphate de calcium14. et, plus récemment, un réactif lipoplex, et une puce microfluidique permettant 280 transfections indépendantes15, mais nécessitant des compétences spécialisées dans ce domaine. Une autre méthode, l'acoustophoresis, permettant la lévitation liquide et conduisant à la manipulation et au mélange de fluides, a été employée pour effectuer la transfection d'ADN dans les formats de plaque de 24 à 96 puits16. Bien que faisable, cette approche souffre d'un débit extrêmement faible car le mélange des cellules avec le mélange de transfection d'ADN nécessite une incubation de 60 s pour chaque point avant l'ensemencement. Cela implique une durée d'au moins 96 min pour une plaque complète de 96 puits. En outre, ce protocole est loin d'être favorable à l'audience globale des biologistes puisque ce travail a été fait avec un dispositif interne conçu et fabriqué qui n'est actuellement pas disponible sur le marché. Au contraire, au cours des dernières années, une technologie de distribution acoustique facile à utiliser par logiciel a vu le jour avec des distributeurs de nanovolume. Utilisant l'énergie acoustique focalisée, ces dispositifs permettent l'éjection étroitement contrôlée de petits volumes liquides de 2.5 nL à 500 nL d'une plaque source à une destination17. Cette technologie, appelée éjection acoustique de gouttelettes (ADE), présente de nombreux avantages : elle est entièrement automatisée, sans contact, sans pointe, précise, précise et hautement reproductible, et elle a un débit élevé18. D'abord consacré à la livraison de solutions de sulfoxide de diméthyle (DMSO), les paramètres ont été améliorés pour distribuer des tampons aqueux19. Les nanodistributeurs acoustiques semblent donc adaptés aux protocoles de transfection des cellules inversées et pourraient contourner la plupart des limitations manuelles mentionnées ci-dessus. Comme aucune tentative de transfection plasmique n'a été précédemment décrite à l'aide de cette technologie, nous avons récemment évalué la pertinence d'un système de distribution acoustique pour effectuer la transfection des cellules inversées.
Profitant du débit nanodispenser et de la facilité d'utilisation, nous avons optimisé un protocole de transfection inverse pour les cellules HeLa en testant plusieurs paramètres qui peuvent influencer la transfection de l'ADN sur une plaque unique de 384 puits, à savoir la quantité totale d'ADN et concentration de départ de l'ADN source, volume diluant, réactif de transfection, et nombre de cellules de propagation. Le protocole développé contourne les limites manuelles décrites ci-dessus de la transfection cellulaire et présente plusieurs avantages par rapport à d'autres tentatives de transfection automatisées. Tout d'abord, il est miniaturisé, permettant ainsi un réactif de transfection rentable en économisant les préparations de plasmide d'ADN et le réactif de transfection. Deuxièmement, il est beaucoup plus haut débit et reproductible que le protocole manuel (même pour les débutants), comme la transfection d'une plaque entière de 384 puits peut être atteint en moins de 1 h. Enfin, il est piloté par un logiciel, permettant le contrôle de la quantité d'ADN distribué et le multiplexage de plusieurs plasmides. En effet, grâce au logiciel nanodispenser (Tableaudes Matériaux),l'utilisateur peut élaborer un plan d'étude pour contrôler les volumes à distribuer à partir d'une plaque de puits de source définie à une plaque de destination.
Le protocole présenté ici est principalement destiné à ceux qui ont accès à un nanodispenser et qui souhaitent mettre en place des expériences de transfection à haut débit, mais aussi pour ceux qui veulent optimiser rapidement leurs paramètres de transfection pour un type de cellule donné par l'application de ce protocole pour contre-vérifier plusieurs paramètres à haut débit. En effet, nous avons montré que les paramètres optimisés identifiés avec ce protocole à l'échelle nanométrique peuvent être transposés à des expériences de transfection manuelles et à plus grande échelle. Enfin, comme le réactif de transfection utilisé dans le protocole actuel permet la transfection d'ADN ou de siRNA selon le fabricant, le protocole est également d'intérêt pour ceux qui visent à effectuer des approches de tableau pour la surexpression ou le knockdown de gène. Les plaques de destination préremplies d'ADN peuvent être conservées jusqu'à 7 jours avant utilisation dans un test de transfection sans perte d'efficacité, ce qui est un autre avantage du protocole suivant pour ce type d'application.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. Préparations préalables
- Préparation des programmes de manutention de liquides péristaltiques
REMARQUE : Pour les étapes de diluant et de distribution cellulaire du protocole, un programme dédié doit être préparé, en tenant compte de la hauteur de la tête distributrice à la plaque utilisée et de l'intention d'étape.- Pour l'étape de distribution de diluant de 1 l,l, montez une cassette de 1 L et préparez un programme avec les paramètres décrits dans les étapes 1.1.1.1 et 1.1.1.2.
- Ajustez le paramètre de débit à Haut pour le meilleur débit car aucun dommage matériel biologique n'est prévu dans cette étape. Ajuster la hauteur de distribution à 9,6 mm (selon la plaque de culture cellulaire utilisée, Figure supplémentaire 1) pour permettre à la goutte de 1 L de toucher le fond des puits pendant la dispensation.
REMARQUE : Cette étape est cruciale pour éviter la rétention des gouttelettes sur la tête de distribution jusqu'à ce qu'elles atteignent un volume suffisant pour tomber. - Ajuster la hauteur claire de la plaque à 14,4 mm pour permettre un déplacement libre de la tête de distribution sur la plaque après la distribution de chaque rangée. Contrôlez visuellement les réglages appropriés de la hauteur de la tête du gestionnaire de liquide péristaltique : assurez-vous qu'aucune goutte n'est retenue sur les extrémités de distribution pendant la distribution et vérifiez que la tête est assez haute pour permettre le déplacement de la tête après distribution de chaque rangée.
REMARQUE : Éviter la rétention des gouttes est un paramètre crucial car il nuira à l'exactitude du volume de la dispensation.
- Ajustez le paramètre de débit à Haut pour le meilleur débit car aucun dommage matériel biologique n'est prévu dans cette étape. Ajuster la hauteur de distribution à 9,6 mm (selon la plaque de culture cellulaire utilisée, Figure supplémentaire 1) pour permettre à la goutte de 1 L de toucher le fond des puits pendant la dispensation.
- Pour la distribution de la suspension cellulaire de 40 l, montez une cassette de 10 L et préparez un programme avec les paramètres décrits dans les étapes 1.1.2.1-1.1.2.2.
- Ajustez le paramètre de débit à Low pour distribuer les cellules à basse vitesse afin d'éviter de favoriser des dommages potentiels aux cellules par le stress de cisaillement et l'impact élevé sur le fond des puits. Ajuster la hauteur de dispensation à 11,43 mm (selon la plaque de culture cellulaire utilisée, Figure supplémentaire 1), assez élevée pour abaisser l'impact cellulaire sur le fond des puits pendant le processus de distribution, mais assez faible pour éviter la rétention des gouttelettes sur le distribuer la tête. Ajuster la hauteur claire de la plaque à 16 mm pour permettre le déplacement libre de la tête de distribution sur la plaque après distribution de chaque rangée.
- Contrôlez visuellement les réglages appropriés de la hauteur de la tête du gestionnaire de liquide péristaltique : assurez-vous qu'aucune goutte n'est retenue sur les extrémités de distribution pendant la distribution et vérifiez que la tête est assez haute pour permettre le déplacement de la tête après distribution de chaque rangée.
REMARQUE : Éviter la rétention des gouttes est un paramètre crucial car il conduira à la distribution d'un numéro de cellule peu fiable.
- Pour l'étape de distribution de diluant de 1 l,l, montez une cassette de 1 L et préparez un programme avec les paramètres décrits dans les étapes 1.1.1.1 et 1.1.1.2.
- Préparation de plasmide d'ADN (protocole classique d'extraction de miniprep)
- Cultivez une souche de bactéries DH5MD transformée dans le milieu LB complétée par un antibiotique de sélection d'ampicilline de 125 g/mL (Tableau des matériaux) pendant la nuit à 37 oC et sous une douce agitation (200 tr/min) sur un shaker orbital (Tableaudes matériaux).
- Récoltez 2 ml de la culture, pelletez les cellules en centrifugeant pendant 5 min à 6 000 x g,et jetez le supernatant.
- Resuspendre le granule cellulaire avec 250 l de tampon de résuspension contenant RNase A (Tableau des matériaux). Ajouter 250 l de tampon de lyse et couver pendant 5 minutes à température ambiante, selon les instructions du fabricant.
- Arrêtez la réaction de lyse en ajoutant 300 l de tampon de neutralisation (Tableau des matériaux) et en vortexant sous peu. Centrifuger les tubes pendant 5 min à 11 000 x g.
- Placer une nouvelle minicolonne plasmide (Table of Materials) dans un tube de collecte de 2 ml et décanter le supernatant dans la colonne en centrifuge pendant 1 min à 11 000 x g.
- Jetez le flux à travers et placez la minicolonne de nouveau dans le tube de collecte.
- Laver la minicolonne plasmide avec 500 l de tampon de lavage optionnel (Table of Materials) et centrifugeuse pendant 1 min à 11 000 x g,selon les instructions du fabricant.
- Jetez le flux à travers et placez la minicolonne plasmide dans le tube de collecte.
- Ajouter 700 l de tampon de lavage (Tableaudes matériaux)complété avec de l'éthanol et centrifugeuse pendant 1 min à 11 000 x g,selon les instructions du fabricant.
- Jetez l'écoulement et la centrifugeuse de la minicolonne plasmide et de son tube de collecte 1x de plus pendant 2 min à 11 000 x g pour sécher la membrane de silice.
- Placer la mini-colonne plasmide séchée dans un nouveau tube de 1,5 ml et ajouter 30 l d'eau distillée préchauffée à 60 oC, l'incuber pendant 2 min à température ambiante, puis la centrifuger pendant 1 min à 11 000 x g.
- Jetez la minicolonne plasmide et gardez l'éluate contenant le plasmide purifié de l'ADN.
- Mesurer la concentration d'ADN de l'ADN élugé à l'aide d'un spectrophotomètre de microvolume (Tableau des matériaux).
- Allumez le spectrophotomètre et choisissez les paramètres de mesure de l'ADN.
- Soulevez le bras d'échantillonnage du spectrophotomètre et de la pipette 1 L d'eau sur le piédestal de mesure pour effectuer un étalonnage vierge.
- Abaissez le bras d'échantillonnage, commencez la mesure vierge et attendez d'être terminé.
- Soulevez le bras d'échantillonnage et essuyez l'échantillon des piédestaux supérieurs et inférieurs.
- Pipette 1 L de la solution d'ADN sur le piédestal inférieur pour le mesurer.
- Abaissez le bras d'échantillonnage, commencez la mesure de la concentration d'ADN et attendez d'être terminé.
- Soulevez le bras d'échantillonnage et essuyez l'échantillon des piédestaux supérieurs et inférieurs.
- Pour d'autres mesures de concentration d'ADN, répétez les étapes 1.2.13.5-1.2.13.7.
- Une fois les mesures terminées, entreposez les solutions d'ADN à 4 oC jusqu'à l'utilisation.
2. Conception expérimentale et génération des listes de sélection pour conduire les dispenses à base d'ADE
REMARQUE : Une macro de feuille de calcul « conviviale » dédiée a été développée pour gérer les quantités d'ADN et mélanger jusqu'à quatre plasmides dans un format de plaque de 384 puits. Basé sur la conception expérimentale entrée, cette macro génère les fichiers nécessaires pour conduire le protocole de transfection d'ADN basé sur L'ADE par nanodispenser. Afin de générer ces fichiers, plusieurs champs doivent être remplis dans la feuille de modèle comme indiqué dans la figure 2.
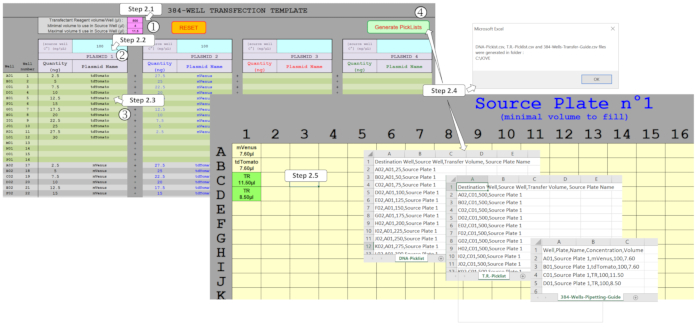
Figure 2 : Génération des listes de sélection pour conduire la dispensation ADE à l'aide de la macro feuille de calcul. Plusieurs paramètres doivent être comblés, à savoir (1) le réactif de transfection (TR) et les volumes minimes/maximaux à utiliser dans la plaque source, (2) les concentrations plasmides initiales à distribuer dans la plaque source, et (3) le conception de la plaque entière, y compris les quantités de plasmides attendues et le multiplexage dans chacun des 384 puits. (4) L'activation des listes de sélection génère permet de vérifier les différents champs et, une fois correctement remplis, des listes de sélection pour la distribution d'ADN et de TR et le modèle de plaque source nécessaire sont automatiquement générés. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
- Entrez les paramètres du protocole nanodispenser dans les champs roses. Définir la valeur du mélange de réactif de transfection (TR) à 500 nL. Définir la valeur de volume minimale dans les puits de plaque source à 4 l. Définir le volume maximal dans les puits de plaque source à 11,25 l.
REMARQUE: Le nanodispenser utilisé ici ne peut transférer un maximum de 500 nL en une seule course de l'ADE. Ces champs roses sont préremplis avec les valeurs recommandées mais peuvent être modifiés en fonction des besoins de l'utilisateur. - Entrez des concentrations de 100 ng/L d'ADN de départ dans les champs bleus correspondant à l'ADN sous-jacent.
REMARQUE : Cette valeur est la concentration optimale précédemment définie mais peut cependant être modifiée pour différents besoins de l'utilisateur. - Entrez la quantité d'ADN désirée dans les champs gris/verts. Entrez les montants et les noms plasmides pour les 384 puits, assurant la même orthographe si le même plasmide est utilisé dans plusieurs puits.
- Générez la conception de la plaque source, les fichiers picklists et le fichier guide de pipetage. Cliquez sur Générer des listes de sélection pour permettre à la macro de générer l'ADN-Picklist.csv, le T.R.-Picklist.csv et le 384-Wells-Pipetting-Guide.csv fichiers à partir des données recueillies sur la feuille correspondante. Si demandé, corrigez les valeurs de cellules remplies d'orange car elles indiquent les erreurs ou les volumes qui ne peuvent pas être manipulés par le nanodistributeur.
- Imprimez le modèle (s) à partir de la feuille de plaque source. Les noms de plasmides et le volume minimal pour remplir les puits sont indiqués. De même, les volumes de mélange de réactifs transfection qui devront ensuite être remplis dans les puits suivants sont indiqués comme TR et mis en évidence en vert.
3. Préparation de la plaque source d'ADN à l'aide de l'application de guide de tuyauterie de 384 puits
- Diluer le plasmide d'ADN stocké de l'étape 1.2.14 à 100 ng/L à l'aide d'eau distillée.
- Calibrer la grille de 384 puits aux dimensions de la plaque : ouvrez l'application de guide de tuyauterie de 384 puits sur une tablette (Figure 3). Placez la plaque source sur la grille sur l'écran inférieur, et dans le menu d'étalonnage supérieur gauche, cliquez sur ou - ou utilisez le curseur rouge) pour améliorer ou réduire la taille de la grille et des puits afin d'ajuster les puits verts aux quatre puits d'angle de la plaque .

Figure 3 : Utilisation de l'application de guide de tuyauterie de 384 puits. (1) Calibration de la grille de 384 puits à la taille de la plaque; (2) ) Mont d'un adaptateur universel de plaque imprimé en 3D sur la tablette à l'aide de ruban à double face; (3) Placement de la plaque sur l'adaptateur; (4) Déplacement de la grille pour la centrer sur la plaque montée. (5) Verrouillage de l'étape d'étalonnage. (6) Ouverture du fichier 384 puits pipetting guide.csv. (7) Compte tenu de la liste des fichiers, l'application indiquera le nom de la plaque source prévue, le réactif (ADN ou réactif de transfection), la concentration et le volume à distribuer dans les puits cibles, qui seront éclairés un par un. (8) Boutons de flèche gauche et droite permettent à l'utilisateur de suivre le guide de pipetage pour distribuer facilement les réactifs selon le modèle de plaque macro-source de feuille de calcul. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
- À l'aide d'un ruban à double face, montez l'adaptateur de plaque imprimé en 3D sur l'écran pour éviter les mouvements de plaque source pendant la distribution. Si nécessaire, déplacez la grille calibrée à l'aide des flèches de rotation et des boutons Up/ Down/Right/Left pour ajuster la grille de l'écran à la position de la plaque. Une fois que la grille et la taille des puits sont correctement calibrées et localisées, cochez la case d'étalonnage de l'écluse.
- Cliquez sur FILE et ouvrez le fichier 384 wells pipetting guide.csv. Suivez les instructions de l'écran pour distribuer manuellement le volume indiqué du plasmide indiqué à la concentration indiquée dans le puits de surbrillance blanc correspondant à la destination cible appropriée de la plaque prévue. Utilisez - ou - flèches pour revenir en arrière ou plus loin dans le processus de distribution d'ADN. Cessez de distribuer lorsque vous atteignez la première solution de réactif Transfection à charger.
- Une fois les dispenses d'ADN terminées, retirez la plaque source de l'adaptateur. Si plusieurs plaques sources doivent être remplies, placez une nouvelle plaque source sur l'adaptateur et suivez les instructions de distribution. Une fois la dispensation d'ADN terminée, centrifuger la plaque source remplie d'ADN (à 1 500 x g pendant 2 min) pour assurer un nivellement liquide adéquat et pour éliminer les bulles conduisant à une inexactitude dans les transferts à base d'ADE.
4. Distribution de diluant à base de liquide peristaltique 1 -L de diluant dans la plaque de destination
REMARQUE : Effectuer les étapes 4.1-4.5 dans un coffret de sécurité biologique.
- Désinfecter la tête de cassette de 1 l l en la pulvérisant avec un désinfectant par pulvérisation (tableaudes matériaux),et permettre à cette solution d'entrer dans le support de pointe. Absorber le désinfectant restant sur du papier absorbant. Montez la cassette de 1 L sur le dispositif de manutention de liquide péristaltique. Allumez l'appareil et assurez-vous que le réglage du type de cassette est correct (1 l), ainsi que le format de la plaque (384 puits).
- Désinfecter l'ensemble du lumen du tube : insérez l'organisateur du tube (en tenant les huit tubes ensemble) dans un récipient stérile et remplissez-le de 5 ml d'alcool à 70 %. En utilisant la fonction d'amorçage du gestionnaire de liquide péristtaltique, rincer d'abord l'alcool dans le tube, puis le rincer en passant 5 ml d'eau distillée et 5 ml de milieu sans sérum (le milieu modifié de l'aigle de Dulbecco [DMEM] complété par 100 U/mL pénicilline-streptomycine; Table de Matériel), remplissant successivement le même récipient. Assurez-vous qu'aucune des pointes n'est obstruée en inspectant visuellement le flux liquide de chacun d'eux.
- Premier le tube avec milieu sans sérum en remplissant un nouveau navire stérile avec 10 ml de milieu préchauffé sans sérum et la plongée de l'organisateur de tube en elle. Appuyez sur le bouton principal du gestionnaire de liquide péristtique pendant environ 10 s. Encore une fois, assurez-vous qu'aucune pointe n'est obstruée en inspectant visuellement le flux liquide de chacun d'eux.
- Remplir la plaque d'une ll de diluant. Placez une plaque de culture stérile de 384 puits (destination) sur le porte-plaques de manutention de liquide péristaltique et retirez son couvercle.
- Exécuter le programme précalibré pour distribuer 1 L dans chaque puits de la plaque de 384 puits. Le temps de distribution est d'environ 8 s. Remplacez ensuite le couvercle de la plaque de 384 puits.
REMARQUE : Alternativement, cette étape peut être traitée manuellement, dans une armoire de sécurité, à l'aide d'une micropipette multicanal.
5. Exécution d'une enquête pour contrôler les volumes distribués manuellement
REMARQUE : Pour plus de détails, voir Figure 4.
- Exécutez le programme de nanodispenser, allez à l'onglet diagnostique, cochez la plaque source La case d'sortie, chargez la plaque source sur le support de plaque, et cochez dedans pour entrer dans la plaque. Lorsque vous êtes invité, sélectionnez 384LDV-AQ-B2 pour définir le nanodispenser au mode de distribution tampon aqueuse, et appuyez sur Ok.
- Sélectionnez Sondage dans le menu divers et cliquez sur Lancement. Sélectionnez les puits préremplis pour analyser et cliquez sur le bouton Go. Vérifier que les volumes mesurés correspondent aux volumes attendus et s'assurer qu'aucun puits n'a été chargé avec des volumes de plus de 12 L, car cela évitera les transferts.
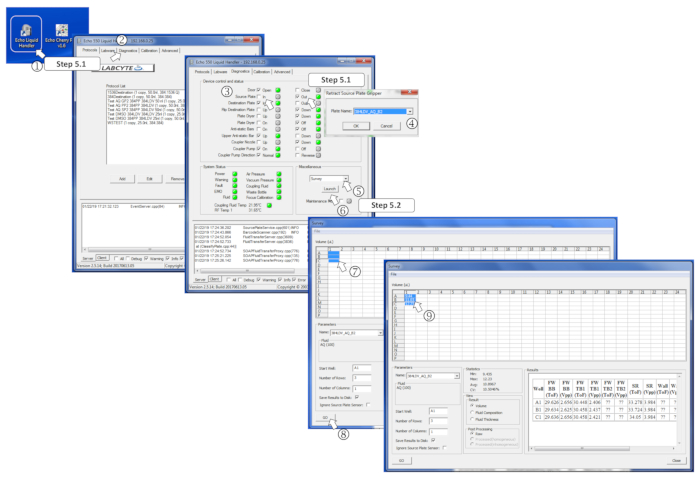
Figure 4 : Définition des paramètres logiciels d'enquête. (1) Démarrer le programme nanodispenser. (2) Ouvrez l'onglet Diagnostics. (3) Insérez la plaque source en cochant pour la plaque source et, ensuite, En. (4) Définir le type de plaque source dans le menu lorsqu'il est invité. (5) Dans la boîte divers, sélectionnez Sondage dans le menu déroulant. (6) Lancer le programme d'enquête en cliquant sur Launch. (7) Sélectionnez les puits préremplis sur mesure. (8) Commencez l'analyse en cliquant sur Go. (9) Une fois l'enquête effectuée, les volumes mesurés sont écrits dans les puits sélectionnés correspondants. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
6. Dispensation d'ADN pilotée par l'ADE dans la plaque de destination
- Exécutez le logiciel de liste de sélection, fixez les types de plaques de source et de destination de 384 puits à 384-LDV et Greiner 384PS-781096, respectivement (figure 5). Placez l'appareil en mode de distribution tampon aqueuse en sélectionnant 384LDV-AQ-B2, et décochez « optimiser le débit de transfert ».

Figure 5 : Performance des dispensations basées sur la liste de sélection. (1) Démarrer le logiciel nanodispenser. Dans l'onglet Protocole, sélectionnez (2) le format de plaque d'échantillon, (3) le type de plaque de destination et (4) unticks "optimiser le débit de transfert". (5) Sélectionnez l'onglet Liste de sélection. (6) Cliquez sur L'importation et sélectionnez le fichier approprié '.csv 'DNA-PickList ou T.R.-Picklist). (7) Une fois sélectionné, cliquez sur Import. (8) Cliquez sur Le jeu et enregistrer le protocole. (9) Effectuer une simulation de dispensation en cliquant sur Simuler, ou (10) Démarrer la dispensation programmée en cliquant sur Run. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
- Sélectionnez l'onglet "Liste de sélection", cliquez sur Import, sélectionnez le fichier DNA-Picklist.csv. Cliquez sur Jouer et enregistrer le protocole. Cliquez sur Simuler pour effectuer une simulation des dispensations programmées pour vous assurer que la liste de sélection correspond à la conception expérimentale attendue. Une fois terminé, cliquez sur Fermer.
- Cliquez sur Play, puis Run, pour commencer le programme de distribution: lorsqu'on vous le demande, insérez la plaque source demandée (solutions D'ADN remplies manuellement) et la plaque de destination (rempli de diluants) dans le nanodispenser.
REMARQUE : Le temps de distribution est d'environ 5-20 min pour une plaque complète de 384 puits, selon les volumes sélectionnés et le nombre total de dispenses dans la conception expérimentale. - Sinon, faites une pause dans le protocole, car les plaques remplies de diluant et d'ADN peuvent gérer le stockage sec ou congelé jusqu'à 7 jours. Pour le stockage à sec, laissez sécher les assiettes sur le banc à température ambiante, puis entreposez-les de la même façon. Décongeler et centrifugeuse (à 1 500 x g pendant 2 min) les plaques stockées congelées avant d'être utilisées dans une étape de transfection (section 7).
7. Dispensation de réactif de transfection ade-conduite
- Dans un coffret de biosécurité, diluer extemporaneusement le réactif de transfection de lipopolyplex dans le milieu sérique-libre à une concentration finale 1x. Vortex et dispenser immédiatement ce mélange de réactif de transfection selon la plaque source prédéfinie (s) conçue par la macro et en utilisant l'application précalibrée 384-puits guide de tuyauterie comme décrit à l'étape 3.4.
REMARQUE : Ne pas centrifier la plaque source une fois qu'elle est chargée avec le réactif car aucune transfection n'est remarquée après centrifugation. - Exécuter le programme de nanodispenser pour effectuer une « enquête » telle que décrite à la section 5, afin de contrôler les volumes de tous les puits TR remplis manuellement de la plaque source afin d'éviter les erreurs de distribution dues à des volumes supérieurs à 12 L.
- Cliquez sur Reset pour effacer la liste d'échantillons de la liste de sélection de l'ADN dans le logiciel de liste de sélection, et vérifier que les paramètres de l'appareil sont toujours réglés sur des tampons aqueurs et sur les types de plaques de source et de destination utilisés, comme à l'étape 6.1.
- Cliquez sur Import et choisissez le fichier TR-Picklist.csv. Cliquez sur Jouer et enregistrer le protocole si vous êtes invité, et (c'est optionnellement mais fortement recommandé) effectuer une simulation des dispensations programmées de mélange de réactifde transfection pour assurer une conception appropriée des dispensations en cliquant sur le Bouton de simulation. Une fois terminé, cliquez sur Fermer.
- Cliquez sur Le bouton Play, puis Exécutez le bouton pour démarrer le programme de distribution : comme demandé, placez la plaque source (s) (TR-mélange-remplie) et la plaque de destination (diluent- et ADN-remplie) dans le nanodispenser.
REMARQUE : Le temps de distribution est inférieur à 20 min pour une plaque complète de 384 puits lors de la distribution de 500 nL de mélange TR. - Incuber 15-30 min à température ambiante après avoir ajouté le TR à l'ADN comme indiqué par le protocole du fabricant.
8. Dispensation de cellules à base de liquide peristaltique
- Préparer le gestionnaire de liquide péristtique pour la distribution des cellules. Désinfecter une tête de cassette de 10 l l en la pulvérisant avec du désinfectant Aniospray Surf 29 et en absorbant le reste sur papier. Montez la cassette sur le dispositif de manutention de liquide péritaltique, changez le réglage de type de cassette à 10 L, et assurez-vous que le format de plaque est réglé à 384 puits.
- Désinfecter le tube de cassette de 10 L tel que décrit précédemment à l'étape 4.2. Plongez l'organisateur de tube dans un récipient stérile et rincer le tube avec 5 ml d'alcool de 70%, puis avec 5 ml d'eau distillée, et enfin, avec 5 ml de milieu sans sérum, successivement rempli dans le même récipient et jusqu'à ce que chaque tube est vide.
- Préparer la suspension de la cellule à distribuer. À partir d'un plat de culture De la cellule B10 confluent, lavez les cellules 1x avec une solution saline tamponnée par phosphate 1x (PBS), puis dissoisez les cellules avec de la trypsine/EDTA pendant 5 min à 37 oC.
- Vérifier la dissociation cellulaire au microscope et arrêter l'action trypsine/EDTA en ajoutant 10 ml de milieu complet (DMEM complété par 10 % de sérum bovin fœtal et 100 U/mL de pénicilline-streptomycine; voir le tableau des matériaux) dans le plat de culture. Récoltez les cellules dans un tube de 50 ml et comptez les cellules au microscope, à l'aide d'une cellule de Malassez ou d'un compteur cellulaire automatique.
- Préparer au moins 25 ml de suspension cellulaire HeLa à une concentration de 37 500 cellules/mL dans un milieu complet (c.-à-d. 1 500 cellules/40 l) pour une plaque complète de 384 puits, afin d'assurer l'amorçage du tube et la distribution de 40 l/puits.
- Pour dispenser les cellules, remplissez un nouveau vaisseau stérile de la suspension de cellules préparée et remuez-le pour éviter la sédimentation conduisant à une inexactitude dans la densité cellulaire de la dispensation. Insérez l'organisateur de tube dans cette solution et appuyez sur le bouton Prime jusqu'à ce que la suspension de la cellule commence à rincer de la tête de distribution. Assurez-vous qu'aucune de la pointe n'est obstruée en inspectant visuellement le flux liquide de chacun d'eux, et assurez-vous que chaque tube est chargé avec la suspension cellulaire.
- Chargez la plaque de destination 384 puits remplie d'ADN et de TR sur le porte-plaques de manutention de liquide péristaltique et retirez son couvercle. Exécuter le programme précalibré pour distribuer 40 l de la suspension cellulaire sur la plaque complète de 384 puits (c.-à-d., 1 500 cellules/puits). Le temps de distribution est d'environ 8 s. Remplacer le couvercle de la plaque de 384 puits.
REMARQUE : Alternativement, la suspension cellulaire de 40 l peut être distribuée manuellement à l'aide d'une micropipette multicanal.
9. Etoiles biologiques personnalisées (surveillance de l'efficacité de la transfection cellulaire)
REMARQUE : Après les paramètres expérimentaux et l'intention de l'expérience, utilisez les méthodes requises pour la luminescence, la fluorescence, le dépistage à haute teneur et la réaction quantitative en chaîne de polymérase de transcription inversée (RT-qPCR). Dans cette section du protocole, l'efficacité de la transfection cellulaire est évaluée par microscopie automatisée de fluorescence et analyse d'image.
- Incuber la plaque à 37 oC avec 5 % de CO2 dans une atmosphère saturée d'eau et jusqu'à ce qu'elle soit correctement accompagnée de protéines.
REMARQUE : Ici, un temps d'incubation de 48 h est utilisé pour les cellules HeLa pour surveiller l'efficacité de la transfection, à l'aide de plasmides exprimant le tdTomato et le mVenus. - Retirez le milieu de culture 48 h après la transfection en inversant la plaque, ajoutez 30 l/puits de formaline de 10 % à l'aide du gestionnaire de liquide péristaltique (10 l cassette), et incubez pendant 15 min à température ambiante.
- Retirer la formaline en inversant la plaque; puis, incuber les cellules pendant 15 min à température ambiante avec 0,1 ng/mL Hoechst dilué dans 1x PBS solution.
- Laver les cellules 3x pendant 15 min avec 80 OL de 1x PBS ajusté au pH 8 afin de récupérer le signal de fluorescence élevé perdu par les 6,9 pH de l'étape d'incubation de la solution formaline.
- À l'aide d'un microscope fluorescent automatisé, acquérir des images de deux ou trois canaux fluorescents (Hoechst, tdTomato et mVenus) de façon séquentielle avec des objectifs 10x et un ensemble de filtres d'émission approprié (4,6-diamidino-2-phenylindole [DAPI], dsRed, et fluorescein isothiocyanate [FITC], respectivement).
- Pour évaluer l'efficacité de la transfection, utilisez un logiciel d'analyse d'image pour déterminer l'efficacité de la transfection à l'aide de l'analyse de script basée sur la coloration des noyaux.
Access restricted. Please log in or start a trial to view this content.
Résultats
fAfin de déterminer si la technologie ADE pourrait être utilisée pour un protocole automatisé de transfection inverse, nous avons surveillé l'efficacité de la transfection cellulaire par microscopie fluorescence, à l'aide d'un tdTomato fluorescent rouge exprimant plasmide. D'abord visant à déterminer les meilleurs paramètres de transfection, différents volumes de diluants et quantités totales d'ADN ont été testés. Le volume diluant a été utilisé pour permettre aux goutte...
Access restricted. Please log in or start a trial to view this content.
Discussion
L'établissement et l'optimisation d'une méthode précise de transfection à haut débit pour une lignée cellulaire donnée exigent des scientifiques qu'ils suivent certains paramètres clés décrits dans cette section. Nous encourageons fortement en commençant par les valeurs recommandées tout au long du protocole que ces paramètres optimisés pour les cellules HeLa s'est également avéré efficace pour les cellules HEK. Cependant, comme les meilleurs paramètres peuvent dépendre des lignées cellulaires et des ...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Les auteurs ont révélé un reçu du soutien financier suivant pour la recherche, la paternité et/ou la publication de cet article : Inserm, Université de Lille, Institut Pasteur de Lille, Conseil Régional du Nord, et PRIM-HCV1 et 2 (Pôle de Recherche Interdisciplinaire sur le Médicament), l'Agence Nationale de la Recherche (ANR-10-EQPX-04-01), le Feder (12001407 (D-AL) Equipex Imaginex BioMed) et la Communauté européenne (ERC-STG INTRACELLTB n ' 260901). Les auteurs tiennent à remercier le Dr S. Moureu, le Dr B. Villemagne, le Dr R. Ferru-Clément et le Dr H. Groult pour leur examen critique et les corrections du manuscrit.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 384LDV Microplate | Labcyte | LP-0200 | |
| 384-well Microplate μClear Black | Greiner | 781906 | |
| Ampicilin | Sigma | A9393-5G | Selection antibiotic for bacteria transformed with ampicilin expressing vector |
| Android Tablet | Samsung | Galaxy Note 8 | used to guide the user while the source plate manual dispense |
| Aniospray Surf 29 | Anios | 2421073 | disinfectant to clean the MicroFlo head |
| Columbus software | Perkin Elmer | image analysis software | |
| Dulbecco's Modified Eagle Medium (DMEM), high glucose, GlutaMAX Supplement, pyruvate | Thermo Fisher Scientific | 10566032 | cell culture medium |
| Echo Cherry Pick 1.5.3 software | Labcyte | Software enabling ADE-based dispenses by the Echo550 device from a *.csv file; nanodispenser software | |
| Echo550 | Labcyte | ADE-based dispenser | |
| Fetal Bovine Serum | Thermo Fisher Scientific | 16000044 | to add in cell culture medium |
| Formalin solution, neutral buffered, 10% | Sigma-Aldrich | HT501128-4L | to fix cell |
| HeLa cells | ATCC | HeLa (ATCC® CCL-2™) | |
| Hoechst 33342, Trihydrochloride, Trihydrate | Thermo Fisher Scientific | H3570 | 10 mg/mL Solution in Water |
| INCell Analyzer 6000 | GE Healthcare | 29043323 | automated laser-based confocal imaging platform |
| LB medium | Thermoischer Scientific LB Broth Base (Lennox L Broth Base)®, powder | 12780052 | culture medium for bacteria growth |
| Lysis Buffer (A2) | Macherey-Nagel | 740912.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| MicroFlo 10µL cassette | Biotek Instruments Inc | 7170013 | to use with the Microflo Dispenser |
| MicroFlo 1μL cassette | Biotek Instruments Inc | 7170012 | to use with the Microflo Dispenser |
| MicroFlo Dispenser | Biotek Instruments Inc | 7171000 | peristaltic pump-based liquid handler device |
| Microvolume spectrophotometer | Denovix | DS-11 Spectrophotometer | Measure the DNA concentration of samples |
| mVenus plasmid | mVenus cDNA was cloned by enzymatic restriction digestion and ligation in Age1/BsrG1 sites of the tdTomato-N1 plasmid | Vector type: Mammalian Expression, Fusion Protein: mVenus | |
| Neutralization Buffer (A3) | Macherey-Nagel | 740913.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| NucleoSpin Plasmid kit | Macherey-Nagel | 740588.50 | used to prepare plasmid from bacterial culture |
| Optimal-Modified Eagle Medium (Opti-MEM) Medium | Thermo Fisher Scientific | 31985070 | |
| optional Wash bufferWash Buffer (A4) | Macherey-Nagel | 740914.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| orbital shaker | incubated large capacity shaker | 444-7084 | Used to grow bacteria under gentle agitation and 37°C |
| Penicillin-Streptomycin | Thermo Fisher Scientific | 15140122 | 10,000 U/mL |
| Phosphate-Buffered Saline | Thermo Fisher Scientific | 10010001 | |
| Plasmid mini-columns | Macherey-Nagel | 740499.250 | Silica membrane mini-column to prepare plasmid from bacterial culture |
| Resuspension Buffer (A1) | Macherey-Nagel | 740911.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| RNAse A | Macherey-Nagel | 740505 | Enzyme from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| tdTomato-N1 plasmid | Addgene | Plasmid #54642 | Vector type: Mammalian Expression, Fusion Protein: tdTomato |
| TransIT-X2 Dynamic Delivery System | Mirus Bio | MIR 6000 | |
| Wash Buffer (AW) | Macherey-Nagel | 740916.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| 3D printer | Creality | CR10S | used to print the plate adapter |
| Blender Software | https://www.blender.org/ Free software under GNU General Public License (GPL). | version 2.79b | used to design the plate adapter |
Références
- Colin, B., Deprez, B., Couturier, C. High-Throughput DNA Plasmid Transfection Using Acoustic Droplet Ejection Technology. SLAS Discovery: Advancing Life Sciences R & D. , 2472555218803064(2018).
- Mirus Bio. Optimising Transfection Performance. , (2019).
- Thermo Fisher Scientific. Factors Influencing Transfection Efficiency | Thermo Fisher Scientific - FR. , https://www.thermofisher.com/fr/fr/home/references/gibco-cell-culture-basics/transfection-basics/factors-influencing-transfection-efficiency.html (2019).
- Boussif, O., et al. A versatile vector for gene and oligonucleotide transfer into cells in culture and in vivo: polyethylenimine. Proceedings of the National Academy of Sciences of the United States of America. 92 (16), 7297-7301 (1995).
- Figueroa, E., et al. A mechanistic investigation exploring the differential transfection efficiencies between the easy-to-transfect SK-BR3 and difficult-to-transfect CT26 cell lines. Journal of Nanobiotechnology. 15 (1), 36(2017).
- Kirchenbuechler, I., Kirchenbuechler, D., Elbaum, M. Correlation between cationic lipid-based transfection and cell division. Experimental Cell Research. 345 (1), 1-5 (2016).
- Zhang, Z., Qiu, S., Zhang, X., Chen, W. Optimized DNA electroporation for primary human T cell engineering. BMC Biotechnology. 18 (1), 4(2018).
- Cao, D., et al. Transfection activity and the mechanism of pDNA-complexes based on the hybrid of low-generation PAMAM and branched PEI-1.8k. Molecular bioSystems. 9 (12), 3175-3186 (2013).
- Bos, A. B., et al. Development of a semi-automated high throughput transient transfection system. Journal of Biotechnology. 180, 10-16 (2014).
- Colosimo, A., et al. Transfer and expression of foreign genes in mammalian cells. BioTechniques. 29 (2), 314-318 (2000).
- Villa-Diaz, L. G., Garcia-Perez, J. L., Krebsbach, P. H. Enhanced transfection efficiency of human embryonic stem cells by the incorporation of DNA liposomes in extracellular matrix. Stem Cells and Development. 19 (12), 1949-1957 (2010).
- Sabatini, D. M. Reverse transfection method. , WO2001020015A1(2001).
- Raymond, C., et al. A simplified polyethylenimine-mediated transfection process for large-scale and high-throughput applications. Methods (San Diego, CA). 55 (1), 44-51 (2011).
- Junquera, E., Aicart, E. Recent progress in gene therapy to deliver nucleic acids with multivalent cationic vectors. Advances in Colloid and Interface Science. 233, 161-175 (2016).
- Woodruff, K., Maerkl, S. J. A High-Throughput Microfluidic Platform for Mammalian Cell Transfection and Culturing. Scientific Reports. 6, 23937(2016).
- Vasileiou, T., Foresti, D., Bayram, A., Poulikakos, D., Ferrari, A. Toward Contactless Biology: Acoustophoretic DNA Transfection. Scientific Reports. 6, 20023(2016).
- Hadimioglu, B., Stearns, R., Ellson, R. Moving Liquids with Sound: The Physics of Acoustic Droplet Ejection for Robust Laboratory Automation in Life Sciences. Journal of Laboratory Automation. 21 (1), 4-18 (2016).
- Grant, R. J., et al. Achieving accurate compound concentration in cell-based screening: validation of acoustic droplet ejection technology. Journal of Biomolecular Screening. 14 (5), 452-459 (2009).
- Sackmann, E. K., et al. Technologies That Enable Accurate and Precise Nano- to Milliliter-Scale Liquid Dispensing of Aqueous Reagents Using Acoustic Droplet Ejection. Journal of Laboratory Automation. 21 (1), 166-177 (2016).
- Day, R. N., Davidson, M. W. The fluorescent protein palette: tools for cellular imaging. Chemical Society Reviews. 38 (10), 2887-2921 (2009).
- Zielinski, D., Gordon, A., Zaks, B. L., Erlich, Y. iPipet: sample handling using a tablet. Nature Methods. 11 (8), 784-785 (2014).
- Brunner, S., et al. Cell cycle dependence of gene transfer by lipoplex, polyplex and recombinant adenovirus. Gene Therapy. 7 (5), 401-407 (2000).
- Nii, T., et al. Single-Cell-State Culture of Human Pluripotent Stem Cells Increases Transfection Efficiency. BioResearch Open Access. 5 (1), 127-136 (2016).
- Noonan, D. J., Henry, K., Twaroski, M. L. A High-Throughput Mammalian Cell-Based Transient Transfection Assay. Signal Transduction Protocols. 284, 051-066 (2004).
- Transfection | TransIT Transfection Reagents | Mirus Bio. , https://www.mirusbio.com/products/transfection (2015).
- American Type Culture Collection. General protocol for transfection of stem cells, primary cells, and continuous cell lines with ATCC TransfeX Transfection Reagent. , https://www.atcc.org/~/media/Transfection%20protocols/TransfeX/TransfeX%20General%20Protocol%20on%20letterhead.ashx (2017).
- American Type Culture Collection. Transfection Reagents for Nucleic Acid Transfer into ATCC Cells. , http://www.lgcstandards-atcc.org/~/media/PDFs/Marketing%20Material/Cell%20Biology/Transfection%20reagents%20for%20nucleic%20acid.ashx (2017).
- de Los Milagros Bassani Molinas, M., Beer, C., Hesse, F., Wirth, M., Wagner, R. Optimizing the transient transfection process of HEK-293 suspension cells for protein production by nucleotide ratio monitoring. Cytotechnology. 66 (3), 493-514 (2014).
- Promega. FuGENE® 6 Transfection Reagent. , https://www.promega.com/-/media/files/resources/protocols/technical-manuals/101/fugene-6-transfection-reagent-protocol.pdf (2019).
- Olden, B. R., Cheng, Y., Yu, J. L., Pun, S. H. Cationic polymers for non-viral gene delivery to human T cells. Journal of Controlled Release: Official Journal of the Controlled Release Society. 282, 140-147 (2018).
- Park, E., Cho, H. B., Takimoto, K. Effective gene delivery into adipose-derived stem cells: transfection of cells in suspension with the use of a nuclear localization signal peptide-conjugated polyethylenimine. Cytotherapy. 17 (5), 536-542 (2015).
- Wood, R. W., Loomis, A. L. The physical and biological effects of high-frequency sound-waves of great intensity. The London, Edinburgh, and Dublin Philosophical Magazine and Journal of Science. 4 (22), 417-436 (1927).
- Eliminating Endotoxin at the Source - A Novel Competent Cell Line with Modified Lipopolysaccharide for Low-Endotoxin Plasmid Production. Mamat, U., et al. 28th Annual Symposium of the Protein Society, San Diego, CA, , (2014).
- Ivanova, N. V., Kuzmina, M. L. Protocols for dry DNA storage and shipment at room temperature. Molecular Ecology Resources. 13 (5), 890-898 (2013).
- Lesnick, J., Lejeune-Dodge, A., Ruppert, N., Jarman, C. High-Precision Cell Dispensing with the Labcyte Echo® Liquid Handler. , https://www.labcyte.com/content/applications/high-precision-cell-dispensing-with-the-labcyte-echo-liquid-handler (2017).
- Yang, X., et al. A public genome-scale lentiviral expression library of human ORFs. Nature Methods. 8 (8), 659-661 (2011).
- Peng, J., Zhou, Y., Zhu, S., Wei, W. High-throughput screens in mammalian cells using the CRISPR-Cas9 system. The FEBS journal. 282 (11), 2089-2096 (2015).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon