É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Multiplexação e transfecção de plasmídeo de DNA de alta produtividade usando tecnologia de Nanodosagem acústica
Neste Artigo
Resumo
Este protocolo descreve o transfection do plasmídeo da elevado-produção de pilhas de mamíferos em uma placa 384-well usando a tecnologia acústica da ejeção da gota. O tempo-demorado, erro-propenso a dispensar e Multiplexing do ADN, mas igualmente a dispensação do reagente do transfection, são software-driven e executado por um dispositivo do nanodispenser. As células são então semeadas nestes poços pré-preenchidos.
Resumo
O transfection da pilha, indispensável para muitos estudos biológicos, exige controlar muitos parâmetros para uma realização exata e bem sucedida. Na maioria das vezes realizada em baixa taxa de transferência, é, além disso demorado e propenso a erros, ainda mais quando multiplexar vários plasmídos. Nós desenvolvemos um método fácil, rápido, e exato executar o transfection da pilha em um layout da placa de 384 poços usando a tecnologia acústica da ejeção da gota (ADE). O dispositivo do nanodispenser usado neste estudo é baseado nesta tecnologia e permite a entrega precisa do nanovolume na alta velocidade de uma placa do poço da fonte a um destino um. Pode dispensar e Multiplex o ADN e o reagente do transfection de acordo com uma folha de cálculo pré-definidos. Aqui nós apresentamos um protocolo óptimo para executar o transfection do plasmídeo da elevado-produção baseada em ADE que faz possível alcangar uma eficiência de até 90% e um Cotransfection de quase 100% em experimentos do Cotransfection. Estendemos o trabalho inicial propondo uma macro baseada em planilhas de fácil utilização, capaz de gerenciar até quatro plasmímetros/poços de uma biblioteca contendo até 1.536 plasmímetros diferentes e uma aplicação de guia de pipetagem baseada em Tablets. A macro projeta os modelos necessários da placa de origem (s) e gera os arquivos prontos para uso para o aplicativo baseado em Tablet e nanodispenser. O protocolo da transfecção de quatro etapas envolve i) um diluente dispensar com um manipulador líquido clássico, II) distribuição e multiplexação do plasmídeo, III) um reagente do transfection dispense pelo nanodispenser, e IV) chapeamento de pilha nos poços pré-preenchidos. O controle software-baseado descrito da multiplexação e do transfection do plasmídeo do Ade permite mesmo especialistas no campo de executar um transfection de pilha de confiança em uma maneira rápida e segura. Esse método permite a rápida identificação de configurações ideais para um determinado tipo de célula e pode ser transposta para abordagens de maior escala e manual. O protocolo facilita aplicações, como a proteína ORFeome humana (conjunto de quadros de leitura abertos [ORFs] em um genoma) ou a validação da função gênica baseada em CRISPR-Cas9, em estratégias de triagem não agrupadas.
Introdução
O método aqui apresentado descreve detalhadamente como realizar a multiplexação e transfecção de plasmídeo de DNA em células de mamíferos com alta taxa de transferência usando um nanodispensador líquido acústico em uma placa de 384 poços, mesmo para não especialistas no campo. Este método publicado recentemente1 permite executar tanto quanto 384 condições independentes da multiplexação e do transfection do ADN do plasmídeo em um experimento, em menos de 1 h. os únicos ou os experimentos do Cotransfection eram bem sucedidos, alcançando uns 100% próximos Cotransfection dentro da população transfected das pilhas. Esse protocolo facilita a transfecção porque a maioria dos passos tediosos, demorados e propensos a erros agora são acionados por software (consulte a Figura 1 para obter uma visão geral). Mais esforços foram feitos para desenvolver ferramentas dedicadas para melhorar a facilidade de uso, evitando erros humanos durante o processo geral e para promover a transfecção bem-sucedida, mesmo para os não-especialistas no campo. O protocolo descrito inclui uma planilha de macro "fácil de usar" que desenvolvemos a fim de gerenciar 384 condições de transfecção independentes com possibilidades de multiplexação de até quatro plasmídos em cada poço. A macro gera automaticamente modelos de placas de origem para carregar o volume de plasmídeo de DNA esperado de iniciar soluções de estoque e os arquivos necessários para conduzir o software nanodispenser sobre o experimento que foi introduzido. Como a dispensação manual do DNA em uma placa de fonte 384-well é tedioso e propenso a erros, também desenvolvemos um aplicativo baseado em Tablet dedicado para orientar o usuário ao dispensar a solução de DNA de acordo com o modelo.
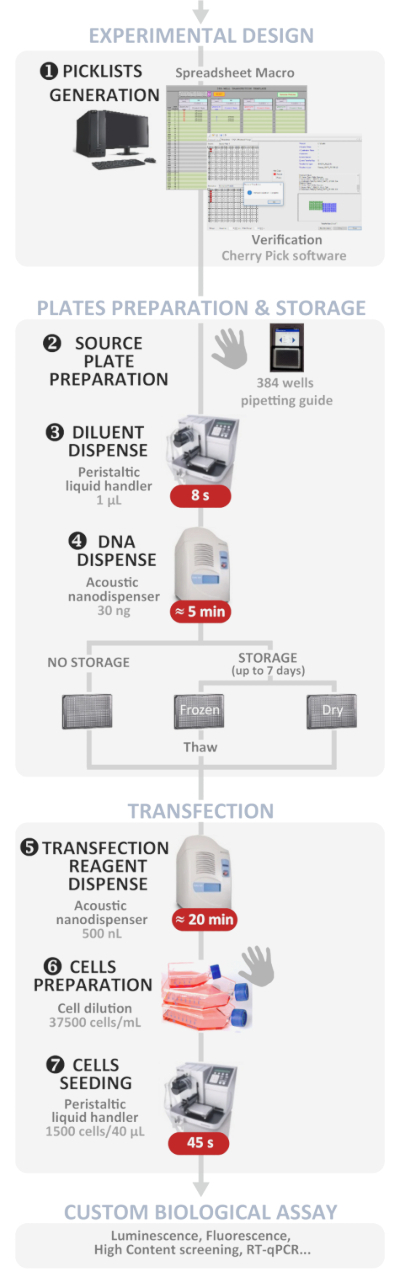
Figura 1: fluxo de trabalho experimental. Representação esquemática do protocolo de transfecção reversa de alta taxa de transferência automatizada ideal (do experimento experimental para o ensaio biológico personalizado). As etapas manuais são indicadas pelo símbolo da mão e o tempo aproximado para cada etapa é escrito em uma caixa vermelha. Por favor clique aqui para ver uma versão maior desta figura.
Muitas experiências Cell-Based começam com o transfection do ADN do plasmídeo, e mesmo se muitos reagentes dedicados foram e estão sendo desenvolvidos ainda para realçar a eficiência do transfection e/ou facilitar o procedimento, muito resta ser feito2,3 , 4. o transfection da pilha do plasmídeo do ADN envolve diversas etapas para alcangar a eficiência elevada, tal como uma tomada complexa inicial, um escape endosomal, e um transporte cytoplasmic ao núcleo5,6. Além de precipitação de cálcio ou técnicas físicas, como eletroporação ou microinjeção usando dispositivos dedicados7, métodos químicos modernos têm focado em melhorar a entrega de células de DNA, reduzindo a citoxicidade celular8, a 9. O uso de lipídios ou polímeros catiônicos formando complexos liposemelhantes e, mais recentemente, os sistemas de química polimérico não-Liposomal tornaram a transfecção mais fácil e eficiente10. Apesar destes desenvolvimentos, o transfection da pilha ainda exige habilidades específicas ser executado exatamente como a maioria destes protocolos físicos ou químicos do transfection exigem cientistas de preparar manualmente cada condição da reação do transfection do ADN, assim prejudicar a taxa de transferência. Para contornar esse problema, os protocolos de transfecção reversa foram desenvolvidos usando reagentes de transfecçãoquímica 11,12,13, permitindoao usuário testar ou combinar vários plasmídos de forma mais rápida. Nestes protocolos, os complexos de ácidos nucleicos com reagentes de transfecção são formados antes da semeadura das células nos complexos. No entanto, esses protocolos inversos ainda são limitados pelo manuseio manual de soluções de DNA e pela combinação de cada uma das condições independentes. Embora seja viável para realizá-los em um formato de placa 96-well, a preparação de DNA e dispensa será tedioso, e provavelmente haverá erros. Quando as quantidades diferentes de diversos plasmídeos do ADN são exigidas e multiplexados com se, o transfection da pilha se torna ainda mais duro de conseguir e mais demorado, e os erros humanos tornam-se completamente inevitáveis. Escalar até o formato da placa 384-well em uma aproximação reversa do transfection, apesar de poucas condições multiplexados do transfection do ADN, transforma-se um desafio impossível devido às seguintes razões. i) as quantidades de ADN, o reagente de transfecção ou os volumes de mistura de reacção a gerir são inferiores a 1 μL para cada poço. II) a multiplexação de plasmídeos para 384 condições independentes torna-se extremamente complicada. A entrega em cada um dos 384 poços também é III) altamente demorado e IV) propenso a erros. De fato, dispensar a solução certa nos poços esperados é difícil de administrar, pois os volumes baixos já dispensados não permitem o monitoramento visual entre os poços vazios e já preenchidos. v) finalmente, há um alto risco de secagem da mistura por evaporação antes que as células sejam adicionadas devido ao tempo necessário para realizar as etapas de dispensação necessárias. Em resumo, o fator limitante para configurar ensaios de transfecção de plasmídeo de DNA de alta taxa de transferência parece ser a miniaturização do ensaio, o que implica a multiplexação e o gerenciamento de baixo volume que não podem ser manuseados manualmente, mas também dificilmente alcançáveis em um maneira de confiança por manipuladores líquidos Peristatic clássicos.
Como prova de dificuldade para automatizar tais ensaios e obter alta taxa de transferência, apenas algumas tentativas de automatização da transfecção foram publicadas até agora: um formato de placa de 96 poços usando um dispositivo comercial de manuseio de líquidos e uma precipitação de fosfato de cálcio14 e, mais recentemente, um reagente lipoplex, e um chip microfluídico permitindo 280 Transfections independentes15 , mas exigindo habilidades especializadas neste campo. Outro método, acoustophoresis, permitindo a levitação líquida e conduzindo à manipulação e à mistura fluidas, foi usado para executar o transfection do ADN em 24-aos formatos da placa de 96-well16. Embora viável, esta aproximação sofre de uma taxa de transferência extremamente baixa como a mistura das pilhas com a mistura do transfection do ADN exige uma incubação de 60 s para cada único ponto antes de semear. Isso implica uma duração de pelo menos 96 min para uma placa completa de 96 poços. Além disso, este protocolo está longe de ser passíveis ao público geral dos biólogos como este trabalho foi feito com um dispositivo interno projetado e manufacturado que não esteja atualmente disponível no mercado. Pelo contrário, nos últimos anos, uma tecnologia de dispensação acústica baseada em software fácil de usar surgiu com dispositivos de dispensador de nanovolume. Usando a energia acústica focalizada, estes dispositivos permitem a ejeção firmemente controlada de volumes líquidos pequenos de 2,5 nL a 500 nL de uma placa da fonte a um destino um17. Esta tecnologia, chamada de ejeção de gotas acústicas (ADE), tem inúmeras vantagens: é totalmente automatizada, sem contato, tipless, precisa, precisa e altamente reprodutível, e tem uma alta taxa de transferência18. Primeiro dedicado à entrega de soluções de dimetil sulfóxido (DMSO), as configurações foram aprimoradas para dispensar buffers aquosos19. Os nanodispensadores acústicos, então, parecem adequados para protocolos de transfecção de células reversas e podem contornar a maioria das limitações manuais acima mencionadas. Como nenhuma tentativa do transfection do plasmídeo foi descrita previamente usando esta tecnologia, nós avaliamos recentemente a adequação de um sistema de distribuição acústico-baseado para executar o transfection reverso da pilha.
Aproveitando-se da taxa de transferência do nanodispenser e da facilidade de utilização, nós otimizamos um protocolo reverso do transfection para pilhas de HeLa por testes cruzados diversos parâmetros que podem influenciar o transfection do ADN em uma placa 384-well, única, a saber, a quantidade total do ADN e concentração inicial do ADN da fonte, volume do diluente, reagente do transfection, e número de pilhas da propagação. O protocolo desenvolvido contorna as limitações manuais acima descritas do Transfection da pilha e apresenta diversas vantagens sobre outras tentativas automatizadas do transfection. Em primeiro lugar, é miniaturizado, permitindo assim o reagente de transfecção rentável, salvando as preparações de plasmídeo de DNA e o reagente de transfecção. Em segundo lugar, é muito mais elevado-throughput e reprodutível do que o protocolo manual (mesmo para novatos), porque o transfection de uma placa inteira de 384-well pode ser conseguido em menos de 1 h. Finalmente, é orientado por software, permitindo o controle da quantidade de DNA dispensado e a multiplexação de vários plasmídos. Certamente, agradecimentos ao software do nanodispenser (tabela dos materiais), o usuário pode elaborar uma planta do estudo para controlar os volumes a ser dispensados de uma placa bem definida da fonte a um destino um.
O protocolo aqui apresentado destina-se principalmente àqueles que têm acesso a um nanodispenser e gostaria de configurar experimentos de transfecção em alta taxa de transferência, mas também para aqueles que desejam otimizar rapidamente seus parâmetros de transfecção para um determinado tipo de célula por Aplicando este protocolo para Cross-Test vários parâmetros em alta taxa de transferência. Na verdade, mostramos que os parâmetros otimizados identificados com este protocolo de nanoescala podem ser transpostos para experimentos de transfecção de maior escala e manuais. Finalmente, como o reagente do transfection usado no protocolo atual permite o transfection do ADN ou do siRNA de acordo com o fabricante, o protocolo é igualmente do interesse àqueles que visam executar aproximações da disposição para o superexpressão ou o knockdown do gene. As placas do destino pré-enchidas com o ADN podem ser conservadas até 7 dias antes do uso em um ensaio do transfection sem perda de eficácia, que é uma outra vantagem do seguinte protocolo para este tipo da aplicação.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. preparações antecipadas
- Preparação dos programas de tratamento de líquidos peristálticos
Nota: para o diluente e as etapas de dispensação da célula do protocolo, um programa dedicado deve ser preparado, tendo em conta a altura da cabeça dispensadora para a placa utilizada e a intenção da etapa.- Para o passo de dosagem de diluente de 1 μL, monte uma gaveta de 1 μL e prepare um programa com as definições descritas nas etapas 1.1.1.1 e 1.1.1.2.
- Ajuste o parâmetro de vazão para alto para obter a melhor taxa de transferência, pois não é esperado nenhum dano de material biológico nesta etapa. Ajuste a altura de dosagem para 9,6 mm (de acordo com a placa de cultura de células utilizada, suplementar Figura 1) para permitir que a gota de 1 μl toque na parte inferior dos poços durante a dispensação.
Nota: esta etapa é crucial para evitar a retenção das gotas na cabeça dispensadora até atingir um volume suficiente para soltar. - Ajuste a altura desobstruída da placa a 14,4 milímetros para permitir um deslocamento livre da cabeça de dispensação sobre a placa após dispensar cada fileira. Controle visualmente as configurações apropriadas da altura da cabeça do manipulador líquido peristáltico: Certifique-se de que nenhuma gota seja mantida nas pontas de dosagem enquanto dispensa e verifique se a cabeça é suficientemente alta para permitir o deslocamento da cabeça após dispensar cada linha.
Observação: evitar a retenção de queda é um parâmetro crucial, pois prejudicará a precisão do volume da dispensação.
- Ajuste o parâmetro de vazão para alto para obter a melhor taxa de transferência, pois não é esperado nenhum dano de material biológico nesta etapa. Ajuste a altura de dosagem para 9,6 mm (de acordo com a placa de cultura de células utilizada, suplementar Figura 1) para permitir que a gota de 1 μl toque na parte inferior dos poços durante a dispensação.
- Para dispensar a suspensão da célula 40 μL, monte uma gaveta de 10 μL e prepare um programa com as definições descritas nas etapas 1.1.2.1-1.1.2.2.
- Ajuste o parâmetro de vazão para baixo para dispensar células com uma velocidade baixa para evitar a promoção de danos potenciais para as células por tensão de cisalhamento e alto impacto na parte inferior dos poços. Ajuste a altura de dispensação para 11,43 mm (de acordo com a placa de cultura de células utilizada, suplementar Figura 1), suficientemente alta para diminuir o impacto da célula na parte inferior dos poços durante o processo de dosagem, mas suficientemente baixo para evitar a retenção das gotas no cabeça de dosagem. Ajuste a altura desobstruída da placa a 16 milímetros para permitir o deslocamento livre da cabeça de dispensação sobre a placa após dispensar cada fileira.
- Controle visualmente as configurações apropriadas da altura da cabeça do manipulador líquido peristáltico: Certifique-se de que nenhuma gota seja mantida nas pontas de dosagem enquanto dispensa e verifique se a cabeça é suficientemente alta para permitir o deslocamento da cabeça após dispensar cada linha.
Observação: evitar a retenção de queda é um parâmetro crucial, pois levará a dispensar um número de célula não confiável.
- Para o passo de dosagem de diluente de 1 μL, monte uma gaveta de 1 μL e prepare um programa com as definições descritas nas etapas 1.1.1.1 e 1.1.1.2.
- Preparação do plasmídeo do ADN (protocolo clássico da extração do protocolo)
- Cresça uma cepa de bactérias DH5α transformada em meio LB suplementado com 125 μg/mL de antibiótico de seleção de ampicilina (tabela de materiais) durante a noite a 37 ° c e agitação suave (200 rpm) em um agitador orbital (tabela de materiais).
- Colher 2 mL da cultura, pellet as células por centrifugação por 5 min em 6.000 x g, e descartar o sobrenadante.
- Ressuscitem o pellet celular com 250 μL de tampão de ressuscipensão contendo RNase A (tabela de materiais). Adicionar 250 μL de tampão de Lise e incubar durante 5 min à temperatura ambiente, de acordo com as instruções do fabricante.
- Pare a reação de Lise adicionando 300 μL de tampão de neutralização (tabela de materiais) e vortexting em breve. Centrifugar os tubos durante 5 min a 11.000 x g.
- Coloque uma nova minicoluna de plasmídeo (tabela de materiais) em um tubo de coleta de 2 ml e decantar o sobrenadante na coluna por centrifugação por 1 min a 11.000 x g.
- Descarte o fluxo e coloque a minicoluna de volta no tubo de coleta.
- Lave a minicoluna de plasmídeo com 500 μL de tampão de lavagem opcional (tabela de materiais) e centrifugue durante 1 min a 11.000 x g, de acordo com as instruções do fabricante.
- Descarte o fluxo e coloque a minicoluna de plasmídeo de volta no tubo de coleta.
- Adicionar 700 μL de tampão de lavagem (tabela de materiais) suplementado com etanol e centrifugador por 1 min a 11.000 x g, de acordo com as instruções do fabricante.
- Descarte o fluxo e centrifugue a minicoluna de plasmídeo e seu tubo de coleta 1x mais por 2 min a 11.000 x g para secar a membrana de sílica.
- Coloque a minicoluna de plasmídeo seco num novo tubo de 1,5 mL e adicione 30 μL de água destilada pré-aquecido a 60 ° c, incubar-o durante 2 min à temperatura ambiente e, em seguida, centrifugue-o durante 1 min a 11.000 x g.
- Descarte a minicoluna de plasmídeo e mantenha o eluato contendo o plasmídeo de DNA purificado.
- Meça a concentração do ADN do ADN eluída usando um espectrofotômetro do microespectrômetro (tabela dos materiais).
- Ligue o espectrofotômetro e escolha as configurações de medição de DNA .
- Levante o braço de amostragem do espectrofotômetro e Pipete 1 μL de água para o pedestal de medição para realizar uma calibração em branco.
- Abaixe o braço de amostragem, inicie a medição em branco e aguarde a conclusão.
- Levante o braço de amostragem e limpe a amostra dos pedestais superiores e inferiores.
- Pipetar 1 μL da solução de ADN para o pedestal inferior para medi-lo.
- Abaixe o braço da amostragem, comece a medida da concentração do ADN, e espere a conclusão.
- Levante o braço de amostragem e limpe a amostra dos pedestais superiores e inferiores.
- Para medidas mais adicionais da concentração do ADN, repita etapas 1.2.13.5-1.2.13.7.
- Uma vez que as medidas são terminadas, armazene as soluções do ADN em 4 ° c até o uso.
2. projeto experimental e geração das listas de opções para impulsionar as dispensas baseadas em ADE
Nota: uma macro de planilha dedicada "amigável" foi desenvolvida para gerenciar quantidades de DNA e misturar até quatro plasmídos em um formato de placa de 384 poços. Baseado no projeto experimental entrado, esta macro gera os arquivos necessários para conduzir o protocolo ADE-based do transfection do ADN pelo nanodispenser. A fim gerar estes arquivos, diversos campos têm que ser preenchidos na folha do molde como mostrado em Figura 2.
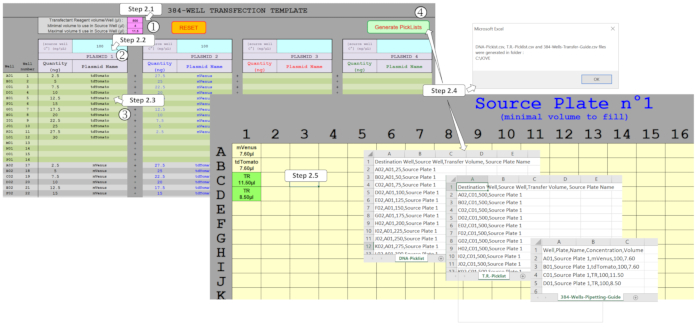
Figura 2 : Geração das listas de opções para conduzir a dispensação Ade usando a macro de planilha. Vários parâmetros devem ser preenchidos, ou seja, (1) o reagente de TRANSFECÇÃO (TR) e os volumes mínimos/máximos a serem utilizados na chapa de origem, (2) as concentrações iniciais de plasmídeo a serem dispensadas na chapa de origem e (3) a projeto da inteiro-placa, incluindo as quantidades esperadas do plasmídeo e multiplexação em cada um dos 384-Wells. (4) gerar listas de seleção ativação permite que os diferentes campos a serem verificados e, uma vez devidamente preenchido, listas de opções para a dispensação de DNA e Tr e o modelo de placa de fonte necessária são gerados automaticamente. Por favor clique aqui para ver uma versão maior desta figura.
- Insira os parâmetros do protocolo nanodispenser nos campos rosa. Defina o valor da mistura de reagente de transfecção (TR) para 500 nL. Defina o valor mínimo de volume nos poços da chapa de origem para 4 μL. defina o volume máximo nos poços da chapa de origem para 11,25 μL.
Nota: o nanodispenser utilizado aqui só pode transferir um máximo de 500 nL em uma corrida de ADE. Esses campos rosa são preenchidos com os valores recomendados, mas podem ser modificados de acordo com as necessidades do usuário. - Insira 100 ng/μL de concentrações iniciais de DNA nos campos azuis correspondentes ao DNA subjacente.
Observação: esse valor é a concentração ideal definida anteriormente, mas pode, no entanto, ser modificada para diferentes necessidades do usuário. - Introduza a quantidade de ADN desejada nos campos cinzento/Verde. Insira os valores e os nomes de plasmídeo para os 384 poços, assegurando a mesma ortografia se o mesmo plasmídeo for usado em vários poços.
- Gere o design da placa de origem, os arquivos de listas de opções e o arquivo de guia de pipetagem. Clique em gerar listas de opções para permitir que a macro gere o DNA-picklist. csv, o T. R.-picklist. csv e os arquivos 384-Wells-pipetting-Guide. csv dos dados coletados na planilha correspondente. Se solicitado, corrija os valores de célula com preenchimento de laranja, pois indica erros ou volumes que não podem ser manipulados pelo nanodispenser.
- Imprima o modelo (s) da folha de placa de origem . Nomes de plasmídeo e volume mínimo para preencher os poços são indicados. Da mesma forma, os volumes de mistura de reagentes de transfecção que, em seguida, terão de ser preenchidos nos seguintes poços são indicados como TR e destacados em verde.
3. preparação da placa da fonte do ADN usando a aplicação de guia de pipetagem 384-well
- Diluir o plasmídeo de DNA armazenado do passo 1.2.14 para 100 ng/μL usando água destilada.
- Calibre a grelha 384-well nas dimensões da chapa: Abra a aplicação de guia de pipetagem 384-well num comprimido (Figura 3). Coloque a placa de origem na grade na tela inferior e, no menu de calibração superior esquerdo, clique em + ou - (ou use o cursor vermelho) para aumentar ou reduzir o tamanho da grade e poços para ajustar os poços verdes aos quatro poços de canto da placa .

Figura 3 : Utilização da aplicação de guia de pipetagem 384-well. (1) calibração da grade 384-well ao tamanho da placa; (2)) montagem de um adaptador de placa 3D universal impresso para o Tablet usando fita dupla face; (3) colocação da placa no adaptador; (4) deslocamento da grelha para CentrÃ-lo na placa montada. (5) bloqueio da etapa de calibração. (6) abertura do arquivo Guide. csv de pipetagem de 384 poços. (7) dada a lista de arquivos, o aplicativo indicará o nome da placa fonte esperada, reagente (DNA ou reagente de transfecção), a concentração e o volume para dispensar os poços alvo, que serão iluminados um por um. (8) as teclas de seta esquerda e direita permitem que o usuário siga o guia de pipetagem para dispensar facilmente os reagentes de acordo com o modelo (s) da placa de fonte macro da planilha. Por favor clique aqui para ver uma versão maior desta figura.
- Usando fita dupla face, monte o adaptador de placa impressa em 3D na tela para evitar movimentos de placa de origem durante a dispensação. Se necessário, mova a grade calibrada usando as setas de rotação e botões para cima/para baixo/direita/esquerda para ajustar a grade na tela para a posição da placa. Uma vez que a grade e os tamanhos de poço são calibrados corretamente e situados, assinale a caixa da calibração do fechamento .
- Clique em arquivo e abra o arquivo 384 Wells pipetagem Guide. csv . Siga as instruções do ecrã para dispensar manualmente o volume indicado do plasmídeo indicado na concentração indicada no poço branco realçado correspondente ao destino adequado da chapa esperada. Use - ou + setas para voltar ou mais no processo de dispensação de DNA. Pare de dispensar ao alcançar a primeira solução do reagente do transfection para carregar.
- Uma vez que as dispensações do ADN são terminadas, remova a placa da fonte do adaptador. Se várias placas de origem tiverem que ser preenchidas, coloque uma nova placa de fonte no adaptador e siga as instruções de dispensação. Uma vez que a dispensação do ADN terminou, centrifugue a placa (s) da fonte DNA-enchida (em 1.500 x g por 2 minutos) para assegurar o nivelamento líquido apropriado e para remover as bolhas que conduzem à imprecisão nas transferências Ade-baseadas.
4. dispensação de diluente com base em manipulador líquido peristáltico 1 μL na placa de destino
Nota: execute as etapas 4.1-4.5 em um armário de segurança biológico.
- Desinfete a cabeça da gaveta de 1 μL pulverizando-a com um desinfetante de pulverização (tabela de materiais) e permita que esta solução entre no suporte da ponta. Absorva o desinfetante remanescente em papel absorvente. Monte a gaveta de 1 μL no dispositivo de tratamento de líquidos peristáltico. Ligue o dispositivo e certifique-se de que a definição do tipo de gaveta está correcta (1 μL), bem como o formato da chapa (384 poços).
- Desinfete todo o lúmen da tubagem: Insira o organizador do tubo (segurando os oito tubos juntos) numa embarcação estéril e encha-o com 5 mL de álcool a 70%. Usando a função de escorva do manipulador líquido peristáltico, primeiro lave o álcool na tubulação e depois enxague-o passando 5 mL de água destilada e 5 mL de meio livre de soro (o meio de Eagle modificado de Dulbecco [DMEM] suplementado com 100 U/mL penicilina-estreptomicina; Tabela de material), sucessivamente preenchendo o mesmo navio. Certifique-se de que nenhuma das pontas está obstruído visualmente inspecionando o fluxo de líquido de todos eles.
- Prime a tubagem com meio livre de soro preenchendo uma nova embarcação estéril com 10 mL de meio livre de soro pré-aquecido e mergulhando no organizador do tubo. Pressione o botão Prime do manipulador líquido peristáltico por cerca de 10 s. Mais uma vez, assegure-se de que nenhuma ponta esteja obstruído inspecionando visualmente o fluxo líquido de todos eles.
- Encha a placa com 1 μL de diluente. Coloque uma placa de cultura 384-well estéril (destino) no portador de placa de manipulador líquido peristáltico e retire a tampa.
- Execute o programa pré-calibrado para dispensar 1 μL em cada poço da placa 384-well. O tempo de dispensação é de aproximadamente 8 s. Em seguida, substitua a tampa da placa 384-well.
Nota: Alternativamente, esta etapa pode ser tratada manualmente, em um gabinete de segurança, usando uma micropipeta multicanal.
5. desempenho de uma pesquisa para controlar os volumes manualmente dispensados
Observação: para obter detalhes, consulte a Figura 4.
- Execute o programa nanodispenser, vá para a guia de diagnóstico, marque a placa de origem para fora da caixa, coloque a placa de origem no suporte da placa, e marque in para entrar na placa. Quando solicitado, selecione 384Ldv_aq_b2 para definir o nanodispenser para o modo de distribuição de buffer aquoso e pressione OK.
- Selecione pesquisa no menu Miscelânea e clique em Launch. Selecione os poços pré-preenchidos para analisar e clique no botão ir . Verifique se os volumes medidos correspondem aos esperados e assegure-se de que nenhum poço tenha sido carregado com volumes de mais de 12 μL, pois isso evitará transferências.
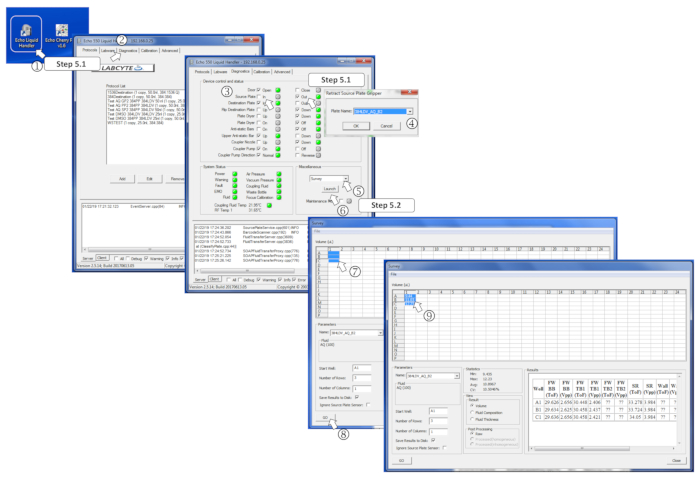
Figura 4 : Definindo os parâmetros do software de pesquisa. (1) inicie o programa nanodispenser. (2) Abra a guia diagnósticos . (3) Insira a placa de origem marcando a placa de origem e, em seguida, em. (4) defina o tipo de placa de origem no menu quando solicitado. (5) na caixa diversos, selecione pesquisa no menu suspenso. (6) inicie o programa de pesquisa clicando no lançamento. (7) Selecione os poços pré-preenchidos para medir. (8) inicie a análise clicando em ir. (9) uma vez realizada a pesquisa, os volumes medidos são escritos nos poços selecionados correspondentes. Por favor clique aqui para ver uma versão maior desta figura.
6. dispensação ADE-driven do ADN na placa do destino
- Execute o software de lista de opções, defina os tipos de placa de origem e destino 384-well para 384_LDV e Greiner 384PS_781096, respectivamente (Figura 5). Defina o dispositivo para o modo de distribuição de buffer aquoso selecionando 384ldv_aq_b2 e desmarque "otimizar o throughput de transferência".

Figura 5 : Desempenho das dispensações baseadas em picklist. (1) inicie o software nanodispenser. Na guia Protocolo, selecione (2) o formato da placa de amostra, (3) o tipo de placa de destino e (4) unticks "otimizar o throughput de transferência". (5) Selecione a guia lista de seleção . (6) clique em importar e selecione o arquivo *. csv adequado (DNA-picklist ou-lista de opções). (7) uma vez selecionado, clique em Import. (8) clique em Play e salve o protocolo. (9) realize uma simulação de dispensação clicando em simular, ou (10) inicie a dispensação programada clicando em executar. Por favor clique aqui para ver uma versão maior desta figura.
- Selecione a guia "lista de seleção", clique em importar, selecione o arquivo DNA-picklist. csv. Clique em Play e salve o protocolo. Clique em simular para realizar uma simulação das dispensações programadas para garantir que a lista de opções corresponda ao experimento experimental esperado. Depois de concluído, clique em fechar.
- Clique em Playe, em seguida, executar, para iniciar o programa de dispensação: quando solicitado, insira a placa de origem solicitada (soluções de DNA preenchidas manualmente) e a placa de destino (diluente-preenchido) no nanodispenser.
Nota: o tempo de dispensação é de aproximadamente 5-20 min para uma placa completa de 384 poços, dependendo dos volumes selecionados e do número total de dispensações no experimento. - Alternativamente, pause o protocolo aqui como as placas Diluent-e ADN-enchidas podem segurar o armazenamento seco ou congelado por até 7 dias. Para o armazenamento seco, deixe as placas secar no banco em temperatura ambiente e, em seguida, armazená-los da mesma maneira. Descongelar e centrifugar (a 1.500 x g por 2 min) placas armazenadas congeladas antes da utilização numa etapa de transfecção (secção 7).
7. dispensação do reagente do transfection ADE-driven
- Em um armário do biossegurança, extemporaneamente dilui o reagente do transfection do lipopolyplex no meio soro-livre a uma concentração final 1x. Vortex e imediatamente dispensar esta mistura de reagente de transfecção de acordo com a placa (s) de origem predefinida concebida pela macro e utilizando a aplicação de guia de pipetagem pré-calibrada 384-well, conforme descrito no passo 3,4.
Nota: Não centrifugue a placa de fonte uma vez que é carregada com o reagente como nenhuma transfecção é observada após a centrifugação. - Execute o programa nanodispenser para realizar uma "pesquisa" conforme descrito na seção 5, a fim de controlar os volumes de todos os poços de TR preenchidos manualmente da (s) chapa (ões) de origem para evitar erros de dispensação devido a volumes superiores a 12 μl.
- Clique em Redefinir para limpar a lista de amostras da lista de opções de DNA no software de lista de opções e verifique se os parâmetros do dispositivo ainda estão configurados para buffers aquosos e para os tipos de placa de origem e destino usados, como na etapa 6,1.
- Clique em importar e escolha o arquivo TR-picklist. csv. Clique em Play e salve o protocolo, se solicitado, e (isto é, opcionalmente, mas fortemente recomendado) realizar uma simulação das dispensações de mistura de reagente de transfecção programada para garantir o design adequado das dispensações, clicando no Botão simular . Depois de concluído, clique em fechar.
- Clique em Playe , em seguida, executar o botão para iniciar o programa de dispensação: conforme solicitado, coloque a placa (s) de origem (TR-mistura-preenchido) e a placa de destino (diluente-e cheia de DNA) no nanodispenser.
Nota: o tempo de dispensação é inferior a 20 min para uma placa completa de 384 poços ao dispensar 500 nL da mistura TR. - Incubar 15-30 min à temperatura ambiente após a adição do TR ao DNA, conforme indicado pelo protocolo do fabricante.
8. dispensação de célula baseada em manipulador líquido peristáltico
- Prepare o manipulador líquido peristáltico para a distribuição de células. Desinfete uma cabeça de gaveta de 10 μL pulverizando-a com Aniospray surf 29 desinfetante e absorvendo o remanescente no papel. Monte a gaveta no dispositivo de manipulador líquido peristalótico, mude a configuração do tipo de gaveta para 10 μL e certifique-se de que o formato da chapa esteja definido para 384 poços.
- Desinfete o tubo de gaveta de 10 μL como descrito anteriormente no passo 4,2. Mergulhe o organizador do tubo em uma embarcação estéril e lave a tubagem com 5 mL de álcool 70%, depois com 5 mL de água destilada e, finalmente, com 5 mL de meio livre de soro, sucessivamente preenchido na mesma embarcação e até que cada tubo esteja vazio.
- Prepare a suspensão da célula para dispensar. A partir de uma célula HeLa confluentes B10-cultura prato, lave as células 1x com 1x fosfato-tampão solução salina (PBS) e, em seguida, dissociar as células com Trypsin/EDTA para 5 min a 37 ° c.
- Verificar a dissociação celular um microscópio e interromper a ação de Trypsin/EDTA adicionando 10 mL de meio completo (DMEM suplementado com soro bovino fetal a 10% e 100 U/mL de penicilina-estreptomicina; ver a tabela de material) no prato de cultura. Colha pilhas em um tubo de 50 mL e conte as pilhas o microscópio, usando uma pilha de Malassez ou um contador de pilha automático.
- Prepare pelo menos 25 mL da suspensão da pilha de HeLa em uma concentração de 37.500 Cells/mL no meio completo (isto é, 1.500 Cells/40 μL) para uma placa completa de 384 poços, para assegurar a escorva do tubo e a dispensação de 40 μL/well.
- Para dispensar as pilhas, encha uma embarcação estéril nova com a suspensão preparada da pilha e mexa-a para evitar o sedimentação que conduz à inexatidão na densidade da pilha da dispensação. Insira o organizador do tubo nesta solução e pressione o botão Prime até que a suspensão da célula esteja começando a se liberar da cabeça dispensadora. Certifique-se que nenhum da ponta está obstruído inspecionando visualmente o fluxo líquido de todos eles, e assegure-se de que cada tubo esteja carregado com suspensão da pilha.
- Carregue o DNA e a placa de destino 384-well preenchida no transportador líquido peristalótico da placa do alimentador e remova sua tampa. Execute o programa pré-calibrado para dispensar 40 μL da suspensão da célula na placa completa de 384 poços (i.e., 1.500 células/poço). O tempo de dispensação é de cerca de 8 s. Substitua a tampa da placa 384-well.
Nota: Alternativamente, a suspensão da célula 40 μL pode ser manualmente dispensada utilizando uma micropipeta multicanal.
9. teste biológico feito encomenda (monitoração da eficiência do Transfection da pilha)
Nota: seguindo as configurações experimentais e a intenção do experimento, use os métodos necessários para luminescência, fluorescência, triagem de alto conteúdo e transcrição reversa da reacção em cadeia quantitativa da polimerase (RT-qPCR). Nesta seção do protocolo, a eficiência da transfecção celular é avaliada por microscopia de fluorescência automatizada e análise de imagem.
- Incubar a placa a 37 ° c com 5% de CO2 em uma atmosfera saturada de água e até expressão protéica adequada.
Nota: aqui, um tempo de incubação de 48 h é usado para células HeLa para monitorar a eficiência de transfecção, usando plasmíos de expressão de tdTomato e mVenus. - Retire o meio de cultura 48 h pós-transfecção invertendo a placa, adicione 30 μL/poço de formalina a 10% utilizando o manipulador líquido peristáltico (gaveta de 10 μL) e incubar durante 15 min à temperatura ambiente.
- Retire a formalina invertendo a placa; em seguida, incubar as células durante 15 min à temperatura ambiente com 0,1 ng/mL de Hoechst diluído em solução de PBS 1x.
- Lave as células 3x por 15 min com 80 μL de 1X PBS ajustado para pH = 8 a fim de recuperar o sinal de alta fluorescência perdido pelo pH 6,9 da etapa de incubação da solução de formalina.
- Usando um microscópio fluorescente automatizado, adquira imagens de duas ou três canaletas fluorescentes (Hoechst, tdtomato, e mvenus) sequencialmente com os objetivos 10x e um jogo apropriado do filtro da emissão (4 ′, 6-diamidino-2-phenylindole [DAPI], dsred, e fluoresceína isotiocianato [FITC], respetivamente).
- Para avaliar eficiências de transfecção, use o software de análise de imagem para determinar as eficiências de transfecção usando a análise de script baseada na coloração dos núcleos.
Access restricted. Please log in or start a trial to view this content.
Resultados
fIn para determinar se a tecnologia ADE poderia ser usada para um protocolo de transfecção reversa automatizada, monitoramos a eficiência da transfecção celular por microscopia de fluorescência, usando um tdTomato fluorescente vermelho expressando plasmídeo. Primeiramente visando determinar os melhores parâmetros de transfecção, diferentes volumes de diluentes e quantidades totais de DNA foram testados em cruz. O volume de diluente foi utilizado para permitir que as gotas de DNA...
Access restricted. Please log in or start a trial to view this content.
Discussão
O estabelecimento e a otimização de um método de transfecção de alta taxa de transferência precisa para uma determinada linha de célula exigem que os cientistas sigam alguns dos principais parâmetros descritos nesta seção. Nós encorajamos a começar com os valores recomendados em todo o protocolo como essas configurações otimizadas para células HeLa também provou ser eficiente para células HEK. No entanto, como os melhores parâmetros podem depender das linhas celulares e reagentes de transfecção, as co...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Os autores divulgaram um recebimento do seguinte apoio financeiro para a pesquisa, autoria e/ou publicação deste artigo: INSERM, Lille University, Lille Pasteur Institute, Conseil Régional du Nord, e PRIM-HCV1 e 2 (Pôle de Recherche Interdisciplinaire sur le Médicament), Agence Nationale de la Recherche (ANR-10-EQPX-04-01), o FEDER (12001407 (D-AL) Equipex Imaginex BioMed) e a Comunidade Européia (ERC-STG INTRACELLTB n ° 260901). Os autores desejam agradecer ao Dr. S. Moureu, Dr. B. Villemagne, Dr. R. ferru-Clément, e Dr. H. Groult por sua revisão crítica e correções do manuscrito.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| 384LDV Microplate | Labcyte | LP-0200 | |
| 384-well Microplate μClear Black | Greiner | 781906 | |
| Ampicilin | Sigma | A9393-5G | Selection antibiotic for bacteria transformed with ampicilin expressing vector |
| Android Tablet | Samsung | Galaxy Note 8 | used to guide the user while the source plate manual dispense |
| Aniospray Surf 29 | Anios | 2421073 | disinfectant to clean the MicroFlo head |
| Columbus software | Perkin Elmer | image analysis software | |
| Dulbecco's Modified Eagle Medium (DMEM), high glucose, GlutaMAX Supplement, pyruvate | Thermo Fisher Scientific | 10566032 | cell culture medium |
| Echo Cherry Pick 1.5.3 software | Labcyte | Software enabling ADE-based dispenses by the Echo550 device from a *.csv file; nanodispenser software | |
| Echo550 | Labcyte | ADE-based dispenser | |
| Fetal Bovine Serum | Thermo Fisher Scientific | 16000044 | to add in cell culture medium |
| Formalin solution, neutral buffered, 10% | Sigma-Aldrich | HT501128-4L | to fix cell |
| HeLa cells | ATCC | HeLa (ATCC® CCL-2™) | |
| Hoechst 33342, Trihydrochloride, Trihydrate | Thermo Fisher Scientific | H3570 | 10 mg/mL Solution in Water |
| INCell Analyzer 6000 | GE Healthcare | 29043323 | automated laser-based confocal imaging platform |
| LB medium | Thermoischer Scientific LB Broth Base (Lennox L Broth Base)®, powder | 12780052 | culture medium for bacteria growth |
| Lysis Buffer (A2) | Macherey-Nagel | 740912.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| MicroFlo 10µL cassette | Biotek Instruments Inc | 7170013 | to use with the Microflo Dispenser |
| MicroFlo 1μL cassette | Biotek Instruments Inc | 7170012 | to use with the Microflo Dispenser |
| MicroFlo Dispenser | Biotek Instruments Inc | 7171000 | peristaltic pump-based liquid handler device |
| Microvolume spectrophotometer | Denovix | DS-11 Spectrophotometer | Measure the DNA concentration of samples |
| mVenus plasmid | mVenus cDNA was cloned by enzymatic restriction digestion and ligation in Age1/BsrG1 sites of the tdTomato-N1 plasmid | Vector type: Mammalian Expression, Fusion Protein: mVenus | |
| Neutralization Buffer (A3) | Macherey-Nagel | 740913.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| NucleoSpin Plasmid kit | Macherey-Nagel | 740588.50 | used to prepare plasmid from bacterial culture |
| Optimal-Modified Eagle Medium (Opti-MEM) Medium | Thermo Fisher Scientific | 31985070 | |
| optional Wash bufferWash Buffer (A4) | Macherey-Nagel | 740914.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| orbital shaker | incubated large capacity shaker | 444-7084 | Used to grow bacteria under gentle agitation and 37°C |
| Penicillin-Streptomycin | Thermo Fisher Scientific | 15140122 | 10,000 U/mL |
| Phosphate-Buffered Saline | Thermo Fisher Scientific | 10010001 | |
| Plasmid mini-columns | Macherey-Nagel | 740499.250 | Silica membrane mini-column to prepare plasmid from bacterial culture |
| Resuspension Buffer (A1) | Macherey-Nagel | 740911.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| RNAse A | Macherey-Nagel | 740505 | Enzyme from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| tdTomato-N1 plasmid | Addgene | Plasmid #54642 | Vector type: Mammalian Expression, Fusion Protein: tdTomato |
| TransIT-X2 Dynamic Delivery System | Mirus Bio | MIR 6000 | |
| Wash Buffer (AW) | Macherey-Nagel | 740916.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| 3D printer | Creality | CR10S | used to print the plate adapter |
| Blender Software | https://www.blender.org/ Free software under GNU General Public License (GPL). | version 2.79b | used to design the plate adapter |
Referências
- Colin, B., Deprez, B., Couturier, C. High-Throughput DNA Plasmid Transfection Using Acoustic Droplet Ejection Technology. SLAS Discovery: Advancing Life Sciences R & D. , 2472555218803064(2018).
- Mirus Bio. Optimising Transfection Performance. , (2019).
- Thermo Fisher Scientific. Factors Influencing Transfection Efficiency | Thermo Fisher Scientific - FR. , https://www.thermofisher.com/fr/fr/home/references/gibco-cell-culture-basics/transfection-basics/factors-influencing-transfection-efficiency.html (2019).
- Boussif, O., et al. A versatile vector for gene and oligonucleotide transfer into cells in culture and in vivo: polyethylenimine. Proceedings of the National Academy of Sciences of the United States of America. 92 (16), 7297-7301 (1995).
- Figueroa, E., et al. A mechanistic investigation exploring the differential transfection efficiencies between the easy-to-transfect SK-BR3 and difficult-to-transfect CT26 cell lines. Journal of Nanobiotechnology. 15 (1), 36(2017).
- Kirchenbuechler, I., Kirchenbuechler, D., Elbaum, M. Correlation between cationic lipid-based transfection and cell division. Experimental Cell Research. 345 (1), 1-5 (2016).
- Zhang, Z., Qiu, S., Zhang, X., Chen, W. Optimized DNA electroporation for primary human T cell engineering. BMC Biotechnology. 18 (1), 4(2018).
- Cao, D., et al. Transfection activity and the mechanism of pDNA-complexes based on the hybrid of low-generation PAMAM and branched PEI-1.8k. Molecular bioSystems. 9 (12), 3175-3186 (2013).
- Bos, A. B., et al. Development of a semi-automated high throughput transient transfection system. Journal of Biotechnology. 180, 10-16 (2014).
- Colosimo, A., et al. Transfer and expression of foreign genes in mammalian cells. BioTechniques. 29 (2), 314-318 (2000).
- Villa-Diaz, L. G., Garcia-Perez, J. L., Krebsbach, P. H. Enhanced transfection efficiency of human embryonic stem cells by the incorporation of DNA liposomes in extracellular matrix. Stem Cells and Development. 19 (12), 1949-1957 (2010).
- Sabatini, D. M. Reverse transfection method. , WO2001020015A1(2001).
- Raymond, C., et al. A simplified polyethylenimine-mediated transfection process for large-scale and high-throughput applications. Methods (San Diego, CA). 55 (1), 44-51 (2011).
- Junquera, E., Aicart, E. Recent progress in gene therapy to deliver nucleic acids with multivalent cationic vectors. Advances in Colloid and Interface Science. 233, 161-175 (2016).
- Woodruff, K., Maerkl, S. J. A High-Throughput Microfluidic Platform for Mammalian Cell Transfection and Culturing. Scientific Reports. 6, 23937(2016).
- Vasileiou, T., Foresti, D., Bayram, A., Poulikakos, D., Ferrari, A. Toward Contactless Biology: Acoustophoretic DNA Transfection. Scientific Reports. 6, 20023(2016).
- Hadimioglu, B., Stearns, R., Ellson, R. Moving Liquids with Sound: The Physics of Acoustic Droplet Ejection for Robust Laboratory Automation in Life Sciences. Journal of Laboratory Automation. 21 (1), 4-18 (2016).
- Grant, R. J., et al. Achieving accurate compound concentration in cell-based screening: validation of acoustic droplet ejection technology. Journal of Biomolecular Screening. 14 (5), 452-459 (2009).
- Sackmann, E. K., et al. Technologies That Enable Accurate and Precise Nano- to Milliliter-Scale Liquid Dispensing of Aqueous Reagents Using Acoustic Droplet Ejection. Journal of Laboratory Automation. 21 (1), 166-177 (2016).
- Day, R. N., Davidson, M. W. The fluorescent protein palette: tools for cellular imaging. Chemical Society Reviews. 38 (10), 2887-2921 (2009).
- Zielinski, D., Gordon, A., Zaks, B. L., Erlich, Y. iPipet: sample handling using a tablet. Nature Methods. 11 (8), 784-785 (2014).
- Brunner, S., et al. Cell cycle dependence of gene transfer by lipoplex, polyplex and recombinant adenovirus. Gene Therapy. 7 (5), 401-407 (2000).
- Nii, T., et al. Single-Cell-State Culture of Human Pluripotent Stem Cells Increases Transfection Efficiency. BioResearch Open Access. 5 (1), 127-136 (2016).
- Noonan, D. J., Henry, K., Twaroski, M. L. A High-Throughput Mammalian Cell-Based Transient Transfection Assay. Signal Transduction Protocols. 284, 051-066 (2004).
- Transfection | TransIT Transfection Reagents | Mirus Bio. , https://www.mirusbio.com/products/transfection (2015).
- American Type Culture Collection. General protocol for transfection of stem cells, primary cells, and continuous cell lines with ATCC TransfeX Transfection Reagent. , https://www.atcc.org/~/media/Transfection%20protocols/TransfeX/TransfeX%20General%20Protocol%20on%20letterhead.ashx (2017).
- American Type Culture Collection. Transfection Reagents for Nucleic Acid Transfer into ATCC Cells. , http://www.lgcstandards-atcc.org/~/media/PDFs/Marketing%20Material/Cell%20Biology/Transfection%20reagents%20for%20nucleic%20acid.ashx (2017).
- de Los Milagros Bassani Molinas, M., Beer, C., Hesse, F., Wirth, M., Wagner, R. Optimizing the transient transfection process of HEK-293 suspension cells for protein production by nucleotide ratio monitoring. Cytotechnology. 66 (3), 493-514 (2014).
- Promega. FuGENE® 6 Transfection Reagent. , https://www.promega.com/-/media/files/resources/protocols/technical-manuals/101/fugene-6-transfection-reagent-protocol.pdf (2019).
- Olden, B. R., Cheng, Y., Yu, J. L., Pun, S. H. Cationic polymers for non-viral gene delivery to human T cells. Journal of Controlled Release: Official Journal of the Controlled Release Society. 282, 140-147 (2018).
- Park, E., Cho, H. B., Takimoto, K. Effective gene delivery into adipose-derived stem cells: transfection of cells in suspension with the use of a nuclear localization signal peptide-conjugated polyethylenimine. Cytotherapy. 17 (5), 536-542 (2015).
- Wood, R. W., Loomis, A. L. The physical and biological effects of high-frequency sound-waves of great intensity. The London, Edinburgh, and Dublin Philosophical Magazine and Journal of Science. 4 (22), 417-436 (1927).
- Eliminating Endotoxin at the Source - A Novel Competent Cell Line with Modified Lipopolysaccharide for Low-Endotoxin Plasmid Production. Mamat, U., et al. 28th Annual Symposium of the Protein Society, San Diego, CA, , (2014).
- Ivanova, N. V., Kuzmina, M. L. Protocols for dry DNA storage and shipment at room temperature. Molecular Ecology Resources. 13 (5), 890-898 (2013).
- Lesnick, J., Lejeune-Dodge, A., Ruppert, N., Jarman, C. High-Precision Cell Dispensing with the Labcyte Echo® Liquid Handler. , https://www.labcyte.com/content/applications/high-precision-cell-dispensing-with-the-labcyte-echo-liquid-handler (2017).
- Yang, X., et al. A public genome-scale lentiviral expression library of human ORFs. Nature Methods. 8 (8), 659-661 (2011).
- Peng, J., Zhou, Y., Zhu, S., Wei, W. High-throughput screens in mammalian cells using the CRISPR-Cas9 system. The FEBS journal. 282 (11), 2089-2096 (2015).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados