Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Analyse et spécification des distributions de taille granule d’amidon
Dans cet article
Résumé
Présenté ici est une procédure pour les déterminations reproductibles et statistiquement valides des distributions de taille granule d’amidon, et pour spécifier les distributions déterminées de taille lognormal de granule utilisant une forme multiplicative à deux paramètres. Il s’applique à toutes les analyses de dimensionnement granulés d’échantillons d’amidon à l’échelle graminale pour la recherche en sciences végétales et alimentaires.
Résumé
L’amidon de toutes les sources végétales est composé de granulés dans une gamme de tailles et de formes ayant des fréquences d’occurrence différentes, c’est-à-dire présentant une taille et une répartition desformes. Les données sur la taille des granules d’amidon déterminées à l’aide de plusieurs types de techniques de dimensionnement des particules sont souvent problématiques en raison d’une faible reproductibilité ou d’un manque de signification statistique résultant de certaines erreurs systématiques insurmontables, y compris la sensibilité aux formes granuleuses et aux limites des tailles des échantillons de granules. Nous avons décrit une procédure pour les déterminations reproductibles et statistiquement valides des distributions de taille granule d’amidon utilisant la technique de zone de détection électrique, et pour spécifier les distributions déterminées de taille lognormal de granule utilisant une forme multiplicative adoptée à deux paramètres avec une précision et une comparabilité améliorées. Il s’applique à toutes les analyses de dimensionnement granuleux d’échantillons d’amidon à l’échelle graminée et, par conséquent, pourrait faciliter les études sur la façon dont la taille des granules d’amidon est moulée par l’appareil et les mécanismes de biosynthèse de l’amidon; et comment ils ont un impact sur les propriétés et la fonctionnalité des amidons pour les utilisations alimentaires et industrielles. Les résultats représentatifs sont présentés à partir d’analyses de reproduction des distributions granulées d’échantillons d’amidon de la patate douce à l’aide de la procédure décrite. Nous avons en outre discuté de plusieurs aspects techniques clés de la procédure, en particulier, la spécification multiplicative des distributions de taille lognormal granule et certains moyens techniques pour surmonter le blocage fréquent de l’ouverture par granulés.
Introduction
Les granules d’amidon sont la structure physique dans laquelle deux polymères homoglucanes de réserve principaux dans les tissus de photosynthèse et de stockage des plantes, l’amylose linéaire ou peu ram ram ramée et l’amylopectine fortement ram ramée, sont emballés de façon ordonnée avec quelques composants mineurs, y compris les lipides et les protéines. Les granules d’amidon de diverses espèces végétales présentent de nombreuses formes tridimensionnelles (3D) (examinées en réf.1,2),y compris des sphères, des ellipsoides, des polyèdres, des plaquettes, des cubes, des cuboïdes et des tubules irréguliers. Même ceux du même tissu ou de tissus différents d’une même espèce végétale pourraient avoir un ensemble de formes avec des fréquences d’occurrence variables. En d’autres termes, les granules d’amidon d’une espèce végétale peuvent avoir une répartition statistique caractéristique de la forme, plutôt qu’une forme spécifique. Les formes granuleuses non uniformes et non sphériques rendent difficile la mesure et la définition adéquates de la taille des granules d’amidon. En outre, les granules d’amidon provenant des mêmes tissus d’une espèce végétale sont d’une gamme de tailles avec des proportions différentes, c’est-à-dire présentant une répartition caractéristique de la taille. Cette distribution de taille complique encore l’analyse et la description des tailles de granules d’amidon.
La taille des granules d’amidon a été analysée à l’aide de plusieurs catégories de techniques de dimensionnement des particules (examinées àl’réf. 3),y compris la microscopie, la sédimentation/fractionnement du débit de champ stérique (Sd/StFFF), la diffraction laser et la zone de détection électrique (ESZ). Cependant, ces techniques ne sont pas également adaptées à la détermination des tailles de granules d’amidon en présence d’une forme granule et d’une distribution de taille. La microscopie, y compris la microscopie électronique légère, confoccale et à balayage, est excellente pour les études de morphologie4,5,6,7, structure8,9 et développement10,11 de granules d’amidon, mais à peine adapté pour définir leurs distributions de taille en raison de certaines lacunes inhérentes. Les mesures directes d’images microscopiques de granules ou d’analyses d’images assistées par logiciel de données de microscopie optique (IAOM), qui ont été utilisées pour la détermination de la taille des granules d’amidons de plusieurs espèces, dontle maïs 12,le blé 13,le 14,la pomme de terre 15 et l’orge 16,ne peuvent mesurer que des images 1D (généralement de longueur maximale) ou 2D (surface) de nombres très limités (de dizaines à quelques milliers) d’images de granule d’amidon. Les petites tailles d’échantillonnage de granules qui sont intrinsèquement limitées par les techniques pourraient rarement être statistiquement représentatives, compte tenu des énormes nombres de granules par unité de poids de l’amidon (~120 x10 6 par gramme, en supposant que les sphères de 10 μm à 1,5 g/cm³ de densité) et, par conséquent, pourraient conduire à une mauvaise reproductibilité des résultats. La technique Sd/StFFF peut avoir une vitesse et une résolution élevées, et des fractions de taille étroite de granules d’amidon17, mais a été rarement utilisé probablement parce que sa précision pourrait être gravement affectée par des dommages, des formes différentes, et la densité des granules d’amidon. La technique de diffraction laser est la plus largement utilisée, et a été appliquée pour des analyses de taille granule d’amidon pour toutes les grandes espècesde cultures 3,14,16. Bien que la technique présente de nombreux avantages, elle n’est en fait pas adaptée à la détermination de la taille des granules d’amidon en présence d’une distribution de forme granulée. La plupart des instruments simultanés de diffraction laser s’appuient sur la théorie mie de diffusion de lalumière 18 pour les particules sphériques uniformes et sur la théorie modifiée de Mie18 pour d’autres formes d’uniformité. La technique est donc intrinsèquement très sensible aux formes de particules, et n’est pas tout à fait adaptée, même pour certaines formes d’uniformité19, et encore moins pour les granules d’amidon ayant un ensemble de formes de proportions différentes. La technique ESZ mesure la perturbation du champ électrique proportionnelle au volume de la particule passant par une ouverture. Il fournit des tailles de volume granulés, ainsi que les informations de distribution de nombre et de volume, etc., à haute résolution. Étant donné que la technique ESZ est indépendante des propriétés optiques des particules, y compris la couleur, la forme, la composition ou l’indice réfractif, et que les résultats sont très reproductibles, elle est particulièrement adaptée pour déterminer les distributions de taille des granules d’amidon ayant un ensemble de formes.
La taille des granules d’amidon a également été définie à l’aide de nombreux paramètres. Ils ont souvent été décrits de façon simpliste par diamètres moyens, qui dans certains cas étaient les moyens arithmétiques des longueurs maximales microscopiquement mesurées des images 2D12,20, ou moyennes de diamètres de sphère équivalents3. Dans d’autres cas, les distributions de la taille du granule ont été spécifiées en utilisant des plagesde taille 21,22, le volume moyen de distribution ou le diamètre moyen (équivalent sphère, pondéré par nombre, volume ou surface) en supposant une distribution normale14,23,24,25,26. Ces descripteurs de granules d’amidon provenant de diverses analyses sont d’une nature très différente, et pas strictement comparables. Il pourrait être très trompeur si ces « tailles » de granules d’amidon de différentes espèces ou même les mêmes tissus de la même espèce ont été directement comparés. De plus, le paramètre de propagation (ou de forme) des distributions normales présumées, c’est-à-dire l’écart type σ (ou écart type graphique σg)mesurant la largeur de la distribution (c.-à-d. la propagation des tailles), a été ignoré dans la plupart des études.
Afin de résoudre les problèmes critiques susmentionnés liés aux analyses de dimensionnement du granule d’amidon, nous avons décrit une procédure de détermination reproductible et statistiquement valide des distributions de granules des échantillons d’amidon à l’aide de la technique ESZ, et pour bien spécifier les distributions de taillenormales de granules déterminées à l’aide d’une forme multiplicative à deux paramètresadoptée 27 avec une précision et une comparabilité améliorées. Pour la validation et la démonstration, nous avons effectué des analyses de dimensionnement de granules de réplication d’échantillons d’amidon de sweetpotato à l’aide de la procédure, et spécifié les distributions différentielles lognormal volume-pourcentage volume-équivalent-sphère diamètre en utilisant leurs moyens 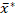 géométriques graphiques et multiplicatifs écarts types s* dans
géométriques graphiques et multiplicatifs écarts types s* dans 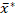 un x/ (multiplier et diviser) s* forme.
un x/ (multiplier et diviser) s* forme.
Protocole
1. Préparation d’échantillons d’amidon
- Préparer deux (ou trois) échantillons d’amidon de reproduction à l’échelle du gramme provenant de tissus accumulant de l’amidon de diverses espèces végétales selon les procédures établies (p. ex., pommesde terre 15,patatesdouces 28,grainsde blé 13,29et grains demaïs 30,etc.).
- Lavez soigneusement les échantillons d’amidon avec de l’acétone ou du toluène 3-4x pour minimiser les granulés et les sécher complètement.
REMARQUE : Utilisez des procédures d’extraction qui produisent plus de 1 g d’amidon par préparation. Un ou deux aliquots de 0,5 g de chacun des trois ou deux extraits de reproduction, respectivement, sont échantillonnés pour l’analyse de dimensionnement de granule d’un extrait d’amidon.
2. Préparation d’électrolyte
- Préparer 500 mL de chlorure de lithium de 50 g/L dans le méthanol pour quatre séries de dimensionnement pour les échantillons d’amidon de réplication (100 mL par course plus 100 mL supplémentaires). De préférence, faire l’électrolyte en lots à grand volume, par exemple, 4 à 8 L à la fois, afin de minimiser la variation de concentration.
- Refroidir le récipient sur de la glace ou dans une armoire à 4 °C pour accélérer la dissolution du chlorure de lithium.
3. Mise en place de l’analyseur
- Choisissez un tube d’ouverture avec une plage de diamètre de particule couvrant la gamme connue (dans la littérature ou par des essais) de taille granule d’échantillons d’amidon à analyser, par exemple, une ouverture de 100 μm pour les amidons de la patate douce. Pour les échantillons d’amidon de taille granule inconnue, sélectionnez une ouverture appropriée par le biais de courses d’essai à l’aide de plusieurs tubes d’ouverture ayant des plages de diamètre de particules qui se chevauchent.
REMARQUE : La plage de diamètre des particules d’un tube d’ouverture est sa plage de dimensionnement précise entre 2 et 60 % et avec une plage de dimensionnement étendue à 80 % de son diamètre d’orifice. Le tableau 1 énumère les propriétés de trois tubes d’ouverture les plus utiles pour dimensionner les granules des amidons des grandes cultures. Si la gamme de taille granuleuse d’un échantillon d’amidon est plus large que la plage de dimensionnement d’un seul tube d’ouverture, effectuez une analyse de chevauchement multi-tube combinant jusqu’à cinq distributions de taille de particules mesurées avec des ouvertures de différentes tailles. Chaque ouverture est identifiable par son diamètre et son numéro de pièce étiqueté sur le tube. Son diamètre et son numéro de série contenus dans un code à barres sur le tube peuvent être numérisés dans le logiciel de l’analyseur à l’aide du lecteur de code à barres sur le panneau de contrôle de l’analyseur. - Choisissez un bécher analytique de 100 ou 200 mL (au-dessus des cuvettes) pour la détermination de la taille des granules d’amidon, et installez l’agitation automatique (ci-dessous) pour maintenir une bonne suspension granuleuse pendant la mesure.
- Créez une méthode d’exploitation standard (SOM) pour spécifier les paramètres d’exécuter et un fichier préférences pour analyser, visualiser et imprimer les résultats. Combinez le fichier SOM et Préférences dans une procédure d’exploitation standard (SOP) au besoin.
REMARQUE : Pour les analyses non standardisables, utilisez SOM pour exécuter les analyses et ajustez les paramètres SOM entre les courses via la fenêtre SOM (voir ci-dessous) au besoin. Après l’achèvement de l’exécuteur, analysez, visualisez et imprimez les résultats de l’exécuteur en changeant les préférences comme vous le souhaitez. Pour les analyses standardisables de dimensionnement des granulés, utilisez un SOP pour exécuter les analyses.- Démarrez le logiciel d’analyseur. Sur le Manu principal, cliquez sur SOP | Créez SOM Wizard ou Modifiez le SOM, ou sur le panneau d’état, cliquez sur Modifier SOM. Utilisez l’assistant ou la fenêtre Modifier la fenêtre SOM pour sélectionner les paramètres d’un SOM. Les paramètres généralement utilisés pour dimensionner les granules d’échantillons d’amidon de sweetpotato sont résumés dans le tableau 2.
- Enregistrez le SOM créé dans un fichier dans la fenêtre SOM Wizard-Summary of Settings ou dans la fenêtre Modifier la fenêtre SOM.
- Sur le Manu principal, cliquez sur SOP | Créer des préférences Assistant ou Modifier les préférences. Utilisez l’assistant ou les onglets de la fenêtre modifier les préférences pour sélectionner les paramètres de préférence comme ceux du tableau 3 ou d’autres comme vous le souhaitez.
- Enregistrez les préférences sélectionnées pour un fichier dans la fenêtre Créer des préférences Wizard-Summary of Settings ou dans la fenêtre Préférences d’édition.
- Sur le menu principal, cliquez sur SOP | Créer SOP Wizard. En suivant le guide étape par étape de l’assistant, entrez une description, sélectionnez le fichier SOM et Préférences pour créer et enregistrer un SOP.
4. Analyses de dimensionnement granule des échantillons d’amidon
- Préparer l’analyseur
- Allumez l’analyseur, ouvrez le logiciel dans l’ordinateur et vérifiez l’état Ready en haut du panneau d’état après sa connexion automatique à l’analyseur.
- Remplir le pot d’électrolyte d’électrolyte, vider le pot à déchets si nécessaire.
- Installez et fixez correctement le tube d’ouverture choisi en suivant le guide dans le manuel de l’utilisateur. Pour un nouveau tube d’ouverture non calibré, calibrez-le en suivant le guide étape par étape sous Calibration | Ouverture calibrée sur le menu principal. Pour un tube d’ouverture calibré, vérifiez l’étalonnage en suivant le guide étape par étape de l’assistant de tube d’ouverture de changement sous la course ou l’étalonnage | Vérifiez l’étalonnage d’ouverture sur le menu principal.
- Déverrouillez la plate-forme d’analyse en poussant le clip de verrouillage-libération (sur le devant central du mur gauche de compartiment d’échantillon) et abaissez manuellement la plate-forme vers le bas. Placez un bécher analytique contenant 100 mL d’électrolyte sur la plate-forme, déplacez l’agitateur à la position d’agitation et soulevez manuellement la plate-forme à la position supérieure auto-verrouillage pour immerger le tube d’ouverture et l’agitateur dans l’électrolyte.
- Cliquez sur Remplir sur la barre d’outils de l’instrument inférieur pour que l’analyseur remplisse automatiquement le système avec l’électrolyte et cliquez sur Flush pour que l’analyseur rince automatiquement le système.
- Chargez le SOM en cliquant sur SOP | chargez un SOM sur le menuprincipal, et utilisez le SOM pour exécuter une analyse sans fichier préférences. Alternativement, chargez un SOP en cliquant sur SOP | Chargez un SOP sur le menu principal ou chargez le SOP sur le panneau d’état, et utilisez le SOP pour exécuter une analyse.
- Si vous utilisez un SOP, cliquez sur SOP | som info ou des informations de préférence sur le menu principal pour vérifier les paramètres SOM et Préférence. Cliquez sur Exemple | Entrez des informations d’échantillon sur le menu principal ou modifiez des informations sur le panneau d’état pour saisir les informations de l’échantillon pour l’exécuter.
- Préparer l’échantillon d’amidon-méthanol et dimensionner les suspensions
- Pesez deux ou un échantillon de 0,5 g de chacun des deux ou trois extraits d’amidon répliqués, respectivement.
- Ajouter chacun des aliquots d’amidon de 0,5 g au méthanol de 5 mL dans un tube de centrifugeuse conique de 50 mL, et disperser complètement les granules d’amidon à l’aide de plusieurs impulsions d’ultrasons de faible intensité (12-24 W/cm2) à partir d’un processeur ultrasonique.
- À l’aide d’une pipette de transfert jetable, appliquer une petite goutte de suspension amidon-méthanol (~0,2 mL) sur les 100 mL d’électrolyte de méthanol LiCl de 50 g/L en remuant constamment dans le bécher. Fermez la porte du compartiment échantillonné.
- Effectuer une course de dimensionnement
- Cliquez sur Aperçu dans la barre d’outils de l’instrument en bas pour démarrer un aperçu. Sur le panneau d’état, vérifiez que la barre de concentration affichée dynamiquement est en vert et affiche une plage de concentration nominale de 5 à 8 % pour la suspension.
- Cliquez sur Arrêtez-vous sur la barre d’outils inférieure pour arrêter l’exécuter Preview. Si nécessaire, diluer la suspension amidon-électrolyte en remplaçant un aliquot de la suspension par l’électrolyte, puis répéter une course Preview.
REMARQUE : La plage de concentration nominale de 5 % à 8 % de la suspension est essentielle à l’achèvement d’une course sans arrêt en raison d’un blocage de l’ouverture par granules agrégés. Au besoin, ajustez la taille de l’échantillon de goutte et/ou la concentration de la suspension amidon-méthanol pour faire une nouvelle suspension amidon-électrolyte ayant la concentration nominale dans la plage optimale. - Après la vérification, cliquez sur Démarrer sur la barre d’outils inférieure pour démarrer l’exécuter. L’analyseur termine automatiquement la course une fois que le nombre total de granules de taille, qui est affiché avec le temps d’exécuteur sur le panneau d’état dans une course, atteint le nombre total défini (125.000 ou 250.000) par le mode de contrôle de la SOM. Selon la concentration de suspension (dans une plage de 5-8% ou moins), une seule course prend 2 à 5 min ou plus.
REMARQUE : Lorsque l’analyseur détecte automatiquement un blocage d’ouverture par zone de détection de blocage du SOM, il interrompt la course, rince pour débloquer l’ouverture et démarrer une nouvelle course. Cette action de blocage est réglée pour répéter au maximum pendant quatre fois avant que l’analyseur annule l’opération d’exécuter. Ce problème de blocage par interruption peut être surmonté en utilisant deux méthodes techniques, comme indiqué dans le tableau 2 et détaillé dans la discussion. - Si nécessaire, effectuez une répétition technique (voir tableau 2 et détaillée dans Discussion) à l’aide de la même suspension amidon-électrolyte en cliquant simplement sur Démarrer ou répéter sur la barre d’outils inférieure.
- Après l’achèvement d’une course ou d’une répétition, videz le bécher, rincez-le avec du méthanol et remplissez-le d’une solution d’électrolyte frais de 100 mL pour la prochaine course.
- Au cours d’une exécution, si un dialogue de notification de plage de taille étendue apparaît lorsque le nombre de granules de plus de 60 μm dépasse 0,1 % du nombre total (par paramètre SOM), cliquez sur Exécuter 60 % à 80 % pour exécuter une plage de dimensionnement dynamique étendue à 80 % du diamètre de l’ouverture.
REMARQUE : Le réglage de la plage de taille étendue contrôle les actions pour les granulés de plus de 60 % du diamètre de l’ouverture (100 μm, dans ce cas). Le réglage du SOM précise l’inclusion de granules d’amidon de plus de 60 μm lorsque leur dénombrement atteint plus de 0,1 % du nombre total. L’achèvement de la course est toujours contrôlé par le nombre total, et peut prendre un peu moins de temps qu’autrement sans l’inclusion des granules plus grands totalisant moins de 0,1% (quantité présumée statiquement insignifiante) du nombre total.
- Analyser les résultats de l’exécuter
- Si un SOM a été utilisé pour contrôler les tirages, sélectionnez les paramètres préférences comme vous le souhaitez pour la visualisation, l’impression et les analyses statistiques des résultats à l’aide de l’assistant Créer des préférences ou des préférences d’édition sous le SOP sur le menu principal.
- La superposition résulte de plusieurs séries sur un seul graphique à comparer.
- Cliquez sur Superposition sur la barre d’outils principale ou le fichier | Superposition sur le menu principal pour accéder à la fenêtre Superposition. Accédez et sélectionnez plusieurs fichiers de résultats souhaités dans la boîte Fichiers, cliquez sur Ajouter pour les déplacer vers la case Fichiers sélectionnés, et cliquez sur OK pour superposer les résultats sélectionnés sur un seul graphique.
- Pour ajouter un fichier à une superposition ouverte, cliquez sur RunFile | Ouvrez pour superposition sur le menu Exécuter pour accéder à la fenêtre Superposition, naviguer vers le fichier désiré, et cliquez pour ajouter.
- Résultats moyens des analyses de reproduction (2 extraits x 2 prélèvements d’amidon ou 3 extraits x 1 échantillon d’amidon), et afficher ou imprimer la distribution moyenne de la taille du granule et les statistiques dans une liste ou un graphique.
- Sur le menu principal, cliquez sur File | FileTool | Moyenne pour ouvrir la fenêtre Moyenne. Accédez et sélectionnez plusieurs fichiers de résultats souhaités dans la boîte Fichiers, cliquez sur Ajouter pour les déplacer vers la case Fichiers sélectionnés, et cliquez sur OK pour faire la moyenne des résultats sélectionnés et afficher la moyenne sur un seul graphique.
- Pour inclure un fichier de résultat supplémentaire dans une distribution moyenne, sur le menu Exécuter, cliquez sur RunFile | Open et Ajouter à la moyenne pour ouvrir la fenêtre Ajouter à la moyenne, naviguer et ajouter le fichier. La nouvelle moyenne apparaît sur le graphique de la fenêtre ou de la liste Run (résultat).
5. Spécifier la distribution moyenne
- Dans la fenêtre Run-Menu affichant la distribution moyenne, cliquez sur Calculer | Statistiques moyennes sur le menu Exécuter pour ouvrir la fenêtre de résumé des statistiques, qui affiche les statistiques moyennes en lignes, et les statistiques graphiques pour la distribution moyenne dans les colonnes.
- Utilisez la moyenne géométrique graphique (
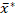 ) et S.D. (s*) dans la colonne de statistiques graphique pour spécifier la distribution moyenne dans
) et S.D. (s*) dans la colonne de statistiques graphique pour spécifier la distribution moyenne dans 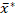 le x/ s* forme. Calculer les variations de mesure du CV entre les distributions moyennes de répliques en divisant la moyenne (μ, la même que la distribution moyenne) des moyens
le x/ s* forme. Calculer les variations de mesure du CV entre les distributions moyennes de répliques en divisant la moyenne (μ, la même que la distribution moyenne) des moyens 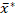 géométriques des distributions moyennes avec le S.D. moyen (σ) indiqué dans la ligne moyenne des statistiques.
géométriques des distributions moyennes avec le S.D. moyen (σ) indiqué dans la ligne moyenne des statistiques.
REMARQUE : Le S.D. moyen (pour μ) évaluant les variations entre les moyens des distributions de répétition est différent de la S.D. géométrique graphique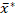 (pour) mesurant l’écart de la distribution moyenne.
(pour) mesurant l’écart de la distribution moyenne.
Résultats
Pour valider la procédure, et démontrer la reproductibilité de la distribution déterminée de taille de granule, nous avons exécuté reproduire des analyses de dimensionnement des échantillons d’amidon de sweetpotato. Nous avons préparé des échantillons d’amidon de reproduction (S1 et S2) provenant de patates douces cultivées en champ d’une lignée de reproduction SC1149-19 à un âge de développement similaire à l’aide d’une procédureprécédemment décrite 28. De chaque ex...
Discussion
La procédure décrite a résolu certains problèmes critiques dans plusieurs méthodes existantes pour les analyses de la taille des granules d’amidon, y compris le dimensionnement 1D ou 2D inapproprié des granules 3D, la distorsion des mesures de dimensionnement en raison de formes granulés non uniformes, une faible reproductibilité et une validité statistique douteuse en raison de tailles limitées échantillon granuleux, spécification inexacte ou incorrecte (en particulier l’utilisation de la taille moyenne)...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer
Remerciements
Ces travaux sont en partie appuyés par le Cooperative Agriculture Research Center et le Integrated Food Security Research Center du College of Agriculture and Human Sciences de l’Université Prairie View A&M. Nous remercions Hua Tian pour son soutien technique.
matériels
| Name | Company | Catalog Number | Comments |
| Analytical beaker | Beckman Coulter Life Sciences | A35595 | Smart-Technology (ST) beaker |
| Aperture tube, 100 µm | Beckman Coulter Life Sciences | A36394 | For the MS4E |
| Disposable transfer pipettor, | Fisher Scientific (Fishersci.com) | 13-711-9AM | Other disposable transfer pipettors with similar orifice can also be used. |
| Fisherbrand Conical Polypropylene Centrifuge Tubes, 50 ml | Fisher Scientific (Fishersci.com) | 05-539-13 | Any other similar types of tubes can be used. |
| Glass beakers, 150 to 250 ml | Fisher Scientific (Fishersci.com) | 02-540K | These beakers are used to contain methanol for washing the aperture tube and stirrer between runs. |
| LiCl | Fisher Chemical | L121-100 | |
| Methanol | Fisher Chemical | A412-500 | Buy in bulk as the analysis uses a large quantity of methanol. |
| Mettler Toledo ML-T Precision Balances | Mettler Toledo | 30243412 | Any other precision balance with a readability 0.01 g to 1 mg will work. |
| Multisizer 4e Coulter Counter | Beckman Coulter Life Sciences | B23005 | The old model, Multisizer 3 can also be used with slight adjustment of parameters. The 4e model comes with a 100 μm aperture tube. Other aperture tubes of different diameter can be purchased separately from the company. |
| Ultrasonic processor UP50H | Hielscher Ultrasound Technology | UP50H | Other laboratory sonicator having a low-power (<50 Watt) output can be also used. Both MS1 and MS2 sonotrodes for the particular sonicator can be used to disperse starch granules in 5 ml methanol. Always use the lowest setting first, 20% amplitude and 0.1 or 0.2 cycle, and raise the setting if aggregates persist in suspension. |
Références
- Shannon, J. C., Garwood, D. L., Boyer, C. D., BeMiller, J., Whistler, R. . Starch:Chemistry and Technology Food Science and Technology. , 23-82 (2009).
- Singh, N., Singh, J., Kaur, L., Singh Sodhi, N., Singh Gill, B. Morphological, thermal and rheological properties of starches from different botanical sources. Food Chemistry. 81 (2), 219-231 (2003).
- Lindeboom, N., Chang, P. R., Tyler, R. T. Analytical, biochemical and physicochemical aspects of starch granule size, with emphasis on small granule starches: a review. Starch - Stärke. 56 (34), 89-99 (2004).
- Baldwin, P. M., Davies, M. C., Melia, C. D. Starch granule surface imaging using low-voltage scanning electron microscopy and atomic force microscopy. International Journal of Biological Macromolecules. 21 (1-2), 103-107 (1997).
- Jane, J. L., Kasemsuwan, T., Leas, S., Zobel, H., Robyt, J. F. Anthology of starch granule morphology by scanning electron microscopy. Starch-Stärke. 46 (4), 121-129 (1994).
- Matsushima, R., Nakamura, Y. . Starch: Metabolism and Structure. , 425-441 (2015).
- Wang, S. -. q., Wanf, L. -. l., Fan, W. -. h., Cao, H., Cao, B. -. s. Morphological analysis of common edible starch granules by scanning electron microscopy. Food Science. 32 (15), 74-79 (2011).
- Baldwin, P. M., Adler, J., Davies, M. C., Melia, C. D. Holes in starch granules: confocal, SEM and light microscopy studies of starch granule structure. Starch-Stärke. 46 (9), 341-346 (1994).
- Chakraborty, I., Pallen, S., Shetty, Y., Roy, N., Mazumder, N. Advanced microscopy techniques for revealing molecular structure of starch granules. Biophysical Reviews. 12 (1), 105-122 (2020).
- Bechtel, D. B., Wilson, J. D. Amyloplast formation and starch granule development in hard red winter wheat. Cereal Chemistry. 80 (2), 175-183 (2003).
- Evers, A. Scanning electron microscopy of wheat starch. III. Granule development in the endosperm. Starch-Stärke. 23 (5), 157-162 (1971).
- Wang, Y. J., White, P., Pollak, L., Jane, J. L. Characterization of starch structures of 17 maize endosperm mutant genotypes with Oh43 inbred line background. Cereal Chemistry. 70, 171-179 (1993).
- Peng, M., Gao, M., Abdel-Aal, E. S. M., Hucl, P., Chibbar, R. N. Separation and characterization of A-and B-type starch granules in wheat endosperm. Cereal Chemistry. 76, 375-379 (1999).
- Wilson, J. D., Bechtel, D. B., Todd, T. C., Seib, P. A. Measurement of wheat starch granule size distribution using image analysis and laser diffraction technology. Cereal Chemistry. 83 (3), 259-268 (2006).
- Liu, Q., Weber, E., Currie, V., Yada, R. Physicochemical properties of starches during potato growth. Carbohydrate Polymers. 51 (2), 213-221 (2003).
- Chmelik, J., et al. Comparison of size characterization of barley starch granules determined by electron and optical microscopy, low angle laser light scattering and gravitational field-flow fractionation. Journal of the Institute of Brewing. 107 (1), 11-17 (2001).
- Moon, M. H., Giddings, J. C. Rapid separation and measurement of particle size distribution of starch granules by sedimentation/steric field-flow fractionation. Journal of Food Science. 58 (5), 1166-1171 (1993).
- Wriedt, T., Wolfram, H., Wriedt, T. . The Mie Theory: Basics and Applications. , 53-71 (2012).
- Schuerman, D. W., Wang, R. T., Gustafson, B. &. #. 1. 9. 7. ;. S., Schaefer, R. W. Systematic studies of light scattering. 1: Particle shape. Applied Optics. 20 (23), 4039-4050 (1981).
- Goering, K. J., Fritts, D. H., Eslick, R. F. A study of starch granule size and distribution in 29 barley varieties. Starch-Stärke. 25 (9), 297-302 (1973).
- Chen, Z., Schols, H. A., Voragen, A. G. J. Starch granule size strongly determines starch noodle processing and noodle quality. Journal of Food Sciences. 68 (5), 1584-1589 (2003).
- Dai, Z. M. Starch granule size distribution in grains at different positions on the spike of wheat (Triticum aestivum L.). Starch-Starke. 61 (10), 582-589 (2009).
- Edwards, M. A., Osborne, B. G., Henry, R. J. Effect of endosperm starch granule size distribution on milling yield in hard wheat. Journal of Cereal Science. 48 (1), 180-192 (2008).
- Karlsson, R., Olered, R., Eliasson, A. C. Changes in starch granule size distribution and starch gelatinization properties during development and maturation of wheat, barley and rye. Starch - Starke. 35 (10), 335-340 (1983).
- Li, W. -. Y., et al. Comparison of starch granule size distribution between hard and soft wheat cultivars in Eastern China. Agricultural Sciences China. 7 (8), 907-914 (2008).
- Park, S. H., Wilson, J. D., Seabourn, B. W. Starch granule size distribution of hard red winter and hard red spring wheat: Its effects on mixing and breadmaking quality. Journal of Cereal Science. 49 (1), 98-105 (2009).
- Limpert, E., Stahel, W. A., Abbt, M. Log-normal distributions across the sciences: keys and clues. Bioscience. 51 (5), 341-352 (2001).
- Gao, M., et al. Self-preserving lognormal volume-size distributions of starch granules in developing sweetpotatoes and modulation of their scale parameters by a starch synthase II (SSII). Acta Physiologiae Plantarum. 38 (11), 259 (2016).
- Wattebled, F., et al. STA11, a Chlamydomonas reinhardtii locus required for normal starch granule biogenesis, encodes disproportionating enzyme. Further evidence for a function of alpha-1,4 glucanotransferases during starch granule biosynthesis in green algae. Plant Physiology. 132 (1), 137-145 (2003).
- Ji, Y., Seetharaman, K., White, P. J. Optimizing a Small-Scale Corn-Starch Extraction Method for Use in the Laboratory. Cereal Chemistry. 81 (1), 55-58 (2004).
- Halloy, S., Whigham, P. The lognormal as universal descriptor of unconstrained complex systems: a unifying theory for complexity. Proceedings of the 7th Asia-Pacific Complex Systems Conference. , 309-320 (2004).
- Furusawa, C., Suzuki, T., Kashiwagi, A., Yomo, T., Kaneko, K. Ubiquity of log-normal distributions in intra-cellular reaction dynamics. Biophysics (Nagoya-shi). 1, 25-31 (2005).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon