È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Analisi e specificazione delle distribuzioni delle dimensioni dei granuli di amido
In questo articolo
Riepilogo
Qui è presentata una procedura per determinare in modo riproducibile e statisticamente valido le distribuzioni delle dimensioni dei granuli di amido e per specificare le distribuzioni di dimensioni lognormali del granulo determinate utilizzando una forma moltiplicativa a due parametri. È applicabile a tutte le analisi di dimensionamento dei granuli di campioni di amido su scala grammo per la ricerca sulle piante e sulle scienze alimentari.
Abstract
L'amido proveniente da tutte le fonti vegetali è costituito da granuli in una gamma di dimensioni e forme con frequenze di occorrenza diverse, cioè che presentano una dimensione e una distribuzione dellaforma. I dati sulle dimensioni dei granuli di amido determinati utilizzando diversi tipi di tecniche di dimensionamento delle particelle sono spesso problematici a causa della scarsa riproducibilità o mancanza di significatività statistica derivante da alcuni errori sistematici insormontabili, tra cui la sensibilità alle forme dei granuli e i limiti delle dimensioni del campione di granuli. Abbiamo delineato una procedura per determinare in modo riproducibile e statisticamente valido le distribuzioni delle dimensioni dei granuli di amido utilizzando la tecnica della zona di rilevamento elettrico e per specificare le distribuzioni di dimensioni lognormali del granulo determinate utilizzando una forma moltiplicativa a due parametri adottata con una maggiore precisione e comparabilità. È applicabile a tutte le analisi di dimensionamento dei granuli di campioni di amido su scala grammo e, pertanto, potrebbe facilitare gli studi su come le dimensioni dei granuli di amido sono modellate dall'apparato e dai meccanismi di biosintesi dell'amido; e come hanno un impatto sulle proprietà e sulla funzionalità degli amidi per usi alimentari e industriali. I risultati rappresentativi sono presentati da analisi replicate delle distribuzioni delle dimensioni dei granuli di campioni di amido di sweetpotato utilizzando la procedura delineata. Abbiamo inoltre discusso diversi aspetti tecnici chiave della procedura, in particolare la specifica moltiplicativa delle distribuzioni di dimensioni lognormali del granulo e alcuni mezzi tecnici per superare il blocco frequente dell'apertura da parte degli aggregati di granuli.
Introduzione
I granuli di amido sono la struttura fisica in cui due principali polimeri omoglucani di riserva nei tessuti di fotosintesi e stoccaggio delle piante, l'amilosio lineare o scarsamente ramificata e l'amiloetina altamente ramificata, sono imballati in modo ordinato insieme ad alcuni componenti minori, inclusi lipidi e proteine. I granuli di amido di varie specie vegetali presentano molte forme tridimensionali (3D) (riviste nel ref.1,2), tra cui sfere, ellissoidi, poliedri, piastrine, cubi, cuboidi e tubuli irregolari. Anche quelli dello stesso tessuto o tessuti diversi della stessa specie vegetale potrebbero avere un insieme di forme con frequenze di occorrenza variabili. In altre parole, i granuli di amido di una specie vegetale possono avere una distribuzione statistica caratteristica della forma, piuttosto che una forma specifica. Le forme granuli non uniformi e non sferiche rendono difficile misurare e definire correttamente le dimensioni dei granuli di amido. Inoltre, i granuli di amido degli stessi tessuti di una specie vegetale sono di una gamma di dimensioni con proporzioni diverse, cioè presentano una distribuzione caratteristica delle dimensioni. Questa distribuzione delle dimensioni complica ulteriormente l'analisi e la descrizione delle dimensioni dei granuli di amido.
Le dimensioni dei granuli di amido sono state analizzate utilizzando diverse categorie di tecniche di dimensionamento delle particelle (esaminate nella rif. 3 ), tra cui microscopia, sedimentazione/frazionamento sterico campo-flusso (Sd/StFFF), diffrazione laser e zona dirilevamentoelettrico (ESZ). Tuttavia, queste tecniche non sono ugualmente adatte per la determinazione delle dimensioni dei granuli di amido in presenza di una forma di granulo e di una distribuzione delle dimensioni. La microscopia, compresa la microscopia elettronica leggera, confocale e a scansione, è eccellente per gli studidi morfologia 4,5,6,7,struttura8,9 e sviluppo10,11 di granuli di amido, ma difficilmente adatta a definire le loro distribuzioni di dimensioni a causa di alcune carenze intrinseche. Misurazioni dirette di immagini microscopiche di granuli o analisi di immagini assistite da software di dati di microscopia ottica (IAOM), che sono stati utilizzati per la determinazione delle dimensioni granuli degli amidi di diverse specie, compreso il mais12,ilgrano 13,14,la patata 15 e l'orzo16,può misurare solo dimensioni 1D (di solito di lunghezza massima) o 2D (superficie) di numeri molto limitati (da decine a poche migliaia) di immagini di granuli di amido. Le piccole dimensioni di campionamento del granulo che sono intrinsecamente vincolate dalle tecniche potrebbero raramente essere statisticamente rappresentative, considerando gli enormi numeri di granuli per unità di peso dell'amido (~120 x 106 per grammo, assumendo tutte le sfere di 10 μm a densità di 1,5 g/cm³) e, quindi, potrebbero portare alla scarsa riproducibilità dei risultati. La tecnica Sd/StFFF può avere alta velocità e risoluzione, e frazioni di dimensioni ridotte di granuli di amido17, ma è stata usata raramente probabilmente perché la sua accuratezza potrebbe essere gravemente influenzata da danni, forme diverse e densità di granuli di amido. La tecnica di diffrazione laser è la più utilizzata ed è stata applicata per analisi delle dimensioni dei granuli di amido per tutte le principali specievegetali 3,14,16. Sebbene la tecnica abbia molti vantaggi, in realtà non è adatta per determinare le dimensioni dei granuli di amido in presenza di una distribuzione della forma del granulo. La maggior parte degli strumenti di diffrazione laser simultanei si basano sulla teoria dello scattering della luce mie18 per particelle sferiche uniformi e sulla teoria di Miemodificata 18 per alcune altre forme di uniformità. La tecnica è, quindi, intrinsecamente molto sensibile alle forme delle particelle, e non del tutto adatta anche per alcune forme diuniformità 19,figuriamoci per granuli di amido aventi una serie di forme di proporzioni variabili. La tecnica ESZ misura il disturbo del campo elettrico proporzionale al volume della particella che passa attraverso un'apertura. Fornisce dimensioni del volume del granulo, nonché informazioni sulla distribuzione del numero e del volume, ecc., ad alte risoluzioni. Poiché la tecnica ESZ è indipendente da qualsiasi proprietà ottica di particelle tra cui colore, forma, composizione o indice di rifrazione, e i risultati sono molto riproducibili, è particolarmente adatta per determinare la distribuzione delle dimensioni dei granuli di amido con un insieme di forme.
Anche le dimensioni dei granuli di amido sono state definite utilizzando molti parametri. Erano spesso descritti simplisticamente da diametri medi, che in alcuni casi erano i mezzi aritmetici delle lunghezze massime misurate microscopicamente delle immagini2D 12,20o medie di diametri disfera equivalenti 3. In altri casi, le distribuzioni delle dimensioni del granulo sono state specificate utilizzando gli intervallidi dimensioni 21,22, il volume medio di distribuzione o il diametro medio (equivalente sfera, ponderato per numero, volume o superficie) assumendo una distribuzionenormale 14,23,24,25,26. Questi descrittori delle dimensioni dei granuli di amido di varie analisi sono di natura molto diversa e non strettamente comparabili. Potrebbe essere molto fuorviante se queste "dimensioni" di granuli di amido di specie diverse o anche gli stessi tessuti della stessa specie fossero confrontati direttamente. Inoltre, il parametro di diffusione (o forma) delle distribuzioni normali ipotizzate, cioè la deviazione standard σ (o deviazione standard grafica σg) che misura la larghezza della distribuzione (cioè la diffusione delle dimensioni), è stato ignorato nella maggior parte degli studi.
Per risolvere i suddetti problemi critici relativi alle analisi di dimensionamento dei granuli di amido, abbiamo delineato una procedura per determinare in modo riproducibile e statisticamente valido le distribuzioni delle dimensioni dei granuli dei campioni di amido utilizzando la tecnica ESZ e per specificare correttamente le distribuzioni di dimensioni lognormali del granulo determinate utilizzando una forma moltiplicativa a due parametriadottata 27 con maggiore precisione e comparabilità. Per la convalida e la dimostrazione, abbiamo eseguito analisi di dimensionamento dei granuli replicati di campioni di amido sweetpotato utilizzando la procedura e specificato le distribuzioni di diametro del volume differenziale lognormale volume-percentuale equivalente-sfera utilizzando i loro mezzi 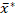 geometrici grafici e deviazioni standard moltiplicative s* in
geometrici grafici e deviazioni standard moltiplicative s* in 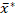 forma x/ (moltiplica e dividi) s*.
forma x/ (moltiplica e dividi) s*.
Protocollo
1. Preparazione di campioni di amido
- Preparare due (o tre) campioni di amido replicati in scala grammo da tessuti che accumulano amido di varie specie vegetali seguendo le procedure stabilite (ad esempio, patate15,dolci28,chicchi digrano 13,29e chicchi di mais30,ecc.).
- Lavare accuratamente i campioni di amido con acetone o toluene 3-4x per ridurre al minimo gli aggregati di granuli e asciugarli completamente.
NOTA: Utilizzare procedure di estrazione che producono più di 1 g di amido per preparazione. Una o due aliquote da 0,5 g di ciascuno dei tre o due estratti replicati, rispettivamente, sono campioate per l'analisi di dimensionamento dei granuli di un estratto di amido.
2. Preparazione elettrolita
- Preparare 500 mL di cloruro di litio da 50 g/L in metanolo per quattro serie di dimensionamento per campioni di amido di replicazione (100 mL per corsa più altri 100 mL). Preferibilmente, fare l'elettrolita in lotti di grande volume, ad esempio da 4 a 8 L alla volta, per ridurre al minimo la variazione di concentrazione.
- Raffreddare il contenitore sul ghiaccio o in un armadio a 4 °C per accelerare la dissoluzione del cloruro di litio.
3. Impostazione dell'analizzatore
- Scegli un tubo di apertura con un intervallo di diametro delle particelle che copra la gamma nota (in letteratura o attraverso prove) di granuli di campioni di amido da analizzare, ad esempio un'apertura di 100 μm per gli amidi dolci. Per campioni di amido di portata di granuli sconosciuti, selezionare un'apertura appropriata attraverso le prove utilizzando diversi tubi di apertura con intervalli di diametro delle particelle sovrapposti.
NOTA: L'intervallo di diametro delle particelle di un tubo di apertura è il suo intervallo di dimensionamento accurato tra il 2 e il 60% di e con un intervallo di dimensionamento esteso all'80% del suo diametro dell'orifizio. La tabella 1 elenca le proprietà di tre tubi di apertura più utili per il dimensionamento dei granuli dei principali amidi di colture. Se l'intervallo di dimensioni del granulo di un campione di amido è più ampio dell'intervallo di dimensionamento di un singolo tubo di apertura, eseguire un'analisi di sovrapposizione multitubo combinando fino a cinque distribuzioni granulose misurate con aperture di dimensioni diverse. Ogni apertura è identificabile per il suo diametro e numero di parte etichettato sul tubo. Il suo diametro e il numero di serie contenuti in un codice a barre sul tubo possono essere scansionati nel software dell'analizzatore utilizzando il lettore di codici a barre sul pannello di controllo dell'analizzatore. - Scegliere un bicchiere analitico da 100 o 200 mL (sopra le cuvette) per la determinazione delle dimensioni dei granuli di amido e impostare l'agitazione automatica (sotto) per mantenere una buona sospensione del granulo durante la misurazione.
- Creare un metodo operativo standard (SOM) per specificare le impostazioni di esecuzione e un file Preferenze per l'analisi, la visualizzazione e la stampa dei risultati. Combinare il file SOM e Preferences in una procedura operativa standard (SOP) in base alle esigenze.
NOTA: per le analisi non standardizzabili, utilizzate SOM per eseguire le analisi e regolate le impostazioni SOM tra le esecuzioni attraverso la finestra Modifica som (vedere sotto) in base alle esigenze. Dopo il completamento dell'esecuzione, analizzare, visualizzare e stampare i risultati dell'esecuzione modificando le preferenze come desiderato. Per le analisi di dimensionamento dei granuli standardizzabili, utilizzare un SOP per eseguire le analisi.- Avviare il software dell'analizzatore. Nel Manu principale fare clic su SOP | Creare la Creazione guidata SOM o Modificare il SOMoppure nel Pannello di stato fare clic su Modifica SOM. Utilizzare la procedura guidata o la finestra Modifica som per selezionare le impostazioni per un SOM. Le impostazioni tipicamente utilizzate per dimensionare i granuli di campioni di amido dolcepotato sono riassunte nella tabella 2.
- Salvare il SOM creato in un file nella finestra Som Wizard-Summary of Settings o nella finestra Modifica som.
- Nel Manu principale, fare clic su SOP | Creazione guidata preferenze o Preferenze di modifica. Utilizzare la procedura guidata o le schede nella finestra di modifica Preferenze per selezionare le impostazioni delle preferenze come quelle desiderate nella tabella 3 o in altre.
- Salvare le preferenze selezionate in un file nella finestra Crea Preferenze-Riepilogo delle impostazioni o nella finestra Modifica preferenze.
- Scegliere SOP dal menu principale | Creare la Creazione guidata SOP. Seguendo la guida dettagliata della procedura guidata, immettere una descrizione, selezionare il file SOM e Preferenze per creare e salvare un SOP.
4. Analisi di dimensionamento del granulo dei campioni di amido
- Preparare l'analizzatore
- Accendere l'analizzatore, aprire il software nel computer e verificare lo stato Pronto nella parte superiore del pannello di stato dopo la connessione automatica all'analizzatore.
- Riempire il barattolo elettrolita con elettrolita, svuotare il barattolo di scarico, se necessario.
- Installare e fissare correttamente il tubo di apertura scelto seguendo la guida nel manuale dell'utente. Per un nuovo tubo di apertura non calibrato, calibrarlo seguendo la guida dettagliata in Calibrazione | Calibrare l'apertura nel menu principale. Per un tubo di apertura calibrato, verificare la calibrazione seguendo la guida dettagliata della Change Aperture Tube Wizard sotto la procedura di esecuzione o calibrazione | Verificare la calibrazione dell'apertura nel menu principale.
- Sblocca la piattaforma di dosaggio spingendo la clip di rilascio del blocco (nella parte anteriore centrale della parete del vano campione sinistro) e abbassa manualmente la piattaforma verso il basso. Posizionare un becher analitico contenente 100 ml di elettrolita sulla piattaforma, spostare l'agitatore nella posizione di agitazione e sollevare manualmente la piattaforma nella posizione superiore autobloccante per immergere il tubo di apertura e l'agitatore nell'elettrolita.
- Fate clic su Riempimento (Fill) sulla barra degli strumenti inferiore (Toolbar) per fare in modo che l'analizzatore riempia automaticamente il sistema con l'elettrolita e fate clic su Scarica (Flush) per fare in modo che l'analizzatore sciacqua automaticamente il sistema.
- Caricare il SOM facendo clic su SOP | Caricare un SOM nel menu principalee utilizzare som per eseguire un'analisi senza un file Preferenze. In alternativa, caricare un SOP facendo clic su SOP | Caricare un SOP nel menu principale o caricare sop nel pannello stato e utilizzare il SOP per eseguire un'analisi.
- Se si utilizza un SOP, fare clic su SOP | info SOM o informazioni sulle preferenze nel menu principale per verificare le impostazioni SOM e Preferenze. Fare clic su Esempio | Immettete informazioni di esempio nel menu principale o modificate le informazioni nel pannello stato per immettere le informazioni di esempio per l'esecuzione.
- Preparare il campione di amido-metanolo e le sospensioni di dimensionamento
- Pesare rispettivamente due o uno campione da 0,5 g da ciascuno dei due o tre estratti di amido replicati.
- Aggiungere ciascuna delle aliquote di amido da 0,5 g a metanolo da 5 ml in un tubo di centrifuga conico da 50 ml e disperdere completamente i granuli di amido utilizzando diversi impulsi di ultrasuoni a bassa intensità (12-24 W/cm2)da un processore ad ultrasuoni.
- Utilizzando una pipetta di trasferimento usa e getta, applicare una piccola goccia della sospensione amido-metanolo (~0,2 mL) sui 100 mL di 50 g/L elettrolita di metanolo LiCl sotto agitazione costante nel bicchiere. Chiudere la porta del vano campione.
- Eseguire un'esecuzione di ridimensionamento
- Fare clic su Anteprima nella barra degli strumenti inferiore per avviare un'esecuzione di anteprima. Nel pannello Stato verificare che la barra di concentrazione visualizzata dinamicamente sia in verde e che mostri un intervallo di concentrazione nominale dal 5 all'8% per la sospensione.
- Fare clic su Interrompi sulla barra degli strumenti inferiore per interrompere l'esecuzione dell'anteprima. Se necessario, diluire la sospensione amido-elettrolita sostituendo un'aliquota della sospensione con l'elettrolita, quindi ripetere una corsa di anteprima.
NOTA: L'intervallo di concentrazione nominale dal 5% all'8% della sospensione è fondamentale per il completamento di una corsa senza arresto a causa del blocco dell'apertura da parte di granuli aggregati. Se necessario, regolare la dimensione del campione di goccia e/o la concentrazione della sospensione amido-metanolo per effettuare una nuova sospensione amido-elettrolita con la concentrazione nominale nell'intervallo ottimale. - Dopo la verifica, fare clic su Avvia sulla barra degli strumenti inferiore per avviare l'esecuzione. L'analizzatore completa automaticamente l'esecuzione una volta che il conteggio totale dei granuli di dimensioni, visualizzato insieme al tempo di esecuzione sul Pannello di stato in un'esecuzione, raggiunge il conteggio totale impostato (125.000 o 250.000) dalla modalità di controllo del SOM. A seconda della concentrazione di sospensione (entro un intervallo del 5-8% o inferiore), una singola corsa richiede da 2 a 5 minuti o più.
NOTA: quando l'analizzatore rileva automaticamente un blocco dell'apertura per impostazioni di rilevamento del blocco del SOM, interromperà l'esecuzione, si scaricherà per sbloccare l'apertura e avviare una nuova esecuzione. Questa azione di blocco è impostata per ripetere al massimo per quattro volte prima che l'analizzatore annulli l'operazione di esecuzione. Questo problema di blocco che si interrompe può essere superato utilizzando due metodi tecnici, come indicato nella tabella 2 e dettagliato nella discussione. - Se necessario, eseguire una ripetizione tecnica (vedere la tabella 2 e dettagliata nella discussione) utilizzando la stessa sospensione starch-electrolyte semplicemente facendo clic su Start o Repeat sulla barra degli strumenti inferiore.
- Dopo il completamento di una corsa o ripetizione, svuotare il becher, sciacquarlo con metanolo e riempirlo con una soluzione di elettrolita fresca da 100 mL per la corsa successiva.
- Durante un'esecuzione, se viene visualizzata una finestra di dialogo di notifica intervallo di dimensioni estese quando il numero di granuli superiori a 60 μm supera lo 0,1% del conteggio totale (secondo l'impostazione SOM), fare clic su Esegui dal 60% all'80% per eseguire un intervallo di ridimensionamento dinamico esteso all'80% del diametro dell'apertura.
NOTA: l'impostazione Extended Size Range controlla le azioni per i granuli superiori al 60% del diametro dell'apertura (100 μm, in questo caso). L'impostazione nel SOM specifica l'inclusione di granuli di amido superiori a 60 μm quando i loro conteggi raggiungono oltre lo 0,1% del conteggio totale. Il completamento della corsa è ancora controllato dal conteggio totale e può richiedere un tempo leggermente inferiore rispetto al contrario senza includere i granuli più grandi per un totale inferiore allo 0,1% (presunta quantità staticamente insignificante) del conteggio totale.
- Analizzare i risultati dell'esecuzione
- Se per controllare le esecuzioni è stato utilizzato un SOM, selezionare Le impostazioni preferenze desiderate per la visualizzazione, la stampa e le analisi statistiche dei risultati utilizzando la Creazione guidata preferenze o le preferenze di modifica nel SOP del menu principale.
- La sovrapposizione risulta da più esecuzioni su un singolo grafico per il confronto.
- Fare clic su Sovrapposizione sulla barra degli strumenti principale o sul file | Sovrapposizione nel menu principale per accedere alla finestra Sovrapposizione. Passare a e selezionare più file dei risultati desiderati nella casella File, fare clic su Aggiungi per spostarli nella casella File selezionati e quindi su OK per sovrapporre i risultati selezionati in un singolo grafico.
- Per aggiungere un file a una sovrapposizione aperta, fare clic su Eseguifile | Aprire per Sovrapposizione nel menu Esegui per accedere alla finestra Sovrapposizione, passare al file desiderato e fare clic per aggiungere.
- Risultati medi delle analisi replicate (2 estratti x 2 campioni di amido o 3 estratti x 1 prelievo di amido) e visualizzare o stampare la distribuzione media delle dimensioni dei granuli e le statistiche in un elenco o in un grafico.
- Scegliere File | FileTool dal menu Principale | Media per aprire la finestra Media. Passare a e selezionare più file dei risultati desiderati nella casella File, fare clic su Aggiungi per spostarli nella casella File selezionati e quindi su OK per visualizzare in media i risultati selezionati e la media in un singolo grafico.
- Per includere un file dei risultati aggiuntivo in una distribuzione media, scegliere Eseguifile | Apri e Aggiungi alla media per aprire la finestra Aggiungi alla media, passare a e aggiungere il file. La nuova media viene visualizzata nel grafico nella finestra Esegui (risultato) o nell'elenco.
5. Specificare la distribuzione media
- Nella finestra Run-Menu che visualizza la distribuzione media fare clic su Calcola | Statistiche medie nel menu Esegui per aprire la finestra di riepilogo delle statistiche, che visualizza le statistiche medie nelle righe, e le statistiche del grafico per la distribuzione media nelle colonne.
- Utilizzare la media geometrica grafica (
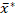 ) e S.D. (s*) nella colonna delle statistiche del grafico per specificare la distribuzione media nella
) e S.D. (s*) nella colonna delle statistiche del grafico per specificare la distribuzione media nella 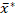 forma x/s*. Calcolare le variazioni di misura CV tra le distribuzioni di replica media dividendo la media (μ, la stessa
forma x/s*. Calcolare le variazioni di misura CV tra le distribuzioni di replica media dividendo la media (μ, la stessa 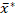 della distribuzione media) dei mezzi geometrici delle distribuzioni medie con la S.D. media (σ) elencata nella riga media delle statistiche.
della distribuzione media) dei mezzi geometrici delle distribuzioni medie con la S.D. media (σ) elencata nella riga media delle statistiche.
NOTA: La S.D. media (per μ) che valuta le variazioni tra i mezzi delle distribuzioni replicate è diversa dalla S.D. geometrica grafica (per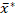 ) che misura la diffusione della distribuzione media.
) che misura la diffusione della distribuzione media.
Risultati
Per convalidare la procedura e dimostrare la riproducibilità della distribuzione determinata delle dimensioni del granulo, abbiamo eseguito analisi di dimensionamento replicate di campioni di amido sweetpotato. Abbiamo preparato campioni di amido replicati (S1 e S2) da dolci coltivati sul campo di una linea di riproduzione SC1149-19 in un'età di sviluppo simile utilizzando una proceduraprecedentemente descritta 28. Da ogni estratto di amido sono state campionato due aliquote da 0,5 g (a e b), so...
Discussione
La procedura delineata ha risolto alcune criticità in diversi metodi esistenti per l'analisi delle dimensioni dei granuli di amido, tra cui il dimensionamento 1D o 2D inappropriato dei granuli 3D, la distorsione delle misure di dimensionamento a causa di forme di granuli non uniformi, scarsa riproducibilità e dubbia validità statistica a causa di dimensioni limitate del campione di granuli, specifiche imprecise o improprie (in particolare l'uso della dimensione media) delle dimensioni dei granuli in presenza sia di fo...
Divulgazioni
Gli autori non hanno nulla da rivelare
Riconoscimenti
Questo lavoro è in parte supportato dal Centro di ricerca sull'agricoltura cooperativa e dal Centro integrato di ricerca sulla sicurezza alimentare del College of Agriculture and Human Sciences, Prairie View A&M University. Ringraziamo Hua Tian per il suo supporto tecnico.
Materiali
| Name | Company | Catalog Number | Comments |
| Analytical beaker | Beckman Coulter Life Sciences | A35595 | Smart-Technology (ST) beaker |
| Aperture tube, 100 µm | Beckman Coulter Life Sciences | A36394 | For the MS4E |
| Disposable transfer pipettor, | Fisher Scientific (Fishersci.com) | 13-711-9AM | Other disposable transfer pipettors with similar orifice can also be used. |
| Fisherbrand Conical Polypropylene Centrifuge Tubes, 50 ml | Fisher Scientific (Fishersci.com) | 05-539-13 | Any other similar types of tubes can be used. |
| Glass beakers, 150 to 250 ml | Fisher Scientific (Fishersci.com) | 02-540K | These beakers are used to contain methanol for washing the aperture tube and stirrer between runs. |
| LiCl | Fisher Chemical | L121-100 | |
| Methanol | Fisher Chemical | A412-500 | Buy in bulk as the analysis uses a large quantity of methanol. |
| Mettler Toledo ML-T Precision Balances | Mettler Toledo | 30243412 | Any other precision balance with a readability 0.01 g to 1 mg will work. |
| Multisizer 4e Coulter Counter | Beckman Coulter Life Sciences | B23005 | The old model, Multisizer 3 can also be used with slight adjustment of parameters. The 4e model comes with a 100 μm aperture tube. Other aperture tubes of different diameter can be purchased separately from the company. |
| Ultrasonic processor UP50H | Hielscher Ultrasound Technology | UP50H | Other laboratory sonicator having a low-power (<50 Watt) output can be also used. Both MS1 and MS2 sonotrodes for the particular sonicator can be used to disperse starch granules in 5 ml methanol. Always use the lowest setting first, 20% amplitude and 0.1 or 0.2 cycle, and raise the setting if aggregates persist in suspension. |
Riferimenti
- Shannon, J. C., Garwood, D. L., Boyer, C. D., BeMiller, J., Whistler, R. . Starch:Chemistry and Technology Food Science and Technology. , 23-82 (2009).
- Singh, N., Singh, J., Kaur, L., Singh Sodhi, N., Singh Gill, B. Morphological, thermal and rheological properties of starches from different botanical sources. Food Chemistry. 81 (2), 219-231 (2003).
- Lindeboom, N., Chang, P. R., Tyler, R. T. Analytical, biochemical and physicochemical aspects of starch granule size, with emphasis on small granule starches: a review. Starch - Stärke. 56 (34), 89-99 (2004).
- Baldwin, P. M., Davies, M. C., Melia, C. D. Starch granule surface imaging using low-voltage scanning electron microscopy and atomic force microscopy. International Journal of Biological Macromolecules. 21 (1-2), 103-107 (1997).
- Jane, J. L., Kasemsuwan, T., Leas, S., Zobel, H., Robyt, J. F. Anthology of starch granule morphology by scanning electron microscopy. Starch-Stärke. 46 (4), 121-129 (1994).
- Matsushima, R., Nakamura, Y. . Starch: Metabolism and Structure. , 425-441 (2015).
- Wang, S. -. q., Wanf, L. -. l., Fan, W. -. h., Cao, H., Cao, B. -. s. Morphological analysis of common edible starch granules by scanning electron microscopy. Food Science. 32 (15), 74-79 (2011).
- Baldwin, P. M., Adler, J., Davies, M. C., Melia, C. D. Holes in starch granules: confocal, SEM and light microscopy studies of starch granule structure. Starch-Stärke. 46 (9), 341-346 (1994).
- Chakraborty, I., Pallen, S., Shetty, Y., Roy, N., Mazumder, N. Advanced microscopy techniques for revealing molecular structure of starch granules. Biophysical Reviews. 12 (1), 105-122 (2020).
- Bechtel, D. B., Wilson, J. D. Amyloplast formation and starch granule development in hard red winter wheat. Cereal Chemistry. 80 (2), 175-183 (2003).
- Evers, A. Scanning electron microscopy of wheat starch. III. Granule development in the endosperm. Starch-Stärke. 23 (5), 157-162 (1971).
- Wang, Y. J., White, P., Pollak, L., Jane, J. L. Characterization of starch structures of 17 maize endosperm mutant genotypes with Oh43 inbred line background. Cereal Chemistry. 70, 171-179 (1993).
- Peng, M., Gao, M., Abdel-Aal, E. S. M., Hucl, P., Chibbar, R. N. Separation and characterization of A-and B-type starch granules in wheat endosperm. Cereal Chemistry. 76, 375-379 (1999).
- Wilson, J. D., Bechtel, D. B., Todd, T. C., Seib, P. A. Measurement of wheat starch granule size distribution using image analysis and laser diffraction technology. Cereal Chemistry. 83 (3), 259-268 (2006).
- Liu, Q., Weber, E., Currie, V., Yada, R. Physicochemical properties of starches during potato growth. Carbohydrate Polymers. 51 (2), 213-221 (2003).
- Chmelik, J., et al. Comparison of size characterization of barley starch granules determined by electron and optical microscopy, low angle laser light scattering and gravitational field-flow fractionation. Journal of the Institute of Brewing. 107 (1), 11-17 (2001).
- Moon, M. H., Giddings, J. C. Rapid separation and measurement of particle size distribution of starch granules by sedimentation/steric field-flow fractionation. Journal of Food Science. 58 (5), 1166-1171 (1993).
- Wriedt, T., Wolfram, H., Wriedt, T. . The Mie Theory: Basics and Applications. , 53-71 (2012).
- Schuerman, D. W., Wang, R. T., Gustafson, B. &. #. 1. 9. 7. ;. S., Schaefer, R. W. Systematic studies of light scattering. 1: Particle shape. Applied Optics. 20 (23), 4039-4050 (1981).
- Goering, K. J., Fritts, D. H., Eslick, R. F. A study of starch granule size and distribution in 29 barley varieties. Starch-Stärke. 25 (9), 297-302 (1973).
- Chen, Z., Schols, H. A., Voragen, A. G. J. Starch granule size strongly determines starch noodle processing and noodle quality. Journal of Food Sciences. 68 (5), 1584-1589 (2003).
- Dai, Z. M. Starch granule size distribution in grains at different positions on the spike of wheat (Triticum aestivum L.). Starch-Starke. 61 (10), 582-589 (2009).
- Edwards, M. A., Osborne, B. G., Henry, R. J. Effect of endosperm starch granule size distribution on milling yield in hard wheat. Journal of Cereal Science. 48 (1), 180-192 (2008).
- Karlsson, R., Olered, R., Eliasson, A. C. Changes in starch granule size distribution and starch gelatinization properties during development and maturation of wheat, barley and rye. Starch - Starke. 35 (10), 335-340 (1983).
- Li, W. -. Y., et al. Comparison of starch granule size distribution between hard and soft wheat cultivars in Eastern China. Agricultural Sciences China. 7 (8), 907-914 (2008).
- Park, S. H., Wilson, J. D., Seabourn, B. W. Starch granule size distribution of hard red winter and hard red spring wheat: Its effects on mixing and breadmaking quality. Journal of Cereal Science. 49 (1), 98-105 (2009).
- Limpert, E., Stahel, W. A., Abbt, M. Log-normal distributions across the sciences: keys and clues. Bioscience. 51 (5), 341-352 (2001).
- Gao, M., et al. Self-preserving lognormal volume-size distributions of starch granules in developing sweetpotatoes and modulation of their scale parameters by a starch synthase II (SSII). Acta Physiologiae Plantarum. 38 (11), 259 (2016).
- Wattebled, F., et al. STA11, a Chlamydomonas reinhardtii locus required for normal starch granule biogenesis, encodes disproportionating enzyme. Further evidence for a function of alpha-1,4 glucanotransferases during starch granule biosynthesis in green algae. Plant Physiology. 132 (1), 137-145 (2003).
- Ji, Y., Seetharaman, K., White, P. J. Optimizing a Small-Scale Corn-Starch Extraction Method for Use in the Laboratory. Cereal Chemistry. 81 (1), 55-58 (2004).
- Halloy, S., Whigham, P. The lognormal as universal descriptor of unconstrained complex systems: a unifying theory for complexity. Proceedings of the 7th Asia-Pacific Complex Systems Conference. , 309-320 (2004).
- Furusawa, C., Suzuki, T., Kashiwagi, A., Yomo, T., Kaneko, K. Ubiquity of log-normal distributions in intra-cellular reaction dynamics. Biophysics (Nagoya-shi). 1, 25-31 (2005).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon