Method Article
Génération de monocouches épithéliales primaires murines du côlon à partir de cryptes intestinales
Dans cet article
Résumé
Dans ce protocole, nous décrivons comment générer des monocouches épithéliales primaires murines de deux points directement des cryptes intestinales. Nous fournissons des approches expérimentales pour générer des monocouches confluentes sur des filtres perméables, des monocouches confluentes pour la cicatrisation des plaies à gratter et des études biochimiques, et des monocouches clairsemés et confluentes pour l’analyse d’immunofluorescence.
Résumé
L’épithélium intestinal est composé d’une seule couche de cellules qui agissent comme une barrière entre la lumière intestinale et l’intérieur du corps. La perturbation de la continuité de cette barrière peut entraîner des troubles inflammatoires tels que des maladies inflammatoires de l’intestin. L’une des limites de l’étude de la biologie épithéliale intestinale a été l’absence de modèles de culture cellulaire primaire, ce qui a obligé les chercheurs à utiliser des lignées cellulaires modèles dérivées de carcinomes. L’avènement des entéroïdes tridimensionnels (3D) a donné aux biologistes épithéliaux un outil puissant pour générer des cultures cellulaires primaires, néanmoins, ces structures sont intégrées dans la matrice extracellulaire et manquent de la maturité caractéristique des cellules épithéliales intestinales différenciées. Plusieurs techniques pour générer des monocouches épithéliales intestinales ont été publiées, mais la plupart sont dérivées d’entéroïdes 3D établis rendant le processus laborieux et coûteux. Ici nous décrivons un protocole pour générer les monocouches épithéliales primaires de deux points directement des cryptes intestinales murines. Nous détaillons également les approches expérimentales qui peuvent être utilisées avec ce modèle telles que la génération de cultures confluentes sur des filtres perméables, la monocouche confluente pour les études de cicatrisation des plaies rayures et les monocouches clairsemée et confluente pour l’analyse d’immunofluorescence.
Introduction
Les cellules épithéliales intestinales (IEC) tapsent les intestins formant une barrière sélectivement perméable qui permet l’absorption des nutriments et de l’eau tout en empêchant les micro-organismes et les toxines de pénétrer dans le corps 1. La muqueuse intestinale est composée de projections luminales appelées villosités (présentes uniquement dans l’intestin grêle) et d’invaginations appelées cryptes. Les villosités et la surface des cryptes du côlon sont couvertes de cellules épithéliales différenciées tandis que la base des cryptes est composée de cellules souches qui font le renouvellement rapide de l’épithélium intestinal, qui a un renouvellement de 3 à 7 jours. Les cellules souches intestinales (ISC) ne sont pas seulement importantes pour le maintien de l’homéostasie intestinale, mais aussi pour une réparation adéquate de l’épithélium endommagé2.
L’étude de la biologie intestinale d’epithelia a été limitée par le manque de cultures cellulaires primaires avec des variétés de cellule transformées étant le seul outil disponible. Les lignées cellulaires modèles épithéliales intestinales ne sont pas capables de reproduire avec précision la physiologie de l’épithélium intestinal normal. Le développement de cultures 3D dérivées d’ISC a fourni aux biologistes des muqueuses intestinales des modèles in vitro qui ressemblent à des conditions muqueuses intestinales in vivo3. Les cryptes peuvent être facilement isolées à partir d’échantillons murins, intégrées dans un milieu matriciel membranaire du sous-sol (par exemple, Matrigel) et cultivées dans des milieux conditionnés contenant Wnt3a, R-spondin et Noggin, générant des structures 3D connues sous le nom d’entéroïdes (intestin grêle) ou de coloïdes (gros intestin)4. Les entéroïdes et les colonoïdes sont des structures sphéroïdales polarisées où le domaine apical fait face à une lumière interne et la région basolatérale est en contact direct avec la matrice extracellulaire. Les entéroïdes et les coléoïdes contiennent tous les principaux sous-types épithéliaux intestinaux différenciés tels que les entérocytes / colonocytes, paneth, entéroendocrine et les cellules gobelines et ils apparaissent dans des proportions relativement identiques à celles qu’ils présentent dans la section de l’intestin où ils ont été isolés à partir de5. Même si les entéroïdes et les coloïdes 3D représentent une avancée majeure dans l’étude du développement intestinal et de la physiologie, ces modèles présentent certains inconvénients tels que l’accès limité à la surface apicale des cellules épithéliales (lumen) et la capacité de mettre à l’échelle les cultures vers le haut ou vers le bas pour réaliser un criblage à haut débit des molécules d’intérêt. Pour surmonter ces limitations, des protocoles pour obtenir des cultures 2D primaires d’IEC dérivées d’entéroïdes/coloïdes 3D ont été générés. Les entéroïdes/coloïdes 2D se développent comme une feuille de cellules tout comme les lignées cellulaires modèles et sont idéaux pour étudier la réparation des plaies intestinales, les interactions hôte-pathogène et la médecine régénérative, entre autres. Plusieurs articles publiés décrivent comment générer des monocouches 2D à partir de structures 3D ou directement à partir de cryptes intestinales (voir6,7,8,9,10,11),mais ces méthodes ont tendance à être laborieuses et difficiles à reproduire. Une méthode rapide, simple, et reproductible pour obtenir des monocouches directement des cryptes intestinales fraîchement isolées de souris est décrite dans ce protocole.
Ici, nous expliquons en détail le processus d’extraction de la crypte avec une génération minimale de débris, un choix de matrice extracellulaire et différentes surfaces et applications pour cette technique. Cette approche expérimentale a été optimisée pour les cryptes de deux points, mais des résultats semblables sont obtenus une fois appliqués pour le petit intestin.
Protocole
Toutes les procédures décrites ci-dessous ont été approuvées et menées conformément aux lignes directrices établies par le Comité institutionnel de protection et d’utilisation des animaux de l’Université du Michigan.
1. Préparation de réactifs pour l’isolement et la culture en crypte (préparation dans la hotte de culture tissulaire)
- Acide tétraacétique éthylènediamine (EDTA) 50 mM : Ajouter 50 mL 0,5 M de stock à 450 mL de solution saline tamponnée au phosphate, sans calcium(Ca2+) et magnésium(Mg2+)pour préparer 500 mL. Dans ce protocole, pbs fera référence au PBS sans calcium et magnésium, sauf indication contraire.
- Tampon de secouage : Dissoudre 7,4 g de saccharose (43,3 mM) et 5 g de sorbitol (54,9 mM) dans du PBS pour préparer 500 mL.
- Milieu L-WRN (L-Wnt-3A, R-spondin et Noggin) : Supplément du produit Eagle Medium/Ham’s F-12 (DMEM/F12) d’Advanced Dulbecco (780 mL) avec 20 % de sérum fœtal bovin (FBS) (200 mL), 1x supplément de glutamine disponible dans le commerce (10 mL), pénicilline de 100 U/mL et 100 g/mL de streptomycine (10 mL), et stériliser le filtre avec un filtre de 0,22 μm.
- Obtenez des cellules L-WRN via ATCC, cultivez dans des flacons T175 et sélectionnez en utilisant Geneticin et Hygromycin. Les médias sont modifiés et collectés pendant 12 jours.
REMARQUE: Chaque lot de supports est testé pour l’activité Wnt à l’aide d’un test TOPflash Wnt Reporter. Dans ce cas-ci les protocoles translationnels de laboratoire de modélisation de tissu de médecine de Michigan (https://www.umichttml.org/protocols) ont été suivis. La lignée cellulaire TOPflash HEK 293 est cultivée en confluence dans une fiole T75, trypsinisée et plaquée sur une plaque de 96 puits. Le lendemain, différentes dilutions du milieux collectés sont ajoutées aux cellules et incubées dans un incubateur à 5% deCO2 à 37 °C pendant la nuit. Le lendemain, les cellules sont lysées et le test de lucifly luciférase est effectué conformément aux instructions du fabricant. Le test est normalisé à l’aide de Wnt-3A recombinant. Le milieu est divisé en aliquotes de 25 mL dans des tubes coniques de 50 mL et se conserve à -80 °C.
- Obtenez des cellules L-WRN via ATCC, cultivez dans des flacons T175 et sélectionnez en utilisant Geneticin et Hygromycin. Les médias sont modifiés et collectés pendant 12 jours.
- Milieu de base : Pour 500 mL, supplément Advanced DMEM/F12 (448 mL) avec 2x supplément de glutamine disponible dans le commerce (10 mL), HEPES 20 mM (10 mL), pénicilline 100 U/mL et 100 g/mL de streptomycine (10 mL), 2 mM de N-acétyl-L-cystéine (2 mL), supplément de N2 (10 mL) et supplément de B27 (20 mL), filtre stériliser avec un filtre de 0,22 μm. Diviser les milieux en aliquotes de 25 mL dans un tube conique de 50 mL et les conserver à -80 °C.
- Milieu complet LWRN : Combiner 25 mL de milieux LWRN avec 25 mL de milieu de base et compléter avec 200 ng/mL de facteur de croissance épidermique (EGF) (20 μL) et 2x solution antibiotique-antimycosique (1 mL). Conservez le support complet à 4 °C.
- Collagène et laminine : Dissoudre 5 mg de poudre dans 5 mL d’acide acétique stérilisé par filtre de 100 mM (ajouter 60 μL de stock d’acide acétique à 9,94 mL d’eau de qualité moléculaire) pour produire une concentration en stock de 1 mg/mL. Tourner à 4 °C pendant 4 h et faire des aliquotes de 100 μL dans des tubes de 0,2 mL. Congeler à -20 °C. La laminine est achetée à une concentration en stock de 100 μg/mL.
- Milieu complet sans facteurs de croissance (CMGF-) : Supplément avancé DMEM/F12 (500 mL) avec 1x supplément de glutamine disponible dans le commerce (5 mL), HEPES 10 mM (5 mL), pénicilline 100 U/mL et streptomycine 100 g/mL (5 mL).
- Milieu de différenciation : À 9,2 mL de milieu CMGF-, ajouter 200 μL de supplément de B27, 100 μL de supplément de N2, 20 μL de N-acétyl-L-cystéine, 500 μL de milieu de Noggin12 (fabriqué à partir de cellules productrices de Noggin) et 2 μL d’EGF pour faire 10 mL de milieux de différenciation.
2. Préparation de plaques, de lames de chambre et d’inserts de membrane de culture cellulaire
- Revêtement de plaques de 48 puits et de lames de chambre pour placage monocouches 2D: Utilisez la solution de revêtement qui constitue la laminine (dilution 1:40, voir table des matériaux)et le collagène (dilution 1:30) dans la solution saline tamponnée au phosphate de Dulbecco froide, avec Ca2 + et Mg2 + (DPBS). Ajouter 200 μL de solution de revêtement à chaque puits et pré-incuber la lame de la plaque/chambre dans un incubateur à 5 % deCO2 à 37 °C pendant au moins 2 h.
- Revêtement d’inserts de membrane de culture cellulaire de 0,4 μm : Faire une dilution 1:30 du collagène dans de l’eau de qualité moléculaire et ajouter 200 μL à chaque insert. Maintenir la plaque contenant l’insert membranaire sur de la glace à 4 °C pendant 30 min. Après 30 min d’incubation, conserver la plaque dans un incubateur à 5% deCO2 à 37 °C pendant 1,5-2 h. Les inserts à membrane en polyester et en polycarbonate donnent des résultats comparables.
REMARQUE: N’importe quelle plaque de culture tissulaire peut être ensemencée (upscaling et downscaling peut être effectuée) en utilisant ce protocole en ajustant le volume de collagène / laminine pour obtenir une couverture complète de la surface de placage.
3. Isolation de la crypte
REMARQUE: Avant de commencer la dissection, préparez le collagène et / ou laminine enduit de plaque / membrane inserts / lame lame et laissez-les dans un incubateur à 5% de CO2 à 37 ° C. Préparez un banc de travail propre et des instruments chirurgicaux stériles appropriés pour la chirurgie, ainsi qu’une armoire de sécurité biologique pour la culture de monocouches 2D. Confirmez que tous les autres équipements standard pour la culture monocouche 2D tels que l’incubateur de CO2 humidifié, les centrifugeuses de table (maintenues à 4 °C), les microscopes et les pipettes (y compris les pipettes sérologiques) sont prêts.
- Utilisez des souris C57Bl/6 âgées de 8 à 12 semaines. Euthanasier des souris à l’aide d’une méthode d’euthanasie approuvée.
- Vaporisez les carcasses de souris avec une solution d’éthanol à 70% (EtOH) pour nettoyer la zone de dissection et éliminer l’excès d’EtOH à l’aide de mouchoir en papier.
REMARQUE: Assurez-vous que les réactifs d’isolation de crypte (tels que PBS, EDTA 50 mM, tampon de secousse) sont maintenus au froid. Ces réactifs peuvent être préparés un jour avant et peuvent être utilisés pendant au moins 3 mois lorsqu’ils sont conservés à 4 °C. Assurez-vous également que le support complet du LWRN est conservé dans un bain de perles ou d’eau maintenu à 37 °C jusqu’à son utilisation. - À l’aide de ciseaux et de pinces de dissection propres, disséquer le côlon du rectum au caecum. Tenez l’extrémité du côlon à l’aide d’une pince et rincez très doucement les matières fécales à l’aide de PBS glacé dans une seringue de 10 mL, munie d’une sonde d’alimentation de 20 G(figure 1A). Assurez-vous de ne pas rompre le côlon.
- Enlever le côlon proximal. Il s’agira de la partie du côlon la plus proche de la jonction ileocecal.
- Avec une pince, faites glisser doucement le côlon distal sur la sonde d’alimentation de 20 G, en attachant le côlon à l’extrémité du tube par la pointe avec un fil de suture de soie 4-0(figure 1B).
- Inversez le côlon à l’envers à l’aide de ses doigts sur l’extrémité attachée et attachez l’autre extrémité avec un fil de suture de soie 4-0. À l’aide de ciseaux chirurgicaux, couper le côlon sous l’extrémité de la sonde d’alimentation (Figure 1C, D, E).
- À l’aide du piston d’une seringue de répétition de 1,25 mL, ouvrez doucement l’extrémité non déliée du côlon inversé sur l’extrémité d’une seringue de répétition de 1,25 mL. Faites glisser le côlon inversé sur l’extrémité de la seringue et attachez-le fermement avec un fil de suture de soie 4-0(figure 1F,G).
- Insérez le piston dans la seringue et gonflez le côlon pour former une saucisse. Gonfler jusqu’à ce que la saucisse du côlon semble turgente sans rides visibles( Figure 1H).
- Placer la seringue/le côlon dans un tube de 15 mL avec 5 mL de solution de récupération cellulaire sur de la glace pendant 20 min, gonfler et dégonfler le côlon une fois toutes les 5 minutes(figure 1I). La saucisse doit rester gonflée pendant l’incubation.
- Attachez à l’aide d’un fil de suture de soie 4-0 sous le bout de la seringue de répétition avec le côlon gonflé. Couper la saucisse de la seringue à répétition et placer dans un tube de 15 mL contenant 10 mL d’EDTA de 50 mM (2 mM pour l’intestin grêle) pendant 40 min et tourner à 4 °C(figure 1J,K).
- Décanter la solution d’EDTA et la remplacer par 5 mL de tampon de secousse. Agiter la saucisse manuellement en position verticale (vigoureusement) pendant 2 min.
- Décanter la solution de secousse dans un nouveau tube de 15 mL et répéter l’étape de secousse pour un total de 10 mL de cryptes dans un tampon de secousse.
- Prenez 20 μL de la suspension de la crypte dans une boîte de Petri et comptez le nombre de cryptes au microscope. Calculer la concentration de cryptes dans les cryptes/μL. En fonction de la concentration, diluer les échantillons pour obtenir 5cryptes/μL au moment du placage (1000 cryptes/cm2).
- Faire tourner le tube avec des cryptes isolées à l’aide d’une centrifugeuse de table à 400 x g pendant 10 min à 4 °C.
- En attendant, retirez la plaque/l’insert/lame de chambre de 48 puits de l’incubateur et placez-la dans une armoire de biosécurité. Aspirer la solution de revêtement à l’aide de P200 et laisser la plaque avec le couvercle légèrement décalé jusqu’à ce que les cellules soient prêtes à être plaquées.
4. Culture de la monocouche 2D
REMARQUE: Pour un protocole détaillé sur la façon de générer des monocouches épithéliales intestinales à partir de coloïdes 3D, vérifiez les protocoles par les laboratoires Estes et Kovbasnjuk (7,11).
- Retirer la zone tampon de secousse à l’aide d’une pipette sérologique de 10 mL. Assurez-vous que le culot est intact et peut utiliser P1000 pour éliminer tout liquide restant. Re-suspendre la pastille dans 3 mL de milieu complet LWRN et pipetter de haut en bas avec P1000. Ajouter 200 μL de cryptes à chaque puits d’une plaque/chambre pré-revêtue de 48 puits et incuber dans un incubateur à 5 % de CO2 à 37 °C.
- Le lendemain, aspirez le média à l’aide du P200 et ajoutez de nouveaux médias. Les cellules deviennent confluentes en 24-48 h.
- Pour les inserts à membrane de culture cellulaire, ajouter 200 μL de cryptes (5 cryptes/μL) au-dessus des inserts et 600 μL de milieu L-WRN complet à la partie inférieure. Le lendemain, aspirez le média en utilisant le P200 et ajoutez des médias frais uniquement à la chambre supérieure. Incuber la plaque dans un incubateur à 5 % deCO2 à 37 °C. La résistance électrique transépithéliale (TEER) est mesurée tous les jours à l’aide d’un compteur de volts/ohm épithéliaux (EVOM).
REMARQUE: Si la culture a une lecture TEER supérieure à 300 Ω,cm2,ils sont censés être confluents. La confluence est atteinte en 3-4 jours. Changez de média tous les 2 jours.
Résultats
Pour illustrer la fiabilité des cultures monocouches épithéliales primaires de deux points, un résumé de l’isolement de crypte et des images représentatives dérivées du protocole est montré. L’utilisateur doit avoir à l’esprit que les cryptes isolées sont cultivées dans des conditions stériles, de sorte qu’une dissection correcte et le nettoyage du côlon est une priorité. La figure 1 présente les étapes clés pendant l’isolation de la crypte. Les cryptes isolées(figure 2A)sont maintenant comptées et concentrées(figure 2B)pour obtenir une concentration de 5 cryptes/μL. Après une préparation concentrée de cryptes, les cellules seront plaquées dans le format souhaité (boîte de culture, inserts membranaires ou lames de chambre) et incubées avec le milieu approprié en fonction des besoins expérimentaux. La figure 3 montre la progression de la culture après 24 et 48 h de culture dans des plaques de 48 puits. Les cellules sont incubées jusqu’à ce que la confluence désirée soit atteinte. Pour illustrer les applications possibles de cette méthode, nous avons permis à des puits de plaque de 48 puits d’atteindre la confluence et avons procédé à un essai de rayure-blessure. La figure 4 représente une rayure fraîchement créée(figure 4A)dans une monocouche colonoïde 2D et la même plaie 24 h après(figure 4B). Il est clair que la culture est toujours saine et viable et qu’il y a une réparation des plaies. Pour générer des monocouches différenciées, les médias viennent du LWRN au média de différenciation. La différenciation est obtenue en montrant des valeurs élevées de TEER(figure 5A),une diminution des marqueurs ISC ((récepteur couplé à la protéine G contenant de la leucine et répétitive 5 (Lgr5) et Achaete-scute comme 2 (Ascl2)) et une augmentation des marqueurs de différenciation ((Alanyl aminopeptidase (Anpep), Mucine 2 (Muc2), lysozyme 1 (Lyz1), saccharose iso-maltase (Sis) et Chrmogranin A (Chga)) (Figure 5B,C)par PCR. D’autres marqueurs comme CDX2 et KRT20 peuvent également être inclus dans ce panneau. En plus des niveaux d’expression de l’ARNm, l’apparition de sous-types de cellules épithéliales différenciées dans les coloïdes 2D cultivés dans des conditions de différenciation est également démontrée par immunofluorescence (Muc2 et Chga; Figure 5D).
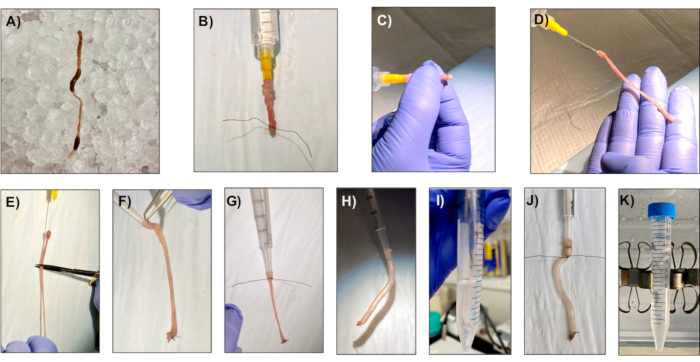
Figure 1: Préparation de l’échantillon pour la génération de monocouches saines. La préparation des échantillons est cruciale pour la génération de monocouches saines. Les étapes clés du processus d’isolation sont illustrées dans cette figure pour faciliter la tâche du lecteur. Le côlon est excisé de la souris, en s’assurant qu’il n’y a pas de restes de fourrure. Retirez soigneusement les matières fécales en vous assurant de ne pas perforer le côlon; ceci est d’une importance vitale car le côlon doit pouvoir retenir l’air et être gonflé et dégonflé. Veuillez cliquer ici pour voir une version plus grande de cette figure.
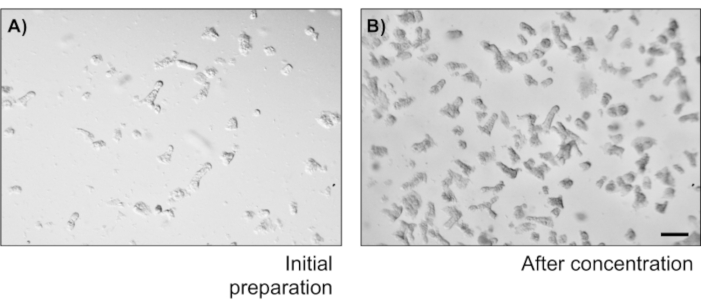
Figure 2: Nombre et concentration d’isolement de la crypte. (A) Cryptes du côlon après avoir secoué les saucisses du côlon. L’image représente un champ d’une goutte de 20 μL. (B) Concentration de crypte afin d’obtenir 5 cryptes/μL. Barre d’échelle : 1 mm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
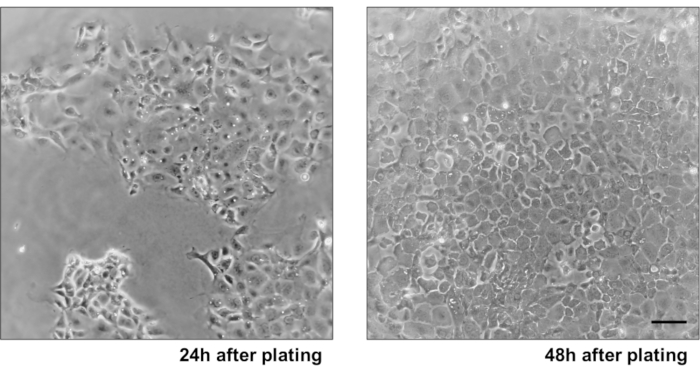
Figure 3: Croissance de la monocouche PRIMAIRE IEC . (A) Monocouches IEC 2D après 24 h de placage et d’élimination des débris cellulaires. (B) Monocouches confluentes 2D IEC 48 h après placage. Barre d’échelle: 10 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
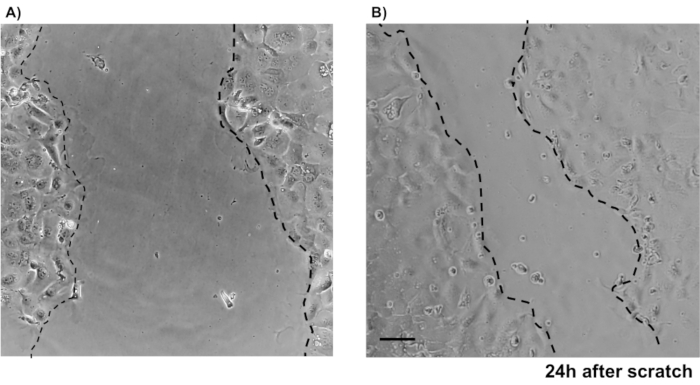
Figure 4: Dosages des plaies à gratter à l’aide de l’IEC primaire murin. Images montrant la blessure guérissant après qu’une égratignure ait été faite dans la monocouche épithéliale de deux points. Barre d’échelle: 5 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
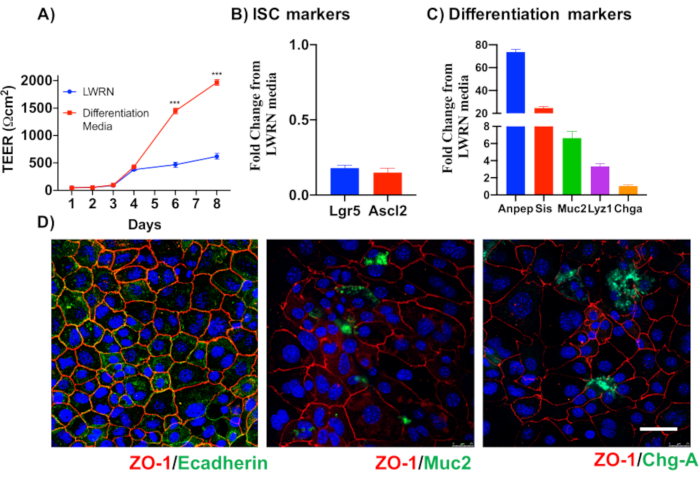
Figure 5: Différenciation des monocouches épithéliales du côlon( A)Les milieux de différenciation créent des barrières épithéliales serrées, comme l’a démontré la TEER. Les milieux de différenciation provoquent une baisse de l’expression de l’ARNm des marqueurs de cellules souches(B)et une régulation à la hausse des marqueurs de différenciation(C). (D) Les marqueurs de cellules épithéliales différenciées spécialisées telles que Muc2 et Chromogranin-A peuvent également être détectés par immunofluorescence de colonoids 2D directs. Barre d’échelle: 5 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Notre protocole fournit une méthode rapide, reproductible et fiable pour générer des monocouches IEC 2D primaires directes. L’une des principales différences dans notre protocole par rapport aux protocoles précédemment publiés pour générer des monocouches épithéliales du côlon est que nous ne coupons pas le côlon en petits morceaux pour libérer les cryptes. Au lieu de cela, nous avons adapté un protocole pour séparer l’épithélium intestinal du mésenchyme13 par une combinaison de forces chimiques et mécaniques pour libérer des cryptes dans une préparation extrêmement propre , (Figure 2), fournissant au chercheur un matériau idéal pour générer des cultures primaires. Notre méthode d’isolation peut également être utilisée pour générer des entéroïdes et des coloïdes 3D. Il est important de faire un comptage de cryptes chaque fois qu’une expérience est effectuée pour normaliser le nombre de cryptes qui sont plaquées. Des variables telles que la turgescence de la saucisse du côlon, la vitesse d’isolement, l’expertise de l’utilisateur peuvent affecter le nombre de cryptes isolées. La concentration de crypte de placage suggérée mentionnée dans le protocole est un point de départ, mais chaque utilisateur pour tenir compte des variables pilotées par l’utilisateur doit l’optimiser. Nous avons utilisé cette technique avec des souris WT mâles et femelles allant de 8 à 20 semaines et nous n’avons pas vu de différences majeures dans la survie cellulaire, en théorie les cryptes isolées de souris plus jeunes ont de meilleures chances de survivre. Les cryptes sont plaquées en excès car seul un petit pourcentage d’entre elles se fixent à la surface et survivent. Un équilibre où il y a assez de cryptes pour avoir une confluence de 50% un jour après le placage mais pas trop de cryptes où les cryptes mourantes auront un effet cytotoxique est l’objectif. Les supports LWRN doivent être soigneusement enlevés 24 heures après le placage pour éliminer les cryptes et les débris morts, cela doit être fait avec soin pour éviter de détacher les cellules qui poussent déjà comme une monocouche.
Après le retrait initial du support LWRN, l’utilisateur doit décider si les conditions expérimentales nécessitent des monocouches IEC primaires qui restent plus proches des cellules souches et ajoutent des milieux LWRN frais ou si la différenciation de la monocouche est souhaitée, remplacez-le par un support de différenciation. Le seul facteur le plus important dans ce protocole est d’assurer l’intégrité des deux points pendant le processus d’isolement. En cas de rupture, la saucisse peut être raccourcie pour éliminer la zone endommagée. Avant de mettre la saucisse dans de l’EDTA, assurez-vous que les nœuds sont aussi serrés que possible. Si la saucisse est dégonflée après l’incubation de l’EDTA, le protocole peut être poursuivi avec peu ou pas d’effet sur le rendement global de la crypte. Si la seringue à répétition n’est pas disponible, une pointe de micropipette régulière fixée à une seringue ordinaire peut également être utilisée pour le processus de gonflage et de déflation. De plus, si aucune solution de récupération cellulaire n’est disponible, les étapes d’inflation et de déflation peuvent être effectuées dans l’EDTA (intestin grêle 2mM, côlon 50 mM), mais cette substitution n’est pas recommandée. Si la confluence n’est pas nécessaire, les cryptes peuvent se développer dans des plaques recouvertes uniquement de collagène et même dans des plaques non enduits. N’utilisez que des plaques non revêtues, il n’y a pas d’autre option, mais les cellules ne se développeront pas en bonne santé comme elles le feraient dans les plaques recouvertes de collagène. En ce qui concerne la santé et la stabilité de la culture, les monocouches plaquées en plastique sont saines pendant 4 à 5 jours tandis que les monocouches plaquées dans des puits de transbordement peuvent être transportées jusqu’à 8 jours.
L’une des principales limitations de cette méthode est le support cellulaire requis pour cultiver les monocouches épithéliales du côlon. Les cellules LWRN sont disponibles chez ATCC, mais la génération de médias conditionnés par LWRN nécessite beaucoup de travail et nécessite l’accès à un spectromètre fluorescent pour déterminer l’activité Wnt. Les milieux de différenciation nécessitent un certain nombre de réactifs qui sont ajoutés frais avant utilisation, ce qui en fait un processus fastidieux. Enfin, la plupart de ces réactifs sont coûteux et faciles à brûler à un rythme rapide. Si un laboratoire souhaite établir cette technique sans culture cellulaire primaire intestinale préalable, il est fortement recommandé de trouver un collaborateur /collège avec de l’expérience et de former l’un de leurs membres.
L’entretien de la culture 3D pourrait être coûteux en raison du coût de la matrice membranaire du sous-sol moyenne et des volumes élevés de milieux conditionnés nécessaires pour les cultures organoïdes, mais il a l’avantage d’utiliser un nombre réduit de souris et les structures générées peuvent être transmises plusieurs fois. Les entéroïdes (dérivés de l’intestin grêle) sont relativement faciles à isoler et à maintenir tandis que les coloïdes sont plus délicats, se développent à un rythme plus lent et ont une capacité de passage plus limitée. La génération de monocouche à partir de coloïdes 3D nécessite une quantité disproportionnée de structures 3D, ce qui rend ce genre d’expériences long et coûteux. Au contraire, la préparation monocouche directe du côlon épithélial est rapide et est un moyen rapide d’obtenir les résultats. Une préparation du côlon peut générer une zone confluente de 75cm2 (10 à 15 mL de milieux conditionnés, remplace une fois) 2 à 3 jours après le placage (cette zone nécessiterait 144 puits de coloïdes 3D, ce qui signifie près de 6 ml de Matrigel et plus de 250 mL de milieux conditionnés). La consommation plus faible de milieux, l’entretien à faible coût de la culture cellulaire et la capacité d’effectuer des tests fonctionnels et un traitement rapide en aval sont de grands avantages des monocouches épithéliales du côlon.
Ce protocole est un outil précieux dans l’étude de la biologie intestinale de cellules épithéliales dans des domaines tels que la cellule-adhérence, la polarité, et la différentiation. Il donne l’avantage de générer des cultures cellulaires primaires à partir de souris génétiquement modifiées (knock-out, surexprimant, reporters). Les monocouches épithéliales intestinales primaires permettent un accès facile aux surfaces apicales et basolatérales (lorsqu’elles sont plaquées sur des puits trans), ce qui permet l’étude de la perméabilité, de la barrière et de la migration transépithéliale de différents types de cellules. Enfin, ce modèle peut être utile dans différents domaines tels que les interactions hôte-pathogène, les dommages épithéliaux et la réparation et la découverte de médicaments.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts.
Remerciements
Ce travail a été soutenu par un prix de développement de carrière de la Crohn’s and Colitis Foundation (544599, à MQ) et les subventions des NIH (DK055679, DK089763, DK059888, à AN). Nous tenons à remercier le Michigan Medicine Translational Tissue Modeling Laboratory pour son aide continue et son accès à ses réactifs et protocoles.
matériels
| Name | Company | Catalog Number | Comments |
| Advanced DMEM/F12 | Gibco | 12634-010 | |
| Antibiotic Antimycotic solution | Corning | 30-004CI | |
| B27 supplement (50X) | Gibco | 12587-010 | |
| Cell Recovery Solution | Corning | 354253 | |
| Collagen from human placenta (type IV) | Sigma-Aldrich | C5533 | |
| D-Sorbitol | Sigma | 85529-250G | |
| D-Sucrose | Fisher Scientific | BP220-1 | |
| Dulbecco’s phosphate buffered saline, with Ca2+ and Mg2+ (DPBS) | Corning | 21-030-CV | |
| Epithelial Volt/Ohm meter | World Precision Instruments | 0-10KΩ with STX2 (EVOM2) | |
| Ethylenediamine tetraacetic acid (EDTA) | Lonza | 51201 | |
| Fetal Bovine Serum (FBS) | Corning | 35-016-CV | |
| Firefly Luciferase assay | Biotium | 30085-2 | |
| Geneticin | Gibco | 10131-035 | |
| GlutaMAX (100X) | Gibco | 35050-061 | |
| HEPES (1M) | Corning | 25060CI | |
| Human recombinant EGF | R&D systems | 236-EG | Stock Concentration: 500µg/mL |
| Human recombinant Wnt-3A | R&D systems | W3a-H-005 | |
| Hygromycin B | Invitrogen | 10687010 | |
| LWRN cells | ATCC | CRL-3276 | |
| Molecular grade water | Corning | 46-000-CV | |
| N2 supplement (100X) | Gibco | 17502-048 | |
| N-acetyl-L-cysteine | Sigma-Aldrich | A9165-5G | Stock Concentration: 500mM |
| Noggin | Conditioned media | - | |
| Nunc Lab-Tek Chamber slide system | Sigma-Aldrich | C7182-1PAK | |
| Pencillin-Streptomycin (10,000U/mL) | Corning | 30002CI | |
| Phospahte buffered saline, Ca2+ and Mg2+ free (PBS) | Corning | 21-040-CV | |
| Plastic 20G feeding tube | Fisher Scientific | 50-810-46 | |
| rh-laminin-521 | Gibco | A29248 | Stock concentration: 100µg/mL |
| Roboz Surgical 4-0 Silk Black Braided 100YD | Fisher Scientific | NC9452680 | |
| TOPflash HEK293 cells | ATCC | CRL-3249 | |
| Transwell Permeable supports (0.4µm) | Corning | 3470 |
Références
- Quiros, M., Nusrat, A. Contribution of wound-associated cells and mediators in orchestrating gastrointestinal mucosal wound repair. Annual Reviews in Physiology. 81, 189-209 (2019).
- Blutt, S. E., et al. Use of organoids to study regenerative responses to intestinal damage. American Journal of Physiology-Gastrointestinal and Liver Physiology. 317 (6), 845-852 (2019).
- Zhang, M., Liu, Y., Chen, Y. G. Generation of 3D human gastrointestinal organoids: principle and applications. Cell Regeneration. 9 (1), 6(2020).
- Sato, T., et al. Single Lgr5 stem cells build crypt-villus structures in vitro without a mesenchymal niche. Nature. 459 (7244), 262-265 (2009).
- Zachos, N. C., et al. Human enteroids/colonoids and intestinal organoids functionally recapitulate normal intestinal physiology and pathophysiology. Journal of Biological Chemistry. 291 (8), 3759-3766 (2016).
- Kozuka, K., et al. Development and characterization of a human and mouse intestinal epithelial cell monolayer platform. Stem Cell Reports. 9 (6), 1976-1990 (2017).
- In, J. G., Foulke-Abel, J., Clarke, E., Kovbasnjuk, O. Human colonoid monolayers to study interactions between pathogens, commensals, and host intestinal epithelium. Journal of Visualized Experiments. (146), e59357(2019).
- Cardenas, D., et al. Two- and three-dimensional bioengineered human intestinal tissue models for cryptosporidium. Methods in Molecular Biology. 2052, 373-402 (2020).
- Moon, C., VanDussen, K. L., Miyoshi, H., Stappenbeck, T. S. Development of a primary mouse intestinal epithelial cell monolayer culture system to evaluate factors that modulate IgA transcytosis. Mucosal Immunology. 7 (4), 818-828 (2014).
- VanDussen, K. L., et al. Development of an enhanced human gastrointestinal epithelial culture system to facilitate patient-based assays. Gut. 64 (6), 911-920 (2015).
- Zou, W. Y., et al. Human intestinal enteroids: New models to study gastrointestinal virus infections. Methods in Molecular Biology. 1576, 229-247 (2019).
- Heijmans, J., et al. ER stress causes rapid loss of intestinal epithelial stemness through activation of the unfolded protein response. Cell Reports. 3 (4), 1128-1139 (2013).
- Nik, A. M., Carlsson, P. Separation of intact intestinal epithelium from mesenchyme. Biotechniques. 55 (1), 42-44 (2013).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon