Method Article
Protocoles d’échantillonnage osseux optimisés pour la récupération d’ADN ancien à partir de vestiges archéologiques
Dans cet article
Résumé
Le protocole présente une série de protocoles de meilleures pratiques pour la collecte de poudre osseuse à partir de huit sites d’échantillonnage anatomique recommandés (emplacements spécifiques sur un élément squelettique donné) sur cinq éléments squelettiques différents d’individus médiévaux (datés au radiocarbone à une période d’environ 1040-1400 CE, gamme calibrée 2-sigma).
Résumé
Les méthodes présentées ici cherchent à maximiser les chances de récupération de l’ADN humain à partir de vestiges archéologiques anciens tout en limitant le matériel d’échantillon d’entrée. Cela a été fait en ciblant les emplacements d’échantillonnage anatomique précédemment déterminés pour produire les plus grandes quantités d’ADN ancien (ADNa) dans une analyse comparative de la récupération de l’ADN à travers le squelette. Des recherches antérieures ont suggéré que ces protocoles maximisent les chances de récupération réussie de l’ADN humain ancien et pathogène à partir de vestiges archéologiques. Les rendements en ADN ont déjà été évalués par Parker et al. 2020 dans le cadre d’une vaste étude de la préservation de l’ADN sur plusieurs éléments squelettiques de 11 individus récupérés dans le cimetière médiéval (daté au radiocarbone d’une période d’environ 1040-1400 de notre ère, plage calibrée de 2 sigmas) à Krakauer Berg, une colonie médiévale abandonnée près de Peißen en Allemagne. Ces huit points d’échantillonnage, qui couvrent cinq éléments squelettiques (pars petrosa, molaires permanentes, vertèbre thoracique, phalange distale et talus) ont permis d’obtenir un ADN humain ancien de haute qualité, où les rendements étaient significativement supérieurs à la moyenne globale pour tous les éléments et individus. Les rendements étaient adéquats pour être utilisés dans la plupart des analyses génétiques de populations en aval courantes. Nos résultats soutiennent l’utilisation préférentielle de ces lieux d’échantillonnage anatomique pour la plupart des études impliquant des analyses d’ADN humain ancien à partir de vestiges archéologiques. La mise en œuvre de ces méthodes aidera à minimiser la destruction de précieux spécimens archéologiques.
Introduction
L’échantillonnage de restes humains anciens à des fins de récupération et d’analyse de l’ADN est intrinsèquement destructeur 1,2,3,4. Les échantillons eux-mêmes sont des spécimens précieux et la conservation morphologique doit être préservée dans la mesure du possible. Par conséquent, il est impératif que les pratiques d’échantillonnage soient optimisées pour éviter la destruction inutile de matériel irremplaçable et maximiser les chances de succès. Les techniques actuelles de bonnes pratiques sont basées sur une petite cohorte d’études limitées soit à des enquêtes médico-légales5,6, soit à des études de spécimens anciens où le développement d’un échantillonnage optimal n’est pas l’objectif direct de l’étude7, soit à des études spécifiques utilisant soit des restes non humains8, soit ciblant une très petite sélection de lieux d’échantillonnage anatomique (utilisés ici pour désigner une zone spécifique d’un élément squelettique à partir de laquelle provient la poudre osseuse, pour une utilisation dans les analyses d’ADN en aval, a été généré)9,10. Les protocoles d’échantillonnage présentés ici ont été optimisés dans la première étude systématique à grande échelle de la préservation de l’ADN sur plusieurs éléments squelettiques de plusieurs individus11. Tous les échantillons provenaient d’éléments squelettiques récupérés sur 11 individus excavés du cimetière de l’église de la colonie médiévale abandonnée de Krakauer Berg, près de Peißen, en Saxe-Anhalt, en Allemagne (voir le tableau 1 pour les données démographiques détaillées de l’échantillon) et, en tant que tels, peuvent nécessiter une modification pour être utilisés avec des échantillons en dehors de cette plage géographique / temporelle.
| Individu | Sexe | Âge estimé au décès | 14 Dates C (CE, Cal 2-sigma) |
| KRA001 | Mâle | 25-35 | 1058-1219 |
| KRA002 | Femelle | 20-22 | 1227-1283 |
| KRA003 | Mâle | 25 | 1059-1223 |
| KRA004 | Mâle | 15 | 1284-1392 |
| KRA005 | Mâle | 10-12 | 1170-1258 |
| KRA006 | Femelle | 30-40 | 1218-1266 |
| KRA007 | Femelle | 25-30 | 1167-1251 |
| KRA008 | Mâle | 20 | 1301-1402 |
| KRA009 | Mâle | Inconnu | 1158-1254 |
| KRA010 | Mâle | 25 | 1276-1383 |
| KRA011 | Femelle | 30-45 | 1040-1159 |
Tableau 1 : Sexe génétiquement déterminé, âge estimé au décès déterminé archéologiquement et datation au radiocarbone (14C Cal 2-sigma) pour les 11 individus échantillonnés. Ce tableau a été adapté de Parker, C. et al. 202011.
Ces protocoles permettent une génération relativement simple et efficace de poudre osseuse à partir de huit sites d’échantillonnage anatomique à travers cinq éléments squelettiques (y compris la pars petrosa) avec une contamination limitée de l’ADN induite par le laboratoire. De ces cinq éléments squelettiques, sept emplacements d’échantillonnage anatomique trouvés sur quatre éléments squelettiques ont été jugés être des alternatives viables à l’échantillonnage destructif de la pyramide pétreuse11,12. Ceux-ci comprennent le cément, la dentine et la chambre pulpaire des molaires permanentes; os cortical recueilli à partir de l’encoche vertébrale supérieure ainsi que du corps des vertèbres thoraciques; os cortical provenant de la surface inférieure de la touffe apicale et de la tige des phalanges distales ; et l’os cortical dense le long de la partie extérieure du tali. Bien qu’il existe plusieurs méthodes largement appliquées pour l’échantillonnage de la pars petrosa 4,12,13,14, de la dentine et de la chambre de pulpe dentaire 1,2,15, les méthodes publiées décrivent la génération réussie de poudre d’os à partir du cément 16 , le corps vertébral, l’encoche vertébrale inférieure et le talus peuvent être difficiles à obtenir. En tant que tel, nous démontrons ici des protocoles d’échantillonnage optimisés pour la pyramide pétrauque (étape 3.1); le cément (étape 3.2.1), la dentine (étape 3.2.2) et la pulpe dentaire (étape 3.2.3) des molaires adultes; os cortical du corps vertébral (étape 3.3.1) et arc vertébral supérieur (étape 3.3.2); la phalange distale (étape 3.4); et le talus (étape 3.5) afin de rendre l’utilisation efficace de ces éléments squelettiques pour la recherche sur l’ADNa et la recherche médico-légale plus largement accessible.
Protocole
Toutes les recherches présentées ici ont été effectuées conformément aux directives établies par l’Institut Max Planck pour la science de l’histoire humaine, Iéna, Allemagne pour travailler avec des restes humains anciens. Avant d’effectuer toute étape de ce protocole, assurez-vous de respecter toutes les exigences éthiques locales / étatiques / fédérales relatives à l’obtention d’une autorisation pour l’étude scientifique et à l’utilisation de restes humains pour l’échantillonnage destructif dans votre région. Toutes les procédures et tous les stockages de produits chimiques doivent être effectués conformément aux directives de sécurité de chaque établissement.
1. Considérations préalables au traitement des échantillons
- Traitez les échantillons avec soin, car les restes anciens sont une ressource irreproductible et limitée (p. ex., l’échantillonnage doit être aussi inutile que possible, et tous les restes doivent être retournés à leurs fournisseurs respectifs et légaux si possible).
- Effectuer toutes les étapes dans un environnement de salle blanche, de préférence dans une installation dédiée à l’ADN ancien17,18,19. Utilisez de l’équipement de protection individuelle (EPI) composé d’une combinaison microporeuse stérile avec capuche, de gants stériles (deux paires), d’un masque chirurgical, de lunettes de protection et de bottes stériles ou de chaussures antidérapantes avec couvre-vêtements stériles (voir le tableau des matériaux). Changez de gants fréquemment, surtout entre les échantillons.
- Nettoyez et désinfectez soigneusement tout l’équipement et les surfaces avec de l’eau de Javel/une solution de décontamination de l’ADN/de l’éthanol et de l’irradiation UV (longueur d’onde : 254 nm) dans la mesure du possible (p. ex. forets, forets, étaux/pinces, etc.). Enfin, il est fortement recommandé de prendre des pauses ergonomiques régulières (toutes les 2-3 h si possible) pour éviter l’épuisement excessif dû à l’environnement de la salle blanche.
REMARQUE : Tous les restes squelettiques doivent être documentés de façon appropriée (p. ex., photographiés, pesés et, si possible, scannés par micro-TDM, imagés en 3D, etc.) avant l’échantillonnage (les protocoles pour la documentation appropriée ne sont pas couverts dans ce manuscrit). Tous les protocoles d’échantillonnage peuvent être suspendus entre les itérations d’échantillonnage et les échantillons peuvent être stockés indéfiniment dans un environnement sec, à température contrôlée (25 °C) et stérile.
2. Prétraitement
- Décontaminer tous les sites d’échantillonnage anatomique avant la production de poudre osseuse afin de minimiser le risque de contamination18.
REMARQUE : L’efficacité de l’eau de Javel et/ou de l’élimination de surface (voir la NOTE à l’étape 3.3.2 pour les étapes d’élimination de surface) pour la décontamination des échantillons fait encore l’objet d’un débat parmi les chercheurs en ADNa 8,19,20,21,22,23,24,25, car les deux peuvent influencer les rendements globaux en ADN, en particulier dans les échantillons fortement dégradés. À ce titre, les étapes suivantes sont considérées comme facultatives et sont incluses ici car elles ont été utilisées dans tous les échantillons pour générer les résultats représentatifs présentés dans le présent document. Il est recommandé que l’utilisation de ces protocoles de prétraitement soit déterminée au cas par cas en fonction de l’application moléculaire, de l’âge, de la rareté et du niveau de dégradation morphologique de chaque ensemble d’échantillons.- Effectuer tous les prélèvements dans une salle blanche dédiée sous une hotte de réaction en chaîne par polymérase (PCR) équipée de lumière UV ou une armoire de biosécurité avec le flux d’air désactivé. Étalez une feuille d’aluminium stérile sur la paillasse pour attraper toute poudre / fragment d’os errant.
- S’assurer que tous les fragments d’os sont récupérés (pour rapatriement) avant de se débarrasser de la feuille. Changez la feuille entre le traitement de chaque élément squelettique. Jeter le papier d’aluminium usagé dans un sac/récipient autoclavable pour risque biologique.
- Enlevez autant de saleté ou de détritus que possible des lieux d’échantillonnage anatomique en essuyant doucement la zone avec une lingette sèche stérile non pelucheuse (voir le tableau des matières). Jetez les lingettes dans des sacs ou des récipients autoclavables pour risques biologiques.
- Décontaminer la surface nettoyée en essuyant avec une lingette stérile imbibée d’eau de Javel commerciale diluée (~0,01% v/v, diluée avec de l’eau ultrapure sans DNase/RNase) et laisser incuber pendant 5 min. Jetez les lingettes dans des sacs ou des récipients autoclavables pour risques biologiques.
ATTENTION : L’eau de Javel est un produit chimique hautement corrosif et réactif; Par conséquent, des précautions de sécurité appropriées devraient être mises en place avant son utilisation. - Retirez autant d’eau de Javel résiduelle que possible de l’emplacement d’échantillonnage anatomique à l’aide d’une lingette stérile humidifiée avec de l’eau ultrapure sans DNase/RNase. Jetez les lingettes dans des sacs ou des récipients autoclavables pour risques biologiques.
- Exposer tous les sites d’échantillonnage anatomique nettoyés aux rayons UV pendant 30 minutes (longueur d’onde: 254 nm), puis laisser sécher complètement à température ambiante. S’assurer que les emplacements d’échantillonnage anatomique sont complètement secs avant de procéder à l’échantillonnage ou de retourner à l’entreposage non seulement pour faciliter la production de poudre osseuse, mais aussi pour prévenir la dégradation de l’échantillon (p. ex. moisissure).
ATTENTION : L’exposition aux rayons UV peut être nocive pour les yeux. - Procéder immédiatement à l’échantillonnage ou entreposer les éléments squelettiques dans un environnement stérile sec et à température contrôlée (25 °C).
3. Génération de poudre osseuse
REMARQUE: Les protocoles suivants sont destinés à être utilisés dans l’extraction d’ADN conformément au protocole26 de Dabney et al. 2019.
- Échantillonnage de pars petrosa
REMARQUE : Ce protocole est adapté des procédures décrites dans Pinhasi et al. 20194 et est présenté ici pour faciliter l’utilisation. Ce protocole ne représente pas la méthode actuelle, la moins destructrice, pour l’échantillonnage des pars petrosa. En tant que tel, il est recommandé d’utiliser le protocole décrit par Sirak et al. 201713 ou Orfanou et al. 202014 pour les échantillons dont la conservation morphologique est d’une importance maximale.- Effectuer tous les prélèvements dans une salle blanche dédiée sous une hotte PCR équipée de lumière UV ou une enceinte de biosécurité (longueur d’onde : 254 nm) avec le flux d’air désactivé. Étalez une feuille d’aluminium stérile sur la paillasse pour attraper toute poudre / fragment d’os errant.
- S’assurer que tous les fragments d’os et autant de poudre que possible sont récupérés (pour le rapatriement) avant de jeter le papier d’aluminium. Changez la feuille entre chaque échantillonnage. Jeter la feuille usagée dans un sac ou un récipient autoclavable pour risques biologiques.
- Fixez l’élément sec et décontaminé à l’aide d’une pince ou d’un étau stérilisé.
- Couper le pars petrosa en deux le long du sulcus petrosus supérieur (voir figure 1) à l’aide d’une scie de bijoutier standard équipée d’une lame de 0,6 mm (voir le tableau des matériaux) à vitesse moyenne pour éviter la surchauffe (voir la NOTE ci-dessous étape 3.1.6).
ATTENTION: Le pars petrosa est très dense et, en tant que tel, peut être difficile à couper. Prenez soin de garder l’élément bien serré pour éviter les blessures. Jetez les lames de scie cassées dans le récipient pour objets tranchants appropriés. - Retirez les parties pétreuses de la pince. Récupérez et conservez tout matériau en vrac / en excès.
- Placez le papier de pesage dans un bateau de pesage stérile
- Tenez la partie pétreuse sur le papier de pesée, côté coupé incliné vers le plateau de pesée. Percer dans l’os cortical dense entre le canal facial et l’antre mastoïdien (qui semble plus brillant que le matériau environnant, voir la figure 1) à l’aide d’une perceuse dentaire équipée d’un taillant de petit calibre (voir le tableau des matériaux) et régler à vitesse moyenne et à couple moyen pour produire de la poudre osseuse.
REMARQUE : Le forage/la coupe doit être effectué en courtes rafales à des vitesses faibles à moyennes pour éviter la surchauffe de l’os et la destruction ou l’endommagement de l’ADN. De manière anecdotique, lorsque la partie dense du pétreux commence à surchauffer, une odeur décrite comme du bacon cuit peut être observée. Cesser immédiatement de percer/scier et laisser reposer l’os jusqu’à ce qu’il refroidisse suffisamment avant de reprendre. - Répéter le forage jusqu’à ce qu’environ 50 à 100 mg de poudre soient recueillis dans le papier de pesée, mesuré à l’aide d’une balance fermée d’une précision d’au moins 0,01 mg (voir le tableau des matériaux).
REMARQUE: Dans la mesure du possible, il est suggéré de recueillir 100 mg de poudre d’os pour permettre deux réplications d’extraction d’ADN de 50 mg chacune. Cependant, cela n’est pas toujours possible en raison de la limitation des emplacements d’échantillonnage anatomique eux-mêmes (p. ex., la phalange distale, la chambre de la pulpe dentaire) ou de la nécessité d’une préservation morphologique. Pour d’autres endroits, tels que le ciment, beaucoup moins de 50 mg du matériau peuvent être disponibles. Cependant, il a été démontré que le cément, la chambre de la pulpe dentaire et la phalange distale produisent tous un apport significatif d’ADN endogène 11,27,28, malgré un apport initial plus faible de poudre osseuse provenant du processus d’extraction. - Transférer la poudre du papier de pesée dans un tube de 2 mL étiqueté à faible liaison, à verrouillage sûr pour l’extraction ou le stockage. Conservez les échantillons à -20 °C, indéfiniment.
- Entreposer le reste de la poudre ou de l’excès de poudre dans un environnement sec et stérile à température contrôlée (25 °C) jusqu’à ce que le retour ou le rapatriement puisse être effectué.
- Jetez tous les déchets dans des sacs ou des récipients autoclavables pour risques biologiques. Stériliser/décontaminer tout l’équipement réutilisable (p. ex. pinces, forets, perceuses, scies, etc.) en utilisant de l’eau de Javel/solution de décontamination de l’ADN/éthanol et une exposition aux UV (longueur d’onde : 254 nm), selon le cas, entre chaque échantillonnage.
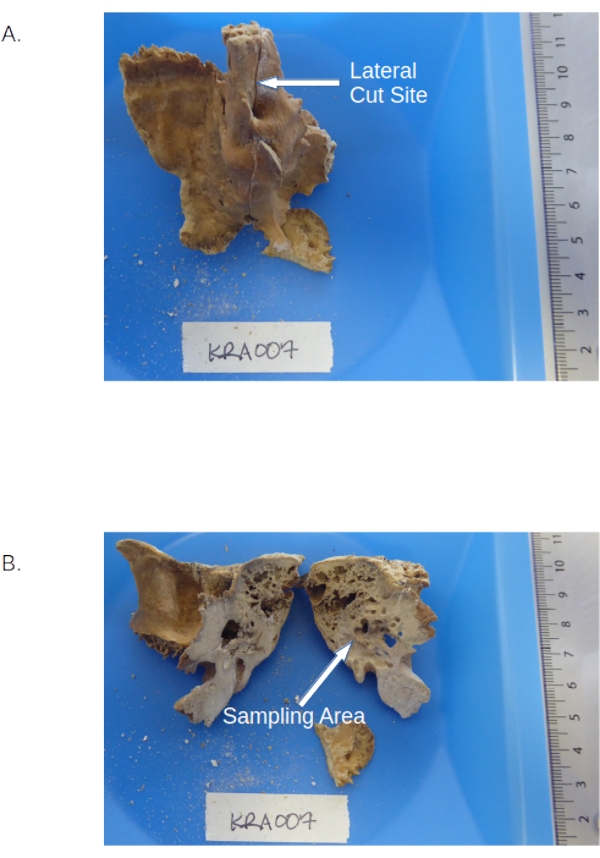
Figure 1 : Os temporal, y compris le pars petrosa. (A) Prédécoupe de l’échantillon montrant les emplacements de la pyramide pétrouse et du sulcus petrosa. (B) Portion pétreuse post-découpe mettant en évidence les zones denses à forer. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Échantillonnage des molaires permanentes
REMARQUE : Pour l’échantillonnage des molaires permanentes, présélectionner in situ molaires avec racines fusionnées et idéalement dépourvues de caries, de fissures dans l’émail ou d’usure excessive pour de meilleurs résultats. Retirer tout prélèvement de tartre dentaire et conserver à -20 °C pour d’éventuelles analyses futures du microbiome oral (procédure non couverte ici).- Échantillonnage du cément
- Effectuer tous les prélèvements dans une salle blanche dédiée sous une hotte PCR équipée de lumière UV ou une enceinte de biosécurité (longueur d’onde : 254 nm) avec le flux d’air désactivé. Étalez une feuille d’aluminium stérile sur la paillasse pour attraper toute poudre / fragment d’os errant.
- S’assurer que tous les fragments d’os et autant de poudre que possible sont récupérés (pour le rapatriement) avant de jeter le papier d’aluminium. Changez la feuille entre chaque échantillonnage. Jeter le papier d’aluminium usagé dans un sac/récipient autoclavable pour risque biologique.
- Placez une feuille de papier de pesée dans un plateau de pesée stérile.
- Tenez/fixez la molaire décontaminée par l’émail, racine vers le bas, sur un plateau de pesée à l’aide d’une pince à main telle qu’une clé réglable (voir le tableau des matériaux).
- Équipez une perceuse dentaire d’une roue de coupe circulaire à bords diamantés. Lorsque la perceuse est réglée sur un réglage vitesse/couple moyen, touchez légèrement le bord du taillant à la racine à un angle d’environ -20°.
- Grattez vers le bas dans le plateau pour enlever / recueillir le matériau jaune le plus externe de la racine (cément). Arrêtez la collecte lorsque le matériau plus léger (blanc) de la dentine devient visible.
REMARQUE: Il est important de faire correspondre le sens de rotation du trépan de coupe par rapport au plateau de collecte pour éviter que la poudre ne devienne aérosolisée et ne gaspille potentiellement l’échantillon en manquant complètement le plateau. Le cément est particulièrement riche en ADN; Cependant, les rendements typiques de matériel sont beaucoup plus faibles que les autres emplacements d’échantillonnage anatomique (~7-20 mg)11,27,28. - Enregistrer la masse de poudre recueillie dans du papier de pesée à l’aide d’une balance fermée avec une précision d’au moins 0,01 mg (voir le tableau des matières).
- Transférer la poudre du papier de pesée dans un tube de verrouillage sûr et à faible liaison de 2 ml pour l’extraction. Conserver indéfiniment à -20 °C.
- Échantillonnage de la chambre pulpaire
- Une fois le cément recueilli (si désiré), sectionnez la molaire le long de la jonction cémento-émail à l’aide d’une scie de bijoutier pour retirer la couronne (voir la figure 2).
- Placez une nouvelle feuille de papier de pesée dans un nouveau plateau de pesée.
- Fixez la section de la couronne dans une pince à main ou un étau, sur le plateau de pesée. Maintenir le côté coupé incliné vers le bas et percer/gratter le matériau lors de la première passe avec une perceuse dentaire munie d’un foret de petit calibre (voir le tableau des matériaux) le long des bords de la chambre à pâte à l’intérieur de la partie couronne (voir la figure 2).
REMARQUE: Seul le premier passage de l’intérieur de la chambre pulpaire doit être recueilli et étiqueté comme matériau de pulpe (rendement typique de 5-15 mg), tout ce qui est plus profond dans la dent est considéré comme de la dentine. - Tournez la dent avec la partie inférieure vers le bas, tapotez la pince avec un marteau et recueillez la poudre libérée sur le papier de pesée.
- Notez le poids de la poudre recueillie dans le papier de pesée à l’aide d’une balance jointe avec une précision d’au moins 0,01 mg (voir le tableau des matières).
- Transférer la poudre du papier de pesage dans un tube à faible liaison et à verrouillage sûr de 2 ml pour l’extraction. Conserver indéfiniment à -20 °C.
- Échantillonnage de la dentine
- Placez une nouvelle feuille de papier de pesée dans un nouveau plateau de pesée.
- Tenir la section de la couronne au-dessus du plateau de pesée (conformément à l’étape 3.2.2.3), forer et recueillir 50 à 100 mg supplémentaires de dentine, mesurée à l’aide d’une balance fermée d’une précision de 0,01 mg (voir le tableau des matériaux) à l’intérieur de la chambre de pâte de la même manière pour un échantillonnage plus poussé de la dentine (voir la figure 2).
- Transférer la poudre d’os du papier de pesée dans un tube à faible liaison et à verrouillage sécuritaire de 2 ml pour l’extraction. Conserver indéfiniment à -20 °C.
- Conservez les dents restantes/l’excès de poudre dans un environnement stérile sec et à température contrôlée (25 °C) jusqu’à ce que le retour ou le rapatriement puisse être terminé.
- Jetez tous les déchets dans des sacs ou des récipients autoclavables pour risques biologiques. Stériliser/décontaminer tout l’équipement réutilisable (p. ex. pinces, forets, perceuses, scies, etc.) en utilisant une solution d’eau de Javel/solution de décontamination de l’ADN/éthanol et des expositions aux UV (longueur d’onde : 254 nm), selon le cas, entre chaque échantillonnage.
- Échantillonnage du cément
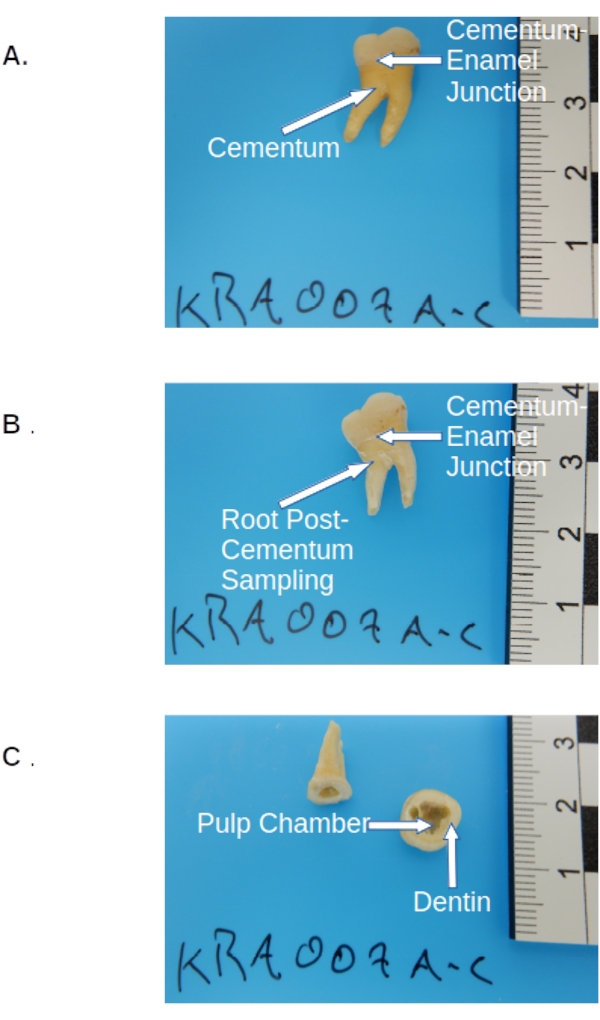
Figure 2 : Prééchantillonnage molaire permanent. (A) Molaire prétraitée avant l’échantillonnage, montrant la couronne, le cément (couche jaunâtre de la racine) et le site de coupe à la jonction cémento-émail. (B) La même collection molaire post-cément, montrant le site de coupe à la jonction cémento-émail. (C) Post-coupe molaire et échantillonnage montrant les emplacements anatomiques d’échantillonnage de la chambre pulpaire dentaire et de la dentine à l’intérieur de la couronne. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Échantillonnage des vertèbres thoraciques
- Prélèvement du corps vertébral
- Effectuer tous les prélèvements dans une salle blanche dédiée sous une hotte PCR équipée de lumière UV ou une enceinte de biosécurité (longueur d’onde : 254 nm) avec le flux d’air désactivé. Étalez une feuille d’aluminium stérile sur la paillasse pour attraper toute poudre / fragment d’os errant.
- S’assurer que tous les fragments d’os et autant de poudre que possible sont récupérés (pour le rapatriement) avant de jeter le papier d’aluminium. Changez la feuille entre chaque échantillonnage. Jeter le papier d’aluminium usagé dans un sac/récipient autoclavable pour risque biologique.
- Placez une petite feuille de papier de pesée dans un plateau de pesée standard.
- Fixez les vertèbres avec une pince ou un étau à main, avec le corps vertébral vers l’extérieur.
- Tenez les vertèbres au-dessus du plateau de pesée avec le corps vertébral incliné vers le bas. À l’aide d’une perceuse dentaire équipée d’un foret de petit calibre (voir le tableau des matériaux) réglé à un couple élevé à basse vitesse, percez le long du bord le plus externe (inférieur et supérieur) de l’os cortical entourant le tissu interne spongieux du corps vertébral (voir la figure 3).
- Grattez le trépan contre la couche corticale sur un plateau de pesée standard jusqu’à ce que 50 à 100 mg de matériau soient recueillis, mesurés à l’aide d’une balance fermée précise à 0,01 mg (voir le tableau des matériaux).
- Transférer la poudre d’os du papier de pesée vers un tube de verrouillage sûr et à faible liaison de 2 ml pour l’extraction. Conserver indéfiniment à -20 °C.
- Échantillonnage de l’arc vertébral supérieur
Remarque : Cette étape est facultative. Enlevez et jetez la couche la plus externe de l’os cortical de l’arc vertébral supérieur à l’aide d’une perceuse dentaire munie d’un foret de petit calibre (voir Tableau des matériaux) en la grattant le long de la surface19. Ceci n’est pas suggéré pour l’échantillonnage du corps vertébral, car la couche d’os cortical est généralement très mince et susceptible d’être entièrement épuisée par ce processus (voir NOTE dans la rubrique 2).- Placez une petite feuille de papier de pesée dans un plateau de pesée standard.
- Fixez les vertèbres dans une pince à main / étau avec le processus vertébral vers l’extérieur, aspect supérieur vers le bas.
- Tout en maintenant les vertèbres, aspect supérieur vers le bas, sur un plateau de pesée, percez vers le haut au centre de l’encoche en forme de V formée par la fusion de l’apophyse épineuse, avec les lamelles (voir la figure 3) à l’aide d’une perceuse dentaire avec un taillant de petit calibre (voir le tableau des matériaux) réglé à basse vitesse et à couple élevé.
- Cesser de forer lorsqu’il y a une baisse notable de la résistance. Modifier légèrement la position de forage et répéter jusqu’à ce que 50 à 100 mg de poudre d’os soient recueillis, mesurés à l’aide d’une balance fermée précise à 0,01 mg (voir le tableau des matériaux).
- Transférer la poudre d’os du papier de pesée dans un tube à faible liaison de 2 ml pour l’extraction. Conserver indéfiniment à -20 °C.
- Conserver le reste de la poudre ou de l’excès dans un environnement sec et stérile à température contrôlée (25 °C) jusqu’au retour ou au rapatriement.
- Jetez tous les déchets dans des sacs ou des récipients autoclavables pour risques biologiques. Stériliser/décontaminer tout l’équipement réutilisable (p. ex. pinces, forets, perceuses, scies, etc.) à l’aide d’une solution d’eau de Javel/d’ADN/éthanol et d’une exposition aux UV (longueur d’onde : 254 nm), selon le cas, entre chaque échantillonnage.
- Prélèvement du corps vertébral

Figure 3 : Emplacements anatomiques de prélèvement anatomique du corps vertébral et de l’os cortical de l’arc vertébral supérieur de la vertèbre thoracique. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Échantillonnage de la phalange distale
Remarque : Cette étape est facultative. Enlever et jeter la couche la plus externe de l’os cortical de la tige et/ou de la touffe apicale à l’aide d’une perceuse dentaire équipée d’un foret de petit calibre en la grattant le long de la surface19. Cela peut ne pas être possible pour les échantillons présentant des restes osseux corticaux ou juvéniles excessivement minces (voir NOTE à la rubrique 2).- Effectuer tous les prélèvements dans une salle blanche dédiée, sous une hotte PCR équipée de lumière UV ou une armoire de biosécurité (longueur d’onde UV : 254 nm) avec le flux d’air désactivé. Étalez une feuille d’aluminium stérile sur la paillasse pour attraper toute poudre / fragment d’os errant.
- S’assurer que tous les fragments d’os et autant de poudre que possible sont récupérés (pour le rapatriement) avant de jeter le papier d’aluminium. Changez la feuille entre chaque échantillonnage. Jeter le papier d’aluminium usagé dans un sac/récipient autoclavable pour risque biologique.
- Placez une petite feuille de papier de pesée dans un plateau de pesée standard.
- Fixez l’échantillon dans une pince portative / étau, côté supérieur vers le haut.
- Tenir l’échantillon au-dessus de la plate-forme de pesée, recueillir la poudre d’os de l’os cortical du côté inférieur de la touffe apicale et de la tige en forant à travers les couches denses les plus externes (voir la figure 4) à l’aide d’une perceuse dentaire équipée d’un foret de petit calibre (voir le tableau des matériaux).
- Cessez de forer lorsqu’il y a une diminution marquée de la résistance, car cela signifie un matériau plus léger et spongieux. Répétez ce processus, en rayonnant vers l’extérieur à partir du forage initial jusqu’à ce qu’au moins 50 à 100 mg de poudre d’os soient recueillis, mesurés à l’aide d’une balance fermée précise à 0,01 mg (voir le tableau des matériaux).
- Transférer la poudre d’os du papier de pesée dans un tube à faible liaison et à verrouillage sécuritaire de 2 ml pour l’extraction. Conserver indéfiniment à -20 °C.
- Conserver le reste de l’os/excès de poudre dans un environnement sec et stérile à température contrôlée (25 °C) jusqu’au retour ou au rapatriement.
- Jetez tous les déchets dans des sacs ou des récipients autoclavables pour risques biologiques. Stériliser/décontaminer tout l’équipement réutilisable (p. ex. pinces, forets, perceuses, scies, etc.) à l’aide d’eau de Javel/d’une solution de décontamination de l’ADN/d’éthanol et d’une exposition aux UV, selon le cas, entre chaque échantillonnage.
REMARQUE : Pour les échantillons plus petits (p. ex. échantillons juvéniles), il peut y avoir beaucoup moins que les 50 à 100 mg d’os cortical suggérés disponibles pour l’échantillon. Cependant, même en faible quantité, ce lieu d’échantillonnage anatomique s’est avéré particulièrement riche en ADN11.

Figure 4 : Phéloange distale montrant les emplacements de l’os cortical dense le long de la tige et du côté inférieur de la touffe apicale. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Échantillonnage du Talus
- Effectuer tous les prélèvements dans une salle blanche dédiée sous une hotte PCR équipée de lumière UV ou une enceinte de biosécurité (longueur d’onde : 254 nm) avec le flux d’air désactivé. Étalez une feuille d’aluminium stérile sur la paillasse pour attraper toute poudre / fragment d’os errant.
- S’assurer que tous les fragments d’os et autant de poudre que possible sont récupérés (pour le rapatriement) avant de jeter le papier d’aluminium. Changez la feuille entre chaque échantillonnage. Jeter le papier d’aluminium usagé dans un sac/récipient autoclavable pour risque biologique.
- Placez une petite feuille de papier de pesée dans un plateau de pesée standard.
- Fixez l’échantillon dans une pince / étau portatif, dôme vers le haut.
- Tenez le talus, le dôme vers le haut et la surface médiale vers le collecteur, au-dessus du plateau de pesée. Grattez l’os cortical du col du talus jusqu’à une profondeur de ~1 mm (voir la figure 5) à l’aide d’une perceuse dentaire avec un taillant de faible calibre (voir le tableau des matériaux) réglé à basse vitesse et à couple élevé.
- Modifier légèrement la position de forage et répéter jusqu’à ce qu’environ 50 à 100 mg de poudre d’os soient recueillis, mesurés à l’aide d’une balance fermée précise à 0,01 mg (voir le tableau des matériaux).
- Transférer la poudre d’os du papier de pesée dans un tube à faible liaison de 2 ml pour l’extraction. Conserver indéfiniment à -20 °C.
- Entreposer le reste de l’os/excès de poudre dans un environnement sec et stérile à température contrôlée (25 °C) jusqu’à ce que le retour ou le rapatriement puisse être terminé.
- Jetez tous les déchets dans des sacs ou des récipients autoclavables pour risques biologiques. Stériliser/décontaminer tout l’équipement réutilisable (p. ex. pinces, forets, perceuses, scies, etc.) en utilisant de l’eau de Javel/solution de décontamination de l’ADN/éthanol et une exposition aux UV (longueur d’onde : 254 nm), selon le cas, entre chaque échantillonnage.
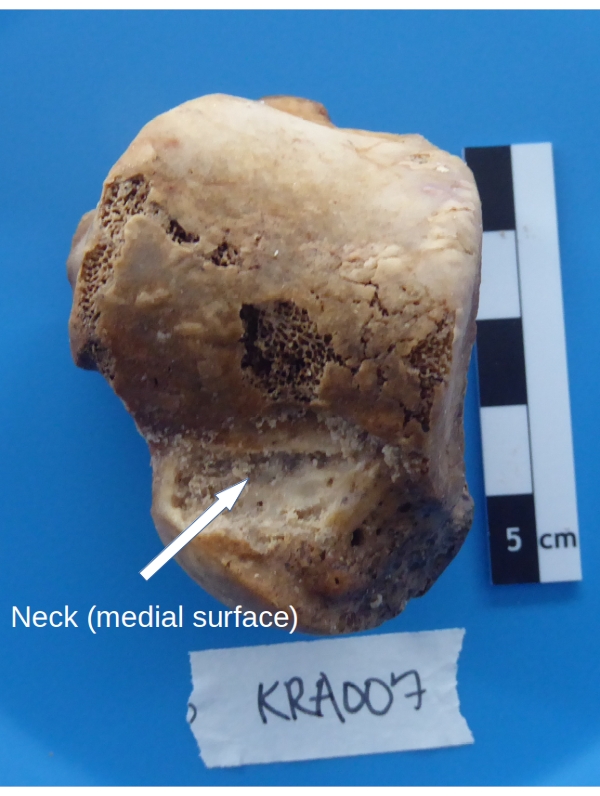
Figure 5 : Zone d’échantillonnage du talus pour la récupération de l’os cortical. Veuillez cliquer ici pour voir une version agrandie de cette figure.
REMARQUE: Le talus a très peu d’os cortical (une fine couche externe). Le matériau doit non seulement être recueilli à la surface, mais également à la couche dense sous-jacente d’os spongieux.
Résultats
Dans une étude distincte 11, l’ADN a été extrait de la poudre d’os générée à partir de chaque emplacement d’échantillonnage anatomique chez11 individus, en utilisant un protocole d’extraction d’ADN standard optimisé pour de courts fragments de tissu calcifié2. Des bibliothèques simple brin ont ensuite été produites28 et séquencées sur un HiSeq 4000 (extrémité appariée de 75 pb) à une profondeur de ~20 000 000 lectures par échantillon. Les données de séquence résultantes ont ensuite été évaluées pour le contenu endogène de l’ADN humain à l’aide du pipeline EAGER29 (paramètres BWA: longueur de la graine de 32, pénalité de non-concordance de 0,1, filtre de qualité de cartographie de 37). Tous les résultats représentatifs sont présentés en utilisant les mêmes paramètres que Parker et al. 202011 pour la cohérence. Les bibliothèques des parties en poudre de la pars petrosa ont produit, en moyenne, un ADN endogène plus élevé que n’importe lequel des 23 autres sites d’échantillonnage anatomique étudiés (figure 6A-B). Les sept autres sites d’échantillonnage anatomique présentés dans ce protocole (le cément, le premier passage de la chambre pulpaire dentaire et la dentine des molaires permanentes; l’os cortical du corps vertébral et l’arc vertébral supérieur de la vertèbre thoracique; l’os cortical de la touffe apicale de la phalange distale; et l’os cortical du col du talus) ont produit les rendements les plus élevés suivants (sans signification statistique entre ces emplacements d’échantillonnage anatomique; Figure 6A-B; Fichier supplémentaire 1 : EndogenousDNAPreCap). Ces emplacements alternatifs ont tous produit de manière cohérente des rendements d’ADN adéquats pour les analyses de génétique des populations standard telles que les analyses mitochondriales et les analyses de polymorphisme mononucléotidique (SNP). Les taux de duplication dans les bibliothèques provenant de tous les emplacements d’échantillonnage anatomique étaient faibles (facteurs de grappe < 1,2 en moyenne, calculés comme le rapport entre toutes les lectures cartographiques et les lectures cartographiques uniques, tableau 2; Fichier supplémentaire 1 : ClusterFactor), indiquant que toutes les bibliothèques examinées étaient d’une très grande complexité. De même, les estimations moyennes de la contamination exogène de l’ADN humain étaient faibles, atteignant en moyenne < 2 % (contamination par le chromosome X chez les mâles, n = 7, telle que rapportée par le pipeline ANGSD30) dans tous les sites d’échantillonnage anatomique, à l’exception de l’arc vertébral supérieur (contamination moyenne estimée : 2,11 %, avec un échantillon prélevé comme valeur aberrante; KRA005: 19,52%, voir tableau 2; Fichier supplémentaire 1 : Xcontamination). La longueur moyenne des fragments (après filtrage pour éliminer toutes les lectures < 30 pb) était la plus faible dans le matériau prélevé dans la chambre pulpaire dentaire et la dentine, sans variation significative entre les autres emplacements d’échantillonnage anatomique (55,14 pb et 60,22 pb, respectivement, par rapport à une médiane moyenne de 62,87, valeurs p par paires < 0,019, tableau 2; Fichier supplémentaire 1 : AvgFragLength). De plus, les dents et les vertèbres thoraciques contiennent chacune plusieurs emplacements d’échantillonnage anatomique où une récupération élevée de l’ADN endogène a été observée, ce qui les rend particulièrement appropriées comme alternatives à la pars petrosa.
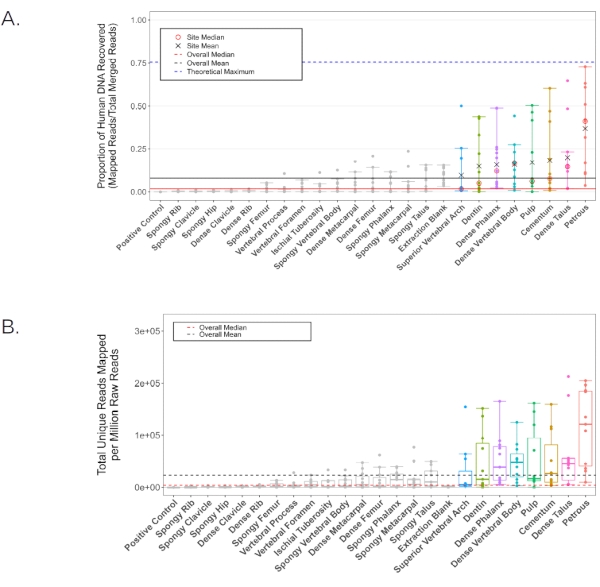
Figure 6 : Contenu de l’ADN humain pour tous les échantillons sélectionnés. Les lignes noires représentent la moyenne globale, tandis que les lignes rouges représentent la médiane (solide: proportion d’ADN humain, en pointillés: lectures humaines cartographiées par million de lectures générées). Les emplacements d’échantillonnage anatomique individuels avec une proportion moyenne d’ADN humain supérieure à la moyenne globale (8,16%) sont colorisés dans toutes les analyses. (A) La proportion de lectures cartographiées avec le génome de référence hg19. La ligne pointillée bleue représente le maximum théorique compte tenu des paramètres de cartographie du pipeline (générés à l’aide de Gargammel31 pour simuler une distribution aléatoire de 5 000 000 de lectures du génome de référence hg19 avec des dommages simulés). Les moyennes individuelles (X noir) et les médianes (cercle rouge) sont rapportées pour les échantillons dont la proportion moyenne d’ADN humain est supérieure à la moyenne globale. Les intervalles de confiance indiquent les limites supérieure et inférieure à l’exclusion des valeurs statistiques aberrantes. (B) Le nombre de lectures uniques correspondant au génome de référence hg19 par million de lectures d’effort de séquençage (extrémité appariée de 75 pb). Les intervalles de confiance indiquent les limites supérieure et inférieure à l’exclusion des valeurs statistiques aberrantes. Cette figure a été adaptée de Parker, C. et al. 202011. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau 2 : Niveaux moyens de duplication (lectures cartographiques/lectures uniques), longueurs moyennes et médianes des fragments et estimations de la contamination du chromosome X pour tous les emplacements d’échantillonnage anatomique. Erreur signalée comme erreur-type de la moyenne. Ce tableau a été adapté de Parker, C. et al. 202011.
| Lieu d’échantillonnage | Facteur de duplication moyen (# lectures mappées /# lectures mappées uniques) | Longueur moyenne des fragments en pb | Proportion moyenne estimée de contamination par le chromosome X |
| Pyramide pétreuse | 1,188 ± 0,006 | 65,40 ± 1,36 | 0,000 ± 0,003 |
| Cément | 1,197 ± 0,028 | 67,28 ± 1,76 | 0,011 ± 0,003 |
| Dentine | 1,188 ± 0,061 | 60,22 ± 2,37 | 0,002 ± 0,007 |
| Pulpe | 1,179 ± 0,024 | 55,14 ± 2,90 | 0,013 ± 0,006 |
| phalange distale | 1,191 ± 0,049 | 65,95 ± 1,08 | 0,013 ± 0,005 |
| Corps vertébral | 1,194 ± 0,037 | 66,14 ± 1,03 | 0,008 ± 0,003 |
| Arc vertébral supérieur | 1,19 ± 0,017 | 63,02 ± 1,23 | 0,021 ± 0,009* |
| Astragale | 1,198 ± 0,010 | 68,20 ± 1,24 | 0,011 ± 0,003 |
| *Échantillon KRA005 retiré comme valeur aberrante à 0,1952 |
Disponibilité du code
Tous les programmes d’analyse et modules R utilisés dans les analyses de ce manuscrit sont disponibles gratuitement auprès de leurs auteurs respectifs. Tous les codes R personnalisés sont disponibles sur demande.
Disponibilité des données
Toutes les données brutes utilisées dans le calcul des résultats représentatifs sont disponibles gratuitement dans le dépôt de données ENA European Nucleotide Archive (numéro d’acquisition PRJ-EB36983) ou dans des documents supplémentaires de Parker, C. et al.11.
Fichier supplémentaire 1. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
La pratique actuelle en génétique des populations humaines anciennes consiste à prélever des échantillons préférentiels à partir de la pars petrosa (étape 2.1) chaque fois que cela est possible. Cependant, la pars petrosa peut être un échantillon difficile à obtenir, car elle est très appréciée pour une myriade d’évaluations du squelette (par exemple, l’histoire de la population32, l’estimation de l’âge fœtal au décès 33 et la détermination du sexe34) et, historiquement, l’échantillonnage de la pars petrosa pour l’analyse de l’ADN peut être très destructeur3,4 (y compris le protocole présenté ici, Bien que de nouveaux protocoles mini-invasifs13,14 aient maintenant été largement adoptés pour atténuer cette préoccupation). Cette situation est aggravée par le fait que, jusqu’à très récemment, aucune étude systématique à grande échelle de la récupération de l’ADN humain à travers le squelette n’avait été tentée11, ce qui rend difficile la recherche d’une stratégie d’échantillonnage appropriée lorsque la pyramide pétreuse n’est pas disponible.
Les protocoles présentés ici aident à atténuer ce défi en fournissant un ensemble de procédures optimisées pour l’échantillonnage de l’ADN à partir de restes squelettiques archéologiques / médico-légaux, y compris le pars petrosa, ainsi que sept emplacements anatomiques alternatifs d’échantillonnage sur quatre éléments squelettiques supplémentaires. Les étapes critiques incluses visent toutes à minimiser la possibilité de perte/dommage à l’ADN dû à un échantillonnage inefficace (étapes 2.1.6 et 3.2.1.3) ou à une surchauffe des échantillons pendant le forage ou la découpe (étape 3.1.6). De plus, il a été noté tout au long du protocole qu’il peut être nécessaire de modifier ou d’omettre les étapes de prétraitement pour assurer la meilleure performance dans les échantillons fortement dégradés. Il convient également de noter que même parmi les éléments sélectionnés présentés ici, il reste plusieurs techniques d’échantillonnage alternatives possibles (en particulier pour les pars petrosa13,14), ainsi que de nombreuses possibilités d’optimisation des emplacements d’échantillonnage anatomiques sous-exploités présentés ici (c’est-à-dire le talus: étape 2.5 et les vertèbres: étape 2.3).
Il est également important de garder à l’esprit que ces protocoles ont été conçus et testés à l’aide d’anciens restes juvéniles-adultes de haute qualité (bonne conservation morphologique) à des fins d’analyses endogènes de l’ADN humain. Les résultats présentés peuvent ne pas s’étendre à des matériaux plus fortement dégradés, à d’autres contextes de préservation, à des restes de nourrissons, à des restes non humains ou à des études sur les agents pathogènes ou le microbiome, car une exploration plus approfondie de l’utilisation de ces protocoles dans d’autres contextes est encore nécessaire. De plus, les éléments squelettiques alternatifs présentés ici (les dents, les vertèbres, la phalange distale et le tali) peuvent être difficiles à attribuer à un seul individu parmi les restes mélangés, nécessitant un échantillonnage à partir de plusieurs éléments pour assurer une origine unique. Malgré ces limites, le fait de rendre ces protocoles largement accessibles peut aider à atténuer une partie de l’hétérogénéité entourant la sélection et le traitement des échantillons en fournissant un cadre généralisé et quantitativement optimisé pour une utilisation dans un large éventail d’études futures sur l’ADNa / médico-légale sur les restes humains.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à signaler.
Remerciements
Les auteurs tiennent à remercier le personnel de laboratoire de l’Institut Max Planck pour la science de l’histoire humaine pour leur aide dans l’élaboration et la mise en œuvre de ces protocoles. Ce travail n’aurait pas été possible sans l’apport et le travail acharné du Dr Guido Brandt, de la Dre Elizabeth Nelson, d’Antje Wissegot et de Franziska Aron. Cette étude a été financée par la Société Max Planck, le Conseil européen de la recherche (CER) dans le cadre du programme de recherche et d’innovation Horizon 2020 de l’Union européenne en vertu des conventions de subvention n ° 771234 - PALEoRIDER (WH, ABR) et de la subvention de démarrage n ° 805268 CoDisEASe (à KIB).
matériels
| Name | Company | Catalog Number | Comments |
| #16 Dental Drill Bit | NTI | H1-016-HP | example drilling bit |
| 0.6 mm scroll saw blade | Fisher Scientific | 50-949-097 | blade for Jewellers Saw |
| 22mm diamond cutting wheel | Kahla | SKU 806 104 358 514 220 | Dremel cutting attachment |
| Commercial Bleach | Fisher Scientific | NC1818018 | |
| Control Company Ultra-Clean Supreme Aluminum Foil | Fisher Scientific | 15-078-29X | |
| DNA LoBind Tubes (2 mL) | Eppendorf | 22431048 | |
| Dremel 225-01 Flex Shaft Attachment | Dremel | 225-01 | Dremel flexible extension |
| Dremel 4300 Rotary Tool | Dremel | 4300 | Example drill |
| Dremel collet and nut kit | Dremel | 4485 | Adapters for various Dremel tool attachments/bits |
| Eagle 33 Gallon Red Biohazard Waste Bag | Fisher Scientific | 17-988-501 | |
| Eppendorf DNA LoBind 2 mL microcentrifuge tube | Fisher Scientific | 13-698-792 | |
| Ethanol (Molecular Biology Grade) | Millipore Sigma | 1.08543 | |
| FDA approved level 2 Surgical Mask | Fisher Scientific | 50-206-0397 | PPE |
| Fisherbrand Comfort Nitrile Gloves | Fisher Scientific | 19-041-171X | PPE |
| Fisherbrand Safety Glasses | Fisher Scientific | 19-130-208X | PPE |
| Granger Stationary Vise | Fisher Scientific | NC1336173 | benchtop vise |
| Invitrogen UltraPure DNase/Rnase free distilled water | Fisher Scientific | 10-977-023 | |
| Jewellers Saw | Fisher Scientific | 50-949-231 | |
| Kimwipes | Sigma-Aldritch | Z188956 | |
| Labconco Purifier Logic Biosafety cabinet | Fisher Scientific | 30-368-1101 | |
| LookOut DNA Erase | Millipore Sigma | L9042-1L | |
| Medium weighing boat | Heathrow Scientific | HS120223 | |
| MSC 10pc plier/clamp set | Fisher Scientific | 50-129-5352 | Miscellaneous clamps/vise grips for securely holding samples while drilling/cutting |
| Sartorius Quintix Semi-Micro Balance | Fisher Scientific | 14-560-019 | enclosed balance |
| Tyvek coveralls with hood | Fisher Scientific | 01-361-7X | PPE |
| Weigh paper | Heathrow Scientific | HS120116 |
Références
- Adler, C. J., Haak, W., Donlon, D., Cooper, A. Survival and recovery of DNA from ancient teeth and bones. Journal of Archaeological Science. 38 (5), 956-964 (2011).
- Dabney, J., Meyer, M. Extraction of highly degraded DNA from ancient bones and teeth. Ancient DNA: Methods and Protocols. , 25-29 (2019).
- Palsdottir, A. H., Bläuer, A., Rannamäe, E., Boessenkool, S., Hallsson, J. Not a limitless resource: ethics and guidelines for destructive sampling of archaeofaunal remains. Royal Society Open Science. 6 (10), 191059 (2019).
- Pinhasi, R., Fernandes, D. M., Sirak, K., Cheronet, O. Isolating the human cochlea to generate bone powder for ancient DNA analysis. Nature Protocols. 14 (4), 1194-1205 (2019).
- Latham, K. E., Miller, J. J. DNA recovery and analysis from skeletal material in modern forensic contexts. Forensic Sciences Research. 4 (1), 51-59 (2019).
- Mundorff, A. Z., Bartelink, E. J., Mar-Cash, E. DNA preservation in skeletal elements from the World Trade Center disaster: Recommendations for mass fatality management. Journal of Forensic Sciences. 54 (4), 739-745 (2009).
- Gamba, C., et al. Genome flux and stasis in a five millennium transect of European prehistory. Nature Communications. 5 (1), 1-9 (2014).
- Alberti, F., et al. Optimized DNA sampling of ancient bones using Computed Tomography scans. Molecular Ecology Resources. 18 (6), 1196-1208 (2018).
- Hansen, H. B., et al. Comparing ancient DNA preservation in petrous bone and tooth cementum. PLoS ONE. 12 (1), 0170940 (2017).
- Sirak, K., et al. Human auditory ossicles as an alternative optimal source of ancient DNA. Genome Research. 30 (3), 427-436 (2020).
- Parker, C., et al. A systematic investigation of human DNA preservation in medieval skeletons. Scientific Reports. 10 (1), 18225 (2020).
- Pinhasi, R., et al. Optimal ancient DNA yields from the inner ear part of the human petrous bone. PLoS ONE. 10 (6), 0129102 (2015).
- Sirak, K. A., et al. A minimally-invasive method for sampling human petrous bones from the cranial base for ancient DNA analysis. BioTechniques. 62 (6), 283-289 (2017).
- . Minimally-invasive sampling of pars petrosa (os temporale) for ancient DNA extraction. protocols.io Available from: https://www.protocols.io/view/minimally-invasive-sampling-of-pars-petrosa-os-tem-bqd8ms9w (2020)
- Damgaard, P. B., et al. Improving access to endogenous DNA in ancient bones and teeth. Scientific Reports. 5 (1), 1-12 (2015).
- Harney, &. #. 2. 0. 1. ;., et al. A minimally destructive protocol for DNA extraction from ancient teeth. Genome Research. 31 (3), 472-483 (2021).
- Cooper, A., Poinar, H. N. Ancient DNA: Do it right or not at all. Science. 289 (5482), 1139 (2000).
- Llamas, B., et al. From the field to the laboratory: Controlling DNA contamination in human ancient DNA research in the high-throughput sequencing era. STAR: Science & Technology of Archaeological Research. 3 (1), 1-14 (2017).
- Llamas, B., et al. From the field to the laboratory: Controlling DNA contamination in human ancient DNA research in the high-throughput sequencing era. STAR: Science & Technology of Archaeological Research. 3 (1), 1-14 (2017).
- Boessenkool, S., et al. Combining bleach and mild predigestion improves ancient DNA recovery from bones. Molecular Ecology Resources. 17 (4), 742-751 (2017).
- García-Garcerà, M., et al. Fragmentation of contaminant and endogenous DNA in ancient samples determined by shotgun sequencing; Prospects for human palaeogenomics. PLoS ONE. 6 (8), 24161 (2011).
- Malmström, H., et al. More on contamination: The use of asymmetric molecular behavior to identify authentic ancient human DNA. Molecular Biology and Evolution. 24 (4), 998-1004 (2007).
- Basler, N., et al. Reduction of the contaminant fraction of DNA obtained from an ancient giant panda bone. BMC Research Notes. 10, 754 (2017).
- Kemp, B. M., Smith, D. G. Use of bleach to eliminate contaminating DNA from the surface of bones and teeth. Forensic Science International. 154 (1), 53-61 (2005).
- Korlević, P., et al. Reducing microbial and human contamination in DNA extractions from ancient bones and teeth. BioTechniques. 59 (2), 87-93 (2015).
- Dabney, J., Meyer, M. Extraction of highly degraded DNA from ancient bones and teeth. Methods in Molecular Biology. 1963, 25-29 (2019).
- Hansen, H. B., et al. Comparing ancient DNA preservation in petrous bone and tooth cementum. PLoS ONE. 12 (1), 0170940 (2017).
- Gansauge, M. -. T., et al. Single-stranded DNA library preparation from highly degraded DNA using T4 DNA ligase. Nucleic Acids Research. 45 (10), 79 (2017).
- Peltzer, A., et al. EAGER: efficient ancient genome reconstruction. Genome Biology. 17 (1), 60 (2016).
- Korneliussen, T. S., Albrechtsen, A., Nielsen, R. ANGSD: analysis of next generation sequencing data. BMC Bioinformatics. 15, 356 (2014).
- Renaud, G., Hanghøj, K., Willerslev, E., Orlando, L. Gargammel: A sequence simulator for ancient DNA. Bioinformatics. 33 (4), 577-579 (2017).
- Ponce de León, M. S., et al. Human bony labyrinth is an indicator of population history and dispersal from Africa. Proceedings of the National Academy of Sciences of the United States of America. 115 (16), 4128-4133 (2018).
- Nagaoka, T., Kawakubo, Y. Using the petrous part of the temporal bone to estimate fetal age at death. Forensic Science International. 248, 188 (2015).
- Norén, A., Lynnerup, N., Czarnetzki, A., Graw, M. Lateral angle: A method for sexing using the petrous bone. American Journal of Physical Anthropology. 128 (2), 318-323 (2005).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon