Method Article
Protocolli di campionamento osseo ottimizzati per il recupero di DNA antico da resti archeologici
In questo articolo
Riepilogo
Il protocollo presenta una serie di protocolli di best practice per la raccolta di polvere ossea da otto posizioni di campionamento anatomico raccomandate (posizioni specifiche su un dato elemento scheletrico) attraverso cinque diversi elementi scheletrici di individui medievali (datato al radiocarbonio a un periodo di circa 1040-1400 CE, calibrato intervallo 2-sigma).
Abstract
I metodi qui presentati cercano di massimizzare le possibilità di recupero del DNA umano da antichi resti archeologici, limitando il materiale campione in ingresso. Ciò è stato fatto prendendo di mira posizioni di campionamento anatomico precedentemente determinate per produrre le più alte quantità di DNA antico (aDNA) in un'analisi comparativa del recupero del DNA attraverso lo scheletro. Ricerche precedenti hanno suggerito che questi protocolli massimizzano le possibilità di successo del recupero di DNA umano antico e patogeno dai resti archeologici. Le rese del DNA sono state precedentemente valutate da Parker et al. 2020 in un'ampia indagine sulla conservazione dell'aDNA attraverso più elementi scheletrici di 11 individui recuperati dal cimitero medievale (datato al radiocarbonio a un periodo di circa (ca.) 1040-1400 CE, calibrato intervallo 2-sigma) a Krakauer Berg, un insediamento medievale abbandonato vicino a Peißen in Germania. Questi otto punti di campionamento, che si estendono su cinque elementi scheletrici (pars petrosa, molari permanenti, vertebra toracica, falange distale e astragalo) hanno prodotto con successo DNA umano antico di alta qualità, dove i rendimenti erano significativamente superiori alla media complessiva di tutti gli elementi e gli individui. Le rese erano adeguate per l'uso nelle analisi genetiche più comuni della popolazione a valle. I nostri risultati supportano l'uso preferenziale di questi luoghi di campionamento anatomici per la maggior parte degli studi che coinvolgono l'analisi del DNA umano antico da resti archeologici. L'implementazione di questi metodi contribuirà a ridurre al minimo la distruzione di preziosi esemplari archeologici.
Introduzione
Il campionamento di antichi resti umani ai fini del recupero e dell'analisi del DNA è intrinsecamente distruttivo 1,2,3,4. I campioni stessi sono esemplari preziosi e la conservazione morfologica dovrebbe essere preservata ove possibile. Pertanto, è imperativo che le pratiche di campionamento siano ottimizzate sia per evitare inutili distruzioni di materiale insostituibile sia per massimizzare la probabilità di successo. Le attuali tecniche di best practice si basano su una piccola coorte di studi limitati a indagini forensi5,6, studi su campioni antichi in cui lo sviluppo di un campionamento ottimale non è l'obiettivo diretto dello studio7, o studi dedicati che utilizzano resti non umani8 o mirati a una selezione molto piccola di posizioni di campionamento anatomico (utilizzate qui per indicare un'area specifica di un elemento scheletrico da cui polvere ossea, per l'uso nelle analisi del DNA a valle, è stato generato)9,10. I protocolli di campionamento qui presentati sono stati ottimizzati nel primo studio sistematico su larga scala della conservazione del DNA su più elementi scheletrici di più individui11. Tutti i campioni provenivano da elementi scheletrici recuperati da 11 individui scavati dal cimitero della chiesa dell'insediamento medievale abbandonato di Krakauer Berg vicino a Peißen, Sassonia-Anhalt, Germania (vedi Tabella 1 per i dati demografici dettagliati del campione) e, come tali, potrebbero richiedere modifiche per l'uso con campioni al di fuori di questo intervallo geografico / temporale.
| Individuo | Sesso | Età stimata alla morte | 14 Date C (CE, Cal 2-sigma) |
| KRA001 | Maschio | 25-35 | 1058-1219 |
| KRA002 | Femmina | 20-22 | 1227-1283 |
| KRA003 | Maschio | 25 | 1059-1223 |
| KRA004 | Maschio | 15 | 1284-1392 |
| KRA005 | Maschio | 10-12 | 1170-1258 |
| KRA006 | Femmina | 30-40 | 1218-1266 |
| KRA007 | Femmina | 25-30 | 1167-1251 |
| KRA008 | Maschio | 20 | 1301-1402 |
| KRA009 | Maschio | Sconosciuto | 1158-1254 |
| KRA010 | Maschio | 25 | 1276-1383 |
| KRA011 | Femmina | 30-45 | 1040-1159 |
Tabella 1: Sesso geneticamente determinato, età stimata archeologicamente determinata alla morte e datazione al radiocarbonio (14C Cal 2-sigma) per tutti gli 11 individui campionati. Questa tabella è stata adattata da Parker, C. et al. 202011.
Questi protocolli consentono una generazione relativamente semplice ed efficiente di polvere ossea da otto posizioni di campionamento anatomico attraverso cinque elementi scheletrici (inclusa la pars petrosa) con una limitata contaminazione del DNA indotta dal laboratorio. Di questi cinque elementi scheletrici, sette posizioni di campionamento anatomico trovate su quattro elementi scheletrici sono state determinate come valide alternative al campionamento distruttivo della piramide petrosa11,12. Questi includono il cemento, la dentina e la camera pulpare dei molari permanenti; osso corticale raccolto dalla tacca vertebrale superiore e dal corpo delle vertebre toraciche; osso corticale derivante dalla superficie inferiore del ciuffo apicale e dall'asta delle falangi distali; e l'osso corticale denso lungo la porzione esterna del tali. Mentre ci sono diversi metodi ampiamente applicati per il campionamento della pars petrosa 4,12,13,14, della dentina e della camera pulpare dentale 1,2,15, i metodi pubblicati descrivono la generazione di successo della polvere ossea dal cemento 16 , corpo vertebrale, tacca vertebrale inferiore e astragalo possono essere difficili da ottenere. Pertanto, qui dimostriamo protocolli di campionamento ottimizzati per la piramide petrosa (passo 3.1); cemento (fase 3.2.1), dentina (fase 3.2.2) e polpa dentale (fase 3.2.3) dei molari adulti; osso corticale del corpo vertebrale (fase 3.3.1) e arco vertebrale superiore (fase 3.3.2); la falange distale (passo 3.4); e l'astragalo (passo 3.5) al fine di rendere più ampiamente accessibile l'uso efficace di questi elementi scheletrici sia per l'aDNA che per la ricerca forense.
Protocollo
Tutte le ricerche qui presentate sono state eseguite in conformità con le linee guida stabilite dal Max Planck Institute for the Science of Human History, Jena, Germania per lavorare con antichi resti umani. Prima di eseguire qualsiasi passaggio di questo protocollo, assicurati di aderire a tutti i requisiti etici locali / statali / federali relativi sia all'ottenimento del permesso per lo studio scientifico che all'uso di resti umani per il campionamento distruttivo nella tua zona. Tutte le procedure/stoccaggio chimico devono essere eseguite secondo le singole linee guida istituzionali di sicurezza.
1. Considerazioni prima dell'elaborazione del campione
- Trattare i campioni con cura poiché i resti antichi sono una risorsa irreplicabile e finita (ad esempio, il campionamento dovrebbe essere il meno dispendioso possibile e tutti i resti restituiti ai rispettivi e legittimi fornitori, se possibile).
- Eseguire tutti i passaggi in un ambiente di camera bianca, preferibilmente in una struttura dedicata al DNA antico17,18,19. Utilizzare dispositivi di protezione individuale (DPI) costituiti da tute sterili microporose con cappuccio, guanti sterili (due paia), maschera chirurgica, occhiali protettivi e stivali sterili o scarpe antiscivolo con coperture sterili (vedere Tabella dei materiali). Cambiare frequentemente i guanti, soprattutto tra un campione e l'altro.
- Pulire e disinfettare accuratamente tutte le apparecchiature e le superfici con candeggina/soluzione di decontaminazione del DNA/etanolo e irradiazione UV (lunghezza d'onda: 254 nm) ove possibile (ad esempio, punte da trapano, trapani, morse/morsetti, ecc.). Infine, si consiglia vivamente di fare pause ergonomiche regolari (ogni 2-3 ore se possibile) per evitare l'eccessivo esaurimento dovuto all'ambiente della camera bianca.
NOTA: Tutti i resti scheletrici devono essere adeguatamente documentati (ad esempio, fotografati, pesati e, se possibile, scansionati con micro-CT, immagini 3D, ecc.) prima del campionamento (i protocolli per la documentazione appropriata non sono trattati in questo manoscritto). Tutti i protocolli di campionamento possono essere sospesi tra le iterazioni di campionamento e i campioni possono essere conservati indefinitamente in un ambiente asciutto, a temperatura controllata (25 °C), sterile.
2. Pretrattamento
- Decontaminare tutte le posizioni di campionamento anatomico prima della generazione di polvere ossea per ridurre al minimo il rischio di contaminazione18.
NOTA: L'efficacia della candeggina e/o della rimozione superficiale (vedere NOTA al punto 3.3.2 per le fasi di rimozione delle superfici) per la decontaminazione del campione è ancora oggetto di dibattito tra i ricercatori di aDNA 8,19,20,21,22,23,24,25 poiché entrambi possono influenzare le rese complessive del DNA, specialmente in campioni altamente degradati. Pertanto, i seguenti passaggi sono considerati facoltativi e sono inclusi qui poiché sono stati utilizzati in tutti gli esempi per generare i risultati rappresentativi presentati in questo documento. Si raccomanda di determinare caso per caso l'uso di questi protocolli di pretrattamento in base all'applicazione molecolare, all'età, alla rarità e al livello di degradazione morfologica di ciascun set di campioni.- Eseguire tutti i campionamenti in una camera bianca dedicata sotto una cappa di reazione a catena della polimerasi (PCR) dotata di luce UV o un armadio di biosicurezza con flusso d'aria disattivato. Stendere un foglio di alluminio sterile sul piano di lavoro per catturare eventuali polvere / frammenti di ossa vaganti.
- Assicurarsi che tutti i frammenti ossei siano recuperati (per il rimpatrio) prima di smaltire il foglio. Cambia la pellicola tra il trattamento di ciascun elemento scheletrico. Smaltire la pellicola usata in una borsa/recipiente autoclavabile a rischio biologico.
- Rimuovere quanto più sporco/detrito sciolto possibile dalle posizioni di campionamento anatomico strofinando delicatamente l'area con una salvietta sterile asciutta priva di lanugine (vedere Tabella dei materiali). Smaltire le salviette in sacchetti o recipienti autoclavabili a rischio biologico.
- Decontaminare la superficie pulita strofinando con una salvietta sterile inumidita con candeggina commerciale diluita (~0,01% v/v, diluita con acqua ultrapura priva di DNasi/RNasi) e lasciare incubare per 5 minuti. Smaltire le salviette in sacchetti o recipienti autoclavabili a rischio biologico.
ATTENZIONE: La candeggina è una sostanza chimica altamente corrosiva e reattiva; Pertanto devono essere prese adeguate precauzioni di sicurezza prima del suo uso. - Rimuovere quanta più candeggina residua possibile dalla posizione di campionamento anatomico con una salvietta sterile inumidita con acqua ultrapura priva di DNasi/RNasi. Smaltire le salviette in sacchetti o recipienti autoclavabili a rischio biologico.
- Esporre tutte le posizioni di campionamento anatomico pulite alle radiazioni UV per 30 minuti (lunghezza d'onda: 254 nm), quindi lasciare asciugare completamente a temperatura ambiente. Assicurarsi che le posizioni di campionamento anatomico siano completamente asciutte prima di procedere con il campionamento o tornare allo stoccaggio non solo per facilitare la generazione di polvere ossea, ma anche per prevenire un'ulteriore degradazione del campione (ad esempio, muffa).
ATTENZIONE: L'esposizione alle radiazioni UV può essere dannosa per gli occhi. - Passare immediatamente al campionamento o conservare gli elementi scheletrici in un ambiente sterile asciutto e a temperatura controllata (25 °C).
3. Generazione di polvere ossea
NOTA: I seguenti protocolli sono destinati all'uso nell'estrazione del DNA seguendo il protocollo26 di Dabney et al. 2019.
- Campionamento di pars petrosa
NOTA: Questo protocollo è adattato dalle procedure descritte in Pinhasi et al. 20194 ed è presentato qui per facilità d'uso. Questo protocollo non rappresenta l'attuale metodo meno distruttivo per il campionamento di pars petrosa. Pertanto, si raccomanda di utilizzare il protocollo descritto da Sirak et al. 201713 o Orfanou et al. 202014 per i campioni in cui la conservazione morfologica è della massima importanza.- Eseguire tutti i campionamenti in una camera bianca dedicata sotto una cappa PCR dotata di luce UV o un armadio di biosicurezza (lunghezza d'onda: 254 nm) con il flusso d'aria disattivato. Stendere un foglio di alluminio sterile sul piano di lavoro per catturare eventuali polvere / frammenti di ossa vaganti.
- Assicurarsi che tutti i frammenti ossei e quanta più polvere possibile siano recuperati (per il rimpatrio) prima di smaltire il foglio. Cambiare la pellicola tra un campionamento e l'altro. Smaltire la pellicola usata in una borsa/recipiente autoclavabile a rischio biologico.
- Fissare l'elemento asciutto e decontaminato utilizzando un morsetto o una morsa sterilizzata.
- Tagliare la pars petrosa a metà lungo il solco petroso superiore (vedi figura 1) usando una sega da gioielliere standard dotata di una lama da 0,6 mm (vedi tabella dei materiali) a velocità media per evitare il surriscaldamento (vedi NOTA sotto il punto 3.1.6).
ATTENZIONE: La pars petrosa è molto densa, e come tale può essere difficile da tagliare. Fare attenzione a mantenere l'elemento saldamente bloccato per evitare lesioni. Smaltire le lame rotte nell'apposito recipiente per oggetti taglienti. - Rimuovere le parti petrose dal morsetto. Recuperare e salvare qualsiasi materiale sciolto / in eccesso.
- Posizionare la carta pesata in un contenitore sterile
- Tenere la parte petrosa sulla carta da pesare, con il lato tagliato inclinato verso il vassoio. Forare l'osso corticale denso tra il canale facciale e l'antro mastoideo (appare più lucido del materiale circostante, vedi Figura 1) usando un trapano dentale dotato di una piccola punta di misura (vedi Tabella dei materiali) e impostato su velocità media, coppia media per produrre polvere ossea.
NOTA: La perforazione / taglio deve essere eseguita in brevi raffiche a basse e medie velocità per evitare il surriscaldamento dell'osso e potenzialmente distruggere / danneggiare il DNA. Aneddoticamente, quando la parte densa del petroso inizia a surriscaldarsi, si può osservare un odore descritto come pancetta da cucina. Cessare immediatamente la perforazione/segatura e lasciare riposare l'osso fino a quando non si raffredda sufficientemente prima di riprendere. - Ripetere la perforazione fino a quando circa 50-100 mg di polvere vengono raccolti nella carta pesata, misurati utilizzando una bilancia chiusa con una precisione di almeno 0,01 mg (vedere Tabella dei materiali).
NOTA: Ove possibile, si consiglia di raccogliere 100 mg di polvere ossea per consentire l'estrazione di due repliche del DNA di 50 mg ciascuna. Tuttavia, ciò potrebbe non essere sempre possibile in base alla limitazione delle posizioni di campionamento anatomico stesse (ad esempio, la falange distale, la camera pulpare dentale) o alla necessità di conservazione morfologica. Per altre località, come il cemento, possono essere disponibili molto meno di 50 mg di materiale. Tuttavia, il cemento, la camera pulpare dentale e la falange distale hanno tutti dimostrato di produrre DNA endogeno significativo 11,27,28, nonostante un minore apporto iniziale di polvere ossea dal processo di estrazione. - Trasferire la polvere dalla carta pesata a un tubo di sicurezza etichettato con 2 ml a bassa legatura per l'estrazione o lo stoccaggio. Conservare i campioni a -20 °C, a tempo indeterminato.
- Conservare l'osso residuo/polvere in eccesso in un ambiente sterile asciutto e a temperatura controllata (25 °C) fino al completamento del rimpatrio/rimpatrio.
- Smaltire tutti i rifiuti in sacchetti o recipienti autoclavabili a rischio biologico. Sterilizzare/decontaminare tutte le attrezzature riutilizzabili (ad esempio, morsetti, punte da trapano, trapani, seghe, ecc.) utilizzando candeggina/soluzione di decontaminazione del DNA/etanolo ed esposizione UV (lunghezza d'onda: 254 nm), a seconda dei casi, tra ciascun campionamento.
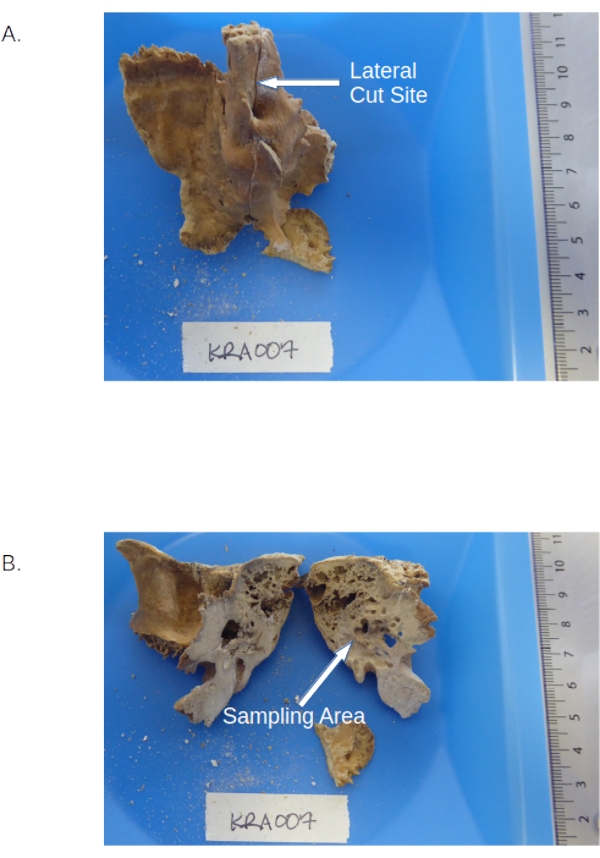
Figura 1: Osso temporale inclusa la pars petrosa. (A) Pretaglio del campione che mostra l'ubicazione della piramide petrosa e del solco petrosa. (B) Porzione petrosa post-taglio evidenziando le aree dense da perforare. Fare clic qui per visualizzare una versione ingrandita di questa figura.
- Campionamento dei molari permanenti
NOTA: Per il campionamento dei molari permanenti, preselezionare in situ Molari con radici fuse e idealmente privi di carie, crepe nello smalto o usura eccessiva per ottenere i migliori risultati. Rimuovere qualsiasi campionamento di calcoli dentali e conservare a -20 °C per eventuali analisi future del microbioma orale (procedura non trattata qui).- Campionamento del cemento
- Eseguire tutti i campionamenti in una camera bianca dedicata sotto una cappa PCR dotata di luce UV o un armadio di biosicurezza (lunghezza d'onda: 254 nm) con il flusso d'aria disattivato. Stendere un foglio di alluminio sterile sul piano di lavoro per catturare eventuali polvere / frammenti di ossa vaganti.
- Assicurarsi che tutti i frammenti ossei e quanta più polvere possibile siano recuperati (per il rimpatrio) prima di smaltire la pellicola. Cambiare la pellicola tra un campionamento e l'altro. Smaltire la pellicola usata in una borsa/recipiente autoclavabile a rischio biologico.
- Introdurre un foglio di carta da pesare in una teglia sterile.
- Tenere/fissare il molare decontaminato per lo smalto, radicare verso il basso, sopra un vassoio di pesata usando un morsetto portatile come una chiave regolabile (vedi Tabella dei materiali).
- Equipaggiare un trapano dentale con una ruota da taglio circolare diamantata. Con il trapano impostato su un'impostazione di velocità/coppia media, toccare leggermente il bordo della punta fino alla radice con un angolo di circa -20°.
- Raschiare verso il basso nel vassoio per rimuovere / raccogliere il materiale giallo più esterno dalla radice (cemento). Interrompere la raccolta quando il materiale più leggero (bianco) della dentina diventa visibile.
NOTA: È importante abbinare la direzione di rotazione della punta di taglio rispetto al vassoio di raccolta per evitare che la polvere si aerosolizzi e potenzialmente sprechi il campione mancando completamente il vassoio. Il cemento è particolarmente ricco di DNA; Tuttavia, le rese tipiche del materiale sono molto più piccole rispetto ad altre posizioni di campionamento anatomico (~7-20 mg)11,27,28. - Registrare la massa di polvere raccolta in carta pesata utilizzando una bilancia allegata con precisione di almeno 0,01 mg (vedere la tabella dei materiali).
- Trasferire la polvere dalla carta pesata a un tubo di bloccaggio sicuro da 2 mL per l'estrazione. Conservare a -20 °C, per un periodo indefinito.
- Campionamento della camera pulpare
- Dopo che il cemento è stato raccolto (se lo si desidera), sezionare il molare lungo la giunzione cemento-smalto usando una sega da gioielliere per rimuovere la corona (vedi Figura 2).
- Inserire un nuovo foglio di carta pesata in una nuova vaschetta di pesatura.
- Fissare la sezione della corona in un morsetto o morsa portatile, sopra il vassoio di pesatura. Tenere il lato tagliato inclinato verso il basso e perforare/raschiare il materiale come prima passata con un trapano dentale dotato di una punta di perforazione di piccolo spessore (vedere la tabella dei materiali) lungo i bordi della camera di polpa all'interno della porzione di corona (vedere Figura 2).
NOTA: Solo il primo passaggio dell'interno della camera pulpare deve essere raccolto ed etichettato come materiale pulpare (resa tipica 5-15 mg), qualsiasi cosa più profonda nel dente è considerata dentina. - Girare il dente con la parte inferiore rivolta verso il basso, picchiettare il morsetto con un martello e raccogliere la polvere liberata sulla carta da pesare.
- Registrare il peso della polvere raccolta nella carta pesata utilizzando una bilancia allegata accurata ad almeno 0,01 mg (vedere Tabella dei materiali).
- Trasferire la polvere dalla carta pesata a un tubo di sicurezza a bassa legatura da 2 ml per l'estrazione. Conservare a -20 °C, per un periodo indefinito.
- Campionamento della dentina
- Inserire un nuovo foglio di carta pesata in una nuova vaschetta di pesatura.
- Tenere la sezione della corona sopra il vassoio di pesata (come da punto 3.2.2.3), forare e raccogliere altri 50-100 mg di dentina misurati utilizzando una bilancia chiusa accurata a 0,01 mg (vedere la tabella dei materiali) dall'interno della camera pulpare nello stesso modo per un ulteriore campionamento della dentina (vedere figura 2).
- Trasferire la polvere ossea dalla carta pesata a un tubo di sicurezza a basso legame da 2 ml per l'estrazione. Conservare a -20 °C, per un periodo indefinito.
- Conservare i restanti pezzi di denti/polvere in eccesso in un ambiente asciutto e a temperatura controllata (25 °C) sterile fino al completamento del rimpatrio/rimpatrio.
- Smaltire tutti i rifiuti in sacchetti o recipienti autoclavabili a rischio biologico. Sterilizzare/decontaminare tutte le attrezzature riutilizzabili (ad esempio, morsetti, punte da trapano, trapani, seghe, ecc.) utilizzando candeggina/soluzione di decontaminazione del DNA/etanolo ed esposizioni UV (lunghezza d'onda: 254 nm), a seconda dei casi, tra ogni campionamento.
- Campionamento del cemento
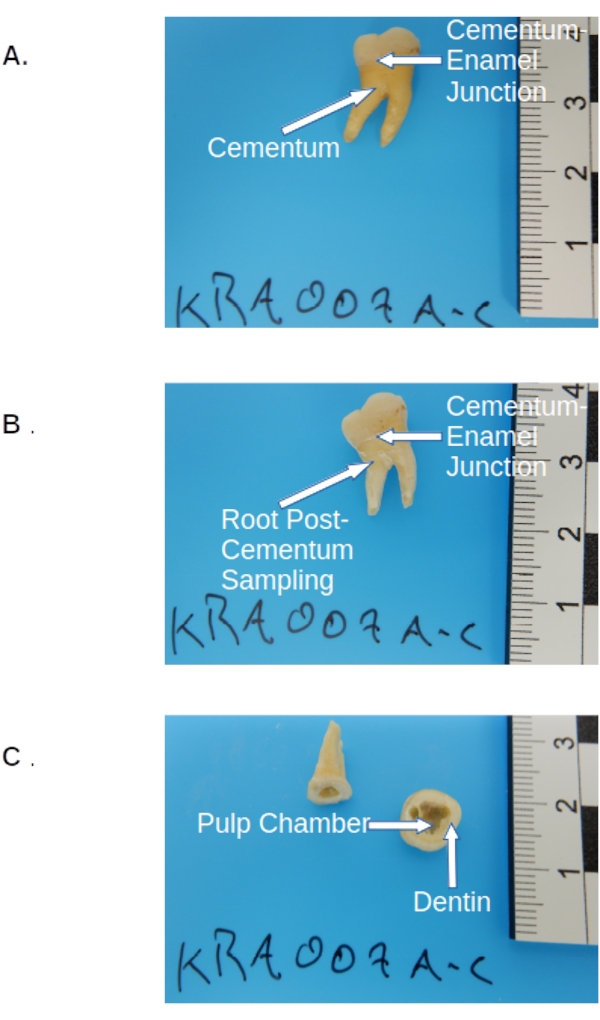
Figura 2: Precampionamento molare permanente . (A) Molare pretrattato prima del campionamento, con indicazione della corona, del cemento (strato giallastro della radice) e del sito di taglio alla giunzione cemento-smalto. (B) La stessa collezione molare post-cemento, che mostra il sito di taglio alla giunzione cemento-smalto. (C) Post-taglio e campionamento molare che mostra le posizioni anatomiche di campionamento per la camera pulpare dentale e la dentina all'interno della corona. Fare clic qui per visualizzare una versione ingrandita di questa figura.
- Campionamento delle vertebre toraciche
- Campionamento del corpo vertebrale
- Eseguire tutti i campionamenti in una camera bianca dedicata sotto una cappa PCR dotata di luce UV o un armadio di biosicurezza (lunghezza d'onda: 254 nm) con il flusso d'aria disattivato. Stendere un foglio di alluminio sterile sul piano di lavoro per catturare eventuali polvere / frammenti di ossa vaganti.
- Assicurarsi che tutti i frammenti ossei e quanta più polvere possibile siano recuperati (per il rimpatrio) prima di smaltire la pellicola. Cambiare la pellicola tra un campionamento e l'altro. Smaltire la pellicola usata in una borsa/recipiente autoclavabile a rischio biologico.
- Inserire un piccolo foglio di carta pesata in una vaschetta di pesata standard.
- Fissare le vertebre con un morsetto o una morsa a mano, con il corpo vertebrale verso l'esterno.
- Tenere le vertebre sopra il vassoio di pesata con il corpo vertebrale inclinato verso il basso. Utilizzando un trapano dentale dotato di una punta di perforazione di piccolo calibro (vedi Tabella dei materiali) impostata su una coppia elevata a bassa velocità, forare lungo il bordo più esterno (inferiore e superiore) dell'osso corticale che circonda il tessuto interno spugnoso del corpo vertebrale (vedere Figura 3).
- Raschiare la punta contro lo strato corticale su un vassoio di pesatura standard fino a raccogliere 50-100 mg di materiale, misurati utilizzando una bilancia chiusa accurata a 0,01 mg (vedere Tabella dei materiali).
- Trasferire la polvere ossea dalla carta pesata a un tubo di bloccaggio sicuro da 2 mL per l'estrazione. Conservare a -20 °C, per un periodo indefinito.
- Campionamento dell'arco vertebrale superiore
Nota : questo passaggio è facoltativo. Rimuovere e scartare lo strato più esterno dell'osso corticale dell'arcata vertebrale superiore utilizzando un trapano dentale dotato di una punta di perforazione di piccolo calibro (vedi Tabella dei materiali) raschiandolo lungo la superficie19. Ciò non è raccomandato per il campionamento dal corpo vertebrale, poiché lo strato di osso corticale è generalmente molto sottile e probabilmente sarà completamente impoverito da questo processo (vedere NOTA nella sezione 2).- Inserire un piccolo foglio di carta pesata in una vaschetta di pesata standard.
- Fissare le vertebre in un morsetto / morsa della mano con il processo vertebrale verso l'esterno, aspetto superiore verso il basso.
- Tenendo le vertebre, aspetto superiore verso il basso, sopra un vassoio di pesatura, forare verso l'alto il centro della tacca a forma di V formata dalla fusione del processo spinoso con le lamelle (vedi Figura 3) utilizzando un trapano dentale con una piccola punta di calibro (vedi Tabella dei materiali) impostata a bassa velocità e coppia elevata.
- Cessare la perforazione quando c'è un notevole calo della resistenza. Cambiare leggermente la posizione di foratura e ripetere fino a raccogliere 50-100 mg di polvere ossea, misurata utilizzando una bilancia chiusa accurata a 0,01 mg (vedere la tabella dei materiali).
- Trasferire la polvere ossea dalla carta pesata a un tubo a basso legame da 2 ml per l'estrazione. Conservare a -20 °C, per un periodo indefinito.
- Conservare l'osso residuo/polvere in eccesso in un ambiente sterile asciutto e a temperatura controllata (25 °C) fino al ritorno/rimpatrio.
- Smaltire tutti i rifiuti in sacchetti o recipienti autoclavabili a rischio biologico. Sterilizzare/decontaminare tutte le attrezzature riutilizzabili (ad esempio, morsetti, punte da trapano, trapani, seghe, ecc.) utilizzando candeggina/soluzione di decontaminazione del DNA/etanolo ed esposizione UV (lunghezza d'onda: 254 nm), a seconda dei casi, tra ciascun campionamento.
- Campionamento del corpo vertebrale

Figura 3: Corpo vertebrale e arco vertebrale superiore posizioni di campionamento anatomico dell'osso corticale della vertebra toracica. Fare clic qui per visualizzare una versione ingrandita di questa figura.
- Campionamento della falange distale
Nota : questo passaggio è facoltativo. Rimuovere e scartare lo strato più esterno dell'osso corticale dell'albero e/o del ciuffo apicale utilizzando un trapano dentale dotato di una punta di perforazione di piccolo calibro raschiandolo lungo la superficie19. Ciò potrebbe non essere possibile per campioni con osso corticale eccessivamente sottile o resti giovanili (vedere NOTA nel paragrafo 2).- Eseguire tutti i campionamenti in una camera bianca dedicata, sotto una cappa PCR dotata di luce UV o un armadio di biosicurezza (lunghezza d'onda UV: 254 nm) con flusso d'aria disattivato. Stendere un foglio di alluminio sterile sul piano di lavoro per catturare eventuali polvere / frammenti di ossa vaganti.
- Assicurarsi che tutti i frammenti ossei e quanta più polvere possibile siano recuperati (per il rimpatrio) prima di smaltire la pellicola. Cambiare la pellicola tra un campionamento e l'altro. Smaltire la pellicola usata in una borsa/recipiente autoclavabile a rischio biologico.
- Inserire un piccolo foglio di carta pesata in una vaschetta di pesata standard.
- Fissare il campione in morsetto/morsa portatile, lato superiore verso l'alto.
- Tenere il campione sopra il vassoio di pesatura, raccogliere la polvere ossea dall'osso corticale dal lato inferiore del ciuffo apicale e dell'albero perforando gli strati più densi più esterni (vedere figura 4) utilizzando un trapano dentale dotato di una punta di perforazione di piccolo calibro (vedere la tabella dei materiali).
- Cessare la perforazione quando c'è una marcata diminuzione della resistenza, poiché ciò significa materiale più leggero e spugnoso. Ripetere questo processo, irradiando verso l'esterno dalla perforazione iniziale fino a raccogliere almeno 50-100 mg di polvere ossea, misurata utilizzando una bilancia chiusa accurata a 0,01 mg (vedere Tabella dei materiali).
- Trasferire la polvere ossea dalla carta pesata a un tubo di sicurezza a basso legame da 2 ml per l'estrazione. Conservare a -20 °C, per un periodo indefinito.
- Conservare l'osso rimanente/polvere in eccesso in un ambiente sterile asciutto e a temperatura controllata (25 °C) fino al ritorno/rimpatrio.
- Smaltire tutti i rifiuti in sacchetti o recipienti autoclavabili a rischio biologico. Sterilizzare/decontaminare tutte le attrezzature riutilizzabili (ad esempio, morsetti, punte da trapano, trapani, seghe, ecc.) utilizzando candeggina/soluzione di decontaminazione del DNA/etanolo ed esposizione ai raggi UV, a seconda dei casi, tra un campionamento e l'altro.
NOTA: Per campioni più piccoli (ad esempio, campioni giovanili) ci può essere considerevolmente meno dei 50-100 mg di osso corticale suggeriti disponibili per il campionamento. Tuttavia, anche in piccole quantità, questa posizione di campionamento anatomico ha dimostrato di essere particolarmente ricca di DNA11.

Figura 4: Falange distale che mostra le posizioni dell'osso corticale denso lungo l'asta e il lato inferiore del ciuffo apicale. Fare clic qui per visualizzare una versione ingrandita di questa figura.
- Campionamento dell'astragalo
- Eseguire tutti i campionamenti in una camera bianca dedicata sotto una cappa PCR dotata di luce UV o un armadio di biosicurezza (lunghezza d'onda: 254 nm) con il flusso d'aria disattivato. Stendere un foglio di alluminio sterile sul piano di lavoro per catturare eventuali polvere / frammenti di ossa vaganti.
- Assicurarsi che tutti i frammenti ossei e quanta più polvere possibile siano recuperati (per il rimpatrio) prima di smaltire la pellicola. Cambiare la pellicola tra un campionamento e l'altro. Smaltire la pellicola usata in una borsa/recipiente autoclavabile a rischio biologico.
- Inserire un piccolo foglio di carta pesata in una vaschetta di pesata standard.
- Fissare il campione in morsetto/morsa portatile, cupola verso l'alto.
- Tenere l'astragalo, la cupola verso l'alto e la superficie mediale verso il collettore, sopra il vassoio di pesatura. Raschiare l'osso corticale dal collo dell'astragalo ad una profondità di ~ 1 mm (vedi Figura 5) usando un trapano dentale con una punta a basso spessore (vedi Tabella dei materiali) impostata su bassa velocità e coppia elevata.
- Cambiare leggermente la posizione di perforazione e ripetere fino a raccogliere circa 50-100 mg di polvere ossea, misurata utilizzando una bilancia chiusa con precisione di 0,01 mg (vedere la tabella dei materiali).
- Trasferire la polvere ossea dalla carta pesata a un tubo a basso legame da 2 ml per l'estrazione. Conservare a -20 °C, per un periodo indefinito.
- Conservare l'osso rimanente/polvere in eccesso in un ambiente sterile asciutto e a temperatura controllata (25 °C) fino al completamento del rimpatrio/rimpatrio.
- Smaltire tutti i rifiuti in sacchetti o recipienti autoclavabili a rischio biologico. Sterilizzare/decontaminare tutte le attrezzature riutilizzabili (ad esempio, morsetti, punte da trapano, trapani, seghe, ecc.) utilizzando candeggina/soluzione di decontaminazione del DNA/etanolo ed esposizione UV (lunghezza d'onda: 254 nm), a seconda dei casi, tra ciascun campionamento.
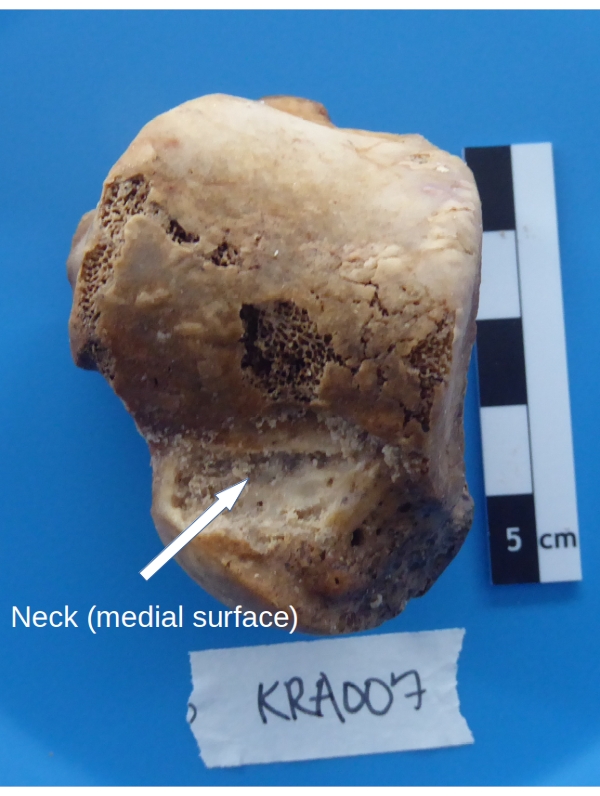
Figura 5: Area di campionamento dell'astragalo per il recupero dell'osso corticale. Fare clic qui per visualizzare una versione ingrandita di questa figura.
NOTA: L'astragalo ha pochissimo osso corticale (uno strato esterno sottile). Il materiale non dovrebbe essere raccolto solo dalla superficie, ma anche dallo strato denso sottostante di osso spugnoso.
Risultati
In uno studio separato 11, il DNA è stato estratto dalla polvere ossea generata da ciascuna posizione di campionamento anatomico in11 individui, utilizzando un protocollo di estrazione del DNA standard ottimizzato per brevi frammenti di tessuto calcificato2. Le librerie a singolo filamento sono state quindi prodotte28 e sequenziate su un HiSeq 4000 (75 bp paired-end) ad una profondità di ~ 20.000.000 di letture per campione. I dati di sequenza risultanti sono stati quindi valutati per il contenuto di DNA umano endogeno utilizzando la pipeline EAGER29 (impostazioni BWA: lunghezza del seme di 32, penalità di mancata corrispondenza di 0,1, filtro di qualità della mappatura di 37). Tutti i risultati rappresentativi sono riportati utilizzando le stesse metriche di Parker et al. 202011 per coerenza. Le librerie delle porzioni in polvere della pars petrosa hanno prodotto, in media, DNA endogeno superiore rispetto a qualsiasi altra delle altre 23 posizioni di campionamento anatomico esaminate (Figura 6A-B). Le sette ulteriori sedi di campionamento anatomico presentate in questo protocollo (il cemento, il primo passaggio della camera pulpare dentale e la dentina dei molari permanenti; l'osso corticale dal corpo vertebrale e l'arco vertebrale superiore della vertebra toracica; l'osso corticale dal ciuffo apicale della falange distale; e l'osso corticale dal collo dell'astragalo) hanno prodotto le rese successive più elevate (senza alcuna significatività statistica tra queste posizioni di campionamento anatomico; Figura 6A-B; File supplementare 1: DNAPreCap endogeno). Queste posizioni alternative producono DNA in modo coerente adeguate per le analisi standard di genetica delle popolazioni, come le analisi mitocondriali e le analisi del polimorfismo a singolo nucleotide (SNP). I tassi di duplicazione nelle librerie derivanti da tutte le posizioni di campionamento anatomico erano bassi (fattori di cluster < 1,2 in media, calcolati come rapporto tra tutte le letture di mappatura e letture di mappatura univoche, Tabella 2; Supplemental File 1: ClusterFactor), indicando che tutte le librerie sottoposte a screening erano di complessità molto elevata. Allo stesso modo, le stime medie di contaminazione del DNA umano esogeno erano basse, con una media < 2% (contaminazione del cromosoma X nei maschi, n = 7, come riportato dalla pipeline ANGSD30) in tutte le sedi di campionamento anatomico ad eccezione dell'arco vertebrale superiore (contaminazione media stimata: 2,11%, con un campione rimosso come anomalia; KRA005: 19,52%, cfr. tabella 2; File supplementare 1: Xcontamination). La lunghezza media del frammento (dopo il filtraggio per rimuovere tutte le letture < 30 bp) era più bassa nel materiale raccolto dalla camera pulpare dentale e dalla dentina, senza variazioni significative tra le altre posizioni di campionamento anatomico (55,14 bp e 60,22 bp, rispettivamente rispetto a una mediana media di 62,87, valori p a coppie < 0,019, Tabella 2; File supplementare 1: AvgFragLength). Inoltre, i denti e le vertebre toraciche contengono ciascuno più punti di campionamento anatomico in cui è stato osservato un elevato recupero endogeno del DNA, rendendoli particolarmente adatti come alternative alla pars petrosa.
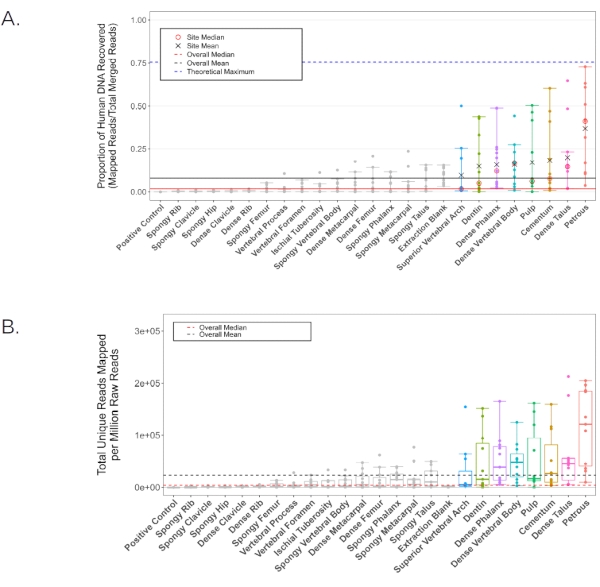
Figura 6: Contenuto di DNA umano per tutti i campioni esaminati. Le linee nere rappresentano la media complessiva, mentre le linee rosse rappresentano la mediana (solido: proporzione del DNA umano, tratteggiato: letture umane mappate per milione di letture generate). Le singole posizioni di campionamento anatomico con una proporzione media di DNA umano superiore alla media complessiva (8,16%) sono colorate in tutte le analisi. (A) La proporzione di letture mappate al genoma di riferimento hg19. La linea tratteggiata blu rappresenta il massimo teorico dati i parametri di mappatura della pipeline (generati utilizzando Gargammel31 per simulare una distribuzione casuale di 5.000.000 di letture dal genoma di riferimento hg19 con danno simulato). Le medie individuali (X nera) e le mediane (cerchio rosso) sono riportate per quei campioni con una proporzione media di DNA umano più elevata rispetto alla media complessiva. Gli intervalli di confidenza indicano i limiti superiore e inferiore escludendo i valori anomali statistici. (B) Il numero di letture uniche mappate al genoma di riferimento hg19 per milione di letture dello sforzo di sequenziamento (75 bp accoppiati end). Gli intervalli di confidenza indicano i limiti superiore e inferiore escludendo i valori anomali statistici. Questa figura è stata adattata da Parker, C. et al. 202011. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Tabella 2: Livelli medi di duplicazione (letture di mappatura/letture univoche), lunghezze medie e mediane dei frammenti e stime della contaminazione del cromosoma X per tutte le posizioni di campionamento anatomico. Errore segnalato come errore standard della media. Questa tabella è stata adattata da Parker, C. et al. 202011.
| Luogo di campionamento | Fattore di duplicazione medio (# letture mappate /# letture mappate univoche) | Lunghezza media del frammento in bp | Percentuale media stimata di contaminazione del cromosoma X |
| Piramide petrosa | 1,188 ± 0,006 | 65,40 ± 1,36 | 0,000 ± 0,003 |
| Cementum | 1,197 ± 0,028 | 67,28 ± 1,76 | 0,011 ± 0,003 |
| Dentina | 1,188 ± 0,061 | 60,22 ± 2,37 | 0,002 ± 0,007 |
| Polpa | 1,179 ± 0,024 | 55,14 ± 2,90 | 0,013 ± 0,006 |
| Falange distale | 1,191 ± 0,049 | 65,95 ± 1,08 | 0,013 ± 0,005 |
| Corpo vertebrale | 1,194 ± 0,037 | 66,14 ± 1,03 | 0,008 ± 0,003 |
| Arco vertebrale superiore | 1,19 ± 0,017 | 63,02 ± 1,23 | 0,021 ± 0,009* |
| Astragalo | 1,198 ± 0,010 | 68,20 ± 1,24 | 0,011 ± 0,003 |
| *Campione KRA005 rimosso come valore anomalo a 0,1952 |
Disponibilità del codice
Tutti i programmi di analisi e i moduli R utilizzati nelle analisi di questo manoscritto sono liberamente disponibili presso i rispettivi autori. Tutti i codici R personalizzati sono disponibili su richiesta.
Disponibilità dei dati
Tutti i dati grezzi utilizzati nel calcolo dei risultati rappresentativi sono liberamente disponibili nell'archivio europeo dei nucleotidi ENA (numero di adesione PRJ-EB36983) o nei materiali supplementari di Parker, C. et al.11.
File supplementare 1. Clicca qui per scaricare questo file.
Discussione
La pratica corrente nell'antica genetica delle popolazioni umane è quella di campionare preferenzialmente dalla pars petrosa (passo 2.1) quando possibile. Tuttavia, la pars petrosa può essere un campione difficile da ottenere, in quanto è molto apprezzata per una miriade di valutazioni scheletriche (ad esempio, la storia della popolazione32, la stima dell'età fetale alla morte 33 e la determinazione del sesso34), e, storicamente, il campionamento della pars petrosa per l'analisi del DNA può essere altamente distruttivo3,4 (incluso il protocollo qui presentato, anche se nuovi protocolli minimamente invasivi13,14 sono stati ora ampiamente adottati per alleviare questa preoccupazione). Ciò è aggravato dal fatto che, fino a poco tempo fa, uno studio sistematico su larga scala del recupero del DNA umano attraverso lo scheletro non era stato tentato11, rendendo difficile trovare una strategia di campionamento appropriata quando la piramide petrosa non è disponibile.
I protocolli presentati qui aiutano ad alleviare questa sfida fornendo una serie di procedure ottimizzate per il campionamento del DNA da resti scheletrici archeologici / forensi tra cui la pars petrosa e sette posizioni di campionamento anatomico alternative attraverso quattro elementi scheletrici aggiuntivi. Le fasi critiche incluse sono tutte intese a ridurre al minimo la possibilità di perdita/danno del DNA a causa di un campionamento inefficiente (fasi 2.1.6 e 3.2.1.3) o del surriscaldamento dei campioni durante la perforazione/taglio (fase 3.1.6). Inoltre, è stato notato in tutto il protocollo che potrebbe essere necessario modificare/omettere le fasi di pre-trattamento per garantire le migliori prestazioni in campioni altamente degradati. Va anche notato che anche tra gli elementi selezionati qui presentati, rimangono diverse possibili tecniche di campionamento alternative (in particolare per la pars petrosa13,14), nonché ampio spazio per un'ulteriore ottimizzazione delle posizioni di campionamento anatomico sottosfruttate qui presentate (ad esempio, l'astragalo: fase 2.5 e le vertebre: fase 2.3).
È anche importante tenere presente che questi protocolli sono stati progettati e testati utilizzando antichi resti giovani-adulti di alta qualità (buona conservazione morfologica) ai fini dell'analisi endogena del DNA umano. I risultati presentati potrebbero non estendersi a materiali più altamente degradati, altri contesti di conservazione, resti infantili, resti non umani o studi di agenti patogeni o del microbioma, poiché è ancora necessaria una maggiore esplorazione dell'uso di questi protocolli in ulteriori contesti. Inoltre, gli elementi scheletrici alternativi qui presentati (i denti, le vertebre, la falange distale e i tali) possono essere difficili da assegnare a un singolo individuo tra i resti mescolati, richiedendo il campionamento da più elementi per garantire una singola origine. Nonostante queste limitazioni, rendere questi protocolli ampiamente disponibili può aiutare ad alleviare parte dell'eterogeneità che circonda la selezione e l'elaborazione dei campioni fornendo un quadro generalizzato e quantitativamente ottimizzato per l'uso in una vasta gamma di futuri studi aDNA / forensi su resti umani.
Divulgazioni
Gli autori non hanno conflitti di interesse da segnalare.
Riconoscimenti
Gli autori desiderano ringraziare il personale di laboratorio dell'Istituto Max Planck per la scienza della storia umana per il loro aiuto nello sviluppo e nell'attuazione di questi protocolli. Questo lavoro non sarebbe stato possibile senza il contributo e il duro lavoro del Dr. Guido Brandt, Dr. Elizabeth Nelson, Antje Wissegot e Franziska Aron. Questo studio è stato finanziato dalla Max Planck Society, dal Consiglio europeo della ricerca (CER) nell'ambito del programma di ricerca e innovazione Horizon 2020 dell'Unione europea nell'ambito degli accordi di sovvenzione n. 771234 - PALEoRIDER (WH, ABR) e Starting Grant No. 805268 CoDisEASe (to KIB).
Materiali
| Name | Company | Catalog Number | Comments |
| #16 Dental Drill Bit | NTI | H1-016-HP | example drilling bit |
| 0.6 mm scroll saw blade | Fisher Scientific | 50-949-097 | blade for Jewellers Saw |
| 22mm diamond cutting wheel | Kahla | SKU 806 104 358 514 220 | Dremel cutting attachment |
| Commercial Bleach | Fisher Scientific | NC1818018 | |
| Control Company Ultra-Clean Supreme Aluminum Foil | Fisher Scientific | 15-078-29X | |
| DNA LoBind Tubes (2 mL) | Eppendorf | 22431048 | |
| Dremel 225-01 Flex Shaft Attachment | Dremel | 225-01 | Dremel flexible extension |
| Dremel 4300 Rotary Tool | Dremel | 4300 | Example drill |
| Dremel collet and nut kit | Dremel | 4485 | Adapters for various Dremel tool attachments/bits |
| Eagle 33 Gallon Red Biohazard Waste Bag | Fisher Scientific | 17-988-501 | |
| Eppendorf DNA LoBind 2 mL microcentrifuge tube | Fisher Scientific | 13-698-792 | |
| Ethanol (Molecular Biology Grade) | Millipore Sigma | 1.08543 | |
| FDA approved level 2 Surgical Mask | Fisher Scientific | 50-206-0397 | PPE |
| Fisherbrand Comfort Nitrile Gloves | Fisher Scientific | 19-041-171X | PPE |
| Fisherbrand Safety Glasses | Fisher Scientific | 19-130-208X | PPE |
| Granger Stationary Vise | Fisher Scientific | NC1336173 | benchtop vise |
| Invitrogen UltraPure DNase/Rnase free distilled water | Fisher Scientific | 10-977-023 | |
| Jewellers Saw | Fisher Scientific | 50-949-231 | |
| Kimwipes | Sigma-Aldritch | Z188956 | |
| Labconco Purifier Logic Biosafety cabinet | Fisher Scientific | 30-368-1101 | |
| LookOut DNA Erase | Millipore Sigma | L9042-1L | |
| Medium weighing boat | Heathrow Scientific | HS120223 | |
| MSC 10pc plier/clamp set | Fisher Scientific | 50-129-5352 | Miscellaneous clamps/vise grips for securely holding samples while drilling/cutting |
| Sartorius Quintix Semi-Micro Balance | Fisher Scientific | 14-560-019 | enclosed balance |
| Tyvek coveralls with hood | Fisher Scientific | 01-361-7X | PPE |
| Weigh paper | Heathrow Scientific | HS120116 |
Riferimenti
- Adler, C. J., Haak, W., Donlon, D., Cooper, A. Survival and recovery of DNA from ancient teeth and bones. Journal of Archaeological Science. 38 (5), 956-964 (2011).
- Dabney, J., Meyer, M. Extraction of highly degraded DNA from ancient bones and teeth. Ancient DNA: Methods and Protocols. , 25-29 (2019).
- Palsdottir, A. H., Bläuer, A., Rannamäe, E., Boessenkool, S., Hallsson, J. Not a limitless resource: ethics and guidelines for destructive sampling of archaeofaunal remains. Royal Society Open Science. 6 (10), 191059(2019).
- Pinhasi, R., Fernandes, D. M., Sirak, K., Cheronet, O. Isolating the human cochlea to generate bone powder for ancient DNA analysis. Nature Protocols. 14 (4), 1194-1205 (2019).
- Latham, K. E., Miller, J. J. DNA recovery and analysis from skeletal material in modern forensic contexts. Forensic Sciences Research. 4 (1), 51-59 (2019).
- Mundorff, A. Z., Bartelink, E. J., Mar-Cash, E. DNA preservation in skeletal elements from the World Trade Center disaster: Recommendations for mass fatality management. Journal of Forensic Sciences. 54 (4), 739-745 (2009).
- Gamba, C., et al. Genome flux and stasis in a five millennium transect of European prehistory. Nature Communications. 5 (1), 1-9 (2014).
- Alberti, F., et al. Optimized DNA sampling of ancient bones using Computed Tomography scans. Molecular Ecology Resources. 18 (6), 1196-1208 (2018).
- Hansen, H. B., et al. Comparing ancient DNA preservation in petrous bone and tooth cementum. PLoS ONE. 12 (1), 0170940(2017).
- Sirak, K., et al. Human auditory ossicles as an alternative optimal source of ancient DNA. Genome Research. 30 (3), 427-436 (2020).
- Parker, C., et al. A systematic investigation of human DNA preservation in medieval skeletons. Scientific Reports. 10 (1), 18225(2020).
- Pinhasi, R., et al. Optimal ancient DNA yields from the inner ear part of the human petrous bone. PLoS ONE. 10 (6), 0129102(2015).
- Sirak, K. A., et al. A minimally-invasive method for sampling human petrous bones from the cranial base for ancient DNA analysis. BioTechniques. 62 (6), 283-289 (2017).
- Orfanou, E., Himmel, M., Aron, F., Haak, W. Minimally-invasive sampling of pars petrosa (os temporale) for ancient DNA extraction. protocols.io. , Available from: https://www.protocols.io/view/minimally-invasive-sampling-of-pars-petrosa-os-tem-bqd8ms9w (2020).
- Damgaard, P. B., et al. Improving access to endogenous DNA in ancient bones and teeth. Scientific Reports. 5 (1), 1-12 (2015).
- Harney, É, et al. A minimally destructive protocol for DNA extraction from ancient teeth. Genome Research. 31 (3), 472-483 (2021).
- Cooper, A., Poinar, H. N. Ancient DNA: Do it right or not at all. Science. 289 (5482), 1139(2000).
- Llamas, B., et al. From the field to the laboratory: Controlling DNA contamination in human ancient DNA research in the high-throughput sequencing era. STAR: Science & Technology of Archaeological Research. 3 (1), 1-14 (2017).
- Llamas, B., et al. From the field to the laboratory: Controlling DNA contamination in human ancient DNA research in the high-throughput sequencing era. STAR: Science & Technology of Archaeological Research. 3 (1), 1-14 (2017).
- Boessenkool, S., et al. Combining bleach and mild predigestion improves ancient DNA recovery from bones. Molecular Ecology Resources. 17 (4), 742-751 (2017).
- García-Garcerà, M., et al. Fragmentation of contaminant and endogenous DNA in ancient samples determined by shotgun sequencing; Prospects for human palaeogenomics. PLoS ONE. 6 (8), 24161(2011).
- Malmström, H., et al. More on contamination: The use of asymmetric molecular behavior to identify authentic ancient human DNA. Molecular Biology and Evolution. 24 (4), 998-1004 (2007).
- Basler, N., et al. Reduction of the contaminant fraction of DNA obtained from an ancient giant panda bone. BMC Research Notes. 10, 754(2017).
- Kemp, B. M., Smith, D. G. Use of bleach to eliminate contaminating DNA from the surface of bones and teeth. Forensic Science International. 154 (1), 53-61 (2005).
- Korlević, P., et al. Reducing microbial and human contamination in DNA extractions from ancient bones and teeth. BioTechniques. 59 (2), 87-93 (2015).
- Dabney, J., Meyer, M. Extraction of highly degraded DNA from ancient bones and teeth. Methods in Molecular Biology. 1963, 25-29 (2019).
- Hansen, H. B., et al. Comparing ancient DNA preservation in petrous bone and tooth cementum. PLoS ONE. 12 (1), 0170940(2017).
- Gansauge, M. -T., et al. Single-stranded DNA library preparation from highly degraded DNA using T4 DNA ligase. Nucleic Acids Research. 45 (10), 79(2017).
- Peltzer, A., et al. EAGER: efficient ancient genome reconstruction. Genome Biology. 17 (1), 60(2016).
- Korneliussen, T. S., Albrechtsen, A., Nielsen, R. ANGSD: analysis of next generation sequencing data. BMC Bioinformatics. 15, 356(2014).
- Renaud, G., Hanghøj, K., Willerslev, E., Orlando, L. Gargammel: A sequence simulator for ancient DNA. Bioinformatics. 33 (4), 577-579 (2017).
- Ponce de León, M. S., et al. Human bony labyrinth is an indicator of population history and dispersal from Africa. Proceedings of the National Academy of Sciences of the United States of America. 115 (16), 4128-4133 (2018).
- Nagaoka, T., Kawakubo, Y. Using the petrous part of the temporal bone to estimate fetal age at death. Forensic Science International. 248, 188(2015).
- Norén, A., Lynnerup, N., Czarnetzki, A., Graw, M. Lateral angle: A method for sexing using the petrous bone. American Journal of Physical Anthropology. 128 (2), 318-323 (2005).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon