Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Caractérisation de la péritonite septique induite par Salmonella Typhimurium chez la souris
Dans cet article
Résumé
Ce protocole décrit l’induction d’une septicémie monobactérienne à Gram négatif dans un système modèle murin. Le modèle est utile pour étudier les réponses inflammatoires et létales de l’hôte pendant la septicémie.
Résumé
La septicémie est une réponse immunitaire dérégulée de l’hôte à une invasion microbienne ou à des lésions tissulaires, entraînant des lésions organiques à un site éloigné de celui de l’infection ou des dommages. Actuellement, les modèles de septicémie largement utilisés chez la souris comprennent l’endotoxémie induite par les lipopolysaccharides (LPS), la ligature et la ponction cæcales (CLP) et les systèmes modèles d’infection monobactérienne. Ce protocole décrit une méthode pour étudier les réponses de l’hôte au cours de la péritonite septique induite par l’infection à Salmonella Typhimurium chez la souris. S. Le typhimurium, un agent pathogène intracellulaire à Gram négatif, provoque une maladie semblable à la typhoïde chez la souris.
Ce protocole élabore la préparation de la culture, l’induction de la péritonite septique chez la souris par injection intrapéritonéale et les méthodes d’étude des réponses systémiques de l’hôte. En outre, l’évaluation de la charge bactérienne dans différents organes et l’analyse cytométrique en flux de l’augmentation du nombre de neutrophiles dans le lavage péritonéal sont présentées. Salmonelle La septicémie induite par le typhimurium chez la souris entraîne une augmentation des cytokines pro-inflammatoires et une infiltration rapide des neutrophiles dans la cavité péritonéale, entraînant une survie plus faible.
Chaque étape de ce protocole a été optimisée, ce qui a entraîné une reproductibilité élevée de la pathogenèse de la péritonite septique. Ce modèle est utile pour étudier les réponses immunologiques au cours de la septicémie bactérienne, les rôles de différents gènes dans la progression de la maladie et les effets des médicaments pour atténuer la septicémie.
Introduction
La septicémie est définie comme une réponse inflammatoire et immunitaire systémique dérégulée à une invasion microbienne ou à des lésions tissulaires, entraînant une lésion d’organe éloignée du site de l’infection ou des dommages. Le choc septique est un sous-ensemble de septicémie caractérisé par une hypotension persistant pendant la réanimation volumique, avec un risque considérablement accru de mortalité1. Le grand public est devenu plus conscient de ce trouble pendant la pandémie de COVID-19. Malgré la forte mortalité associée, des données épidémiologiques complètes sur le fardeau mondial de la septicémie font défaut en raison de la complexité de son diagnostic. En 2017, il y a eu 48,9 millions d’incidents de septicémie et 11 millions de décès dans le monde, ce qui représente 19,7 % de tous les décès dans le monde2. En outre, une étude sur la prévalence étendue de l’infection et de la septicémie associée chez les patients en unité de soins intensifs a révélé que 62% des isolats positifs des patients étaient des organismes à Gram négatif3.
Initialement, les recherches sur la septicémie se sont concentrées sur la délimitation de la pathogenèse microbienne. Cependant, la compréhension de « l’hypothèse du danger », qui dicte la façon dont l’hôte se distingue de soi et de non-soi, a conduit à faire pencher la balance de la recherche sur la septicémie vers la compréhension de la réponse de l’hôte à un agent pathogène envahissant. Les modèles de septicémie largement utilisés chez la souris comprennent le modèle d’endotoxémie induite par les lipopolysaccharides (LPS), les modèles de septicémie polymicrobienne, la ligature et la ponction cæcales (CLP) et la péritonite de stent ascendante du côlon (CASP) et les modèles d’infection monobactérienne4.
Nous avons normalisé un système de modèle murin en induisant une septicémie péritonéale à l’aide de Salmonella Typhimurium. Ce modèle est avantageux par rapport à d’autres parce que Salmonella Typhimurium est un agent pathogène intracellulaire qui imite l’état cliniquement pertinent de la septicémie à Gram négatif. Le résultat de la septicémie péritonite dans ce modèle est systémique, avec une mortalité de 100% dans les 96 heures suivant l’infection. Par conséquent, ce modèle joue un rôle déterminant dans l’étude des réponses inflammatoires et létales de l’hôte. Dans ce modèle, la septicémie est induite par l’injection intrapéritonéale de 0,5 million d’unités formant des colonies (UFC) de Salmonella Typhimurium dans une souris C57BL/6 âgée de 8 à 10 semaines. L’infection systémique peut être confirmée en évaluant la charge bactérienne des organes ~ 16 h après l’infection. Cet article démontre la septicémie de péritonite induite par Salmonella Typhimurium chez la souris, caractérise les altérations résultantes de la composition cellulaire péritonéale et quantifie la charge bactérienne dans différents organes.
Protocole
Toutes les expériences utilisant Salmonella Typhimurium ont été menées dans des installations de niveau de biosécurité 2 (BSL-2). Il faut veiller à utiliser un équipement de protection individuelle (EPI) approprié, à assurer la sécurité et à suivre les méthodes standard d’élimination des risques biologiques BSL-2. Toutes les expériences sur les souris ont été menées conformément aux directives énoncées par le Comité institutionnel d’éthique animale, IISc. Les souris ont été élevées et entretenues à l’installation animale centrale de l’IISc (numéro d’enregistrement: 48/1999/CPCSEA, daté du 1/3/1999), approuvé par le ministère de l’Environnement et des Forêts du gouvernement de l’Inde. Les protocoles expérimentaux ont été approuvés par le Comité des fins et du contrôle et de la supervision des expériences sur les animaux avec le numéro de permis approuvé CAF/Éthique/797/2020.
Définition BSL2 : Une cote BSL2 indique que les agents biodangereux constituent une menace modérée pour l’environnement et le personnel de laboratoire5.
1. Préparation de la culture de Salmonella Typhimurium
- Ajouter 100 μL de bouillon de glycérol Salmonella Typhimurium NCTC 12023 à 3 mL de bouillon Luria Bertani (LB). Incuber la culture à 160 tr/min à 37 °C pendant la nuit.
- Strier 50 μL de la culture cultivée pendant la nuit dans un bouillon LB sur une plaque de gélose Salmonella Shigella (SS) et incuber à 37 °C pendant environ 12 h. Conserver la plaque de gélose SS avec les colonies bactériennes à 4 °C pendant plusieurs jours avant l’expérience d’infection in vivo .
- Choisissez une seule colonie dans la plaque de gélose SS striée à l’aide d’une micro-pointe. Éjecter la micro-pointe dans 3 mL de bouillon LB et la cultiver à 160 tr/min à 37 °C pendant la nuit.
- Ajouter 0,1 mL de culture bactérienne à 50 mL de bouillon LB et incuber à 37 °C dans un incubateur agitateur à 160 tr/min pendant 3-4 h pour atteindre la phase logarithmique de croissance. Diluer la culture d’un facteur 2 en utilisant du bouillon LB.
REMARQUE: Pendant la phase logarithmique, les cellules bactériennes sont en meilleure santé et se divisent activement. - Mesurer la densité optique (OD) de la culture à une longueur d’onde de lumière de 600 nm dans un spectrophotomètre ou un lecteur de microplaques. Une fois que la DO atteint 1,0, faire deux aliquotes de 1 mL de culture dans des tubes de microfuge de 1,5 mL.
- Centrifugez les tubes à 7 750 × g pendant 15 min. Jetez le surnageant et lavez la pastille avec 1 mL de 1x PBS 2x. Centrifugez les tubes à 7 750 × g pendant 15 min.
- Remettre en suspension la pastille dans 0,5 mL de 1x PBS dans deux tubes de microfuge de 1,5 mL différents. Combinez les suspensions des deux tubes en un tube de 1,5 mL contenant maintenant ~ 2 × 108 unités formant des colonies (UFC) / mL.
- Préparer une suspension de cellules bactériennes de 1 × 106 UFC/mL en diluant cette solution mère.
ATTENTION : Optimiser l’UFC correspondant à la DO dans des conditions de laboratoire spécifiques afin de déterminer l’UFC pour la DO 1.0 avant de lancer les expériences.
2. Souris et infections
- Hébergez des souris mâles C57BL/6 âgées de 8 à 10 semaines pesant environ 20 g dans la salle d’air pur de l’animalerie pendant plusieurs jours pour l’acclimatation.
- Le jour de l’infection, tenez la souris d’une main, essuyez la peau abdominale avec 70% d’éthanol et écartez les pattes postérieures pour une meilleure accessibilité de la paroi abdominale.
- Injecter 0,5 mL de 1 × 106 UFC/mL de suspension bactérienne par voie intrapéritonéale à l’aide d’une seringue de 1 mL. Par conséquent, chaque souris reçoit 5 × 105 UFC. Témoin, les souris non infectées reçoivent 0,5 mL de PBS seul. Après l’infection, plaquez la culture pour vérifier l’UFC injectée, qui peut varier de 0,2 à 0,8 million d’UFC / 0,5 mL.
- Remettez les souris dans les cages comme assigné.
- Sacrifiez les souris en utilisant l’asphyxie au CO2 ~ 12-18 h après l’infection pour la meilleure réponse. Habituellement, toutes les souris infectées meurent dans les 96 heures. Dans le cadre de certaines interventions expérimentales, certaines souris peuvent survivre. Euthanasier ces souris après 96 h. De plus, euthanasier toute souris dont la température corporelle est inférieure à 33,2 °C et dont la détresse aiguë est ressentie à 96 h comme point de terminaison sans cruauté.
REMARQUE: Dans ce modèle, certaines souris peuvent commencer à mourir après 12 h d’injection de Salmonella . Par conséquent, planifiez correctement les expériences impliquant plusieurs points temporels.
3. Évaluation des organes par l’UFC
- Sacrifiez la souris infectée par asphyxie au CO2 et essuyez l’abdomen avec un morceau de coton trempé dans de l’éthanol à 70%. Coupez la peau abdominale. Reportez-vous à l’article de Ray et Dittel pour un protocole vidéo sur la façon de recueillir le liquide de lavage péritonéal6. Coupez la cavité péritonéale et collectez les organes d’intérêt dans des tubes de microfuge. De plus, comme le sang chez les souris atteintes de septicémie se coagule rapidement et que les quantités sont faibles, collectez-le rapidement après le sacrifice.
REMARQUE: Cette vidéo démontre le dénombrement de l’UFC d’organe du foie alors que le foie subit des dommages histopathologiques importants dans ce modèle de septicémie. - Coupez un petit morceau du foie et placez-le dans un tube de microfuge.
REMARQUE: Cela peut être stocké sur de la glace pendant un maximum de 2-3 h avant de passer à l’étape suivante. - Peser et transférer les pièces dans des tubes de microcentrifugation. De préférence, couper des morceaux pesant ~10-15 mg pour une homogénéisation correcte. Utilisez des organes entiers dans le cas d’organes plus petits tels que les ganglions lymphatiques mésentériques (MLN) ou le thymus.
- Ajouter 0,5 mL de 1x PBS au tube et homogénéiser les organes à l’aide d’un homogénéisateur à main. Assurez-vous que les organes sont complètement homogénéisés. Augmentez le volume à 1 mL en ajoutant 0,5 mL de 1x PBS.
- Centrifuger les tubes à 200 × g pendant 5 min à 4 °C.
- Recueillir le surnageant dans des tubes de microfuge frais et préparer des dilutions de 1 × 10−1 et 10−2 dans une plaque de 96 puits.
- Étaler 50 μL du diluant sur des plaques de gélose SS fraîches et incuber les plaques à 37 °C pendant 12 h.
- Comptez le nombre de colonies qui apparaissent dans chaque condition et normalisez les données avec le poids de l’organe à l’aide de l’équation (1) :
UFC/mg =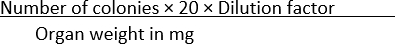 (1)
(1)
REMARQUE: Le nombre 20 est utilisé dans la formule pour convertir les colonies par plaque en UFC / mL. Ce nombre est obtenu en divisant 1 mL par la quantité d’un volume donné de la culture plaquée - dans ce cas, 50 μL.
Par exemple, si 100 colonies se trouvent dans une plaque de gélose SS, où 50 μL d’une dilution 1 × 10-1 d’un organe homogénéisé pesant 10 mg est étalée, alors
UFC/mg =
4. Analyse cytométrique en flux de diverses populations de cellules immunitaires dans l’exsudat péritonéal
- Prélevez les cellules péritonéales comme décrit précédemment par Ray et Dittel6.
- Remettre en suspension la pastille cellulaire du liquide de lavage péritonéal dans 1 mL de RPMI complété par 10% de sérum fœtal bovin (FBS). Dénombrer le nombre total de cellules dans le lavage péritonéal à l’aide d’un hémocytomètre. Ajustez le nombre de cellules de sorte que chaque tube reçoive 0,2 à 0,5 million de cellules.
REMARQUE: Le lavage péritonéal chez les souris atteintes de septicémie peut contenir des globules rouges, qui apparaissent probablement en raison d’une hémorragie. Veillez à exclure les globules rouges lors du comptage des cellules péritonéales. Dans le microscope à champ lumineux, les globules rouges apparaissent beaucoup plus petits que les cellules immunitaires. Ceux-ci apparaissent sous forme de disques plats ou de beignets, ronds, avec une indentation au centre, mais pas creux. Une étape pour lyser les globules rouges peut être ajoutée7. - Faites tourner les cellules à 200 × g à 4 °C pendant 10 min, jetez le surnageant et lavez les cellules 1x avec 1x PBS froid. Centrifuger les cellules à 200 × g à 4 °C pendant 10 min.
- Bloquer les récepteurs Fc sur les PEC à l’aide d’un bloqueur FcR (dilution 1:400), préparé dans un tampon bloquant composé de 5% de FBS et de 0,02% d’azoture de sodium dans le PBS. Incuber sur de la glace pendant 15 min.
- Centrifuger les cellules à 200 × g à 4 °C pendant 10 min. Jetez le surnageant. Diluer les anticorps conjugués au fluorochrome d’intérêt dans le tampon bloquant. Utilisez une dilution 1:500 de LY6G anti-souris pour tacher les neutrophiles.
REMARQUE: D’autres populations de cellules immunitaires peuvent également être détectées en utilisant, par exemple, anti-souris B220 pour les cellules B, anti-souris CD3 pour les cellules T et anti-souris F4/80 pour les macrophages. - Incuber environ 0,2 million de cellules dans 200 μL de solutions diluées d’anticorps dans des tubes séparés. Comme témoin négatif, réserver un tube dans chaque type de fluorochrome pour le contrôle non coloré afin d’incuber les cellules avec 200 μL de tampon bloquant sans anticorps.
- Incuber les échantillons sur de la glace pendant 45 minutes avec un tapotement intermittent toutes les 15 minutes.
- Centrifuger les cellules à 200 × g pendant 10 min à 4 °C. Jetez le surnageant. Fixez les cellules avec 4% de paraformaldéhyde pendant environ 15 minutes à température ambiante si elles doivent être stockées pendant plusieurs jours. Cependant, il est préférable d’acquérir des données à partir d’échantillons fraîchement colorés dans un cytomètre en flux.
- Remettre en suspension les cellules dans 200 μL de tampon de coloration FACS (2% FBS dans PBS). Acquérir les données dans un cytomètre en flux.
Résultats
Une caractérisation détaillée de la réponse immunitaire de l’hôte à l’aide de ce modèle particulier est présentée dans les publications précédentes 8,9. Quelques résultats représentatifs du protocole décrit sont présentés dans cette section. Ce modèle vise à induire une infection systémique de S. Typhimurium par injection intrapéritonéale de la culture bactérienne pour induire une septicémie. Pour confirmer l’infection, les l...
Discussion
Cet article décrit une méthode d’induction d’une forme grave de septicémie bactérienne par injection intrapéritonéale de Salmonella Typhimurium. Ce modèle est avantageux par rapport à d’autres car Salmonella Typhimurium est un agent pathogène intracellulaire et, par conséquent, hautement pathogène, imitant l’état cliniquement pertinent de la septicémie à Gram négatif. Le résultat de la septicémie péritonite dans ce modèle est systémique, avec une mortalité de 100% dans les 9...
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Nous remercions l’Installation centrale pour animaux, IISc, de nous avoir fourni des souris pour la recherche. Cette étude a été financée par des subventions au DpN du Department of Biotechnology and Science and Engineering Research Board, gouvernement de l’Inde. Le soutien infrastructurel du programme DBT-IISc et des subventions DST-FIST est grandement reconnu. Nous remercions tous les membres précédents et actuels du laboratoire DpN pour leur soutien.
matériels
| Name | Company | Catalog Number | Comments |
| Consumables | |||
| 1 mL Sterile Syringe with 26 G needle | Beckton Dickinson, Singapore | 303060 | |
| 1.5 mL Microcentrifuge Tube | Tarsons, USA | 500010 | |
| 10 mL Sterile Syringe with 21 G needle | Beckton Dickinson, Spain | 307758 | |
| 50 mL Conical Flask | Tarsons, USA | 441150 | |
| 50 mL Graduated Centrifuge Tube | Tarsons, USA | 546041 | |
| 50 mL Graduated Centrifuge Tube | Tarsons, USA | 546021 | |
| Cell spreader | VWR, USA | VWRU60828-680 | |
| Dulbecco’s Phosphate Buffered Saline | HiMedia, Mumbai, India | TS1006 | |
| Ethanol | Merck | 100983 | |
| FcR blocker | BD Biosciences | 553142 | |
| Fetal Bovine Serum | Gibco | 10270-106 | |
| FITC Rat anti-mouse Ly6G (Clone 1A8) | BD Pharmingen | 551460 | |
| Glycerol | Sigma-Aldrich | G9012 | |
| Hand based Homogenizer | - | - | |
| Hemocytometer (Neubauer counting chamber) | Rohem, India | I.S. 10269 | |
| Luria Bertani Broth | HiMedia, Mumbai, India | M1245 | |
| Paraformaldehyde | Sigma-Aldrich | 158127 | |
| Petriplates | Tarsons, USA | 460091 | |
| RPMI | Himedia, Mumbai, India | AT060-10X1L | |
| Salmonella-Shigella Agar | HiMedia, Mumbai, India | M108 | |
| Sodium azide | Sigma-Aldrich | S2002 | |
| Equipments | |||
| Centrifuge | Kubota | ||
| Flow cytometer | BD FACSverse | ||
| Incubator | N-biotek | ||
| Spectrophotometer | Shimadzu | ||
| Weighing machine | Sartorius |
Références
- Hotchkiss, R. S., et al. Sepsis and septic shock. Nature Reviews Disease Primers. 2 (1), 1-21 (2016).
- Rudd, K. E., et al. regional, and national sepsis incidence and mortality, 1990-2017: Analysis for the Global Burden of Disease Study. The Lancet. 395 (10219), 200-211 (2020).
- Vincent, J. L., et al. International study of the prevalence and outcomes of infection in intensive care units. JAMA. 302 (21), 2323-2329 (2009).
- Lewis, A. J., Seymour, C. W., Rosengart, M. R. Current murine models of sepsis. Surgical Infections. 17 (4), 385-393 (2016).
- Ta, L., Gosa, L., Nathanson, D. A. Biosafety and biohazards: Understanding biosafety levels and meeting safety requirements of a biobank. Biobanking. 1897, 213-225 (2019).
- Ray, A., Dittel, D. N. Isolation of Mouse Peritoneal Cavity Cells. Journal of Visualized Experiments: JoVE. (35), e1488 (2010).
- Liu, X., Quan, N. Immune cell isolation from mouse femur bone marrow. Bio-protocol. 5 (20), 1631 (2015).
- Yadav, S., et al. Nitric oxide synthase 2 enhances the survival of mice during Salmonella Typhimurium infection-induced sepsis by increasing reactive oxygen species, inflammatory cytokines and recruitment of neutrophils to the peritoneal cavity. Free Radical Biology & Medicine. 116, 73-87 (2018).
- Verma, T., et al. Cell-free hemoglobin is a marker of systemic inflammation in mouse models of sepsis: A Raman spectroscopic study. Analyst. 146 (12), 4022-4032 (2021).
- Cassado, A. D. A., Lima, M. R. D., Bortoluci, K. R. Revisiting mouse peritoneal macrophages: Heterogeneity, development, and function. Frontiers in Immunology. 6, 225 (2015).
- Yadav, S., Verma, T., Chattopadhyay, A., Nandi, D. Factors affecting the pathophysiology of sepsis, an inflammatory disorder: Key roles of oxidative and nitrosative stress. Indian Journal of Inflammation Research. 3 (1), 2 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon