È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Caratterizzazione della peritonite settica indotta da Salmonella Typhimurium nei topi
In questo articolo
Riepilogo
Questo protocollo descrive l'induzione della sepsi monobatterica Gram-negativa in un sistema di modelli murini. Il modello è utile per studiare le risposte infiammatorie e letali dell'ospite durante la sepsi.
Abstract
La sepsi è una risposta immunitaria dell'ospite disregolata all'invasione microbica o al danno tissutale, che porta a lesioni d'organo in un sito distante da quello dell'infezione o del danno. Attualmente, i modelli murini ampiamente utilizzati di sepsi includono endotossiemia indotta da lipopolisaccaride (LPS), legatura e puntura cecale (CLP) e sistemi modello di infezione monobatterica. Questo protocollo descrive un metodo per studiare le risposte dell'ospite durante la peritonite settica indotta da Salmonella Typhimurium nei topi. S. Il typhimurium, un agente patogeno intracellulare Gram-negativo, causa la malattia simile al tifo nei topi.
Questo protocollo elabora la preparazione della coltura, l'induzione della peritonite settica nei topi attraverso l'iniezione intraperitoneale e i metodi per studiare le risposte sistemiche dell'ospite. Inoltre, viene presentata la valutazione della carica batterica in diversi organi e l'analisi citometrica a flusso dell'aumento del numero di neutrofili nel lavaggio peritoneale. Salmonella La sepsi indotta da typhimurium nei topi porta ad un aumento delle citochine proinfiammatorie e alla rapida infiltrazione di neutrofili nella cavità peritoneale, portando a una minore sopravvivenza.
Ogni fase di questo protocollo è stata ottimizzata, con conseguente elevata riproducibilità della patogenesi della peritonite settica. Questo modello è utile per studiare le risposte immunologiche durante la sepsi batterica, i ruoli dei diversi geni nella progressione della malattia e gli effetti dei farmaci per attenuare la sepsi.
Introduzione
La sepsi è definita come una risposta infiammatoria e immunitaria sistemica disregolata all'invasione microbica o al danno tissutale, che porta a lesioni d'organo distanti dal sito di infezione o danno. Lo shock settico è un sottogruppo di sepsi caratterizzata da ipotensione persistente durante la rianimazione di volume, con un rischio sostanzialmente aumentato di mortalità1. Il pubblico in generale è diventato più consapevole di questo disturbo durante la pandemia di COVID-19. Nonostante la sua elevata mortalità associata, mancano dati epidemiologici completi sul carico globale della sepsi a causa della complessità della sua diagnosi. Nel 2017, ci sono stati 48,9 milioni di incidenze di sepsi e 11 milioni di morti in tutto il mondo, pari al 19,7% di tutti i decessi globali2. Inoltre, uno studio sulla prevalenza estesa dell'infezione e della sepsi correlata nei pazienti delle unità di terapia intensiva ha rilevato che il 62% degli isolati positivi dei pazienti erano organismi Gram-negativi3.
Inizialmente, le indagini sulla sepsi si sono concentrate sulla delineazione della patogenesi microbica. Tuttavia, la comprensione dell'"ipotesi del pericolo", che determina il modo in cui l'ospite distingue se stesso e non sé, ha portato all'inclinazione dell'equilibrio della ricerca sulla sepsi verso la comprensione della risposta dell'ospite a un agente patogeno invasore. I modelli di sepsi murina ampiamente utilizzati includono il modello di endotossiemia indotta da lipopolisaccaride (LPS), modelli di sepsi polimicrobica, legatura cecale e puntura (CLP) e peritonite dello stent ascendente del colon (CASP) e modelli di infezione monobatterica4.
Abbiamo standardizzato un sistema di modelli murini inducendo la sepsi peritoneale utilizzando Salmonella Typhimurium. Questo modello è vantaggioso rispetto ad altri perché Salmonella Typhimurium è un patogeno intracellulare che imita la condizione clinicamente rilevante della sepsi Gram-negativa. L'esito della sepsi da peritonite in questo modello è sistemico, con una mortalità del 100% entro 96 ore dall'infezione. Pertanto, questo modello è strumentale nello studio delle risposte infiammatorie e letali dell'ospite. In questo modello, la sepsi è indotta dall'iniezione intraperitoneale di 0,5 milioni di unità formanti colonie (CFU) di Salmonella Typhimurium in un topo C57BL/6 di 8-10 settimane. L'infezione sistemica può essere confermata valutando la carica batterica dell'organo ~ 16 ore dopo l'infezione. Questo articolo dimostra la sepsi da peritonite indotta da Salmonella Typhimurium nei topi, caratterizza le alterazioni risultanti nella composizione delle cellule peritoneali e quantifica la carica batterica in diversi organi.
Protocollo
Tutti gli esperimenti con Salmonella Typhimurium sono stati condotti in strutture di livello di sicurezza biologica 2 (BSL-2). È necessario prestare attenzione all'uso di adeguati dispositivi di protezione individuale (DPI), garantire la sicurezza e seguire i metodi standard di smaltimento a rischio biologico BSL-2. Tutti gli esperimenti sui topi sono stati condotti seguendo le linee guida stabilite dal Comitato etico animale istituzionale, IISc. I topi sono stati allevati e mantenuti presso la Central Animal Facility di IISc (numero di registrazione: 48/1999 / CPCSEA, datato 1/3/1999), approvato dal Ministero dell'Ambiente e delle Foreste, Governo dell'India. I protocolli sperimentali sono stati approvati dal Comitato per lo scopo e il controllo e la supervisione degli esperimenti sugli animali con il numero di autorizzazione approvato CAF/Ethics/797/2020.
Definizione BSL2: una classificazione BSL2 rappresenta che gli agenti biopericolosi rappresentano una minaccia moderata per l'ambiente e il personale di laboratorio5.
1. Preparazione della coltura di Salmonella Typhimurium
- Aggiungere 100 μL di Salmonella Typhimurium NCTC 12023 glicerolo a 3 mL di brodo Luria Bertani (LB). Incubare la coltura a 160 giri/min a 37 °C durante la notte.
- Strisciare 50 μL della coltura coltivata durante la notte in brodo LB su una piastra di agar Salmonella Shigella (SS) e incubare a 37 °C per ~ 12 ore. Conservare la piastra di agar SS con le colonie batteriche a 4 °C per diversi giorni prima dell'esperimento di infezione in vivo .
- Scegli una singola colonia dalla piastra di agar SS striata usando un microtip. Espellere il microtipo in 3 mL di brodo LB e coltivare a 160 giri/min a 37 °C durante la notte.
- Aggiungere 0,1 mL di coltura batterica a 50 mL di brodo LB e incubare a 37 °C in un incubatore shaker a 160 rpm per 3-4 h per raggiungere la fase logaritmica di crescita. Diluire la coltura di un fattore 2 usando il brodo LB.
NOTA: Durante la fase logaritmica, le cellule batteriche sono al loro meglio e si dividono attivamente. - Misurare la densità ottica (OD) della coltura a 600 nm di lunghezza d'onda della luce in uno spettrofotometro o in un lettore di micropiastre. Una volta che l'OD raggiunge 1,0, fare due aliquote di 1 mL di coltura in tubi di microfuga da 1,5 mL.
- Centrifugare i tubi a 7.750 × g per 15 min. Scartare il surnatante e lavare il pellet con 1 mL di 1x PBS 2x. Centrifugare i tubi a 7.750 × g per 15 min.
- Risospesciare il pellet in 0,5 mL di 1x PBS in due diversi tubi microfuga da 1,5 mL. Combinare le sospensioni di entrambi i tubi in un tubo da 1,5 mL ora contenente ~ 2 × 108 unità formanti colonie (CFU) / mL.
- Preparare una sospensione cellulare batterica di 1 × 106 CFU/mL diluendo questa soluzione madre.
ATTENZIONE: Ottimizzare il CFU corrispondente a OD in specifiche condizioni di laboratorio per determinare il CFU per OD 1.0 prima di iniziare gli esperimenti.
2. Topi e infezioni
- Casa topi maschi C57BL / 6 di 8-10 settimane del peso di ~ 20 g nella stanza dell'aria pulita della struttura per animali per diversi giorni per l'acclimatazione.
- Il giorno dell'infezione, tenere il mouse con una mano, pulire la pelle addominale con etanolo al 70% e diffondere le zampe posteriori per una migliore accessibilità della parete addominale.
- Iniettare 0,5 mL di 1 × 106 CFU/mL sospensione batterica per via intraperitoneale con l'aiuto di una siringa da 1 mL. Pertanto, ogni mouse riceve 5 × 105 CFU. Controllo, i topi non infetti ricevono 0,5 ml di PBS da solo. Dopo l'infezione, placcare la coltura per controllare l'effettivo CFU iniettato, che può variare da 0,2-0,8 milioni di CFU / 0,5 ml.
- Rimetti i topi nelle gabbie come assegnato.
- Sacrificare i topi usando l'asfissia co2 ~ 12-18 ore dopo l'infezione per la migliore risposta. Di solito, tutti i topi infetti muoiono entro 96 ore. Sotto alcuni interventi sperimentali, alcuni topi possono sopravvivere. Eutanasia di questi topi dopo 96 h. Inoltre, l'eutanasia di qualsiasi topo con una temperatura corporea inferiore a 33,2 °C e angoscia acuta a 96 ore come endpoint umano.
NOTA: In questo modello, alcuni topi possono iniziare a morire dopo 12 ore di iniezione di Salmonella . Pertanto, pianificare correttamente gli esperimenti che coinvolgono più punti temporali.
3. Valutazione degli organi dei CFU
- Sacrifica il topo infetto per asfissia di CO2 e pulisci l'addome con un pezzo di cotone immerso nel 70% di etanolo. Tagliare la pelle addominale. Fare riferimento all'articolo di Ray e Dittel per un protocollo video su come raccogliere il liquido di lavaggio peritoneale6. Tagliare la cavità peritoneale e raccogliere gli organi di interesse in tubi di microfuga. Inoltre, poiché il sangue nei topi con sepsi si coagula velocemente e le quantità sono basse, raccoglierlo rapidamente dopo il sacrificio.
NOTA: Questo video mostra l'enumerazione dei CFU d'organo dal fegato mentre il fegato subisce un ampio danno istopatologico in questo modello di sepsi. - Tagliare un piccolo pezzo di fegato e metterlo in un tubo di microfuga.
NOTA: Questo può essere conservato su ghiaccio per un massimo di 2-3 ore prima di procedere al passaggio successivo. - Pesare e trasferire i pezzi in tubi microcentrifuga. Preferibilmente, tagliare pezzi del peso di ~ 10-15 mg per una corretta omogeneizzazione. Utilizzare interi organi nel caso di quelli più piccoli come i linfonodi mesenterici (MLN) o timo.
- Aggiungere 0,5 ml di 1x PBS al tubo e omogeneizzare gli organi usando un omogeneizzatore a mano. Assicurarsi che gli organi siano completamente omogeneizzati. Aumentare il volume a 1 mL aggiungendo 0,5 mL di 1x PBS.
- Centrifugare i tubi a 200 × g per 5 min a 4 °C.
- Raccogliere il surnatante in provette di microfuga fresche e preparare diluizioni di 1 × 10−1 e 10−2 in una piastra a 96 pozzetti.
- Distribuire 50 μL del diluente su piastre di agar SS fresche e incubare le piastre a 37 °C per 12 ore.
- Contare il numero di colonie che appaiono in ogni condizione e normalizzare i dati con il peso dell'organo usando l'equazione (1):
CFU/mg =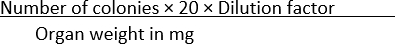 (1)
(1)
NOTA: Il numero 20 viene utilizzato nella formula per convertire le colonie per piastra in CFU/mL. Questo numero si ottiene dividendo 1 mL per la quantità di un dato volume della coltura placcata, in questo caso 50 μL.
Ad esempio, se si trovano 100 colonie in una piastra di agar SS, dove vengono diffusi 50 μL di una diluizione di 1 × 10-1 di un organo omogeneizzato del peso di 10 mg, allora
CFU/mg =
4. Analisi citometrica a flusso di varie popolazioni di cellule immunitarie nell'essudato peritoneale
- Raccogliere le cellule peritoneali come descritto in precedenza da Ray e Dittel6.
- Risospesso il pellet cellulare dal liquido di lavaggio peritoneale in 1 mL di RPMI integrato con il 10% di siero bovino fetale (FBS). Enumerare i numeri totali delle cellule nel lavaggio peritoneale usando un emocitometro. Regolare il numero di cella in modo che ogni tubo riceva 0,2-0,5 milioni di celle.
NOTA: Il lavaggio peritoneale nei topi con sepsi può contenere globuli rossi, che probabilmente compaiono a causa di emorragia. Fare attenzione a escludere i globuli rossi durante il conteggio delle cellule peritoneali. Nel microscopio a campo luminoso, i globuli rossi appaiono molto più piccoli delle cellule immunitarie. Questi appaiono come dischi piatti o ciambelle, rotondi, con una rientranza al centro, ma non vuoti. È possibile aggiungere un passaggio per lisare i globuli rossi7. - Far girare le cellule a 200 × g a 4 °C per 10 minuti, scartare il surnatante e lavare le cellule 1x con 1x PBS freddo. Centrifugare le celle a 200 × g a 4 °C per 10 min.
- Bloccare i recettori Fc sui PEC utilizzando il bloccante FcR (diluizione 1:400), preparato in tampone di blocco costituito da 5% FBS e 0,02% di azide di sodio in PBS. Incubare su ghiaccio per 15 min.
- Centrifugare le celle a 200 × g a 4 °C per 10 min. Scartare il surnatante. Diluire gli anticorpi coniugati al fluorocromo di interesse nel tampone bloccante. Utilizzare una diluizione 1:500 di anti-topo LY6G per macchiare i neutrofili.
NOTA: Altre popolazioni di cellule immunitarie possono anche essere rilevate utilizzando, ad esempio, anti-topo B220 per le cellule B, anti-topo CD3 per le cellule T e anti-topo F4/80 per i macrofagi. - Incubare ~ 0,2 milioni di cellule in 200 μL delle soluzioni diluite di anticorpi in tubi separati. Come controllo negativo, mettere da parte un tubo in ogni tipo di fluorocromo per il controllo non macchiato per incubare le cellule con 200 μL di tampone bloccante senza anticorpi.
- Incubare i campioni sul ghiaccio per 45 minuti con un tocco intermittente ogni 15 minuti.
- Centrifugare le celle a 200 × g per 10 min a 4 °C. Scartare il surnatante. Fissare le cellule con il 4% di paraformaldeide per ~ 15 minuti a temperatura ambiente se devono essere conservate per diversi giorni. Tuttavia, è meglio acquisire dati da campioni appena macchiati in un citometro a flusso.
- Risospesere le cellule in 200 μL di tampone di colorazione FACS (2% FBS in PBS). Acquisire i dati in un citometro a flusso.
Risultati
Una caratterizzazione dettagliata della risposta immunitaria dell'ospite utilizzando questo particolare modello è mostrata nelle precedenti pubblicazioni 8,9. In questa sezione sono riportati alcuni risultati rappresentativi del protocollo descritto. Questo modello mira a indurre l'infezione sistemica di S. Typhimurium mediante iniezione intraperitoneale della coltura batterica per indurre la sepsi. Per confermare l'infezione, i lisati del fegato e dell...
Discussione
Questo articolo descrive un metodo per indurre una forma grave di sepsi batterica mediante iniezione intraperitoneale di Salmonella Typhimurium. Questo modello è vantaggioso rispetto ad altri in quanto la Salmonella Typhimurium è un patogeno intracellulare e, quindi, altamente patogeno, imitando la condizione clinicamente rilevante della sepsi Gram-negativa. L'esito della sepsi da peritonite in questo modello è sistemico, con una mortalità del 100% entro 96 ore dall'infezione. Pertanto, questo model...
Divulgazioni
Gli autori non hanno conflitti di interesse da divulgare.
Riconoscimenti
Ringraziamo la Central Animal Facility, IISc per averci fornito topi per la ricerca. Questo studio è stato finanziato da sovvenzioni a DpN dal Dipartimento di Biotecnologia e Scienza e Ingegneria Research Board, Governo dell'India. Il sostegno infrastrutturale del programma DBT-IISc e le sovvenzioni DST-FIST sono ampiamente riconosciuti. Ringraziamo tutti i membri precedenti e attuali del laboratorio DpN per il loro supporto.
Materiali
| Name | Company | Catalog Number | Comments |
| Consumables | |||
| 1 mL Sterile Syringe with 26 G needle | Beckton Dickinson, Singapore | 303060 | |
| 1.5 mL Microcentrifuge Tube | Tarsons, USA | 500010 | |
| 10 mL Sterile Syringe with 21 G needle | Beckton Dickinson, Spain | 307758 | |
| 50 mL Conical Flask | Tarsons, USA | 441150 | |
| 50 mL Graduated Centrifuge Tube | Tarsons, USA | 546041 | |
| 50 mL Graduated Centrifuge Tube | Tarsons, USA | 546021 | |
| Cell spreader | VWR, USA | VWRU60828-680 | |
| Dulbecco’s Phosphate Buffered Saline | HiMedia, Mumbai, India | TS1006 | |
| Ethanol | Merck | 100983 | |
| FcR blocker | BD Biosciences | 553142 | |
| Fetal Bovine Serum | Gibco | 10270-106 | |
| FITC Rat anti-mouse Ly6G (Clone 1A8) | BD Pharmingen | 551460 | |
| Glycerol | Sigma-Aldrich | G9012 | |
| Hand based Homogenizer | - | - | |
| Hemocytometer (Neubauer counting chamber) | Rohem, India | I.S. 10269 | |
| Luria Bertani Broth | HiMedia, Mumbai, India | M1245 | |
| Paraformaldehyde | Sigma-Aldrich | 158127 | |
| Petriplates | Tarsons, USA | 460091 | |
| RPMI | Himedia, Mumbai, India | AT060-10X1L | |
| Salmonella-Shigella Agar | HiMedia, Mumbai, India | M108 | |
| Sodium azide | Sigma-Aldrich | S2002 | |
| Equipments | |||
| Centrifuge | Kubota | ||
| Flow cytometer | BD FACSverse | ||
| Incubator | N-biotek | ||
| Spectrophotometer | Shimadzu | ||
| Weighing machine | Sartorius |
Riferimenti
- Hotchkiss, R. S., et al. Sepsis and septic shock. Nature Reviews Disease Primers. 2 (1), 1-21 (2016).
- Rudd, K. E., et al. regional, and national sepsis incidence and mortality, 1990-2017: Analysis for the Global Burden of Disease Study. The Lancet. 395 (10219), 200-211 (2020).
- Vincent, J. L., et al. International study of the prevalence and outcomes of infection in intensive care units. JAMA. 302 (21), 2323-2329 (2009).
- Lewis, A. J., Seymour, C. W., Rosengart, M. R. Current murine models of sepsis. Surgical Infections. 17 (4), 385-393 (2016).
- Ta, L., Gosa, L., Nathanson, D. A. Biosafety and biohazards: Understanding biosafety levels and meeting safety requirements of a biobank. Biobanking. 1897, 213-225 (2019).
- Ray, A., Dittel, D. N. Isolation of Mouse Peritoneal Cavity Cells. Journal of Visualized Experiments: JoVE. (35), e1488 (2010).
- Liu, X., Quan, N. Immune cell isolation from mouse femur bone marrow. Bio-protocol. 5 (20), 1631 (2015).
- Yadav, S., et al. Nitric oxide synthase 2 enhances the survival of mice during Salmonella Typhimurium infection-induced sepsis by increasing reactive oxygen species, inflammatory cytokines and recruitment of neutrophils to the peritoneal cavity. Free Radical Biology & Medicine. 116, 73-87 (2018).
- Verma, T., et al. Cell-free hemoglobin is a marker of systemic inflammation in mouse models of sepsis: A Raman spectroscopic study. Analyst. 146 (12), 4022-4032 (2021).
- Cassado, A. D. A., Lima, M. R. D., Bortoluci, K. R. Revisiting mouse peritoneal macrophages: Heterogeneity, development, and function. Frontiers in Immunology. 6, 225 (2015).
- Yadav, S., Verma, T., Chattopadhyay, A., Nandi, D. Factors affecting the pathophysiology of sepsis, an inflammatory disorder: Key roles of oxidative and nitrosative stress. Indian Journal of Inflammation Research. 3 (1), 2 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon