Methods Article
Analyse de l’hérédité épigénétique transgénérationnelle chez C. elegans à l’aide d’un rapporteur fluorescent et de l’immunoprécipitation de la chromatine (ChIP)
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ce protocole décrit un test d’interférence de l’ARN et de ChIP pour étudier l’hérédité épigénétique du silençage induit par l’ARNi et des modifications de la chromatine associées chez C. elegans.
Résumé
L’héritage épigénétique transgénérationnel (TEI) permet la transmission d’informations à travers la lignée germinale sans modifier la séquence du génome, par le biais de facteurs tels que les ARN non codants et les modifications de la chromatine. Le phénomène d’hérédité par interférence ARN (ARNi) chez le nématode Caenorhabditis elegans est un modèle efficace pour étudier la TEI qui tire parti du court cycle de vie, de l’auto-propagation et de la transparence de cet organisme modèle. Dans l’hérédité de l’ARNi, l’exposition des animaux à l’ARNi entraîne un silençage génique et une altération des signatures de la chromatine au locus cible qui persistent pendant plusieurs générations en l’absence du déclencheur initial. Ce protocole décrit l’analyse de l’hérédité de l’ARNi chez C. elegans à l’aide d’un rapporteur de protéine fluorescente verte nucléaire (GFP) exprimée par la lignée germinale. Le silençage des rapporteurs est initié par l’alimentation des animaux avec des bactéries exprimant de l’ARN double brin ciblant la GFP. À chaque génération, les animaux sont passés pour maintenir un développement synchronisé, et le silençage des gènes rapporteurs est déterminé par microscopie. À certaines générations, les populations sont prélevées et traitées pour l’immunoprécipitation de la chromatine (ChIP) et la réaction en chaîne par polymérase quantitative (qPCR) afin de mesurer l’enrichissement en modification des histones au locus rapporteur de la GFP. Ce protocole d’étude de l’hérédité de l’ARNi peut être facilement modifié et combiné avec d’autres analyses pour étudier plus en détail les facteurs TEI dans les petites voies de l’ARN et de la chromatine.
Introduction
L’héritage épigénétique permet la transmission de l’information régulatrice des gènes à travers les générations et peut donc permettre à l’environnement ou aux expériences des parents d’affecter leur progéniture. Chez C. elegans, le silençage génique germinal initié par l’ARN double brin exogène (ARNdb) peut être hérité pendant plusieurs générations chez la descendance non exposée au déclencheur d’origine 1,2,3,4. Ce processus, appelé hérédité par interférence ARN (ARNi), est l’un des nombreux phénomènes de silençage épigénétique apparentés chez C. elegans, y compris le silençage multigénérationnel initié par les piARN 2,5, la paramutation/ARNe (silençage épigénétique induit par l’ARN)6,7,8 et le silençage initié par la matrice multicopie9, qui ont des exigences qui se chevauchent mais qui sont distinctes pour les petites machines de régulation de l’ARN et de la chromatine . Dans l’ARNi exogène de C. elegans, l’ARNdb est transformé en petits ARN interférents (siRNA) qui agissent en complexe avec les protéines primaires d’Argonaute pour reconnaître leur ARNm cible. Ce ciblage conduit à l’amplification des siARN secondaires qui s’associent aux Argonautes secondaires pour faire taire l’ARNm cible par des voies de silençage cytoplasmique et nucléaire. Pour les cibles d’ARNi exprimées par la lignée germinale, l’Argonaute secondaire nucléaire HRDE-1 et d’autres facteurs d’ARNi nucléaires ciblent les transcrits naissants, ce qui entraîne une répression transcriptionnelle et le recrutement d’histones méthyltransférases pour déposer des marques de chromatine répressives, y compris H3K9me310. L’histone H3K9me3 favorise l’établissement d’un silençage héréditaire des transgènes de la protéine fluorescente verte (GFP) exprimée par la lignée germinale par l’hérédité de l’ARNi11,12.
L’objectif de ce protocole est d’utiliser un transgène exprimant une protéine de fusion GFP-histones dans la lignée germinale comme rapporteur pour l’hérédité de l’ARNi et de tester les changements dans les modifications des histones au locus du transgène rapporteur en utilisant l’immunoprécipitation de la chromatine et la réaction en chaîne par polymérase quantitative (ChIP-qPCR). Ce protocole décrit une approche standardisée d’alimentation en ARNi à base de plaques pour initier le silençage du rapporteur. Il fournit également une chronologie détaillée du passage des animaux d’une génération à l’autre en isolant in utero les embryons d’adultes gravides par un traitement à l’hypochlorite alcalin (« blanchiment »). Des méthodes et des données représentatives pour le suivi de la fréquence du silençage de la GFP dans un sous-ensemble de la population par microscopie à fluorescence et pour l’histone H3K9me3 ChIP-qPCR sont également décrites. Les tests d’hérédité de l’ARNi basés sur des rapporteurs fournissent un système très facile à disséquer fonctionnellement les rôles des facteurs génétiques et environnementaux dans la régulation épigénétique 13,14, et les criblages génétiques utilisant de tels rapporteurs ont identifié à la fois les gènes nécessaires à 2,3,15 et les gènes qui régulent négativement la durée de l’héritage épigénétique transgénérationnel.
Protocole
REMARQUE : La figure 1 présente un calendrier d’analyse.
1. Préparation de plaques de milieu de croissance des nématodes à ARNi (RNAi-NGM)
- Filtrer E. coli HT115(DE3) contenant des vecteurs GFP ou ARNi témoins à partir de stocks de glycérol sur des plaques de gélose Luria-Bertani (LB) complétées par 100 μg/mL d’ampicilline. Incuber les bactéries pendant une nuit à 37 °C.
- Le lendemain, préparer des plaques d’ARNi-NGM (gélose à 1,7 % [p/v], NaCl à 0,3 % [p/v], peptone à 0,25 % [p/v], 1 mM de CaCl2, 5 μg/mL de cholestérol, 25 mM de tampon de phosphate de potassium [pH 6,0], 1 mM de MgSO4, 25 μg/mL de carbénicilline et 5 mM d’isopropyle β-d-1-thiogalactopyranoside [IPTG]) selon le protocole standard18.
- Pour chaque souche de l’essai, préparer au moins six plaques d’ARNi-NGM ensemencées d’ARNi GFP (deux plaques pour chacune des trois répétitions biologiques) et deux plaques d’ARNi-NGM ensemencées d’ARNi témoin (une répétition biologique).
- Recouvrez les assiettes de papier d’aluminium sans serrer pour les protéger de la lumière et laissez-les sécher toute la nuit à température ambiante.
- Le même jour que le versement de la plaque d’ARNi-NGM, préparer 4 mL de cultures liquides de bactéries ARNi.
- Dans des conditions stériles, aliquote 4 mL de bouillon LB complété par 10 μg/mL de tétracycline et 100 μg/mL d’ampicilline dans des tubes de culture. Prélever des colonies individuelles dans les plaques de gélose LB dans chaque tube et incuber toute la nuit en agitant pendant environ 16 h à 37 °C.
- Semez les bactéries ARNi sur les plaques RNAi-NGM.
- Après la croissance pendant la nuit, mesurez la densité optique (OD600) des cultures à l’aide d’une dilution de 1 :5 des bactéries avec du bouillon LB.
- Diluer les bactéries ARNi à un DO relatif de 600 de 2 en utilisant du bouillon LB complété par 10 μg/mL de tétracycline et100 μg/mL d’ampicilline.
- Dans des conditions stériles, ajouter 150 μL de bactéries ARNi témoins ou GFP sur chaque plaque d’ARNi-NGM.
- Recouvrez les assiettes de papier d’aluminium et laissez les bactéries se développer pendant au moins 24 h à température ambiante avant de les utiliser.
REMARQUE : Les plaques d’ARNi-NGM non utilisées peuvent être conservées à l’envers jusqu’à 1 semaine à 4 °C dans un récipient scellé à l’obscurité.
2. Démarrage du test d’hérédité de l’ARNi : blanchiment et placage des embryons pour la génération P0
NOTE : Avant de commencer l’essai d’hérédité de l’ARNi, les vers contenant le rapporteur GFP mjIs134 [mex-5p ::gfp-h2b ::tbb-2 3'UTR]7 doivent être maintenus non affamés à 21 °C pendant au moins deux générations.
- À 4 jours (96 h) avant le début de l’essai d’hérédité de l’ARNi (figure 1), prélever trois ou quatre adultes gravides sur chaque plaque NGM standard de 35 mm ensemencée avec la bactérie OP50-1.
REMARQUE : Trois plaques NGM par souche suffisent. Pour les souches dont la fertilité est compromise, plus de quatre animaux par plaque peuvent être nécessaires. - Après 4 jours, assurez-vous que la population adulte a commencé à pondre des embryons dans l’assiette. Laver les vers des plaques dans des tubes de 1,5 mL en utilisant 800 μL de tampon M9 complété par Triton X-100 (TX-100) (22 mM KH 2 PO 4, 42 mM Na2HPO 4, 86 mM NaCl, 1mM MgSO 4, 0,01 % (v/v) TX-100). Répétez le lavage et la piscine dans le même tube.
- Isolez les embryons de la population en les blanchissant comme suit.
- Préparez une solution de blanchiment avec de l’eau de Javel (hypochlorite de sodium à 6 %) et 10 N NaOH dans un rapport de volume de 1,5 :1. Préparez une quantité suffisante pour que 250 μL puissent être utilisés pour chaque échantillon.
- Centrifuger les tubes de 1,5 ml avec les vis sans fin à 1 000 x g pendant 1,5 à 2 min.
- Aspirer le surnageant en laissant 100 μL sans perturber la « pastille » de ver. Alternativement, les tubes peuvent être laissés pendant environ 2 minutes pour permettre aux vers de se déposer avant d’aspirer.
- Ajouter 650 μL de ddH2O dans chaque tube pour obtenir un volume final de 750 μL.
REMARQUE : Les étapes suivantes sont urgentes. - Ajoutez 250 μL de solution de blanchiment dans chaque tube et lancez une minuterie.
- Toutes les 1 à 2 minutes, faites tourner les tubes à fond. Après 5 min, vérifiez les vers sous le stéréoscope pour surveiller la dégradation des adultes.
- Continuez à vortex jusqu’à ce que les vers soient complètement dissous et qu’il ne reste que des embryons. Centrifuger immédiatement les tubes à 1 000 x g pendant 1,5 min.
REMARQUE : Le blanchiment est généralement terminé en 6 à 7 minutes, mais doit être surveillé sous un stéréoscope à un grossissement suffisant pour observer les embryons libérés (40x). Ne laissez pas les vers dans l’eau de Javel pendant une période prolongée, car cela compromettrait les embryons. - Après centrifugation, aspirer le surnageant et laisser environ 50 à 100 μL. Ajouter 1 mL de tampon M9 avec TX-100 pour laver et mélanger par vortex. Centrifugez à nouveau à 1 000 x g pendant 1,5 à 2 minutes et lavez au moins deux fois de plus.
- Aspirer le surnageant du lavage final et laisser environ 100 μL dans le tube. Mélanger par vortex. Pipeter 2 μL de chaque tube deux fois sur une lame de verre étiquetée. Comptez les embryons à l’aide d’un compteur de comptage pour estimer la concentration d’embryons par microlitre.
REMARQUE : Les lames avec plusieurs puits givrés peuvent être utilisées ici pour compter les répétitions de plusieurs souches. Les lames de comptage peuvent être lavées et réutilisées.
- Pour démarrer une croissance semi-synchronisée de la population, mélangez et pipetez un volume contenant 250 embryons sur chaque plaque d’ARNi-NGM. Utilisez deux plaques pour chaque répétition. Laisser le liquide absorber.
- Pour la génération P0 traitée à l’ARNi, incuber à 21 °C pendant 4 jours (96 h) (de l’embryon à l’âge adulte). Les périodes peuvent varier selon le génotype.
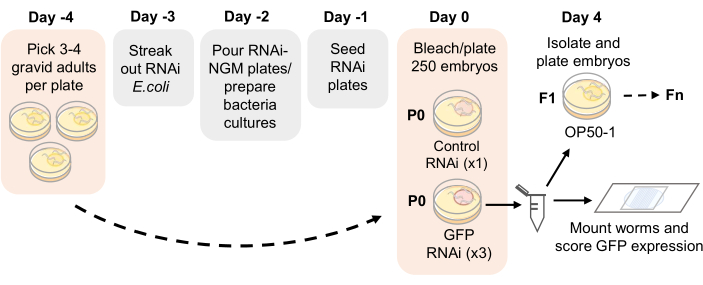
Figure 1 : Schéma du test d’hérédité de l’ARNi. Échéancier proposé pour la préparation de la plaque d’ARNi-NGM et la mise en place d’un test d’hérédité de l’ARNi. Les adultes gravides sont cueillis au jour -4 sur des plaques NGM ensemencées avec OP50-1. Après 4 jours, la progéniture adulte est blanchie et les embryons sont plaqués sur des plaques d’ARNi-NGM. La génération P0 est exposée à l’ARNi pendant 4 jours à 21 °C. Une fois que les vers atteignent l’âge adulte, les répétitions sont transmises par blanchiment et notées pour l’expression germinale de la GFP à chaque génération. Veuillez cliquer ici pour voir une version agrandie de cette figure.
3. Passage et notation de chaque génération pour l’expression de la GFP germinale
REMARQUE : Pour faciliter le marquage, utilisez un disque vinyle pour créer des tampons d’agarose avec des crêtes linéaires pour aligner les vers. Cette méthode a été adaptée de Rivera Gomez et Schvarzstein19.
- Préparer l’agarose à 1 % (p/v) en dissolvant l’agarose dans ddH2O dans une petite fiole en la chauffant au micro-ondes. Ajouter une barre de mélange et couvrir de papier d’aluminium. En cas de refonte, chauffer l’agarose sur une plaque chauffante à 200 °C tout en remuant. Une fois fondu, réduisez le feu à 80 °C.
- Laver les vers des plaques NGM à l’aide de 800 μL de tampon M9 avec TX-100 dans un tube de 1,5 mL. Lavez les assiettes deux fois pour éliminer tous les adultes gravides. Vérifiez les plaques sous le stéréoscope pour confirmer que les animaux ont été collectés efficacement.
- Centrifugez les tubes à 1 000 x g pendant 1,5 à 2 minutes ou laissez les vers se déposer pendant 2 minutes. Aspirer le surnageant et laisser 100 μL sans perturber la « pastille » de ver.
- Préparez les tampons d’agarose pour le montage des vers comme suit.
- Placez une goutte d’agarose fondue à 1 % (p/v) sur le disque vinyle (décrit précédemment19) à l’aide d’une pipette Pasteur en verre (avec l’extrémité étroite cassée).
- Orientez une lame de verre de manière à ce que les lignes du disque soient horizontales ou verticales et placez rapidement la diapositive sur la goutte d’agarose. Attendez environ 30 s avant de retirer la lame de verre du disque.
REMARQUE : Si vous marquez plusieurs génotypes, il peut être utile de faire un coussinet d’agarose plus grand sur une lame et de le couper en deux à l’aide d’une lame.
- Montez les vers sur les tampons d’agarose.
- Sous le stéréoscope, ajouter environ 5 μL de lévamisole de 5 mM au tampon d’agarose. La dilution du lévamisole doit être faite fraîche chaque semaine.
- Effleurez doucement les tubes de vers pour mélanger et transférer 5 à 10 μL de vers sur le tampon d’agarose. Estimez le nombre de vers à l’œil nu au fur et à mesure qu’ils sont ajoutés afin qu’il y ait environ 40 vers par répétition.
- Alignez les vers en rangées à l’aide d’un cure-cils pour faciliter le marquage. Ajoutez une lamelle en verre.
REMARQUE : Les toboggans avec des animaux montés de cette manière peuvent durer plusieurs heures avant de sécher.
- Isolez les embryons des animaux restants en suivant le protocole de blanchiment (voir l’étape 2.3) avant de marquer les lames. Plaquer 250 embryons sur des plaques NGM de 35 mm ensemencées avec OP50-120. Une fois le liquide absorbé, retournez les plaques et remettez-les dans l’incubateur à 21 °C.
- Utilisez un stéréoscope à florescence avec un jeu de filtres GFP. Comptez et notez le nombre de vers GFP positifs et négatifs GFP sur les lames pour chaque répétition à l’aide d’un compteur de comptage.
REMARQUE : Les vers positifs à la GFP auront une expression nucléaire de la GFP dans leur lignée germinale et les embryons in utero. Les lignées germinales des vers GFP négatifs ne seront pas fluorescentes, cependant, lorsque la GFP commence à se réactiver dans les générations ultérieures, la fluorescence peut être faible. Il peut être utile de monter d’autres souches de vers non transgéniques pour tenir compte de toute autofluorescence observée. - Passage de la population par blanchiment, comme décrit ci-dessus, environ tous les 4 jours (96 h) à 21 °C. Alternativement, le passage peut être effectué selon un horaire alterné de 3 jours et de 4 jours pour plus de commodité. Plaquez ~50 embryons supplémentaires pour les générations de passage plus courtes si l’alternance, car il peut y avoir un rendement plus faible d’embryons après 72 h.
REMARQUE : Maintenir le temps de passage de la génération P0 constant à 4 jours après le placage des embryons.
4. Collecte d’animaux pour ChIP
REMARQUE : Le nombre d’animaux et le moment dépendent de la souche, du stade de développement, de l’épitope et du nombre de cibles d’immunoprécipitation (PP). Dans l’exemple ci-dessous, collectez des animaux pour trois adresses IP : H3K9me3, l’histone H3 et le contrôle IgG. Le protocole ChIP a été adapté d’Askjaer et al.21.
- Avant le prélèvement de l’échantillon ChIP, élargissez la génération précédente du test d’hérédité de l’ARNi de trois plaques NGM supplémentaires (à au moins quatre plaques par répétition). Cultivez pendant 4 jours à 21 °C.
- Blanchir les adultes gravides pour isoler les embryons (voir l’étape 2.3).
- Pour chaque souche, plaquer environ 3 500 embryons sur 14 plaques (250 par plaque) et les faire croître pendant 3 jours à 21 °C jusqu’au stade de jeune adulte.
- Laver les animaux dans des tubes de 1,5 ml avec une solution saline tamponnée au phosphate (PBS) contenant 0,01 % (v/v) TX-100 (PBS/TX). Centrifuger à 1 000 x g pendant 2 min. Aspirez et regroupez les animaux d’une souche dans un tube et lavez-les trois fois de plus avec au moins 1 mL de PBS/TX.
- Aspirer jusqu’à 1 000 μL, inverser pour mélanger et compter trois fois le nombre d’animaux dans 3 μL (voir l’étape 2.3.9).
- Calculer la concentration d’animaux et transférer un volume correspondant à 3 000 animaux dans un nouveau tube de réticulation du formaldéhyde.
- Assurez-vous que le volume total est au moins 10 fois le volume de granulés de ver.
5. Réticulation du formaldéhyde
ATTENTION : Travaillez avec du formaldéhyde dans une hotte pour éviter l’exposition à la vapeur.
- Ajouter du formaldéhyde (concentration finale de 1,8 %) dans le tube contenant les vers. Laisser tourner à température ambiante pendant 6 min.
- Congeler immédiatement dans de l’azote liquide. L’expérience peut être interrompue à cette étape et les échantillons peuvent être stockés à -80 °C.
- Décongeler l’échantillon réticulé dans un bain-marie à température ambiante pendant 3 min et le faire tourner pendant 16 min à température ambiante.
- Ajouter 1,25 M de glycine à une concentration finale de 125 mM et tourner pendant 5 min.
REMARQUE : Jusqu’à l’élution des billes magnétiques, conserver les échantillons sur de la glace ou à 4 °C et utiliser des tampons glacés. - Centrifuger l’échantillon à 1 000 x g pendant 3 min. Laver trois fois avec 1 mL de PBS/TX à chaque fois. Laver deux fois avec 1 mL de tampon de remise en suspension (150 mM de NaCl, 50 mM D’HEPES-KOH [pH 7,5], 1 mM d’acide éthylènediaminetétraacétique [EDTA], 0,01 % TX-100, inhibiteur de protéase [un comprimé pour 5 mL]).
- Après le dernier lavage, laissez suffisamment de tampon pour vous assurer que le volume total est au moins trois fois le volume des granulés et un minimum de ~100 μL.
6. Sonication
REMARQUE : Les paramètres de sonication dépendent du type et du modèle de sonicateur et du stade animal. Des paramètres tels que le volume et la concentration de l’échantillon, les intervalles marche/arrêt, le nombre de cycles et le réglage de la puissance doivent être optimisés empiriquement. Par exemple, au cours d’une période de sonication, surveillez la lyse des vers à l’aide d’un test protéique et déterminez quand la concentration atteint un plateau. De plus, surveiller lorsque la taille moyenne du cisaillement de l’ADN génomique est d’environ 200 à 1 000 pb par électrophorèse de l’ADN, purifié après inversion de réticulation, sur un gel d’agarose/tris-acétate-EDTA (TAE) à 1,5 %.
- Mesurez le volume d’échantillons de l’étape précédente. Mélanger et aliquote 90-120 μL dans un tube de sonication en polystyrène.
- Ajouter un volume égal de tampon de remise en suspension contenant 2 détergents (150 mM de NaCl, 50 mM DE HEPES-KOH [pH 7,5], 1 mM d’EDTA, 0,2 % de désoxycholate de sodium, 0,7 % de sarkosyl).
- Soniser dans un sonicateur à bain-marie à 50 % de puissance pendant 7 min (20 s marche/40 s éteint) à 4 °C. Mélanger délicatement par pipetage. Répétez la sonication pendant 7 minutes supplémentaires.
- Transférez le lysat sonique dans un tube de 1,5 ml. Ajouter 0,5 volume de tampon de remise en suspension sans détergents (150 mM de NaCl, 50 mM DE HEPES-KOH [pH 7,5], 1 mM d’EDTA) (par exemple, ajouter 100 μL de tampon à 200 μL d’échantillon).
- Centrifuger à 13 000 x g pendant 15 min à 4 °C. Conservez le surnageant de lysat et transférez-le dans un nouveau tube.
- Si vous le souhaitez, effectuez un test protéique pour déterminer les concentrations de chaque lysat. Cette étape peut être utile pour les grandes quantités d’échantillons ou les comparaisons entre essais. Cependant, la standardisation du nombre d’animaux telle que décrite ci-dessus fonctionne bien pour les quantités d’échantillons décrites ici, car il y a une meilleure corrélation avec le rendement de la chromatine/de l’ADN tel que déterminé par qPCR.
7. Immunoprécipitation
REMARQUE : Adaptez la quantité d’anticorps et de billes magnétiques au volume et à la concentration de lysat.
- Divisez le surnageant de lysat en quatre portions : trois volumes égaux pour chaque IP et 10 % du volume d’un IP en entrée (p. ex., 100 μL de IP #1, 100 μL de IP #2, 100 μL de IP #3, 10 μL d’entrée). Transférez le lysat IP dans un tube PCR de 200 μL et stockez le lysat d’entrée à -20 °C dans un tube de 1,5 mL.
- Ajouter 0,5 μg d’anticorps anti-H3K9me3, d’anticorps anti-histone H3 ou d’IgG à l’échantillon IP approprié. Incuber à 4 °C pendant la nuit avec rotation.
- Le lendemain, aliquote 9 μL de billes magnétiques enrobées de protéines G par IP dans un seul tube de 1,5 mL. Lavez les billes deux fois avec 1 mL de FA-150 (150 mM de NaCl, 50 mM D’HEPES-KOH [pH 7,5], 1 mM d’EDTA, 1 % de TX-100, 0,1 % de désoxycholate de sodium).
- Remettez les billes magnétiques en suspension dans le tampon FA-150 jusqu’au volume d’origine prélevé sur le stock à l’étape 7.3 ci-dessus. Ajoutez 7,5 μL à chaque IP. Incuber à 4 °C pendant 2 h en rotation.
8. Lavages et élution
REMARQUE : Pour vous assurer que les billes magnétiques ne sèchent pas, ajoutez chaque tampon de lavage ou d’élution rapidement après avoir aspiré le lavage précédent.
- Utilisez 0,2 à 1 ml des solutions tampons suivantes à chaque fois pour laver les billes. Récupérez les perles sur un support magnétique pour aspirer les lavages. Incuber chaque lavage à 4 °C pendant 5 min avec rotation. Suivez l’ordre ci-dessous.
- Laver deux fois avec le FA-150.
- Laver une fois avec FA-1M (FA-150 avec 1 M de NaCl).
- Laver une fois avec FA-0,5M (FA-150 avec 0,5 M NaCl). Transférer dans un nouveau tube PCR.
- Laver une fois avec TE-LiCl (250 mM LiCl, 10 mM Tris-Cl [pH 8,0], 1 mM EDTA, 1 % d’IGEPAL CA-630, 1 % de désoxycholate de sodium).
- Laver deux fois avec TE+ (50 mM de NaCl, 10 mM de Tris-Cl [pH 8,0], 1 mM d’EDTA, 0,005 % d’IGEPAL CA-630).
REMARQUE : Poursuivre le traitement de l’échantillon à température ambiante, sauf indication contraire.
- Aspirez le dernier tampon de lavage. Remettre les billes magnétiques en suspension avec 50 μL de tampon d’élution ChIP (200 mM de NaCl, 10 mM de Tris-Cl [pH 8,0], 1 mM d’EDTA, 1 % de dodécylsulfate de sodium [SDS]) et transférer dans un tube de 1,5 mL.
- Éluer à 65 °C pendant 15 min dans un thermomélangeur avec mélange à 1 000 tr/min pendant 5 s par minute.
- Récupérez les perles sur un support magnétique et transférez le surnageant dans un nouveau tube.
- Répétez l’élution avec 50 μL supplémentaires de tampon d’élution ChIP. Regrouper les surnageants pour un total de 100 μL. Procéder à la réticulation inverse et à l’élution de l’ADN, comme indiqué ci-dessous.
9. Réticulation inverse et élution de l’ADN
- Décongeler l’échantillon de lysat d’entrée. Complétez jusqu’à 100 μL avec le tampon d’élution ChIP.
- Ajouter 16,5 μg de RNase A à chaque échantillon IP et d’entrée. Incuber à 37 °C pendant 1 h.
- Ajouter 40 μg de protéinase K et incuber à 55 °C pendant 2 h. Ensuite, incubez à 65 °C pendant la nuit.
- Refroidissez les échantillons à température ambiante et purifiez l’ADN à l’aide d’un kit de colonne de centrifugation.
10. Configuration et exécution de la réaction qPCR
REMARQUE : Les paramètres de l’amorce, de la configuration de la réaction et du thermocycleur doivent être modifiés pour correspondre aux recommandations du fabricant pour le mélange réactionnel qPCR utilisé.
- Préparer des ensembles d’amorces ciblant le rapporteur d’ARNi GFP et les régions de contrôle d’enrichissement positif et négatif H3K9me3. La température de fusion de l’apprêt est de 60 °C. Voir le tableau des matériaux pour les séquences d’amorces.
- Pour l’ADN d’entrée, faites quatre dilutions en série quadruples (par exemple, 1 :5, 1 :20, 1 :80, 1 :320).
REMARQUE : L’ADN IP peut être utilisé directement ou dilué (p. ex., 1 :2 ou 1 :3) pour permettre plus de réactions. La pertinence de la dilution doit être déterminée empiriquement, car les performances de la qPCR peuvent être affectées. - Organiser toutes les réactions correspondant à un jeu d’amorces sur une plaque PCR. Configurez des réactions techniques dupliquées pour chaque dilution d’ADN d’entrée et chaque échantillon d’ADN IP. Chaque réaction de 10 μL contient 1,5 μL d’ADN d’entrée ou IP (ou tampon d’élution comme témoin), un mélange maître qPCR (1x concentration finale), une amorce directe et une amorce inverse (concentration finale de 400 nM pour chaque amorce).
- Sur un thermocycleur temps réel, exécuter le programme suivant : dénaturation initiale : 95 °C pendant 4 min ; amplification et détection de fluorescence : 40 cycles de 95 °C pendant 10 s et 60 °C pendant 30 s avec lecture de plaque ; extension finale : 60 °C pendant 5 min ; courbe de fusion : de 60 °C à 90 °C par incréments de 0,5 °C, 5 s par pas.
11. Détermination de l’efficacité de l’amplification et vérification de la spécificité du produit
- Pour chaque ensemble de dilutions d’ADN d’entrée, tracez les quatre points de données avec [log10(1/dilution)] sur l’axe des abscisses et [Cq d’entrée] sur l’axe des ordonnées. Déterminez la pente de la ligne de meilleur ajustement.
- Calculez l’efficacité de l’amplification. Les ensembles d’apprêts idéaux doivent afficher une efficacité constante de 95 % à 100 %.
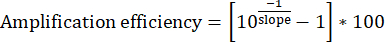
- Vérifiez que toutes les réactions ont un pic de courbe de fusion aigu et que les réactions avec le même jeu d’apprêts ont la même température de fusion. Des pics multiples ou des températures de fusion différentes peuvent indiquer une amplification non spécifique.
- Si vous le souhaitez, effectuez des réactions sur un gel standard d’agarose/TAE à 2 % à température ambiante pour vérifier la taille de la bande du produit.
12. Calcul du pourcentage d’intrants
- Calculez le facteur de dilution d’entrée. Étant donné que 10 % du volume de lysat IP a été enregistré en entrée, le facteur de dilution pour la dilution 1 :5 de l’ADN d’entrée est de 50.
- Déterminez le facteur de dilution de l’IP. Si l’ADN IP n’est pas dilué avant la qPCR, le facteur de dilution est de 1.
- Calculez la différence de Cq entre l’IP et l’entrée ajustée pour les facteurs de dilution.
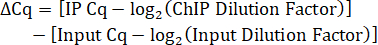
- Calculez le pourcentage d’entrée.
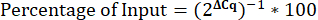
Résultats
Les animaux porteurs de l’histone Gfp-H2B exprimée par la lignée germinale [mex-5p ::gfp-h2b ::tbb-2 3'UTR]7 rapporteur (Figure 2A) ont été exposés à l’ARNi GFP ou à l’ARNi témoin en se nourrissant et en passant comme décrit dans le protocole et dans la Figure 1. Le signal de GFP nucléaire dans la lignée germinale a été évalué manuellement à l’aide d’un microscope à dissection à fluorescence pour un échantillon de la population à chaque génération. L’inhibition du transgène a été complètement pénétrante chez les animaux P0 et F1 marqués traités avec l’ARNi GFP (Figure 2B). Dans la génération F2, la proportion de la population présentant une hérédité du silençage de la GFP était d’environ 50 %. À la génération F5, la majorité de la population n’a pas montré d’hérédité de silençage, et à la génération F10, aucune hérédité n’a été détectée, car tous les animaux exprimaient la GFP.
Pour déterminer le changement dans l’enrichissement de l’histone H3K9me3 correspondant au silençage induit par l’ARNi, la ChIP-qPCR a été réalisée sur des animaux de génération F1 après un traitement par ARNi GFP ou ARNi témoin. Comme on pouvait s’y attendre, la population traitée par l’ARNi GFP présentait des niveaux plus élevés d’histone H3K9me3 à la cible GFP et à la région aval de 1,3 kb par rapport aux animaux témoins traités à l’ARNi (Figure 2C). La spécificité de l’histone H3K9me3 ChIP est étayée par l’enrichissement à un locus témoin positif (clec-18) connu pour être enrichi en cette marque, mais pas à un locus témoin négatif voisin (hrp-2). L’enrichissement en histone H3 et l’enrichissement proche du bruit de fond dans les immunoprécipitations de contrôle des IgG ont également été détectés à tous les loci qPCR, comme prévu. Lorsque l’enrichissement en ChIP au niveau du rapporteur est normalisé au locus témoin positif clec-18 , un enrichissement plus élevé en histone H3K9me3 sur l’ARNi GFP est montré, tandis que l’enrichissement en histone H3 est similaire entre le traitement par ARNi témoin et GFP (Figure 2D). Étant donné qu’on ne s’attend pas à ce que l’ARNi GFP affecte l’histone H3K9me3 ou l’occupation totale de l’histone H3 au locus clec-18 , cette normalisation atténue les variations techniques, telles que les différences d’efficacité ChIP entre les échantillons d’ARNi GFP et d’ARNi témoin. Le changement de pli des niveaux d’histone H3K9me3 et d’histone H3 entre les traitements à ARNi montre un enrichissement de l’histone H3K9me3 spécifique du rapporteur GFP, indépendant de l’occupation des histones, lors de l’inactivation induite par l’ARNi GFP (Figure 2E).
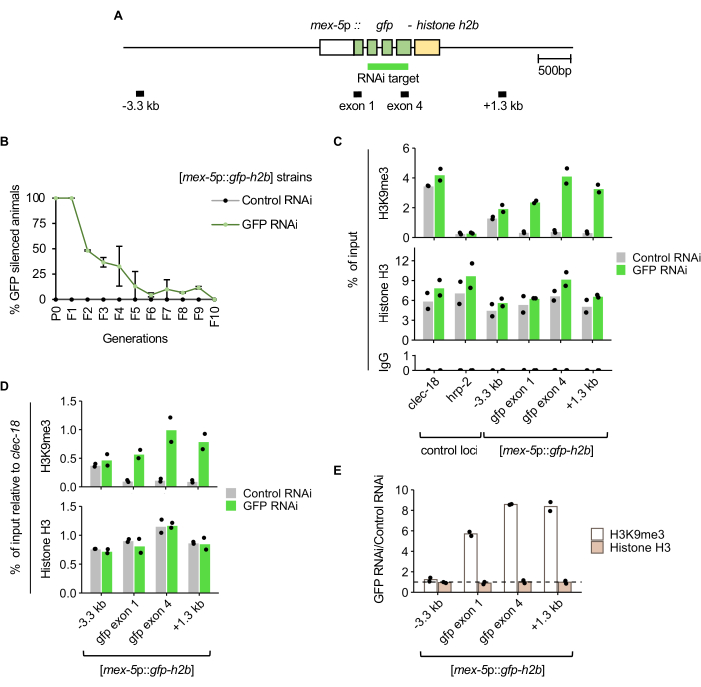
Figure 2 : Le silençage induit par l’ARNi de la GFP correspond à un enrichissement élevé de H3K9me3 au niveau de la cible de l’ARNi. (A) Diagramme de l’ARNi GFP exprimé par la lignée germinale mjIs134[mex-5p ::gfp-h2b ::tbb-2 3'UTR] avec des régions d’amplicon qPCR marquées. (B) Expression de la GFP notée sur plusieurs générations après des traitements à l’ARNi à 21 °C. Les barres d’erreur représentent l’écart-type de deux répétitions biologiques. (C) ChIP-qPCR de H3K9me3, de l’histone H3 et du contrôle IgG chez les jeunes adultes F1 à partir de deux répétitions biologiques. clec-18 et hrp-2 sont les loci de contrôle positifs et négatifs pour l’enrichissement en H3K9me3, respectivement. (D) Enrichissement de H3K9me3 et de l’histone H3 au rapporteur d’ARNi GFP normalisé au locus témoin positif clec-18. (E) Modification de l’enrichissement en H3K9me3 et en histone H3 entre les animaux traités à l’ARNi GFP et les animaux traités à l’ARNi témoin, avec normalisation à clec-18. La ligne pointillée représente un changement de pli de 1. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Dans ce protocole, l’ARNdb est introduit par l’alimentation, ce qui est devenu une méthode standard chez C. elegans18. Pour les tests d’hérédité de l’ARNi, l’approche d’alimentation fournit une méthode facile pour obtenir une grande population de P0 2,11,12,22,23,24,25. Cependant, le moment et la durée de l’exposition à l’ARNi affectent l’efficacité du silençage des transgènes26, et la concentration des bactéries ARNi affecte la persistance du silençage de l’ARNi héréditaire1. Par conséquent, la croissance standardisée des bactéries ARNi et des vers est importante pour obtenir un niveau constant de silençage de la GFP et une durée d’hérédité. Ici, les embryons sont plaqués sur des plaques d’ARNi afin que les animaux P0 soient exposés aux bactéries ARNi dès l’éclosion. D’autres approches ont permis de plaquer des animaux synchronisés de stade L1 24,27 ouL4 25 sur des plaques RNAi-NGM. De plus, étant donné que la famine et d’autres stress affectent le maintien de l’hérédité de l’ARNi13, les plaques doivent être surveillées pour éviter le surpeuplement et l’épuisement de l’approvisionnement alimentaire. Comme alternative à l’initiation de l’ARNi par l’alimentation, certaines des études pionnières sur l’hérédité de l’ARNi de C. elegans ont induit l’ARNi par injection de gonades, ce qui permet un meilleur contrôle de la concentration d’ARNdb 1,28.
Dans ce protocole, chaque génération est transmise en tant que population avec un traitement à l’hypochlorite alcalin, comme décrit précédemment 16,22,23,24. Le traitement à l’hypochlorite garantit que la génération F1 ne sera pas contaminée par des bactéries ARNi provenant de l’environnement parental22 et empêche un éventuel goulot d’étranglement indésirable de la population. Cependant, le passage en masse peut également être une limitation, car les animaux individuels peuvent avoir des schémas de transmission distincts 1,29. Une autre méthode pour établir chaque génération consiste à sélectionner des animaux individuels 1,2,9,11,25. Cette approche permet de suivre les phénotypes au sein des lignées et d’incorporer des croisements génétiques. L’analyse de la lignée peut également être avantageuse pour les phénotypes à faible pénétrance12.
L’expression de la protéine fluorescente est une lecture puissante et pratique du silençage induit par l’ARNi 2,3,4,12,14,15,16,25,30. Comme décrit dans ce protocole, l’expression GFP peut être évaluée manuellement comme ON ou OFF 11,12,15,16,25. La notation manuelle peut être affinée en attribuant des niveaux d’intensité qualitatifs 3,4,14. Alternativement, le score d’intensité automatisé des images microscopiques 11,12,14,25,27 ou la mesure de fluorescence par cytométrie en flux d’animaux vivants2 peuvent fournir une lecture quantitative et à haut débit. Cependant, étant donné que l’expression rapporteur est limitée à la lignée germinale, une mise en garde des approches automatisées est qu’elles doivent également faire la différence entre les animaux dont l’expression de la GFP est réduite au silence et les animaux sans lignée germinale, en particulier si l’étude intègre des mutants ayant un développement anormal de la lignée germinale. Comme alternative à la fluorescence GFP, la transcription inverse (RT)-qPCR peut être utilisée pour quantifier les niveaux de pré-ARNm et d’ARNm ciblesde l’ARNm 2,16,23,24. Cette approche fournit une lecture plus directe du silençage, qui cible l’ARN, et est particulièrement utile pour d’autres cibles d’ARNi où le silençage ne produit pas de phénotype visible. L’une des limites de l’utilisation de rapporteurs GFP artificiels est que les séquences exogènes et endogènes sont régulées de manière différentielle dans l’ARNi transgénérationnel11. Les études portant sur des cibles endogènes, telles que l’allèle létal létal embryonnaire oma-1(zu405)1,11,16,25 sensible à la température, doivent donc être considérées comme une approche complémentaire aux rapporteurs de transgènes fluorescents.
L’analyse de ChIP dans le cadre des tests d’hérédité de l’ARNi nécessite une comparaison entre les traitements et les répétitions. Tout d’abord, pour tenir compte des différences de matériau de départ entre les échantillons, le signal ChIP est normalisé en signal d’entrée au même locus que le « pourcentage d’entrée ». Le traitement d’une histone H3 ChIP en parallèle aidera à déterminer si les altérations de modification des histones correspondent à des changements dans la densité des nucléosomes. De plus, étant donné que ChIP est un processus en plusieurs étapes, l’efficacité peut varier d’un échantillon à l’autre. La sélection de loci de contrôle positifs et négatifs appropriés est utile pour évaluer et comparer le rapport signal/bruit entre les échantillons et les expériences. De plus, pour faciliter les comparaisons entre les échantillons, les valeurs seuils du cycle de qPCR de l’ADN ChIP à la cible de l’ARNi sont souvent normalisées à un locus de contrôle 3,11,15,23,30,31. Pour évaluer les effets du traitement à l’ARNi, le rapport entre le signal ChIP dans l’ARNi de traitement et les conditions de contrôle ou d’absence d’ARNi est également comparé. L’une des limites de l’approche actuelle est que la CHIP est réalisée sur des animaux entiers, alors que la réponse au traitement par ARNi peut être unique à la lignée germinale. Une approche pour surmonter cette mise en garde consiste à effectuer une ChIP à l’aide de noyaux germinaux isolés. D’autres considérations techniques pour l’optimisation de ChIP ont également été discutées en détail ailleurs32,33.
Dans l’ensemble, ce protocole d’hérédité de l’ARNi et de ChIP fournit une base détaillée et facile à adapter qui peut être intégrée à d’autres techniques pour explorer davantage la régulation épigénétique transgénérationnelle. Par exemple, des banques de séquençage à haut débit peuvent être construites à partir de l’ADN ChIP (ChIP-seq) pour une vue plus détaillée du paysage de la chromatine à la fois à proximité de la cible de l’ARNi et à l’échelle du génome.
Déclarations de divulgation
Tous les auteurs confirment qu’ils n’ont aucun conflit à divulguer.
Remerciements
Nous tenons à remercier les laboratoires de la communauté C. elegans qui ont développé et partagé les outils et dont le travail est cité dans ce manuscrit. Certaines souches ont été fournies par le CCG, qui est financé par le Bureau des programmes d’infrastructure de recherche des NIH (P40 OD010440). Ces travaux ont été financés par une subvention Projet des Instituts de recherche en santé du Canada (IRSC) à A.L.S. (PJT-175245). C.L. bénéficie d’une bourse d’études supérieures du Conseil de recherches en sciences naturelles et en génie du Canada (CRSNG).
matériels
| Name | Company | Catalog Number | Comments |
| Agarose | Bioshop | AGA002 | |
| Ampicillin | Bioshop | AMP201 | Make a 100 mg/mL solution in ultrapure water. Filter-sterilize and store at -20 °C. |
| Anti-H3K9me3 Rabbit Polyclonal Antibody | Abcam | ab8898 | Concentration is batch-dependent (0.9 - 1 mg/mL). |
| Anti-Histone H3 Rabbit Polyclonal Antibody | Abcam | ab1791 | Concentration is batch-dependent (0.7 - 1 mg/mL). |
| Bleach (6% Sodium hypochlorite) | Lavo | 02358107 | |
| C. elegans strain with GFP RNAi Reporter | NA | SX1263 | Sapetschnig et al. 2015 (ref. 7). A gift from E. Miska lab, University of Cambridge. |
| Carbenicillin | BioShop | CAR544 | Make a 25 mg/mL solution in ultrapure water. Filter-sterilize and store at -20 °C. |
| Dynabeads Protein G Magnetic Beads | Invitrogen | 10003D | |
| E. coli strain HT115(DE3) | Caenorhabditis Genetics Center (CGC) | HT115(DE3) | |
| E. coli strain OP50-1 | Caenorhabditis Genetics Center (CGC) | OP50-1 | |
| EDTA (0.5 M, pH 8.0) | Invitrogen | 15575020 | |
| Fluorescence Stereoscope | Zeiss | Axio Zoom.V16 | |
| Formaldehyde (37%) | Sigma | F8775 | |
| Glycine | Sigma | 50046 | Make a 1.25 M solution and store at 4 °C. |
| HEPES-KOH (1 M, pH 7.5) | Teknova | H1035 | |
| Hydrophobic Printed Slides, 10 wells | VWR | 100488-904 | |
| IGEPAL CA-630 (Octylphenol ethoxylate) | BioShop | NON999 | Make a 10% (v/v) solution in ultrapure water and store at room temperature. |
| IPTG (Isopropyl-β-D-thiogalactoside) | BioShop | IPT001 | Make a 0.2 g/mL solution in ultrapure water. Filter-sterilize and store at -20 °C. |
| iTaq Universal SYBR Green Supermix | Bio-Rad | 1725122 | |
| LB Agar Plates supplemented with 100 µg/mL Ampicillin | NA | NA | Standard lab recipe. |
| Levamisole (Tetramisole hydrochloride) | Sigma | L9756 | Make a 200 mM solution in ultrapure water. Store at -20 °C. |
| LiCl (8 M) | Sigma | L7026 | |
| M9 Buffer | NA | NA | 22 mM KH2PO4, 42 mM Na2HPO4, 86 mM NaCl, 1 mM MgSO4. |
| Magnetic Separator (1.5 mL tubes) | Applied Biosystems | A13346 | |
| Magnetic Separator (0.2 mL tubes) | Permagen | MSR812 | |
| Microscope Cover Glass | Fisher Scientific | 12541B | |
| Microscope Slide | Technologist Choice | LAB-037 | |
| NaCl (5 M) | Promega | V4221 | For ChIP buffers. |
| NaOH | Sigma | S5881 | Make a 10 M solution and store at room temperature. |
| NGM Plates | NA | NA | 1.7% (w/v) agar, 0.3% (w/v) NaCl, 0.25% (w/v) peptone, 1 mM CaCl2, 5 μg/mL cholesterol, 25 mM Potassium phosphate pH 6.0, 1 mM MgSO4, 50 µg/mL streptomycin. |
| Normal Rabbit IgG (1 mg/mL) | Cell Signaling Technology | 2729 | |
| Petri Dishes (35 mm x 10 mm) | Sarstedt | 82.1135.500 | |
| Phosphate Buffered Saline (10X) | Fisher BioReagents | BP3991 | |
| Plasmid - Control RNAi | Addgene | L4440 (Plasmid #1654) | |
| Plasmid - GFP-targetting RNAi | Addgene | L4417 (Plasmid #1649) | Note, alternative L4440-derived plasmids targeting GFP can be used. |
| Primer pair [-3.3 kb upstream of gfp] | Integrated DNA Technologies | NA | F: AAACCAAAGGACGAGAGATTCA, R: GGCTCGATCAAGTAAAATTTCG |
| Primer pair [+1.3 kb downstream of gfp] | Integrated DNA Technologies | NA | F: TCGACCAGTTCTAAAGTCACCG, R: ACGTGCGGGATCATTTCTTACT |
| Primer pair [clec-18] | Integrated DNA Technologies | NA | F: TGCTCCATGACCTCAACAACA, R: AGTACAGTTCACCGATCCAGA |
| Primer pair [gfp exon 1] | Integrated DNA Technologies | NA | F: CTGGAGTTGTCCCAATTCTTGT, R: GGGTAAGTTTTCCGTATGTTGC |
| Primer pair [gfp exon 4] | Integrated DNA Technologies | NA | F: GATGGCCCTGTCCTTTTACCA, R: ATGCCATGTGTAATCCCAGCA |
| Primer pair [hrp-2] | Integrated DNA Technologies | NA | F: CGTCAACAGGGAGCAGCTG, R: CCTCCGAACTTTCTCTGTCCA |
| Protease Inhibitor Cocktail Tablet | Roche | 11836170001 | |
| Proteinase K | Bioline | BIO-37084 | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| Real-Time PCR Detection System | Bio-Rad | CFX96 | |
| RNAi-NGM plates | NA | NA | 1.7% (w/v) agar, 0.3% (w/v) NaCl, 0.25% (w/v) peptone, 1 mM CaCl2, 5 µg/mL cholesterol, 25 mM Potassium phosphate buffer pH 6.0, 1 mM MgSO4, 25 µg/mL carbenicillin and 5 mM IPTG. |
| RNase A | Sigma | R4642 | |
| Sarkosyl (N-Lauroylsarcosine sodium salt) | Sigma | L5777 | Make a 10% (w/v) solution and store at room temperature protected from light for a maximum of 1 month. |
| SDS | Sigma | 74255 | Make a 10% (w/v) solution and store at room temperature. |
| Sodium deoxycholate | Sigma | 30970 | Make a 5% (w/v) solution and store at room temperature protected from light for a maximum of 1 month. |
| Sonication Tube | Evergreen | 214-3721-010 | |
| Sonication Tube Cap | Evergreen | 300-2911-020 | |
| Sonicator | Qsonica | Q800R3-110 | |
| Streptomycin sulfate | Bioshop | STP101 | Make a 50 mg/mL solution in ultrapure water. Filter-sterilize and store at -20 °C. |
| TAE buffer (1X) | NA | NA | 40 mM Tris, 20 mM acetate, 1 mM EDTA |
| Tally counter clicker | Uline | H-7350 | |
| Tetracycline | Bioshop | TET701 | Make a 5 mg/mL solution in ethanol and store at -20 °C. |
| Thermomixer | Eppendorf | 05-400-205 | |
| Tris-HCl (1 M, pH 8.0) | Invitrogen | 15568025 | |
| Triton X-100 | Sigma | T8787 | Make a 10% (v/v) solution in ultrapure water and store at room temperature. |
Références
- Alcazar, R. M., Lin, R., Fire, A. Z. Transmission dynamics of heritable silencing induced by double-stranded RNA in Caenorhabditis elegans. Genetics. 180 (3), 1275-1288 (2008).
- Ashe, A., et al. piRNAs can trigger a multigenerational epigenetic memory in the germline of C. elegans. Cell. 150 (1), 88-99 (2012).
- Buckley, B. A., et al. A nuclear Argonaute promotes multigenerational epigenetic inheritance and germline immortality. Nature. 489 (7416), 447-451 (2012).
- Vastenhouw, N. L., et al. Long-term gene silencing by RNAi. Nature. 442 (7105), 882 (2006).
- Lee, H. -. C., et al. C. elegans piRNAs mediate the genome-wide surveillance of germline transcripts. Cell. 150 (1), 78-87 (2012).
- Luteijn, M. J., et al. Extremely stable Piwi-induced gene silencing in Caenorhabditis elegans. The EMBO Journal. 31 (16), 3422-3430 (2012).
- Sapetschnig, A., Sarkies, P., Lehrbach, N. J., Miska, E. A. Tertiary siRNAs mediate paramutation in C. elegans. PLoS Genetics. 11 (3), e1005078 (2015).
- Shirayama, M., et al. piRNAs initiate an epigenetic memory of nonself RNA in the C. elegans germline. Cell. 150 (1), 65-77 (2012).
- Minkina, O., Hunter, C. P. Stable heritable germline silencing directs somatic silencing at an endogenous locus. Molecular Cell. 65 (4), 659-670 (2017).
- Seroussi, U., et al. Mechanisms of epigenetic regulation by C. elegans nuclear RNA interference pathways. Seminars in Cell & Developmental Biology. 127, 142-154 (2022).
- Lev, I., Gingold, H., Rechavi, O. H3K9me3 is required for inheritance of small RNAs that target a unique subset of newly evolved genes. eLife. 8, e40448 (2019).
- Woodhouse, R. M., et al. Chromatin modifiers SET-25 and SET-32 are required for establishment but not long-term maintenance of transgenerational epigenetic inheritance. Cell Reports. 25 (8), 2259-2272 (2018).
- Houri-Zeevi, L., Teichman, G., Gingold, H., Rechavi, O. Stress resets ancestral heritable small RNA responses. eLife. 10, e65797 (2021).
- Houri-Ze'evi, L., et al. A tunable mechanism determines the duration of the transgenerational small RNA inheritance in C. elegans. Cell. 165 (1), 88-99 (2016).
- Spracklin, G., et al. The RNAi inheritance machinery of Caenorhabditis elegans. Genetics. 206 (3), 1403-1416 (2017).
- Perales, R., et al. Transgenerational epigenetic inheritance is negatively regulated by the HERI-1 chromodomain protein. Genetics. 210 (4), 1287-1299 (2018).
- Shukla, A., Perales, R., Kennedy, S. piRNAs coordinate poly(UG) tailing to prevent aberrant and perpetual gene silencing. Current Biology. 31 (20), 4473-4485 (2021).
- Ahringer, J. Reverse GeneticsWormBook: the Online Review of C. elegans Biology. WormBook. , (2006).
- Rivera Gomez, K., Schvarzstein, M. Immobilization of nematodes for live imaging using an agarose pad produced with a Vinyl Record. microPublication Biology. 2018, (2018).
- Stiernagle, T. Maintenance of C. elegans. WormBook: The Online Review of C. Elegans Biology. , 1-11 (2006).
- Askjaer, P., Ercan, S., Meister, P. Modern techniques for the analysis of chromatin and nuclear organization in C. elegans. WormBook: the Online Review of C. elegans Biology. , 1-35 (2014).
- Gu, S. G., et al. Amplification of siRNA in Caenorhabditis elegans generates a transgenerational sequence-targeted histone H3 lysine 9 methylation footprint. Nature Genetics. 44 (2), 157-164 (2012).
- Kalinava, N., Ni, J. Z., Peterman, K., Chen, E., Gu, S. G. Decoupling the downstream effects of germline nuclear RNAi reveals that H3K9me3 is dispensable for heritable RNAi and the maintenance of endogenous siRNA-mediated transcriptional silencing in Caenorhabditis elegans. Epigenetics & Chromatin. 10, 6 (2017).
- Kalinava, N., et al. elegans heterochromatin factor SET-32 plays an essential role in transgenerational establishment of nuclear RNAi-mediated epigenetic silencing. Cell Reports. 25 (8), 2273-2284 (2018).
- Lev, I., et al. MET-2-dependent H3K9 methylation suppresses transgenerational small RNA inheritance. Current Biology. 27 (8), 1138-1147 (2017).
- Kamath, R. S., Martinez-Campos, M., Zipperlen, P., Fraser, A. G., Ahringer, J. Effectiveness of specific RNA-mediated interference through ingested double-stranded RNA in Caenorhabditis elegans. Genome Biology. 2 (1), (2001).
- Xu, F., et al. A cytoplasmic Argonaute protein promotes the inheritance of RNAi. Cell Reports. 23 (8), 2482-2494 (2018).
- Grishok, A., Tabara, H., Mello, C. C. Genetic requirements for inheritance of RNAi in C. elegans. Science. 287 (5462), 2494-2497 (2000).
- Houri-Zeevi, L., Korem Kohanim, Y., Antonova, O., Rechavi, O. Three rules explain transgenerational small RNA inheritance in C. elegans. Cell. 182 (5), 1186-1197 (2020).
- Burton, N. O., Burkhart, K. B., Kennedy, S. Nuclear RNAi maintains heritable gene silencing in Caenorhabditis elegans. Proceedings of the National Academy of Sciences. 108 (49), 19683-19688 (2011).
- Mao, H., et al. The Nrde pathway mediates small-RNA-directed histone H3 lysine 27 trimethylation in Caenorhabditis elegans. Current Biology. 25 (18), 2398-2403 (2015).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Research. 22 (9), 1813-1831 (2012).
- Mukhopadhyay, A., Deplancke, B., Walhout, A. J. M., Tissenbaum, H. A. Chromatin immunoprecipitation (ChIP) coupled to detection by quantitative real-time PCR to study transcription factor binding to DNA in Caenorhabditis elegans. Nature Protocols. 3 (4), 698-709 (2008).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon