Cartographie quantitative de la déformation d'un anévrisme de l'aorte abdominale
Vue d'ensemble
Source: Hannah L. Cebull1, Arvin H. Soepriatna1, John J. Boyle2 et Craig J. Goergen1
1 Fois Weldon School of Biomedical Engineering, Purdue University, West Lafayette, Indiana
2 (en) Génie mécanique et science des matériaux, Université de Washington à St. Louis, St Louis, Missouri
Le comportement mécanique des tissus mous, tels que les vaisseaux sanguins, la peau, les tendons et d'autres organes, sont fortement influencés par leur composition d'élastine et de collagène, qui fournissent élasticité et force. L'orientation des fibres de ces protéines dépend du type de tissu mou et peut aller d'une seule direction préférée à des réseaux maillés complexes, qui peuvent devenir altérés dans les tissus malades. Par conséquent, les tissus mous se comportent souvent anisotropically au niveau cellulaire et d'organe, créant un besoin pour la caractérisation tridimensionnelle. Il est important de développer une méthode d'estimation fiable des champs de souches dans des tissus ou des structures biologiques complexes pour caractériser et comprendre mécaniquement la maladie. La souche représente la façon dont les tissus mous se déforment relativement au fil du temps, et il peut être décrit mathématiquement à travers diverses estimations.
L'acquisition de données d'image au fil du temps permet d'estimer la déformation et la tension. Cependant, toutes les modalités d'imagerie médicale contiennent une certaine quantité de bruit, ce qui augmente la difficulté d'estimer avec précision la souche in vivo. La technique décrite ici permet de surmonter ces problèmes avec succès en utilisant une méthode d'estimation de déformation directe (DDE) pour calculer spatialement différents champs de souches 3D à partir de données d'image volumétriques.
Les méthodes actuelles d'estimation des souches comprennent la corrélation d'image numérique (DIC) et la corrélation numérique du volume. Malheureusement, DIC ne peut estimer avec précision la souche d'un avion 2D, limitant gravement l'application de cette méthode. Bien qu'utiles, les méthodes 2D telles que le DIC ont de la difficulté à quantifier la souche dans les régions qui subissent une déformation 3D. C'est parce que le mouvement hors plan crée des erreurs de déformation. La corrélation numérique du volume est une méthode plus applicable qui divise les données de volume initiale en régions et trouve la région la plus similaire du volume déformé, réduisant ainsi l'erreur hors plan. Cependant, cette méthode s'avère sensible au bruit et nécessite des hypothèses sur les propriétés mécaniques du matériau.
La technique démontrée ici élimine ces problèmes en utilisant une méthode DDE, ce qui la rend très utile dans l'analyse des données d'imagerie médicale. En outre, il est robuste à souche élevée ou localisée. Ici, nous décrivons l'acquisition de données d'échographie 4D fermées et volumétriques, sa conversion en format analysable, et l'utilisation d'un code Matlab personnalisé pour estimer la déformation 3D et les souches green-Lagrange correspondantes, un paramètre qui décrit mieux les grandes déformations. Le tenseur de souche Green-Lagrange est mis en œuvre dans de nombreuses méthodes d'estimation des souches 3D, car il permet de calculer F à partir d'un Least Squares Fit (LSF) des déplacements. L'équation ci-dessous représente le tenseur de souche Green-Lagrange, E, où F et moi représentons respectivement le gradient de déformation et le tenseur d'identité de deuxième ordre.
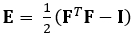 (1)
(1)
Principles
L'échographie 4D est un volume dynamique qui est acquis à l'aide d'un moteur de traduction linéaire attaché à un transducteur d'ultrasons, permettant l'acquisition de boucles vidéo séquentielles cardiaques et respiratoires fermées dans une région d'intérêt. Cette méthode est utile pour visualiser des structures complexes telles que le cœur, où l'hypertrophie ou l'infarctus provoque des géométries uniques, ou des anévrismes aortiques, où l'expansion et la dissection asymétriques des vaisseaux se produisent souvent dans les vaisseaux tortueux. En outre, les données 4D peuvent fournir des informations spatiales et temporelles à haute résolution, ce qui est également important pour l'imagerie cardiovasculaire.
La méthode DDE appliquée aux données d'échographie 4D est supérieure à d'autres méthodes parce qu'elle utilise l'enregistrement d'image non rigide. Les tenseurs de gradient de déformation sont traditionnellement estimés à partir des champs de déplacement suivant la corrélation numérique du volume. En revanche, la méthode DDE estime intrinsèquement les tenseurs de gradient de déformation lors de l'enregistrement du volume en optimisant une fonction de déformation soigneusement choisie pour être directement analogue au tenseur de déformation. La fonction de déformation dépend à la fois de la position spatiale et du paramètre de déformation (p):
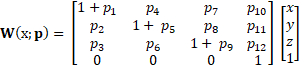 (2)
(2)
Les trois premiers éléments de cette fonction représentent le tenseur de gradient de déformation, F, permettant de intégrer directement le calcul de la déformation dans la fonction de déformation. Cette méthode de déformation a été prouvée pour augmenter la précision et la précision de l'estimation de la souche par rapport à des techniques précédentes similaires, car elle permet des déformations grandes ou localisées couramment trouvés dans les tissus mous.
Procédure
1. Set-up d'ultrason4D
- Lorsque vous utilisez le logiciel d'imagerie, utilisez un ordinateur portable capable d'exécuter un logiciel informatique mathématique pour automatiser le processus d'acquisition 4D. Connectez l'ordinateur portable avec ce code personnalisé au système d'échographie via le port USB. Notez que le logiciel d'imagerie a une fonction d'échographie 4D intégrée dans le logiciel.
- Après avoir allumé le système d'échographie, configurez l'unité de surveillance physiologique tout en veillant à ce que la fréquence cardiaque et les boutons de température soient allumés. Initialisez l'étage moteur 3D attaché au support du transducteur.
- Utilisez le transducteur d'étape et d'ultrason approprié pour l'acquisition d'images. Assurez-vous que toutes les connexions appropriées sont faites.
- Procéder à l'anesthésie et la préparation de l'animal pour l'imagerie. Ajoutez l'onuleur ophtalmique aux yeux pour empêcher la dessiccation cornéenne, fixez les pattes aux électrodes de scène, et insérez une sonde de température rectale lubrifiée. Retirer la fourrure dans la zone d'intérêt à l'aide d'une crème dépilatoire.
- Assurez-vous que la crème dépilatoire est terminée. Ensuite, appliquez une quantité généreuse du gel transduisant ultrasonique réchauffé à l'animal. Ceci est particulièrement important pour créer une bonne connexion sur toute la région d'intérêt pour l'imagerie 4D.
2. Acquisition d'ultrasons 4D
- Commencez une nouvelle étude sur le système d'échographie et ouvrez la fenêtre d'imagerie en mode B (mode luminosité). Abaissez le transducteur sur l'animal et localisez la région d'intérêt à l'aide des boutons x et y-axe sur la scène, en s'assurant que la fréquence respiratoire ne diminue pas considérablement. Surveillez cela en bas de l'écran.
- Placez le transducteur au milieu de la région d'intérêt. À partir de là, approximatif de la distance nécessaire pour que le transducteur se déplace de haut en bas de sorte que toute la région d'intérêt est incluse.
- Entrez les dimensions approximatives dans le code logiciel informatique, y compris une taille d'étape qui est généralement de 0,08 mm pour l'imagerie aurifysm aortique abdominale. Commencez à exécuter le code après s'assurer que les fréquences cardiaques et respiratoires de l'animal sont stables. Ceci est important pour réduire les erreurs lors de la reconstruction d'images.
- Après avoir terminé l'acquisition d'image, exportez les données sous forme de fichiers XML bruts.
3. Conversion de données d'ultrason 4D
- Entrez les fichiers XML bruts dans un logiciel qui peut convertir les données dans le format approprié pour l'analyse des taches 3D. Ici, nous utilisons Matlab pour convertir les fichiers XML en fichiers MAT. Le script Matlab complet est disponible ici.
- Pour une conversion appropriée, le nombre d'images, la taille de l'étape et la résolution de sortie souhaitée devront également être saisis.
- Après avoir rééchantillonné la matrice par avion, importer le nouveau fichier MAT dans le code d'analyse des souches 3D.
4. Analyse 3D du code de contrainte
- Commencez l'analyse en ajustant correctement le fichier MAT importé. Par exemple, le volume d'image peut devoir être redimensionné pour réduire le temps de calcul.
- Entrez la région à analyser et déterminez le modèle de maillage approprié pour segmenter les données d'image sous forme de boîtes simples ou de polygones choisis manuellement. La taille de la boîte des régions et l'espacement entre les points du centre peuvent devoir être modifiés pour chaque jeu de données. Les nombres optimaux choisis pour la taille de la boîte seront autour de l'ordre des pixels de la fonctionnalité en cours de suivi, qui peut être approximative en regardant le nombre de pixels en deux dimensions dans une tranche. L'espacement des boîtes déterminera la résolution des champs de contrainte. Plus de boîtes augmenteront la résolution mais peuvent également augmenter considérablement le temps de calcul.
- Commencez à calculer les Jacobiens et les gradients itérativement dans chacune de ces régions. Une fois la précomputation terminée, appliquez la fonction de déformation.
- Calculer le tenseur de gradient de déformation. Calculez d'abord la souche, puis calculez les eigenvalues et les eigenvectors à l'aide de la méthode d'estimation de la déformation directe.
- Tracez ces résultats dans les plans désirés en utilisant une technique telle que la cartographie des couleurs d'un plan coupé pour représenter le champ de tension au-dessus de votre région d'intérêt.
Résultats
Utilisant la procédure décrite ci-dessus, l'ultrason 4D d'une angiotensine II-induite aorcisme aortique aortique de dissection (AAA) d'une souris a été acquise. Plusieurs boucles vidéo EKV à axe court ont été acquises le long de l'aorte et combinées pour créer des données 4D, comme le montre la figure 1. Ces données ont ensuite été converties en fichier MAT à l'aide d'un code personnalisé, qui a ensuite été analysé dans un code de calcul de la souche 3D à l'aide d'une fonction de déformation. Après avoir optimisé les paramètres du code pour un jeu de données spécifique, une vue représentative à long axe avec les valeurs de contrainte correspondantes a été produite ainsi qu'une parcelle de visualisation de tranches 3D avec une carte de couleur de souche superposée (Figure 2). Cette technique DDE et les données de souche mettent en évidence les variations spatiales hétérogènes de la souche, en particulier lorsqu'un thrombus est présent. Ces résultats peuvent ensuite être corrélés avec la structure du vaisseau pour déterminer la relation entre la déformation in vivo et la composition de l'anévrisme.
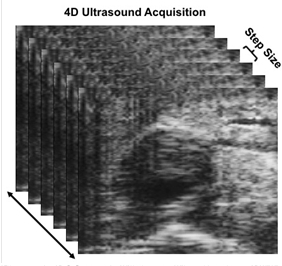
Figure 1 : Les boucles de visualisation kilohertz (EKV) à la porte ECG sont acquises à partir d'emplacements de démarrage et de terminaison entrés manuellement, suivant une taille d'étape de 0,2 mm.
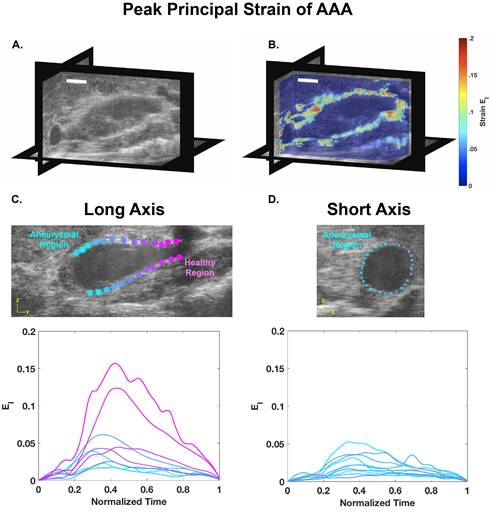
Figure 2 : Données d'échographie à haute fréquence 4D d'un anévrisme aortique abdominal disséquant murine représenté au systole (A) avec les principaux champs de tension estimés et superposés (B) (Scalebar - 5 mm). Vues à axe long et court représentant à la fois des régions anévrismes et des régions saines correspondant à la souche principale sur un cycle cardiaque (systole : t - 0,4) (C, D). Ces données montrent des niveaux de tension relativement élevés dans des régions saines et des valeurs de souche réduites dans l'anévrisme disséquant.
Applications et Résumé
La caractérisation mécanique in vivo localisée est une partie importante de la compréhension de la croissance et du remodelage des tissus biologiques. Par rapport aux approches existantes, la procédure de quantification des souches décrite ici utilise une méthode améliorée de calcul précis de la souche 3D en déviant de façon optimale l'image non déformée avant la corrélation croisée. Cette méthode n'utilise aucune hypothèse matérielle pour déterminer les souches dans les volumes de tissus. Malheureusement, l'estimation de la souche n'est fiable que jusqu'à une taille de noyau de 15x15x15 voxels lors de l'utilisation des données d'ultrason, ce qui suggère que cette approche DDE peut ne pas détecter des caractéristiques subtiles dans un champ de tension. Malgré cette limitation, il demeure un outil important pour étudier les réponses mécaniques, diagnostiquer la pathologie et améliorer les modèles de maladies.
De nombreux domaines de recherche au-delà des anévrismes aortiques peuvent bénéficier de cet outil de mesure de la souche. La souche cardiaque peut également être facilement quantifiée à l'aide de cette méthode. Puisque le myocarde subit la déformation 3D pendant le cycle cardiaque, la tension de quantification dans trois dimensions fait partie intégrante de caractériser de manière fiable la dynamique de ce tissu. Des données fiables sur les souches sont particulièrement importantes pour suivre la progression de la maladie dans les modèles animaux.
L'analyse 3D de la souche peut également être appliquée à l'imagerie par ultrasons intestinaux. La caractérisation mécanique du tissu intestinal est le plus souvent effectuée in vitro. Cependant, ce n'est pas toujours une véritable représentation du comportement réel des intestins in vivo en raison des effets des structures environnantes. Comme exemple de traduire cliniquement cette approche, calculer la souche des images de la fibrose intestinale due à la pression luminale anormale pourrait fournir la détection tôt des secteurs problématiques qui exigent l'intervention chirurgicale.
Au-delà des applications à plus grande échelle, cette méthode peut également être appliquée au niveau cellulaire en utilisant des techniques d'imagerie à plus haute résolution, telles que la microscopie confocale. Caractériser la matrice extracellulaire est important pour comprendre comment les cellules communiquent. Beaucoup de recherches ont été menées sur la caractérisation biochimique, mais la compréhension de la façon dont la communication peut être menée par des réponses mécaniques exige une compréhension de la déformation et de la souche. La souche en vrac n'est pas bénéfique parce qu'il n'y a aucun moyen de déterminer l'origine du changement de déformation. L'application d'une approche DDE à haute résolution pourrait révéler directement comment la matrice extracellulaire réagit aux changements mécaniques.
Remerciements
Nous tenons à remercier John Boyle, Guy Genin et Stavros Thomopoulos pour la contribution du code Matlab personnalisé DDE capable d'estimer directement la souche Lagrange-Green.
Tags
Passer à...
Vidéos de cette collection:

Now Playing
Cartographie quantitative de la déformation d'un anévrisme de l'aorte abdominale
Biomedical Engineering
4.6K Vues

Imagerie d'échantillons biologiques par microscopie optique et confocale
Biomedical Engineering
36.2K Vues

Imagerie d'échantillons biologiques par MEB
Biomedical Engineering
24.0K Vues

Biodistribution des nanomédicaments : Application de la microscopie à balayage électronique
Biomedical Engineering
9.5K Vues

Imagerie par ultrasons à haute fréquence de l'aorte abdominale
Biomedical Engineering
14.8K Vues

Tomographie photoacoustique pour l'imagerie du sang et des lipides dans l'aorte infrarénale
Biomedical Engineering
5.9K Vues

Imagerie par résonance magnétique cardiaque
Biomedical Engineering
15.0K Vues

Simulations numériques de la dynamique des fluides du flux sanguin lors d'un anévrisme cérébral
Biomedical Engineering
11.9K Vues

Imagerie des anévrismes de l'aorte abdominale par fluorescence dans le proche infrarouge
Biomedical Engineering
8.4K Vues

Techniques de mesure non invasive de la pression artérielle
Biomedical Engineering
12.1K Vues

Acquisition et analyse d'un signal ECG (électrocardiographie)
Biomedical Engineering
106.7K Vues

Résistance à la traction des biomatériaux résorbables
Biomedical Engineering
7.7K Vues

Imagerie Micro-CT de la moelle épinière d'une souris
Biomedical Engineering
8.3K Vues

Visualisation de la dégénérescence de l'articulation du genou après une blessure non invasive du LCA chez le rat
Biomedical Engineering
8.3K Vues

Imagerie combinée SPECT et CT pour visualiser la fonctionnalité cardiaque
Biomedical Engineering
11.2K Vues