Conjugaison : méthode de transfert de la résistance à l'ampicilline du donneur à l'hôte E. coli
Vue d'ensemble
Source: Alexander S. Gold1, Tonya M. Colpitts1
1 Département de microbiologie, Boston University School of Medicine, National Emerging Infections Diseases Laboratories, Boston, MA
Découverte pour la première fois par Lederberg et Tatum en 1946, la conjugaison est une forme de transfert horizontal de gènes entre bactéries qui repose sur un contact physique direct entre deux cellules bactériennes (1). Contrairement à d'autres formes de transfert de gènes, comme la transformation ou la transduction, la conjugaison est un processus naturel dans lequel l'ADN est sécrété d'une cellule de donneur à une cellule recevable d'une manière unidirectionnelle. Cette directionnalité et la capacité de ce processus d'augmenter la diversité génétique des bactéries a donné à la conjugaison la réputation comme une forme d'« accouplement » bactérien, qui aurait grandement contribué à l'augmentation récente de la résistance aux antibiotiques. bactéries (2, 3). En utilisant des pressions sélectives, par exemple l'utilisation d'antibiotiques, la conjugaison a été manipulée pour une utilisation en laboratoire, ce qui en fait un outil puissant pour le transfert horizontal de gènes entre les bactéries, et dans certains cas des bactéries à la levure, les plantes et les animaux cellules (4). Outre les applications en laboratoire, le transfert de gène bactérierium-eucaryote par conjugaison est une avenue passionnante de transfert d'ADN avec une multitude d'applications biotechniques possibles et des implications naturelles (5).
On pense que la conjugaison fonctionne par un « mécanisme en deux étapes » (6). Tout d'abord, avant que l'ADN puisse être transféré, la cellule du donneur doit établir un contact direct de cellule à cellule avec le receveur. Ce processus a été caractérisé le mieux dans les bactéries gram-négatives, dont la plus étudiée est Escherichia coli. Le contact cellule-cellule est établi par la présence d'un réseau complexe de filaments extracellulaires sur le donneur connu sous le nom de pilus sexuel, un élément conjugal codé par le gène transférable connu sous le nom de facteur F (fertilité) (7, 8). En plus d'établir le contact entre le donneur et le receveur, plusieurs protéines sont transportées par le pilus sexuel au cytoplasme receveur, formant un conduit de système de sécrétion de type IV (T4SS) entre les deux cellules, une structure nécessaire pour la deuxième étape de conjugaison, transfert d'ADN (6). En combinant cette fonction du pilus sexuel avec la réplication du cercle roulant de l'ADN, la cellule du donneur est capable de transférer l'ADN sous la forme d'un élément transposable, tel qu'un plasmide ou un transposon, au destinataire par un modèle de « tirer et pomper » (6). Dans ce cas, le « tir » est le transport de la protéine pilote, avec l'ADN lié, par le T4SS dans la cellule récepteur, et le « pompage » est le transport actif de l'ADN au destinataire, un processus dépendant du T4SS et catalysé par des protéines de couplage (6). La machinerie utilisée dans ce processus est composée d'une origine de séquence de transfert (oriT), qui doit être fournie par l'ADN dans les gènes cis et trans, qui codent une relaxase, complexe de formation de paire de compagnon, et le type IV protéine de couplage, et peuvent être présents en cis ou trans (9). Cette relaxase fend le site nic dans la séquence oriT et se fixe de façon covalente à l'extrémité 5' du brin transféré pour produire le relaxosome, un complexe d'ADN-relaxase à brin unique avec d'autres protéines auxiliaires (9). Une fois formé, le relaxosome se connecte au complexe de formation de paires d'accouplement, via la protéine de couplage de type IV, qui permet le transfert du complexe ssDNA-relaxase en cellules réceptrices par le T4SS (10). Une fois dans le cytoplasme du receveur, l'ADN peut s'intégrer dans le génome du receveur ou exister séparément sous la forme d'un plasmide, qui permettent l'expression de ses gènes.
Dans cette expérience, la souche de donneur de conjugaison largement utilisée E. coli WM3064 a été utilisée pour transférer l'encodage génétique pour la résistance à l'ampicilline à la souche bénéficiaire E. coli J53. Tandis que les deux souches des bactéries gram-négatives étaient résistantes à la tétracycline, seulement la souche de distributeur WM3064 a eu le gène pour la résistance d'ampicilline, codépour dans le vecteur de navette de pWD2-oriT, et était auxotrophique à l'acide diaminomiélique (DAP) (11-13). Cette expérience consistait en deux étapes principales, la préparation des souches du donneur et du receveur, suivie du transfert du gène de résistance à l'ampicilline du donneur au receveur par conjugaison (figure 1).
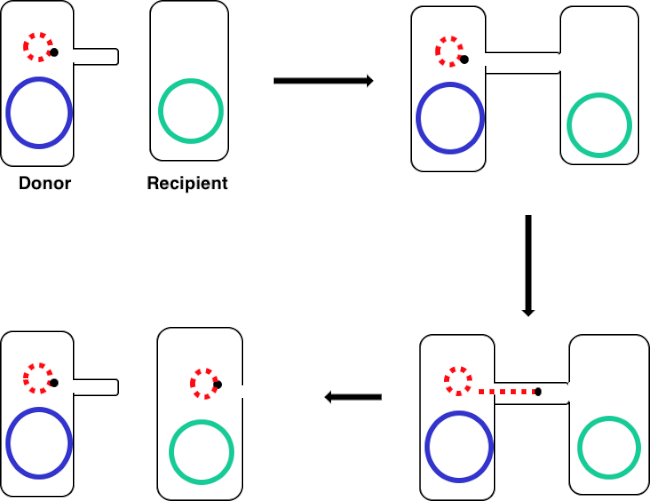
Figure 1 : Schéma de conjugaison. Ce schéma montre le transfert réussi d'un plasmide, un seul exemple d'un élément d'ADN transposable, d'une cellule de donneur à une cellule receveure utilisant la conjugaison. Au contact de la cellule réceptrice par la cellule du donneur par l'intermédiaire du pilus sexuel, le plasmide se reproduit par la réplication du cercle roulant, se déplace à travers le complexe multiprotéiné en joignant les deux cellules, et forme un nouveau plasmide complet dans la cellule réceptrice.
En couvant un mélange de cellules de donneur et de receveur, puis en placageant successivement ces cellules en présence de la tétracycline et du DAP, cela a permis le transfert réussi du gène de résistance à l'ampicilline. Les cellules de placage successivement cultivées à partir de ce mélange en présence de tétracycline et d'ampicilline, enlevé toutes les cellules de donneur en raison de l'absence de DAP et toutes les cellules receveuses qui peuvent ne pas avoir gagné le gène de résistance à l'ampicilline, donnant strictement destinataire J53 souche bactéries qui ont acquis une résistance à l'ampicilline (figure 2). Une fois effectué, le transfert réussi du gène de résistance d'ampicilline a été confirmé par PCR. Depuis que la conjugaison a été réussie, la souche J53 de E. coli a contenu pWD2-oriT et était résistante à l'ampicilline, et l'encodage de gène pour cette résistance est détectable par PCR. Cependant, en cas d'échec, il n'y aurait pas eu de détection du gène de résistance à l'ampicilline et l'ampicilline fonctionnerait toujours comme un antibiotique efficace contre la souche J53.
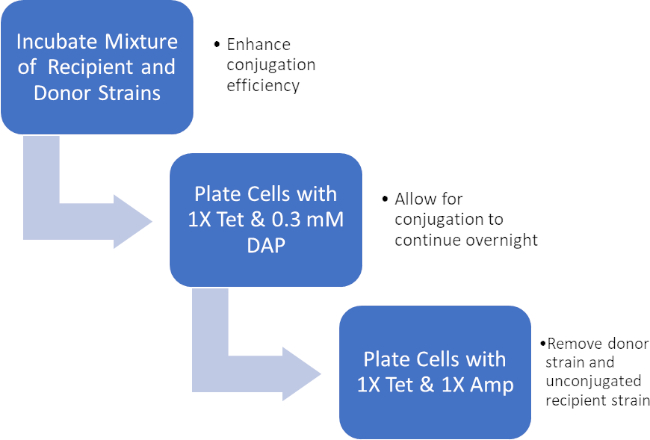
Figure 2 : Schéma de protocole. Ce schéma montre un aperçu du protocole présenté.
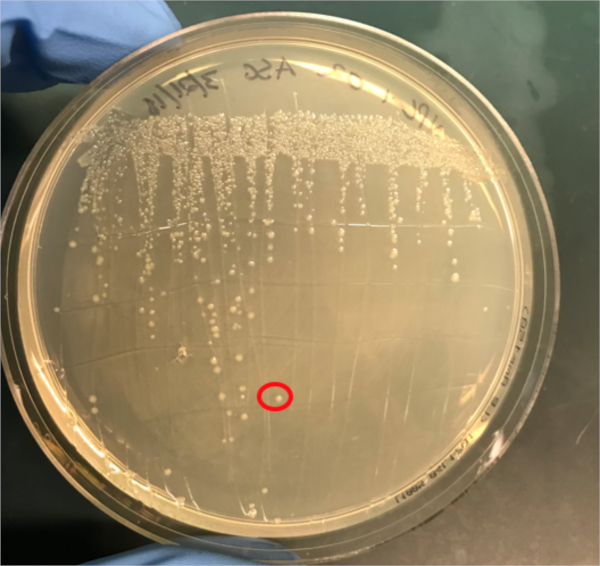
Figure 3A : Confirmation d'une conjugaison réussie par PCR. A) Les stocks de congélation des échantillons témoins conjugués et négatifs ont été striés sur des plaques d'agar et une colonie a été sélectionnée (rouge) pour l'isolement de l'ADN.
Procédure
1. Mise en place
- Autoclave environ 1L du milieu de Luria-Bertani (LB). Ce LB stérile sera utilisé pour fabriquer environ 5 ml de LB contenant 0,3 mM d'acide diaminopiélique (DAP).
- Recueillir les plaques suivantes: plaques d'agar LB avec 1X Tet et 0,3 mM DAP, plaques d'agar LB avec 1X Tet seulement, et plaques d'agar LB avec 1X Amp / Tet seulement.
- Assurez-vous qu'un peu de glycérol et une boîte de pointes de pipette en plastique pré-stérilisée sont à portée de main.
- Avant de commencer tout travail impliquant des microbes, stériliser l'espace de travail avec 70% d'éthanol. Portez toujours l'équipement de protection individuelle nécessaire, y compris une blouse de laboratoire et des gants.
- Une fois terminé, stériliser toutes les surfaces et les gants avec 70% d'éthanol et se laver les mains.
2. Préparation des souches du donneur et du receveur
- Préparer 5 ml de cultures bactériennes des souches du donneur et du receveur et les cultiver pendant la nuit à 37 oC avec l'aération et les secousses à 220 tr/min. La souche de donneur devrait être cultivée en LB avec 0.3 mM DAP.
- Faites tourner 1 ml des deux cultures (3 000 tr/min pendant 5 minutes) et lavez les cellules avec du PBS.
- Resuspendre les cellules dans 500 l de phosphate tamponné saline (PBS).
3. Conjugaison
- Combiner 50 l de cellules réceptrices avec 50 l de cellules donneuses dans un tube de microcentrifuge et mélanger par pipetting.
- Incuber le mélange cellulaire pendant 1 heure à 37 oC. Cela permettra d'améliorer l'efficacité de la conjugaison avant et pendant la prochaine étape de placage.
- Pipet 100 l du mélange cellulaire sur une plaque d'agar avec 1X Tet et 0,3 mM DAP. Ne pas étendre sur la plaque.
- Pipet 100 l de culture cellulaire receveur sur une plaque d'agar avec 1X Tet et 0,3 mM DAP. Ne pas étendre sur la plaque.
- Incuber les deux assiettes pendant la nuit à 37 oC.
- Gratter le mélange des cellules de conjugaison et la culture cellulaire réceptrice avec un grattoir à cellules stériles. Transférer les cellules dans un tube microcentrifuge stérile et les suspendre à 1 ml de PBS.
- Vortex les cellules et les faire tourner doucement vers le bas (3000 tr/min pendant 5 minutes).
- Resuspendre les cellules dans 1 ml de PBS.
- Plaquer le mélange de cellules de réaction de conjugaison sur une plaque d'agar LB avec 1X Amp/Tet seulement. Sur cette plaque, seules les bactéries récepteuses qui ont réussi à obtenir le gène de résistance de l'ampli par conjugaison sont censées se développer.
- Déposer les cellules réceptrices sur une plaque d'agar LB avec 1X Tet seulement. Sur cette plaque, seules les bactéries récepteuses non conjuguées, qui n'ont pas le gène de résistance de l'ampli, devraient se développer.
- Incuber les assiettes toute la nuit à 37 oC.
- Choisissez des colonies simples des deux plaques et utilisez-les pour faire pousser des cultures de nuit dans 5 ml de support (37 oC avec aération à 220 tr/min).
4. Isolement de l'ADN
- Isoler l'ADN des cultures précédemment préparées en utilisant 4,5 ml du volume total de culture par miniprep d'ADN.
- Pour ce faire, éluter l'ADN à l'aide de 35 L d'eau sans nucléane.
- L'ADN pur générera un rapport d'absorption (A260/280) d'environ 1,8.
- Utilisez les 0,5 ml restants de chaque culture pour préparer les stocks de glycérol en faisant un mélange 1:1 de culture bactérienne et 100% de glycérol.
- Conserver les stocks de congélateurs à -80 oC.
5. Confirmation de la conjugaison de transfert de Plasmid par PCR
- Préparer deux mélanges maîtres PCR, chacun avec un ensemble différent d'amorces avant et inverses, l'un ciblant un segment de 500 paires de base au sein du gène de résistance à l'ampicilline et l'autre ciblant un segment au sein d'un gène d'entretien ménager.
- Les amorces de gène d'entretien ménager ont été conçues pour amplifier un segment de l'ADN dans l'encodage de gène bactérien pour le gyrase B d'ADN (14).
- Les volumes de réactifs suivants ont été utilisés pour préparer 90 L de chaque mix master :
7,5 L de 10 m d'apprêt avant
7,5 L de 10 m d'apprêt inversé
75 L de 2X PCR Master Mix
- Préparer les six réactions PCR suivantes à l'aide d'un mix maître de 15 l, de 10 ng d'ADN de modèle et d'eau exempte de nucléane jusqu'à un volume final de 25 l.
Apprêtdedede de réaction de conjugaison et d'ampicilline
Réaction de conjugaison ADN et amorces d'entretien ménager
Amorces d'ADN et d'ampicilline de contrôle négatif
ADN de contrôle négatif et amorces d'entretien ménager
Pas d'amorces d'ADN et d'ampicilline
Pas d'ADN et d'amorces d'entretien ménager - Transférez ces réactions à une machine PCR avec le bloc préchauffé à 98 C et commencez le thermocyclisme dans les conditions suivantes :
98 oC pendant 30 secondes
25-35 cycles de 98 oC pendant 5-10 secondes, 45-72 oC pendant 10 à 30 secondes, et 72 oC pendant 15-30 secondes par kb
72 oC pendant 5-10 minutes
Tenir à 4 oC - Chargez les six réactions PCR sur un gel agarose de 1% et exécutez à 150V pendant environ 20 minutes.
- Visualisez le produit PC à l'aide d'un illuminateur UV.
Résultats
Si la conjugaison a été couronnée de succès, un produit PCR de la taille d'une paire de 500 de base sera observé dans le puits où la réaction PCR 1 a été chargée (bien #2 à la figure 3B), alors qu'aucune bande ne sera observée dans le puits où la réaction PCR 3 a été chargée (bien #4 à la figure 3B). La présence de cette bande confirme le transfert réussi du gène de résistance à l'ampicilline, conférant ainsi une résistance à l'ampicilline à la souche J53 d'E. coli.
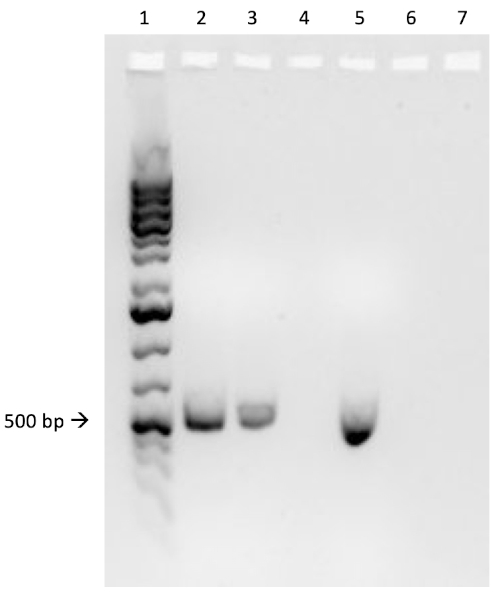
Figure 3B : Confirmation d'une conjugaison réussie par PCR. B) L'analyse de PCR a été faite utilisant l'ADN isolé de la colonie choisie. Le contenu de chaque puits est le suivant : 1) échelle d'ADN, 2) Amorces d'ADN de conjugaison et d'ampicilline, 3) ADN de conjugaison et amorces d'entretien ménager, 4) Amorces de contrôle négatif et amorces d'ampicilline, 5) ADN de contrôle négatif et amorces d'entretien ménager, 6) Aucun ADN et ampicilline amorces, et 7) Pas d'ADN et d'amorces de contrôle négatif. La présence d'un produit PCR de bande de base-paire de 500 de 500 de PCR de la réaction de PCR 1 (bien 2), et l'absence de ce produit de la réaction de PCR 3 (bien 4), confirme la conjugaison réussie.
Applications et Résumé
La conjugaison est un processus naturel de transfert horizontal de gènes qui repose sur le contact direct cellule-cellule d'une cellule donneuse et d'une cellule recevable. Ce processus est partagé entre toutes sortes de bactéries et a joué un rôle déterminant dans l'évolution bactérienne, notamment la résistance aux antibiotiques. En laboratoire, la conjugaison peut être utilisée comme une méthode efficace de transfert de gènes qui est beaucoup moins perturbateur par rapport à d'autres techniques. En dehors du laboratoire, la capacité de transférer l'ADN des bactéries aux eucaryotes par conjugaison offre une nouvelle avenue passionnante de thérapie génique et de compréhension des implications de ces transferts de gènes naturels, par exemple la relation entre l'infection bactérienne et le cancer, est un domaine de recherche qui émerge rapidement.
Tags
Passer à...
Vidéos de cette collection:

Now Playing
Conjugaison : méthode de transfert de la résistance à l'ampicilline du donneur à l'hôte E. coli
Microbiology
38.5K Vues

Création d'une colonne de Winogradsky : une méthode pour enrichir les espèces microbiennes dans un échantillon de sédiments.
Microbiology
130.4K Vues

Dilutions en série et ensemencement des géloses : numération des micro-organismes
Microbiology
317.7K Vues

Cultures enrichies : Cultiver des micro-organismes aérobies et anaérobies dans des milieux sélectifs et différentiels
Microbiology
132.5K Vues

Cultures pures et ensemencement des géloses : isolement des colonies bactériennes pures à partir d'un échantillon mixte
Microbiology
166.8K Vues

Séquençage de l'ARNr 16S : une technique basée sur la PCR pour l'identification d'espèces bactériennes
Microbiology
190.2K Vues

Courbes de croissance : Générer des courbes de croissance en comptant les unités formant colonies (UFC) et en mesurant l'absorbance
Microbiology
298.9K Vues

Tests de sensibilité aux antibiotiques : Utilisation du ETEST pour déterminer la CMI de deux antibiotiques et évaluer la synergie des antibiotiques
Microbiology
94.3K Vues

Microscopie et coloration : Gram, Capsule et endospores.
Microbiology
364.6K Vues

Test de la plaque : méthode de détermination de la charge virale exprimée en unités formant des plaques
Microbiology
186.9K Vues

Transformation des cellules E. coli en utilisant le chlorure de calcium
Microbiology
87.3K Vues

Transduction via bactériophage : méthode de transfert de la résistance à l'ampicilline du donneur au receveur E. coli
Microbiology
29.3K Vues