Method Article
אינטגרציה של Wet ותהליכי ספסל יבשים מבצעת אופטימיזציה ממוקד רצף הדור הבא של איכות נמוכה וביופסיות גידול נמוכה כמות
In This Article
Summary
An integrated system for targeted next-generation sequencing of oncology specimens is described. This cross-platform system is optimized for low-quality and low-quantity tumor biopsies, accommodates low DNA inputs, includes well-characterized multi-variant controls, and features a novel variant caller that is informed by quantitative pre-analytical quality control measures.
Abstract
כל רצף של הדור הבא (NGS) הנהלים כוללים מבחנים בצעו ליד שולחן המעבדה ( "ספסל רטוב") ונתונים ניתוחיים שנעשה בעזרת צינורות ביואינפורמטיקה ( "ספסל יבש"). שני האלמנטים החיוניים כדי להפיק תוצאות מדויקות ואמינות, אשר הן קריטיות במיוחד למעבדות קליניות. טכנולוגיות ממוקדות NGS מצאו לטובה ויותר ביישומי אונקולוגיה שיאפשר לך לעמוד ביעדי רפואת דיוק מראש, עדיין השיטות לעתים קרובות כרוכות מנותק ומשתנים זרימות עבודת ספסל רטובות ויבשות וערכות מגיבות לא מתואמות. בדו"ח זה, אנו מתארים שיטה רצף מאתגר דגימות סרטן עם פאנל 21-גן כדוגמה של מערכת NGS ממוקד מקיף. המערכת משלבת כימות DNA פונקציונלית הסמכה, העשרה חד צינור מרובב PCR, וטיהור ספרייה ונורמליזציה באמצעות מאומת אנליטית, חומרים נגד מקור יחיד עם חבילה ביואינפורמטיקה עצמאית.כתוצאה מכך, חלופה מדויקת שיחות באיכות נמוכה פורמלין-קבוע נמוכה כמות, פרפין מוטבע (FFPE) ושאיפה-מחט דקה (FNA) ביופסיות גידול יכולות להיות מושגת. השיטה יכולה שגרתי להעריך גרסאות סרטן הקשורים מתוך קלט של 400 עותקים DNA amplifiable, והוא מודולרי בעיצוב כדי להכיל תוכן גן חדש. שני סוגים שונים של בקרות אנליטית מוגדרות לספק אבטחת איכות לסייע בהגנת שיחת דיוק עם דגימות קליניות רלוונטיות. צעד PCR גמיש "תג" מטביע מתאמים וקודי מדד ספציפי פלטפורמה כדי לאפשר barcoding מדגם ותאימות עם מכשירי NGS המעבדתיים משותפים. חשוב לציין, הפרוטוקול הוא יעילים יכול לייצר 24 ספריות רצף מוכן ביום אחד. לבסוף, הגישה מקשרת תהליכי ספסל רטובים ויבשים על ידי שילוב תוצאות בקרת איכות מדגם מראש אנליטיים ישירות לתוך הגרסה קורא אלגוריתמים כדי לשפר את דיוק זיהוי מוטציה ולבדל שווא שלילי indetermשיחות inate. שיטת NGS ממוקד זה משתמשת להתקדמויות wetware ותוכנה להשיג מעמיק גבוה, רצף מרובב וניתוח רגישות של דגימות סרטן הטרוגנית עבור יישומים אבחוניים.
Introduction
רפואת Precision מסתמכת על אינדיבידואליזציה של אפשרויות אבחון וטיפול בחולים. ההבטחה של טיפולים מותאמים היא תוצאה ישירה של הבנה משופרת של מסלולי מחלה שיכולה ליידע את ההצמדה של אבחון מולקולרי חקר טיפולים. לדוגמא, שימוש טיפולים מולקולריים במיקוד עלה מ -11% ל -46% מ 2003 עד 2013 1, ותרופות נגד סרטן כגון vemurafenib ו crizotinib הם פינו FDA עם בדיקות אבחון לוויה. עם יכולתה במדויק לשחזר מטרות רצף נמוך שפע פני קבוצות מדגם מרובבות מאוד, רצף של הדור הבא (NGS) התפתח שיטת הבחירה להערכת סטיות גנטיות הקשורות בסרטן וזיהוי מטרות מולקולריות לרפואת דיוק.
ביופסיות הגידולים המוצקות השכיחות ביותר לבדיקה מולקולרית כוללות פורמלין-קבוע, פרפין מוטבע (FFPE) ושאיפה בסדר מחט (FNA) SPECIMens. דגימות אלה טומנת בחובה-כמות נמוכה ו / או חומצות גרעין באיכות נמוכה המאתגרות NGS מדויק שומות 2-5. NGS שיטות המסחרי נוכחיות לניתוח הדגימות אלה מבוססות על טלאים של חומרים כימיים שונים, פרוטוקולים, וכלי אינפורמטיקה המייצגים מטרות נעות של שיפורים מתמשכים. לדוגמא, שינויים כימיים assay ו / או תוכנה התרחשו כל 1-2 חודשים עבור ערכות NGS הנפוצה בשימוש הממוקד ביותר 6. חוסר יציבות זו משקפת חוסר קוהרנטיות בבניית ואימות מערכת NGS מאוחדת לסוגי דגימה מאתגרים, במיוחד עבור בדיקות סרטן, ומניח ניטל מיותר על מעבדות לפתח פרוטוקולים מלוכדים שאינם מותאמים ממדגם ל-תוצאות. ואכן, בסקר שנערך לאחרונה של משתמשי NGS הדגיש את הקשיים של אלה "המשתנים במהירות" טכנולוגיות, יחד עם הדרישות שנקבע, תוכן רפואי בת תביעה, מומחיות ביואינפורמטיקה מושרשת, solidifiאד הליך משולב שניתן ליישם במהירות, זרימות עבודה יעילה ופרוטוקולים פשוטים המאפשרים ההכשרה תוך כדי עבודת 7. במאמר זה, מערכת כוללת NGS ממוקד מתוארת כי כתובות פערים אלה.
המתודולוגיה הציג משלב את כל הצעדים פרוצדורליים לקוחות pre-אנליטי פוסט-אנליטיות הוא ספסלים רטובים ויבשים כדי לשפר את הדיוק, רגישות, והאמינות של כימות יעד וזיהוי עבור NGS של לוקוסי גן קליני רלוונטיים הסרטן. גישה זו מתחילה עם הכימות של "הפונקציונלי" DNA 4 להעריך איכות DNA, להדריך קלט לתוך צעד עשרה PCR, וגם להישמר מפני שיחות חיוביות שגויות שיכול להתעורר מהחקירה של עותקי תבנית נמוכים מאוד. זמנית חד צינור PCR אז מעשירה עבור 46 לוקוסים ב 21 גנים סרטניים באמצעות רק 400 עותקים DNA amplifiable, ואחריו ההתאגדות של רצפים ספציפיים פלטפורמה usin NGSמכשירי רצף משותף שולחן עבודה גרם. ספריות הם מטוהרים באמצעות הליך חרוז מגנטי פשוט לכמת עם assay qPCR רומן, כיול חינם. סוויטה ביואינפורמטיקה עצמאית, הודיעה על ידי תוצאות QC DNA במדגם כדי לשפר את ביצועי קריאה, מספקת ניתוח הרצף הבא NGS. אנו מציגים נתונים באמצעות גישת מערכות זה ממוקד NGS לחשוף מוטציות בסיס החלפה, כניסה / מחיקות (indels), ולהעתיק מספר הגירסות (CNVs) בביופסיה נמוכה באיכות נמוכה כמות גידול כגון FFPE ודוגמאות FNA, וברחו שולט.
Protocol
הערה: פרוטוקול זה מתאר העיבוד בו הזמני של דגימות באמצעות מערכת MiSeq NGS אבל יכול להיות מותאם עבור מכשיר מכונת הגנום האישי (PGM). עבור קלט DNA המינימלי המומלץ של 400 עותקים תבנית amplifiable, assay הוא מסוגל לייצר לפחות כיסוי החציוני 3,000x עבור כל אחד 96 דגימות לכל בטווח NGS, ועומק כיסוי שווה ערך ל -24 דגימות באמצעות PGM על שבב 318. השיטה גם מחייב שימוש של מכשיר PCR בזמן אמת.
כימות 1. DNA פונקציונלית ובקרת איכות (QC)
- ריאגנטים הפשרה: מיקס מאסטר 2x, מערבבי Probe פריימר, מערבבי Probe פריימר עיכוב, 6-carboxy-X-rhodamine (רוקס), ממס, וארבעה סטנדרטים עקומי כיול DNA האנושי גנומית (תקן ה- DNA (50 ng / μl), DNA רגיל (10 ng / μl), תקן ה- DNA (2 ng / μl), ו- DNA רגיל (0.4 ng / μl)) (טבלה 1). וורטקס כל ריאגנטים עבור 10 שניות ו צנטריפוגות במהירות המרבית למשך 10 שניות כדי לאסוף תוכן.שמור את תערובת מאסטר 2x על הקרח.
- הכן כמות מספקת של תערובת אב המספר הכולל של דגימות להיבדק וכולל 10% יותר נפח כדי להימנע ממחסור בשל pipetting. מכינים את תערובת מאסטר בצינור microcentrifuge באמצעות הכרכים הבאים לדגימה: 5 μl מיקס מאסטר 2x, 0.5 μl פריימר Probe Mix, 0.5 μl עיכוב פריימר Probe Mix, 0.05 רוקס μl ו 2.95 הממס μl. וורטקס למשך 10 שניות ו צנטריפוגות במהירות המרבית למשך 10 שניות כדי לאסוף תוכן.
- להוסיף תערובת אמן 9 μl לתוך הבארות של צלחת 96-היטב.
- הוסף 1 μl של תקני ה- DNA בשני עותקים כדי ליצור עקומת כיול. מערבבים על ידי pipetting מעלה ומטה 5 פעמים.
- ודא מדגם חומצות הגרעין הוא מעורב היטב לפני השימוש. הוסף 1 מדגם μl לתערובת מאסטר ומערבבים ידי pipetting למעלה ולמטה 5 פעמים.
- חותם את הצלחת, מערבולת עבור 10 שניות ו צנטריפוגות במהירות המרבית למשך 10 שניות כדי לאסוף תוכן.
- מניחים את הצלחת לתוך מערכת ה- PCR. קצה הוא FAM (כימות תפקודית) וויק (עיכוב תפקודי) גלאי עבור כל דגימה על פי הוראות היצרן. בצעו מחזורי PCR של 10 דקות ב 95 מעלות צלזיוס, ו -40 מחזורים (15 שניות על 95 מעלות צלזיוס, 1 דקות ב 60 מעלות צלזיוס).
- לנתח את הנתונים qPCR על ידי יצירת העלילה רגרסיה ליניארית עבור כל הסטנדרטים DNA משוכפל באמצעות פרוטוקולים תוכנה.
- מגרש את יומן 10 של מספר עותק עבור כל תקן ה- DNA על ציר ה- x לבין הערך q המקביל FAM C על ציר ה- y.
- אשר שהתוצאות מנפילת מדגם חומצות הגרעין בתוך הטווח הדינמי של עקומת כיול תקני ה- DNA, ולאחר מכן לחשב את הריכוז של ה- DNA הידוע מספר עותק "פונקציונליים" או amplifiable לכל μl מהעמדה המתאימה לו על עקומת סטנדרט התייחסות. איור 1 מציג דוגמאות של עקומות כיול שעברו ונכשלו.
- קבע אם הגברה התרחשה כל תגובה על ידי בדיקת הנוכחות של amplicon היעד הלא-האנושי בערוץ VIC.
הערה: כביקורת חיובית, פריימר העיכוב Probe Mix מכיל פריימרים ספציפיים עבור מטרה לא אנושית אקסוגניים ואת התבנית המקבילה. מיקס Probe פריימר העיכוב הוא מרכיב של מיקס מאסטר אשר מתווסף כל תגובה, כולל הבקרה לא-תבנית (NTC). בהיעדרו של מעכב, המוצר PCR עבור היעד שאינם בני אדם תמיד צריך להתגלות בערוץ VIC. Q "מבלי שיבחין" C למדגם בערוץ VIC מעיד על קיומו של מעכבי ה- PCR שעשוי להפיק תועלת לנקות הבא של המדגם לפני עיבוד נוסף.
2. הכנת הספרייה: ג'ין-ספציפי (GS) PCR
- הכן כמות מספקת של תערובת אב המספר הכולל של דגימות להיבדק וכולל 10% יותר נפח כדי להימנע ממחסור בשל pipetting. מכינים את תערובת מאסטר PCR GS בצינור microcentrifuge באמצעות הכרכים הבאים לדגימה: 5 μl מיקס מאסטר הגברה 2x (טבלה 1) ו 1 μl פאן לוח פריימר סרטן (טבלה 1). מערבבים על ידי pipetting מעלה ומטה, מערבולת עבור 10 שניות ו צנטריפוגות במהירות המרבית למשך 10 שניות כדי לאסוף תוכן.
- Aliquot מיקס מאסטר 6 μl GS PCR לתוך הבארות של צלחת 96-היטב. הוסף 4 μl של כל דגימת חומצות גרעין לתוך בארות בודדות. כדי בארות אחרים, להוסיף 4 μl של הפיקוח FFPE (טבלה 1), 4 μl של שליטה רב-Variant (טבלה 1), ו -4 μl מים nuclease חינם למשך NTC פרוצדורליים. לכל תוספת, ומערבבים על ידי pipetting מעלה ומטה 5 פעמים.
- חותם את הצלחת, מערבולת עבור 10 שניות ו צנטריפוגות במהירות המרבית למשך 10 שניות כדי לאסוף תוכן.
- מניחים את הצלחת thermocycler עבור מחזורי PCR הבאים: 5 דקות ב 95 מעלות צלזיוס,2 מחזורים (15 שניות על 95 מעלות צלזיוס, 4 דק 'ב 60 ° C), 23 מחזורים (15 שניות על 95 מעלות צלזיוס, 4 דק' ב 72 ° C), ותוסף סופי של 10 דקות ב 72 מעלות צלזיוס. החזק על 4 מעלות צלזיוס.
הערה: לאחר השלים שלב 2.4, הצלחת תיקרא כמו צלחת PCR GS.
הכנה הספרייה 3.: תגית PCR
- ריאגנטים ההפשרה: מיקס מאסטר אינדקס 2x (טבלה 1) וקודים אינדקס (טבלה 1). וורטקס למשך 10 שניות ו צנטריפוגות במהירות המרבית למשך 10 שניות כדי לאסוף תוכן.
הערה: קודי מדד הם premixed לספק מערך ייחודי של מדדי pairwise (ברקוד) עבור כל דגימה. - צלחת 96-היטב, להוסיף 7.5 μl של מיקס מאסטר אינדקס 2x ו -5.5 μl של קוד מפתח שצוין גם ומערבבים ידי pipetting למעלה ולמטה 5 פעמים.
- בזהירות לפתוח את צלחת ה- PCR GS, ולהוסיף 2 μL GS PCR מוצר לצלחת החדשה עם מיקס מאסטר. מערבבים על ידי pipetting מעלה ומטה 5 פעמים. עבור כל דגימה, להקליט את מזהה לדוגמא ואת קודי אינדקס pairwise המקבילים. חותם את הצלחת, מערבולת עבור 10 שניות ו צנטריפוגות במהירות המרבית למשך 10 שניות כדי לאסוף תוכן.
- מניחים את הצלחת thermocycler ו- PCR במשך 5 דקות ב 95 מעלות צלזיוס, 10 מחזורים (30 שניות על 95 מעלות צלזיוס, 30 שניות על 55 מעלות צלזיוס, 1 דקות ב 72 מעלות צלזיוס), ותוסף סופי של 10 דקות ב 72 מעלות צלזיוס. החזק על 4 מעלות צלזיוס.
הערה: לאחר השלים שלב 3.4, הצלחת תיקרא כמו צלחת PCR תג.
טיהור ספריית 4. בחירת גודל
- הסר את חרוזים מגנטיים טהור הכנה הספרייה (טבלה 1) ו הצפת elution (טבלה 1) 2-8 מעלות צלזיוס ולאפשר לאזן RT למשך 30 דקות. להוסיף 9.6 מ"ל 100% אתנול למאגר לשטוף (טבלה 1) מיכל, כובע ומערבבים על ידי היפוך הבקבוק כמה פעמים.
- וורטקס חרוזים מגנטיים עבור 10 שניות ומוסיפים 11 μl לתוך בארות נפרדות של 96-גם צלחת.
- פתח את צלחת תג PCR ולהוסיף 10 μl של מוצר תג PCR לתערובת חרוזים pipet 5 פעמים. דגירה את התערובת במשך 4 דקות ב RT.
- מניחים את צלחת 96-גם על מעמד מגנטי (טבלה 1) למשך 4 דקות. עם צלחת 96-היטב עדיין על דוכן העדים, להסיר ולסלק את supernatant עם טפטפת.
- הסר את הצלחת 96-היטב מהדוכן המגנטי ולהוסיף 100 המכיל אתנול μl הצפתי לשטוף היטב כל ומערבבים ידי pipetting למעלה ולמטה 5 פעמים. דגירה של 2 דקות.
- מניחים את צלחת 96-גם על מעמד מגנטי למשך 2 דקות, ולאחר מכן הסר וזורקים supernatant עם טפטפת.
- חזור על שלב 4.5, עבור סכום כולל של 2 שוטף אתנול, הסרת פתרון לשטוף כמה שיותר לאחר לשטוף את השני.
- עם צלחת 96-היטב על המעמד המגנטי, לייבש את החרוזים עבור 2 דקות ב RT, ולאחר מכן להסיר את הצלחת מהדוכן.
- Resuspend את החרוזים על ידי הוספת 20 μl הצפת elution לכל well ו pipet מעלה ומטה 5 פעמים.
- דגירה של 2 דקות ב RT.
- מניחים את צלחת 96-גם חזרה על מעמד מגנטי למשך 4 דקות, ובזהירות להסיר ולהעביר 18 μl של supernatant ברור באר חדשה.
הערה: ההליך עשוי להיפסק בבטחה בשלב זה ודוגמאות לאחסן ב -15 עד -30 °. כדי להפעיל מחדש, להפשיר דגימות קפואות על קרח לפני שתמשיך.
5. כימות הספרייה
- ריאגנטים ההפשרה הספרייה קוואנט (LQ): מיקס מאסטר 2x LQ, פריימר LQ / Probe Mix, LQ רגיל, בקרה חיובית LQ, LQ הממס, ו LQ רוקס (טבלה 1). וורטקס למשך 10 שניות ו צנטריפוגות במהירות המרבית למשך 10 שניות כדי לאסוף תוכן.
- שימוש במוצרי ספרייה מטוהר, לבצע דילול סדרה של כל דגימה בודדת LQ ממס.
- הוסף 2 μl (מוצר מטוהרים הספרייה) 198 μl LQ הממס ומערבבים עד ומטה עם pipet 10 פעמים.
- הוסף 2 μl (1: 100 דילול) עד 198 μl LQ הממס ומערבבים עד ומטה עם pipet 10 פעמים.
- הכן כמות מספקת של תערובת אמן LQ עבור המספר הכולל של דגימות להיבדק וכולל 10% יותר נפח כדי להימנע ממחסור בשל pipetting. הכן את מיקס מאסטר LQ בצינור microcentrifuge באמצעות הכרכים הבאים לדגימה: 5 μl 2X LQ מיקס מאסטר, 2 μl LQ פריימר / Probe Mix, 0.5 μl LQ Standard ו- 0.5 μl LQ רוקס. מערבבים על ידי pipetting מעלה ומטה, מערבולת עבור 10 שניות ו צנטריפוגות במהירות המרבית למשך 10 שניות כדי לאסוף תוכן.
- להוסיף תערובת אמן LQ 8 μl אל באר של צלחת 96-היטב אופטית.
- בבארות נפרדות, להוסיף 2 μl ספרייה מדולל, 2 μl LQ הבקרה חיובית 2 μl ממיס LQ (NTC) ומערבב על ידי pipetting מעלה ומטה 5 פעמים. חותם את הצלחת עם סרט דבק אופטי, מערבולת עבור 10 שניות ו צנטריפוגות ב 400 XG במשך 10 שניות כדי לאסוף תוכן.
- קצה הוא FAM וויק detectors עבור כל דגימה. בצע הגברת PCR באמצעות תנאי אופניים של 5 דקות ב 95 מעלות צלזיוס, ו -40 מחזורים (15 שניות על 95 מעלות צלזיוס, 1 דקות ב 60 מעלות צלזיוס).
- קבע את הריכוז של כל דגימה (ננומטר) בשיטת q C ההשוואתית. חישוב ההפרש של LQ הרגילה (VIC C q) הידוע לספרייה הידועה (FAM q C). ריכוז המדגם המדולל (PM) מחושב באמצעות המשוואה הבאה:
[Lib Conc] ננומטר = 12.5 x 2 Δ Cq
אם באמצעות גורם לדילול למעט 10,000, לחשב את היחס בין הגורם לדילול היעד ל -10,000, ולהתרבות גורם זה על פי מבחן התוצאה של המשוואה ב 5.7.
6. נורמליזציה הספרייה איגום לדוגמא
- קבע את הריכוז החציוני (ננומטר), מעבר לכל הדוגמאות (שכל אחת מהן מכילה מדד pairwise ייחודי) כדי להיות ונקווה.
- קבע את volum המדגם הבודדדואר (μl) כדי הבריכה על ידי הכפלת הריכוז החציוני מעבר לכל הדוגמאות ידי 5, אז חלוקת ידי ריכוז הפרט שלה (ננומטר). סיבוב הערך שהתקבל למספר השלם הקרוב ביותר. כרכים עגולים עם ערכים של <2 μl 2 μl, ואמצעי אחסון> 15 μl 15 μl.
- מוסיפים את נפח מנורמל (μl) עבור כל דגימה לצינור microcentrifuge יחיד כדי ליצור את הבריכה המדגם.
- חשבתי את הריכוז החדש עבור כל דגימה באמצעות ערכים שלמים מעוגלים ולהקליט את התוצאות.
- כדי לקבוע את הריכוז של הברכה המדגמת, לחשב את הסכום של כל ריכוזי הפרט ולהקליט את הערך המתקבל (ננומטר).
- לדלל את הבריכה מדגם 1.25 ננומטר באמצעות רצף הממס (טבלה 1).
הערה: ההליך עשוי להיפסק בבטחה בשלב זה ודוגמאות לאחסן ב -15 עד -30 °. כדי להפעיל מחדש, להפשיר דגימות קפואות על קרח לפני שתמשיך.
רצף 7.
- Denatיור הבריכה מדגם בנוכחות v3 בקרה PhiX (טבלה 1) על ידי הוספת את הדברים הבאים: 15 μl של בריכת לדוגמא 1.25 ננומטר, 3 μl של 0.5 ננומטר PhiX ו -2 μl של 1 N NaOH. וורטקס ואחריו בקצרה על ידי צנטריפוגה קצר דגירה במשך 5 דקות ב RT.
- מניח את ברכת המדגם המפוגלת על קרח.
- הוסף 8 μl של הספרייה מפוגל כדי 992 μl של חיץ מראש צונן HT1-היב לצינור microcentrifuge. וורטקס בקצרה לערבב, ואחריו צנטריפוגה קצר לאסוף תוכן. שמור על הקרח.
- להוסיף 600 μl של הספרייה המפוגלת ומדוללת למקם 17 # של המחסנית המגיבה.
- Primers רצף ההפשרה 1 קרא (טבלה 1), Primers רצף קרא אינדקס (טבלה 1), וכן קרא Primers רצף 2 (טבלה 1). צינורות microcentrifuge, בנפרד לדלל 4 Primers רצף μl עם 636 חיץ μl HT1-היב. הערה: חיץ HT1-היב הוא שנינות הניתניםh בערכה מגיב רצף (טבלה 1).
- מערבבים על ידי vortexing למשך 10 שניות ו צנטריפוגות במהירות המרבית למשך 10 שניות כדי לאסוף תוכן. להוסיף 600 μl של פריימרים רצף מדולל 1 קרא למצב # 18 של מחסנית מגיב רצף, 600 μl של פריימרים בדילול מדד קרא למקם 19 #, ו -600 μl של פריימרים רצף בדילול קרא 2 למקם 20 #.
- טען את ריאגנטים על המכשיר NGS (טבלה 1) ורצף פי הוראות היצרן. בצע ריצת רצף מחזור 2 לזווג סוף x 150.
ניתוח 8. נתונים
הערה: תוכנת כלי NGS ממירה תמונות אשכול לקריאות בסיס ועשרות איכות, demultiplexes מדדי pairwise ליצור FASTQ gzip דחוס פרט (* .fastq.gz) קבצים עבור כל דגימה. לפני ניתוח קבצי demultiplexed, הקורא חייב להוריד ולהתקין את התוכנה ביואינפורמטיקה קשורה (לוח1). התוכנה יכולה להיות מותקנת על מחשב Windows כיתת צרכן ואינו דורשת חומרת מחשוב מיוחדת או חיבור לאינטרנט כדי לבצע את ניתוח הנתונים.
- לחץ לחיצה כפולה על סמל שולחן העבודה תוכנה.
- התחבר למערכת באמצעות שם המשתמש והסיסמה שסופק במדריך התוכנה.
- פתח את מרכז השליטה של הפרויקט, ולחץ על "פרויקט חדש".
- שם הפרויקט לספק תיאור פרויקט אופציונאלי. בחר את סוג לוח NGS מיקוד וסוגי כלי NGS. לחץ על "שמור והמשך".
- העלה את קבצי FASTQ הדחוסים עבור קדימה לאחור קורא. אל תעלה את FASTQs "לא המסומן", אשר מכיל קורא שלא הצליח demultiplex. לחץ על "שמור והמשך".
- הזן את מספר עותקי קלט הפונקציונליים להכין כל ספרייה כפי שנקבע על ידי assay הכימות פונקציונלי DNA (ראה שלב 1 לעיל). הוספה ידנית של ערכים או עותק וערכים הדבקה מתוך התפשטותגיליון לטבלת הביאור. לחץ על "שמור והמשך".
- סקור את הספריות המבוארות נטענות לניתוח ולחץ על "שלח ניתוח" ליזום את הניתוח.
- מעקב אחר ההתקדמות של הניתוח מוצג דרך מרכז השליטה של הפרויקט.
הערה: אינדיקצית מעמד ניתוח מלאה מוצגת כאשר התוצאות מוכנות לבדיקה. - סקור את התוצאות נותחו על מדדי מדגם QC כולל כיסוי מוחלט לכל ספרייה, אחוז קורא מסננים חולפים, עומק כיסוי amplicon ואחידות. סקור את הגרסה קוראה לכל ספרייה רצף עם dbSNP, COSMIC, 1,000 הגנום ומקורות אחרים של ביאור ברמה פונקציונלי ואוכלוסייה.
- לייצא את תוצאות גלם טבלאות גיליון אלקטרוניים לסיכום, * קבצי .bam ו * קבצים .vcf עבור אחסון לטווח ארוך או ניתוח המשך בעלי כלי אינפורמטיקה משלימה.
תוצאות
סך של 90 דגימות (74 ייחודיים) המייצג בקרות חיוביות ושליליות, שורות תאים שאפיינו בעבר, וביופסיות גידול FFPE קליניות שיורית הוערך עבור DNA amplifiable, קלטת לתוך העשרה זמנית PCR, מתויג עם מתאמי רצף, המינימרקטים, ונתח יחיד מכשיר המעבדתיים NGS לרוץ (איור 2) שהניב 19.1 M קורא עובר סינון. איגום מדגם equimolar הביא רצף עומק גבוה (3,692x כניסות) וכיסוי אחיד (97.8% של amplicons מכוסה בתוך פי 5 של העומק לקריאה החציוני). חריגים מורכבים ללא תבנית שולטת, DNA אונליין תא אחד עם הגברת מספר עותק גדולה, ו- DNA אחד FFPE כי סומן בשל עיכוב PCR ידי assay QC מראש אנליטי (איור 3). אחידות כיסוי פני 46 amplicons נשמרה באמצעות שלושה מפעילים שונים (איור 4 א), וכן עבור דגימות DNA FFPE באיכות נמוכה שונות ( איור 4B). פקד ה- DNA הגידול FFPE, ניסח מתערובת של דגימות קליניות שיורית להשיג V600E BRAF 5% (לכמת ידי אגל דיגיטלי PCR), דווח כי המוטציה BRAF היעד ב שכיחותם של 3.9, 5.3, ו -6.5% על ידי שלושה מפעילים באמצעות הזנה של 400 עותקים amplifiable (ובכך 20 רק עותקים מוטציה) (איור 4 ב ו "FFPE3" של השולחן הבלעה). יתר על כן, תערובת של 12 תבניות סינטטיות DNA, שכל אחד מהם מייצגת מוטציה "נהג" ידועת בסיס החלפה, חשפה את המוטציות הצפויות בטווח המיועד של 9 - 17% תדירות אלל ממוצע (טבלה 2). דילול של שורת תאי דגימות FFPE DNA עם רחבות מספר עותק הפגין במינון תלות עבור משתני EGFR ו KRAS, בהתאמה (איור 5). חשוב לציין, קלט DNA FFPE יצומצמו כמה כמו 50 עותקים amplifiable או 1.2 ng של DNA בתפזורת תוך שמירה על זיהוי של מוטציות ידועות ללא חיוביות שגויותשיחות (איור 6). תשומות DNA שוכנו על פני טווח של פי 100 עד לפחות 50,000 עותקים amplifiable (לוח 3). בניסויים זה וצדדים קשורים, וריאנט שיחות 22 FFPE ו -20 דגימות FNA דווחו הסכם עם שיטות עצמאיות עם כיסוי מוטציה משותף (לוח 4).
הרגישות וערך מנבא חיובי עבור assay נקבע מניתוח 97 דגימות, כולל FFPE, FNA, טרי קפוא, ותא אונליין DNA, וכן סך של 195 תוצאות רצף. התוצאות הראו 365 שיחות גרסה חיוביות אמיתיות, 4 שיחות שליליות כוזבות, ו 1 שיחה חיובית כוזבת עם רגישות של 98.9% (95% CI: 97.1-99.7%) ו ערך מנבא חיובי (PPV) של 99.7% (95% CI : 98.2-99.99%). ניתוח של indels בוצעו שתי גרסאות EGFR משותף (p.E746_A750delELREA ו p.V769_D770insASV) ב 33 מדגם-ריצות, מפגין רגישות 93.9% (95% CI: 78.4-98.9%) וכן PPV של 100% (95% CI: 86.3-100%) עם גרסאות זוהה על פני טווח של 2.4-84.8%.
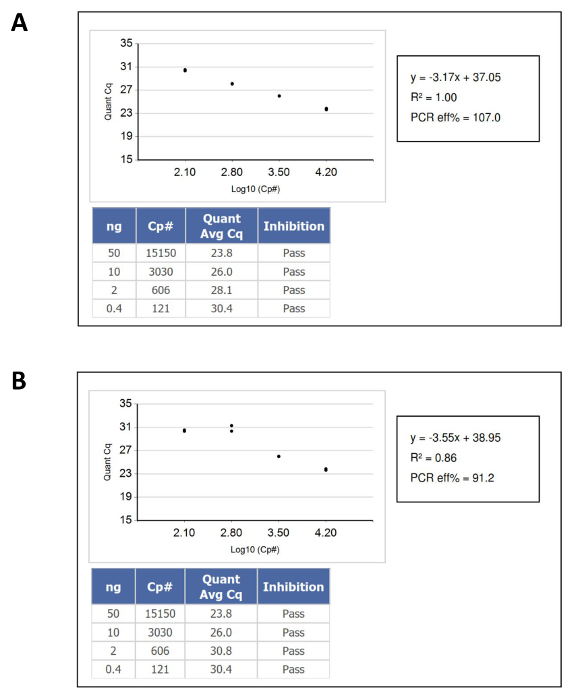
איור 1: דוגמה Curves DNA כימות כיול שעוברים Fail QC קריטריונים (א) עקומת סטנדרט חולף.. (ב) עקום סטנדרט כושל. במקרה זה, הכישלון נגרם על ידי pipetting הכפול של תקן ה- DNA הקלט הנמוך ביותר. נא ללחוץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
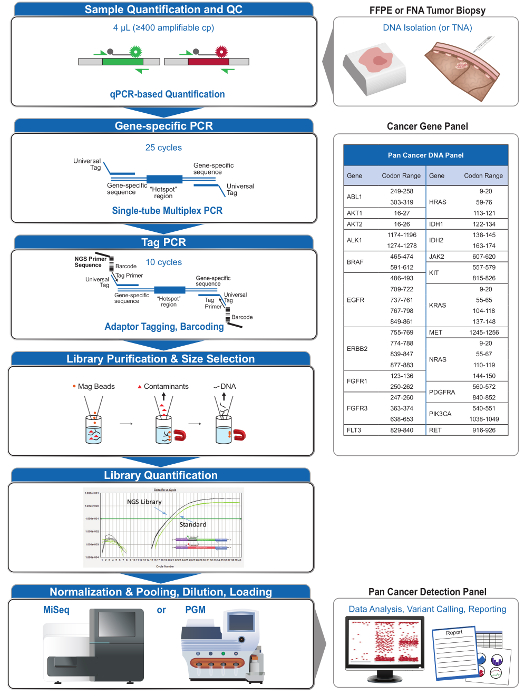
איור 2: מבט כולל של מערכת מקיפה ממוקד NGS עבור יישומים אונקולוגיה המשלב טרום אנליטיתאל, אנליטית, ופוסט-אנליטי זרימות עבודה. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
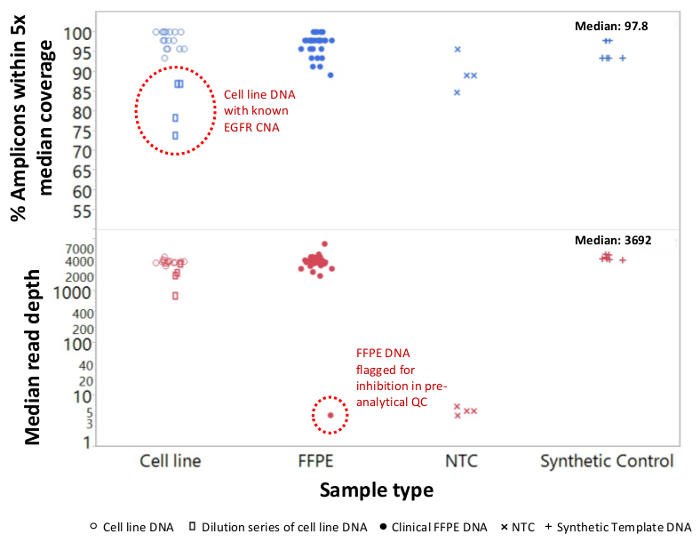
איור 3: סיקור קראו ואחידות עבור NGS הממוקדת של גנים סרטניים בדנ"א FFPE באיכות נמוכה בהשוואה ל- DNA ובקרות הסלולרית אונליין Intact סך של 90 דגימות שכלל FFPE הקליני שיורית (בחוגים סגורים), תא אונליין (עיגולים פתוחים. ), ו- DNA תבנית סינתטי (בתוספת סימנים) עובדו באמצעות יחיד צינור, 21-גן זמנית PCR העשרה. כל ספריית amplicon תויגה עם רצפי מתאם עבור מכשיר NGS, המינימרקטים עם קוד כפול מדד מובהק, מטוהרים, לכמת, מנורמל לריכוז של 2.5 ננומטר. ספריית DNA הייתה רצף ונותחה על ידי התוכנה ביואינפורמטיקה הלוויה. דווי לדוגמאations כלל סדרת דילול של DNA מד"א- MB-468 התאים אונליין ועליו הגברת מספר עותק EGFR גדול (למעלה, מלבנים פתוחים בתוך המעגל המקווקו) שעיוותו אחידות כיסוי מדגם FFPE אחד מלנומה כי הצליחו ליצור מספר ניכר של קורא בשל כדי שריד של מעכבי ה- PCR מתוך מיצוי DNA. הכישלון מדגם מלנומה (למטה, מנוקד מעגל) נחזה על ידי assay DNA QC qPCR מראש אנליטי. NTC, ללא תבנית מלאה (סימני x). נא ללחוץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
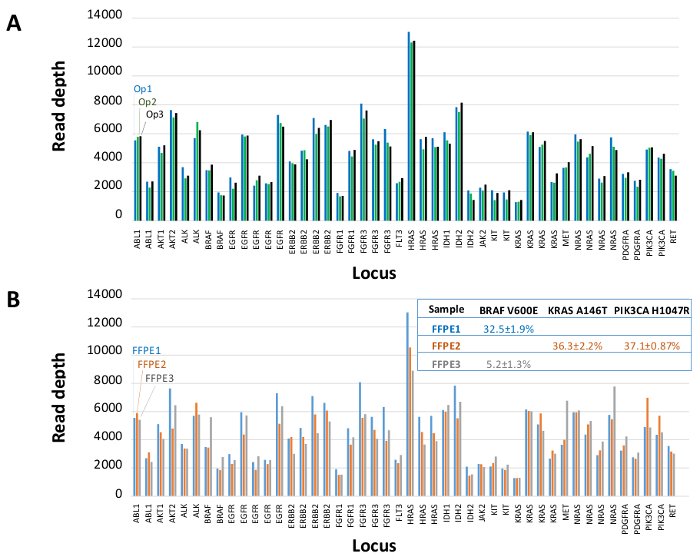
איור 4: Amplicon-ידי-amplicon קראו כיסוי, אחידות וזיהוי Variant בדנ"א גידול הקליני FFPE שיורית. (א) קראו כיסוי בכל הלוקוסים המועשרים דגימת DNA FFPE נציג נמדדועל פני שלושה מפעילים שונים. מפעיל 1, OP1 (סורגים כחולים); מפעיל 2, Op2 (פסים ירוקים); מפעיל 3, Op3 (פסים שחורים). (ב) סיקור אחיד ושיחות גרסה הוערכו באמצעות שלוש דגימות גידולי FFPE, כולל תערובת מלאה (FFPE3, פסים אפורים וטקסט) המורכבים c.1799T BRAF 5% ידועים> מוטציה. FFPE1 (מוטות וטקסט כחולים), FFPE2 (ברי טקסט כתומים). נא ללחוץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
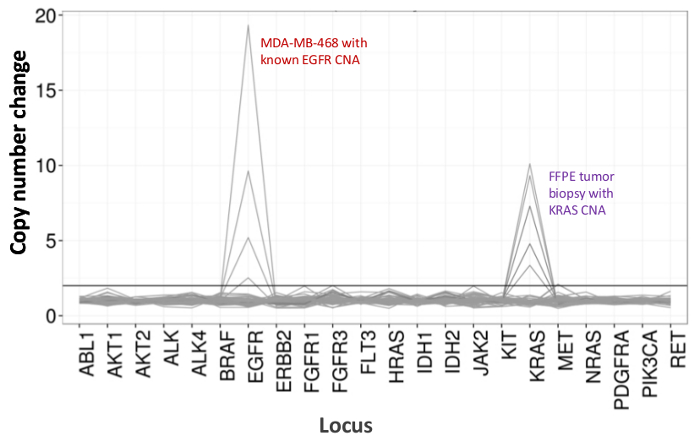
איור 5:. איתור התלוי במינון של וריאנטים המספר העתיקו את שורת תאי FFPE DNA מד"א- MB-468 התאים אונליין DNA עם הגברת מספר עותק EGFR היטב מאופיינת דולל בהדרגת רקע של תאים הלא-מוטצית הפניה DNA -line כדי להמחיש את הירידה שינוי מספר עותק בתורפונקציה של דילול. האחוז של כל דגימת DNA התא אונליין מוצג עם קו ברור (0, 12.5, 25, 50, ו 100%). דילול של מדגם גידול FFPE שחלות עם הגברת KRAS ידועה גילה פרופיל דומה באמצעות אותה הסדרה טיטרציה אבל עם משכפל הדנ"א 100% FFPE עבור שני הקווים העליונים. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
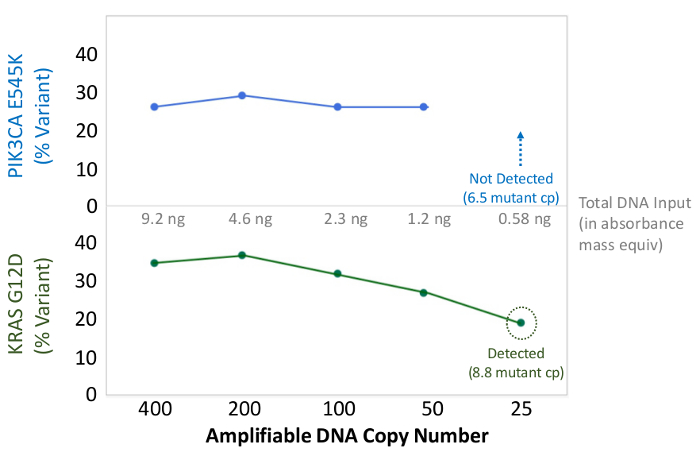
איור 6: מדויק מוטציה איתור וכימות 50 עותקים Amplifiable FFPE DNA, או 1.2 ng DNA גורפת מספר עותק amplifiable של סרטן המעי הגס FFPE DNA נקבע על ידי assay QC מבוססי qPCR, ומדולל מן 400 עד 25 עותקים בתור. לפני רצף קלט לתוך העשרה זמנית PCR. הצינור ביואינפורמטיקה כראוי בשם שתיהן וריאן הידועTS עד 50 עותקים, או שווה הערך של ~ 10 תבניות מוטציה. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
| שם חומר / חומר | חֶברָה | מספר קטלוגי |
| מערבבים 2x Quantidex מאסטר | Asuragen | 145,345 |
| קוואנט פריימר Probe Mix | Asuragen | 145,336 |
| עיכוב פריימר Probe Mix | Asuragen | 145,344 |
| רוקס | Asuragen | 145,346 |
| diluent | Asuragen | 145,339 |
| תקן ה- DNA (50) | Asuragen | 145,340 |
| תקן ה- DNA (10) | Asuragen | 145,341 |
| DNA תקן (2) | Asuragen | 145,342 |
| תקן ה- DNA (0.4) | Asuragen | 145,343 |
| מיקס מאסטר הגברת 2x | Asuragen | 145,348 |
| לוח פאן פריימר הסרטן | Asuragen | 145,347 |
| בקרה פאן סרטן FFPE | Asuragen | 145,349 |
| בקרה רב-Variant סרטן פאן | Asuragen | 145,350 |
| חרוזי הכנה טהורים ספרייה | Asuragen | 145,351 |
| מאגר Wash | Asuragen | 145,352 |
| הצפת elution | Asuragen | 145,353 |
| מערבבים 2x LQ מאסטר | Asuragen | 145,358 |
| LQ הממס | Asuragen | 145,354 |
| LQ חיובילִשְׁלוֹט | Asuragen | 145,355 |
| תקן LQ | Asuragen | 145,356 |
| LQ פריימר / Probe Mix (ILMN) | Asuragen | 145,357 |
| LQ רוקס | Asuragen | 145,359 |
| קודי אינדקס (ILMN) - קבוצה א ' | Asuragen | 150,004 |
| AIL001 - AIL048 (48) | ||
| קודי אינדקס (ILMN) - קבוצה ב ' | Asuragen | 150,005 |
| AIL049 - AIL096 (48) | ||
| מערבבים 2x אינדקס מאסטר | Asuragen | 145,361 |
| קרא 1 Primers רצף | Asuragen | 150,001 |
| Primers רצף קרא אינדקס | Asuragen | 150,002 |
| קרא 2 Primers רצף | Asuragen | 150,003 |
| רצף ממס | Asuragen | 145,365 |
| Illumina MiSeq | Illumina | |
| v3 קיט מגיב MiSeq (600-מחזור) | Illumina | MS-102-3003 |
| v2 ערכת ננו מגיב MiSeq (300-מחזור) | Illumina | MS-103-1001 |
| PhiX בקרה v3 | Illumina | FC-110-3001 |
| Stand-96 המגנטי (או מכשיר מקביל) | Ambion | AM10027 |
| תוכנת Reporter Quantidex | Asuragen |
טבלה 1:. ריאגנטים ערכות עם השימוש הראשון של רוקס, לאחסן את הבקבוקון ב 2-8 מעלות צלזיוס. אל תקפיאו. התוכנה ניתן להוריד בכתובת www.asuragen.com.
| Gפנטן | גרסת COSMIC | חומצת אמינו COSMIC | Variant% |
| NRAS | c.182A> G | p.Q61R | 13.3 |
| NRAS | c.35G> | p.G12D | 15.2 |
| HRAS | c.182A> G | p.Q61R | 17.8 |
| HRAS | c.35G> | p.G12D | 9.2 |
| KRAS | c.182A> G | p.Q61R | 13.5 |
| KRAS | c.35G> | p.G12D | 19.1 |
| PIK3CA | c.1633G> | p.E545K | 9.3 |
| PIK3CA | c.3140A> G | p.H1047R | 9.1 |
| קִיט | c.2447A> T | p.D816V | 14.6 |
| EGFR | c.2369C> T | p.T790M | 11.3 |
| EGFR | c.2573T> G | p.L858R | 14.9 |
| BRAF | c.1799T> | p.V600E | 17.3 |
טבלה 2: פקד סינטטי משולב מורכב 12 "נהג" וריאנטים גנטיים לסרטן כי הם לכמת ב 9-17% שפע תערובת של 12 תבניות סינטטיות שונות פעמים תקועות נושאת 12 מוטציות נפרדות הוערכה הבאה רצף.. כל הגרסאות נקראו באופן תקין וללא תוצאות חיוביות שגויות.
| מזהה לדוגמא | פוּנקצִיוֹנָלִי CPS | גֵן | גרסת COSMIC | חומצת אמינו COSMIC | Variant% | עומק לקריאת חציון | % בתוך 5x של חציון |
| BCPAP | 400 | BRAF | c.1799T> | p.V600E | 99.5 | 3289 | 96% |
| BCPAP | 10,000 | BRAF | c.1799T> | p.V600E | 99.7 | 4040 | 98% |
| BCPAP | 25,000 | BRAF | c.1799T> | p.V600E | 99.4 | 3687 | 96% |
| BCPAP | 50,000 | BRAF | c.1799T> | p.V600E | 99.7 | 4611 | 93% |
טבלה 3: סיקור Calling Variant נשמרים במשך> פי 100 טווח מחירי תשומה DNA. DNA Amplifiable משורת תאים BCPAP היה קלט לתוך העשרה זמנית PCR ב 400 50,000 עותקים להפיק ולרצף. עומק קרא, אחידות כיסוי, זיהוי המוטציה, ודיוק מוטציה השתמרו על פני טווח קלט.
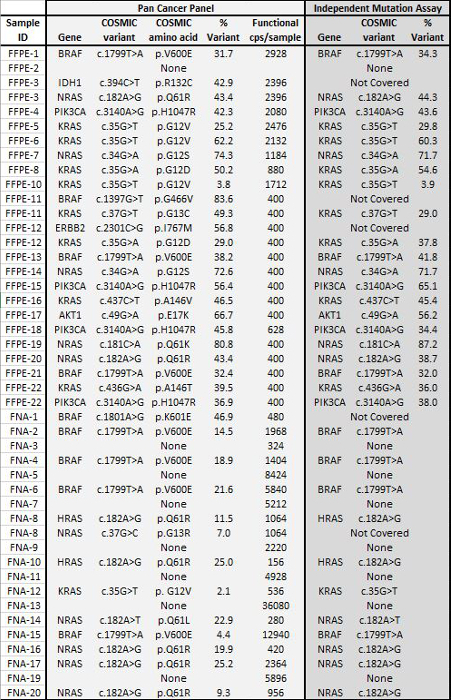
לוח 4: Variant שיחות ב -22 FFPE ו -20 FNA ביופסיות גידול מסכים עם תוצאות עצמאי מוטציה מבחני סט של 22 FFPE הגידול DNA עם מעמד מוטציה נקבע בעבר על ידי מבחני NGS ממוקד מאונך היה קלט ב 400 עד 2,928 עותקים amplifiable לתוך העשרה PCR. צעד להפיק ולרצף באמצעות לוח סרטן 21-גן פאן. בנוסף, קבוצה של 20 דגימות DNA FNA מאופיינת בעבר באמצעות assay מוטצית מערך חרוז נוזל 8 היו PCR מוגבר באמצעות 156 ל 36,080 עותקי קלט amplifiable להפיק ולרצף. כל השיחות החופפות בין פנל NGS סרטן פאן ואת refereNCE שיטות היו בהסכמה. אנא לחצו כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
Discussion
טכנולוגיות NGS הגדירו מחדש ציפיות לחקור את הפרופילים המולקולריים של ביופסיות גידול במסגרות קליניות 9. מספר לוחות NGS ממוקד פותחו טכנולוגיות מחקר, בדיקות שפותחה במעבדה, ומוצרים זמינים מסחרית ופאנלים מנהג להערכת סוגים שונים של דגימות קליניות 3,5,10-15. מדיווחי מחקרים רבים הוכיחו את הערך של NGS ככלי קליני רגיש וספציפי לצורך זיהוי של שינויים גנומיים 3,10-12,14. עם זאת מחקרים הוכיחו גם את הסיכון של חפצים אשר יכול לגרום לתוצאות חיוביות שגויות ביופסיות החולים בסרטן קשות כגון FFPE דגימות 2-5,12,14. בנוסף, פרסומים אחרונים הדגישו שיעורי כישלון גבוהים עבור NGS באמצעות אונקולוגיה דגימות 16 ושיחות חיוביות שגויים עם לוחות מסחריים ממוקדים NGS כי הן מחמירות על ידי השימוש DNA נמוכה קלט 12. כתוצאה מכך, כמה laboraטורים או שינית או הוספת בדיקות נוספות ויתרות לטכנולוגיות NGS ממוקדות זמינות מסחרי כדי לשפר את ביצועים, לעתים קרובות כדי להבטיח דיוק או לאשר את הממצאים מן bioinformatic מנתח 5,10,11.
פנל 21-הגן (האיור 2) פותח כתוכן ממוקד עבור מערכת NGS מקיפה לחקור מבוסס-ראיות, מוטציות תביעה בסוגי דגימה מאתגרות כגון FFPE וביופסיות גידול FNA. זרימת העבודה יש מספר היתרונות: 1) עקבי על ידי מתן כיסוי amplicon אחיד (איורים 3 ו -4, טבלה 3); 2) וקלות לשימוש על ידי מתן טרום ניסח, ערכות מגיב אופטימיזציה, ולפשט את דרישות ביואינפורמטיקה; 3) יעיל על ידי ייעול העבודה והפחתת מספר צעדי pipetting לעומת שיטות NGS המסחרי אחרות; דיוק ו -4) על ידי שילוב של assay QC DNA להערכת amplifiמספר עותק דנ"א מסוגל שמנווט תבנית מקובל למנוע תנודות סטוכסטיים זיהוי גרסה 4. השילוב של נתוני QC מראש אנליטי עם ניתוח ביואינפורמטיקה מאפשר לתשומות נמוכות של דנ"א FFPE. זו הושגה על ידי אימון אלגוריתם עץ החלטות באמצעות עותקים פונקציונליים שונים של קלט DNA על פני 400 דגימות FFPE באמצעים עצמאיים של אמת, ושילוב אלגוריתם זה לתוך תוכנת bioinformatic. כתוצאה מכך, הקלט המומלץ של 400 עותקים amplifiable, בדרך כלל שווים ערך ל ~ 5-20 ng של DNA FFPE, משווה לטובה לשיטות אחרות 10-12, כולל העשרת הכלאה המבוססת שבו ~ 250 ng של DNA FFPE מומלץ 17,18 . למרות הטכניקה מתוארת לשימוש בפלטפורמת MiSeq, זה יכול להיות שונה באמצעות פריימרים PCR תג עם מתאמים ספציפי מכשיר, שנועד לאפשר ניתוח רצף בפלטפורמות NGS אחרים.
מספר צעדים חיוניים כדי להבטיח הצלחהשל ההליך. את assay QC מראש אנליטיים מקובע את מספר עותק amplifiable של עיכוב תפקודי DNA ודוחות. עם זאת, אם פחות מ -400 עותקי DNA amplifiable משמשים צעד עשרה PCR, קיים סיכון מוגבר שיחת false-negative ממדגמים עם מוטציות נמוך שפע (איור 6). בנוסף, יש להקפיד במהלך טיהור ספרייה כדי למנוע יתר ייבוש של החרוזים המגנטיים במהלך שלבי כביסה או elution. יתר על כן, כימות ספרייה מוצלחות תלויות מאוד על הדילול המדויק של ה- DNA ספרייה. לקבלת התוצאה הטובה ביותר, את ההבדל של qPCR תוצאות עבור הספריה מדגם (FAM CQ) לעומת התקן LQ (CQ VIC) צריך להיות ≤3.3 Cq. אם ההבדל הוא גדול מ 3.3 CQ, מחדש דילול בדיקת הדגימה מומלץ. למרות קורלציה מצוינת נצפתה בין שיטת qPCR התחרותית זה וערכות מסחריות המציעות כימות מוחלטות באמצעות עקומת סטנדרט, בקיזוז קלט הספרייה לתוך ביחס צעד הגברה המשובטים לשיטות אחרות עשוי להיות נחוץ כדי להשיג צפיפות זריעה אופטימלית.
כמה דגימות סרטן במיוחד הם מאתגרים שממפה בגלל מעכבים כי נמשכים לאחר בידוד DNA. כדי לזהות דגימות אלה לפני הכנת ספרייה, assay QC qPCR גם מזהה עיכוב הגברה ידי הכללת תבנית אקסוגניים המשמשת הוא בקרה פנימית לבין זקיף עבור עיכוב תפקודי. דוגמה מוצגת באיור 3 שבו דגימת DNA מלנומה לא עברה את עיכוב QC מטרי לפני רצף ולאחר מכן הצליחו ליצור ספריה שיכול להיות רצף. הכישלון היה צפוי תוצאה של זיהום מלנין, מעכב PCR ידוע, נשא משלב בידוד DNA FFPE. דוגמאות המזוהות על ידי assay QC להיות בסיכון לכישלון הגברה ניתן להציל באמצעות t צעד לנקות נוסףo להסיר מעכבים פוטנציאליים.
פנל 21-הגן הממוקד מתמקד בנקודות חמות גן מבוסס ראיות מספק מערכת שלמה עם ריאגנטים ובקרות אופטימיזציה עבור DNA QC, תוכנת NGS וביואינפורמטיקה כי הוא הודיע על ידי מראש אנליטיים תוצאות כימות DNA "פונקציונליות". השיטה במדויק מזהה מוטציות בסיס-החלפת indels מ- DNA נמוך קלט, ומספקת דוגמא מערכת NGS עם האפשרות להרחיב את תכני פנל, כדי לזהות גרסות נוספות כגון CNVs ולהיות מותאמים רצף RNA ממוקד.
Disclosures
JH, AH, RZ, BCH, ואת GJL הם עובדים ויש להם בעלות המניות Asuragen, Inc. RZ, BCH, ואת GJL הם-ממציאים שיתוף על בקשת פטנט לשיפור גרסה מתקשר שימוש במידע מספר עותק amplifiable נקבע עבור כל דגימה.
Acknowledgements
אנו מודים לד"ר אנט שלאגטר לבדיקה של כתב היד. עבודה זו נתמכה בחלקה על ידי CP120017 מענק מטעם למניעת סרטן מכון המחקר של טקסס (PI: GJL).
Materials
| Name | Company | Catalog Number | Comments |
| 2x Quantidex Master Mix | Asuragen | 145345 | |
| Quant Primer Probe Mix | Asuragen | 145336 | |
| Inhibition Primer Probe Mix | Asuragen | 145344 | |
| ROX | Asuragen | 145346 | |
| Diluent | Asuragen | 145339 | |
| DNA Standard (50) | Asuragen | 145340 | |
| DNA Standard (10) | Asuragen | 145341 | |
| DNA Standard (2) | Asuragen | 145342 | |
| DNA Standard (0.4) | Asuragen | 145343 | |
| 2X Amplification Master Mix | Asuragen | 145348 | |
| Pan Cancer Primer Panel | Asuragen | 145347 | |
| Pan Cancer FFPE Control | Asuragen | 145349 | |
| Pan Cancer Multi-Variant Control | Asuragen | 145350 | |
| Library Pure Prep Beads | Asuragen | 145351 | |
| Wash Buffer | Asuragen | 145352 | |
| Elution Buffer | Asuragen | 145353 | |
| 2X LQ Master Mix | Asuragen | 145358 | |
| LQ Diluent | Asuragen | 145354 | |
| LQ Positive Control | Asuragen | 145355 | |
| LQ Standard | Asuragen | 145356 | |
| LQ Primer/Probe Mix (ILMN) | Asuragen | 145357 | |
| LQ ROX | Asuragen | 145359 | |
| Index Codes (ILMN) - Set A, AIL001 - AIL048 (48) | Asuragen | 150004 | |
| Index Codes (ILMN) - Set B, AIL049 - AIL096(48) | Asuragen | 150005 | |
| 2x Index Master Mix | Asuragen | 145361 | |
| Read 1 Sequencing Primers | Asuragen | 150001 | |
| Index Read Sequencing Primers | Asuragen | 150002 | |
| Read 2 Sequencing Primers | Asuragen | 150003 | |
| Sequencing Diluent | Asuragen | 145365 | |
| Illumina MiSeq | Illumina | ||
| MiSeq Reagent Kit v3 (600-cycle) | Illumina | MS-102-3003 | |
| MiSeq Reagent Nano Kit v2 (300-cycle) | Illumina | MS-103-1001 | |
| PhiX Control v3 | Illumina | FC-110-3001 | |
| Magnetic Stand-96 (Or equivalent device) | Ambion | AM10027 | |
| Quantidex Reporter Software | Asuragen |
References
- Chen, G., Mosier, S., Gocke, C. D., Lin, M. T., Eshleman, J. R. Cytosine deamination is a major cause of baseline noise in next-generation sequencing. Mol Diagn Ther. 18 (5), 587-593 (2014).
- Choudhary, A., et al. Evaluation of an integrated clinical workflow for targeted next-generation sequencing of low-quality tumor DNA using a 51-gene enrichment panel. BMC Med Genomics. 7, 62 (2014).
- Sah, S., et al. Functional DNA quantification guides accurate next-generation sequencing mutation detection in formalin-fixed, paraffin-embedded tumor biopsies. Genome Med. 5 (8), 77 (2013).
- Zhang, L., et al. Profiling cancer gene mutations in clinical formalin-fixed, paraffin-embedded colorectal tumor specimens using targeted next-generation sequencing. Oncologist. 19 (4), 336-343 (2014).
- Latham, G. J. Next-generation sequencing of formalin-fixed, paraffin-embedded tumor biopsies: navigating the perils of old and new technology to advance cancer diagnosis. Expert Rev Mol Diagn. 13 (8), 769-772 (2013).
- Crawford, J. M., et al. The business of genomic testing: a survey of early adopters. Genet Med. 16 (12), 954-961 (2014).
- Smith, D. L., et al. A multiplex technology platform for the rapid analysis of clinically actionable genetic alterations and validation for BRAF p.V600E detection in 1549 cytologic and histologic specimens. Arch Pathol Lab Med. 138 (3), 371-378 (2014).
- Thomas, F., Desmedt, C., Aftimos, P., Awada, A. Impact of tumor sequencing on the use of anticancer drugs. Curr Opin Oncol. 26 (3), 347-356 (2014).
- Singh, R. R., et al. Clinical validation of a next-generation sequencing screen for mutational hotspots in 46 cancer-related genes. J Mol Diagn. 15 (5), 607-622 (2013).
- Beadling, C., et al. Combining highly multiplexed PCR with semiconductor-based sequencing for rapid cancer genotyping. J Mol Diagn. 15 (2), 171-176 (2013).
- McCall, C. M., et al. False positives in multiplex PCR-based next-generation sequencing have unique signatures. J Mol Diagn. 16 (5), 541-549 (2014).
- Schleifman, E. B., et al. Next generation MUT-MAP, a high-sensitivity high-throughput microfluidics chip-based mutation analysis panel. PLoS One. 9 (3), e90761 (2014).
- Wong, S. Q., et al. Sequence artefacts in a prospective series of formalin-fixed tumours tested for mutations in hotspot regions by massively parallel sequencing. BMC Med Genomics. 7, 23 (2014).
- Narayan, A., et al. Ultrasensitive measurement of hotspot mutations in tumor DNA in blood using error-suppressed multiplexed deep sequencing. Cancer Res. 72 (14), 3492-3498 (2012).
- Hagemann, I. S., et al. Clinical next-generation sequencing in patients with non-small cell lung cancer. Cancer. 121 (4), 631-639 (2015).
- Won, H. H., Scott, S. N., Brannon, A. R., Shah, R. H., Berger, M. F. Detecting somatic genetic alterations in tumor specimens by exon capture and massively parallel sequencing. J Vis Exp. (80), e50710 (2013).
- Simen, B. B., et al. Validation of a next-generation-sequencing cancer panel for use in the clinical laboratory. Arch Pathol Lab Med. 139 (4), 508-517 (2015).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved