Method Article
낮은 품질과 낮은 수량 종양 생검의 습식 및 건식 벤치 프로세스의 통합을 대상으로 최적화하여 차세대 시퀀싱
요약
An integrated system for targeted next-generation sequencing of oncology specimens is described. This cross-platform system is optimized for low-quality and low-quantity tumor biopsies, accommodates low DNA inputs, includes well-characterized multi-variant controls, and features a novel variant caller that is informed by quantitative pre-analytical quality control measures.
초록
모든 차세대 시퀀싱 (NGS) 절차는 생물 정보학 파이프 라인 ( "건조 벤치")를 사용하여 수행 분석 ( "젖은 벤치")와 데이터를 실험실 벤치에서 수행 분석을 포함한다. 이 두 요소는 임상 실험실에 특히 중요 정확하고 신뢰할 수있는 결과를 생산하는 것이 필수적이다. 대상 NGS 기술은 점점 사전 정밀 의학 목표를 도와 종양학 응용 프로그램에서 은혜를 발견, 아직 방법은 자주 끊어과 습식 및 건식 벤치 워크 플로우 및 조정되지 않은 시약 세트 변수를 포함한다. 이보고에서는, 서열이 광범위한 표적 NGS 시스템의 일례로서 21 유전자 패널 암 시료를 도전하기위한 방법을 설명한다. 이 시스템은 독립형 생물 정보학 제품군과 분석적 검증, 단일 소스 시약을 사용하여 기능 DNA 정량 및 자격, 단일 튜브 멀티 플렉스 PCR 농축 및 라이브러리 정화 및 정상화를 통합합니다.그 결과, 정확한 변형에서 통화 품질이 낮은 저 량 포르말린 고정 파라핀 포매 (FFPE) 및 미세 침 흡인 (FNA) 종양 생검을 달성 할 수있다. 상기 방법은 통상적으로 400 증폭 DNA 카피의 입력에서 암 - 관련 변형을 평가할 수 있고, 새로운 유전자 콘텐츠를 수용하는 모듈 식으로 설계이다. 분석적으로 정의 컨트롤의 두 가지 종류가 임상 적으로 관련 샘플 품질 보증 및 도움 보전과 전화 정확도를 제공한다. 유연한 "태그"PCR 단계는 일반적으로 벤치 탑 NGS 장비 샘플 바코드와의 호환성을 할 수 있도록 플랫폼 별 어댑터와 인덱스 코드를 포함합니다. 중요한 것은,이 프로토콜은 유선형이며, 하루에 24 서열 준비 라이브러리를 생산할 수있다. 마지막으로, 방법은 돌연변이 검출 정밀도를 향상시키고 위음성 및 indeterm 차별화 알고리즘을 호출 변형에 직접 미리 분석 샘플의 품질 관리 결과를 통합하여 건식 및 습식 벤치 처리 링크가하는 통화. 이 타겟 NGS 방법은 그리고 wetware 높은 깊이 다중화 시퀀싱 및 진단 애플리케이션 이종 암 샘플 민감한 분석을 달성하기 위해 소프트웨어 모두의 진보를 이용한다.
서문
정밀 의학은 환자에 대한 진단 및 치료 옵션의 개별화에 의존한다. 맞춤형 치료의 약속은 분자 진단 및 표적 치료제의 링크를 알릴 수 질병 경로의 개선 된 이해의 직접적인 결과이다. 예를 들어, 분자 표적 치료의 사용을 2013 년 1 2003 년 46 %로 11 % 증가하고, vemurafenib 및 crizotinib와 같은 항암 약물 동반자 진단 테스트와 FDA-삭제됩니다. 정확하게 매우 다중화 된 샘플 세트를 통해 미량 시퀀스 타겟을 복구하는 능력과 함께, 차세대 시퀀싱 (NGS)는 암과 관련된 유전자의 수차를 평가하는 정밀 의학 분자 표적을 식별하기위한 선택의 방법으로 떠오르고있다.
분자 테스트를위한 가장 일반적인 고체 종양 생검은 포르말린 고정 포함, 파라핀 (FFPE) 및 미세 침 흡인 (FNA) specimENS. 이 샘플은 낮은 수량 및 / 또는 낮은 품질의 정확한 NGS는 2-5 발생 평가에 도전 핵산을 내포한다. 이들 시료의 분석을위한 현재 상업적 NGS 방법은 다른 시약, 프로토콜, 지속적인 개선의 움직이는 대상을 나타내는 정보학 도구의 패치 워크에 기초한다. 예를 들어, 분석 화학 및 / 또는 소프트웨어의 변경은 가장 일반적으로 사용되는 타겟 NGS 키트 6 매 1-2개월 일어났다. 이 불안정성 구성 특히 암 테스트 도전 표본 유형 통일 NGS 시스템 확인의 일관성 결여를 반영하고, 시료 대 결과에서 최적화 응집 프로토콜을 개발 실험실에 과도한 부담을 둔다. 사실, NGS의 사용자 중 한 최근 조사에 설립, 의료 - 실행 가능한 내용, 확고한 생물 정보학 전문 지식을하는 solidifi에 대한 요구 사항과 함께,이 "빠르게 변화하는"기술의 어려움을 강조온 - 더 - 직업 훈련 (7) 촉진 에드 빠르게 구현 될 수 통합 절차 및 간소화 된 워크 플로우를 간소화 프로토콜. 이 기사에서는 대상 NGS를위한 포괄적 인 시스템은 이러한 격차를 해결하는 기술되어있다.
제시된 방법은 임상 적으로 관련 암 유전자 좌위의 NGS 할 대상 정량 검출 정밀도, 감도, 및 신뢰성을 향상시키기 위해 건식 및 습식 모두 벤치에서 사전 분석에 게시 및 분석 모든 절차 단계를 통합한다. 이 방법은 DNA의 품질을 평가하는 매우 낮은 템플릿 복사 심문에서 발생할 수 위양성 호출에 대해 PCR 농축 단계에 입력하고, 가드 안내 "기능"DNA (4)의 정량화로 시작한다. 단일 튜브 멀티 플렉스 PCR은 NGS의 날기위한 플랫폼 별 서열의 혼입 한 후, 400 증폭 DNA 복사본을 사용하여 46 궤적 (21)의 암 유전자에 대한 풍부g 공통 데스크탑 시퀀싱 장비. 라이브러리는 단순 마그네틱 비드 절차를 사용하여 정제하고, 신규 교정없이 qPCR을 분석하여 정량화한다. 통화 성능을 향상시키기 위해 샘플 DNA의 품질 관리 결과에 의해 통보 독립 생물 정보학 스위트 룸, NGS 다음 서열 분석을 제공합니다. 우리는 염기 치환 돌연변이, 삽입 / 결실 (삽입과 삭제를) 공개 대상 NGS이 시스템 접근 방식을 사용하여 데이터를 표시하고, 숫자는 FFPE와 FNA 샘플 및 실행 낮은 품질과 낮은 수량 종양 생검에서 (CNVs)를 변형 복사 통제 수단.
프로토콜
주 :이 프로토콜은 MiSeq NGS 시스템을 사용하여 샘플의 동시 처리를 설명하지만, 개인 게놈 머신 (PGM) 기기에 적합 할 수있다. 400 증폭 템플릿 복사본 권장 최소 DNA 입력의 경우, 분석은 318 칩상의 PGM을 사용하여 24 샘플 NGS 실행 당 96 개의 샘플들 각각에 대해, 적어도, 3,000 중앙값에 따르면, 등가 따르면 깊이를 제조 할 수있다. 또한,이 방법은 실시간 PCR 기기의 사용을 필요로한다.
1. DNA 기능 정량 및 품질 관리 (QC)
- 해동 시약 : 배 마스터 믹스 프라이머 프로브 믹스 억제 프라이머 프로브 믹스 -6- 카르복시 X-로다 민 (ROX), 희석제, 4 개의 인간 게놈 DNA 검량선 표준 (DNA 표준 (50 NG / μL), DNA 표준 (10 NG / μL), DNA 표준 (이 NG / μL) 및 DNA 표준 (0.4 NG / μL)) (표 1). 10 초 동안 최대 속도에서 10 초 원심 분리기에 대한 모든 시약이 내용을 수집하는 소용돌이.얼음의 2 배 마스터 믹스를 유지합니다.
- 시험 될 샘플의 총 수에 대한 마스터 믹스의 충분한 양을 준비하고 피펫으로 인해 부족을 방지하는 10 % 이상의 볼륨을 포함한다. 샘플 당 다음 볼륨을 사용하여 microcentrifuge 관의 마스터 믹스를 준비 : 5 ㎕의 2 배 마스터 믹스, 0.5 ㎕를 프라이머 프로브 믹스, 0.5 ㎕를 억제 프라이머 프로브 믹스, 0.05 μL의 ROX 및 2.95 ㎕의 희석제. 10 초는 내용을 수집하는 최대 속도 10 초 원심 분리기를위한 소용돌이.
- 96 웰 플레이트의 우물에 9 ㎕를 마스터 믹스를 추가합니다.
- 교정 곡선을 생성하기 위해 중복의 DNA 표준 1 μl를 추가합니다. 상하 5 번 피펫 팅하여 섞는다.
- 핵산 샘플을 사용하기 전에 잘 혼합해야합니다. 상하 최대 5 번 마스터 믹스 1 μL 샘플을 추가하고 피펫으로 혼합한다.
- 내용을 수집하기 위해 10 초 동안 최대 속도로 10 초 원심 분리기에 대한 플레이트, 소용돌이 인감.
- PCR을 시스템에 접시를 놓습니다. FAM (기능 정량화) 및 VIC (기능 억제) 제조업체의 지침에 따라 각 샘플에 대한 감지기를 모두 할당합니다. (95 ° C, 60 ℃에서 1 분 15 초)을 95 ° C에서 10 분의 PCR 사이클을 수행하고, 40 사이클.
- 소프트웨어 프로토콜을 사용하여 중복 된 DNA 표준들 각각에 대한 선형 회귀 곡선을 생성함으로써 qPCR의 데이터를 분석한다.
- X 축의 각 DNA 표준 및 Y 축에 대응 FAM의 C의 Q 값의 카피 수의 로그 (10)를 그린다.
- 다음, 상기 DNA 표준 검량선의 동적 범위 내의 핵산 시료 가을 결과 및 상기 기준 표준 곡선에 대응하는 위치로부터 μL 단위 "기능"또는 증폭 카피 수 미지의 DNA 농도를 계산하는 것을 확인한다. 그림 1은 통과 실패 교정 곡선의 예를 보여줍니다.
- 증폭은 VIC 채널의 비 - 인간 대상의 증폭의 유무를 확인하여 각각의 반응 발생 여부를 결정.
주 : 양성 대조군으로 억제 프라이머 프로브 믹스 비인간 외래 타겟 및 대응하는 템플릿에 대한 특이 적 프라이머를 포함한다. 억제 프라이머 프로브 믹스 노 템플릿 대조군 (NTC)를 포함하여 각각의 반응에 첨가 된 마스터 믹스의 구성 요소이다. 억제제의 부재에있어서, 인간이 아닌 대상의 PCR 생성물은 항상 VIC 채널에서 검출되어야한다. 피해자 채널 내의 샘플에 대한 "발견되지 않은"C의 Q 공정이 진행되기 전에 샘플의 후속 정리 이점있다 PCR 억제제의 존재를 나타낸다.
2. 라이브러리 준비 : 유전자 별 (GS) PCR
- 시험 될 샘플의 총 수에 대한 마스터 믹스의 충분한 양을 준비하고, 10 %를 pipett 인해 부족을 피하기 위해 더 볼륨을 포함ING. 샘플 당 다음 볼륨을 사용하는 미세 원심 분리 튜브에 GS PCR 마스터 믹스를 제조 : 5 μL 배 증폭 마스터 믹스 (표 1), 1 μL 팬 암 패널 프라이머 (표 1). 내용을 수집하기 위해 10 초 동안 최대 속도에서 10 초 원심 분리기에 대한 상하, 소용돌이을 피펫 팅하여 섞는다.
- 96- 웰 플레이트의 웰에 6 μL GS PCR 마스터 믹스 나누어지는. 개별 우물에 각 핵산 시료의 4 μl를 추가합니다. 다른 우물 위해, FFPE 제어의 4 μL (표 1), 멀티 변형 제어 (표 1)의 4 μL 및 절차 NTC에 대한 클레아없는 물 4 μl를 추가합니다. 각 추가의 경우, 상하 최대 5 회 피펫 팅하여 혼합한다.
- 내용을 수집하기 위해 10 초 동안 최대 속도로 10 초 원심 분리기에 대한 플레이트, 소용돌이 인감.
- 다음의 PCR 사이클에 대한 열 순환기에서 플레이트를 놓고 5 분간 95 ℃에서이주기 (95 ℃에서 15 초, 60 ℃에서 4 분), 23 사이클 (95 ° C, 72 ℃에서 4 분 15 초), 72 ℃에서 10 분간 최종 연장. 4 ° C에서 잡으십시오.
주의 : 단계 2.4이 완료된 후, 플레이트는 GS PCR 플레이트로 언급된다.
3. 라이브러리 준비 : 태그 PCR
- 해동 시약 : 배 지수 마스터 믹스 (표 1) 및 인덱스 코드 (표 1). 10 초는 내용을 수집하는 최대 속도 10 초 원심 분리기를위한 소용돌이.
주 : 인덱스 코드는 각 샘플에 대한 페어 지수 (바코드)의 고유 한 세트를 제공하기 위해 미리 혼합되어있다. - 상하 최대 5 시간을 96 웰 플레이트에 잘 지정에 배 지수 마스터 믹스의 7.5 μL 및 인덱스 코드의 5.5 μl를 추가하고 피펫으로 혼합한다.
- 조심스럽게 GS PCR 플레이트를 열고 마스터 믹스와 새로운 판에 2 μL GS PCR 제품을 추가 할 수 있습니다. 상하로 5 회 피펫으로 섞어. 각 샘플에 대해, 샘플 ID와 해당 페어 색인 코드를 기록한다. 내용을 수집하기 위해 10 초 동안 최대 속도로 10 초 원심 분리기에 대한 플레이트, 소용돌이 인감.
- , 72시 10 분 최종 연장 (95 ° C, 30 ~ 55 ° C, 72 ° C에서 1 분에서 초에서 30 초) 95 ° C, 10 사이클에서 5 분 동안 열 순환기 및 PCR에 접시를 놓고 기음. 4 ° C에서 잡으십시오.
주의 : 단계 3.4이 완료된 후, 플레이트는 태그 PCR 플레이트로 언급된다.
4. 도서관 정제 및 크기 선택
- 2-8 ° C에서 라이브러리 순수 준비 자석 구슬 (표 1)과 용출 버퍼 (표 1)를 제거하고 30 분 동안 실온으로 평형 수 있습니다. 워시 버퍼 (표 1) 용기, 캡 9.6 ml를 100 % 에탄올을 추가하고 병을 여러 번 반전 섞는다.
- 소용돌이 자기 10 초 동안 구슬은 9 별도의 우물에 11 μl를 추가6 웰 플레이트.
- 태그 PCR 플레이트를 열고 구슬과 피펫 믹스에 5 번 태그 PCR 제품의 10 μl를 추가합니다. RT에서 4 분 동안 인큐베이션 혼합물.
- 4 분 동안 자기 스탠드 (표 1)에 96 웰 플레이트를 놓습니다. 스탠드에 여전히 96 웰 플레이트로 제거하고 피펫으로 상층 액을 버린다.
- 상하 최대 5 번 자기 스탠드에서 96 웰 플레이트를 제거하고 각 웰에 100 ㎕의 에탄올 함유 워시 버퍼를 추가하고 피펫으로 혼합한다. 2 분 동안 품어.
- 2 분 동안 자기 스탠드에 96 웰 플레이트를 배치 한 다음 제거하고 피펫으로 상층 액을 버린다.
- 두 번째 세척 후 가능한 한 많이 세척 용액을 제거하는 2 에탄올 세척의 전체 단계를 반복 4.5.
- 자기 스탠드에 96 웰 플레이트, 다음, 실온에서 2 분 동안 구슬을 건조 스탠드에서 플레이트를 제거합니다.
- 각 아 20 μL 용출 완충액을 첨가하여 비드를 재현 탁리터와 피펫 5 회 상하입니다.
- 실온에서 2 분 동안 품어.
- 다시 4 분 동안 자기 스탠드에 96 웰 플레이트를 놓고, 조심스럽게 제거하고 잘 새에 맑은 상층 액 18 μl를 전송합니다.
주 : 절차 안전하게 -15 -30 ° C에서 저장이 단계 및 샘플에서 정지 될 수있다. 다시 시작 진행하기 전에 얼음에 냉동 샘플을 해동합니다.
5. 도서관 정량화
- 해동 도서관 퀀트 (LQ) 시약 : 배 LQ 마스터 믹스, LQ 프라이머 / 프로브 믹스, LQ 표준, LQ 긍정적 인 제어, LQ 희석제 및 LQ ROX (표 1). 10 초는 내용을 수집하는 최대 속도 10 초 원심 분리기를위한 소용돌이.
- 정제 라이브러리 제품을 사용, LQ 희석제 각 시료의 일련의 희석을 수행한다.
- 198 μL LQ 희석제 2 μL (라이브러리 정제 제품)을 추가하고 상하 피펫 10 배를 혼합한다.
- 1 : (100 희석 1) 2 μl를 추가98 μl를 LQ 희석제 및 피펫 10 배와 상하를 섞는다.
- 시험 될 샘플의 총 수에 대한 LQ 마스터 믹스의 충분한 양을 준비하고 피펫으로 인해 부족을 방지하는 10 % 이상의 볼륨을 포함한다. 샘플 당 다음 볼륨을 사용하여 microcentrifuge 관에서 LQ 마스터 믹스를 준비 : 5 ㎕의 2 배 LQ 마스터 믹스, 2 μL LQ 프라이머 / 프로브 믹스, 0.5 μl를 LQ 표준 0.5 μL LQ ROX합니다. 내용을 수집하기 위해 10 초 동안 최대 속도에서 10 초 원심 분리기에 대한 상하, 소용돌이을 피펫 팅하여 섞는다.
- 광학 96 웰 플레이트의 웰에 8 ㎕를 LQ 마스터 믹스를 추가합니다.
- 별도의 우물에서 2 μL 희석 도서관, 2 μL LQ 긍정적 인 제어 및 2 μL LQ의 희석제 (NTC)를 추가하고 상하 최대 5 회 피펫 팅하여 혼합한다. 내용을 수집하기 위해 10 초 동안 400 XG에 10 초 원심 분리기에 대한 광 접착 필름, 소용돌이와 함께 접시를 밀봉합니다.
- FAM과 VIC 디텍 모두 할당각 샘플에 대한 RS. (95 ° C, 60 ℃에서 1 분 15 초)을 95 ° C에서 5 분의 사이클링 조건을 사용하여 PCR 증폭을 수행하고, 40 사이클.
- 비교 C Q의 방법을 사용하여 각 시료 (㎚)의 농도를 결정한다. 미지의 라이브러리 (C Q의 FAM)로 알려진 표준 LQ (C의 Q VIC)의 차이를 계산한다. 희석 된 샘플 (PM)의 농도는 다음의 방정식을 사용하여 계산된다 :
[이 용액에 농축 lib 디렉토리] nM의 = 12.5 × 2 Δ Cq와
10,000 이외의 희석 배수를 사용하는 경우, 10,000 대상 희석 계수의 비율을 계산하고 570에서 수학 식의 결과가이 비율을 곱한다.
6. 도서관 정상화 및 샘플 풀링
- 모든 샘플에서 평균 농도 (㎚) (각각 고유 페어 인덱스를 포함) 풀링 할을 결정합니다.
- 각각의 샘플 있습니다 volum를 결정전자 (μL)는 개별 농도 (㎚)로 나누어 다음, (5)에 의해 모든 샘플에서 평균 농도를 곱하여 풀 수 있습니다. 가장 가까운 정수에 결과 값 라운드. 라운드의 값으로 볼륨 <2 μL 2 μL 및 볼륨> 15 μl를 15 μl를.
- 시료 풀을 생성하기 위해 단일 마이크로 원심 튜브에 각 샘플에 대한 정규화 된 양 (μL)을 추가한다.
- 둥근 정수 값을 사용하여 각 시료에 대한 새로운 농도를 계산하고 그 결과를 기록한다.
- 시료 풀의 농도를 결정하기 위해 모든 개별적인 농도의 합을 계산하여 그 결과 값 (㎚)을 기록한다.
- 연속 희석액 (표 1)을 사용하여 125 ㎚에 시료 풀을 희석.
주 : 절차 안전하게 -15 -30 ° C에서 저장이 단계 및 샘플에서 정지 될 수있다. 다시 시작 진행하기 전에 얼음에 냉동 샘플을 해동합니다.
7. 시퀀싱
- DenatURE 다음 추가하여 PhiX 제어 V3 (표 1)의 존재 샘플 수영장 : 1.25 nM의 샘플 수영장, 0.5 nM의 PhiX의 3 ㎕의 1 N NaOH를 2 μL의 15 μl를. 소용돌이는 간단히 간단한 원심 분리 및 RT에서 5 분 동안 품어.
- 얼음에 변성 샘플 풀을 배치합니다.
- 미세 원심 튜브에 미리 냉장 HT1-HYB 버퍼의 992 μL에 변성 라이브러리의 8 μl를 추가합니다. 소용돌이 간략 내용을 수집 할 간략한 원심 분리 한 다음, 혼합한다. 얼음에 보관하십시오.
- 시약 카트리지 # 17의 위치를 변성 및 희석 라이브러리의 600 μl를 추가합니다.
- 해동 한 읽기 시퀀싱 프라이머 (표 1), 인덱스 읽기 시퀀싱 프라이머 (표 1), (2) 및 읽기 시퀀싱 프라이머 (표 1). 마이크로 원심 튜브에 별도로 636 ㎕를 HT1-HYB 버퍼 4 μL 시퀀싱 프라이머를 희석. 참고 : HT1-HYB 버퍼가 제공 기지입니다시간 시퀀싱 시약 키트 (표 1).
- 내용을 수집하기 위해 10 초 동안 최대 속도로 10 초 원심 분리기에 대한 텍싱에 의해 섞는다. 시퀀싱 시약 카트리지의 # 18를 위치 희석 읽기 1 시퀀싱 프라이머의 600 μl를 추가, 희석 인덱스 읽기 프라이머 600 μL는 # 20의 위치를 # 19, 희석 읽기 2 시퀀싱 프라이머의 600 μl를 배치합니다.
- 제조업체의 지침에 따라 NGS 장비 (표 1)과 순서에 시약을 넣습니다. 페어링 엔드 2 × 150 사이클 시퀀싱 실행을 수행합니다.
8. 데이터 분석
주 : NGS 구 소프트웨어는 각 샘플에 대해 (.fastq.gz *) 파일을 기본 통화 품질 스코어를 클러스터 화상을 변환하고, 각각의 GZIP 압축 FASTQ를 생성 페어 인덱스를 역 다중화한다. 역 다중화 된 파일을 분석하기에 앞서, 리더 (표 다운로드와 관련된 바이오 인포 매틱스 소프트웨어를 설치해야1). 소프트웨어는 소비자 용 윈도우 PC에 설치 될 수 있고, 특수한 컴퓨팅 하드웨어 또는 데이터 분석을 수행하기 위해 인터넷 연결을 필요로하지 않는다.
- 소프트웨어 바탕 화면 아이콘을 두 번 클릭합니다.
- 소프트웨어 설명서에서 제공하는 사용자 이름과 암호를 사용하여 시스템에 로그인합니다.
- 프로젝트 대시 보드를 열고 "새 프로젝트"를 클릭합니다.
- 프로젝트 이름을 지정하고 선택적인 프로젝트 설명을 제공한다. 대상 NGS 패널 유형 및 NGS 장비 유형을 선택합니다. "저장하고 계속하기"를 클릭합니다.
- 앞으로의 압축 FASTQ 파일을 업로드하고 읽어 역. 그 역 다중화하는 데 실패 읽기 포함 "할당되지 않은"FASTQs을 업로드하지 마십시오. "저장하고 계속하기"를 클릭합니다.
- 입력 기능 DNA 정량 분석 (상기 단계 1 참조)에 의해 결정되는 각 라이브러리를 제조하는데 사용되는 기능 입력 매수. 수동 확산의 값 또는 복사 및 붙여 넣기 값을 추가주석 테이블에 시트입니다. "저장하고 계속하기 '를 클릭합니다.
- 분석을 위해 업로드 주석 라이브러리를 검토하고 분석을 시작합니다 "분석을 제출"을 클릭합니다.
- 프로젝트 대시 보드를 통해 표시되는 분석의 진행 상황을 모니터링합니다.
참고 : 결과를 검토 할 준비가되면 분석 완료 상태 표시가 표시됩니다. - 의 비율을 라이브러리 당 전체 범위를 포함하는 샘플 품질 관리 측정에 대한 분석 결과를 검토 통과 필터, 앰플 리콘 범위의 깊이와 균일 성을 읽습니다. 변형이 dbSNP, COSMIC, 1000 게놈 및 기능과 인구 수준 주석의 다른 소스와 각각의 염기 서열을 라이브러리에 대한 호출을 검토합니다.
- 보완 정보학 도구를 사용하여 장기 저장 또는 다운 스트림 분석을위한 요약 스프레드 시트 테이블, * .bam 파일과 *가 .vcf 파일로 원시 결과를 내 보냅니다.
결과
양성 및 음성 대조군 이전 특징 세포주 잔류 임상 FFPE 종양 생검을 나타내는 90 샘플 (74 고유의 합계)는 시퀀스 어댑터 바코드 태그로 증폭 DNA, 멀티 플렉스 PCR 농축에 입력 대해 평가하고, 단일 분석 하였다 19.1 M을 생산 벤치 탑 NGS 장비 실행 (그림 2) 필터를 통과 읽습니다. 몰 샘플 풀링은 높은 깊이 시퀀싱 (3,692x 읽기) 및 균일 한 커버리지 (중간 읽기 깊이의 5 배 내에서 적용 증폭의 97.8 %)에서 발생했습니다. 특이점은 템플릿 컨트롤, 대형 카피 수 증폭 한 셀 라인 DNA 및 사전 분석 QC 분석에 의해 PCR 억제을 받았습니다 된 한 FFPE의 DNA (그림 3)를 구성합니다. 46 증폭 걸쳐 커버리지 균일 성 (세 가지 연산자 (도 4a), 그리고 다른 저품질 FFPE의 DNA 샘플에 대한 사용을 유지 그림 4B). 잔류 임상 검체의 혼합물로부터 제제화 FFPE 종양 DNA 제어, 3.9, 5.3의 존재비의 목표 BRAF 돌연변이가보고되었다 (액적 디지털 PCR로 정량) 5 % BRAF의 V600E 달성, 3 사업자가 6.5 %를 사용하는 400 증폭 사본 (따라서 20 돌연변이 부) (그림 4B 및 삽입 테이블의 "FFPE3")의 입력. 17 % 평균 대립 유전자 주파수 (표 2) - 또한, 각 알려진 "드라이버"기본-대체 돌연변이를 나타내는 12 합성 DNA 템플릿의 혼합물 (9)의 의도 된 범위에서 예상되는 돌연변이를 한 것으로 밝혀졌습니다. 카피 수 증폭과 세포 라인과 FFPE DNA 샘플을 희석 EGFR 및 KRAS, 각각 (그림 5)의 변형에 대한 선량 의존성을 보여 주었다. 중요한 FFPE의 DNA 입력 위양성없이 적은 50 증폭 카피 나 대량 DNA 1.2 NG 알려진 돌연변이의 검출을 유지하면서 감소 될 수있다호출 (그림 6). DNA 입력은 적어도 50,000 증폭 카피 (표 3) 내지 100 배의 범위까지 위에 수용 하였다. 이것과 관련된 실험에서 변형 22 FFPE 호출에서는 20 FNA 표본 공유 돌연변이 범위 (표 4)와 독립적 인 방법으로 계약에보고되었다.
분석에 대한 민감도와 양성 예측도는 신선한 냉동 FFPE, FNA, 및 셀 라인 DNA, 195 시퀀싱 결과 총 포함 97 샘플의 분석에서 결정되었다. 99.7 %의 및 양성 예측도 (PPV) (95 % CI : 결과는 365 진정한 긍정적 인 변형 통화, 4 위음성 통화 및 98.9 %의 민감도 (97.1-99.7 %, 95 % CI) 1 가양 통화를 한 것으로 밝혀졌습니다 : 98.2-99.99 %). 삽입과 삭제의 분석은 93.9 %의 민감도를 보여, 33 샘플 실행의 두 가지 일반적인 EGFR 변이 (p.E746_A750delELREA 및 p.V769_D770insASV)에 대해 수행되었다 (95 % CI : 78.4-98.9 %)와 100 % (95 % CI의 PPV : 86.3-100 %) 변종은 2.4-84.8 %의 범위에서 검출.
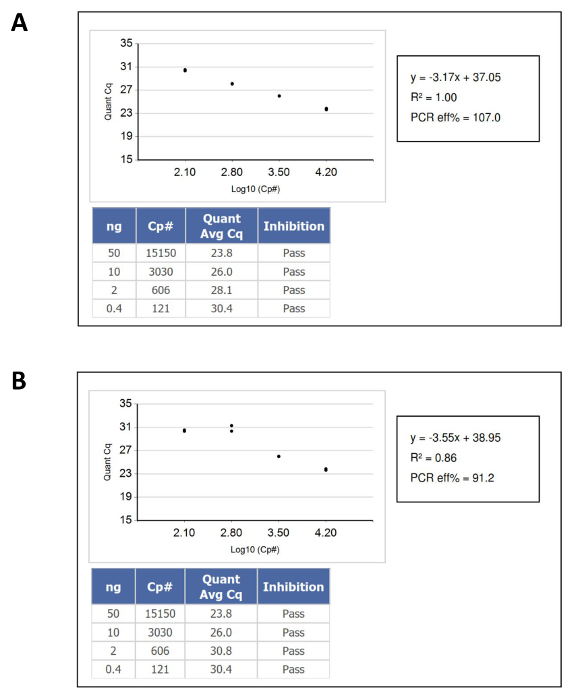
그림 1 :. 패스 및 실패 품질 관리 기준 (A) 통과 표준 곡선 DNA 정량 교정 곡선의 예. (B) 실패한 표준 곡선. 이 경우, 오류가 가장 낮은 입력 DNA 표준의 중복 피펫 팅에 의해 발생했다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
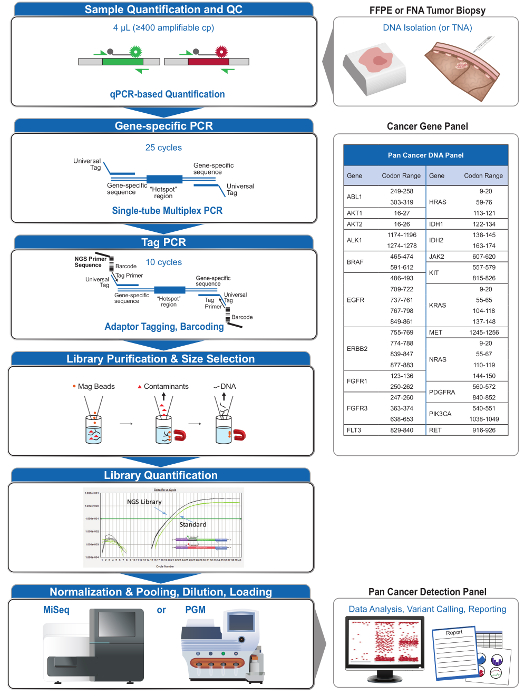
그림 2 : 종양학 응용 프로그램에 대한 대상 종합 NGS 시스템의 개요 사전 분석 통합등, 분석 및 사후 분석 워크 플로우. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
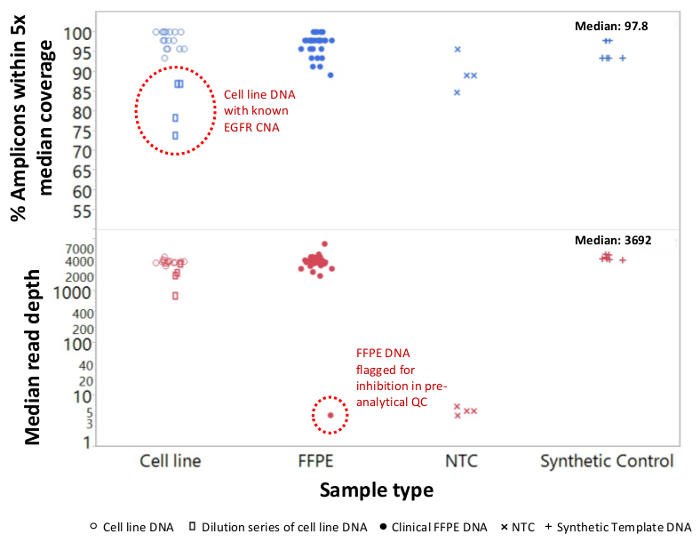
그림 3 : 읽기 범위 및 균일 본래 셀 라인 DNA 및 컨트롤에 비해 낮은 품질의 FFPE의 DNA에 암 유전자의 표적으로 NGS에 대한 잔류 임상 FFPE (폐쇄 원), 셀 라인을 포함 90 샘플의 총 (오픈 원. ), 및 합성 주형 DNA (더하기 기호) 단일 튜브, 21 유전자 멀티 플렉스 PCR 농축을 사용하여 처리 하였다. 각 앰플 리콘 라이브러리는 2.5 nM의 농도로 구별 듀얼 인덱스 정량 정제 번호, 및 정규화하여 바코드 NGS 악기 용 어댑터 서열 태그 하였다. 한 DNA 라이브러리 컴패니언 바이오 인포 매틱스 소프트웨어에 의해 서열 분석 하였다. 샘플 데비관리 포인트 큰 EGFR의 카피 수 증폭 (위, 점선 원에서 열린 사각형)의 상당한 번호를 생성하는 데 실패 커버리지 균일 한 흑색 종 FFPE 샘플을 왜곡 때문에 읽기 베어링 MDA-MB-468 셀 라인 DNA의 희석 시리즈를 포함 DNA의 추출로부터 PCR 억제제의 이월한다. 흑색 종 샘플 실패 (아래 점선 원)는 사전 분석 qPCR에 DNA QC 분석에 의해 예측했다. NTC, 아니 템플릿 컨트롤 (X 기호). 이 그림의 더 큰 버전을 보려면 여기를 클릭하여 주시기 바랍니다.
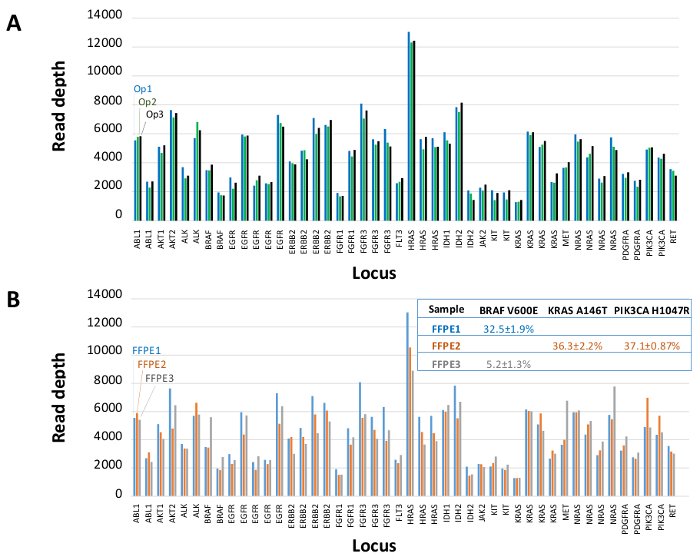
그림 4 : 앰플 리콘 별 앰플 리콘 읽기 범위, 균일 성 및 잔류 임상 FFPE 종양 DNA에 변형 탐지. (A)를 측정 대표 FFPE의 DNA 샘플의 모든 농축 궤적에 걸쳐 적용 범위를 읽고세 가지 다른 사업자에 걸쳐. 운영자 1, OP1 (파란색 막대); 운영자 2, OP2 (녹색 막대); 운영자 3, OP3 (검은 막대). 공지 된 5 % BRAF c.1799T> 돌연변이 구성된 제어 혼합물 (FFPE3, 회색 막대 및 텍스트) 등 3 FFPE 종양 샘플을 사용하여 평가 (B) 범위의 균일 변형 통화. FFPE1 (파란색 막대 및 텍스트), FFPE2 (오렌지 바 및 텍스트). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
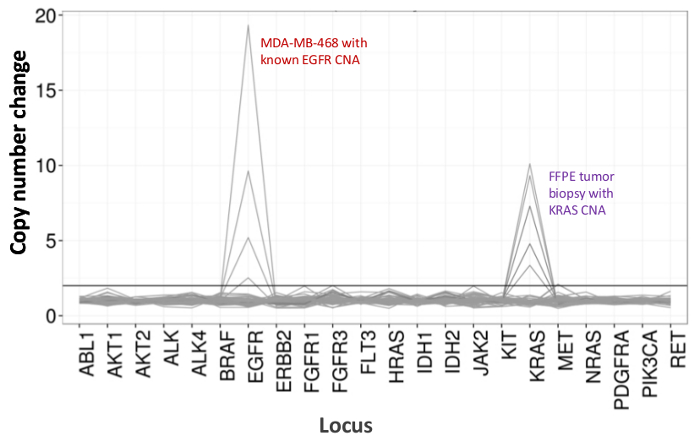
그림 5 :. 잘 특징 EGFR의 카피 수 증폭과 세포 라인과 FFPE DNA MDA-MB-468 셀 라인 DNA 복사 번호 변종의 용량 의존적 검출은 점진적으로 기준이 아닌 돌연변이 세포의 배경에 희석 - 라인 DNA 등은 카피 수 변화의 감소를 설명 할희석의 기능. 각 셀에 행 DNA 샘플의 백분율은 별개 라인 (0, 12.5, 25, 50, 100 %)을 나타낸다. 알려진 KRAS 증폭과 난소 FFPE 종양 샘플의 희석은 동일한 적정 시리즈를 사용하여 유사한 프로필을 공개하지만이 상단 라인의 100 % FFPE의 DNA의 복제와. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
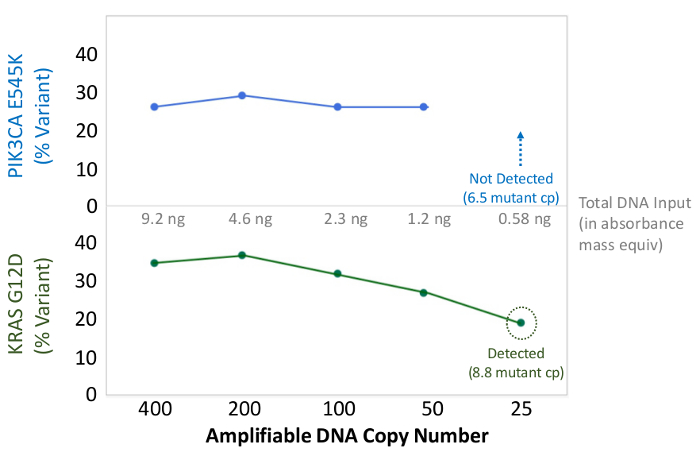
그림 6 : 정확한 돌연변이 검출 및 정량 대량 DNA를 ng를 50 증폭 FFPE DNA 복사, 또는 1.2 FFPE DNA가 qPCR에 기반 QC 분석에 의해 결정 된 대장 암의 증폭 사본 번호와 같은 25 복사본 (400)에서 희석. 시퀀싱하기 전에 멀티 플렉스 PCR 농축에 입력. 생물 정보학 파이프 라인은 정확하게 알려진 리언 모두라고아래로 50 사본, 또는 ~ 10 돌연변이 템플릿의 상당 화면 반전. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 소재 / 재료의 이름 | 회사 | 카탈로그 번호 |
| 배 Quantidex 마스터 믹스 | Asuragen | 145345 |
| 퀀트 프라이머 프로브 믹스 | Asuragen | 145336 |
| 억제 프라이머 프로브 믹스 | Asuragen | 145344 |
| ROX | Asuragen | 145346 |
| 희석제 | Asuragen | 145339 |
| DNA 표준 (50) | Asuragen | 145340 |
| DNA 표준 (10) | Asuragen | 145341 |
| DNA 표준 (2) | Asuragen | 145342 |
| DNA 표준 (0.4) | Asuragen | 145343 |
| 배 증폭 마스터 믹스 | Asuragen | 145348 |
| 팬 암 프라이머 패널 | Asuragen | 145347 |
| 팬 암 FFPE 제어 | Asuragen | 145349 |
| 팬 암 멀티 변형 제어 | Asuragen | 145350 |
| 도서관 순수 준비 비즈 | Asuragen | 145351 |
| 세척 버퍼 | Asuragen | 145352 |
| 용출 버퍼 | Asuragen | 145353 |
| 배 LQ 마스터 믹스 | Asuragen | 145358 |
| LQ 희석제 | Asuragen | 145354 |
| LQ 긍정적제어 | Asuragen | 145355 |
| LQ 표준 | Asuragen | 145356 |
| LQ 프라이머 / 프로브 믹스 (ILMN) | Asuragen | 145357 |
| LQ ROX | Asuragen | 145359 |
| 인덱스 코드 (ILMN) - 세트 A | Asuragen | 150004 |
| AIL001 - AIL048 (48) | ||
| 인덱스 코드 (ILMN) - 세트 B | Asuragen | 150005 |
| AIL049 - AIL096 (48) | ||
| 배 지수 마스터 믹스 | Asuragen | 145361 |
| 1 시퀀싱 프라이머 읽기 | Asuragen | 150001 |
| 인덱스 읽기 시퀀싱 프라이머 | Asuragen | 150002 |
| 2 시퀀싱 프라이머 읽기 | Asuragen | 150003 |
| 시퀀싱 희석제 | Asuragen | 145365 |
| Illumina의 MiSeq | 일루미나 | |
| MiSeq 시약 키트 v3의 (600 사이클) | 일루미나 | MS-102-3003 |
| MiSeq 시약 나노 키트 v2를 (300 사이클) | 일루미나 | MS-103-1001 |
| PhiX 제어 V3 | 일루미나 | FC-110-3001 |
| 마그네틱 스탠드-96 (또는 동등한 장치) | Ambion이 | AM10027 |
| Quantidex 리포터 소프트웨어 | Asuragen |
표 1 :. 시약 및 키트 ROX의 최초 사용시, 2-8 ℃에서 유리 병을 저장합니다. 재 냉동하지 마십시오. 소프트웨어 www.asuragen.com에서 다운로드 할 수 있습니다.
| 지엔 | COSMIC 변형 | COSMIC 아미노산 | %의 변형 |
| NRAS | c.182A> G | p.Q61R | 13.3 |
| NRAS | c.35G>를 | p.G12D | 15.2 |
| HRAS | c.182A> G | p.Q61R | 17.8 |
| HRAS | c.35G>를 | p.G12D | 9.2 |
| KRAS | c.182A> G | p.Q61R | 13.5 |
| KRAS | c.35G>를 | p.G12D | 19.1 |
| PIK3CA | c.1633G>를 | p.E545K | 9.3 |
| PIK3CA | c.3140A> G | p.H1047R | 9.1 |
| 전부 | c.2447A> T | p.D816V | 14.6 |
| EGFR | c.2369C> T | p.T790M | 11.3 |
| EGFR | c.2573T> G | p.L858R | 14.9 |
| BRAF | c.1799T>를 | p.V600E | 17.3 |
표 2 : 풀링 합성 컨트롤 9~17%의 풍요로 정량화 12 "드라이버"암 유전자 변형으로 구성되어 12 별개의 돌연변이가 부착 된 12 가지 이중 가닥 합성 템플릿의 혼합물 순서 다음 평가 하였다.. 모든 변종이 제대로없는 오탐 (false positive)으로 불렸다.
| 샘플 ID | 기능의 CPS | 유전자 | COSMIC 변형 | COSMIC 아미노산 | %의 변형 | 중간 읽기 깊이 | 평균의 5 배 이내 % |
| BCPAP | (400) | BRAF | c.1799T>를 | p.V600E | 99.5 | 3,289 | 96 % |
| BCPAP | 만 | BRAF | c.1799T>를 | p.V600E | 99.7 | 4040 | 98 % |
| BCPAP | 25000 | BRAF | c.1799T>를 | p.V600E | 99.4 | 3,687 | 96 % |
| BCPAP | 50000 | BRAF | c.1799T>를 | p.V600E | 99.7 | 4611 | 93 % |
표 3 : 범위 및 변형 콜링은 DNA 입력의> 100 배 범위에서 유지됩니다. 를 BCPAP 세포주에서 증폭 DNA (400) 50,000에 복사 염기 서열에서 멀티 플렉스 PCR 농축에 입력했다. 읽기 깊이 따르면 균일 돌연변이 탐지 및 돌연변이 정밀도가 입력 범위에 걸쳐 보존 하였다.
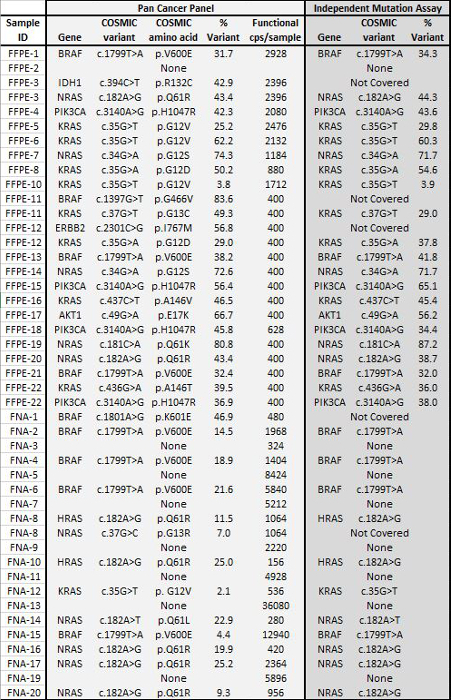
표 4 : 변형 22 FFPE 20 FNA 종양 생검에서 호출 독립 돌연변이 분석 실험의 결과에 동의 이전에 직교 대상 NGS 분석에 의해 결정 돌연변이 상태로 22 FFPE 종양 DNA의 세트는 PCR 농축에 400 2928로 증폭 사본에 입력했다. 공정 및 21 유전자 팬 암 패널을 사용하여 서열화. 또한, 20 FNA DNA 샘플의 코호트 이전 PCR 8 156 36080 입력 증폭 카피 및 서열화를 사용하여 증폭 하였다 액체 비드 어레이 돌연변이 분석을 이용하여 특징으로한다. 팬 암 NGS 패널과 참조하지만 사이의 모든 중복 통화후부 방법은 계약에 있었다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
NGS 기술은 임상 9 종양 생검의 분자 프로파일을 심문에 대한 기대를 재정의했다. 타겟 NGS 패널 수많은 임상 검체 3,5,10-15 복수 종류의 평가를위한 조사 기술 개발 실험실 검사 및 시판품 맞춤 패널로서 개발되어왔다. 여러 연구 보고서는 게놈 변경 3,10-12,14의 검출을위한 민감하고 특정 임상 도구로 NGS의 가치를 증명하고있다. 그러나 연구는 또한 FFPE는 2-5,12,14을 표본으로 도전 암 조직 검사에서 위양성 결과가 발생할 수 있습니다 유물의 위험을 증명하고있다. 또한, 최근 출판물은 낮은 입력 DNA (12)의 사용에 의해 악화되는 상업 대상 NGS의 패널 (16)과 거짓 양성 호출 종양학를 사용하여 NGS 높은 실패율을 표본 강조했다. 따라서 일부 LABORA토리 중 하나를 수정하거나 상업적으로 이용 가능한 대상 NGS 기술은 정확성을 보장하거나 생물 정보학은 5,10,11 분석에서 결과를 확인하기 위해 자주 성능을 향상시키기 위해 추가 견제와 균형을 추가했습니다.
21 유전자 패널 (그림 2)와 같은 FFPE 및 FNA 종양 생검 등의 도전 시편 종류의 증거를 기반으로 실행 가능한 돌연변이를 심문 할 수있는 포괄적 인 NGS 시스템에 대한 대상 콘텐츠로 개발되었다. 워크 플로우는 이점이있다 : 균일 앰플 리콘에 따르면 (도 3 및 4, 표 3)를 설치함으로써 1) 일관성; 2) 사용 용이성을 제공하여, 최적화 된 시약 세트를 미리 배합하고, 생물 정보학 요건을 단순화; 워크 플로우를 간소화 및 다른 상업적인 NGS 방법에 비해 피펫 공정 수를 줄여 3) 효율; 않는 앰프를 평가하기위한 QC DNA 분석을 포함하여, 4) 정확도수 DNA 복사 번호는 허용 템플릿 다양성을 보장하고 변형 감지 4의 확률 적 변동을 방지 할 수 있습니다. 생물 정보학 분석과 사전 분석 QC 데이터의 통합은 FFPE DNA의 낮은 입력이 가능합니다. 이는 진실 독립적 대책 400 FFPE 시료 전체 DNA 입력 상이한 기능 사본을 사용하여 결정 트리 알고리즘을 훈련 및 생물 정보학 소프트웨어에 이러한 알고리즘을 통합함으로써 달성되었다. 따라서, DNA의 FFPE ~ 5-20 NG 전형적 당량 400 증폭 카피 권장 입력은 FFPE DNA의 250 ~ 17,18 NG가 추천 혼성화 기반 농축을 포함하는 다른 방법 10-12 버금가 . 기술이 MiSeq 플랫폼의 사용에 대해 기재되어 있지만, 다른 NGS 플랫폼 서열 분석을 사용하는 기기 별 태그 어댑터 PCR 프라이머를 사용하여 변형 될 수있다.
몇 가지 단계가 성공을 보장하는 것이 중요하다절차. 사전 분석 QC 분석은 DNA 및 보고서 기능 억제의 증폭 카피 수를 결정합니다. 400 미만 증폭 DNA 카피는 PCR 농축 공정에서 사용되는 경우, 미량 돌연변이 샘플에서 위음성 호출 (도 6)의 증가 된 위험이있다. 또한, 치료는 세척 또는 용출 단계에서 자기 구슬을 통해 건조를 방지하기 위해 라이브러리를 정제하는 동안주의해야합니다. 또한, 성공적인 라이브러리 정량화 라이브러리 DNA의 정확한 희석에 크게 의존한다. 최상의 결과를 들어, qPCR에의 차이는 ≤3.3 Cq와해야 샘플 라이브러리 LQ 표준 (Cq와 VIC)에 비해 (된 CQ FAM)에 대해 발생합니다. 그 차이가 3.3 이상 Cq와 재 희석 및 샘플 시험 인 경우에 권장된다. 우수한 상관이 경쟁 qPCR의 방법과 상업용 키트 사이 관찰되었지만, 표준 곡선을 사용하여 절대 정량을 제공하는다른 방법으로 클론의 증폭 단계에 대해 라이브러리의 입력 오프셋 최적 파종 밀도를 달성 할 필요가있다.
일부 암 표본 특히 때문에 DNA 분리 후 지속 억제제의 시퀀스에 도전한다. 라이브러리 준비하기 전에 이러한 샘플을 확인하기 위해, qPCR에 QC 분석은 또한 내부 통제 및 기능 억제를위한 감시 두 역할을하는 외생 템플릿을 포함하여 증폭 억제를 감지합니다. 예는 흑색 종의 DNA 샘플 메트릭 전에 순서에 QC 억제를 통과하는 데 실패하고 염기 서열을 할 수있는 라이브러리를 생성하는 데 실패하는 그림 3에 제시되어있다. 실패 가능성이 FFPE DNA 분리 단계 이월 멜라닌 오염 공지 된 PCR 억제제의 결과였다. 품질 관리 분석에 의해 식별되는 샘플은 별도의 청소 단계 t를 통해 회수 할 수있다 증폭 실패에 대한 위험에 노출 될 수 있습니다오 잠재적 억제제를 제거합니다.
대상 21 유전자 패널은 증거 기반 유전자 핫스팟에 초점을 맞추고 DNA QC, 사전 분석 "기능"DNA 정량 결과에 의해 통보 NGS 및 생물 정보학 소프트웨어에 최적화 된 시약 및 컨트롤 완벽한 시스템을 제공한다. 상기 방법은 정확한 저 입력 DNA의 염기 - 치환 돌연변이 삽입과 삭제를 검출하고, 이러한 CNVs 같은 추가적인 변형을 검출하고, 표적 RNA 서열에 대해 적응 될, 패널의 콘텐츠를 확대하기 위해 옵션으로 NGS 시스템의 일례를 제공한다.
공개
JH, AH, RZ, BCH 및 GJL 직원이며 Asuragen, Inc.의 RZ, BCH의 사주가 있고, GJL는 각 샘플에 대해 결정 증폭 사본 번호 정보를 사용하여 호출 변형을 개선하기위한 특허 출원에 공동 발명가입니다.
감사의 말
우리는 원고의 검토를 위해 박사 아네트 Schlageter 감사합니다. 이 작품은 암 예방과 텍사스 연구소 (: GJL PI)에서 부여 CP120017에 의해 부분적으로 지원되었다.
자료
| Name | Company | Catalog Number | Comments |
| 2x Quantidex Master Mix | Asuragen | 145345 | |
| Quant Primer Probe Mix | Asuragen | 145336 | |
| Inhibition Primer Probe Mix | Asuragen | 145344 | |
| ROX | Asuragen | 145346 | |
| Diluent | Asuragen | 145339 | |
| DNA Standard (50) | Asuragen | 145340 | |
| DNA Standard (10) | Asuragen | 145341 | |
| DNA Standard (2) | Asuragen | 145342 | |
| DNA Standard (0.4) | Asuragen | 145343 | |
| 2X Amplification Master Mix | Asuragen | 145348 | |
| Pan Cancer Primer Panel | Asuragen | 145347 | |
| Pan Cancer FFPE Control | Asuragen | 145349 | |
| Pan Cancer Multi-Variant Control | Asuragen | 145350 | |
| Library Pure Prep Beads | Asuragen | 145351 | |
| Wash Buffer | Asuragen | 145352 | |
| Elution Buffer | Asuragen | 145353 | |
| 2X LQ Master Mix | Asuragen | 145358 | |
| LQ Diluent | Asuragen | 145354 | |
| LQ Positive Control | Asuragen | 145355 | |
| LQ Standard | Asuragen | 145356 | |
| LQ Primer/Probe Mix (ILMN) | Asuragen | 145357 | |
| LQ ROX | Asuragen | 145359 | |
| Index Codes (ILMN) - Set A, AIL001 - AIL048 (48) | Asuragen | 150004 | |
| Index Codes (ILMN) - Set B, AIL049 - AIL096(48) | Asuragen | 150005 | |
| 2x Index Master Mix | Asuragen | 145361 | |
| Read 1 Sequencing Primers | Asuragen | 150001 | |
| Index Read Sequencing Primers | Asuragen | 150002 | |
| Read 2 Sequencing Primers | Asuragen | 150003 | |
| Sequencing Diluent | Asuragen | 145365 | |
| Illumina MiSeq | Illumina | ||
| MiSeq Reagent Kit v3 (600-cycle) | Illumina | MS-102-3003 | |
| MiSeq Reagent Nano Kit v2 (300-cycle) | Illumina | MS-103-1001 | |
| PhiX Control v3 | Illumina | FC-110-3001 | |
| Magnetic Stand-96 (Or equivalent device) | Ambion | AM10027 | |
| Quantidex Reporter Software | Asuragen |
참고문헌
- Chen, G., Mosier, S., Gocke, C. D., Lin, M. T., Eshleman, J. R. Cytosine deamination is a major cause of baseline noise in next-generation sequencing. Mol Diagn Ther. 18 (5), 587-593 (2014).
- Choudhary, A., et al. Evaluation of an integrated clinical workflow for targeted next-generation sequencing of low-quality tumor DNA using a 51-gene enrichment panel. BMC Med Genomics. 7, 62 (2014).
- Sah, S., et al. Functional DNA quantification guides accurate next-generation sequencing mutation detection in formalin-fixed, paraffin-embedded tumor biopsies. Genome Med. 5 (8), 77 (2013).
- Zhang, L., et al. Profiling cancer gene mutations in clinical formalin-fixed, paraffin-embedded colorectal tumor specimens using targeted next-generation sequencing. Oncologist. 19 (4), 336-343 (2014).
- Latham, G. J. Next-generation sequencing of formalin-fixed, paraffin-embedded tumor biopsies: navigating the perils of old and new technology to advance cancer diagnosis. Expert Rev Mol Diagn. 13 (8), 769-772 (2013).
- Crawford, J. M., et al. The business of genomic testing: a survey of early adopters. Genet Med. 16 (12), 954-961 (2014).
- Smith, D. L., et al. A multiplex technology platform for the rapid analysis of clinically actionable genetic alterations and validation for BRAF p.V600E detection in 1549 cytologic and histologic specimens. Arch Pathol Lab Med. 138 (3), 371-378 (2014).
- Thomas, F., Desmedt, C., Aftimos, P., Awada, A. Impact of tumor sequencing on the use of anticancer drugs. Curr Opin Oncol. 26 (3), 347-356 (2014).
- Singh, R. R., et al. Clinical validation of a next-generation sequencing screen for mutational hotspots in 46 cancer-related genes. J Mol Diagn. 15 (5), 607-622 (2013).
- Beadling, C., et al. Combining highly multiplexed PCR with semiconductor-based sequencing for rapid cancer genotyping. J Mol Diagn. 15 (2), 171-176 (2013).
- McCall, C. M., et al. False positives in multiplex PCR-based next-generation sequencing have unique signatures. J Mol Diagn. 16 (5), 541-549 (2014).
- Schleifman, E. B., et al. Next generation MUT-MAP, a high-sensitivity high-throughput microfluidics chip-based mutation analysis panel. PLoS One. 9 (3), e90761 (2014).
- Wong, S. Q., et al. Sequence artefacts in a prospective series of formalin-fixed tumours tested for mutations in hotspot regions by massively parallel sequencing. BMC Med Genomics. 7, 23 (2014).
- Narayan, A., et al. Ultrasensitive measurement of hotspot mutations in tumor DNA in blood using error-suppressed multiplexed deep sequencing. Cancer Res. 72 (14), 3492-3498 (2012).
- Hagemann, I. S., et al. Clinical next-generation sequencing in patients with non-small cell lung cancer. Cancer. 121 (4), 631-639 (2015).
- Won, H. H., Scott, S. N., Brannon, A. R., Shah, R. H., Berger, M. F. Detecting somatic genetic alterations in tumor specimens by exon capture and massively parallel sequencing. J Vis Exp. (80), e50710 (2013).
- Simen, B. B., et al. Validation of a next-generation-sequencing cancer panel for use in the clinical laboratory. Arch Pathol Lab Med. 139 (4), 508-517 (2015).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유