Method Article
Интеграция мокрого и сухого Bench Оптимизирует Targeted Следующее поколение Секвенирование низкого качества и низкого количества Опухолевые Биопсию
В этой статье
Резюме
An integrated system for targeted next-generation sequencing of oncology specimens is described. This cross-platform system is optimized for low-quality and low-quantity tumor biopsies, accommodates low DNA inputs, includes well-characterized multi-variant controls, and features a novel variant caller that is informed by quantitative pre-analytical quality control measures.
Аннотация
Все процедуры следующего поколения секвенирования (NGS) включают анализы, выполненные на лабораторном столе ( "мокрую скамейку") и данные анализов, проводимых с использованием биоинформатики трубопроводов ( "сухой скамейке"). Оба элемента имеют важное значение для получения точных и надежных результатов, которые особенно важны для клинических лабораторий. Целевые технологии NGS все чаще благоволение в онкологических приложений, чтобы помочь достижению целей точной медицины, но методы часто включают отсоединены и изменчивы мокрой и сухой стендовые рабочие процессы и несогласованные наборы реагентов. В этом докладе мы опишем метод секвенирования вызов образцов рака с группой 21-гена в качестве примера комплексной целевой системы NGS. Система интегрирует функциональную количественную ДНК и квалификацию, монотуннеля мультиплексированный обогащение ПЦР и очистку библиотеки и нормализацию с помощью аналитически-Подтвержденные одного источника реагентов с набором автономных биоинформатики.В результате, точный вариант требует от низкого качества и низкого количества фиксированных формалином, парафин (FFPE) и тонкоигольной аспирации (FNA) опухолевые биопсии может быть достигнуто. Метод может регулярно оценивать рак связаны варианты от входа 400 усиливаемых копий ДНК, и имеет модульную конструкцию для размещения нового содержания генов. Два различных типа аналитически определенных элементов управления обеспечивают контроль качества и точности вызова помочь защитить с клинически соответствующих образцов. Гибкий "тег" шаг PCR встраивает специфичные для платформы адаптеры и коды индекса, чтобы образец Barcoding и совместимость с обычными инструментами NGS настольных. Важно отметить, что протокол упрощен и может производить 24 последовательности готовых библиотек в один день. Наконец, подход связывает влажные и сухие стендовые процессы путем включения результатов контроля предварительного аналитического качества образца непосредственно в вариант вызова алгоритмов для повышения точности обнаружения мутации и дифференцировать ложноотрицательных и indeterminate звонки. Этот метод целевой NGS использует достижения в области как Wetware и программного обеспечения для достижения высокой глубины, мультиплексированный секвенирование и чувствительный анализ гетерогенных образцов рака для диагностических применений.
Введение
Прецизионный медицина опирается на индивидуализацию диагностических и терапевтических возможностей для пациентов. Обещание адаптированных лечения является прямым следствием лучшего понимания путей заболевания, которые могут информировать связь молекулярной диагностики и целевых терапевтических средств. Например, использование молекулярно-целенаправленной терапии увеличилась с 11% до 46% с 2003 по 2013 год 1, а также препараты против рака , таких как Vemurafenib и crizotinib являются FDA-очищен с помощью диагностических тестов компаньонов. Благодаря своей способности точно восстанавливать цели последовательности низкого обилия по высоко мультиплексированных наборов образцов, секвенирования следующего поколения (NGS) возникла как метод выбора для оценки генетических аберраций, связанных с раком и идентификации молекулярных мишеней для точной медицины.
Наиболее распространенные твердые опухолевые биопсии для молекулярного тестирования включают в себя фиксированные формалином, парафин (FFPE) и тонкоигольной аспирации (FNA) specimепз. Эти образцы чреваты низким количеством и / или низкого качества нуклеиновых кислот , которые бросают вызов точным NGS оценок 2-5. Современные методы коммерческого NGS для анализа этих образцов основаны на путанице различных реагентов, протоколов и средств информатики, которые представляют собой движущиеся цели по постоянному совершенствованию. Например, изменения в аналитических и химических / или программного обеспечения произошло через каждые 1-2 месяца для наиболее часто используемых целевых NGS наборов 6. Эта неустойчивость отражает отсутствие согласованности в построении и проверке единой системы NGS для сложных типов образцов, в частности, для тестирования рака, и ставит чрезмерную нагрузку на лаборатории по разработке липких протоколы, которые оптимизированы от образца к результатам. Действительно, один недавний опрос пользователей NGS обратили внимание на трудности этих "быстро меняющихся" технологий, наряду с требованиями к установленным, с медицинской точки зрения содержания, Осуществимое укоренившейся опыт биоинформатики, A solidifiред и интегрированная процедура , которая может быть быстро реализованы, и упорядоченные рабочие процессы и упрощенные протоколы , которые облегчают обучение по месту работы 7. В этой статье, комплексная система для целевого NGS описано, что устраняет эти пробелы.
Представленная методология включает в себя все процедурные этапы от предварительной аналитической пост-аналитической на мокрой и сухой скамейками для повышения точности, чувствительности и надежности целевой количественной оценки и обнаружения для NGS клинически-соответствующего гена рака локусов. Этот подход начинается с количественного определения «функциональной» ДНК 4 для оценки качества ДНК, направлять вклад в стадию обогащения ПЦР, и защиты от ложноположительных вызовов , которые могут возникнуть в результате допроса очень низких копий шаблона. Одна трубка мультиплекс ПЦР затем обогащают для 46 локусов в 21 генов рака с использованием только 400 амплифицируемая копий ДНК, с последующим введением конкретных платформ последовательностей для NGS USINг общих настольных секвенирования инструментов. Библиотеки очищают с помощью простого магнитного шарика процедуру и количественно с новой, без калибровки КПЦР анализа. Автономный биоинформатики люкс, сообщил результаты выборочных ДНК QC для повышения производительности вызовов, обеспечивает анализ последовательности следующие NGS. Мы представляем данные, используя этот системный подход для целевого NGS выявить базового замещения мутации, вставки / делеции (вставкам), а также количество копий вариантов (ВКК) в низкокачественных и опухолевой низкой количество биопсий, таких как FFPE и образцы Фна и бега управления.
протокол
Примечание: Этот протокол описывает одновременную обработку образцов с использованием системы MiSeq NGS, но может быть адаптирован для Персональный Геном машины (PGM) прибора. Для получения рекомендованного минимального ввода ДНК 400 усиливаемых копий шаблона, анализ способен производить по крайней мере, 3,000x медианный охват для каждого из 96 образцов в NGS перспективе, и эквивалентную глубину покрытия на 24 образцов с помощью PGM на 318 чипе. Способ также требует использование ПЦР в реальном времени инструмента.
1. ДНК Функциональная Количественное и контроль качества (QC)
- Оттепель реагенты: 2x Master Mix, Грунтовка Probe Mix, Ингибирование Праймер Probe Mix, 6-карбокси-X-родамин (ROX), разбавителем, и четыре человека геномные калибровочных стандартов ДНК кривой (ДНК Стандартные (50 нг / мкл), ДНК Стандартные (10 нг / мкл), стандартной ДНК (2 нг / мкл), и ДНК - стандарт (0,4 нг / мкл)) (таблица 1). Vortex все реагенты в течение 10 секунд и центрифуге при максимальной скорости в течение 10 секунд, чтобы собрать содержимое.Держите 2x мастер смеси на льду.
- Подготовьте достаточное количество основной смеси для общего количества образцов, подлежащих испытанию, и включают в себя 10% больше объема, чтобы избежать дефицита в связи с пипеткой. Подготовка мастер-микс в микроцентрифужных трубки, используя следующие объемы на пробу: 5 мкл 2х Master Mix, 0,5 мкл Грунтовка Probe Mix, 0,5 мкл Ингибирование Праймер Probe Mix, 0,05 мкл ROX и 2,95 мкл Разбавитель. Вихревые в течение 10 сек и центрифуге при максимальной скорости в течение 10 сек, чтобы собрать содержимое.
- Добавить 9 мкл мастер смеси в лунки 96-луночного планшета.
- Добавить 1 мкл Стандартов ДНК в двух экземплярах для создания калибровочной кривой. Смешайте с помощью пипетки вверх и вниз 5 раз.
- Убедитесь, что образец нуклеиновой кислоты хорошо перемешан перед использованием. Добавить образец 1 мкл к основной смеси и перемешать с помощью пипетки вверх и вниз 5 раз.
- Уплотнение пластины, вихрь в течение 10 сек и центрифуге при максимальной скорости в течение 10 секунд, чтобы собрать содержимое.
- Поместите планшет в PCR System. Назначают как FAM (функциональный) и квантификации VIC (функциональное ингибирование) детекторов для каждого образца в соответствии с инструкциями изготовителя. Выполнение циклов ПЦР 10 мин при 95 ° С, и 40 циклов (15 с при 95 ° С, 1 мин при 60 ° C).
- Анализ данных КПЦР путем генерации линейной регрессии участка каждой из дублирующих стандартов ДНК с использованием программных протоколов.
- Участок журнала 10 из числа копий для каждого стандарта ДНК на оси абсцисс и соответствующее FAM с = Q значения на оси у.
- Убедитесь, что результаты из нуклеиновой образца кислоты попадают в динамическом диапазоне калибровочной кривой ДНК-стандартами, а затем рассчитать концентрацию неизвестной ДНК в "функциональной" или амплификаторного числом копий на мкл из соответствующей позиции на исходной стандартной кривой. на рисунке 1 показаны примеры кривых калибровки , которые прошли и потерпели неудачу.
- Определить, если усиление происходит в каждой реакции путем проверки на наличие нечеловеческого мишени ампликона в канале VIC.
Примечание: В качестве положительного контроля, ингибировании Праймер зонд смесь содержит праймерами, специфичными к отличному от человека экзогенного мишени и соответствующим шаблоном. Ингибирование Праймер Probe Mix является составной частью основной смеси, которая добавляется к каждой реакции, в том числе контроль не-шаблона (NTC). В отсутствие ингибитора, ПЦР-продукт для нечеловеческого цели всегда должны быть обнаружены в канале VIC. "Необнаруженным" С Q для образца в канале VIC указывает на наличие ингибиторов ПЦР , которые могут получить выгоду от последующей очистки образца перед дальнейшей обработкой.
2. Подготовка Библиотека: Гено-специфические (GS) ПЦР
- Подготовьте достаточное количество основной смеси для общего количества образцов, подлежащих испытанию, и включают в себя 10% больше объема, чтобы избежать дефицита из-за pipettING. Подготовьте основной смеси , включающей Г.С. ПЦР в микроцентрифужных трубки с использованием следующих объемов на образце: 5 мкл 2x амплификация Master Mix (Таблица 1) и 1 мкл Пань Рак Праймер панель (таблица 1). Смешайте с помощью пипетки вверх и вниз, вихрь в течение 10 сек и центрифуге при максимальной скорости в течение 10 секунд, чтобы собрать содержимое.
- Алиготе 6 мкл Г.С. ПЦР основной смеси в лунки 96-луночного планшета. Добавляют 4 мкл каждого образца нуклеиновой кислоты в отдельные лунки. Для других скважин, добавьте 4 мкл Control FFPE (таблица 1), 4 мкл управления многовариантность (таблица 1) и 4 мкл нуклеазы без воды для процедурного NTC. Для каждого добавления, перемешать с помощью пипетки вверх и вниз 5 раз.
- Уплотнение пластины, вихрь в течение 10 сек и центрифуге при максимальной скорости в течение 10 секунд, чтобы собрать содержимое.
- Поместите пластину в амплификатор для следующих циклов ПЦР: 5 мин при 95 ° С,2 циклов (15 с при 95 ° С, 4 мин при 60 & deg; С), 23 циклов (15 с при 95 ° С, 4 мин при 72 ° С), а окончательное расширение 10 мин при 72 ° С. Выдержка при температуре 4 ° С.
Примечание: После завершения стадии 2.4, пластина будет упоминаться в качестве обкладки Г.С. ПЦР.
3. Подготовка Библиотека: Tag PCR
- Оттепель реагенты: 2x Индекс Master Mix (Таблица 1) и коды индексов (таблица 1). Вихревые в течение 10 сек и центрифуге при максимальной скорости в течение 10 сек, чтобы собрать содержимое.
Примечание: Index Коды предварительно смешивают, чтобы обеспечить уникальный набор попарно индексов (штрих-коды) для каждого образца. - В 96-луночного планшета, добавить 7,5 мкл Индекс 2x Master Mix и 5,5 мкл кода индекса на указанный хорошо и перемешать с помощью пипетки вверх и вниз 5 раз.
- Осторожно откройте пластину GS PCR, и добавьте 2 мкл ПЦР-продукта GS на новой пластинке с мастер-микс. Смешайте с помощью пипетки вверх и вниз 5 раз, Для каждого образца записывают идентификатор образца и соответствующие попарные коды индекса. Уплотнение пластины, вихрь в течение 10 сек и центрифуге при максимальной скорости в течение 10 секунд, чтобы собрать содержимое.
- Поместите пластину в амплификатор и ПЦР в течение 5 мин при 95 ° С, 10 циклов (30 с при 95 ° С, 30 с при 55 ° С, 1 мин при 72 & deg; С) и конечным удлинением 10 мин при температуре 72 ° С. Выдержка при температуре 4 ° С.
Примечание: После завершения стадии 3.4, пластина будет упоминаться как тег PCR пластины.
4. Библиотека Очистка и выбор размера
- Удалить магнитных шариков Библиотека Чистая Prep (таблица 1) и буфера для элюции (таблица 1) от 2-8 ° C и позволяет уравновешиваться до комнатной температуры в течение 30 мин. Добавить 9,6 мл 100% этанола в промывочный буфер (таблица 1) контейнера, крышкой и взболтайте Флакон несколько раз.
- Vortex магнитные шарики в течение 10 секунд и добавить 11 мкл в отдельные лунки 96-луночный планшет.
- Откройте Tag PCR тарелку и добавить 10 мкл Tag продукта ПЦР к шарикам и смеси пипетки 5 раз. Выдержите смесь в течение 4 мин при комнатной температуре.
- Поместите 96-луночного планшета на магнитной подставке (таблица 1) в течение 4 мин. С 96-луночный планшет еще находится на стенде, удалить и отбросить супернатант с пипеткой.
- Удалите 96-луночного планшета с магнитной подставкой и добавляют 100 мкл этанола, содержащего промывочного буфера в каждую лунку и перемешать с помощью пипетки вверх и вниз 5 раз. Инкубировать в течение 2 мин.
- Поместите 96-луночного планшета на магнитной подставке в течение 2 мин, затем снимите и выбросьте супернатант с пипеткой.
- Повторите шаг 4.5, в общей сложности на 2 промывок этанол, удаляя так много промывочного раствора возможно после второй стирки.
- С 96-луночного планшета на магнитной подставке, сушки гранул в течение 2 мин при комнатной температуре, а затем снимите пластину с подставки.
- Ресуспендируют бусин путем добавления 20 мкл буфера для элюции для каждого достл и пипетку вверх и вниз 5 раз.
- Инкубировать в течение 2 мин при комнатной температуре.
- Поместите 96-луночный планшет обратно на магнитной подставке в течение 4 мин и осторожно снять и передать 18 мкл прозрачного супернатанта в новую скважину.
Примечание: Эта процедура может быть безопасно остановлен на этой стадии, и образцы хранили при температуре от -15 до -30 ° С. Для повторного запуска, оттаивать замороженные образцы на льду, прежде чем продолжить.
5. Количественное библиотека
- Оттепель библиотека Quant (LQ) реактивы: 2x LQ Master Mix, LQ Primer / Probe Mix, LQ Standard, LQ Положительный контроль, LQ Разбавитель и LQ ROX (таблица 1). Вихревые в течение 10 сек и центрифуге при максимальной скорости в течение 10 сек, чтобы собрать содержимое.
- С использованием очищенных продуктов библиотеки, выполняют серийное разведение каждого образца в LQ разбавителем.
- Добавьте 2 мкл (библиотека очищенного продукта) до 198 мкл LQ Разбавителя и смешать вверх-вниз с пипеткой 10 раз.
- Добавляют 2 мкл (разведение 1: 100) до 198 мкл LQ Разбавитель и смешать вверх и вниз с пипеткой 10 раз.
- Подготовьте достаточное количество LQ мастер-смеси для общего количества образцов, подлежащих испытанию, и включают в себя 10% больше объема, чтобы избежать дефицита в связи с пипеткой. Подготовьте LQ Master Mix в микроцентрифужных трубки, используя следующие объемы на пробу: 5 мкл 2X LQ Master Mix, 2 мкл LQ Primer / зонда Mix, 0,5 мкл LQ Стандартные и 0,5 мкл LQ ROX. Смешайте с помощью пипетки вверх и вниз, вихрь в течение 10 сек и центрифуге при максимальной скорости в течение 10 секунд, чтобы собрать содержимое.
- Добавить 8 мкл мастер-LQ смесь в лунку оптической 96-луночного планшета.
- В отдельных скважинах, добавьте 2 мкл разведенного библиотеки, 2 мкл LQ Положительный контроль и 2 мкл LQ Разбавитель (NTC) и перемешать с помощью пипетки вверх и вниз 5 раз. Уплотнение пластины с оптической клейкой пленкой, вихря на 10 сек и центрифуге при 400 мкг в течение 10 секунд, чтобы собрать содержимое.
- Назначают как FAM и VIC DetectoRS для каждого образца. Выполните ПЦР-амплификации с использованием велосипедного условия 5 мин при 95 ° С, и 40 циклов (15 секунд при 95 & deg; С, 1 мин при 60 ° C).
- Определить концентрацию каждого образца (нм) с использованием сравнительного метода С Q. Вычислить разность известного LQ Standard (C д ВМЦ) к неизвестной библиотеке (C д FAM). Концентрация разбавленного образца (рм) рассчитывается по следующей формуле:
[Конц Lib] Nm = 12,5 х 2 Δ Cq
При использовании коэффициента разбавления, кроме 10000, вычислить отношение коэффициента разбавления мишени до 10000, и умножить этот фактор в результате уравнения в 5.7.
6. Библиотека Нормализация и образец Аккумулирование
- Определить медиану концентрацию (нМ) во всех образцах (каждая из которых содержит уникальный парный индекс) должны быть объединены.
- Определить индивидуальный образец Volumе (мкл) для объединения путем умножения средняя концентрация во всех пробах на 5, а затем разделив его индивидуальной концентрации (нМ). Вокруг полученное значение до ближайшего целого числа. Круглые объемы со значениями <2 мкл до 2 мкл, а объем> 15 мкл до 15 мкл.
- Добавьте нормализованный объем (мкл) для каждого образца к одной трубки микроцентрифужных для создания пула образца.
- Вычислить новую концентрацию для каждого образца с использованием округлых целочисленных значений и записи результатов.
- Для того, чтобы определить концентрацию пула пробы, вычислить сумму всех индивидуальных концентраций и записать полученное значение (нм).
- Развести бассейн образца до 1,25 нм с использованием секвенирования раствора для разведения (таблица 1).
Примечание: Эта процедура может быть безопасно остановлен на этой стадии, и образцы хранили при температуре от -15 до -30 ° С. Для повторного запуска, оттаивать замороженные образцы на льду, прежде чем продолжить.
7. Секвенирование
- DenatЮр пул образец в присутствии v3 PhiX Control (таблица 1), добавив следующие: 15 мкл 1,25 нМ Sample бассейн, 3 мкл 0,5 нМ PhiX и 2 мкл 1 N NaOH. Вихревой кратко с последующим быстрым центрифугированием и инкубировали в течение 5 мин при комнатной температуре.
- Поместите денатурированный бассейн образец на льду.
- Добавить 8 мкл денатурированного библиотеки 992 мкл предварительно охлажденного буфера HT1-Hyb в микроцентрифужных трубки. Вихревой кратко перемешать, а затем кратким центрифугированием для сбора содержимого. Держите на льду.
- Добавьте 600 мкл денатурированного и разбавленного библиотеки в положение # 17 картриджа с реагентом.
- Оттепель прочитанной 1 праймеры для секвенирования (Таблица 1), индекс прочитанной праймеры для секвенирования (Таблица 1) и читать 2 праймеры для секвенирования (таблица 1). В микропробирок, отдельно развести 4 мкл праймеры для секвенирования с 636 мкл буфера HT1-Hyb. Примечание: HT1-Гибр буфер при условии, остроумиеч набора для секвенирования реагент (таблица 1).
- Смешайте встряхиванием в течение 10 сек и центрифуге при максимальной скорости в течение 10 секунд, чтобы собрать содержимое. Добавьте 600 мкл разбавленных Читать 1 праймеры для секвенирования в позиции № 18 картриджа секвенирование реагента, 600 мкл разбавленных Index Read грунтовок на позицию # 19, и 600 мкл разбавленных Read 2 праймеры для секвенирования в положение # 20.
- Загрузите реагенты на инструменте NGS (таблица 1) и последовательности в соответствии с инструкциями изготовителя. Выполните парноконцевое 2 х 150 циклов секвенирования перспективе.
Анализ 8. Данные
Примечание: Прибор программное обеспечение NGS преобразует изображения кластера в базовых вызовов и показателей качества, и демультиплексирует попарные индексы для создания отдельных GZIP сжатых FASTQ (* .fastq.gz) файлы для каждого образца. До анализа демультиплексированные файлов, читатель должен загрузить и установить программное обеспечение , связанное биоинформатики (таблица1). Программное обеспечение может быть установлено на потребительского класса ПК с Windows и не требует специализированных вычислительных аппаратных средств или подключение к Интернету для выполнения анализа данных.
- Дважды щелкните значок на рабочем столе программного обеспечения.
- Войдите в систему, используя имя пользователя и пароль, указанные в руководстве по программному обеспечению.
- Откройте панель проекта, и нажмите кнопку "Создать проект".
- Назовите проект и предоставить дополнительное описание проекта. Выберите целевой NGS тип панели и NGS тип инструмента. Нажмите кнопку "Сохранить и продолжить".
- Загрузите сжатые файлы FASTQ для прямого и обратного чтения. Не загружать "нераспределенные" FASTQs, которые содержат говорится, что не в состоянии демультиплексирования. Нажмите кнопку "Сохранить и продолжить".
- Введите число функциональных входных копий, используемых для получения каждой библиотеки, как определено с помощью ДНК-анализа функциональной количественной оценки (этап 1 выше). Вручную добавить значения или скопировать и вставить значения из распространениялист в таблицу аннотаций. Нажмите кнопку "Сохранить и продолжить".
- Обзор аннотированные библиотеки загружены для анализа и нажмите кнопку "Отправить анализ", чтобы начать анализ.
- Контроль за ходом анализа отображается через панель управления проекта.
Примечание: Индикация полного состояния анализ представлен, когда результаты будут готовы к рассмотрению. - Просмотрите Проанализировав результаты для выборочных показателей QC включая полное освещение в библиотеке, процент чтений, проходящих фильтров, глубину охвата ампликона и однородность. Просмотрите вариант вызовов для каждой библиотеки секвенировали с dbSNP, КОСМИЧЕСКИХ, 1000 геномов и других источников функционального и населения аннотацию уровня.
- Экспорт необработанных результатов в виде таблиц Резюме работы с электронными таблицами, * .bam файлы и * .vcf файлов для длительного хранения или вниз по течению анализа с помощью взаимодополняющих средств информатики.
Результаты
В общей сложности 90 образцов (74 уникальных), представляющих положительные и отрицательные элементы управления, ранее характеризуемые клеточных линий, а также остаточные клинические опухолевые биопсии FFPE оценивали укрупнения ДНК, ввод в мультиплексной ПЦР обогащения, помеченным адаптеров последовательности, Barcoded и анализировали в одном NGS настольный инструмент запуска (рисунок 2) , который произвел 19.1 M считывает прохождение фильтра. Эквимолярные образец объединения привели к высокой глубины секвенирования (3,692x читает) и равномерное покрытие (97,8% от ампликонов, охватываемых в 5-кратном средней глубины чтения). Выпадающие не состоит ни одного -шаблонов элементов управления, одна клеточная линия ДНК с большим количеством копий усиления, и одна ДНК FFPE, помеченный для ингибирования ПЦР с помощью анализа QC предварительной аналитической (рисунок 3). Равномерность покрытия через 46 ампликонов поддерживалась с использованием трех различных операторов (рис 4а), а также для различных образцов ДНК FFPE низкого качества ( Рисунок 4B). Контрольную ДНК Опухоль FFPE, сформулированный из смеси остаточных клинических образцов для достижения 5% BRAF V600E (количественно капельным цифровой ПЦР), как сообщается, целевой BRAF мутацию в содержаний 3,9, 5,3 и 6,5% тремя операторами с использованием вход 400 усиливаемых копий (и , таким образом , только 20 мутантных копий) (рис 4В и "FFPE3" закладных таблицы). Кроме того, смесь из 12 синтетических ДНК - матриц, каждая из которых представляет собой известную "драйвер" база-заместительную мутации, показал ожидаемые мутации в планируемом диапазоне 9 - 17% средней частоты аллеля (таблица 2). Разбавление клеточной линии и образцы ДНК FFPE с числом копий усилений продемонстрирована доза-зависимость для вариантов в EGFR и KRAS, соответственно (рисунок 5). Важно отметить, что ввод ДНК FFPE может быть уменьшена до всего лишь 50 копий или усиливаемых 1.2 нг объемной ДНК, сохраняя при этом обнаружение известных мутаций без ложноположительныхвызовы (Рисунок 6). Входы ДНК были размещены над 100-кратном диапазоне до по меньшей мере 50000 усиливаемых копий (таблица 3). В этом и связанных с ними экспериментов, вариант вызывает у 22 FFPE и 20 образцов FNA было зарегистрировано в соответствии с независимыми методами с общим охватом мутаций (таблица 4).
Чувствительность и положительное прогностическое значение для анализа был определен из анализа 97 образцов, в том числе FFPE, FNA, свежемороженой и клеточной линии ДНК, и в общей сложности 195 результатов секвенирования. Результаты показали 365 истинных положительных вызовов вариант, 4 ложноотрицательных звонки и 1 ложный положительный призыв к чувствительности 98,9% (95% ДИ: 97.1-99.7%) и положительная прогностическая ценность (PPV) 99,7% (95% ДИ : 98.2-99.99%). Анализы вставкам были выполнены для двух вариантов общей EGFR (p.E746_A750delELREA и p.V769_D770insASV) в 33 выборочных трасс, демонстрируя чувствительность 93,9% (95% ДИ: 78.4-98.9%) и PPV 100% (95% ДИ: 86.3-100%) с вариантами обнаружен по диапазону 2.4-84.8%.
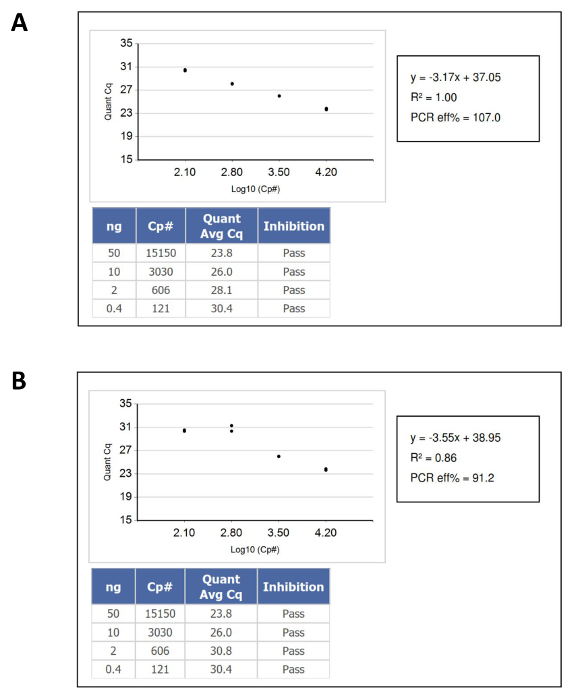
Рисунок 1: Пример кривых ДНК Количественное калибровки, прошедшими и не прошедшими КК Критерии (А) Проходящий стандартной кривой.. (B) провальную стандартной кривой. В этом случае, неисправность была вызвана дубликата пипеткой самого низкого стандартного ввода ДНК. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
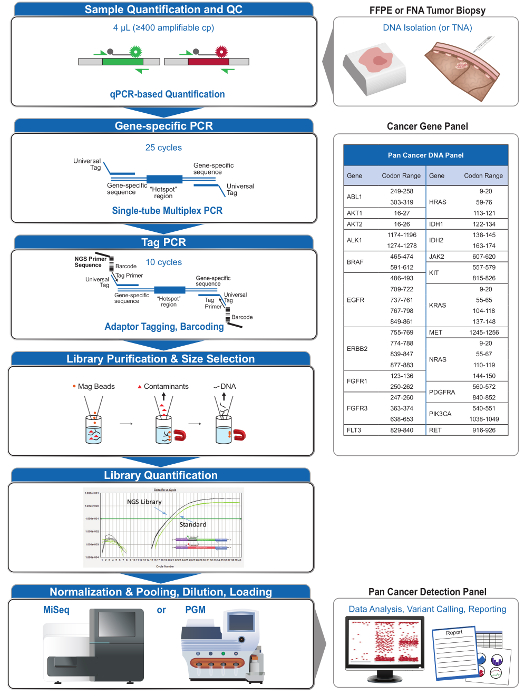
Рисунок 2: Обзор всеобъемлющей Целевые NGS системы для онкологии Приложения, Интегрируется Pre-аналитическаяАль-аналитическая и пост-аналитические рабочие процессы. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
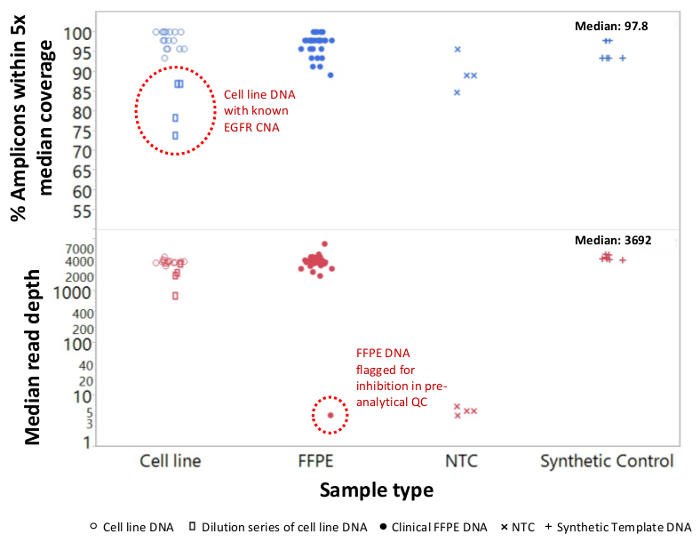
Рисунок 3: Чтение Покрытие и единообразие для борьбы с целевыми NGS генов рака в ДНК FFPE низкого качества по сравнению с интактными ДНК клеточных линий и контроля в общей сложности 90 образцов , которые включали остаточную клиническую FFPE (закрытые кружки), клеточная линия (открытые кружки. ), и синтетические матричной ДНК (плюс символы) были обработаны с использованием одной пробирке, 21-ген мультиплекс ПЦР по обогащению урана. Каждая библиотека ампликона был помечен с адаптером последовательностей для инструмента NGS, Barcoded с отличным кодом двойного индекса, очищают, количественно и нормированы к концентрации 2,5 нМ. Библиотеку ДНК секвенировали и анализировали с помощью программного обеспечения компаньон биоинформатики. Примеры девиations включал смесительный серию MDA-MB-468 клеток линии ДНК, несущий большое EGFR число копий усиление (сверху, открытые прямоугольники в пределах пунктирного круга), что искаженные однородность покрытия, и один образец меланома FFPE, что не удалось создать заметное число чтений из-за с выносом ингибиторов ПЦР из экстракции ДНК. Провал меланома образца (нижний, пунктирный круг) было предсказано заранее аналитического анализа КПЦР ДНК QC. NTC, не-шаблон управления (х символов). Пожалуйста, нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
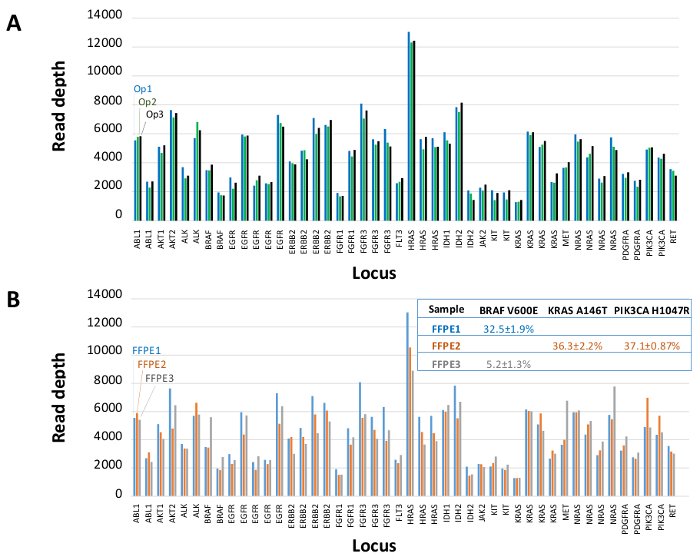
Рисунок 4: Amplicon-на-ампликонов Read Охват, однородность и вариант обнаружения остаточной клинической опухолевой ДНК FFPE. (A) Прочитайте охват по всей обогащенном локусов в представительном образце ДНК FFPE измереннойна трех различных операторов. Оператор 1, Op1 (синие полосы); Оператор 2, Op2 (зеленые столбики); Оператор 3, Op3 (черные столбики). (B) по однородности покрытия и вариантные вызовов оцениваемые с использованием трех FFPE образцов опухолей, в том числе контрольной смеси (FFPE3 серые полосы и текст) , состоящий из известного 5% BRAF c.1799T> мутацией. FFPE1 (синие полосы и текст), FFPE2 (оранжевые полосы и текст). Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
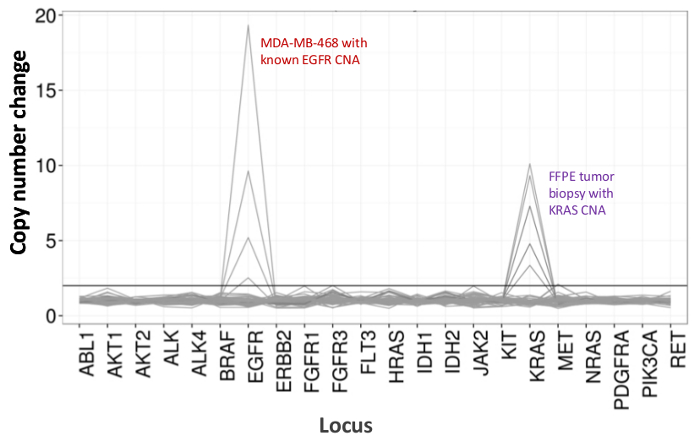
Рис . 5: Дозозависимый Обнаружение числа копий Варианты в клеточной линии и FFPE ДНК MDA-MB-468 клеток линии ДНК с хорошо охарактеризованного EGFR число копий амплификации постепенно разводили в фоне эталонного немутантный клетки -line ДНК, чтобы проиллюстрировать уменьшение копии изменения числа в видеФункция разбавления. Процентное содержание каждого образца ДНК клеточной линии показана с отдельной линией (0, 12,5, 25, 50 и 100%). Разбавление яичника образца опухоли FFPE с известным KRAS усиления показал аналогичный профиль , используя один и тот же серии титрование , но с повторах 100% ДНК FFPE для двух верхних линий. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
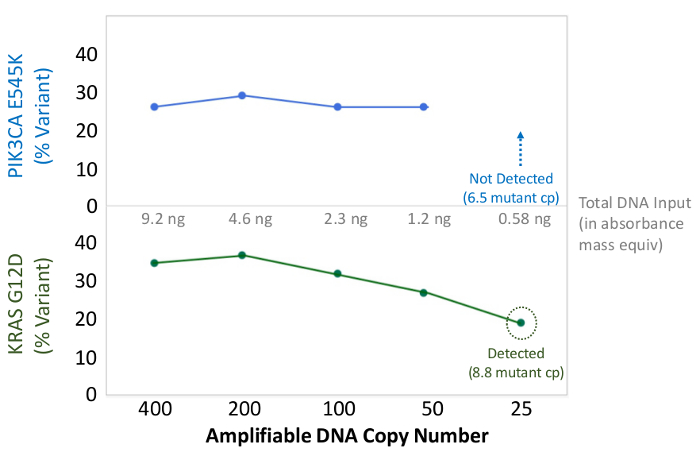
Рисунок 6: Обнаружение Точная Мутация и Количественная 50 копий амплифицируемая FFPE ДНК или 1,2 нг Bulk ДНК амплифицируемая число копий рака толстой кишки FFPE ДНК определяли с помощью анализа QC КПЦР основе, и разводили от 400 до 25 копий как. ввод в мультиплексной ПЦР обогащения перед секвенированием. Трубопровод биоинформатики правильно называют оба известного Varianт.с. до 50 копий или эквивалент ~ 10 мутантных шаблонов. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
| Наименование Материал / Материал | Компания | Номер каталога |
| 2x Quantidex Master Mix | Asuragen | 145345 |
| Quant Праймер Probe Mix | Asuragen | 145336 |
| Ингибирование Праймер Probe Mix | Asuragen | 145344 |
| ROX | Asuragen | 145346 |
| разжижитель | Asuragen | 145339 |
| ДНК-стандарт (50) | Asuragen | 145340 |
| ДНК-стандарт (10) | Asuragen | 145341 |
| ДНК-стандарт (2) | Asuragen | 145342 |
| ДНК-стандарт (0.4) | Asuragen | 145343 |
| 2x Amplification Master Mix | Asuragen | 145348 |
| Панель Pan Рак Primer | Asuragen | 145347 |
| Контроль Pan Рак FFPE | Asuragen | 145349 |
| Управление Pan Рак многовариантность | Asuragen | 145350 |
| Библиотека Pure Бисер Prep | Asuragen | 145351 |
| Промывочный буфер | Asuragen | 145352 |
| элюирующего буфера | Asuragen | 145353 |
| 2x LQ Master Mix | Asuragen | 145358 |
| LQ Разбавитель | Asuragen | 145354 |
| LQ Положительныйконтроль | Asuragen | 145355 |
| LQ Стандартный | Asuragen | 145356 |
| LQ Грунтовка / Probe Mix (ILMN) | Asuragen | 145357 |
| LQ ROX | Asuragen | 145359 |
| Индекс Коды (ILMN) - установка | Asuragen | 150004 |
| AIL001 - AIL048 (48) | ||
| Индекс Коды (ILMN) - Set B | Asuragen | 150005 |
| AIL049 - AIL096 (48) | ||
| 2x Индекс Master Mix | Asuragen | 145361 |
| Прочитайте 1-праймеры для секвенирования | Asuragen | 150001 |
| Индекс прочитанной праймеры для секвенирования | Asuragen | 150002 |
| Читать 2 праймеры для секвенирования | Asuragen | 150003 |
| Секвенирование Разбавитель | Asuragen | 145365 |
| Illumina MiSeq | Illumina | |
| MiSeq Реагент Kit v3 (600-цикл) | Illumina | MS-102-3003 |
| MiSeq Реагент Nano Kit v2 (300 циклов) | Illumina | MS-103-1001 |
| PhiX управления v3 | Illumina | FC-110-3001 |
| Магнитная подставка-96 (или аналогичное устройство) | Амбион | AM10027 |
| Quantidex Reporter Software | Asuragen |
Таблица 1:. Реагенты и наборы При первом использовании ROX, храните флакон при температуре 2-8 ° C. Не замораживать. Программное обеспечение можно загрузить по адресу www.asuragen.com.
| гена | КОСМИЧЕСКИЙ вариант | КОСМИЧЕСКИЙ аминокислота | % Вариант |
| NRAS | c.182A> G | p.Q61R | 13.3 |
| NRAS | c.35G> а | p.G12D | 15,2 |
| HRAS | c.182A> G | p.Q61R | 17,8 |
| HRAS | c.35G> а | p.G12D | 9.2 |
| KRAS | c.182A> G | p.Q61R | 13.5 |
| KRAS | c.35G> а | p.G12D | 19,1 |
| PIK3CA | c.1633G> а | p.E545K | 9.3 |
| PIK3CA | c.3140A> G | p.H1047R | 9.1 |
| КОМПЛЕКТ | c.2447A> T | p.D816V | 14.6 |
| EGFR | c.2369C> T | p.T790M | 11.3 |
| EGFR | c.2573T> G | p.L858R | 14,9 |
| BRAF | c.1799T> а | p.V600E | 17.3 |
Таблица 2: Обобщенный Синтетический управления состоит из 12 "Driver" Рак Генные варианты , которые количественно на 9-17% Изобилия Смесь из 12 различных двухцепочечных синтетических шаблонов несущих 12 различных мутаций оценивали следующие последовательности.. Все варианты были правильно вызваны без ложных срабатываний.
>% Вариант| образец ID | функциональная герц | Ген | КОСМИЧЕСКИЙ вариант | КОСМИЧЕСКИЙ аминокислота | Медиана чтения глубины | % В пределах 5х медианы | |
| BCPAP | 400 | BRAF | c.1799T> а | p.V600E | 99,5 | 3289 | 96% |
| BCPAP | 10000 | BRAF | c.1799T> а | p.V600E | 99,7 | 4040 | 98% |
| BCPAP | 25000 | BRAF | c.1799T> а | p.V600E | 99,4 | 3687 | 96% |
| BCPAP | 50000 | BRAF | c.1799T> а | p.V600E | 99,7 | 4611 | 93% |
Таблица 3: Покрытие и Variant Призвание сохраняются Над> 100-кратный диапазон входного ДНК. укрупнения ДНК из BCPAP клеточной линии был введен в мультиплексной ПЦР обогащения от 400 до 50000 копий и секвенировали. Читайте глубина, однородность покрытия, обнаружение мутации, и точность мутации были сохранены по всему диапазону входного сигнала.
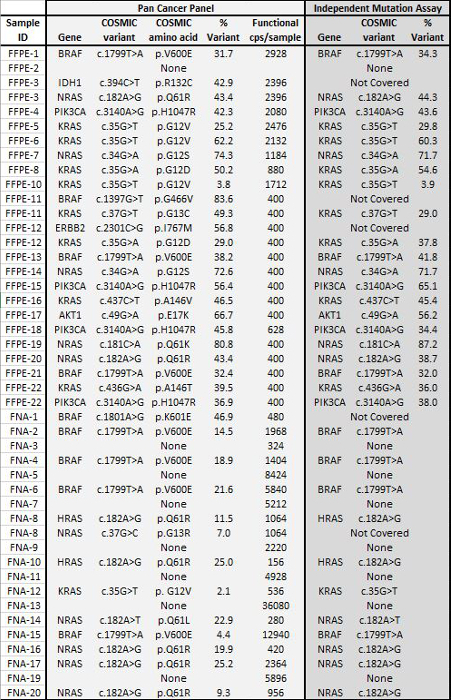
Таблица 4: Вариант Вызовы в 22 FFPE и 20 FNA Опухолевые Биопсию согласен с результатами независимых Мутация Assays Набор 22 опухолевой ДНК FFPE со статусом мутации ранее определенной ортогональными целевых NGS анализов был введен на 400 до 2928 усиливаемых копий в обогащении ПЦР. шаг и секвенировали с использованием 21-гена панели Pan рака. Кроме того, когорты из 20 образцов ДНК ТАБ ранее охарактеризованы с использованием жидкого шарика решетки мутация пробирного 8 подвергали ПЦР амплифицировали с использованием 156 до 36080 входных амплифицируемая копий и секвенировали. Все звонки между перекрывающихся панели Pan Рак NGS и refereМетоды NCE были согласны. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
Обсуждение
Технологии NGS пересмотрели ожидания опрашивая молекулярные профили опухолевых биопсий в клинических условиях 9. Ряд целевых групп NGS были разработаны в качестве научно - исследовательских технологий, лабораторных испытаний разработаны и коммерчески доступных продуктов и пользовательских панелей для оценки нескольких типов клинических образцов 3,5,10-15. Сообщения из нескольких исследований продемонстрировали значение NGS в качестве чувствительного и специфического клинического инструмента для выявления геномных изменений 3,10-12,14. Тем не менее , исследования также показали риск артефактов , которые могут привести к ложно-положительных результатов от сложных биопсий рака , таких как FFPE образцы 2-5,12,14. Кроме того, последние публикации выделены высокие показатели отказов для NGS с использованием онкология образцов 16 и ложноположительных звонки с коммерческими целевых групп NGS, которые усугубляются при использовании низкозатратного ДНК 12. В результате, некоторые Laboraтори либо модифицированы или добавлены дополнительные проверки и балансы коммерчески доступные целевые технологии NGS для повышения производительности, часто для обеспечения точности или подтверждения результатов от биоинформатики анализов 5,10,11.
21-ген панели (Рисунок 2) была разработана в качестве целевого контента для комплексной системы NGS допросить основанных на фактических данных, осуществимые мутации в сложных типах образцов , таких как FFPE и FNA опухолевых биопсий. Рабочий процесс имеет ряд преимуществ: 1) последовательность, обеспечивая равномерное покрытие ампликона (Рисунки 3 и 4, таблица 3); 2) простота в использовании, обеспечивая предварительно сформулировали, оптимизированные наборы реагентов, а также упрощение требований к биоинформатики; 3) коэффициент полезного действия путем рационализации процесса и сокращение количества шагов при использовании пипеток по сравнению с другими методами коммерческие NGS; и 4) точность путем включения анализа ДНК QC для оценки усилителювозможность ДНК количество копий , чтобы обеспечить приемлемое разнообразие шаблонов и избежать стохастических колебаний обнаружения варианта 4. Интеграция предварительно аналитических данных QC с анализом биоинформатики позволяет низким входам FFPE ДНК. Это было достигнуто путем обучения алгоритм принятия дерева с использованием различных функциональных копий входных ДНК через 400 образцов FFPE с независимыми мерами истины, и включение этого алгоритма в биоинформатики программного обеспечения. В результате, рекомендуемый ввод 400 усиливаемых копий, как правило , эквивалентно ~ 5-20 нг FFPE ДНК, выигрывает по сравнению с другими методами 10-12, включая обогащение гибридизацией на основе которых ~ 250 нг ДНК FFPE рекомендуется 17,18 , Хотя методика описана для использования на MiSeq платформе, он может быть модифицирован с помощью Tag праймеров для ПЦР с инструментальными конкретных адаптеров для проведения анализа последовательности на других платформах NGS.
Несколько шагов имеют решающее значение для обеспечения успехапроцедуры. Анализ QC перед аналитическим определяет укрупнения число копий функционального ингибирования ДНК и отчетов. Тем не менее, если он составляет менее 400 амплифицируемые копий ДНК используются на стадии обогащения ПЦР, существует повышенный риск ложноотрицательных вызова из образцов с низким обилием мутаций (рисунок 6). Кроме того, следует соблюдать осторожность во время очистки библиотеки для предотвращения пересушивания магнитных шариков во время стирки или элюции шагов. Кроме того, успешная библиотека Количественное сильно зависит от точного разведения библиотеки ДНК. Для достижения наилучшего результата, разность кПЦР результатов для библиотеки образцов (Cq FAM) по сравнению с LQ Standard (Cq VIC) должно быть ≤3.3 Cq. Если разность больше, чем 3,3 Сч, повторное разбавление и испытание образца рекомендуется. Несмотря на то, отличная корреляция наблюдается между этим методом конкурентного КПЦР и коммерческих комплектов, которые предлагают абсолютное количественное определение с использованием стандартной кривой, Смещение входа библиотеки в клональной стадии амплификации по сравнению с другими методами, которые могут потребоваться для достижения оптимальной плотности посева.
Некоторые образцы рака особенно сложной задачей для последовательности из-ингибиторов, которые сохраняются после выделения ДНК. Для идентификации этих образцов до подготовки библиотеки, анализ КПЦР QC также обнаруживает ингибирование амплификации путем включения экзогенного шаблон, который служит в качестве внутреннего контроля и часовому для функционального торможения. Пример представлен на рисунке 3 , в котором образец ДНК меланома не смогла пройти торможение QC метрики перед секвенированием и затем не удалось создать библиотеку , которая может быть секвенировали. Неудача, вероятно, является следствием загрязнения меланина, известный ингибитор ПЦР, перенесенных из стадии выделения FFPE ДНК. Образцы, которые идентифицированы с помощью анализа QC, чтобы быть в опасности для отказа амплификации может быть спасена через дополнительную очистке шаг то удаления потенциальных ингибиторов.
Целевая группа 21-ген фокусируется на основе фактических данных генов горячих точек и обеспечивает полную систему с оптимизированными реагентами и управления для ДНК QC, NGS и биоинформатики программного обеспечения, которое сообщается путем предварительного анализа «функциональных» результатов Количественное определение ДНК. Метод точно обнаруживает базового замещения мутаций и вставкам из низкозатратного ДНК, и представляет собой пример системы NGS с возможностью расширения содержимого панели, чтобы обнаружить дополнительные варианты, такие как ВКК и быть адаптированы для целевой РНК последовательности.
Раскрытие информации
JH, AH, RZ, ВСН и GJL являются служащими и имеют право собственности на акции в Asuragen, Inc. Р.З., МПБ и GJL являются соавторами по патентной заявке для улучшения вариант призывающую с использованием информации укрупнения числа копий определяется для каждого образца.
Благодарности
Мы благодарим доктора Аннетт Schlageter на рассмотрение рукописи. Эта работа была частично поддержана грантом CP120017 от профилактики рака и Научно-исследовательского института Техаса (PI: GJL).
Материалы
| Name | Company | Catalog Number | Comments |
| 2x Quantidex Master Mix | Asuragen | 145345 | |
| Quant Primer Probe Mix | Asuragen | 145336 | |
| Inhibition Primer Probe Mix | Asuragen | 145344 | |
| ROX | Asuragen | 145346 | |
| Diluent | Asuragen | 145339 | |
| DNA Standard (50) | Asuragen | 145340 | |
| DNA Standard (10) | Asuragen | 145341 | |
| DNA Standard (2) | Asuragen | 145342 | |
| DNA Standard (0.4) | Asuragen | 145343 | |
| 2X Amplification Master Mix | Asuragen | 145348 | |
| Pan Cancer Primer Panel | Asuragen | 145347 | |
| Pan Cancer FFPE Control | Asuragen | 145349 | |
| Pan Cancer Multi-Variant Control | Asuragen | 145350 | |
| Library Pure Prep Beads | Asuragen | 145351 | |
| Wash Buffer | Asuragen | 145352 | |
| Elution Buffer | Asuragen | 145353 | |
| 2X LQ Master Mix | Asuragen | 145358 | |
| LQ Diluent | Asuragen | 145354 | |
| LQ Positive Control | Asuragen | 145355 | |
| LQ Standard | Asuragen | 145356 | |
| LQ Primer/Probe Mix (ILMN) | Asuragen | 145357 | |
| LQ ROX | Asuragen | 145359 | |
| Index Codes (ILMN) - Set A, AIL001 - AIL048 (48) | Asuragen | 150004 | |
| Index Codes (ILMN) - Set B, AIL049 - AIL096(48) | Asuragen | 150005 | |
| 2x Index Master Mix | Asuragen | 145361 | |
| Read 1 Sequencing Primers | Asuragen | 150001 | |
| Index Read Sequencing Primers | Asuragen | 150002 | |
| Read 2 Sequencing Primers | Asuragen | 150003 | |
| Sequencing Diluent | Asuragen | 145365 | |
| Illumina MiSeq | Illumina | ||
| MiSeq Reagent Kit v3 (600-cycle) | Illumina | MS-102-3003 | |
| MiSeq Reagent Nano Kit v2 (300-cycle) | Illumina | MS-103-1001 | |
| PhiX Control v3 | Illumina | FC-110-3001 | |
| Magnetic Stand-96 (Or equivalent device) | Ambion | AM10027 | |
| Quantidex Reporter Software | Asuragen |
Ссылки
- Chen, G., Mosier, S., Gocke, C. D., Lin, M. T., Eshleman, J. R. Cytosine deamination is a major cause of baseline noise in next-generation sequencing. Mol Diagn Ther. 18 (5), 587-593 (2014).
- Choudhary, A., et al. Evaluation of an integrated clinical workflow for targeted next-generation sequencing of low-quality tumor DNA using a 51-gene enrichment panel. BMC Med Genomics. 7, 62 (2014).
- Sah, S., et al. Functional DNA quantification guides accurate next-generation sequencing mutation detection in formalin-fixed, paraffin-embedded tumor biopsies. Genome Med. 5 (8), 77 (2013).
- Zhang, L., et al. Profiling cancer gene mutations in clinical formalin-fixed, paraffin-embedded colorectal tumor specimens using targeted next-generation sequencing. Oncologist. 19 (4), 336-343 (2014).
- Latham, G. J. Next-generation sequencing of formalin-fixed, paraffin-embedded tumor biopsies: navigating the perils of old and new technology to advance cancer diagnosis. Expert Rev Mol Diagn. 13 (8), 769-772 (2013).
- Crawford, J. M., et al. The business of genomic testing: a survey of early adopters. Genet Med. 16 (12), 954-961 (2014).
- Smith, D. L., et al. A multiplex technology platform for the rapid analysis of clinically actionable genetic alterations and validation for BRAF p.V600E detection in 1549 cytologic and histologic specimens. Arch Pathol Lab Med. 138 (3), 371-378 (2014).
- Thomas, F., Desmedt, C., Aftimos, P., Awada, A. Impact of tumor sequencing on the use of anticancer drugs. Curr Opin Oncol. 26 (3), 347-356 (2014).
- Singh, R. R., et al. Clinical validation of a next-generation sequencing screen for mutational hotspots in 46 cancer-related genes. J Mol Diagn. 15 (5), 607-622 (2013).
- Beadling, C., et al. Combining highly multiplexed PCR with semiconductor-based sequencing for rapid cancer genotyping. J Mol Diagn. 15 (2), 171-176 (2013).
- McCall, C. M., et al. False positives in multiplex PCR-based next-generation sequencing have unique signatures. J Mol Diagn. 16 (5), 541-549 (2014).
- Schleifman, E. B., et al. Next generation MUT-MAP, a high-sensitivity high-throughput microfluidics chip-based mutation analysis panel. PLoS One. 9 (3), e90761 (2014).
- Wong, S. Q., et al. Sequence artefacts in a prospective series of formalin-fixed tumours tested for mutations in hotspot regions by massively parallel sequencing. BMC Med Genomics. 7, 23 (2014).
- Narayan, A., et al. Ultrasensitive measurement of hotspot mutations in tumor DNA in blood using error-suppressed multiplexed deep sequencing. Cancer Res. 72 (14), 3492-3498 (2012).
- Hagemann, I. S., et al. Clinical next-generation sequencing in patients with non-small cell lung cancer. Cancer. 121 (4), 631-639 (2015).
- Won, H. H., Scott, S. N., Brannon, A. R., Shah, R. H., Berger, M. F. Detecting somatic genetic alterations in tumor specimens by exon capture and massively parallel sequencing. J Vis Exp. (80), e50710 (2013).
- Simen, B. B., et al. Validation of a next-generation-sequencing cancer panel for use in the clinical laboratory. Arch Pathol Lab Med. 139 (4), 508-517 (2015).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены