A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
מיפוי אתר הקישור של Aptamer על ATP שימוש microscale Thermophoresis
In This Article
Summary
MicroScale Thermophoresis (MST) is a sensitive technology to characterize aptamer-target interactions. This manuscript describes an MST protocol to characterize aptamer-small molecule interactions.
Abstract
Characterization of molecular interactions in terms of basic binding parameters such as binding affinity, stoichiometry, and thermodynamics is an essential step in basic and applied science. MicroScale Thermophoresis (MST) is a sensitive biophysical method to obtain this important information. Relying on a physical effect called thermophoresis, which describes the movement of molecules through temperature gradients, this technology allows for the fast and precise determination of binding parameters in solution and allows the free choice of buffer conditions (from buffer to lysates/sera). MST uses the fact that an unbound molecule displays a different thermophoretic movement than a molecule that is in complex with a binding partner. The thermophoretic movement is altered in the moment of molecular interaction due to changes in size, charge, and hydration shell. By comparing the movement profiles of different molecular ratios of the two binding partners, quantitative information such as binding affinity (pM to mM) can be determined. Even challenging interactions between molecules of small sizes, such as aptamers and small compounds, can be studied by MST. Using the well-studied model interaction between the DH25.42 DNA aptamer and ATP, this manuscript provides a protocol to characterize aptamer-small molecule interactions. This study demonstrates that MST is highly sensitive and permits the mapping of the binding site of the 7.9 kDa DNA aptamer to the adenine of ATP.
Introduction
אינטראקציה בין המולקולות היא הבסיס של הטבע. לפיכך, מדענים בתחומים רבים של מחקר בסיסי ויישומים לנסות להבין את עקרונות היסוד של אינטראקציות מולקולריות מסוגים שונים. Microscale Thermophoresis (MST) מאפשרת למדענים לבצע את מהירה מדויקת, יעילה וחסכונית, ואיכות שבשליטת אפיון של אינטראקציות מולקולריות בתמיסה, עם בחירה חופשית של מאגרים. יש כבר יותר מ -1,000 פרסומים באמצעות MST, משנת 2016 בלבד, המתאר סוגים שונים של ניתוחים, כוללים סריקות ספרייה, מחייב אימותי אירוע, מבחני תחרות, וניסויים עם שותפים מחייבים מספר 1-8. באופן כללי, MST מתיר חקר הפרמטרים המחייבים הקלסיים, כגון זיקה מחייבת (PM כדי מ"מ), ורכב, ותרמודינאמיקה, מכל סוג של אינטראקציה מולקולרית. יתרון גדול של MST הוא היכולת ללמוד אירועים מחייבים תלות בגודל של שותפי האינטראקציה. אפילו chalאינטראקציות lenging בין aptamers חומצות גרעין קטן (15-30 NT) ויעדים כגון מולקולות קטנות, תרופות, אנטיביוטיקה, או מטבוליטים ניתן לכמת.
נוכח המדינה- of-the-art טכנולוגיות לאפיין אינטראקציות aptamer-היעד הם או מעבדה קשות ומורכבות מאוד או להיכשל לכמת aptamer-מולקולה קטנה קשרי גומלין 9,10. Surface Plasmon תהודה (SPR) מבוססי מבחני 11,12 וגישות קלוריות ללא תווית באמת, כגון Calorimetry טיטרציה איזו תרמים (ITC) 13-15, elution isocratic 16, ltration fi שיווי משקל 17,18, ב-קו חיטוט 19, ג'ל משמרת מבחנים, והשתתק fl זרימת uorescence ספקטרוסקופיה 20,21, אנאיזוטרופיה קרינה (FA) 22,23, uorescence ההדמיה fl מולקולה בודדת 24,25, ו interferometry ביו השכבתי (BLI) 26 גם אם לא מדויקים או לא תואמים עם מולקולת aptamer-קטן יחסי גומלין. principa אחרסוגיות l של שיטות אלו רגישות נמוכה, צריכת דגימה גבוהה, חוסר תנועה, מגבלות להסעת המונים על משטחים, ו / או הגבלות חיץ. רק מעטי טכנולוגיות אלה מספקים בקרות משולבות אפקטי צבירת ספיחה.
MST מהווה כלי רב עוצמה עבור המדענים להתגבר על מגבלה זו כדי לחקור את יחסי הגומלין בין aptamers ומולקולות קטנות 27-29, כמו גם מטרות אחרות כגון חלבונים 30-33. הטכנולוגיה מתבססת על תנועת מולקולות באמצעות הדרגתיים טמפרטורה. התנועה מכוונת זו, המכונה "thermophoresis," תלוי בגודל, פריצה, ופצצת הידרציה של המולקולה 34,35. עקידת ליגנד למולקולה תשנה במישרין לפחות אחד הפרמטרים האלה, וכתוצאה מכך ניידות thermophoretic השתנה. הליגנדים עם מידות קטנות לא שיכולים להשפיע בצורה מהותית מבחינת שינוי הגודל מתוך המאוגד אל המדינה הנכנסת, אבל הם יכולים להיות dr תופעות amatic על הקליפה הידרציה ו / או תשלום. השינויים בתנועה thermophoretic של מולקולות לאחר אינטראקציות עם שותף מחייב מאפשרת כימות של פרמטרים מחייבים בסיסיים 2,7,34,36,37.
כפי שמתואר באיור 1A, מכשיר MST מורכב ליזר אינפרא אדום הממוקד על המדגם בתוך נימי הזכוכית באמצעות אותו אופטיקה ובאשר גילוי קרינה. התנועה thermophoretic של חלבונים באמצעות uorescence fl הפנימי של tryptophans 6 או של 3,8 שותף אינטראקציה שכותרתו fluorescently ניתן לנטר בזמן הלייזר קובע שיפוע הטמפרטורה (ΔT של 2-6 מעלות צלזיוס). הבדל הטמפרטורה והתוצאה הוא יותר שטח, ΔT, מוביל הדלדול או ההצטברות של מולקולות בתחום הטמפרטורה גבוהה, אשר ניתן לכמת ידי Soret COEF fi יעיל (S T):
ז "/>
ג חם מייצג את הריכוז באזור המחומם, וקרת c היא הריכוז באזור הקר הראשוני.
כפי שניתן לראות בתרשים 1B, תוצאות ניסוי MST טיפוסיות פרופיל תנועת MST (זכר זמן), מורכב שלבים שונים, אשר יכול להיות מופרד על ידי לוחות הזמנים שלהם. הקרינה הראשונית נמדדה ב -5 s הראשונה בהיעדרו של שיפוע הטמפרטורה כדי להגדיר את הקרינה החל מדויקת כדי לבדוק photobleaching או photoenhancement. קפיצת הטמפרטורה (T-Jump) מייצגת את השלב שבו שינויי הקרינה לפני תנועת thermophoretic. ירידה ראשונית זו קרינה תלויה בשינויים חומים תלויה של fl uorophore תשואת קוונטים. שלב thermophoresis כדלקמן, שבו ירידות הקרינה (או עליות) עקב תנועת thermophoretic של המולקולות עד החלוקה היציבה הוא הגיע.TJump הפוכה דיפוזיה בחזרה קשורה של מולקולות uorescent fl ניתן לצפות כמצוין באיור 1B לאחר הלייזר מכובה. כדי לגשת פרמטרים מחייבים בסיסיים, יחסי טוחנת שונים של השותפים באינטראקציה מנותחים ומשווים. בדרך כלל, 16 יחסים שונים נלמדים בניסוי אחד MST, ואילו מולקולת הגלוי האופטית נשמרה קבועה מסופקת עם כמות הולכת וגדלה של ליגנד ללא תווית. האינטראקציה בין שני השותפים מחייבים גורמת לשינויי thermophoresis, ומכאן גם uorescence fl המנורמל, נורמת F, אשר מחושבת כדלקמן:
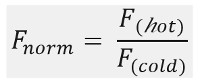
F חם וקר F מייצג ממוצעי עוצמות fl uorescence ב דה fi מועד לפי שעון נד של עקבות MST. זיקות מחייבות (ד K או EC 50 ערכים) יכולות להיות מחושבות על ידי הכמתמ"כהולם דואר (תרשים 1C).
בסך הכל, MST הוא כלי רב עוצמה כדי לחקור אינטראקציות מולקולריות מכל סוג שהוא. כתב יד זה מציע פרוטוקול לאפיין את האינטראקציה המאתגרת בין אדנוזין אדנוזין המולקולה הקטן (ATP; 0.5 KDA) ואת 25-NT הקצר ssDNA aptamer DH25.42 (7.9 KDA). במהלך של כתב היד, אתר הקישור של aptamer על מולקולת ATP ממופה אל קבוצת אדנין של ATP.
Protocol
1. הכנה של Stock עבודת Aptamer
- פעל על פי הוראות היצרן ו לפזר את oligonucleotide (5-Cy5-CCTG GGGGAGTATTGCGGAGGAAGG-3, רצף בהפניה 18) במים, להגיע לריכוז סופי של 100 מיקרומטר.
- הכן את הפתרון עובד aptamer ידי דילול המניות oligonucleotide 200 ננומטר עם חיץ מחייב (20 מ"מ טריס, pH 7.6; 300 מ"מ NaCl; 5 מ"מ MgCl 2; 0.01% Tween20).
- דגירה את התערובת במשך 2 דקות ב 90 מעלות צלזיוס, בואו המדגם מיד להתקרר על קרח, ולהשתמש מדגם בטמפרטורת החדר.
2. הכנת סדרת הדילול ליגנד
- עבור כל ליגנד (אדנוזין אדנוזין (ATP), diphosphate אדנוזין (ADP), monophosphate אדנוזין (AMP), אדנין, אדנוזין guanosine (GTP), אדנוזין ציטוזין (CTP), אדנוזין deoxyadenosine (dATP), ומתיונין S-adenosyl (SAM) ; 10 מ"מ מניות כל אחת), להכין diluti סדר 16 שלביםעל ב 200 צינורות התגובה מיקרו μl.
הערה: צנטריפוגה של מניות ליגנד במשך 5 דקות ב 14,000 XG עשוי לעזור כדי להסיר אגרגטים. נפח נמוך, צינורות תגובה מחייבת נמוכים מומלצים להימנע ספיחה של מולקולות על קירות הצינור. - התחל עם ריכוז מרבי של לפחות 50 פעמים גבוהות יותר מאשר הזיקה המוערכת ולהפחית את הריכוז ליגנד ב -50% בכל שלב דילול.
הערה: הכלי מאתר הריכוז מיושם תוכנות שליטת מדמת נתונים מחייבים ועוזרת במציאת טווח הריכוז הנכון עבור סדרת הדילול. - מלאו 20 μl של המניה ליגנד (10 מ"מ) צינור 1. הוסף 10 μl של חיץ מחייב aptamer לתוך צינורות התגובה מיקרו 2 עד 16.
- העברת 10 μl של צינור 1 צינור 2 ומערבבים היטב על ידי pipetting מעלה ומטה מספר פעמים. העבר 10 μL אל הצינור הבא וחזרו דילול זה עבור הצינורות הנותרים.
- מחק את עודף 10 μl מהצינור האחרון. הימנע מכל bתופעות דילול uffer. המאגר בצינור 1 ו צינורות 2-16 חייב להיות זהה.
3. הכנה של מיקס התגובה האחרון
- הכן את התגובות מחייבות אדם עם נפח של 20 μl (10 μl של פתרון עובד aptamer + 10 μl של הדילול ליגנד בהתאמה) כדי למזער שגיאות pipetting. נפח של רק 4 μl הוא יעיל fi סוף ל- Fi ll הנימים.
- הוסף 10 μl של הפתרון עובד aptamer 200 ננומטר עד 10 μl של דילול כל ליגנד ומערבבים היטב על ידי pipetting מעלה ומטה מספר פעמים.
- דגירת הדגימות במשך 5 דקות בטמפרטורת חדר וסיבי ll הדגימות מתפצלות לנימים סטנדרטיים על ידי טבילת הנימים לתוך המדגם. פעמי דגירה כבר עשויות להיות נחוצות עבור אינטראקציות כמה; עם זאת, 5 דקות הוא מתאים ביותר. גע הנימים רק בצדדים, לא על החלק האמצעי, שבו המדידה האופטית תילקח.
- מניחים את הנימים על הדואר נימי מגש ולהתחיל מכשיר MST.
4. הפעלת התקן MST
הערה: המכשיר מספק שתי חבילות תוכנה שהותקנה מראש, ה ' "תוכנה עבור ההגדרות הטכניות של תנאי הניסוי ואת השליטה' 'ניתוח" "תוכנה עבור הפרשנות של הנתונים המיוצר.
- לפני הצבת מגש הנימים לתוך מכשיר MST, להפעיל את התוכנה מלאה להתאים את הטמפרטורה הכללית רצויה על ידי הבחירה ב '' לאפשר בקרת טמפרטורה ידנית "ב '' בקרת טמפרטורה" התפריט הנפתח. כוונו את הטמפרטורה ל 25 מעלות צלזיוס בדרך זו.
הערה: מכשירי MST יכולים להיות מבוקר טמפרטורה מ 22 עד 45 ° C. - חכה הטמפרטורה להגיע לרמה הצפויה ולאחר מכן למקם את מגש הנימים לתוך מכשיר MST.
- הגדר את ערוץ LED כדי '' אדום "עבור צבע Cy5 ולהתאים את כוח LED להשיג fl uorescencאות e של 300 עד 1,000 יחידות קרינה במכשיר MST עם חיישן רגיל. 25% כוח LED משמש במחקר זה.
הערה: 6,000 עד 18,000 יחידות קרינה מומלצות לשימוש MST עם חיישן רגישות גבוהה.
5. נימי סריקה
- לבצע סריקת נימים לבדוק היבטים שונים באיכות של המדגם על ידי בחירת מיקום הנימים על תוכנת "שליטה" ולחיצה על "התחל סריקת כובע" לפני תחילת מדידת MST.
- בדוק את הסריקה נימי להגברת קרינה / מרווה ואפקטים דבקים (בצורת U או נמחצה על פסגות) בתוכנה.
הערה: פרטים נוספים על זיהוי והטיפול של קרינה ותופעות דבקות ניתן למצוא בדיון.
6. מדידת MST
הערה: לפני שמתחיל את מדידת MST, הקפד לכלול דבק אפקטים, שיפור / אפקטים מרווים, אוטעויות pipetting, ולהבטיח כי סריקת הנימים מציינת כי אות fl uorescence היא הסוף יעיל fi. לפרטים נוספים, ראה דיון.
- הקצה את ריכוז ליגנד מסדרת הדילול לתפקיד נימי בהתאמה בתוכנת '' השליטה "לשקול את צעד הדילול של ערבוב aptamer ליגנד. (1: 1).
- הזן את הריכוז הגבוה ביותר של ליגנד (5 המ"מ) עבור נימי # 1, בחר את סוג הדילול הנכון (כאן, 1: 1), לחץ על הריכוז המרבי, ולהשתמש בפונקציית גרור להקצות הריכוזים הנותרים באופן אוטומטי נימים # 2 16. הריכוז הנמוך ביותר הוא 152.6 ננומטר.
- הזן את הריכוז של aptamer uorescent fl (כאן, 100 ננומטר) בסעיף בהתאמה של תוכנות שליטה.
- השתמש בהגדרות ברירת המחדל, אשר לזהות את uorescence fl למשך 5 שניות, להקליט את MST למשך 30 שניות, ולהקליט את הקרינה במשך שניות 5 נוספות לאחר האיון של t הליזר o לפקח על דיפוזיה האחורי של מולקולות.
- התאם את כוח לייזר עד 20% בגזרה בהתאמה של תוכנות שליטה.
הערה: כדי לקבל את יחס אות לרעש הטוב ביותר ועל מנת למנוע תופעות נוקבות, כוח ליזר של 20-40% מומלץ. במקרים מסוימים, כוח ליזר גבוה יותר עשוי להידרש לקבל הפרדה טובה של מולקולות מאוגדות וכרוכות. - שמור את הניסוי לאחר בחירת תיקיית היעד ולהתחיל מדידת MST ידי לחיצה על כפתור '' התחל MST המדידה ".
הערה: קובץ .ntp יופק בתיקיית היעד. באמצעות התקנה זו, מדידה אחת נמשכת 10-15 דקות. - חזור על ההליך הניסיוני לפחות פעמים לשם קביעה מדויקת יותר של הערך 50 EC.
הערה: על מנת לבחון את השחזור הטכני, באותה הנימים ניתן לסרוק מספר פעמים (חזרות טכניות).
7. MST ניתוח נתונים
NT "> הערה: תוכנת הניתוח מאפשרת ניתוח של נתונים על y fl במהלך מדידת תוכנת ניתוח מגרשי עקבות MST הזמן ושינויי uorescence fl המנורמל (נורמת F) לעומת הריכוז ליגנד 37..- הפעל את תוכנת ניתוח MST (ניתוח MO.Affinity) ולטעון את הקובץ .ntp מתיקיית היעד. בחר "MST" כסוג ניתוח בתפריט מבחר נתונים.
הערה: במקרה של תופעות קרינה תלויה ליגנד, הקרינה הראשונית ניתן לבחור לניתוח. - מוסיף את הריצה טכנית או ביולוגית בהתאמה (ים) ניתוח חדש על ידי גרירה ושחרור או על ידי לחיצה על הכפתור "+" להלן בטווח הניסיון בהתאמה.
- לחץ על לחצן המידע שלהלן בטווח הניסיון בהתאמה כדי להשיג מידע על המאפיינים של הניסוי, עקבות MST, סריקת נימים, צורה נימי, קרינה ראשונית, וקצב לבן.
הערה: נתונים גולמיים אלה יכולים אלכך להיבדק, בשלבים מאוחרים יותר של ניתוח. - ראייה לבדוק את עקבות MST לתופעות צבירה משקעים, נראה כמו בליטות קוצים.
הערה: לקבלת מידע נוסף על זיהוי וטיפול של תופעות צבירה, לקרוא את הדיון. - ראייה לבדוק את הסריקה נימי ואת שכבת צורה נימי עבור אפקטי ספיחה, גלויה כמו פסגות שטוחות או בצורת U. ראייה לבדוק את הסריקה נימי ואת הקרינה הראשונית לתופעות הקרינה. ראייה לבדוק את שיעור ההלבנה עבור photobleaching אפקטים.
- למעבר למצב מנת התגובה ולשנות את הגדרת הניתוח למצב "מומחה" על ידי לחיצה על הכפתור בהתאמה. בחר "T-קפיצה" כאסטרטגית הערכת MST.
- בחר את הדגם "היל" עבור tting fi העקומה. הפרמטרים מחייבים יחושבו אוטומטית. לנרמל את הנתונים על ידי בחירת בהתאמה לסוג של נורמליזציה בתפריט "להשוות את התוצאות". יצואנתונים אם במסגרת .xls או pdf.
הערה: הטבלה מתחת לגרף המחייב מסכמת את הפרמטרים מחייבים המחושבים.
תוצאות
במחקר זה, MST יושמה לאפיין את אתר הקישור של aptamer DH25.42 DNA 18 על ATP. בניגוד למחקרים אחרים המאפיינים את האינטראקציה של ATP או ה- ATP-מחק מולקולות קטנות עם חלבונים שכותרתו אקראית עם fluorophores אחד או יותר 38-40, מחקר זה כולל גרסה שכותרתו של aptamer 7.9 kDa ssDNA עם מול...
Discussion
בקרות איכות:
דבק נוקבים / ספיחה של חומר מדגם למשטחים, וכן תופעות צבירה, יש השפעה דרמטית על איכות נתוני הזיקה. עם זאת, רק המדינה- of-the-art כמה טכנולוגיות מציעות אפשרויות מדויקות ומהירות לפקח למנוע את ההשפעות הללו. MST מציעה בקרת איכות משול...
Disclosures
CE ו- TS הם עובדי 2bind GmbH, המספקת שירותים אנליטיים biophysical. עמלות פרסום עבור מאמר וידאו זה משולמות על ידי 2bind GmbH.
Acknowledgements
יש המחברים לא בתודות.
Materials
| Name | Company | Catalog Number | Comments |
| Aptamer binding buffer | 20 mM Tris pH 7.6; 300 mM NaCl; 5 mM MgCl2; 0.01% Tween-20 | ||
| Fluorescently labeled ATP aptamer | IDT, Leuven, Belgium | sequence: DH25.42 50-Cy5-CCTGGGGGAGT- ATTGCGGAGGAAGG-3 | |
| ATP | Sigma Aldrich, Germany | A2383 | 10 mM stock solutions stored at - 20 °C |
| ADP | Sigma Aldrich, Germany | A2754 | 10 mM stock solutions stored at - 20 °C |
| AMP | Sigma Aldrich, Germany | A2252 | 10 mM stock solutions stored at - 20 °C |
| Adenine | Sigma Aldrich, Germany | A8626 | 10 mM stock solutions stored at - 20 °C |
| SAM | Sigma Aldrich, Germany | A7007 | 10 mM stock solutions stored at - 20 °C |
| dATP | Sigma Aldrich, Germany | 11934511001 | 10 mM stock solutions stored at - 20 °C |
| CTP | Sigma Aldrich, Germany | C1506 | 10 mM stock solutions stored at - 20 °C |
| GTP | Sigma Aldrich, Germany | G8877 | 10 mM stock solutions stored at - 20 °C |
| Monolith NT.115 | NanoTemper Technologies, Munich, Germany | MO-G008 | Blue/Red Channel MST device with standard detector, Monolith NT115 pico is MST device with high sensitivity detector |
| Monolith NT.115 capillaries Standard | NanoTemper Technologies, Munich, Germany | MO-K002 | |
| Eppendorf PCR tubes | Eppendorf, Germany | 30124537 | |
| Monolith control software. 2.1.33, pre-installed on the device | NanoTemper Technologies, Munich, Germany | ||
| MO.affinity analysis v2.1.1 | NanoTemper Technologies, Munich, Germany | ||
| Kaleidagraph 4.5.2 | Synergy Software |
References
- Linke, P., et al. An Automated Microscale Thermophoresis Screening Approach for Fragment-Based Lead Discovery. J Biomol Screen. 21 (4), 414-421 (2015).
- Jerabek-Willemsen, M., et al. MicroScale Thermophoresis: Interaction analysis and beyond. Journal of Molecular Structure. 1077, 101-113 (2014).
- Zillner, K., et al. Microscale thermophoresis as a sensitive method to quantify protein: nucleic acid interactions in solution. Methods Mol Biol. 815, 241-252 (2012).
- Zhang, W., Duhr, S., Baaske, P., Laue, E. Microscale thermophoresis for the assessment of nuclear protein-binding affinities. Methods Mol Biol. 1094, 269-276 (2014).
- Wienken, C. J., Baaske, P., Rothbauer, U., Braun, D., Duhr, S. Protein-binding assays in biological liquids using microscale thermophoresis. Nat Commun. 1, 100 (2010).
- Seidel, S. A., et al. Label-free microscale thermophoresis discriminates sites and affinity of protein-ligand binding. Angew Chem Int Ed Engl. 51 (42), 10656-10659 (2012).
- Seidel, S. A., et al. Microscale thermophoresis quantifies biomolecular interactions under previously challenging conditions. Methods. 59 (3), 301-315 (2013).
- Schubert, T., et al. Df31 protein and snoRNAs maintain accessible higher-order structures of chromatin. Mol Cell. 48 (3), 434-444 (2012).
- McKeague, M., Derosa, M. C. Challenges and opportunities for small molecule aptamer development. J Nucleic Acids. 2012, 748913 (2012).
- Ruscito, A., DeRosa, M. C. Small-Molecule Binding Aptamers: Selection Strategies, Characterization, and Applications. Front Chem. 4, 14 (2016).
- Chang, A. L., McKeague, M., Liang, J. C., Smolke, C. D. Kinetic and equilibrium binding characterization of aptamers to small molecules using a label-free, sensitive, and scalable platform. Anal Chem. 86 (7), 3273-3278 (2014).
- Chang, A. L., McKeague, M., Smolke, C. D. Facile characterization of aptamer kinetic and equilibrium binding properties using surface plasmon resonance. Methods Enzymol. 549, 451-466 (2014).
- Jing, M., Bowser, M. T. Methods for measuring aptamer-protein equilibria: a review. Anal Chim Acta. 686 (1-2), 9-18 (2011).
- Sokoloski, J. E., Dombrowski, S. E., Bevilacqua, P. C. Thermodynamics of ligand binding to a heterogeneous RNA population in the malachite green aptamer. Biochemistry. 51 (1), 565-572 (2012).
- Burnouf, D., et al. kinITC: a new method for obtaining joint thermodynamic and kinetic data by isothermal titration calorimetry. J Am Chem Soc. 134 (1), 559-565 (2012).
- Mannironi, C., Scerch, C., Fruscoloni, P., Tocchini-Valentini, G. P. Molecular recognition of amino acids by RNA aptamers: the evolution into an L-tyrosine binder of a dopamine-binding RNA motif. RNA. 6 (4), 520-527 (2000).
- Jenison, R. D., Gill, S. C., Pardi, A., Polisky, B. High-resolution molecular discrimination by RNA. Science. 263 (5152), 1425-1429 (1994).
- Huizenga, D. E., Szostak, J. W. A DNA aptamer that binds adenosine and ATP. Biochemistry. 34 (2), 656-665 (1995).
- Lee, E. R., Baker, J. L., Weinberg, Z., Sudarsan, N., Breaker, R. R. An allosteric self-splicing ribozyme triggered by a bacterial second messenger. Science. 329 (5993), 845-848 (2010).
- Wickiser, J. K., Cheah, M. T., Breaker, R. R., Crothers, D. M. The kinetics of ligand binding by an adenine-sensing riboswitch. Biochemistry. 44 (40), 13404-13414 (2005).
- Jucker, F. M., Phillips, R. M., McCallum, S. A., Pardi, A. Role of a heterogeneous free state in the formation of a specific RNA-theophylline complex. Biochemistry. 42 (9), 2560-2567 (2003).
- Zhao, Q., Lv, Q., Wang, H. Aptamer fluorescence anisotropy sensors for adenosine triphosphate by comprehensive screening tetramethylrhodamine labeled nucleotides. Biosens Bioelectron. 70, 188-193 (2015).
- Zhang, D., et al. A sensitive fluorescence anisotropy method for detection of lead (II) ion by a G-quadruplex-inducible DNA aptamer. Anal Chim Acta. 812, 161-167 (2014).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule imaging of an in vitro-evolved RNA aptamer reveals homogeneous ligand binding kinetics. J Am Chem Soc. 131 (29), 9866-9867 (2009).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule binding experiments on long time scales. Rev Sci Instrum. 81 (8), 083705 (2010).
- Zichel, R., Chearwae, W., Pandey, G. S., Golding, B., Sauna, Z. E. Aptamers as a sensitive tool to detect subtle modifications in therapeutic proteins. PLoS One. 7 (2), 31948 (2012).
- Baaske, P., Wienken, C. J., Reineck, P., Duhr, S., Braun, D. Optical thermophoresis for quantifying the buffer dependence of aptamer binding. Angew Chem Int Ed Engl. 49 (12), 2238-2241 (2010).
- Entzian, C., Schubert, T. Studying small molecule-aptamer interactions using MicroScale Thermophoresis (MST). Methods. 97, 27-34 (2016).
- Valenzano, S., et al. Screening and Identification of DNA Aptamers to Tyramine Using in Vitro Selection and High-Throughput Sequencing. ACS Comb Sci. 18 (6), 302-313 (2016).
- Jauset Rubio, M., et al. beta-Conglutin dual aptamers binding distinct aptatopes. Anal Bioanal Chem. 408 (3), 875-884 (2016).
- Breitsprecher, D., et al. Aptamer Binding Studies Using MicroScale Thermophoresis. Methods Mol Biol. 1380, 99-111 (2016).
- Stoltenburg, R., Schubert, T., Strehlitz, B. In vitro Selection and Interaction Studies of a DNA Aptamer Targeting Protein A. PLoS One. 10 (7), 0134403 (2015).
- Kinghorn, A. B., et al. Aptamer Affinity Maturation by Resampling and Microarray Selection. Anal Chem. 88 (14), 6981-6985 (2016).
- Duhr, S., Braun, D. Why molecules move along a temperature gradient. Proc Natl Acad Sci U S A. 103 (52), 19678-19682 (2006).
- Braun, D., Libchaber, A. Trapping of DNA by thermophoretic depletion and convection. Phys Rev Lett. 89 (18), 188103 (2002).
- Duhr, S., Arduini, S., Braun, D. Thermophoresis of DNA determined by microfluidic fluorescence. Eur Phys J E Soft Matter. 15 (3), 277-286 (2004).
- Jerabek-Willemsen, M., Wienken, C. J., Braun, D., Baaske, P., Duhr, S. Molecular interaction studies using microscale thermophoresis. Assay Drug Dev Technol. 9 (4), 342-353 (2011).
- He, K., Dragnea, V., Bauer, C. E. Adenylate Charge Regulates Sensor Kinase CheS3 To Control Cyst Formation in Rhodospirillum centenum. MBio. 6 (3), 00546 (2015).
- Brvar, M., et al. Structure-based discovery of substituted 4,5'-bithiazoles as novel DNA gyrase inhibitors. J Med Chem. 55 (14), 6413-6426 (2012).
- Pogorelcnik, B., et al. 4,6-Substituted-1,3,5-triazin-2(1H)-ones as monocyclic catalytic inhibitors of human DNA topoisomerase IIalpha targeting the ATP binding site. Bioorg Med Chem. 23 (15), 4218-4229 (2015).
- Jhaveri, S., Rajendran, M., Ellington, A. D. In vitro selection of signaling aptamers. Nat Biotechnol. 18 (12), 1293-1297 (2000).
- Khavrutskii, L., et al. Protein purification-free method of binding affinity determination by microscale thermophoresis. J Vis Exp. (78), (2013).
- Ramakrishnan, M., et al. Probing cocaine-antibody interactions in buffer and human serum. PLoS One. 7 (7), 40518 (2012).
- Chen, M., et al. Antiviral activity and interaction mechanisms study of novel glucopyranoside derivatives. Bioorg Med Chem Lett. 25 (18), 3840-3844 (2015).
- Wan, C., et al. Insights into the molecular recognition of the granuphilin C2A domain with PI(4,5)P2. Chem Phys Lipids. 186 (4,5), 61-67 (2015).
- Harazi, A., et al. The Interaction of UDP-N-Acetylglucosamine 2-Epimerase/N-Acetylmannosamine Kinase (GNE) and Alpha-Actinin 2 Is Altered in GNE Myopathy M743T Mutant. Mol Neurobiol. , (2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved