Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Kartierung der Bindungsstelle eines Aptamer auf ATP Mit Micro Thermophorese
In diesem Artikel
Zusammenfassung
MicroScale Thermophoresis (MST) is a sensitive technology to characterize aptamer-target interactions. This manuscript describes an MST protocol to characterize aptamer-small molecule interactions.
Zusammenfassung
Characterization of molecular interactions in terms of basic binding parameters such as binding affinity, stoichiometry, and thermodynamics is an essential step in basic and applied science. MicroScale Thermophoresis (MST) is a sensitive biophysical method to obtain this important information. Relying on a physical effect called thermophoresis, which describes the movement of molecules through temperature gradients, this technology allows for the fast and precise determination of binding parameters in solution and allows the free choice of buffer conditions (from buffer to lysates/sera). MST uses the fact that an unbound molecule displays a different thermophoretic movement than a molecule that is in complex with a binding partner. The thermophoretic movement is altered in the moment of molecular interaction due to changes in size, charge, and hydration shell. By comparing the movement profiles of different molecular ratios of the two binding partners, quantitative information such as binding affinity (pM to mM) can be determined. Even challenging interactions between molecules of small sizes, such as aptamers and small compounds, can be studied by MST. Using the well-studied model interaction between the DH25.42 DNA aptamer and ATP, this manuscript provides a protocol to characterize aptamer-small molecule interactions. This study demonstrates that MST is highly sensitive and permits the mapping of the binding site of the 7.9 kDa DNA aptamer to the adenine of ATP.
Einleitung
Wechselwirkung zwischen Molekülen ist die Basis der Natur. Daher Wissenschaftler in vielen Bereichen der Grundlagen- und angewandten Forschung versuchen die grundlegenden Prinzipien der molekularen Wechselwirkungen verschiedener Arten zu verstehen. Micro Thermophorese (MST) können Wissenschaftler die schnelle, präzise und kosteneffiziente und qualitätskontrollierte Charakterisierung von molekularen Wechselwirkungen in Lösung durchzuführen, mit einer freien Wahl von Puffern. Es gibt bereits mehr als 1.000 Publikationen MST mit, ab 2016 allein, beschreibt verschiedene Arten von Analysen, einschließlich Bibliothek Vorführungen, Ereignis Validierungen Bindung, kompetitive Assays, und Experimente mit mehreren Bindungspartnern 1-8. Im Allgemeinen erlaubt MST die Untersuchung der klassischen Bindungsparameter wie Bindungsaffinität (pM bis Mm), Stöchiometrie und Thermodynamik, der jede Art von molekularen Wechselwirkungen. Ein großer Vorteil der MST ist die Fähigkeit, Bindungsereignisse unabhängig von der Größe der Interaktionspartner zu studieren. Auch Heraus-fordernd Wechselwirkungen zwischen kleinen Nukleinsäure-Aptamere (15-30 nt) und Ziele wie kleine Moleküle, Medikamente, Antibiotika oder Metaboliten quantifiziert werden können.
Aktuelle state-of-the-art Technologien Aptamer-Target - Wechselwirkungen sind entweder im Labor intensiv und sehr komplex zu charakterisieren oder scheitern Aptamer-kleines Molekül 9,10 Wechselwirkungen zu quantifizieren. Oberflächenplasmonresonanz (SPR) -basierte Assays 11,12 und wirklich markierungsfreie kalorimetrischen Ansätze wie Isothermen Titrations - Kalorimetrie (ITC) 13-15, isokratische Elution 16, Gleichgewicht Infiltrations 17,18, in-line Sondieren 19, Gel- Shift - Assays, Stopped fl ow Fluoreszenz- Spektroskopie 20,21, Fluoreszenzanisotropiemessungen (FA) 22,23, Einzelmolekül - Fluoreszenz - Bildgebung 24,25 und Bio-Schicht - Interferometrie (BLI) 26 sind ebenfalls entweder ungenau oder nicht kompatibel mit Aptamer-kleines Molekül Wechselwirkungen. Andere principal Probleme dieser Verfahren sind eine geringe Empfindlichkeit, hohe Probenverbrauch, Immobilisation, Massentransportbeschränkungen auf Oberflächen und / oder Puffer Einschränkungen. Nur wenige dieser Technologien bieten integrierte Kontrollen für die Aggregation und Adsorption Effekte.
MST stellt ein leistungsfähiges Werkzeug für Wissenschaftler , diese Einschränkung zu überwinden , um die Wechselwirkungen zwischen Aptamere und kleine Moleküle , 27-29, sowie andere Ziele, wie Proteine 30-33 zu studieren. Die Technologie beruht auf der Bewegung von Molekülen durch Temperaturgradienten. Diese gerichtete Bewegung, die so genannte "Thermophorese," hängt von der Größe, der Ladung und Hydrathülle des Moleküls 34,35. Die Bindung eines Liganden an das Molekül direkt an mindestens einen dieser Parameter zu verändern, in einer veränderten thermophoretische Mobilität führt. Liganden mit kleinen Größen haben nicht erhebliche Auswirkungen in Bezug auf die Größenänderung von der ungebundenen zum gebundenen Zustand, aber sie können dr haben amatic Auswirkungen auf die Hydrathülle und / oder Ladung. Die Veränderungen in der thermophoretische Bewegung der Moleküle nach der Wechselwirkung mit dem Bindungspartner ermöglicht die Quantifizierung der Grundbindungsparameter 2,7,34,36,37.
Wie in 1A dargestellt ist , besteht der MST Vorrichtung eines Infrarotlasers auf die Probe innerhalb der Glaskapillaren fokussiert die gleiche Optik wie für die Fluoreszenzdetektion. Die thermophoretischer Bewegung von Proteinen über die Eigenfluoreszenz von Tryptophane 6 oder eines fluoreszierend 3,8 Interaktionspartner markiert überwacht werden kann , während der Laser einen Temperaturgradienten (AT von 2-6 ° C) herstellt. Die sich ergebende Temperaturdifferenz im Raum, & Delta; T, führt zur Verarmung oder Anreicherung von Molekülen im Bereich der erhöhten Temperatur, die durch die Soret quantifiziert werden kann Koeffizienten (S T):
g "/>
c hot stellt die Konzentration in dem erwärmten Bereich, und c kalt ist die Konzentration in dem anfänglichen Kaltbereich.
Wie in 1B gezeigt , um eine typische MST Versuchsergebnisse in einer MST Bewegungsprofil (Zeitverlauf), die aus verschiedenen Phasen, die durch ihre jeweiligen Zeitrahmen getrennt werden können. Die anfängliche Fluoreszenz wird in den ersten 5 s in Abwesenheit des Temperaturgradienten gemessen, um die genaue Start Fluoreszenz zu definieren und für Photobleaching oder photoenhancement zu überprüfen. Der Temperatursprung (T-Jump) repräsentiert die Phase, in der die Fluoreszenzänderungen vor thermophoretischer Bewegung. Diese anfängliche Abnahme der Fluoreszenz hängt von wärmeabhängige Veränderungen von fl uorophore Quantenausbeute. Die Thermophorese Phase folgt, in der die Fluoreszenz abnimmt (oder zunimmt) aufgrund der thermophoretische Bewegung der Moleküle, bis die Stationärzustandsverteilung erreicht wird.Die umgekehrte TJump und die gleichzeitige Rückdiffusion der fluoreszierenden Moleküle können wie in 1B angegeben beobachtet werden , nachdem der Laser ausgeschaltet ist. Um grundlegende Bindungsparameter, verschiedene Molverhältnisse der Interaktionspartner für den Zugriff werden analysiert und verglichen. Typischerweise werden 16 unterschiedliche Verhältnisse in einem MST Experiment untersucht, wohingegen der optische sichtbaren Molekül konstant gehalten wird und mit zunehmender Menge des unmarkierten Liganden zugeführt wird. Die Wechselwirkung zwischen den beiden Bindungspartner induziert Veränderungen in der Thermophorese, und somit in der normalisierten Fluoreszenz, F Norm, die wie folgt berechnet wird:
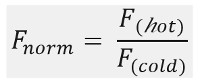
F heiß und F kalt stellen Intensitäten fluoreszenz bei de fi nierten Zeitpunkte der MST Spuren gemittelt. Bindungsaffinitäten (K d oder EC 50 -Werte) können durch curv berechnet werdene Fitting (1C).
Insgesamt ist MST ein leistungsfähiges Werkzeug molekularen Wechselwirkungen jeglicher Art zu studieren. Dieses Manuskript bietet ein Protokoll das herausfordernde Interaktion zwischen dem kleinen Molekül Adenosintriphosphat (ATP; 0,5 kDa) zu charakterisieren und die 25-nt kurze ssDNA Aptamer DH25.42 (7,9 kDa). Im Laufe des Manuskripts wird die Bindungsstelle des Aptamers an das ATP-Molekül an die Adenin-Gruppe des ATP kartiert unten.
Protokoll
1. Vorbereitung der Aptamer-Funktion Stock
- Folgen Sie den Anweisungen des Herstellers und lösen das Oligonukleotid (5-Cy5-CCTG GGGGAGTATTGCGGAGGAAGG-3, Sequenz aus Referenz 18) in Wasser, einer 100-uM Endkonzentration zu erreichen.
- Bereiten Sie die Aptamer - Arbeitslösung durch Verdünnen des Oligonukleotid - Lager auf 200 nM mit Bindungspuffer (20 mM Tris, pH 7,6; 300 mM NaCl, 5 mM MgCl 2; 0,01% Tween 20).
- Inkubieren Sie die Mischung für 2 min bei 90 ° C, lassen Sie sofort die Probe auf Eis abkühlen, und die Probe bei Raumtemperatur verwenden.
2. Herstellung der Ligand Verdünnungsreihe
- Für jeden Liganden (Adenosin-Triphosphat (ATP), Adenosindiphosphat (ADP), Adenosinmonophosphat (AMP), Adenin, Guanosin-Triphosphat (GTP), Cytosin-triphosphat (CTP), Desoxyadenosintriphosphat (dATP) und S-Adenosylmethionin (SAM) ; jeweils) 10 mM Lager, bereiten Sie einen 16-stufigen seriellen dilutiauf in 200 & mgr; l Mikroreaktionsröhrchen.
HINWEIS: Die Zentrifugation von Ligand Bestände für 5 min bei 14000 xg helfen können Aggregate zu entfernen. Geringes Volumen, geringe Bindungsreaktionsrohre werden empfohlen Adsorption von Molekülen an den Rohrwänden zu vermeiden. - Beginnen mit einer maximalen Konzentration von mindestens 50 mal höher ist als die geschätzte Affinität und Verringerung um 50% in jedem Verdünnungsschritt die Ligandenkonzentration.
HINWEIS: Die Konzentration finder Werkzeug in die Steuerungssoftware implementiert simuliert Bindungsdaten und hilft bei der richtigen Konzentrationsbereich für die Verdünnungsreihe finden. - Füllen Sie 20 ul des Liganden Lager (10 mM) in Rohr 1. Fügen Sie 10 ul Aptamer-Bindungspuffer in Mikroreaktionsrohre 2 bis 16.
- Transfer 10 ul Rohr 1 bis Rohr 2 und ordnungsgemäß durch Auf- und Abpipettieren mehrmals mischen. Übertragen Sie 10 & mgr; l in die nächste Röhre und wiederholen Sie diese Verdünnung für die verbleibenden Rohre.
- Entsorgen Sie die 10 ul Überschuss aus dem letzten Rohr. Vermeiden Sie bUffer Verwässerungseffekte. Der Puffer in Rohr 1 und in Röhrchen 2-16 müssen identisch sein.
3. Herstellung der endgültigen Reaktions Mix
- Bereiten Sie die einzelnen Bindungsreaktionen mit einem Volumen von 20 & mgr; l (10 & mgr; l Arbeitslösung Aptamer + 10 & mgr; l des jeweiligen Liganden Verdünnung) Pipettierfehler zu minimieren. Ein Volumen von nur 4 ul reicht aus ll die Kapillare zu fi nden.
- In 10 ul der 200 nM Aptamer-Arbeitslösung zu 10 & mgr; l jedes Liganden Verdünnung und mischen richtig durch Auf- und Abpipettieren mehrmals.
- Inkubiere die Proben für 5 min bei Raumtemperatur und fi ll die Proben in Standard-Kapillaren durch die Kapillaren in die Probe eingetaucht wird. Längere Inkubationszeiten können für einige Wechselwirkungen erforderlich sein; jedoch 5 min ist ausreichend für die meisten. Berühren die Kapillaren nur an den Seiten, nicht auf dem mittleren Teil, wo die optische Messung genommen wird.
- Legen Sie die Kapillaren auf the Kapillar-Fach und die MST-Gerät starten.
4. Starten des MST-Gerät
HINWEIS: Das Gerät verfügt über zwei vorinstallierte Software-Pakete, die '' control "Software für den technischen Aufbau der experimentellen Bedingungen und der '' Analyse" Software für die Interpretation der erzeugten Daten.
- Vor der Platzierung der Kapillare Fach in die MST-Gerät, starten Sie die Steuerungssoftware und stellen Sie die gesamte gewünschte Temperatur von 'Auswahl von' ermöglichen Steuerung manueller Temperatur "in der '' Temperaturregelung" Drop-Down-Menü. Einstellen der Temperatur auf 25 ° C in dieser Weise.
HINWEIS: Die MST Instrumente können von 22 bis 45 ° C temperiert werden. - Warten, bis die Temperatur das erwartete Niveau zu erreichen, und legen Sie dann die Kapillare Fach in die MST-Gerät.
- Stellen Sie den LED-Kanal '' rot "für Cy5 Farbstoffen und stellen Sie die LED-Leistung ein fl uorescenc zu gewinnene Signal von 300 bis 1000 Fluoreszenzeinheiten an der MST-Gerät mit einem Standard-Sensor. 25% LED-Leistung wird in dieser Studie verwendet.
HINWEIS: 6.000 bis 18.000 Fluoreszenzeinheiten für die MST mit einem hochempfindlichen Sensor empfohlen.
5. Kapillar-Scan
- Führen Sie eine Kapillare Scan verschiedene Qualitätsaspekte der Probe zu überprüfen, indem die Kapillare Position auf der "Control" Software ein und klicken auf "Start Kappe scan" bevor die MST-Messung starten.
- Überprüfen Sie die Kapillar-Scan für Fluoreszenzverstärkung / Lösch- und Klebeeffekte (U-förmige oder abgeflachte Spitzen) in der Software.
HINWEIS: Weitere Informationen über die Erkennung und Behandlung von Fluoreszenz und Klebeeffekte können in der Diskussion zu finden.
6. MST Mess
HINWEIS: Vor der Messung MST beginnen, stellen Sie sicher, dass Effekte, Verbesserung / Löscheffekte auszuschließen kleben, oderPipettierfehler, und sicherzustellen, dass die Kapillare Scan zeigt, dass die Fluoreszenz- Signal reicht aus. Weitere Details, die Diskussion.
- Ordnen die Ligandenkonzentrationen aus der Verdünnungsreihe auf die jeweilige Kapillare Position in der '' control "Software Betrachten der Verdünnungsschritt das Aptamer und dem Liganden zu mischen. (1: 1).
- Geben Sie die höchste Konzentration an Ligand (5 mM) für kapillare # 1, wählen Sie die richtige Verdünnung (hier 1: 1), klicken Sie auf die maximale Konzentration, und verwenden Sie die Drag-Funktion, um automatisch die verbleibenden Konzentrationen in Kapillaren zuweisen # 2- 16. Die niedrigste Konzentration ist 152,6 nM.
- Geben Sie die Konzentration des Fluoreszenz- Aptamer (hier 100 nM) in dem jeweiligen Abschnitt der Steuerungssoftware.
- Verwenden Sie die Standardeinstellungen, die die fluoreszenz für 5 Sekunden erkennen, notieren Sie die MST für 30 Sekunden, und notieren Sie die Fluoreszenz für weitere 5 Sekunden nach der Inaktivierung des Lasers t o die Rückdiffusion von Molekülen zu überwachen.
- Stellen Sie die Laserleistung auf 20% in dem jeweiligen Abschnitt der Steuersoftware.
ANMERKUNG: Um das beste Signal-zu-Rausch-Verhältnis zu erhalten und zu unspezifischen Effekte zu vermeiden, wird eine Laserleistung von 20-40% empfohlen. In bestimmten Fällen kann eine höhere Laserleistung eine gute Trennung von ungebundenen und gebundenen Moleküle zu erhalten erforderlich. - Speichern Sie das Experiment nach den Zielordner auswählen und auf die MST Messung starten durch Drücken der '' Start MST Messung "-Taste.
HINWEIS: Die .ntp Datei im Zielordner erzeugt wird. Mit diesem Setup dauert eine Messung 10-15 min. - Wiederholen Sie die experimentellen Verfahren mindestens zweimal für eine genauere Bestimmung des Wertes 50 EC.
HINWEIS: Um die technische Reproduzierbarkeit zu testen, können die gleichen Kapillaren mehrmals gescannt werden (technische Wiederholungen).
7. MST Datenanalyse
nt "> Hinweis: Die Analyse - Software die Analyse von Daten über die fl y während der Messung ermöglicht die Analyse - Software , die MST Zeit Spuren und Veränderungen in der normierten Fluoreszenz- (F Norm) gegen die Ligandenkonzentration 37 aufgetragen ist ..- Starten Sie die MST-Analyse-Software (MO.Affinity Analysis) und laden Sie die .ntp Datei aus dem Zielordner. Wählen Sie "MST" als Analysetyp in der Datenauswahlmenü.
HINWEIS: Bei Liganden-abhängige Fluoreszenzeffekte kann die anfängliche Fluoreszenz zur Analyse ausgewählt werden. - Fügen Sie die entsprechenden technischen oder biologischen Lauf (s) einer neuen Analyse per Drag-and-Drop oder über die Schaltfläche "+" unterhalb der jeweiligen Versuchslauf drücken.
- Drücken Sie die Informationstaste unterhalb der jeweiligen Versuchslauf zu erhalten Informationen über die Eigenschaften des Experiments MST Spuren, Kapillar-Scan, Kapillar-Form, anfängliche Fluoreszenz und Bleichrate.
Hinweis: Diese Rohdaten können also in späteren Schritten der Analyse überprüft werden. - die MST Spuren für die Aggregation und Fällung Effekte sichtbar, wie Beulen und Spikes einer Sichtprüfung.
HINWEIS: Weitere Informationen über die Erkennung und Behandlung von Aggregationseffekten, lesen Sie die Diskussion. - die kapillare Scan und die kapillare Form Overlay für Adsorptionseffekten, sichtbar als abgeflacht oder U-förmigen Spitzen Sichtprüfung. Eine Sichtprüfung des Kapillar-Scan und die anfängliche Fluoreszenz für Fluoreszenzeffekte. Sichtprüfung für Photobleaching Auswirkungen der Bleichrate.
- Wechseln Sie auf die Dosis-Wirkungs-Modus und ändern Sie die Analyse-Einstellung auf "Experten" -Modus indem Sie die entsprechende Taste drücken. Wählen Sie "T-Jump", wie die MST Bewertungsstrategie.
- Wählen Sie den "Hügel" Modell für die Kurve Armatur. Die Bindungsparameter werden automatisch berechnet. Normalisieren der Daten durch die jeweilige Art der Normalisierung in der "vergleichen Sie die Ergebnisse" Menü wählen. exportieren Sie dieDaten entweder als .xls oder .pdf.
HINWEIS: Die unten stehende Tabelle der Bindungs Diagramm fasst die berechneten Bindungsparameter.
Ergebnisse
In dieser Studie wurde MST die Bindungsstelle des DNA - Aptamer DH25.42 18 an ATP zu charakterisieren aufgetragen. Im Gegensatz zu anderen Studien , die die Wechselwirkung von ATP oder ATP-nachahmt kleine Moleküle mit Proteinen zufällig markiert mit einem oder mehreren Fluorophoren 38-40 charakterisierenden umfasst diese Studie eine markierte Version des 7,9 kDa ssDNA Aptamer mit einem Cy5 - Molekül am 5'Ende . Verschiedenen ATP-Derivate und verwandte Molekü...
Diskussion
Qualitätskontrollen:
Unspezifische Klebe- / Adsorption von Probenmaterial an Oberflächen sowie Aggregationseffekte, haben einen dramatischen Einfluss auf die Qualität der Affinitätsdaten. Doch nur wenige state-of-the-art Technologien bieten genaue und schnelle Möglichkeiten zu überwachen und diese Effekte zu vermeiden. MST bietet integrierte Qualitätskontrollen, die erkennen und helfen, diese Probleme zu überwinden, so dass für die schrittweise Optimierung der technischen Einrichtung. ...
Offenlegungen
CE und TS sind Mitarbeiter von 2bind GmbH, die biophysikalischen analytische Dienste zur Verfügung stellt. Publikationsgebühren für dieses Video-Artikel sind für die von 2bind GmbH bezahlt.
Danksagungen
Die Autoren haben keine Bestätigungen.
Materialien
| Name | Company | Catalog Number | Comments |
| Aptamer binding buffer | 20 mM Tris pH 7.6; 300 mM NaCl; 5 mM MgCl2; 0.01% Tween-20 | ||
| Fluorescently labeled ATP aptamer | IDT, Leuven, Belgium | sequence: DH25.42 50-Cy5-CCTGGGGGAGT- ATTGCGGAGGAAGG-3 | |
| ATP | Sigma Aldrich, Germany | A2383 | 10 mM stock solutions stored at - 20 °C |
| ADP | Sigma Aldrich, Germany | A2754 | 10 mM stock solutions stored at - 20 °C |
| AMP | Sigma Aldrich, Germany | A2252 | 10 mM stock solutions stored at - 20 °C |
| Adenine | Sigma Aldrich, Germany | A8626 | 10 mM stock solutions stored at - 20 °C |
| SAM | Sigma Aldrich, Germany | A7007 | 10 mM stock solutions stored at - 20 °C |
| dATP | Sigma Aldrich, Germany | 11934511001 | 10 mM stock solutions stored at - 20 °C |
| CTP | Sigma Aldrich, Germany | C1506 | 10 mM stock solutions stored at - 20 °C |
| GTP | Sigma Aldrich, Germany | G8877 | 10 mM stock solutions stored at - 20 °C |
| Monolith NT.115 | NanoTemper Technologies, Munich, Germany | MO-G008 | Blue/Red Channel MST device with standard detector, Monolith NT115 pico is MST device with high sensitivity detector |
| Monolith NT.115 capillaries Standard | NanoTemper Technologies, Munich, Germany | MO-K002 | |
| Eppendorf PCR tubes | Eppendorf, Germany | 30124537 | |
| Monolith control software. 2.1.33, pre-installed on the device | NanoTemper Technologies, Munich, Germany | ||
| MO.affinity analysis v2.1.1 | NanoTemper Technologies, Munich, Germany | ||
| Kaleidagraph 4.5.2 | Synergy Software |
Referenzen
- Linke, P., et al. An Automated Microscale Thermophoresis Screening Approach for Fragment-Based Lead Discovery. J Biomol Screen. 21 (4), 414-421 (2015).
- Jerabek-Willemsen, M., et al. MicroScale Thermophoresis: Interaction analysis and beyond. Journal of Molecular Structure. 1077, 101-113 (2014).
- Zillner, K., et al. Microscale thermophoresis as a sensitive method to quantify protein: nucleic acid interactions in solution. Methods Mol Biol. 815, 241-252 (2012).
- Zhang, W., Duhr, S., Baaske, P., Laue, E. Microscale thermophoresis for the assessment of nuclear protein-binding affinities. Methods Mol Biol. 1094, 269-276 (2014).
- Wienken, C. J., Baaske, P., Rothbauer, U., Braun, D., Duhr, S. Protein-binding assays in biological liquids using microscale thermophoresis. Nat Commun. 1, 100 (2010).
- Seidel, S. A., et al. Label-free microscale thermophoresis discriminates sites and affinity of protein-ligand binding. Angew Chem Int Ed Engl. 51 (42), 10656-10659 (2012).
- Seidel, S. A., et al. Microscale thermophoresis quantifies biomolecular interactions under previously challenging conditions. Methods. 59 (3), 301-315 (2013).
- Schubert, T., et al. Df31 protein and snoRNAs maintain accessible higher-order structures of chromatin. Mol Cell. 48 (3), 434-444 (2012).
- McKeague, M., Derosa, M. C. Challenges and opportunities for small molecule aptamer development. J Nucleic Acids. 2012, 748913 (2012).
- Ruscito, A., DeRosa, M. C. Small-Molecule Binding Aptamers: Selection Strategies, Characterization, and Applications. Front Chem. 4, 14 (2016).
- Chang, A. L., McKeague, M., Liang, J. C., Smolke, C. D. Kinetic and equilibrium binding characterization of aptamers to small molecules using a label-free, sensitive, and scalable platform. Anal Chem. 86 (7), 3273-3278 (2014).
- Chang, A. L., McKeague, M., Smolke, C. D. Facile characterization of aptamer kinetic and equilibrium binding properties using surface plasmon resonance. Methods Enzymol. 549, 451-466 (2014).
- Jing, M., Bowser, M. T. Methods for measuring aptamer-protein equilibria: a review. Anal Chim Acta. 686 (1-2), 9-18 (2011).
- Sokoloski, J. E., Dombrowski, S. E., Bevilacqua, P. C. Thermodynamics of ligand binding to a heterogeneous RNA population in the malachite green aptamer. Biochemistry. 51 (1), 565-572 (2012).
- Burnouf, D., et al. kinITC: a new method for obtaining joint thermodynamic and kinetic data by isothermal titration calorimetry. J Am Chem Soc. 134 (1), 559-565 (2012).
- Mannironi, C., Scerch, C., Fruscoloni, P., Tocchini-Valentini, G. P. Molecular recognition of amino acids by RNA aptamers: the evolution into an L-tyrosine binder of a dopamine-binding RNA motif. RNA. 6 (4), 520-527 (2000).
- Jenison, R. D., Gill, S. C., Pardi, A., Polisky, B. High-resolution molecular discrimination by RNA. Science. 263 (5152), 1425-1429 (1994).
- Huizenga, D. E., Szostak, J. W. A DNA aptamer that binds adenosine and ATP. Biochemistry. 34 (2), 656-665 (1995).
- Lee, E. R., Baker, J. L., Weinberg, Z., Sudarsan, N., Breaker, R. R. An allosteric self-splicing ribozyme triggered by a bacterial second messenger. Science. 329 (5993), 845-848 (2010).
- Wickiser, J. K., Cheah, M. T., Breaker, R. R., Crothers, D. M. The kinetics of ligand binding by an adenine-sensing riboswitch. Biochemistry. 44 (40), 13404-13414 (2005).
- Jucker, F. M., Phillips, R. M., McCallum, S. A., Pardi, A. Role of a heterogeneous free state in the formation of a specific RNA-theophylline complex. Biochemistry. 42 (9), 2560-2567 (2003).
- Zhao, Q., Lv, Q., Wang, H. Aptamer fluorescence anisotropy sensors for adenosine triphosphate by comprehensive screening tetramethylrhodamine labeled nucleotides. Biosens Bioelectron. 70, 188-193 (2015).
- Zhang, D., et al. A sensitive fluorescence anisotropy method for detection of lead (II) ion by a G-quadruplex-inducible DNA aptamer. Anal Chim Acta. 812, 161-167 (2014).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule imaging of an in vitro-evolved RNA aptamer reveals homogeneous ligand binding kinetics. J Am Chem Soc. 131 (29), 9866-9867 (2009).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule binding experiments on long time scales. Rev Sci Instrum. 81 (8), 083705 (2010).
- Zichel, R., Chearwae, W., Pandey, G. S., Golding, B., Sauna, Z. E. Aptamers as a sensitive tool to detect subtle modifications in therapeutic proteins. PLoS One. 7 (2), 31948 (2012).
- Baaske, P., Wienken, C. J., Reineck, P., Duhr, S., Braun, D. Optical thermophoresis for quantifying the buffer dependence of aptamer binding. Angew Chem Int Ed Engl. 49 (12), 2238-2241 (2010).
- Entzian, C., Schubert, T. Studying small molecule-aptamer interactions using MicroScale Thermophoresis (MST). Methods. 97, 27-34 (2016).
- Valenzano, S., et al. Screening and Identification of DNA Aptamers to Tyramine Using in Vitro Selection and High-Throughput Sequencing. ACS Comb Sci. 18 (6), 302-313 (2016).
- Jauset Rubio, M., et al. beta-Conglutin dual aptamers binding distinct aptatopes. Anal Bioanal Chem. 408 (3), 875-884 (2016).
- Breitsprecher, D., et al. Aptamer Binding Studies Using MicroScale Thermophoresis. Methods Mol Biol. 1380, 99-111 (2016).
- Stoltenburg, R., Schubert, T., Strehlitz, B. In vitro Selection and Interaction Studies of a DNA Aptamer Targeting Protein A. PLoS One. 10 (7), 0134403 (2015).
- Kinghorn, A. B., et al. Aptamer Affinity Maturation by Resampling and Microarray Selection. Anal Chem. 88 (14), 6981-6985 (2016).
- Duhr, S., Braun, D. Why molecules move along a temperature gradient. Proc Natl Acad Sci U S A. 103 (52), 19678-19682 (2006).
- Braun, D., Libchaber, A. Trapping of DNA by thermophoretic depletion and convection. Phys Rev Lett. 89 (18), 188103 (2002).
- Duhr, S., Arduini, S., Braun, D. Thermophoresis of DNA determined by microfluidic fluorescence. Eur Phys J E Soft Matter. 15 (3), 277-286 (2004).
- Jerabek-Willemsen, M., Wienken, C. J., Braun, D., Baaske, P., Duhr, S. Molecular interaction studies using microscale thermophoresis. Assay Drug Dev Technol. 9 (4), 342-353 (2011).
- He, K., Dragnea, V., Bauer, C. E. Adenylate Charge Regulates Sensor Kinase CheS3 To Control Cyst Formation in Rhodospirillum centenum. MBio. 6 (3), 00546 (2015).
- Brvar, M., et al. Structure-based discovery of substituted 4,5'-bithiazoles as novel DNA gyrase inhibitors. J Med Chem. 55 (14), 6413-6426 (2012).
- Pogorelcnik, B., et al. 4,6-Substituted-1,3,5-triazin-2(1H)-ones as monocyclic catalytic inhibitors of human DNA topoisomerase IIalpha targeting the ATP binding site. Bioorg Med Chem. 23 (15), 4218-4229 (2015).
- Jhaveri, S., Rajendran, M., Ellington, A. D. In vitro selection of signaling aptamers. Nat Biotechnol. 18 (12), 1293-1297 (2000).
- Khavrutskii, L., et al. Protein purification-free method of binding affinity determination by microscale thermophoresis. J Vis Exp. (78), (2013).
- Ramakrishnan, M., et al. Probing cocaine-antibody interactions in buffer and human serum. PLoS One. 7 (7), 40518 (2012).
- Chen, M., et al. Antiviral activity and interaction mechanisms study of novel glucopyranoside derivatives. Bioorg Med Chem Lett. 25 (18), 3840-3844 (2015).
- Wan, C., et al. Insights into the molecular recognition of the granuphilin C2A domain with PI(4,5)P2. Chem Phys Lipids. 186 (4,5), 61-67 (2015).
- Harazi, A., et al. The Interaction of UDP-N-Acetylglucosamine 2-Epimerase/N-Acetylmannosamine Kinase (GNE) and Alpha-Actinin 2 Is Altered in GNE Myopathy M743T Mutant. Mol Neurobiol. , (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten