Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
La cartographie du site de liaison d'un aptamère sur ATP utilisant MicroScale thermophorèse
Dans cet article
Résumé
MicroScale Thermophoresis (MST) is a sensitive technology to characterize aptamer-target interactions. This manuscript describes an MST protocol to characterize aptamer-small molecule interactions.
Résumé
Characterization of molecular interactions in terms of basic binding parameters such as binding affinity, stoichiometry, and thermodynamics is an essential step in basic and applied science. MicroScale Thermophoresis (MST) is a sensitive biophysical method to obtain this important information. Relying on a physical effect called thermophoresis, which describes the movement of molecules through temperature gradients, this technology allows for the fast and precise determination of binding parameters in solution and allows the free choice of buffer conditions (from buffer to lysates/sera). MST uses the fact that an unbound molecule displays a different thermophoretic movement than a molecule that is in complex with a binding partner. The thermophoretic movement is altered in the moment of molecular interaction due to changes in size, charge, and hydration shell. By comparing the movement profiles of different molecular ratios of the two binding partners, quantitative information such as binding affinity (pM to mM) can be determined. Even challenging interactions between molecules of small sizes, such as aptamers and small compounds, can be studied by MST. Using the well-studied model interaction between the DH25.42 DNA aptamer and ATP, this manuscript provides a protocol to characterize aptamer-small molecule interactions. This study demonstrates that MST is highly sensitive and permits the mapping of the binding site of the 7.9 kDa DNA aptamer to the adenine of ATP.
Introduction
L'interaction entre les molécules est la base de la nature. Par conséquent, les scientifiques dans de nombreux domaines de la recherche fondamentale et appliquée tentent de comprendre les principes fondamentaux des interactions moléculaires de différents types. MicroScale thermophorèse (MST) permet aux scientifiques d'effectuer la caractérisation précise rapide, rentable et de qualité contrôlée des interactions moléculaires en solution, avec un libre choix de tampons. Il y a déjà plus de 1000 publications en utilisant MST, à partir de 2016 seulement, décrivant les différents types d'analyses, y compris des projections de la bibliothèque, les validations d'événements, des essais de compétition, et les expériences de liaison avec plusieurs partenaires de liaison 1-8. En général, le MST permet l'étude des paramètres de liaison classiques, tels que l'affinité de liaison (pM mM), stoechiométrie, et de la thermodynamique, de tout type d'interaction moléculaire. Un grand avantage de MST est la possibilité d'étudier des événements de liaison indépendants de la taille des partenaires d'interaction. Même chalinteractions lenging entre les petites aptamères d'acides nucléiques (15-30 nt) et des cibles telles que des petites molécules, des médicaments, des antibiotiques, ou métabolites peuvent être quantifiés.
Les technologies actuelles state-of-the-art pour caractériser les interactions aptamère-cibles sont soit en laboratoire intense et très complexe ou ne parviennent pas à quantifier les interactions aptamère-petite molécule 9,10. Surface Plasmon Resonance (SPR) à base de dosages 11,12 et approches colorimétriques vraiment sans étiquette, comme Calorimétrie titration isotherme (ITC) 13-15, élution isocratique 16, l' équilibre fi ltration 17,18, en ligne de sondage 19, gel - passer des tests, arrêt - fl ux fl uorescence spectroscopie 20,21, anisotropie de fluorescence (FA) 22,23, une seule molécule fl uorescence imagerie 24,25, et Bio-couche interférométrie (BLI) 26 sont également soit imprécis ou incompatible avec la molécule d'aptamères-petite interactions. Autres principal enjeux de ces méthodes sont une faible sensibilité, une consommation élevée de l'échantillon, l'immobilisation, les limitations de transport de masse sur les surfaces, et / ou des restrictions de mémoire tampon. Seules quelques-unes de ces technologies offrent des contrôles intégrés pour l'agrégation et d'adsorption des effets.
MST représente un outil puissant pour les scientifiques de surmonter cette limitation pour étudier les interactions entre les aptamères et les petites molécules 27-29, ainsi que d' autres cibles telles que les protéines 30-33. La technologie repose sur le mouvement de molécules à travers des gradients de température. Ce mouvement dirigé, appelé "thermophorèse," dépend de la taille, la charge, et la coquille d'hydratation de la molécule 34,35. La liaison d'un ligand à la molécule directement altérer au moins l'un de ces paramètres, ce qui entraîne une mobilité thermophorétique changé. Ligands avec de petites tailles peuvent ne pas avoir un impact considérable en termes de changement de taille de la non liée à l'état lié, mais ils peuvent avoir dr effets Amatic sur la coque et / ou la charge hydratation. Les changements dans le mouvement thermophorétique des molécules après interactions avec le partenaire de liaison permet la quantification des paramètres de liaison de base 2,7,34,36,37.
Comme cela est représenté sur la figure 1A, le dispositif MST est constitué d'un laser infrarouge focalisé sur l'échantillon à l' intérieur des tubes capillaires en verre optique en utilisant les mêmes que pour la détection de la fluorescence. Le mouvement thermophorétique de protéines via le fl uorescence intrinsèque de tryptophanes 6 ou d'un partenaire d' interaction 3,8 marqué par fluorescence peut être surveillée tandis que le laser établit un gradient de température (AT de 2-6 ° C). La différence de température dans l' espace, AT, conduit à l'épuisement ou l' accumulation de molécules dans le domaine de température élevée, qui peut être quantifiée par la Soret coef fi cient (S T):
g "/>
c représente la concentration à chaud dans la région chauffée et froid c est la concentration initiale dans la zone froide.
Comme le montre la figure 1B, un exemple typique de résultats expérimentaux MST dans un profil de mouvement MST (trace de temps), composé de différentes phases, qui peuvent être séparés par leurs échelles de temps respectives. La fluorescence initiale est mesurée au cours des 5 premières secondes en absence de gradient de température pour définir la fluorescence de départ précis et pour vérifier photoblanchiment ou photoenhancement. Le saut de température (T-Jump) représente la phase dans laquelle les changements de fluorescence avant le mouvement thermophorétique. Cette diminution initiale de la fluorescence dépend des changements de chaleur dépendant de fl uorophore rendement quantique. La phase de thermophorèse suit, dans laquelle les baisses de fluorescence (ou augmente) en raison du mouvement thermophorétique des molécules jusqu'à ce que la distribution à l'état d'équilibre est atteint.Le tjump inverse et rétrodiffusion concomitante de molécules fluorescents peuvent être observées comme indiqué dans la figure 1B après le laser est éteint. Pour accéder aux paramètres de liaison de base, différents rapports molaires des partenaires d'interaction sont analysées et comparées. En règle générale, 16 rapports différents sont étudiés dans une expérience MST, tandis que la molécule visible optique est maintenue constante et est alimentée avec une quantité croissante du ligand non marqué. L'interaction entre les deux partenaires de liaison induit des changements dans la thermophorèse, et donc dans la fluorescence normalisée, F norme, qui est calculé comme suit:
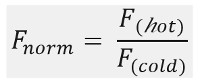
F chaud et froid F représentent en moyenne fl intensités uorescence au dé fi nies points de temps des traces du MST. Les affinités de liaison (K d ou CE 50 valeurs) peuvent être calculées par curve montage (figure 1C).
Dans l'ensemble, le MST est un outil puissant pour étudier les interactions moléculaires de toute nature. Ce manuscrit propose un protocole pour caractériser l'interaction difficile entre la petite triphosphate molécule d'adénosine (ATP; 0,5 kDa) et le 25-nt court ADNss aptamère DH25.42 (7,9 kDa). Au cours du manuscrit, le site de liaison de l'aptamère sur la molécule d'ATP est mappée vers le bas pour le groupe adénine de l'ATP.
Protocole
1. Préparation de l'aptamère de travail Stock
- Suivez les instructions du fabricant et de dissoudre l'oligonucléotide (5-Cy5-CCTG GGGGAGTATTGCGGAGGAAGG-3, la séquence de référence 18) dans l' eau, pour atteindre une concentration finale de 100 uM.
- Préparer la solution de travail en diluant aptamère l'oligonucléotide actions pour 200 nM avec un tampon de liaison (Tris 20 mM, pH 7,6, NaCl 300 mM, MgCl2 5 mM, 0,01% de Tween 20).
- Incuber le mélange pendant 2 min à 90 ° C, laisser refroidir l'échantillon immédiatement vers le bas sur la glace, et d'utiliser l'échantillon à température ambiante.
2. Préparation de la dilution de la série Ligand
- Pour chaque ligand (adénosine triphosphate (ATP), l'adénosine diphosphate (ADP), l'adénosine monophosphate (AMP), l'adénine, la guanosine triphosphate (GTP), cytosine triphosphate (CTP), désoxyadénosine triphosphate (dATP), et S-adénosyl méthionine (SAM) ; 10 mM chacun), préparer une série diluti 16 étapesdans 200 micro-tubes de réaction ul.
NOTE: Centrifugation des stocks de ligand pendant 5 min à 14000 xg peut aider à éliminer les agrégats. Faible volume des tubes à faible réaction de liaison sont recommandés pour éviter l'adsorption de molécules sur les parois du tube. - Commencer avec une concentration maximale d'au moins 50 fois supérieure à l'affinité estimée et réduire la concentration en ligand de 50% à chaque étape de dilution.
REMARQUE: L'outil concentration finder mis en œuvre dans le logiciel de contrôle simule les données de liaison et aide à trouver la plage de concentration juste pour la série de dilution. - Remplir 20 pi de ligand stock (10 mM) dans le tube 1. Ajouter 10 pi de tampon de liaison de l'aptamère dans des tubes de réaction micro 2-16.
- Transfert 10 pi de tube 1 à tube 2 et bien mélanger par pipetage de haut en bas plusieurs fois. Transférer 10 pi au tube suivant et répétez cette dilution pour les tubes restants.
- Jeter l'excès de 10 ul du dernier tube. Évitez tout bUffer effets de dilution. Le tampon dans le tube 1 et 2-16 dans les tubes doivent être identiques.
3. Préparation du mélange final de réaction
- Préparer les réactions de liaison individuelles avec un volume de 20 pi (10 pi de solution de travail aptamère + 10 pl de la dilution du ligand respectif) afin de minimiser les erreurs de pipetage. Un volume de seulement 4 pi est suf fi sante pour fi ll le capillaire.
- Ajouter 10 ul de la solution de travail 200 nM aptamère à 10 ul de chaque dilution du ligand et bien mélanger par pipetage de haut en bas plusieurs fois.
- Incuber les échantillons pendant 5 min à température ambiante et fi ll les échantillons dans les capillaires classiques en plongeant les capillaires dans l'échantillon. De plus longs temps d'incubation peuvent être nécessaires pour certaines interactions; cependant, 5 minutes est suffisante pour la plupart. Touchez les capillaires que sur les côtés, pas sur la partie centrale, où la mesure optique sera prise.
- Placer les capillaires th SUR DESe plateau capillaire et démarrer le périphérique MST.
4. À partir de l'appareil MST
NOTE: Le dispositif fournit deux logiciels pré-installés, le "logiciel pour la configuration technique des conditions expérimentales et de contrôle '' analyse« logiciel pour l'interprétation des données produites.
- Avant de placer le bac capillaire dans le dispositif MST, démarrez le logiciel de commande et de régler la température globale souhaitée en sélectionnant '' activer le contrôle manuel de la température "dans le '' contrôle de la température" menu déroulant. Ajuster la température à 25 ° C de cette manière.
REMARQUE: Les instruments peuvent être MST 22-45 ° C, à température contrôlée. - Attendez que la température pour atteindre le niveau attendu, puis placer le plateau capillaire dans le dispositif MST.
- Réglez le canal LED '' rouge "pour les colorants Cy5 et ajuster la puissance LED pour obtenir un fl uorescencsignal e de 300 à 1000 unités de fluorescence au niveau du dispositif MST avec un capteur standard. 25% de la puissance LED est utilisé dans cette étude.
NOTE: 6.000 à 18.000 unités de fluorescence sont recommandées pour le MST avec un capteur à haute sensibilité.
5. Capillaire Numériser
- Effectuer une analyse capillaire pour vérifier les différents aspects de la qualité de l'échantillon en choisissant la position capillaire sur le logiciel de «contrôle» et en cliquant sur "start cap scan" avant de commencer la mesure MST.
- Inspecter l'analyse capillaire pour la fluorescence d'amélioration / trempe et les effets de collage (U ou aplatie pics) dans le logiciel.
NOTE: Plus de détails sur la détection et la manipulation de la fluorescence et les effets de collage peuvent être trouvés dans la discussion.
6. MST Mesure
NOTE: Avant de commencer la mesure MST, assurez-vous d'exclure coller des effets, l'amélioration / effets de trempe, ouerreurs de pipetage, et veiller à ce que l'analyse capillaire indique que le signal de fluorescence est suffisante. Pour plus de détails, voir la discussion.
- Affecter les concentrations de ligand de la série de dilution à la position respective dans le capillaire "logiciel" "contrôle Considérons l'étape de dilution du mélange de l'aptamère et un ligand. (1: 1).
- Entrez la plus forte concentration de ligand (5 mM) pour capillaire # 1, sélectionnez le type de dilution correct (ici, 1: 1), cliquez sur la concentration maximale, et utiliser la fonction de glisser pour attribuer automatiquement les concentrations restantes dans les capillaires # 2- 16. La concentration la plus faible est 152,6 nm.
- Entrer la concentration de l'aptamère fluorescents (ici 100 nM) dans la section respective du logiciel de commande.
- Utilisez les paramètres par défaut, qui détectent la fl uorescence pendant 5 secondes, enregistrer le MST pendant 30 secondes, et enregistrer la fluorescence pendant encore 5 secondes après l'inactivation du laser t o surveiller la diffusion arrière de molécules.
- Régler la puissance du laser à 20%, dans la partie correspondante du logiciel de commande.
NOTE: Afin de recevoir le meilleur rapport signal-bruit et éviter les effets non spécifiques, une puissance laser de 20-40% est recommandé. Dans certains cas, une puissance laser plus élevée peut être nécessaire pour obtenir une bonne séparation des molécules non liées et liées. - Enregistrer l'expérience après avoir sélectionné le dossier de destination et démarrer la mesure MST en appuyant sur le bouton '' de mesure Démarrer MST ".
REMARQUE: Le fichier .ntp sera généré dans le dossier de destination. Grâce à cette configuration, une mesure dure 10-15 min. - Répétez la procédure expérimentale au moins deux fois pour une détermination plus précise de la valeur CE 50.
NOTE: Afin de tester la reproductibilité technique, les mêmes capillaires peuvent être numérisés à plusieurs reprises (répétitions techniques).
7. MST Analyse des données
nt "> NOTE: Le logiciel d'analyse permet à l'analyse des données sur la fl y pendant la mesure Le logiciel d'analyse trace les traces de temps MST et des changements dans la normalisation de fluorescence (F norme) par rapport à la concentration de ligand 37..- Démarrez le logiciel d'analyse MST (MO.Affinity Analysis) et charger le fichier .ntp à partir du dossier de destination. Sélectionnez "MST" comme le type d'analyse dans le menu de sélection des données.
NOTE: En cas d'effets de fluorescence ligand-dépendante, la fluorescence initiale peut être choisie pour l'analyse. - Ajouter la course technique ou biologique respective (s) à une nouvelle analyse par glisser-déposer ou en appuyant sur le bouton "+" en dessous de la course expérimentale respective.
- Appuyez sur le bouton d'information ci-dessous la course expérimentale respective pour obtenir des informations sur les propriétés de l'expérience, des traces du MST, analyse capillaire, la forme capillaire, fluorescence initiale, et le taux de blanchiment.
NOTE: Ces données brutes peuvent alalors être inspecté dans les étapes ultérieures de l'analyse. - Inspecter visuellement les traces du MST pour l'agrégation et de précipitation effets, visibles comme des bosses et des pointes.
NOTE: Pour plus d'informations sur la détection et le traitement des effets d'agrégation, lisez la discussion. - Inspecter visuellement l'analyse capillaire et le capillaire forme overlay pour les effets d'adsorption, visible sous forme de pics aplatis ou en forme de U. Inspecter visuellement l'analyse capillaire et la fluorescence initiale pour les effets de fluorescence. Inspecter visuellement le taux de blanchiment pour les effets photoblanchiment.
- Passez en mode dose-réponse et modifier le paramètre d'analyse en mode "expert" en appuyant sur la touche correspondante. Sélectionnez "T-Jump» comme stratégie d'évaluation MST.
- Sélectionnez le modèle "Hill" pour la courbe fi Prép. Les paramètres de liaison seront automatiquement calculés. Normaliser les données en choisissant le type respectif de la normalisation dans le menu "comparer les résultats". Export de lales données soit en tant .xls ou .pdf.
NOTE: Le tableau ci-dessous le graphique de liaison résume les paramètres de liaison calculés.
Résultats
Dans cette étude, le MST a été appliquée pour caractériser le site de liaison de l'aptamère ADN DH25.42 18 sur l' ATP. Contrairement à d' autres études caractérisant l'interaction de l' ATP ou d' ATP mimant les petites molécules avec des protéines marquées de façon aléatoire avec un ou plusieurs fluorophores 38-40, cette étude comprend une version marquée de l'aptamère 7,9 kDa ADNsb avec une molécule Cy5 sur l'extré...
Discussion
Les contrôles de qualité:
l'adhérence non spécifique / adsorption de l'échantillon de matériau sur les surfaces, ainsi que des effets d'agrégation, ont une influence considérable sur la qualité des données d'affinité. Cependant, seulement quelques technologies state-of-the-art offrent des options précises et rapides pour surveiller et éviter ces effets. MST offre des contrôles de qualité intégrés qui détectent et aident à surmonter ces problèmes, permettant l...
Déclarations de divulgation
CE et TS sont des employés de 2bind GmbH, qui fournit des services d'analyse biophysiques. frais de publication pour cette vidéo-article sont payés par 2bind GmbH.
Remerciements
Les auteurs ont pas accusés de réception.
matériels
| Name | Company | Catalog Number | Comments |
| Aptamer binding buffer | 20 mM Tris pH 7.6; 300 mM NaCl; 5 mM MgCl2; 0.01% Tween-20 | ||
| Fluorescently labeled ATP aptamer | IDT, Leuven, Belgium | sequence: DH25.42 50-Cy5-CCTGGGGGAGT- ATTGCGGAGGAAGG-3 | |
| ATP | Sigma Aldrich, Germany | A2383 | 10 mM stock solutions stored at - 20 °C |
| ADP | Sigma Aldrich, Germany | A2754 | 10 mM stock solutions stored at - 20 °C |
| AMP | Sigma Aldrich, Germany | A2252 | 10 mM stock solutions stored at - 20 °C |
| Adenine | Sigma Aldrich, Germany | A8626 | 10 mM stock solutions stored at - 20 °C |
| SAM | Sigma Aldrich, Germany | A7007 | 10 mM stock solutions stored at - 20 °C |
| dATP | Sigma Aldrich, Germany | 11934511001 | 10 mM stock solutions stored at - 20 °C |
| CTP | Sigma Aldrich, Germany | C1506 | 10 mM stock solutions stored at - 20 °C |
| GTP | Sigma Aldrich, Germany | G8877 | 10 mM stock solutions stored at - 20 °C |
| Monolith NT.115 | NanoTemper Technologies, Munich, Germany | MO-G008 | Blue/Red Channel MST device with standard detector, Monolith NT115 pico is MST device with high sensitivity detector |
| Monolith NT.115 capillaries Standard | NanoTemper Technologies, Munich, Germany | MO-K002 | |
| Eppendorf PCR tubes | Eppendorf, Germany | 30124537 | |
| Monolith control software. 2.1.33, pre-installed on the device | NanoTemper Technologies, Munich, Germany | ||
| MO.affinity analysis v2.1.1 | NanoTemper Technologies, Munich, Germany | ||
| Kaleidagraph 4.5.2 | Synergy Software |
Références
- Linke, P., et al. An Automated Microscale Thermophoresis Screening Approach for Fragment-Based Lead Discovery. J Biomol Screen. 21 (4), 414-421 (2015).
- Jerabek-Willemsen, M., et al. MicroScale Thermophoresis: Interaction analysis and beyond. Journal of Molecular Structure. 1077, 101-113 (2014).
- Zillner, K., et al. Microscale thermophoresis as a sensitive method to quantify protein: nucleic acid interactions in solution. Methods Mol Biol. 815, 241-252 (2012).
- Zhang, W., Duhr, S., Baaske, P., Laue, E. Microscale thermophoresis for the assessment of nuclear protein-binding affinities. Methods Mol Biol. 1094, 269-276 (2014).
- Wienken, C. J., Baaske, P., Rothbauer, U., Braun, D., Duhr, S. Protein-binding assays in biological liquids using microscale thermophoresis. Nat Commun. 1, 100 (2010).
- Seidel, S. A., et al. Label-free microscale thermophoresis discriminates sites and affinity of protein-ligand binding. Angew Chem Int Ed Engl. 51 (42), 10656-10659 (2012).
- Seidel, S. A., et al. Microscale thermophoresis quantifies biomolecular interactions under previously challenging conditions. Methods. 59 (3), 301-315 (2013).
- Schubert, T., et al. Df31 protein and snoRNAs maintain accessible higher-order structures of chromatin. Mol Cell. 48 (3), 434-444 (2012).
- McKeague, M., Derosa, M. C. Challenges and opportunities for small molecule aptamer development. J Nucleic Acids. 2012, 748913 (2012).
- Ruscito, A., DeRosa, M. C. Small-Molecule Binding Aptamers: Selection Strategies, Characterization, and Applications. Front Chem. 4, 14 (2016).
- Chang, A. L., McKeague, M., Liang, J. C., Smolke, C. D. Kinetic and equilibrium binding characterization of aptamers to small molecules using a label-free, sensitive, and scalable platform. Anal Chem. 86 (7), 3273-3278 (2014).
- Chang, A. L., McKeague, M., Smolke, C. D. Facile characterization of aptamer kinetic and equilibrium binding properties using surface plasmon resonance. Methods Enzymol. 549, 451-466 (2014).
- Jing, M., Bowser, M. T. Methods for measuring aptamer-protein equilibria: a review. Anal Chim Acta. 686 (1-2), 9-18 (2011).
- Sokoloski, J. E., Dombrowski, S. E., Bevilacqua, P. C. Thermodynamics of ligand binding to a heterogeneous RNA population in the malachite green aptamer. Biochemistry. 51 (1), 565-572 (2012).
- Burnouf, D., et al. kinITC: a new method for obtaining joint thermodynamic and kinetic data by isothermal titration calorimetry. J Am Chem Soc. 134 (1), 559-565 (2012).
- Mannironi, C., Scerch, C., Fruscoloni, P., Tocchini-Valentini, G. P. Molecular recognition of amino acids by RNA aptamers: the evolution into an L-tyrosine binder of a dopamine-binding RNA motif. RNA. 6 (4), 520-527 (2000).
- Jenison, R. D., Gill, S. C., Pardi, A., Polisky, B. High-resolution molecular discrimination by RNA. Science. 263 (5152), 1425-1429 (1994).
- Huizenga, D. E., Szostak, J. W. A DNA aptamer that binds adenosine and ATP. Biochemistry. 34 (2), 656-665 (1995).
- Lee, E. R., Baker, J. L., Weinberg, Z., Sudarsan, N., Breaker, R. R. An allosteric self-splicing ribozyme triggered by a bacterial second messenger. Science. 329 (5993), 845-848 (2010).
- Wickiser, J. K., Cheah, M. T., Breaker, R. R., Crothers, D. M. The kinetics of ligand binding by an adenine-sensing riboswitch. Biochemistry. 44 (40), 13404-13414 (2005).
- Jucker, F. M., Phillips, R. M., McCallum, S. A., Pardi, A. Role of a heterogeneous free state in the formation of a specific RNA-theophylline complex. Biochemistry. 42 (9), 2560-2567 (2003).
- Zhao, Q., Lv, Q., Wang, H. Aptamer fluorescence anisotropy sensors for adenosine triphosphate by comprehensive screening tetramethylrhodamine labeled nucleotides. Biosens Bioelectron. 70, 188-193 (2015).
- Zhang, D., et al. A sensitive fluorescence anisotropy method for detection of lead (II) ion by a G-quadruplex-inducible DNA aptamer. Anal Chim Acta. 812, 161-167 (2014).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule imaging of an in vitro-evolved RNA aptamer reveals homogeneous ligand binding kinetics. J Am Chem Soc. 131 (29), 9866-9867 (2009).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule binding experiments on long time scales. Rev Sci Instrum. 81 (8), 083705 (2010).
- Zichel, R., Chearwae, W., Pandey, G. S., Golding, B., Sauna, Z. E. Aptamers as a sensitive tool to detect subtle modifications in therapeutic proteins. PLoS One. 7 (2), 31948 (2012).
- Baaske, P., Wienken, C. J., Reineck, P., Duhr, S., Braun, D. Optical thermophoresis for quantifying the buffer dependence of aptamer binding. Angew Chem Int Ed Engl. 49 (12), 2238-2241 (2010).
- Entzian, C., Schubert, T. Studying small molecule-aptamer interactions using MicroScale Thermophoresis (MST). Methods. 97, 27-34 (2016).
- Valenzano, S., et al. Screening and Identification of DNA Aptamers to Tyramine Using in Vitro Selection and High-Throughput Sequencing. ACS Comb Sci. 18 (6), 302-313 (2016).
- Jauset Rubio, M., et al. beta-Conglutin dual aptamers binding distinct aptatopes. Anal Bioanal Chem. 408 (3), 875-884 (2016).
- Breitsprecher, D., et al. Aptamer Binding Studies Using MicroScale Thermophoresis. Methods Mol Biol. 1380, 99-111 (2016).
- Stoltenburg, R., Schubert, T., Strehlitz, B. In vitro Selection and Interaction Studies of a DNA Aptamer Targeting Protein A. PLoS One. 10 (7), 0134403 (2015).
- Kinghorn, A. B., et al. Aptamer Affinity Maturation by Resampling and Microarray Selection. Anal Chem. 88 (14), 6981-6985 (2016).
- Duhr, S., Braun, D. Why molecules move along a temperature gradient. Proc Natl Acad Sci U S A. 103 (52), 19678-19682 (2006).
- Braun, D., Libchaber, A. Trapping of DNA by thermophoretic depletion and convection. Phys Rev Lett. 89 (18), 188103 (2002).
- Duhr, S., Arduini, S., Braun, D. Thermophoresis of DNA determined by microfluidic fluorescence. Eur Phys J E Soft Matter. 15 (3), 277-286 (2004).
- Jerabek-Willemsen, M., Wienken, C. J., Braun, D., Baaske, P., Duhr, S. Molecular interaction studies using microscale thermophoresis. Assay Drug Dev Technol. 9 (4), 342-353 (2011).
- He, K., Dragnea, V., Bauer, C. E. Adenylate Charge Regulates Sensor Kinase CheS3 To Control Cyst Formation in Rhodospirillum centenum. MBio. 6 (3), 00546 (2015).
- Brvar, M., et al. Structure-based discovery of substituted 4,5'-bithiazoles as novel DNA gyrase inhibitors. J Med Chem. 55 (14), 6413-6426 (2012).
- Pogorelcnik, B., et al. 4,6-Substituted-1,3,5-triazin-2(1H)-ones as monocyclic catalytic inhibitors of human DNA topoisomerase IIalpha targeting the ATP binding site. Bioorg Med Chem. 23 (15), 4218-4229 (2015).
- Jhaveri, S., Rajendran, M., Ellington, A. D. In vitro selection of signaling aptamers. Nat Biotechnol. 18 (12), 1293-1297 (2000).
- Khavrutskii, L., et al. Protein purification-free method of binding affinity determination by microscale thermophoresis. J Vis Exp. (78), (2013).
- Ramakrishnan, M., et al. Probing cocaine-antibody interactions in buffer and human serum. PLoS One. 7 (7), 40518 (2012).
- Chen, M., et al. Antiviral activity and interaction mechanisms study of novel glucopyranoside derivatives. Bioorg Med Chem Lett. 25 (18), 3840-3844 (2015).
- Wan, C., et al. Insights into the molecular recognition of the granuphilin C2A domain with PI(4,5)P2. Chem Phys Lipids. 186 (4,5), 61-67 (2015).
- Harazi, A., et al. The Interaction of UDP-N-Acetylglucosamine 2-Epimerase/N-Acetylmannosamine Kinase (GNE) and Alpha-Actinin 2 Is Altered in GNE Myopathy M743T Mutant. Mol Neurobiol. , (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon