JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
마이크로 열 영동을 사용하여 ATP에 앱 타머의 결합 부위를 매핑
요약
MicroScale Thermophoresis (MST) is a sensitive technology to characterize aptamer-target interactions. This manuscript describes an MST protocol to characterize aptamer-small molecule interactions.
초록
Characterization of molecular interactions in terms of basic binding parameters such as binding affinity, stoichiometry, and thermodynamics is an essential step in basic and applied science. MicroScale Thermophoresis (MST) is a sensitive biophysical method to obtain this important information. Relying on a physical effect called thermophoresis, which describes the movement of molecules through temperature gradients, this technology allows for the fast and precise determination of binding parameters in solution and allows the free choice of buffer conditions (from buffer to lysates/sera). MST uses the fact that an unbound molecule displays a different thermophoretic movement than a molecule that is in complex with a binding partner. The thermophoretic movement is altered in the moment of molecular interaction due to changes in size, charge, and hydration shell. By comparing the movement profiles of different molecular ratios of the two binding partners, quantitative information such as binding affinity (pM to mM) can be determined. Even challenging interactions between molecules of small sizes, such as aptamers and small compounds, can be studied by MST. Using the well-studied model interaction between the DH25.42 DNA aptamer and ATP, this manuscript provides a protocol to characterize aptamer-small molecule interactions. This study demonstrates that MST is highly sensitive and permits the mapping of the binding site of the 7.9 kDa DNA aptamer to the adenine of ATP.
서문
분자 사이의 상호 작용은 자연의 기초가된다. 따라서, 기본 및 응용 연구의 많은 분야에서 과학자들은 서로 다른 종류의 분자 상호 작용의 기본 원리를 이해하려고합니다. 마이크로 열 영동 (MST)는 버퍼의 자유로운 선택으로, 용액에서 분자 상호 작용의 빠르고 정확한 비용 효율성 및 품질 제어 특성을 수행하는 과학자들 수 있습니다. 여러 결합 파트너 1-8으로 이벤트 유효성 검사, 경쟁 분석, 실험을 결합, 도서관 검사를 포함한 분석의 다른 종류를, 기술, 혼자 2016에서 MST를 사용하여 1,000 개 이상의 출판물이 이미 있습니다. 일반적으로, MST는 분자 상호 작용의 어떤 종류의 결합 친화 (mM의 PM), 화학 양론, 열역학, 같은 고전적인 바인딩 매개 변수에 대한 연구를 허용합니다. MST의 큰 장점은 상호 작용 파트너의 크기의 독립적 인 바인딩 이벤트를 공부할 수있는 기능입니다. 심지어 chal같은 작은 분자, 약물, 항생제, 또는 대사 산물로 같은의 작은 핵산 앱 타머 사이의 상호 작용 (NT를 15 ~ 30) 및 목표 정량화 할 수있다.
현재 최첨단 기술은 압 타머 - 대상의 상호 작용이 실험실 강렬하고 매우 복잡한 중 하나입니다 특성화 또는 앱 타머 - 작은 분자가 9,10의 상호 작용을 정량화하는 데 실패합니다. 플라즈몬 공명 (SPR)를 분석 (11, 12) 및 등온 적정 열량계 (ITC) 13 ~ 15, 등용 매 용리 16 평형 Fi를 여과에 17, 18으로 진정으로 라벨없는 열량 접근 방식을 기반 표면에 라인 19, 겔을 프로빙 분석을 이동, stopped- FL 흐름 FL uorescence 분광법 (20, 21), 형광 이방성 (FA) 22, 23, 단일 분자 FL uorescence 영상 24, 25, 및 바이오 층 간섭 (BLI) (26)는 압 타머 - 작은 분자와 부정확 또는 호환되지 않는 중 하나입니다 상호 작용. 기타 principa이러한 방법의 리터 문제는 낮은 민감도, 높은 샘플 소비, 고정, 표면에 대량 전송 제한 및 / 또는 버퍼 제한됩니다. 이러한 기술의 몇 응집 흡착 효과에 대한 통합 제어를 제공합니다.
MST 과학자 단백질 30-33로서 앱 타머 및 소분자 27-29 사이의 상호 작용뿐만 아니라 다른 대상을 연구하는 이러한 한계를 극복하기위한 강력한 도구를 나타낸다. 이 기술은 온도 구배를 통해 분자의 움직임에 의존한다. 라고하는이 감독 운동 "열 영동는"분자 (34, 35)의 크기, 비용 및 수화 쉘에 따라 달라집니다. 분자에 대한 리간드의 결합을 직접 변경 thermophoretic 이동성 결과 이들 파라미터 중 적어도 하나를 변경한다. 작은 크기와 리간드가 결합 된 상태로 결합되지 않은에서 크기 변화의 측면에서 상당한 영향을 미칠하지 않을 수 있습니다,하지만 그들은 닥터를 가질 수 있습니다 수화 쉘 및 / 또는 충전 amatic 효과. 결합 파트너와의 상호 작용 후 분자의 thermophoretic 운동의 변화는 기본 바인딩 매개 변수 2,7,34,36,37의 정량화 할 수 있습니다.
도 1a에 도시 된 바와 같이, MST 장치는 형광 검출과 같은 광학 장치를 이용하여 유리 모세관 내 샘플 상에 초점 적외선 레이저로 구성된다. 레이저 온도 구배 (2-6 ℃의 ΔT)을 설정하면서 tryptophans 6 또는 형광 표지의 상호 작용 파트너의 3,8의 고유의 FL uorescence 통한 단백질의 thermophoretic 움직임 모니터링 할 수있다. 공간 ΔT에서 생성 된 온도 차이는 Soret 의해 정량화 될 수있다 고온의 영역에서 공핍 또는 분자의 축적에 이르게 COEF 효율적인 인터넷 (S T)
g "/>
C 고온 가열 영역의 농도를 나타내고, C 감기 초기 저온 영역에서의 농도이다.
도 1b는 각자의 시간 척도에 의해 분리 될 수있는 여러 단계들로 구성된 MST 운동 프로파일 (시간 추적)에서 전형적인 MST 실험 결과에 도시 된 바와 같이. 초기 형광 정확한 시작 형광을 정의하고 또는 photoenhancement photobleaching에 대해 확인하기 위해 온도 구배의 부재에서 제 5 초 단위로 측정된다. 온도 점프 (T-점프)하는 형광 변화 thermophoretic 운동 전에 위상을 나타냅니다. 형광이 초기 감소는 양자 수율 uorophore 플로리다의 열 의존적 변화에 따라 달라집니다. 열 영동 상 인해 정상 분포까지 분자의 운동 thermophoretic 형광이 감소 (또는 증가)에 도달 한 다음.레이저가 꺼진 후 그림 (b)에 나타낸 바와 같이 역 TJump와 플로리다 uorescent 분자의 수반 다시 확산 관찰 할 수있다. 기본적인 결합 매개 변수를 액세스하기 위해, 상호 작용 파트너의 상이한 몰비를 분석 및 비교된다. 광학 표시 분자가 일정하게 유지되고, 표지 된 리간드의 양의 증가와 함께 공급되는 반면, 일반적으로 16 개의 다른 비율 번 MST 실험에서 연구되었다. 두 결합 파트너 간의 상호 작용은 열 영동의 변화를 유도하고, 따라서 표준화의 FL uorescence에서는, 다음과 같이 계산된다 F 규범 :
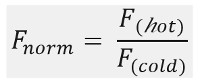
F 뜨거운 F 감기는 MST 추적의 드 Fi를 NED 시점에서 플라이 uorescence 강도 평균 나타냅니다. 결합 친화도 (K d 또는 EC 50 값) CURV 의해 계산 될 수있다E 피팅 (도 1C).
전반적으로, MST는 어떤 종류의 분자 상호 작용을 연구하는 강력한 도구입니다. 그리고 25-NT의 짧은 ssDNA를 앱 타머 DH25.42 (7.9 kDa의)이 원고는 작은 분자 아데노신 트리 포스페이트 (0.5 kDa의 ATP) 사이의 도전적인 상호 작용을 특성화하기위한 프로토콜을 제공합니다. 원고의 과정 동안, ATP 분자의 앱 타머의 결합 부위는 ATP의 아데닌 그룹에 아래로 매핑됩니다.
프로토콜
앱타 머는 작업 증권 1. 준비
- 제조업체의 지침에 따라 100 μm의 최종 농도에 도달, 물 (참조 18에서 5-Cy5에-CCTG GGGGAGTATTGCGGAGGAAGG-3, 순서) 올리고 뉴클레오티드를 녹인다.
- 버퍼를 결합 200 nm의에 올리고 뉴클레오티드 주식을 희석하여 앱 타머 작업 솔루션을 준비합니다 (20 mM 트리스, 산도 7.6, 300 mM의 NaCl을 5 밀리미터의 MgCl 2, 0.01 % 트윈 20).
- 90 ℃에서 2 분 동안 혼합 배양 한 샘플을 즉시 얼음에 식혀 실온에서 시료를 사용한다.
리간드 희석 시리즈 2. 준비
- ) SAM (각 리간드 (아데노신 트리 포스페이트 (ATP), 아데노신 디 포스페이트 (ADP), 아데노신 모노 포스페이트 (AMP), 아데닌, 구아노 신 트리 포스페이트 (GTP), 시토신 트리 포스페이트 (CTP), 아데노신 트리 포스페이트 (dATP 존재) 및 S-아데노 실 메티오닌 10 mM의 스톡 각)는 16 단계 직렬 diluti 준비에 200 ㎕를 마이크로 반응 튜브.
참고 : 집계를 제거하는 데 도움이 될 수 14,000 XG에서 5 분 리간드 주식의 원심 분리. 낮은 볼륨, 낮은 결합 반응 튜브는 튜브 벽 분자의 흡착을 방지하는 것이 좋습니다. - 추정 친화력보다 적어도 50 배의 최대 농도로 시작하여 각 희석 단계에서 50 % 리간드 농도를 감소시킨다.
참고 : 제어 소프트웨어로 구현 농도 측정기 도구는 데이터 바인딩을 시뮬레이션하고 희석 시리즈에 적합한 농도 범위를 찾는 데 도움이됩니다. - 16 마이크로 반응 튜브에 2 앱 타머 바인딩 버퍼의 10 μl를 추가 튜브 1의 리간드 재고 (10 mM)을 20 μl를 입력합니다.
- 이전 10 관 2 관 (1)의 μl의 최대 피펫 팅에 의해 여러 번 아래로 적절하게 혼합한다. 다음 튜브에 10 μL를 전송하고 나머지 관이 희석을 반복합니다.
- 마지막 튜브에서 10 μl를 초과 폐기하십시오. 어떤 B를 피하십시오uffer 희석 효과. 튜브 1 및 튜브 2-16의 버퍼가 동일해야합니다.
최종 반응 믹스 3. 준비
- 피펫 팅의 오차를 최소화하도록 각각의 결합 20 μL의 부피와 반응 (앱 타머 작업 용액 + 각 리간드 희석액 10 ㎕를 10 μL)을 준비한다. 만 4 μL의 볼륨은 모세관 LL Fi에 SUF 과학 효율적인이다.
- 각 리간드 희석의 10 μL에 200 nm의 앱 타머 작업 용액 10 μl를 추가로 pipetting에 의해 여러 번 아래로 적절하게 혼합한다.
- 샘플에 모세관을 담가 모세관 기준으로 샘플 LL 실온 인터넷에서 5 분간 샘플을 인큐베이션. 긴 배양 시간은 약간의 상호 작용에 대한 필요가있을 수있다; 그러나, 5 분은 대부분 적합합니다. NOT 광학 측정이 수행 될 중간 부분에 만 측에 모세 혈관을 터치합니다.
- 일 상 모세 혈관을 배치전자 모세관 트레이와 MST 장치를 시작합니다.
4. MST 장치 시작
참고 :이 장치는 두 개의 사전 설치된 소프트웨어 패키지, 생성 된 데이터의 해석을위한 소프트웨어 '' ''분석 제어 실험 조건과의 기술적 설정을위한 소프트웨어 '를 "제공합니다.
- 산악 표준시 장치에 모세관 트레이를 배치하기 전에 제어 소프트웨어를 시작하고 '드롭 다운 메뉴' ''온도 조절에서 "수동 온도 제어를 가능하게 '선택하여 전체 원하는 온도를 조정합니다. 이 방법으로 25 ° C의 온도를 조정합니다.
참고 : MST 악기 온도 조절 45 ° C에 22에서 할 수있다. - 예상 수준에 도달하는 온도 기다린 다음 MST 장치에 모세관 트레이를 배치합니다.
- Cy5에 염료은 "빨간색 ''에 LED 채널을 설정하고 FL uorescenc 얻을 수있는 LED 전력을 조정표준 센서와 MST 장치 300 1,000 형광 단위의 전자 신호를 출력한다. 25 % LED 전원이 연구에 사용된다.
참고 : 6,000 18,000 형광 단위 고감도 센서와 MST 권장합니다.
5. 모세관 스캔
- "컨트롤"소프트웨어에 대한 모세관의 위치를 선택하고 클릭하여 샘플의 다른 품질의 측면을 확인하는 모세 혈관 검사를 수행합니다 "캡 스캔을 시작" 산악 표준시 측정을 시작하기 전에.
- 형광 향상 / 담금질 및 소프트웨어의 고집 효과 (피크를 U 자형 또는 평평)의 모세관 스캔을 검사합니다.
참고 : 탐지 및 형광 처리 및 접착 효과에 대한 자세한 정보는 토론에서 찾을 수 있습니다.
6. MST 측정
참고 : MST 측정을 시작하기 전에 제외해야합니다 고집 효과, 강화 / 담금질 효과, 또는피펫 오류 및 모세관 스캔이 플라이 uorescence 신호가 SUF 과학 효율적인 있음을 나타냅니다 있는지 확인합니다. 자세한 내용은 설명을 참조하십시오.
- . '제어'소프트웨어의 각 모세관 위치로 희석 시리즈 리간드 농도 할당 앱타 머는 리간드와 혼합 희석 단계 고려 (1 : 1).
- 모세관 # 1에 대한 리간드의 높은 농도 (5 mm)를 입력 올바른 희석 유형을 선택합니다 (여기서, 1 : 1), 최대 농도를 클릭하고 자동으로 모세 혈관의 나머지 농도 # 2를 할당하는 드래그 기능을 사용 16. 가장 낮은 농도는 152.6 ㎚이다.
- 제어 소프트웨어의 각 부분의 FL uorescent 앱 타머 (여기서, 100 ㎚)의 농도를 입력한다.
- 5 초 동안의 FL uorescence 검출의 기본 설정을 사용하여 30 초 동안 MST를 기록하고, 상기 레이저 t의 비활성화 후 상기 5 초 동안 형광을 기록 O 분자의 확산을 다시 모니터링한다.
- 제어 소프트웨어의 각 부분에 20 %의 레이저 파워를 조정한다.
주 : 위해 최상의 신호 대 잡음비를 수신하고 비특이적 효과를 방지하기 위해서는 20 내지 40 %의 레이저 파워가 권장된다. 특정한 경우에있어서, 고출력 레이저는 비 결합 및 결합 분자의 양호한 분리를 얻기 위하여 요구 될 수있다. - 대상 폴더를 선택한 후 실험을 저장하고 '시작 MST 측정 "버튼을 눌러 MST 측정을 시작합니다.
주 : .ntp 파일을 대상 폴더에 생성됩니다. 이 설정을 사용하여, 하나의 측정은 10 ~ 15 분 동안 지속됩니다. - EC의 값이 50보다 정확한 판정을 위해 적어도 두 실험 절차를 반복한다.
참고 기술의 재현성을 시험하기 위해서, 동일한 모세관 스캔 할 수 수회 (기술 반복).
7. MST 데이터 분석
NT "> 주 : 분석 소프트웨어는 측정 동안 플라이에서 데이터의 분석을 가능하게 분석 소프트웨어 리간드 농도 37 대 MST 시간 추적 및 정규화 플로리다 uorescence (F 놈)의 변화를 나타내는..- 산악 표준시 분석 소프트웨어 (MO.Affinity 분석)를 시작하고 대상 폴더에서 .ntp 파일을로드합니다. 데이터 선택 메뉴에서 분석 유형으로 "MST"를 선택합니다.
주 : 리간드 의존적 형광 효과의 경우, 초기 형광 분석을 위해 선택 될 수있다. - 드래그 앤 드롭하거나 각각의 실험 실행 아래의 "+"버튼을 눌러 새로운 분석 각각의 기술 또는 생물학적 실행 (들)을 추가합니다.
- 실험, MST 추적, 모세 혈관 검사, 모세관 모양, 초기 형광, 표백 속도의 특성에 대한 정보를 얻기 위해 각각의 실험 실행 아래 정보 버튼을 누릅니다.
참고 :이 원시 데이터 수 등그래서 분석 이후 단계에서 검사 될 수있다. - 시각적으로 볼 범프와 스파이크로 응집 및 침전 효과에 대한 MST 추적을 검사합니다.
참고 : 검색 및 통합 효과의 처리에 대한 자세한 내용은 설명을 참조하십시오. - 시각적으로 평평하게 또는 U 자 모양의 봉우리로 보이는 모세 혈관 검사 및 흡착 효과 모세관 모양 오버레이를 검사합니다. 시각적으로 모세관 검사 및 형광 효과에 대한 초기 형광을 검사합니다. 시각적 효과를 photobleaching에 대한 표백 속도를 검사합니다.
- 용량 반응 모드로 전환하고, 각각의 버튼을 누름에 의해 "전문가"모드 분석 설정을 변경. 산악 표준시 평가 전략으로 "T-점프"를 선택합니다.
- 곡선 Fi를 tting에 대해 "힐"모델을 선택합니다. 바인딩 매개 변수가 자동으로 계산됩니다. 은 "결과를 비교"메뉴 정규화의 각 유형을 선택하여 데이터를 정규화한다. 내보내.XLS 또는 .PDF 등 중 데이터.
참고 : 바인딩 그래프 아래의 표는 계산 된 바인딩 매개 변수를 요약 한 것입니다.
결과
본 연구에서는, MST는 ATP의 DH25.42 DNA 앱 타머 (18)의 결합 부위의 특성을 적용 하였다. ATP 또는 임의로 하나 이상의 형광 38-40 표지 단백질과 소형 분자 ATP를 흉내 낸의 상호 작용의 특성을 다른 연구와 대조적으로, 본 연구에서는 5 '말단에 한 Cy5의 분자와 7.9 kDa의 ssDNA를 앱 타머에 표지 된 버전을 포함 . 다른 ATP 유도체 및 관련 분자는 다양한 위치에서 ATP ?...
토론
품질 관리 :
비특이적 부착 / 표면에 시료 물질뿐만 아니라, 응집 효과를 흡착, 선호도 데이터의 품질에 큰 영향을 미친다. 그러나, 몇 최첨단 기술을 모니터링하고 이러한 효과를 방지하기 정확하고 빠른 옵션을 제공합니다. MST 감지하고 기술적 인 설치의 단계적 최적화를 허용, 이러한 문제를 극복하는 데 도움이 통합 된 품질 관리를 제공합니다. 응집 / 침전 효과 (단계 7.7)의 ...
공개
CE 및 TS는 생물 리 학적 분석 서비스를 제공 2bind GmbH의, 직원 수 있습니다. 이 비디오 기사에 대한 출판 비용은 2bind GmbH에 의해 지불된다.
감사의 말
저자는 어떤 승인이 없습니다.
자료
| Name | Company | Catalog Number | Comments |
| Aptamer binding buffer | 20 mM Tris pH 7.6; 300 mM NaCl; 5 mM MgCl2; 0.01% Tween-20 | ||
| Fluorescently labeled ATP aptamer | IDT, Leuven, Belgium | sequence: DH25.42 50-Cy5-CCTGGGGGAGT- ATTGCGGAGGAAGG-3 | |
| ATP | Sigma Aldrich, Germany | A2383 | 10 mM stock solutions stored at - 20 °C |
| ADP | Sigma Aldrich, Germany | A2754 | 10 mM stock solutions stored at - 20 °C |
| AMP | Sigma Aldrich, Germany | A2252 | 10 mM stock solutions stored at - 20 °C |
| Adenine | Sigma Aldrich, Germany | A8626 | 10 mM stock solutions stored at - 20 °C |
| SAM | Sigma Aldrich, Germany | A7007 | 10 mM stock solutions stored at - 20 °C |
| dATP | Sigma Aldrich, Germany | 11934511001 | 10 mM stock solutions stored at - 20 °C |
| CTP | Sigma Aldrich, Germany | C1506 | 10 mM stock solutions stored at - 20 °C |
| GTP | Sigma Aldrich, Germany | G8877 | 10 mM stock solutions stored at - 20 °C |
| Monolith NT.115 | NanoTemper Technologies, Munich, Germany | MO-G008 | Blue/Red Channel MST device with standard detector, Monolith NT115 pico is MST device with high sensitivity detector |
| Monolith NT.115 capillaries Standard | NanoTemper Technologies, Munich, Germany | MO-K002 | |
| Eppendorf PCR tubes | Eppendorf, Germany | 30124537 | |
| Monolith control software. 2.1.33, pre-installed on the device | NanoTemper Technologies, Munich, Germany | ||
| MO.affinity analysis v2.1.1 | NanoTemper Technologies, Munich, Germany | ||
| Kaleidagraph 4.5.2 | Synergy Software |
참고문헌
- Linke, P., et al. An Automated Microscale Thermophoresis Screening Approach for Fragment-Based Lead Discovery. J Biomol Screen. 21 (4), 414-421 (2015).
- Jerabek-Willemsen, M., et al. MicroScale Thermophoresis: Interaction analysis and beyond. Journal of Molecular Structure. 1077, 101-113 (2014).
- Zillner, K., et al. Microscale thermophoresis as a sensitive method to quantify protein: nucleic acid interactions in solution. Methods Mol Biol. 815, 241-252 (2012).
- Zhang, W., Duhr, S., Baaske, P., Laue, E. Microscale thermophoresis for the assessment of nuclear protein-binding affinities. Methods Mol Biol. 1094, 269-276 (2014).
- Wienken, C. J., Baaske, P., Rothbauer, U., Braun, D., Duhr, S. Protein-binding assays in biological liquids using microscale thermophoresis. Nat Commun. 1, 100 (2010).
- Seidel, S. A., et al. Label-free microscale thermophoresis discriminates sites and affinity of protein-ligand binding. Angew Chem Int Ed Engl. 51 (42), 10656-10659 (2012).
- Seidel, S. A., et al. Microscale thermophoresis quantifies biomolecular interactions under previously challenging conditions. Methods. 59 (3), 301-315 (2013).
- Schubert, T., et al. Df31 protein and snoRNAs maintain accessible higher-order structures of chromatin. Mol Cell. 48 (3), 434-444 (2012).
- McKeague, M., Derosa, M. C. Challenges and opportunities for small molecule aptamer development. J Nucleic Acids. 2012, 748913 (2012).
- Ruscito, A., DeRosa, M. C. Small-Molecule Binding Aptamers: Selection Strategies, Characterization, and Applications. Front Chem. 4, 14 (2016).
- Chang, A. L., McKeague, M., Liang, J. C., Smolke, C. D. Kinetic and equilibrium binding characterization of aptamers to small molecules using a label-free, sensitive, and scalable platform. Anal Chem. 86 (7), 3273-3278 (2014).
- Chang, A. L., McKeague, M., Smolke, C. D. Facile characterization of aptamer kinetic and equilibrium binding properties using surface plasmon resonance. Methods Enzymol. 549, 451-466 (2014).
- Jing, M., Bowser, M. T. Methods for measuring aptamer-protein equilibria: a review. Anal Chim Acta. 686 (1-2), 9-18 (2011).
- Sokoloski, J. E., Dombrowski, S. E., Bevilacqua, P. C. Thermodynamics of ligand binding to a heterogeneous RNA population in the malachite green aptamer. Biochemistry. 51 (1), 565-572 (2012).
- Burnouf, D., et al. kinITC: a new method for obtaining joint thermodynamic and kinetic data by isothermal titration calorimetry. J Am Chem Soc. 134 (1), 559-565 (2012).
- Mannironi, C., Scerch, C., Fruscoloni, P., Tocchini-Valentini, G. P. Molecular recognition of amino acids by RNA aptamers: the evolution into an L-tyrosine binder of a dopamine-binding RNA motif. RNA. 6 (4), 520-527 (2000).
- Jenison, R. D., Gill, S. C., Pardi, A., Polisky, B. High-resolution molecular discrimination by RNA. Science. 263 (5152), 1425-1429 (1994).
- Huizenga, D. E., Szostak, J. W. A DNA aptamer that binds adenosine and ATP. Biochemistry. 34 (2), 656-665 (1995).
- Lee, E. R., Baker, J. L., Weinberg, Z., Sudarsan, N., Breaker, R. R. An allosteric self-splicing ribozyme triggered by a bacterial second messenger. Science. 329 (5993), 845-848 (2010).
- Wickiser, J. K., Cheah, M. T., Breaker, R. R., Crothers, D. M. The kinetics of ligand binding by an adenine-sensing riboswitch. Biochemistry. 44 (40), 13404-13414 (2005).
- Jucker, F. M., Phillips, R. M., McCallum, S. A., Pardi, A. Role of a heterogeneous free state in the formation of a specific RNA-theophylline complex. Biochemistry. 42 (9), 2560-2567 (2003).
- Zhao, Q., Lv, Q., Wang, H. Aptamer fluorescence anisotropy sensors for adenosine triphosphate by comprehensive screening tetramethylrhodamine labeled nucleotides. Biosens Bioelectron. 70, 188-193 (2015).
- Zhang, D., et al. A sensitive fluorescence anisotropy method for detection of lead (II) ion by a G-quadruplex-inducible DNA aptamer. Anal Chim Acta. 812, 161-167 (2014).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule imaging of an in vitro-evolved RNA aptamer reveals homogeneous ligand binding kinetics. J Am Chem Soc. 131 (29), 9866-9867 (2009).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule binding experiments on long time scales. Rev Sci Instrum. 81 (8), 083705 (2010).
- Zichel, R., Chearwae, W., Pandey, G. S., Golding, B., Sauna, Z. E. Aptamers as a sensitive tool to detect subtle modifications in therapeutic proteins. PLoS One. 7 (2), 31948 (2012).
- Baaske, P., Wienken, C. J., Reineck, P., Duhr, S., Braun, D. Optical thermophoresis for quantifying the buffer dependence of aptamer binding. Angew Chem Int Ed Engl. 49 (12), 2238-2241 (2010).
- Entzian, C., Schubert, T. Studying small molecule-aptamer interactions using MicroScale Thermophoresis (MST). Methods. 97, 27-34 (2016).
- Valenzano, S., et al. Screening and Identification of DNA Aptamers to Tyramine Using in Vitro Selection and High-Throughput Sequencing. ACS Comb Sci. 18 (6), 302-313 (2016).
- Jauset Rubio, M., et al. beta-Conglutin dual aptamers binding distinct aptatopes. Anal Bioanal Chem. 408 (3), 875-884 (2016).
- Breitsprecher, D., et al. Aptamer Binding Studies Using MicroScale Thermophoresis. Methods Mol Biol. 1380, 99-111 (2016).
- Stoltenburg, R., Schubert, T., Strehlitz, B. In vitro Selection and Interaction Studies of a DNA Aptamer Targeting Protein A. PLoS One. 10 (7), 0134403 (2015).
- Kinghorn, A. B., et al. Aptamer Affinity Maturation by Resampling and Microarray Selection. Anal Chem. 88 (14), 6981-6985 (2016).
- Duhr, S., Braun, D. Why molecules move along a temperature gradient. Proc Natl Acad Sci U S A. 103 (52), 19678-19682 (2006).
- Braun, D., Libchaber, A. Trapping of DNA by thermophoretic depletion and convection. Phys Rev Lett. 89 (18), 188103 (2002).
- Duhr, S., Arduini, S., Braun, D. Thermophoresis of DNA determined by microfluidic fluorescence. Eur Phys J E Soft Matter. 15 (3), 277-286 (2004).
- Jerabek-Willemsen, M., Wienken, C. J., Braun, D., Baaske, P., Duhr, S. Molecular interaction studies using microscale thermophoresis. Assay Drug Dev Technol. 9 (4), 342-353 (2011).
- He, K., Dragnea, V., Bauer, C. E. Adenylate Charge Regulates Sensor Kinase CheS3 To Control Cyst Formation in Rhodospirillum centenum. MBio. 6 (3), 00546 (2015).
- Brvar, M., et al. Structure-based discovery of substituted 4,5'-bithiazoles as novel DNA gyrase inhibitors. J Med Chem. 55 (14), 6413-6426 (2012).
- Pogorelcnik, B., et al. 4,6-Substituted-1,3,5-triazin-2(1H)-ones as monocyclic catalytic inhibitors of human DNA topoisomerase IIalpha targeting the ATP binding site. Bioorg Med Chem. 23 (15), 4218-4229 (2015).
- Jhaveri, S., Rajendran, M., Ellington, A. D. In vitro selection of signaling aptamers. Nat Biotechnol. 18 (12), 1293-1297 (2000).
- Khavrutskii, L., et al. Protein purification-free method of binding affinity determination by microscale thermophoresis. J Vis Exp. (78), (2013).
- Ramakrishnan, M., et al. Probing cocaine-antibody interactions in buffer and human serum. PLoS One. 7 (7), 40518 (2012).
- Chen, M., et al. Antiviral activity and interaction mechanisms study of novel glucopyranoside derivatives. Bioorg Med Chem Lett. 25 (18), 3840-3844 (2015).
- Wan, C., et al. Insights into the molecular recognition of the granuphilin C2A domain with PI(4,5)P2. Chem Phys Lipids. 186 (4,5), 61-67 (2015).
- Harazi, A., et al. The Interaction of UDP-N-Acetylglucosamine 2-Epimerase/N-Acetylmannosamine Kinase (GNE) and Alpha-Actinin 2 Is Altered in GNE Myopathy M743T Mutant. Mol Neurobiol. , (2016).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유