A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
זיהוי של קינאז תלוי-Cyclin 1 אתרי זירחון ספציפיים על-ידי קינאז במבחנה Assay
* These authors contributed equally
In This Article
Summary
קינאז תלוי-cyclin 1 (Cdk1) מופעל בשלב G2 של מחזור התא, מסדיר משעולים הסלולר רבים. כאן, אנו מציגים את פרוטוקול להצפנה assay קינאז במבחנה עם Cdk1, אשר מאפשר זיהוי אתרי זירחון ספציפי Cdk1 להקמת מטרות תאיות של קינאז חשוב.
Abstract
קינאז תלוי-cyclin 1 (Cdk1) הוא בקר ראשיים עבור המחזור תאים פרוקריוטים כל ו phosphorylates של % 8-13 המשוערת של פרוטאום; עם זאת, המספר של מטרות מזוהה Cdk1, במיוחד בתאי אדם הוא עדיין נמוך. הזיהוי של אתרי זירחון ספציפי Cdk1 חשוב, כפי שהם מספקים תובנות מכניסטית איך Cdk1 שולט מחזור התא. מחזור התא תקנה הוא קריטי עבור נאמן כרומוזום סגרגציה, פגמים בתהליך מורכב זה להוביל סטיות כרומוזומליות וסרטן.
כאן, אנו מתארים של במבחנה קינאז assay המשמש כדי לזהות אתרי זירחון ספציפית Cdk1. זה assay, חלבון מטוהרים שוכן phosphorylated במבחנה על ידי זמינים מסחרית מוצלחת ב' בני אדם Cdk1/cyclin זרחון אושר על ידי עמודים מרחביות, אתרי זירחון לאחר מכן מזוהים באמצעות ספקטרומטר מסה. נתאר גם פרוטוקולים טיהור המניבים ההכנות חלבון מאוד טהור, הומוגני מתאים וזמינותו קינאז, ואת וזמינותו של איגוד עבור אימות פונקציונלי של האתרים זרחון מזוהה, אשר חוקרת את האינטראקציה בין אות לוקליזציה הגרעינית הקלאסית (cNLS), שלה α karyopherin קולטן של נשק גרעיני. כדי לסייע עם עיצוב ניסיוני, אנו בודקים גישות לחיזוי של אתרי זירחון ספציפי Cdk1 מ רצפי חלבונים. יחד פרוטוקולים אלה מציגים גישה רבת עוצמה מניב אתרי זירחון Cdk1 ספציפית ומאפשרת מחקרים מכניסטית לתוך כמה Cdk1 שולט מחזור התא. מאז בשיטה זו מסתמך על חלבונים מטוהרים, יכול להחיל אותה על כל דגם האורגניזם, התשואות תוצאות אמינות, במיוחד בשילוב עם לימודי תפקודי התא.
Introduction
Kinases הם אנזימים להעביר קבוצות פוספט מ- ATP על גבי מצעים ולווסת תהליכים תאיים רבים. זרחון זה הוא הפיך, מהר, מוסיף שני מטענים שליליים, ו מאחסן אנרגיה חופשית, לאחד השינויים posttranslational הנפוצים ביותר בשימוש על ידי התאים. Cdk1, אשר ידוע גם בשם homolog חלבון 2 מחזור חלוקת התא (cdc2) הוא בקר ראשיים עבור מחזור התא כל פרוקריוטים1,2,3,4,5, phosphorylates משוער 8-13% פרוטאום6,7.
בעוד מחקרים פרוטיאומיה מבנית זיהו אתרים רבים זרחון של חלבונים, ברוב המקרים, קינאז האחראי לבצע שינויים אלה אינו ידוע. מספר יעדים Cdk1 הידוע, במיוחד בתאי אדם הוא נמוך7. הזיהוי של אתרי זירחון ספציפי Cdk1 חשובה, שכן היא מאפשרת מכניסטית מחקרים הקובעים איך Cdk1 שולט מחזור התא. מחזור התא תקנה חשוב עבור נאמן כרומוזום סגרגציה, חלוקת התא, מספר עצום של תהליכים תאיים צריך להתרחש כדי לתמוך תפקיד פיזיולוגי החשוב הזה. זה כולל עצירת תמלול ותרגום לפני תחילתה של מיטוזה, וכן ארגון מחדש דרמטי בתוך המבנה התאי של הארגון, כגון פירוק של מעטפת הגרעין, כרומוזום עיבוי, פלך mitotic הרכבה. והסחר הבינלאומי שגיאות בתהליכים אלה לגרום סרטן, מומים מולדים או מוות של תאים mitotic. מעכבים ספציפיים של Cdk1 כגון RO-3306 היו מפותחות8, אשר מספקים כלים רבי-עוצמה ללימודי פונקציונלי, חלק מעכבי אלה הם כיום בניסויים קליניים לטיפול בסרטן (ראה9 לבדיקה).
כאן, אנו מתארים של במבחנה קינאז assay המאפשר זיהוי אתרי זירחון ספציפית Cdk1. ב הזה assay, זמינים מסחרית B Cdk1/cyclin האנושי משמש phosphorylate יישום יעד מטוהרים חלבון במבחנה. זירחון של מצע מגבירה את המאסה שלו ומוסיף שני מטענים שליליים; לכן, זרחון מוצלחת אושר על ידי משמרת כלפי מעלה של הלהקה ג'ל חלבון בדף-מרחביות. אתרי זירחון ספציפי Cdk1 לאחר מכן מזוהים באמצעות ספקטרומטר מסה ניתוח של החלבון במבחנה phosphorylated. כדי לסייע עם עיצוב ניסיוני, אנו בודקים גם כלים חישוביים והפניות לחיזוי של אתרי זירחון ספציפי Cdk1 מהרצף חלבון. יתר על כן, אנו גם מתארים פרוטוקולים טיהור המניבים ההכנות חלבון מאוד טהור, הומוגני מתאים וזמינותו קינאז. לבסוף, יש צורך לאמת את אתרי זירחון שזוהה על ידי מחקרים פונקציונלי, וזמינותו איגוד פשוט מתואר כאן למטרה זו. משולב, זה בגישה רב עוצמה מניב אתרי זירחון Cdk1 ספציפית ומאפשרת לימודים מכניסטית לתוך כמה Cdk1 שולט מחזור התא7,10,11. מאז בשיטה זו מסתמך על חלבונים מטוהרים, ניתן להחיל אותה על כל דגם האורגניזם, התשואות תוצאות אמינות. עם זאת, אימות פונקציונלי של ה זרחון שהושג אתרי במבחנה מומלץ, כמו תאים יש מנגנונים רגולטוריים נוספים במקום, כמו שינויים posttranslational, אינטראקציה עם שותפים או לוקליזציה הסלולר זה עלול להפוך את אתרי זירחון נגיש או נגיש עבור זיהוי על-ידי Cdk1.
Cdk1 מזהה אתר זרחון הסכמה המורכבת (Ser/חמישי-Pro-X-ליס/Arg), כאשר X הוא שאריות ו סרין או תראונין הוא האתר של זרחון. חשוב במיוחד עבור זיהוי היא הנוכחות של פרולין התנוחה +1. בנוסף, שאריות בסיסי המועדפות בעמדות +2 או +3, עם רוב אתרי זירחון ספציפי Cdk1 המכיל של ליס או Arg ב +3 מקם6,12.
הפעלה של Cdk1, והיא מובילה התחלתה של מיטוזה1,2,3,4,5. הפעילות של kinases תלויי-cyclin באופן כללי תלוי שיוכן cyclins נפרדות (cyclin A, B, C, D ו- E אצל בני אדם), אשר באים לידי ביטוי ב נדנוד רמות לאורך כל מחזור התא13. Cdk1 הביטוי הוא קבוע על פני מחזור התא וכן ברגולציה של פעילותה על השיוך שלו subunits תקינה cyclin A ו- cyclin B5,13,14,15, כמו גם שינויים post-translational. היווצרות הקומפלקס B Cdk1/cyclin נדרש קינאז הפעלה5,14,15,16,17,18. בשלב G2, הוא cyclin B המתורגמת את ציטוזול ולא לייבאו לתוך גרעין איפה זה נקשר Cdk15,14,15,16,17,18; עם זאת, Cdk1/cyclin B מתקיים לא מופעל על ידי זרחון על שאריות Thr14 ו- Tyr15 על ידי kinases Cdk1-מעכבות האנושי Myt1 (ממברנה-הקשורים טירוזין תראונין ספציפיים או cdc2-מעכבות קינאז) ו- Wee1, בהתאמה19, 20,21. בשלב G2 מאוחר, dephosphorylation Thr14, Tyr15 על ידי חלוקת התא מחזור 25 פוספטאז (cdc25) מפעילה את פעילות קינאז של המתחם B Cdk1/cyclin ותפעיל את החניכה של מיטוזה12,14, 18 , 20 , 22 , 23. זרחון של Thr161 גם נדרש עבור הפעלת B Cdk1/cyclin, מתווך על ידי Cdk7, קינאז (CAK) הפעלת Cdk18. השפלה של B cyclin "אנאפאזה" חלבונית Cdk1, המאפשר יציאה מן מיטוזה24,25. הפעלה של Cdk1/cyclin B לכן תהליך מסובך. פרוטוקול המובאת כאן מתבצע באמצעות B Cdk1/cyclin זמינים מסחרית. במהלך ביטוי רקומביננטי של מתחם זה בתאי חרקים, מופעל ויוו kinases אנדוגני14,20 , נותר פעיל במדינה מטוהרים. וכתוצאה מכך פעיל, רקומביננטי האנושי Cdk1/cyclin ב' מתאים במבחנה מבחני קינאז.
כאן, אנו מתארים את פרוטוקול לצורך זיהוי ספציפי Cdk1 אתרי זירחון ב- F (CENP-F) חלבון10צנטרומר אנושי. CENP-F הוא חלבון קינטוכור שוכנת בגרעין במשך רוב לאטמוספרה (G1 ו- S-פאזי), ייצוא של ציטוזול ב- G2 שלב26,27,28 ב באופן תלוי-Cdk110, 11. גרעיני לוקליזציה שהוענקו דו-צדדי cNLS26. cNLSs מזוהים על ידי α karyopherin פקטור נשק גרעיני, אשר מקלה, יחד עם karyopherin β RanGDP, הייבוא של cNLS-מטען לתוך גרעין29. ייצוא גרעיני בשלב G2 הוא הקל דרך מסלול10ייצוא לא ידוע. ברגע CENP-F שוכן ציטוזול, זה הוא גויס על המעטפה גרעיני והוא בתורו מגייס חלבון מוטוריים מורכבים דינאין30,31. מסלול זה חשוב למקם את הגרעין בהתאמה centrosome במהלך בשלבים הראשונים של פלך mitotic הרכבה בצורה דינאין תלויית, וזה חשוב על העיתוי הנכון של הערך mitotic ועל תהליך מהותי במוח פיתוח30,31,32. החל בשלב G2, CENP-F הוא גם לצרפן יחד קינטוכור בהם לו תפקידים חשובים עבור נאמן כרומוזום סגרגציה27,28,33,34,35 . צעד רגולטוריות. מפתח של המסלולים הללו הוא הגרעין הייצוא של CENP-F בשלב G2, אשר תלויה Cdk110,11. נתאר כאן פרוטוקול לצורך זיהוי אתרי זירחון ספציפי Cdk1 cNLS של CENP-F. Phosphomimetic מוטציות של אתרים אלה להאט גרעיניות הייבוא של CENP-F, רומז כי B Cdk1/cyclin ישירות מסדיר הסלולר לוקליזציה של CENP-F על ידי זירחון של שלו cNLS10.
בסך הכל, זה וזמינותו קינאז במבחנה מאפשר הזיהוי של סובסטרטים ספציפי עבור קינאז Cdk1. Purified היעד החלבונים הם phosphorylated במבחנה על-ידי Cdk1/cyclin B המתחם זמינים מסחרית של אתרי זירחון לאחר מכן מזוהים באמצעות ספקטרומטר מסה. הזיהוי של אתרי זירחון ספציפי Cdk1 תומך מחקרים מכניסטית לגלות איך Cdk1 שולט מחזור התא.
Protocol
1. חיזוי של אתרי זירחון ספציפי Cdk1 מהרצף חלבון
- לפני תחילת וזמינותו קינאז, לנתח את רצף החלבון עבור אתרי זירחון ספציפי Cdk1 החזוי, חיפוש הספרות עבור אתרי זירחון השפעול הוקמה עם ירידה לפרטים קינאז לא ידוע. השתמש הבאים כלים, מאגרי מידע, הפניות המסוכמים.
- השתמש iGPS 3.0 תוכנה36,37 (http://gps.biocuckoo.org/online_full.php) לחזות Cdk1 ספציפיים אתרי זירחון ברצף חלבון המטרה. השתמש בקישור כאן עבור חיזוי מקיף הכולל ביאורים של מבנה שניוני ונגישות משטח.
- התוכנה, הזן את רצף החלבון בתבנית FASTA. עבור קינאז ירידה לפרטים, בדוק תראונין/סרין קינאז, CMGC, CDK, CDC2, CDK1, ולהוריד את כל specificities אחרים. בחר בינונית לסף ולחץ על הלחצן Submit .
- להשוות בין האתרים החזוי שנוצר עם אתר זרחון קונצנזוס Cdk1 (Ser/חמישי-Pro-X-ליס/Arg).
- חשוב לבדוק את אתר החזוי עבור פרולין ליד השאריות phosphorylated (כמה מצעים Cdk1 הם phosphorylated6,אתר מינימלי (Ser/חמישי-Pro)12). בנוסף, בדוק שאריות בסיסי, המועדפות בעמדות +2 או +3, עם רוב אתרי זירחון ספציפי Cdk1 המכיל של ליס או Arg בעמדה +36,12.
- גם לבדוק האתר נגיש עבור זיהוי, כמו הנוכחות של אתר תמימות דעים לגבי אינה מספקת עבור זרחון Cdk1.
- לבדוק את הנגישות פני השטח ואת מבנה שניוני ביאור חיזוי בפלט iGPS, אשר קובע אם האתר הוא ניבא יהיה נגיש; טרמיני N ו- C של חלבונים הם במקרים רבים גמיש ונגיש עבור זירחון.
- אם מבנה רנטגן של החלבון יעד זמין, בדיקת נגישות של אתרי זירחון בשם באמצעות בדיקת המיקום שלהם במבנה.
- חיפוש הספרות עבור אתרי זירחון השפעול הוקמה עם ירידה לפרטים קינאז לא ידוע באמצעות מסד הנתונים UniProtKB/שוויצרי-פרוט38 (http://www.uniprot.org).
- הזן את השם של החלבון בתיבת החיפוש ולחץ על הלחצן ' חפש '. בחר את ערך הרצף הנכון. אתרי זירחון מוערת, המוזכרים בסעיף חומצת אמינו שינויים .
- חיפוש ללימודי פרוטאומיקס mitotic תמציות של התא האנושי קווים7,39,40, אשר הם שימושיים במיוחד בגלל Cdk1 הופכת לפעילה בשלב G2 של מחזור התא.
- לזהות דו-צדדי cNLS על ידי NLSmapper (אופציונלי).
הערה: חלבונים המעבורת בין ציטוזול את הגרעין, מנגנון ויסות לוקליזציה הסלולר נפוצה היא זירחון של cNLS דו-צדדי התנוחה-1 של מוטיב מרכזי על-ידי Cdk1. זירחון מבטל לוקליזציה גרעינית ב G2 שלב10,41,42,43.- חלבונים והצוות כזה, לזהות את cNLS ברצף החלבון על ידי NLSmapper43 (http://nls-mapper.iab.keio.ac.jp/cgi-bin/NLS_Mapper_form.cgi). להדביק את רצף חלבון כטקסט בתיבת רצף, בחר ציון ניתוק של 5.0 ו בוחרים לחפש NLS האזור כולו. לחץ על לחצן לחזות NLS .
- להשוות את cNLS החזוי וכתוצאה מכך נגד קונצנזוס על רצף cNLS דו-צדדי, אשר מורכב של המוטיב מינור (Lys Arg) מקשר (linker) לפחות 10 משקעים, מוטיב מרכזי (Lys-ליס/Arg-X-ליס/Arg), כאשר X הוא שאריות זה לא טעון שלילית .
- בדוק אם האתר החזוי זרחון Cdk1 (*) הינו ממוקם בסמוך המוטיב העיקרי במצב-1 של מקשר: Ser/Thr*-Pro/X-X-Lys-Lys/Arg-X-Lys/Arg.
הערה: זירחון של cNLS דו-צדדי התנוחה-1 מאת Cdk1 הוקם מנגנון רגולטורי הסלולר לוקליזציה עבור מספר והצוות חלבונים10,41,42, 43.
2. הבעה של חומרים ב- Escherichia Coli
- השתמש את הביטוי פלסמידים של צעדים 2.1.1-2.1.2 עבור הביטוי של חומרים ב e. coli.
- כדי ליצור את הביטוי האנושי CENP-F (שאריות 2,987-3,065) לבנות, ליצור מוסיף על ידי הגברה שברי DNA על ידי תגובת שרשרת פולימראזית (PCR) מתוך מבנה CENP-F באורך מלא (GenBank ההצטרפות: U19769.1). להשתמש את תחל הבאה: AGTCGGGGATCCCAGCAATCTAAACAAGATTCCCG ו- AGTCGGCTCGAGTCATTATTCTGCAGGGTGAATACCACTCATG.
הערה: פלסמיד CENP-F אנושי באורך מלא בנדיבות סופק על ידי ד ר אקס ז'ו, מכונים עבור מדעי הביולוגיה, האקדמיה הסינית למדעים, שנחאיי, סין.- לשכפל את תותב לתוך וקטור pGEX6p1 עם האתרים הגבלה BamHI, XhoI. השתמש פלסמיד זו כדי להביע את N-מסוף גלוטתיון S-טרנספראז (GST) חלבונים היתוך של CENP-F (שאריות 2,987-3,065).
הערה: GST-התג יכול להיות שהבקיע את מאת פרוטאז PreScission (המכונה מעתה ואילך פרוטאז PS).
- לשכפל את תותב לתוך וקטור pGEX6p1 עם האתרים הגבלה BamHI, XhoI. השתמש פלסמיד זו כדי להביע את N-מסוף גלוטתיון S-טרנספראז (GST) חלבונים היתוך של CENP-F (שאריות 2,987-3,065).
- עבור האדם לבנות ביטוי α karyopherin, ליצור מוסיף על ידי הגברה שברי DNA על ידי ה-PCR מתבנית cDNA α2 karyopherin באורך מלא (רצף הצטרפותן NM_002264.3; תחל: GCACTACATATGTCCACCAACGAGAATGCTAATA ו TCACGCCTCGAGTTATCAAAAGTTAAAGGTCCCAGGAGCC).
הערה: בשל הדמיון של איזופורמים, את המבנה α2 karyopherin הזה הוא המכונה בתוך הטקסט העוקב karyopherin α.- לשכפל את תותב לתוך וקטור pET28a-פרס עם האתרים הגבלה NdeI, XhoI.
הערה: זהו וקטור pET28a ששונה בהם רצף קידוד עבור האתר המחשוף תרומבין הוחלף על ידי רצף מקודד עבור אתר המחשוף פרוטאז PS. להשתמש פלסמיד זו כדי להביע חלבון פיוז'ן של α karyopherin עם N-מסוף שלו6-תג, איפה שלו6-תג יכול להיות שהבקיע את מאת PS פרוטאז.
- לשכפל את תותב לתוך וקטור pET28a-פרס עם האתרים הגבלה NdeI, XhoI.
- כדי ליצור את הביטוי האנושי CENP-F (שאריות 2,987-3,065) לבנות, ליצור מוסיף על ידי הגברה שברי DNA על ידי תגובת שרשרת פולימראזית (PCR) מתוך מבנה CENP-F באורך מלא (GenBank ההצטרפות: U19769.1). להשתמש את תחל הבאה: AGTCGGGGATCCCAGCAATCTAAACAAGATTCCCG ו- AGTCGGCTCGAGTCATTATTCTGCAGGGTGAATACCACTCATG.
- כדי ליצור מוטציות נקודה של חלבון המטרה, לבצע מוטגנזה עם ערכת.
- אקספרס של חומרים ב e. coli לטיהור עוקבות.
- להפוך את המניות הבאות: kanamycin 50 מ"ג/מ"ל (1:1, 000 פתרון מניות), כלורמפניקול 35 מ"ג/מ"ל ב 70% אתנול (1:1, 000), 100 מ"ג/מ"ל אמפיצילין (1:1, 000), ו- 1 מ' איזופרופיל β-D-1-thiogalactopyranoside (IPTG; 1:5, 000).
- סטרילי לסנן את המניות ולאחסן ב-20 ° C. הימנע מחזורים ההקפאה חוזרות ונשנות-הפשרה עבור IPTG.
- להפוך µL 1 של הביטוי פלסמיד µL 50 תאים המוסמכים של המתח pLysS e. coli רוזטה 2 (DE3), על פי הוראות היצרן.
- צלחת התרבות על אגר לוריא-Bertani (ליברות) עם כלורמפניקול (35 µg/mL ליברות), אמפיצילין (100 µg/mL ליברות) כדי להפוך את המבנה CENP-F, או כלורמפניקול kanamycin (תרבות µg/mL 50) עבור הבונה α karyopherin. דגירה הלוחות עבור 12-20 h ב- 37 מעלות צלזיוס.
- לבחור שמושבה בודדת, לחסן 50 מ של LB בתוספת אנטיביוטיקה (ראה שלב 2.3.4). דגירה את preculture תוך כדי טלטול במהירות של 160-180 סל ד עבור 12-20 h ב- 37 מעלות צלזיוס.
- להכנת 1 ליטר של LB במדיום mL 2,800 התפישה Fernbach את הבקבוק. להפוך את שתי המבחנות לכל preculture ולכל החיטוי אותם. כדי לאפשר אוורור, אל תמלא המבחנות שריצתה שני שליש הבקבוק באמצעי האחסון. Erlenmeyer המבחנות יכול לשמש גם כן.
- מוסיפים אנטיביוטיקה (שלב 2.3.4) 10 מיליליטר תמיסת גלוקוז 40% (w/v) סטרילי-מסוננים לכל 1 ליטר של מקורר LB. להוסיף 20 מ של preculture לכל 1 ליטר של מדיום ליברות.
הערה: ספיגת על 600 nm של 1:10 לדילול preculture צריך להיות בין 0.15-0.2. - דגירה המבחנות תוך טלטול במהירות של 160-180 סל ד ב 37 º C. למדוד את ספיגת על 600 nm של התרבות חיידקים באופן קבוע. אם ספיגת מגיע 0.5-0.6, זירוז ביטוי חלבונים על-ידי הוספת 0.2 מ מ IPTG (200 µL של 1 מ' מניות לכל 1 ליטר של מדיום ליברות).
הערה: חשוב כי התאים הם המושרה ב ספיגת הנכון. בדרך כלל זה לוקח 3-4 שעות כדי להגיע בשלב זה. - דגירה המבחנות תוך טלטול עבור h 3-37 מעלות צלזיוס. לקצור את התאים על ידי צנטריפוגה (15 דקות, 4,100 x g, 4 ° C, והזזה רוטור). למחוק את תגובת שיקוע. Resuspend כדורי תא 20 מ ל GST-איגוד מאגר (CENP-F) או שלו6-איגוד מאגר (karyopherin α) לכל 1 ליטר של תרבות. לאחסן את התאים ב- 80 ° c
- הכנת המאגר מחייב GST (10 מ מ טריס pH 8.0 25 ° c, 150 מ מ NaCl, 1 מ dithiothreitol (DTT), 0.5 מ מ EDTA) שלו6-איגוד מאגר (10 מ מ טריס pH 8.0 25 ° c, 250 מ מ NaCl, 5 מ מ Imidazole pH 8.0, 5 מ מ β-mercaptoethanol (BME)) מראש.
3. טיהור של חלבון רקומביננטי מאת גלוטתיון-כרומטוגרפיית זיקה ולסינון ג'ל
- להפוך את המניות הבאות: 1 מ' DTT (1:1, 000 מניות, סטרילי מסונן עם גודל הנקבוביות מיקרומטר 0.2, degassed); 250 מ מ phenylmethylsulfonyl פלואוריד (PMSF; 1:1,000 מניות ב- dimethylsulfoxide). חנות ב-20 ° C. הימנע מחזורים ההקפאה חוזרות ונשנות-הפשרה.
- להפוך את המאגרים הבאים וניתן עד 4 ° C.
- להכין מאגר GST מחייב שימוש 10 מ מ טריס pH 8.0 25 ° c, 150 מ מ NaCl, 1 מ"מ DTT, 0.5 מ מ EDTA.
- להכין מאגר GST-• תנאי שימוש 10 מ מ מופחת גלוטתיון (0.15 גרם של 50 מ"ל) מומס מאגר של 50 מ מ טריס pH 8.0 25 ° c, 150 מ מ NaCl, 1 מ"מ DTT. לאשר ה-pH של המאגר הסופי הוא 8.0 ולהשתמש באותו יום שהיה עשוי.
- להכין מאגר ג'ל-סינון באמצעות 20 מ מ טריס pH 7.5 ב- 25 ° C, 60 מ מ NaCl, 2 מ מ DTT. סטרילי לסנן את המאגר עם מסנן בקבוק-top (0.2 µM גודל הנקבוביות). דגה המאגר תוך ערבוב תחת ואקום (-6 psi) למשך 15 דקות.
- להוסיף DTT לכל מעל מאגרי ביום של שימוש.
- להפשיר את התאים של סעיף 2 המכילים הבונה CENP-F. לכוון את עוצמת הקול עד 40 מ"ל עם המאגר GST מחייב. להוסיף 1 מ"מ DTT, 0.25 מ"מ PMSF, 0.5 מ מ EDTA (פתרון מניות 0.5 M, pH 8.0), 7 מ"ג benzamidine הידרוכלוריד (1 מ מ), ו- 40 מ"ג D/L מתיונין.
הערה: כדי לצמצם את השפלה הפרוטאוליטי, לבצע כל השלבים ב 4 ° C, רצוי בחדר קר, אלא אם נכתב אחרת. לשמור תאים lysates, חלבון מטוהרים שברים בקרח כל הזמן והוספת מעכבי פרוטאז. כדי למנוע חמצון של שאריות ציסטאין ומתיונין, להוסיף reductants כגון DTT ומתיונין ביום של הניסוי. כדי לצמצם את השפלה הפרוטאוליטי, לעבוד דרך השלבים במהירות. - Lyse התאים sonicator כמו עוקב אחר. מילוי גביע זכוכית עם lysate, לטבול אותו באמבט מי קרח. Sonicate התרבות (s 1 דקות 40, ב פלט W (50%) 100/משרעת, עם פולסים s 10 ו 10 מחזורים מנוחה s). להוסיף 250 מיקרומטר PMSF לאחר sonication. לבדוק את התא lysate, אשר צריך להיראות שקוף יותר, כבר לא יהיה צמיגה.
- ברור lysate על ידי צריך שתוציאו g x 12,000-40,000, 25 דקות, 4 מעלות צלזיוס. השתמש צנטריפוגה למטה לסיבוב צינורות, רוטור זווית קבועה.
- לבצע equilibration על-ידי הוספת 2 מ"ל של גלוטתיון agarose 1:1 slurry עמודה כרומטוגרפיה חד פעמיות. העמודה הנוצרת יהיה בעל נפח של 1 מ"ל. לשטוף את העמודה עם 25 מ של מים הנדסה גנטית, 25 מ ל GST-איגוד המאגר.
- לבצע איגוד כדלקמן. לעבוד בחדר קר, או בתיבת קר, decant את lysate של כדורי. לסנן את lysate (0.2 µm גודל הנקבוביות), יוצקים אותו לתוך העמודה פקוק. קאפ העמודה, דגירה זה תוך בעדינות nutating למשך 30 דקות ב 4 º C.
- כדי לשטוף את העמודה, שים את זה על מתלה, תן שרף ליישב, ולאסוף את הזרימה. לשטוף את העמודה פעמיים עם 25 מ ל GST-איגוד המאגר.
-
Elute על-ידי פצילות הפרוטאוליטי.
- חבר את העמודה. הוסף µL 400 מאגר מחייב GST µL 250 של PS פרוטאז (2 מ"ג/מ"ל המניה עם פעילות של 1,000 U/מ ג, או מטוהרים במעבדה או לרכוש; ראה טבלה של חומרים).
- קאפ העמודה ו מערבולת בעדינות כדי resuspend השרף במאגר. דגירה את העמודות עבור 16-20 h-4 מעלות צלזיוס. לאחר מכן להוסיף 4 מ ל GST-איגוד המאגר כדי elute השבר CENP-F proteolytically cleaved מן העמודה.
-
• תנאי גלוטתיון
- לחבר את העמודה, להוסיף 4 מ"ל • GST-תנאי מאגר (עמודה 4 כרכים), תקופת דגירה של 10 דקות elute בתג-GST מאוגד. לאסוף את eluate. צור מחדש את העמודות לפני שימוש חוזר כפי שתואר על ידי היצרן.
- לנתח השבר • תנאי פרוטאז PS לבין השבר • תנאי גלוטתיון מאת מרחביות-דף על 16% אקרילאמיד ג'לים. לבצע אלקטרופורזה ב V 25/ס במשך 45 דקות כתם של ג'לים מאת Coomassie כחול. השבר פרוטאז PS יכיל השבר CENP-F מטוהרים, ואילו השבר גלוטתיון יכיל cleaved GST-התג. בדוק את הג'ל כדי להעריך את יעילות המחשוף הפרוטאוליטי.
- לרכז PS eluate פרוטאז עד 0.5 מ"ל באמצעות יחידות צנטריפוגליות מסנן עם משקל מולקולרי 3 kDa הקיצוץ (ראה טבלה של חומרים). הוסף את eluate בתוך התא העליון של המסנן, centrifuge זה במרווחים של 10-15 דקות ב g x 4100, 4 ° C (והזזה רוטור) לרכז את הדגימה. מערבבים על-ידי pipetting לאחר כל תוספת.
- להתחבר מתאים ג'ל עמודה סינון (ראה טבלה של חומרים עבור רכישת מידע) למערכת כרומטוגרפיה נוזלית (FPLC) חלבון מהיר, ו- equilibrate זה עם עמודה 1 נפח המאגר סינון ג'ל.
- Centrifuge השבר CENP-F מרוכזים ב microcentrifuge (20 דקות, 21,700 x g, 4 ° C). הכנס הדגימה על העמודה ואת elute זה עם עמודה 1 נפח המאגר סינון ג'ל. לאסוף את השברים 0.6 מ"ל.
הערה: המאגר סינון ג'ל ממוטבת עבור תאימות עם וזמינותו קינאז עוקבות. בדוק אם חלבון המטרה יציב במאגר הזה. אם זה לא יציב, נסה להוסיף יותר נתרן כלורי. - לנתח את השברים שיא מאת מרחביות-דף (שלב 3.11). בריכת שברים שיא המכילים שברים CENP-F טהור. . להתרכז השברים CENP-F מטוהרים כדי 3.3 מ"ג/מ"ל (שלב 3.12). לנתח את השברים CENP-F מטוהרים מאת מרחביות-דף (שלב 3.11).
-
קביעת ריכוז חלבון
- לדלל את בטחונות מדגם במים הנדסה גנטית ולמקם אותו מיקרו-cuvette קוורץ. ספקטרה הרשומה במגוון באורך גל של nm 220-300 בספקטרופוטומטר.
הערה: ריכוז חלבון c (mg/mL) נגזר מן המשוואה הבאה: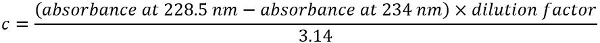
- לדלל את בטחונות מדגם במים הנדסה גנטית ולמקם אותו מיקרו-cuvette קוורץ. ספקטרה הרשומה במגוון באורך גל של nm 220-300 בספקטרופוטומטר.
- להפוך את aliquots 50 µL של החלבון מטוהרים microtubes 0.5 mL, הקפאה אותם תוך חנקן נוזלי. לאחסן את aliquots ב-80 מעלות צלזיוס.
4. טיהור של חלבון רקומביננטי על ידי Ni-נ כרומטוגרפיית זיקה
- המאגרים הבאים ולבצע להצטנן עד 4 ° C.
- להכין שלו6-איגוד מאגר באמצעות 10 מ מ טריס pH 8.0 25 ° c, 250 מ מ NaCl, 5 מ מ Imidazole pH 8.0, 5 מ מ BME. להוסיף BME מאגרי כל יום שימוש.
- להכין שלו6-מאגר לשטוף באותו אופן כמו שלו6-איגוד מאגר אבל באמצעות 20 מ מ Imidazole.
- להכין שלו6-מאגר • תנאי באותו אופן כמו המאגר מחייב אבל באמצעות 150 מ מ Imidazole.
-
להפשיר את התאים של סעיף 2 שמכילים α karyopherin את לבנות ולכוונן את העוצמה עד 40 מ"ל עם שלו6 -איגוד מאגר. הוסף 5 מ מ BME, 0.250 מ מ PMSF, 7 מ"ג benzamidine הידרוכלוריד (1 מ מ) ו- 40 מ"ג D/L מתיונין.
- המשך כמתואר בצעדים 3.4-3.8 עם הווריאציות הבאים: שימוש שלו6-מאגר מחייב במקום GST-איגוד המאגר עבור כל השלבים. במקום agarose גלוטתיון, להשתמש 4 מיליליטר בזיקה ניקל ג'ל (ראה טבלה של חומרים) כדי להפוך את העמודה, אשר יהיה נפח טורים של 2 מ ל.
הערה: ניקל זיקה עמודות שאינם תואמים ל- DTT ו- EDTA. במקום הג'ל זיקה ניקל, ני2 +- nitrilotriacetic חומצה (נ) agarose יכול לשמש, אם ריכוז imidazole במאגר • תנאי עלה ל 250 מ מ (ראה טבלה של חומרים).
- המשך כמתואר בצעדים 3.4-3.8 עם הווריאציות הבאים: שימוש שלו6-מאגר מחייב במקום GST-איגוד המאגר עבור כל השלבים. במקום agarose גלוטתיון, להשתמש 4 מיליליטר בזיקה ניקל ג'ל (ראה טבלה של חומרים) כדי להפוך את העמודה, אשר יהיה נפח טורים של 2 מ ל.
- לשטוף עמודות עם 8 מ ל שלו6-מאגר לשטוף.
- Elute α karyopherin עם 12 מ ל שלו6-• תנאי מאגר ולנתח eluate מאת מרחביות-דף (שלב 3.11).
- להוסיף 250 µL של PS פרוטאז (שלב 3.9) כדי eluate דגירה עבור 16-20 h-4 מעלות צלזיוס. בינתיים, dialyze את eluate פעמיים נגד 1 ליטר של שלו6-איגוד המאגר עבור h 2-20, באמצעות קרום דיאליזה עם kDa 6-8 מולקולרית משקל הקיצוץ.
- צור את העמודה ג'ל זיקה ניקל כפי שתואר על ידי היצרן. Equilibrate את העמודה עם 25 מיליליטר שלו6-מאגר מחייב. להוסיף את החלבון dialyzed לעמודה ולבצע את הצעד מחייב כפי שמתואר בשלב 3.7.
- לאסוף את הזרימה, אשר מכיל את α karyopherin מטוהרים (עם שלו6-תג שהבקיע את), ו elute החלבון הנותרים עם אוטם 4 נוספים של שלו6-מאגר מחייב. בריכת השברים האלה. ואז elute את העמודות 12 מ של שלו6-מאגר • תנאי. שבר זה מכיל זיהומים.
- לנתח את α karyopherin uncleaved ו cleaved מצעדי 4.4-4.5 ושברים • תנאי שני מהשלב הזה מאת מרחביות-דף (שלב 3.11) כדי להעריך את היעילות של שלו6-תג המחשוף וטוהר.
- לרכז את α karyopherin 8 מ"ג/מ"ל, הקפאה של חנקן נוזלי (שלבים 3.15-3.17).
5. במבחנה קינאז Assay עם Cdk1/Cyclin B
-
במבחנה assay קינאז
הערה: עם ההגעה של קינאז, להפוך aliquots קטן, הקפאה אותם בחנקן נוזלי ואחסן אותם ב-80 מעלות צלזיוס. השתמש בתוך 6 חודשים. בעוד משקל מולקולרי הוא 34 kDa עבור Cdk1 ו- 48 kDa עבור cyclin B, המשקל המולקולרי לכאורה של B cyclin בדף-מרחביות הוא כ-60 kDa.- הכינו 10 x באמצעות מאגר (המצורף של קינאז) PK 0.5 מ' טריס-HCl, MgCl 0.1 M2, 1 מ"מ EDTA, 20 מ מ. DTT, 0.1% 35 מוטי ממליה, pH 7.5 ב 25 º C.
- להכין את 10 x ATP מניות: לרכוש פתרון של 4 מ מ ATP 1 x PK מאגר, וכך לבדוק את רמת ה-pH הסופי של המניה. אחסן aliquots קטן ב-20 ° C ולהימנע מחזורים ההקפאה-הפשרה.
- מיקס 40 µL של CENP-F קטע (על ריכוז של 3.3 מ"ג/מ"ל, או 400 מיקרומטר), µL 6 10 x PK מאגר 3 µL הנדסה גנטית, 6 µL ATP 10 x מניות ומים אנושית 5 µL B Cdk1/cyclin (µg 1, 100 U) ב- microtube 0.5 מ ל.
הערה: השבר CENP-F יש מסה טוחנת של 8.6 kDa ו 4 אתרי זירחון ספציפית Cdk1. מצע חדש, להתאים את ריכוז קינאז וזמינותו על פי ריכוז מולרי של החלבון ועל פי מספר אתרי זירחון. הפעילות של המניה B Cdk1/cyclin רקומביננטי האנושית היא 20,000 U/mL, ו הפעילות הספציפי היא 1,000,000 U / מ ג. 1 U מוגדרת בתור הסכום של B Cdk1/cyclin הנדרש כדי לעודד את העברת pmol 1 של פוספט Cdk1 פפטיד המצע PKTPKKAKKL-NH2 (50 מיקרומטר) ב- 1 דקות ב- 30 ° C (ראה טבלה של חומרים). - להכין תגובה שליטה ללא Cdk1/cyclin B. Incubate את התגובות באמבט מים 1-16 h ב- 30 ° C.
- לנתח µL 2.5 התגובה assay קינאז µL 2.5 של הפקד על-ידי מרחביות-דף (שלב 3.11), באמצעות ג'ל עם 16% אקרילאמיד. כדי להגדיל את הרזולוציה, להאריך את זמן הפעולה.
- הוסף אמצעי שווה של 6 מ' guanidine הידרוכלוריד פתרון כדי התגובה assay קינאז הנותרים (וכדי הפקד). ריכוז הידרוכלוריד guanidine הסופי יהיה מ 3 לנתח דגימות אלה באמצעות ספקטרומטר מסה (סעיף 6) כדי לזהות את אתרי זירחון או לשלוח את הדגימות למתקן ספקטרומטר מסה לניתוח, ולאחר מכן לבצע פונקציה אימות ( סעיף 7).
הערה: לניתוח ספקטרומטר מסה עוקבות, חשוב מאוד כי היעילות זירחון של האתרים הבודדים גבוה ככל האפשר, רצוי 100%, אחרת אין אפשרות למפות את אתרי זירחון. כדי לרומם זרחון היעילות, להוסיף כמויות גדולות יותר של B Cdk1/cyclin ולהגדיל את זמן הדגירה ש 16. גם יכול להיות מוכפל הריכוז של ATP. - לפתרון בעיות, ביצוע של assay קינאז במבחנה של CENP-F (שאריות 2,987-3,065) כפקד חיובי. השתמש אצוות טריים של B Cdk1/cyclin עם פעילות גבוהה.
6. זיהוי של אתרי זירחון Cdk1 ספציפי באמצעות ספקטרומטר מסה
- Denature, desalt את דגימות חלבון, לבצע microbore שלב הפוכה כרומטוגרפיה נוזלית עם עמודה של PLRP300.
- כדי לקבל את מספר אתרי זירחון השבר CENP-F, לנתח את שלמה במבחנה phosphorylated CENP-F 84-מר ידי ספקטרומטריית electrospray יינון מלכודת יונים ספקטרומטר מסה (ESI-ITMS)44,45.
- כדי להעריך את מיקום האתר חומצה phosphoamino CENP-F, להכין את מעכל טריפסין של ה במבחנה phosphorylated דגימות חלבון CENP-F. Desalt את מעכל טריפסין עם desalting pipet טיפים.
- לנתח את מעכל tryptic של CENP-F על ידי יינון ספקטרומטריית electrospray פורייה להפוך יון ציקלוטרון תהודה ספקטרומטר מסה (MS ESI-FTICR). בצע ניתוח ספקטרומטר מסה ESI-FTICR עם מועד הצטברות של 169 µs.
הערה: הדיוק של ההמונים נקבע על ידי MS ESI-FTICR נמצא במרחק אמו 0.005. כדי לכמת את היחס של כל מין phosphorylated בהתאמה לכמות החלבון הכולל, לקבוע הגולן שיא הספקטרום המוני.
7. פונקציונלי אימות: בחינת השפעת Phosphomimetic מוטציות על אינטראקציות חלבון-חלבון לפי גודל אנליטי אי-הכללה של כרומטוגרפיה
הערה: לצורך אימות פונקציונלי אתרי זירחון ספציפי Cdk1 מזוהה, phosphomimetic מוטציות של CENP-F קטעים שנוצרו על-ידי החלפת את אתרי זירחון מזוהה aspartates. המטען השלילי של אספרטט מחקה את ההשפעות של זרחון. מוטציה S3048D של השבר CENP-F (שאריות 2,987-3,065) נוצר.
- לצורך אימות פונקציונלי, להשוות את הכריכה של פראי-סוג ולהשתמש קטעים phosphomimetic CENP-F karyopherin α. סינון ג'ל אנליטית של איגוד וזמינותו. סינון ג'ל אנליטית, לטהר שברי CENP-F על ידי גלוטתיון כרומטוגרפיית זיקה (3.1 שלבים-3.12), לדלג על השלב הסינון ג'ל.
- ריכוז החלבון ל 5-6 מ"ג/מ"ל הקפאה aliquots חלבון בחנקן נוזלי (שלבים 3.15-3.17).
- מיקס CENP-F מטוהרים שברי (0.1 מ ג, או פראי-סוג או S3048D variant) לטהר karyopherin α (0.7 מ ג) ביחס 1:1 שן טוחנת. להתאים את אמצעי האחסון מדגם µL 200 עם ג'ל סינון מאגר. דגירה המדגם במשך 30 דקות על קרח, לסנן מדגם (0.2 µm גודל הנקבוביות) ונקה את המדגם על ידי צנטריפוגה (25 דקות, 21,700 x g, 4 ° C)
- הפרד את הדגימה על ידי גודל אי-הכללה של כרומטוגרפיה (שלבים 3.13-3.14). השתמש מאגר סינון ג'ל בהיקף של 20 מ מ HEPES, pH 7.5, 150 מ מ NaCl, ו- 2 מ מ DTT. לבצע את הניסויים סינון ג'ל אנליטי בתנאים זהים עבור כל הדגימות.
- כיילו את העמודה סינון ג'ל אנליטי בתקנים משקל מולקולרי של המסה טוחנת ידוע כפי שתואר על ידי היצרן.
- לנתח את השברים • תנאי 16% מרחביות-דף (שלב 3.11).
תוצאות
השתמשנו לאחרונה במבחנה קינאז assay (איור 1) כדי לזהות אתרי זירחון ספציפי Cdk1 קטע CENP-F שהכיל cNLS10. האות הזה מקנה גרעיני לוקליזציה של CENP-F במשך רוב לאטמוספרה. בשלב G2, CENP-F מיוצא מהגרעין ציטוזול באופן תלוי-Cdk1. כדי לקבל תובנות מכניסטית על איך Cdk1 מווסת ...
Discussion
שלנו assay קינאז במבחנה היא שיטה רבת עוצמה כדי לזהות מטרות מולקולריות עבור קינאז Cdk1, אשר הוא בקר מאסטר של מחזור התא, מווסת תהליכים תאיים חשובים רבים. השיטה קובעת אם חלבון מטוהרים מצע עבור Cdk1, מאפשר זיהוי של אתרי זירחון ספציפיים. זה מקל על מכניסטית ללימודי ויסות תהליכים תאיים על ידי זרחון ?...
Disclosures
המחברים אין לחשוף.
Acknowledgements
אנו מודים ד ר דוד המלך, הווארד יוז במכון הרפואי, האוניברסיטה של קליפורניה בברקלי ספקטרומטר מסה ניתוח והערות מועילות. אנו מודים ז'ו Xuelian ד ר, שנגחאי, מכוני המחקר למדעי הביולוגיה, האקדמיה הסינית למדעים, שנחאיי, סין למתן מבנה CENP-F באורך מלא. לבסוף, אנו מודים ד ר סוזאן ביין, ד ר בריאן קאלהן, ד ר Grewer כריסטוף באוניברסיטת Binghamton על גישה לציוד. מחקר זה מומן על ידי קרן מחקר באוניברסיטת מדינת ניו יורק, במחלקה לכימיה, אוניברסיטת המדינה של ניו יורק ב- Binghamton.
Materials
| Name | Company | Catalog Number | Comments |
| 2800 ml baffled Fernbach flask | Corning | 44232XL | |
| ampicillin | Gold Biotechnology | A-301-25 | |
| ATP | Fisher Scientfiic | BP413-25 | |
| benzamidine hydrochloride | Millipore Sigma | B6506-25 | |
| bottletop filter | Corning | 431161 | |
| Cdk1/cyclin B recombinant, human 20,000 U/mL | New England Biolabs | P6020 | |
| Cdk1/cyclin B (alternate source) | EMD Millipore | 14-450 | |
| Cdk1/cyclin B (alternate source) | Invitrogen | PV3292 | |
| Cdk1/cyclin B + 10x PK buffer | New England Biolabs | P6020 | |
| CENP-F (residues 2987 – 3065) pGEX6P1 plasmid | Available upon request. | ||
| centrifuge: Heraeus Multifuge X3R, cooled, with TX-1000 swing-out rotor | Thermo Scientific | 10033-778 | |
| centrifugal filter units: Amicon Ultra-15 centrifugal filter units, 3 kDa cutoff, Ultracel-PL membranes | EMD Millipore | UFC900324 | |
| chlorampenicol | Gold Biotechnology | C-105-100 | |
| D/L methionine | Agros Organics / Fisher | 125650010 | |
| desalting pipet tips: Zip tips | Millipore Sigma | ZTC18S008 | |
| disposable chromatography columns, Econo-Pac 1.5 x 12 cm | Biorad | 7321010 | |
| dithiothreitol | Gold Biotechnology | DTT50 | |
| E. coli Rosetta 2(DE3)pLysS strain | EMD Millipore | 71403 | |
| electrospray ionization Fourier transform ion | Bruker Amazon | Apex III | |
| cyclotron resonance mass spectrometer | |||
| electrospray ionization ion trap mass spectrometer | Bruker Amazon | custom | |
| fixed angle rotor: Fiberlite F15-8x-50cy | Thermo Scientific | 97040-276 | |
| FPLC system: Äkta Pure FPLC | GE Healthcare | 29032697 | |
| Gel filtration column: Superdex 200 Increase 10/300 GL | GE Healthcare | 28990944 | |
| glutathione agarose | Pierce | 16101 | |
| glutathione, reduced | Millipore Sigma | G4251-50g | |
| incubation shaker: multitron shaker | Infors | I10102 | |
| isopropyl β-D-1-thiogalactopyranoside | Gold Biotechnology | I2481C50 | |
| kanamycin | Gold Biotechnology | K-120-25 | |
| karyopherin α pet-28a pres plasmid | Available upon request. | ||
| Luria Bertani medium | Fisher Scientfiic | BP1426-2 | |
| microcentrifuge 5418R, refrigerated | Eppendorf | 5401000013 | |
| microtubes (0.5 ml) | Eppendorf | 30121023 | |
| microtubes (1.5 ml) | Eppendorf | 30120086 | |
| Nickel affinity gel: His-Select Nickel affinity gel | Millipore Sigma | P6611-100ml | |
| pGEX-6P-1 plasmid | Millipore Sigma | GE28-9546-48 | |
| phenylmethylsulfonyl fluoride | Gold Biotechnology | P470-10 | |
| PS protease: PreScission protease | GE Healthcare | 27084301 | |
| Phos-tag acrylamide | Wako Pure Chem. Ind. | 304-93521 | |
| reduced gluthathione | Millipore Sigma | G4251-50g | |
| roundbottom centrifuge tubes (Oakridge tubes) | Fisher Scientfiic | 055291D | |
| site-directed mutagenesis kit: QuikChange Lightning | Agilent | 210518 | |
| Site-Directed Mutagenesis Kit | |||
| sonifier: Branson S-250D sonifier | Branson | 15 338 125 | |
| Spectra/Por 1RC dialysis membrane (6-8 kDa cutoff) | Spectrum Labs | 08 670B | |
| swing out rotor TX-1000 | Thermo Scientific | 10033-778 |
References
- Nurse, P. Cyclin dependent kinases and cell cycle control (Nobel Lecture). ChemBioChem. 3 (7), 596-603 (2002).
- Lee, M. G., Nurse, P. Complementation used to clone a human homologue of the fission yeast cell cycle control gene cdc2. Nature. 327, 31 (1987).
- Lohka, M. J., Hayes, M. K., Maller, J. L. Purification of maturation-promoting factor, an intracellular regulator of early mitotic events. Proc Natl Acad of Sci U S A. 85 (9), 3009-3013 (1988).
- Gautier, J., Norbury, C., Lohka, M., Nurse, P., Maller, J. Purified maturation-promoting factor contains the product of a Xenopus homolog of the fission yeast cell cycle control gene. Cell. 54 (3), 433-439 (1988).
- Gautier, J., Minshull, J., Lohka, M., Glotzer, M., Hunt, T., Maller, J. L. Cyclin is a component of maturation-promoting factor from Xenopus. Cell. 60 (3), 487-494 (1990).
- Ubersax, J. A., Woodbury, E. L., Quang, P. N., Paraz, M., Blethrow, J. D., Shah, K., Shokat, K. M., Morgan, D. O. Targets of the cyclin-dependent kinase Cdk1. Nature. 425 (6960), 859-864 (2003).
- Petrone, A., Adamo, M. E., Cheng, C., Kettenbach, A. N. Identification of Candidate Cyclin-dependent kinase 1 (Cdk1) Substrates in Mitosis by Quantitative Phosphoproteomics. Mol Cell Proteomics. 15 (7), 2448-2461 (2016).
- Vassilev, L. T., Tovar, C., Chen, S., Knezevic, D., Zhao, X., Sun, H., Heimbrook, D. C., Chen, L. Selective small-molecule inhibitor reveals critical mitotic functions of human CDK1. Proc Natl Acad of Sci U S A. 103 (28), 10660-10665 (2006).
- Balakrishnan, A., Vyas, A., Deshpande, K., Vyas, D. Pharmacological cyclin dependent kinase inhibitors: Implications for colorectal cancer. World J Gastroenterol. 22 (7), 2159-2164 (2016).
- Loftus, K. M., Coutavas, E., Cui, H., King, D., Ceravolo, A., Pereiras, D., Solmaz, S. Mechanism for G2 phase-specific nuclear export of the kinetochore protein CENP-F. Cell Cycle. 16 (15), 1414-1429 (2017).
- Baffet, A. D., Hu, D. J., Vallee, R. B. Cdk1 activates pre-mitotic nuclear envelope dynein recruitment and apical nuclear migration in neural stem cells. Dev Cell. 33 (6), 703-716 (2015).
- Songyang, Z., Blechner, S., Hoagland, N., Hoekstra, M. F., Piwnica-Worms, H., Cantley, L. C. Use of an oriented peptide library to determine the optimal substrates of protein kinases. Curr Biol. 4 (11), 973-982 (1994).
- Malumbres, M. Cyclin-dependent kinases. Genome Biol. 15 (6), 122 (2014).
- Peeper, D. S., Parker, L. L., Ewen, M. E., Toebes, M., Hall, F. L., Xu, M., Zantema, A., van der Eb, A. J., Piwnica-Worms, H. A- and B-type cyclins differentially modulate substrate specificity of cyclin-cdk complexes. EMBO J. 12 (5), 1947-1954 (1993).
- Trembley, J., Ebbert, J., Kren, B., Steer, C. Differential regulation of cyclin B1 RNA and protein expression during hepatocyte growth in vivo. Cell Growth Differ. 7 (7), 903-916 (1996).
- Pines, J., Hunter, T. The differential localization of human cyclins A and B is due to a cytoplasmic retention signal in cyclin B. EMBO J. 13 (16), 3772-3781 (1994).
- Morgan, D. O. Principles of CDK regulation. Nature. 374, 131 (1995).
- Larochelle, S., Merrick, K. A., Terret, M. -. E., Wohlbold, L., Barboza, N. M., Zhang, C., Shokat, K. M., Jallepalli, P. V., Fisher, R. P. Requirements for Cdk7 in the assembly of Cdk1/cyclin B and activation of Cdk2 revealed by chemical genetics in human cells. Mol cell. 25 (6), 839-850 (2007).
- Parker, L. L., Sylvestre, P. J., Byrnes, M. J., Liu, F., Piwnica-Worms, H. Identification of a 95-kDa WEE1-like tyrosine kinase in HeLa cells. Proc Natl Acad of Sci U S A. 92 (21), 9638-9642 (1995).
- Atherton-Fessler, S., Parker, L. L., Geahlen, R. L., Piwnica-Worms, H. Mechanisms of p34cdc2 regulation. Mol Cell Biol. 13 (3), 1675-1685 (1993).
- Liu, F., Stanton, J. J., Wu, Z., Piwnica-Worms, H. The human Myt1 kinase preferentially phosphorylates Cdc2 on threonine 14 and localizes to the endoplasmic reticulum and Golgi complex. Mol Cell Biol. 17 (2), 571-583 (1997).
- McGowan, C. H., Russell, P. Human Wee1 kinase inhibits cell division by phosphorylating p34cdc2 exclusively on Tyr15. EMBO J. 12 (1), 75-85 (1993).
- Strausfeld, U., Labbé, J. C., Fesquet, D., Cavadore, J. C., Picard, A., Sadhu, K., Russell, P., Dorée, M. Dephosphorylation and activation of a p34cdc2/cyclin B complex in vitro by human CDC25 protein. Nature. 351, 242 (1991).
- Leuken, R., Clijsters, L., Wolthuis, R. To cell cycle, swing the APC/C. Biochim Biophys Acta. 1786 (1), 49-59 (2008).
- Acquaviva, C., Pines, J. The anaphase-promoting complex/cyclosome: APC/C. J Cell Sci. 119 (12), 2401-2404 (2006).
- Zhu, X., Chang, K. -. H., He, D., Mancini, M. A., Brinkley, W. R., Lee, W. -. H. The C-terminus of mitosin is essential for its nuclear localization, centromere/kinetochore targeting, and dimerization. J Biol Chem. 270 (33), 19545-19550 (1995).
- Liao, H., Winkfein, R. J., Mack, G., Rattner, J. B., Yen, T. J. CENP-F is a protein of the nuclear matrix that assembles onto kinetochores at late G2 and is rapidly degraded after mitosis. J Cell Biol. 130 (3), 507-518 (1995).
- Rattner, J. B., Rao, A., Fritzler, M. J., Valencia, D. W., Yen, T. J. CENP-F is a ca 400 kDa kinetochore protein that exhibits a cell-cycle dependent localization. Cell Motil Cytoskeleton. 26 (3), 214-226 (1993).
- Christie, M., Chang, C. -. W., Rona, G., Smith, K. M., Stewart, A. G., Takeda, A. A. S., Fontes, M. R. M., Stewart, M., Vertessy, B. G., Forwood, J. K., Kobe, B. Structural biology and regulation of protein import into the nucleus. J Mol Biol. 428 (10A), 2060-2090 (2016).
- Zuccolo, M., Alves, A., Galy, V., Bolhy, S., Formstecher, E., Racine, V., Sibarita, J. B., Fukagawa, T., Shiekhattar, R., Yen, T., Doye, V. The human Nup107/160 nuclear pore subcomplex contributes to proper kinetochore functions. EMBO J. 26, 1853-1864 (2007).
- Bolhy, S., Bouhlel, I., Dultz, E., Nayak, T., Zuccolo, M., Gatti, X., Vallee, R., Ellenberg, J., Doye, V. A Nup133-dependent NPC-anchored network tethers centrosomes to the nuclear envelope in prophase. J Cell Biol. 192 (5), 855-871 (2011).
- Hu, D. J., Baffet, A. D., Nayak, T., Akhmanova, A., Doye, V., Vallee, R. B. Dynein recruitment to nuclear pores activates apical nuclear migration and mitotic entry in brain progenitor cells. Cell. 154 (6), 1300-1313 (2013).
- Vergnolle, M. S., Taylor, S. S. Cenp-F links kinetochores to Ndel1/Nde1/Lis1/Dynein microtubule motor complexes. Curr Biol. 17 (13), 1173-1179 (2007).
- Yang, Z. Y., Guo, J., Li, N., Qian, M., Wang, S. N., Zhu, X. L. Mitosin/CENP-F is a conserved kinetochore protein subjected to cytoplasmic dynein-mediated poleward transport. Cell Res. 13 (4), 275-283 (2003).
- Yang, Z., Guo, J., Chen, Q., Ding, C., Du, J., Zhu, X. Silencing mitosin induces misaligned chromosomes, premature chromosome decondensation before anaphase onset, and mitotic cell death. Mol Cell Biol. 25 (10), 4062-4074 (2005).
- Xue, Y., Ren, J., Gao, X., Jin, C., Wen, L., Yao, X. GPS 2.0, a tool to predict kinase-specific phosphorylation sites in hierarchy. Mol Cell Proteomics. 7 (9), 1598-1608 (2008).
- Song, C., Ye, M., Liu, Z., Cheng, H., Jiang, X., Han, G., Songyang, Z., Tan, Y., Wang, H., Ren, J., Xue, Y., Zou, H. Systematic analysis of protein phosphorylation networks from phosphoproteomic data. Mol Cell Proteomics. 11 (10), 1070-1083 (2012).
- UniProt-Consortium. UniProt: the universal protein knowledgebase. Nucleic Acids Res. 45 (D1), D158-D169 (2017).
- Olsen, J. V., Vermeulen, M., Santamaria, A., Kumar, C., Miller, M. L., Jensen, L. J., Gnad, F., Cox, J., Jensen, T. S., Nigg, E. A., Brunak, S., Mann, M. Quantitative phosphoproteomics reveals widespread full phosphorylation site occupancy during mitosis. Science Signal. 3 (104), ra3 (2010).
- Dephoure, N., Zhou, C., Villén, J., Beausoleil, S. A., Bakalarski, C. E., Elledge, S. J., Gygi, S. P. A quantitative atlas of mitotic phosphorylation. Proc Natl Acad of Sci U S A. 105 (31), 10762-10767 (2008).
- Rona, G., Marfori, M., Borsos, M., Scheer, I., Takacs, E., Toth, J., Babos, F., Magyar, A., Erdei, A., Bozoky, Z., Buday, L., Kobe, B., Vertessy, B. G. Phosphorylation adjacent to the nuclear localization signal of human dUTPase abolishes nuclear import: structural and mechanistic insights. Acta Cryst D. 69 (12), 2495-2505 (2013).
- Harreman, M. T., Kline, T. M., Milford, H. G., Harben, M. B., Hodel, A. E., Corbett, A. H. Regulation of nuclear import by phosphorylation adjacent to nuclear localization signals. J Biol Chem. 279 (20), 20613-20621 (2004).
- Kosugi, S., Hasebe, M., Tomita, M., Yanagawa, H. Systematic identification of cell cycle-dependent yeast nucleocytoplasmic shuttling proteins by prediction of composite motifs. Proc Natl Acad Sci U S A. 106 (25), 10171-10176 (2009).
- McLachlin, D. T., Chait, B. T. Analysis of phosphorylated proteins and peptides by mass spectrometry. Curr Opin Chem Biol. 5 (5), 591-602 (2001).
- Van Berkel, G. J., Glish, G. L., McLuckey, S. A. Electrospray ionization combined with ion trap mass spectrometry. Anal Chem. 62 (13), 1284-1295 (1990).
- Hodel, A. E., Harreman, M. T., Pulliam, K. F., Harben, M. E., Holmes, J. S., Hodel, M. R., Berland, K. M., Corbett, A. H. Nuclear localization signal receptor affinity correlates with in vivo localization in Saccharomyces cerevisiae. J Biol Chem. 281 (33), 23545-23556 (2006).
- Hong, K. U., Kim, H. -. J., Kim, H. -. S., Seong, Y. -. S., Hong, K. -. M., Bae, C. -. D., Park, J. Cdk1-Cyclin B1-mediated Phosphorylation of Tumor-associated Microtubule-associated Protein/Cytoskeleton-associated Protein 2 in Mitosis. J Biol Chem. 284 (24), 16501-16512 (2009).
- Meraldi, P., Lukas, J., Fry, A. M., Bartek, J., Nigg, E. A. Centrosome duplication in mammalian somatic cells requires E2F and Cdk2–Cyclin A. Nature Cell Biol. 1, 88 (1999).
- Heuvel, S., Harlow, E. Distinct roles for cyclin-dependent kinases in cell cycle control. Science. 262 (5142), 2050-2054 (1993).
- Kinoshita, E., Kinoshita-Kikuta, E., Takiyama, K., Koike, T. Phosphate-binding Tag, a New Tool to Visualize Phosphorylated Proteins. Mol Cell Proteomics. 5 (4), 749-757 (2006).
- Takeda, H., Kawasaki, A., Takahashi, M., Yamada, A., Koike, T. Matrix-assisted laser desorption/ionization time-of-flight mass spectrometry of phosphorylated compounds using a novel phosphate capture molecule. Rapid Commun Mass Spectrom. 17 (18), 2075-2081 (2003).
- Linder, M. I., Köhler, M., Boersema, P., Weberruss, M., Wandke, C., Marino, J., Ashiono, C., Picotti, P., Antonin, W., Kutay, U. Mitotic Disassembly of Nuclear Pore Complexes Involves CDK1- and PLK1-Mediated Phosphorylation of Key Interconnecting Nucleoporins. Dev Cell. 43 (2), (2017).
- Arai, T., Haze, K., Iimura-Morita, Y., Machida, T., Iida, M., Tanaka, K., Komatani, H. Identification of β-catenin as a novel substrate of polo-like kinase 1. Cell Cycle. 7 (22), 3556-3563 (2008).
- Hansen, D. V., Tung, J. J., Jackson, P. K. CaMKII and Polo-like kinase 1 sequentially phosphorylate the cytostatic factor Emi2/XErp1 to trigger its destruction and meiotic exit. Proc Natl Acad of Sci U S A. 103 (3), 608-613 (2006).
- Zhang, Y., Dong, Z., Nomura, M., Zhong, S., Chen, N., Bode, A. M., Dong, Z. Signal Transduction Pathways Involved in Phosphorylation and Activation of p70S6K Following Exposure to UVA Irradiation. J Biol Chem. 276 (24), 20913-20923 (2001).
- Richard, D. E., Berra, E., Gothié, E., Roux, D., Pouysségur, J. p42/p44 Mitogen-activated Protein Kinases Phosphorylate Hypoxia-inducible Factor 1α (HIF-1α) and Enhance the Transcriptional Activity of HIF-1. J Biol Chem. 274 (46), 32631-32637 (1999).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved