A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
יצירת חלבון מאוד ספציפי הנגרמת כימית מערכות דימריזציה על ידי בחירת בחירה באמצעות מבחר של קומבינטורית מתחם יחיד בספריית הנוגדן
In This Article
Summary
יצירת מערכות דימרזציה בתחום החלבונים עם הזיקה הרצויה וספציפיות לכל מולקולה קטנה שניתנה לפני הרבה מולקולות ובקשות חישה והגשמה. כאן, אנו מתארים שיטה יעילה, הניתן להכליל עבור דה נובו הנדסה של מערכות דימריזציה הנגרמת כימית דרך הבחירה החורגת של קומבינטורית הציג phage בספרייה נוגדן יחיד בתחום.
Abstract
אירועים מדימריזציה של חלבונים המתרחשים רק בנוכחות של מולקולה קטנה ומאפשרים פיתוח של מולקולה קטנה ביולוגית לניתוח ומניפולציה של מסלולים ביולוגיים. כיום, רק מספר מוגבל של מערכות dimerization בהשפעת כימית (CID) קיימים והנדסה חדשים עם רגישות הרצויה וסלקטיביות עבור ליגניות מולקולות קטנות מסוימות נשאר אתגר בתחום הנדסת חלבונים. אנו מתארים את השיטה הקרנת תפוקה גבוהה, comביננדרס בן ders-e nabled הבחירות שלסיד (משלב סיד), עבור דה נובו הנדסה של סיד מערכות החלים על מגוון רחב של ליגנדס. שיטה זו משתמשת בבחירה בשני השלבים של הספרייה phage מוצג קומבינטורית ננוגוף כדי לקבל 1) "עוגן אוגדן" כי לאגד הראשון כדי להשיג ולאחר מכן 2) "dimerization קלסרים" כי רק לאגד עוגן מאגד-ליגנד מכלולי. כדי לבחור בקלסרים עוגן, ספריית קומבינטורית של מעל 109 האזור משלימה לקביעת (cdr)-ננוגופים אקראיים מוקרן עם ligtinylated וכניסות מאומתים עם ליגנד על ידי ביולוגי אינטרפרומטריה (בלי). כדי להשיג בקלסרים dimerization הספרייה הננו מוקרן עם העוגן קלסר-ligand מכלולי כמטרות עבור הקרנה חיובית ואוגדן עוגן מאוגד עבור הקרנה שלילית. משלבת-CID ישימה באופן כללי כדי לבחור בקלסרים CID עם חיסוני אחרים, non-חיסוני, או בחינה חישובית עוצב מתוכננת הפיגומים כדי ליצור ביוחיישנים עבור מבחנה ב-vivo זיהוי של תרופות, מטבוליטים, מולקולות איתות, וכו '.
Introduction
CID מערכות, שבו שני חלבונים dimerize רק בנוכחות של מולקולה קטנה (איור 1), מציעים כלים רב-תכליתי לניתוח ותפעול מטבולית, איתות, ומסלולים ביולוגיים אחרים1. הם הפגינו את הפוטנציאל בהפעלה ביולוגית, כגון הפעלה מבוקרת סמים T2 ו אפופטוזיס3,4, לשיפור הבטיחות והיעילות של טיפול תא T המאמצת. בנוסף, הם מספקים מתודולוגיה חדשה בvivo או בזיהוי מתורבת של מטרות מולקולות קטנות. לדוגמה, חלבונים CID יכול להיות התמזגו גנטית עם מערכות העיתונאי הניאון (g., העברת אנרגיה בתהודה של אור השמש (למשל)5 ו מעגליות חלבונים פלורסנט מושתק)6 עבור בזמן אמת במדידות vivo, או לשמש ריאגנטים אהדה עבור כריך האנזים מקושר חיסוני כפוף (אליסה)-כמו assays.
למרות היישומים הרחבים שלהם, יצירת מערכות CID חדשות שיכולות להיות נשלטות על-ידי מולקולה קטנה ובעלת אתגרים מרכזיים. הוקמה שיטות הנדסה חלבון קלסר כולל חיסון בעלי חיים7, בחירה מחוץ גופית8,9, ועיצוב חלבון חישובית10 יכול ליצור ליגנד מחייב חלבונים הפועלים באמצעות חלבון בינארי-ליגנד אינטראקציות. עם זאת, שיטות אלה יש קשיים ביצירת מתחם CID ליגארי המושרה. כמה שיטות ליצור סיד על ידי קישור כימי שני ליגניות כי באופן עצמאי לאגד את אותם חלבונים או שונים11,12,13,14,15,16 או להסתמך על בחירת חלבונים קלסר כגון נוגדנים ליעד מולקולה קטנה מולקולת חלבון מתחמים17,18, ולכן יש מבחר מוגבל של ליגנדס.
פיתחנו לאחרונה מקוםהבחירה של החבר ' ה-e NABLEDהבחירות של סיד (משלבת סיד) שיטה עבור דה נובו הנדסה של סיד מערכות19. שיטה זו יכולה להשיג את היחודיות הגבוהה של דימרזציה (לדוגמה, קבוע עוגן-דימרזציה, kd (ללא Ligand)/kd (עם ligand) > 1,000). ספציפיות הדימרזציה מושגת באמצעות קלסרים עוגן עם אתרי קשירה גמישים שיכולים להציג שינויים מתרניים עם ליגנד מחייב, מתן בסיס לבחירה של קלסרים סלקטיבי מבחינה מבצעית רק לזהות ליגור העוגן אוגדן. הדגמנו הוכחה של עקרון על ידי יצירת קנאידיל (CBD) המושרה הטרודימרס של nanobodies, 12 – 15 מקטע נוגדן מסוג kda מתוך גמליים הכוללת פיגום אוניברסלי ושלושה cdr לולאות גמיש (איור 2)20, אשר יכול ליצור כיס מחייב עם הגדלים של מולקולהקטנה מולקולות21. בעיקר, הבחירה מחוץ גופית של ספריית החלבון קומבינטורית צריך להיות חסכוני וכללי עבור הנדסת CID, כי הספרייה באיכות גבוהה ניתן להחיל על ליגניות שונות.
בפרוטוקול זה וידאו, אנו מתמקדים בתיאור שני שלבים בחירת מבחנה ואימות של עוגן (איור 3A) ו dimerization קלסרים (איור 3ב) על ידי הקרנת ספריית קומבינטורית nanobody עם גיוון גבוה יותר 109 באמצעות CBD כיעד, אבל הפרוטוקול צריך להיות ישים לספריות חלבון אחרות או מולקולות קטנות מטרות. ההקרנה של בקלסרים CID לוקח בדרך כלל 6 – 10 שבועות (איור 4).
Access restricted. Please log in or start a trial to view this content.
Protocol
1. בניית ספרייה
- השתמש קומבינטורית סינתטי מתחם יחיד בספריית הנוגדן עם מגוון של ~ 1.23 – 7.14 x 109, כפי שתוארה בעבר19. בעוד שפרוטוקול זה אינו כולל בניית ספריות, ניתן להחילו על ספריות אוגדן אחרות הקומבינטורית.
2. ביוטילציה של ליג, מטרה או ליגטין
- Biotinylate אוחר the שנבחרו ligand, למשל, CBD ו tetrahydrocannabinol (THC)19, באמצעות אסטרטגיות סינתזה כימית שונים, בהתאם לאתרים הביוקטילציה מתאים של מטרה.
3. הקרנת אוגדן עוגן
- תחילת הבחירה
- התחל כל סיבוב של בחירה על ידי הTG1 מושבה אחת-cell, גדל טרי ב 6 מ ל של 2YT ב 37 ° צ' ו 250 מהפכות לדקה (rpm) ל 600 nm (OD600) בספיגת ~ 0.5. דגירה את התאים על הקרח לשימוש בשלב 3.5.1.
- בחירה שלילית עם חרוזי streptavidin מאוגדים בביוטין
- הכינו את "חרוזי בחירה שליליים" על ידי שטיפת 300 μL של חרוזים מגנטיים streptavidin באמצעות מתלה הפרדה מגנטית, 3x עם 0.05% פוספט באגירה מלוחים עם מאגר רצף (PBST, 1 x PBS עם 0.05% vol/vol רצף 20%) ו-2x עם 1 x PBS.
- להשעות את החרוזים עם 1 מ ל של 1% קזאין ב 1 x PBS (pH = 7.4), ולהרוות את החרוזים על ידי הוספת 5x קיבולת האיגוד המדווח באמצעות ביוטין. דגירה בטמפרטורת החדר (RT) על מסובבי עבור 1 h.
- לשטוף את החרוזים 5x באמצעות 0.05% PBST ו 3x באמצעות 1 x PBS, עבור סך של שמונה שוטף.
- הוסיפו ~ 1013 חלקיקים phage ב 1% קזאין/1% bsa ב 1 x PBS (pH = 7.4) ו דגירה ב-RT על מסובבי במשך 1 h.
- לאחר הדגירה, לאסוף את הסופרנטאנט כדי לשמש בשלב 3.3.6.
- בחירה חיובית עם חרוזים streptavidin מאוגדים ביולוגיים
- הכינו את "חרוזי הבחירה החיוביים" ב1/2 את נפח החרוזים המשמשים את "חרוזי הבחירה השליליים" בצעדים הבאים 3.2.1.
- השהה מחדש את החרוזים עם 1 מ"ל 1% קזאין ב-1 × PBS, pH 7.4 ורוויה החרוזים על ידי הוספת 5x קיבולת הכריכה המלאה מחושבת בהתבסס על המדריך באמצעות ליגטילנטיב ביולוגי הבחירה. מודטה ב-RT על מסובבי במשך 1 h.
- לשטוף את החרוזים 5x באמצעות 0.05% PBST ו 3x באמצעות 1 x PBS, עבור סך של שמונה שוטף.
- לחסום את החרוזים עם 1 מ ל של 1% קזאין/1% BSA ב 1 x PBS (pH = 7.4) ו דגירה ב-RT על מסובבי עבור 1 h כדי למנוע קשירה לא ספציפית בין phages לבין חרוזי מגנטי streptavidin מצופה.
- לשטוף את streptavidin מצופה חרוזים מגנטיים 3x באמצעות 0.05% PBST ופעם אחת באמצעות 1 x PBS, עבור סך של ארבעה שוטף.
- השהה מחדש את streptavidin מצופה חרוזים מגנטיים באמצעות phages מאוגד נלקח משלב 3.2.5 ו דגירה ב RT על מסובבי עבור 1 h.
- מבלי להפריע. לחרוזים המגנטיים שמור את ה-phages הלא מאוגדים כקלט, שישמש בשלב 3.5.1.
- לשטוף את החרוזים 10x באמצעות 0.05% PBST ו 5x באמצעות 1 x PBS. בין כל שלוש שוטף להעביר אותם צינור חדש כדי למנוע phages מאוגד במיוחד אל קירות הצינור.
- הימנעות מהננו-רובוטים מוצגים
- מאגד תחרותי מאוגד באמצעות הוספת 450 μL של ליגטין לא-biotinylated, באמצעות ריכוז בטווח micromolar (למשל, 10 – 50 μm) ו דגירה ב RT על מסובבי עבור 30 דקות. ליגיון שנבחרו הריכוז התחרותי של phages מאוגד תלוי ב - K הרצוי של העוגן ". ליגדי ריכוזים יכולים להיות גבוהים יחסית בסיבובים של בחירה ראשונית ולאחר מכן להקטין בסיבובים מאוחרים יותר.
- לאסוף סופרנטאנט ולשמור את phages הפלט כמו תפוקה, כדי לשמש בשלב 3.5.2.
- חדירות קלט/פלט וזיהום
- עבור titration קלט, להכין מדלל טורי 10x ב 1 x PBS עד 109-קיפול עם הקלט phage משלב 3.3.7. השתמש 107-109 מדלל סדרתי כדי לעשות זיהומים על ידי העברת 10 μl קלט phage מכל דילול כדי 70 μl TG1 תאים (OD600 של ~ 0.5). מודקון ב 37 ° c עבור 45 דקות, צלחת TG1 תאים נגועים על 3 90 mm 2YT-אגר מנות המכילות 100 g/mL אמפיצילין ו 2% (wt/vol) גלוקוז, ו הדגירה לילה ב 37 ° c. מלוחות הלילה, ניתן לחשב קלט phage באופן הבא:
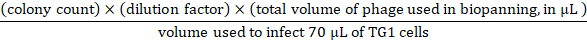
- עבור זיהום הפלט ואת טיטור, העבר את מצנח phages משלב 3.4.2 כדי 3 מ ל של TG1 תאים (OD600 של ~ 0.5). דגירה באמבט מים ב 37 ° c עבור 45 דקות. לאחר מכן להכין מדלל טורי 10x ב 2YT עד 103-קיפול, צלחת כל דילול על 90 MM 2yt-אגר מנות, ו דגירה לילה ב 37 ° c. מלוחות הלילה, ניתן לחשב פלט phage באופן הבא:
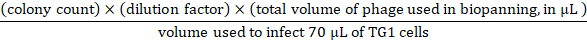
- לחלק את שאר התאים TG1 נגועים ב-3 150 mm 2YT-אגר לוחות המכילים 100 μg/mL אמפיצילין ו 2% (wt/vol) גלוקוז. לוחיות הרישוי של הלילה. ב-37 ° c
- עבור titration קלט, להכין מדלל טורי 10x ב 1 x PBS עד 109-קיפול עם הקלט phage משלב 3.3.7. השתמש 107-109 מדלל סדרתי כדי לעשות זיהומים על ידי העברת 10 μl קלט phage מכל דילול כדי 70 μl TG1 תאים (OD600 של ~ 0.5). מודקון ב 37 ° c עבור 45 דקות, צלחת TG1 תאים נגועים על 3 90 mm 2YT-אגר מנות המכילות 100 g/mL אמפיצילין ו 2% (wt/vol) גלוקוז, ו הדגירה לילה ב 37 ° c. מלוחות הלילה, ניתן לחשב קלט phage באופן הבא:
- הגברה והתאוששות של הספרייה לסיבובים נוספים של בחירה
- הוסף 3 מ ל של 2YT לצלחת, לגרד עם מגרד תא סטרילי ולאסוף את כל התאים בשפופרת 50 mL חרוט. מערבבים את התאים שנאספו עם גליצרול סטרילי (20% wt/כרך הריכוז הסופי). למדוד את OD600 של התערובת ולעשות 3 – 5 המניות מניות. חנות at-80 ° צ' לאחסון לטווח ארוך.
- עבור הצלה phage, לדלל את phagemid המכיל TG1 בקטריאלי באמצעות 25 מ ל של 2YT מדיה בתוספת 2% גלוקוז ו 100 μg/mL אמפיצילין כדי OD600 של ~ 0.1. התאים התרבותיים ב 37 ° צ' ו 250 rpm ל-OD600 של ~ 0.5.
- Superinfect את התאים על ידי הוספת CM13 עוזר phage ב 5 x 109 pfu/mL ו-דגירה ב 37 ° צ' ו 250 rpm עבור 45 דקות. CM13 עוזר phage מספק נדרש חלבונים מעיל phage להרכבה של חלקיקים phage להשלים.
- צנטריפוגה את התרבות ב 8,000 x g עבור 10 דקות כדי להסיר את הגלוקוז. להשעות מחדש את התאים באמצעות 50 mL של 2YT מדיה בתוספת 100 μg/mL אמפיצילין ו 50 μg/mL kanamycin ו-דגירה ב 25 ° צ' ו 250 rpm לילה.
- צנטריפוגה את התאים מן התרבות לילה ב 9,000 x g, 4 ° צ' עבור 30 דקות. העברת supernatant לצינור חדש מזרז phages ב-supernatant באמצעות 1/5 נפח יתד/הרוג פתרון (20% wt/vol-פוליאתילן גליקול-6,000 ו2.5 M הנאל). מערבבים בעדינות ומניחים על קרח במשך 1 h.
- לאסוף חלקיקים phage על ידי צנטריפוגה באמצעות 12,000 x g ב 4 ° c עבור 30 דקות. להשעות את כדורי באמצעות 1 mL של 1 x PBS, ולהעביר את ההשעיה לצינור מיקרוצנטריפוגה. צנטריפוגה את הצינור ב 20,000 x g ו 4 ° צ' עבור 10 דקות כדי להסיר חיידקים שיורית.
- מבלי להפריע. לגלולה החיידקית השתמש בדילול 1:100 כדי למדוד את הספיגה ב 269 nm ו 320 nm. ניתן לחשב את המספר הכולל של phages באמצעותהנוסחההבאה:
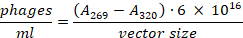
- אחסן בספריית phage ב -4 ° c לשימוש לטווח קצר או עם 25% גליצרול ב-80 ° c לאחסון לטווח ארוך.
- חזור על סבבים של בחירה (שלבים 3.1 – 3.6) עבור 3 – 6 סיבובים או עד לקבלת העשרה רצויה (עיין בסעיף תוצאות). צלחת לבחור שיבוטים יחיד (סעיף 4) על מנת לאפיין את הזיקה שלהם ספציפיות לליטור (סעיפים 5 – 7).
4. בידוד לשכפול יחיד
- כדי לבודד שיבוטים בודדים מספריית משנה מועשר, להכין מדלל סדרתי 10x של תאים TG1 נגוע phage (שלב 3.5.2). מדלל את הלוח על 90 mm 2YT-אגר מנות המכילות 100 μg/mL אמפיצילין ו 2% (wt/vol) גלוקוז ו דגירה ב 37 ° צ' לילה.
- מתוך לוחות הלילה, לבחור מושבות יחיד לתוך 250 μL של מדיה 2YT בתוספת 100 μg/mL אמפיצילין per גם ב לוחיות היטב סטרילי עמוק ולצמוח ב 37 ° c לילה.
- מן התרבויות לילה, האיחסן 10 μL לתוך 500 μL של מדיה 2YT טריים בתוספת 100 μg/mL אמפיצילין.
- לגדול תאים כדי OD600 של ~ 0.5, להוסיף CM13 עוזר phage ב 5 x 109 pfu/mL ו-דגירה ב 37 ° צ' ו 250 סל ד עבור 45 min.
- הוסף 500 μL של 2YT מדיה בתוספת 100 μg/mL אמפיצילין ו 50 μg/mL kanamycin. דגירה ב 25 ° צ' ו 250 rpm לילה.
- צנטריפוגה את צלחות הבאר העמוקה מן התרבויות לילה ב 3,000 x g עבור 10 דקות. לאסוף את supernatant המכיל את חלקיקי phage מבלי להפריע את הגלולה התא.
- חלקיקי phage ניתן להשתמש כדי אליסה כדי לקבוע את הספציפיות של שיבוטים שנבחרו כדי ligand. Biotin או מבנה הומויומן של היעד ניתן להשתמש כפקד שלילי.
5. בדיקת העוגן על ידי אליסה
- מעיל 96 היטב לוחיות הרישוי באמצעות 100 μL של 5 μg/mL streptavidin במאגר ציפוי (100 mM קרבונט מאגר, pH = 8.6) ב 4 ° צלזיוס לילה.
- לשטוף את הצלחות אליסה 3x באמצעות 0.05% PBST ולהוסיף 100 μL של 1 μM היעד biotinylated היעד בארות. הוסף 100 μL של ביוטין 1 μM או מטרה הומובול לבקרת הבקרה. . בסדר.
- לשטוף את הצלחות 5x באמצעות 0.05% PBST ולחסום איגוד ספציפי על ידי הוספת 300 μL של 1% קזאין ב 1 x PBS. . בסדר.
- שוטפים את לוחיות ה-דיאגנוסטיקה 3x באמצעות 0.05%-PBST ומוסיפים את הפייגה הטהורה של phage. . מודטה בשביל 1 h ב-RT
- לשטוף את הצלחות אליסה 10x באמצעות 0.05% PBST ולהוסיף 100 μL peroxidase (HRP)-M13 מעיל הגדול נוגדן חלבון (1:10000 דילול עם 1 x PBS עם 1% קזאין). . בסדר.
- שטוף את צלחות אליסה 3x באמצעות 0.05% PBST ולהוסיף 100 μL טטרמתיד (TMB) המצע. דגירה עבור 10 דקות או עד שינוי צבע גלוי הוא נצפתה. להפסיק את התגובה על ידי הוספת 100 μL של HCl 1 M. לקרוא את הצלחת ב 450 ננומטר על ספקטרוסקופיה.
- עבור ביטוי חלבון וטיהור, בחר את המשובטים מראה אהדה גבוהה וספציפיות עבור המטרה (ראה דיון).
6. ביטוי חלבון, טיהור וביוטילציה
- כפי שדווח בעבר19, משנה שיבוטים שנבחרו מסעיף 5 ולהביע כמו C-טרמינל Avi-tagged ו-מתויג שלו ננוגופים.
- לבטא ננו-רובוטים שנבחרו בפריפלמטר של E. coli WK6 תאים (בדרך כלל בתרבות 1 L), לשחרר על ידי הלם אוסמוטי, ולטהר באמצעות העמודה ניקל-נ. ת. ע (ראה טבלת חומרים).
- מאגר Exchange עם טור התפלה (1 x PBS עם 5% גליצרול; ראו טבלת חומרים).
- ננוגופים בbiotinylate אוחרים באמצעות ערכה מסחרית (ראו טבלת חומרים) לשימוש נוסף.
7. אפיון עוגן על ידי בבלי
- לנתח את האהדה ואת הקינטיקה של קלסרים עוגן נבחר על ידי השתק 200 ננומטר ביוטילנטיל העוגן על streptavidin bioחיישנים (ראה טבלת חומרים) עם מאגר שיטת מחייב (1 x PBS (pH = 7.4), 0.05% יצירת רצף 20, 0.2% bsa, 3% מתנול).
- חישוב קבועי דיסוציאציה (KD) של מאגד עוגן-ליגניות ואינטראקציות באמצעות ניתוח מצב יציב באמצעות תוכנת ניתוח נתונים (ראה טבלת חומרים). התקבלו ערכי K בדרך כלל טווח בין מיקרומטר יחיד לספרה כפולה.
8. הקרנת אוגדן דימרזציה
הערה: הקרנת הביואננינג של "קלסרים לפירוק" דומה לזו של קלסרים עוגן, למעט שני שלבים קריטיים: 1) קלסרים Dimerization נבחרים באמצעות קלסר העוגן הנבחר biotinylated ואת העוגן-ligand מורכב עבור שלילי ובחירות חיוביות, בהתאמה. 2) במהלך הצעד, ה100 מילימטר triethyine משמש כדי להשתמש בפייגים שנבחרו באופן חיובי, שהיו מאוגדים רק לכורך העוגן--ליגס ומתחם היעד. הפתרון 100 mM trimethylamine (pH = 11.5) משמש כדי לשכפל שיבוטים חיוביים על ידי שיבוש אינטראקציות החלבון.
- תחילת הבחירה
- התחל כל סיבוב של בחירה על ידי הTG1 מושבת התא יחיד, גדל טרי על מדיה מינימלית, ב 6 מ ל 2YT ב 37 ° c ו 250 rpm ל-OD600 של ~ 0.5. . התאים האלה בקרח
- הסרה של ננוגופים שלילית שנבחרו
- הכן את "צינור חיסור" באמצעות 400 μL של חרוזים מגנטיים streptavidin מצופה ולאחר שלב 3.2. עם זאת, במקום לטפל בביוטין, הוסף 5x את קיבולת האיגוד המלאה המחושבת באמצעות קלסר העוגן הנבחר של biotinylated ושמור את ה-phages הלא מאוגדים לשימוש בשלב 8.3.3.
- מבחר של ננוגופים שנבחרו באופן חיובי
- הכן את "שפופרת לכידת" באמצעות 1/2 הנפח של חרוזים מגנטיים streptavidin מצופה המשמש "צינור חיסור" והשלבים הבאים 3.3.2 כדי 3.3.3. עם זאת, במקום לשבת עם ligtinylated, הוסף פי חמש את קיבולת האיגוד המלאה המחושבת באמצעות קלסר העוגןהנבחר.
- כדי ליצור את קלסר העוגן-ליגנד מורכב עבור בחירת האוגדן dimerization חיובית, להוסיף ריכוז מספיק גבוה של ligtinylated לא. הדבר יאפשר לstreptavidin העוגן המאוגד ביותר ליצור את המכלול המאוגד ליגולי.
- בצע את השלבים 3.3.3 to 3.3.8, באמצעות phages לא מאוגד נלקח "צינור חיסור".
- הימנעות של ננוגופים שנבחרו באופן חיובי
- Elute phages מאוגד העוגן מאגד-ligand מורכב על ידי הוספת 450 μL של 100 mM triethylamine, ו הדגירה ב RT על מסובבי עבור 10 דקות.
- לאסוף את הפייגים התחרותיים ולעקוב אחר הצעדים ה3.4.1 ל3.4.2.
- סבבים נוספים של בחירת אוגדן דימרזציה
- בצע את השלבים 3.5 ו 3.6 כדי להגביר ולשחזר את הספרייה כדי לבצע סיבובים נוספים של בחירה. חזור על סבבים של בחירה עבור 3 – 6 סיבובים או עד לקבלת העשרה רצויה. צלחת לבחור שיבוטים יחיד (להתייחס לסעיף 4) על מנת לאפיין את הזיקה שלהם ספציפיות למטרה.
9. אפיון של אוגדן דימרוניזציה מאת אליסה
- בצע את השלבים בסעיף 4 כדי לבודד שיבוטים בודדים לאפיון דרך אליסה.
- כדי לבדוק את האהדה של מועמדים באוגדן dimerization כדי לעגן את העוגן-ligand מורכבים, המעיל צלחת היעד של אליסה באמצעות 100 μL של 100 מאגד העוגן biotinylated. לאחר הדגירה של 1 h, להוסיף 1 μm של ליגנד היעד כדי ליצור מאגד עוגן-ליגנד מורכב.
- לוחית הבקרה צריכה להיות מצופה באמצעות קלסר העוגן biotinylated לבד כדי למסך את המשובטים כי יכול גם לאגד לאוגדן עוגן חינם. להוסיף 100 μL של 100 מאגד העוגן biotinylated ו מודטה ב RT עבור 1 h.
- בצע את סעיפים 5.3 – 5.7.
10. אפיון של אוגדן דימרזציה מאת בלי
- הזיקה הכבילה והקינטיקה של אוגדן הדימרזציה לאוגדן העוגן-ניתן לנתח את מורכבות המידע על-ידי שינופי קלסרים ביולוגיים ביולוגיים על streptavidin (SA) bioחיישנים עם מאגר שיטת הכריכה ולאחר מכן מקבל 1 μM עיגון עוגן מראש באמצעות דיעות טוריות של ligand. KD, konו- kמחוץ לאינטראקציות ניתן לחשב באמצעות שיטה מדווחת19.
Access restricted. Please log in or start a trial to view this content.
תוצאות
אנו מתארים את שני השלבים בבחירת מבחנה ואימות של העוגן ו dimerization קלסרים על ידי הקרנת הספרייה קומבינטורית ננוbody עם גיוון גבוה יותר 109 באמצעות CBD כיעד. הערכת העשרת ביואננינג במהלך הסיבובים הרצופים של בחירת העוגן והדימרזציה, חשובה ביותר. תוצאות העשרה טיפוסיות לאחר 4 – 6 סיבובים של בחירה כפ...
Access restricted. Please log in or start a trial to view this content.
Discussion
חשוב לבחור את הריכוזים הנכונים של ספריות phage קלט עבור סיבובים שונים של ביואננינג. אנחנו בדרך כלל התחלנו מספריית הקלט של ~ 1012– 1013 phage חלקיקים עם גיוון > 109, המאפשר ~ 100-1000 עותקים של כל שיבוט phage להיות מוצג בתוך שיטת המשיכה למטה. אם הריכוז phage בתוך הסדר מחייב הוא גבוה מדי או נמוך, ?...
Access restricted. Please log in or start a trial to view this content.
Disclosures
פטנט זמני הקשור לעבודה זו הוגשה על ידי אוניברסיטת וושינגטון.
Acknowledgements
עבודה זו נתמכת על ידי פרס החדשנות אוניברסיטת וושינגטון (כדי L.G.), מענק של U.S. המכונים הלאומיים לבריאות (1R35GM128918 כדי L.G.), ו קרן אתחול של אוניברסיטת וושינגטון (כדי L.G.). H.J. היתה נתמכת על ידי מלגת קרן וושינגטון למחקר. K.W. נתמכת על ידי מלגת לתואר ראשון מהמכון לעיצוב חלבונים באוניברסיטת וושינגטון.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| 1-Step Ultra TMB ELISA substrate solution | Thermo Fisher Scientific | 34029 | |
| Agar | Thermo Fisher Scientific | BP1423-2 | |
| Amicon Ultra-15 Centrifugal Filter unit (3 kDa cutoff) | Millipore | UFC900324 | |
| Ampicillin | Thermo Fisher Scientific | BP1760-25 | |
| Bio-Rad Protein Assay Kit II | Bio-Rad | 5000002 | |
| BirA biotin-protein ligase standard reaction kit | Avidity | BirA500 | |
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A2153-50G | |
| Casein | Sigma-Aldrich | C7078-1KG | |
| CM13 Helper phage | Antibody Design Labs | PH020L | |
| D-(+)-Glucose monohydrate | Alfa Aesar | A11090 | |
| Dynabeads M-280 Streptavidin | Thermo Fisher Scientific | 11205D | |
| DynaMag-2 Magnet | Thermo Fisher Scientific | 12321D | |
| EDTA | Thermo Fisher Scientific | BP120-1 | |
| Fast DNA Ladder | New England Biolabs | N3238S | |
| FastDigest BglI | Thermo Fisher Scientific | FD0074 | |
| Glycerol | Thermo Fisher Scientific | BP229-1 | |
| HiLoad 16/600 Superdex 200 pg | GE Healthcare | 28989335 | |
| HiPrep 26/10 Desalting Column | GE Healthcare | 17508701 | |
| HisTrap-FF-1ml | GE Healthcare | 11000458 | |
| Imidazole | Alfa Aesar | 161-0718 | |
| IPTG | Thermo Fisher Scientific | 34060 | |
| Kanamycin | Thermo Fisher Scientific | BP906-5 | |
| M13 Major Coat Protein Antibody | Santa Cruz Biotechnology | sc-53004 | |
| NaCl | Sigma-Aldrich | S3014-500G | |
| NanoDrop 2000/2000c Spectrophotometers | Thermo Fisher Scientific | ND-2000 | |
| Nunc 96-Well Polypropylene DeepWell Storage Plates | Thermo Fisher Scientific | 260251 | |
| Nunc MaxiSorp | Thermo Fisher Scientific | 44-2404-21 | |
| Octet RED96 | ForteBio | N/A | |
| pADL-23c Phagemid Vector | Antibody Design Labs | PD0111 | |
| PEG-6000 | Sigma-Aldrich | 81260-1KG | |
| Platinum SuperFi DNA Polymerase | Invitrogen | 12351010 | |
| PureLink PCR Purification Kit | Thermo Fisher Scientific | K310001 | |
| QIAprep Spin M13 Kit | Qiagen | 27704 | |
| Recovery Medium | Lucigen | 80026-1 | |
| SpectraMax Plus 384 | Molecular Devices | N/A | |
| Sucrose | Sigma-Aldrich | S0389-1KG | |
| Super Streptavidin (SSA) Biosensors | ForteBio | 18-5057 | |
| Superdex 75 increase 10/300 GL Column | GE Healthcare | 28-9909-44 | |
| T4 DNA Ligase | Thermo Fisher Scientific | 15224-025 | |
| TG1 Electrocompetent Cells | Lucigen | 60502-1 | |
| Triethylamine | Sigma-Aldrich | 471283-100mL | |
| Trizma Base | Sigma-Aldrich | T1503 | |
| Tryptone | Thermo Fisher Scientific | BP9726-5 | |
| Tween 20 | Thermo Fisher Scientific | BP337-500 | |
| Yeast Extract | Thermo Fisher Scientific | BP1422-2 | |
| Zeba Spin Desalting Column | Thermo Fisher Scientific | 89882 |
References
- Stanton, B. Z., Chory, E. J., Crabtree, G. R. Chemically induced proximity in biology and medicine. Science. 359 (6380), (2018).
- Wu, C. Y., Roybal, K. T., Puchner, E. M., Onuffer, J., Lim, W. A. Remote control of therapeutic T cells through a small molecule-gated chimeric receptor. Science. 350 (6258), (2015).
- Straathof, K. C., et al. An inducible caspase 9 safety switch for T-cell therapy. Blood. 105 (11), 4247-4254 (2005).
- Di Stasi, A., et al. Inducible apoptosis as a safety switch for adoptive cell therapy. The New England Journal of Medicine. 365 (18), 1673-1683 (2011).
- Mank, M., et al. A FRET-based calcium biosensor with fast signal kinetics and high fluorescence change. Biophysical Journal. 90 (5), 1790-1796 (2006).
- Nagai, T., Sawano, A., Park, E. S., Miyawaki, A. Circularly permuted green fluorescent proteins engineered to sense Ca2+. Proceedings of the National Academy of Sciences of the United States of America. 98 (6), 3197-3202 (2001).
- Hunter, M. M., Margolies, M. N., Ju, A., Haber, E. High-affinity monoclonal antibodies to the cardiac glycoside, digoxin. Journal of Immunology. 129 (3), 1165-1172 (1982).
- Bradbury, A. R. M., Sidhu, S., Dubel, S., McCafferty, J. Beyond natural antibodies: the power of in vitro display technologies. Nature Biotechnology. 29 (3), 245-254 (2011).
- Chen, G., et al. Isolation of high-affinity ligand-binding proteins by periplasmic expression with cytometric screening (PECS). Nature. Biotechnology. 19 (6), 537-542 (2001).
- Tinberg, C. E., et al. Computational design of ligand-binding proteins with high affinity and selectivity. Nature. 501 (7466), 212-216 (2013).
- Spencer, D. M., Wandless, T. J., Schreiber, S. L., Crabtree, G. R. Controlling signal transduction with synthetic ligands. Science. 262 (5136), 1019-1024 (1993).
- Ho, S. N., Biggar, S. R., Spencer, D. M., Schreiber, S. L., Crabtree, G. R. Dimeric ligands define a role for transcriptional activation domains in reinitiation. Nature. 382 (6594), 822-826 (1996).
- Belshaw, P. J., Ho, S. N., Crabtree, G. R., Schreiber, S. L. Controlling protein association and subcellular localization with a synthetic ligand that induces heterodimerization of proteins. Proceedings of the National Academy of Sciences of the United States of America. 93 (10), 4604-4607 (1996).
- Farrar, M. A., AlberolaIla, J., Perlmutter, R. M. Activation of the Raf-1 kinase cascade by coumermycin-induced dimerization. Nature. 383 (6596), 178-181 (1996).
- Erhart, D., et al. Chemical Development of Intracellular Protein Heterodimerizers. Chemistry & Biology. 20 (4), 549-557 (2013).
- Ballister, E. R., Aonbangkhen, C., Mayo, A. M., Lampson, M. A., Chenoweth, D. M. Localized light-induced protein dimerization in living cells using a photocaged dimerizer. Nature Communications. 17 (5), 5475(2014).
- Hill, Z. B., Martinko, A. J., Nguyen, D. P., Wells, J. A. Human antibody-based chemically induced dimerizers for cell therapeutic applications. Nature Chemical Biology. 14 (2), 112-117 (2018).
- Foight, G. W., et al. Multi-input chemical control of protein dimerization for programming graded cellular responses. Nature Biotechnology. 37 (10), 1209-1216 (2019).
- Kang, S., et al. COMBINES-CID: An efficient method for de novo engineering of highly specific chemically induced protein dimerization systems. Journal of the American Chemical Society. 141 (28), 10948-10952 (2019).
- Muyldermans, S. Nanobodies: natural single-domain antibodies. Annual Review of Biochemistry. 82, 775-797 (2013).
- Fanning, S. W., Horn, J. R. An anti-hapten camelid antibody reveals a cryptic binding site with significant energetic contributions from a nonhypervariable loop. Protein Science. 20 (7), 1196-1207 (2011).
- Zavrtanik, U., Luken, J., Loris, R., Lah, J., Hadzi, S. Structural basis of epitope recognition by heavy-chain camelid antibodies. Journal of Molecular Biology. 430 (21), 4369-4386 (2018).
- Denhardt, D. T., Dressler, D., Ray, D. S. The Single-Stranded DNA Phages. , 605-625 (1978).
- Virnekas, B., et al. Trinucleotide phosphoramidites: ideal reagents for the synthesis of mixed oligonucleotides for random mutagenesis. Nucleic Acids Research. 22 (25), 5600-5607 (1994).
- Gu, L., et al. Multiplex single-molecule interaction profiling of DNA-barcoded proteins. Nature. 515 (7528), 554-557 (2014).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved