Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Création de systèmes de dimerisation des protéines hautement spécifiques induits par des produits chimiques par stepwise Phage Selection of a Combinatorial Single-Domain Un-Domain Un-Domain Un-Domain Body Library
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
La création de systèmes de dimerisation des protéines induits chimiquement avec l'affinité et la spécificité souhaitées pour n'importe quelle petite molécule donnée ligand aurait beaucoup d'applications biologiques de détection et d'actionnement. Ici, nous décrivons une méthode efficace et généralisable pour l'ingénierie de novo des systèmes de dimerization induits chimiquement par l'intermédiaire de la sélection progressive d'une bibliothèque d'anticorps combinatorial de phage-affiché.
Résumé
Les événements de dimerisation des protéines qui se produisent seulement en présence d'un ligand à petites molécules permettent le développement de biocapteurs à petites molécules pour la dissection et la manipulation des voies biologiques. À l'heure actuelle, il n'existe qu'un nombre limité de systèmes de dimerisation induite par des produits chimiques (CID) et l'ingénierie de nouveaux systèmes avec la sensibilité et la sélectivité souhaitées pour des ligands spécifiques à petites molécules demeure un défi dans le domaine de l'ingénierie des protéines. Nous décrivons ici une méthode de criblage à haut débit, combinatorial binders-enabled sélection de CID (COMBINES-CID), pour l'ingénierie de novo des systèmes CID applicables à une grande variété de ligands. Cette méthode utilise la sélection en deux étapes d'une bibliothèque de nanobody combinatorial phage-affichée pour obtenir 1) « liants d'ancrage » qui se lient d'abord à un ligand d'intérêt et puis 2) « liants de dimerisation » qui se lient seulement aux complexes liants-ligands d'ancrage. Pour sélectionner les liants d'ancrage, une bibliothèque combinatoire de plus de 109 nanocorps randomisés de région de complémentarité (CDR) est examinée à l'autorisation d'un ligand biotinylated et les hits sont validés avec le ligand non étiqueté par interférométrie biocouche (BLI). Pour obtenir des classeurs de dimerisation, la bibliothèque nanobody est examinée avec des complexes de liant d'ancrage-ligand comme cibles pour le criblage positif et les liants d'ancrage non liés pour le criblage négatif. COMBINES-CID est largement applicable à certains liants CID avec d'autres immunoglobulines, non-immunoglobuline, ou des échafaudages conçus par calcul pour créer des biocapteurs pour la détection in vitro et in vivo de médicaments, métabolites, molécules de signalisation, etc.
Introduction
Les systèmes CID, dans lesquels deux protéines se dimerisent seulement en présence d'un ligand à petites molécules (figure 1), offrent des outils polyvalents pour disséquer et manipuler les voies métaboliques, de signalisation et autres voies biologiques1. Ils ont démontré le potentiel dans l'actionnement biologique, tel que l'activation de cellule T drogue-commandée2 et l'apoptose3,4, pour améliorer l'innocuité et l'efficacité de la thérapie de cellule T adoptive. En outre, ils fournissent une nouvelle méthodologie pour la détection in vivo ou in vitro des cibles de petites molécules. Par exemple, les protéines CID peuvent être génétiquement fusionnées avec des systèmes de rapporteur de fluorescence (p. ex., transfert d'énergie par résonance fluorescence (FRET)5 et protéines fluorescentes permutées circulairement)6 pour des mesures in vivo en temps réel, ou servir de réactifs d'affinité pour les analyses immunosorbent liées à l'enzyme sandwich (ELISA).
Malgré leurs applications étendues, la création de nouveaux systèmes CID qui peuvent être contrôlés par un ligand à petites molécules donnée présente des défis majeurs. Les méthodes établies d'ingénierie de liant de protéine comprenant la vaccinationanimale 7,la sélection in vitro8,9, et la conception de protéine computationnelle10 peuvent produire des protéines de liaison de ligand qui fonctionnent par l'intermédiaire des interactions binaires de protéine-ligand. Cependant, ces méthodes ont des difficultés à créer un complexe de CID ternaire induit par ligand. Certaines méthodes créent CID en reliant chimiquement deux ligands qui se lient indépendamment aux mêmes protéines ou différentes11,12,13,14,15,16 ou s'appuient sur la sélection de protéines de liant tels que les anticorps ciblant préexistant sa petite molécule-protéines complexes17,18, et ont donc un choix limité de ligands.
Nous avons récemment développé une combinatorial binders-enabled sélection de CID (COMBINES-CID) méthode pour l'ingénierie de novo des systèmes CID19. Cette méthode peut obtenir la grande spécificité de la dimerisation induite par ligand (par exemple, une dissociation de liant d'ancrage-dimerisation constante, KD (sans ligand)/KD (avec ligand) -gt; 1.000). La spécificité de la dimerisation est obtenue à l'aide de liants d'ancrage avec des sites de liaison flexibles qui peuvent introduire des changements conformationnels lors de la liaison ligand, fournissant une base pour la sélection de liants sélectifs conformationnellement ne reconnaissant que les liants d'ancrage ligand-liés. Nous avons démontré une preuve de principe en créant des hétérodimères induits par le cannabidiol (CBD) de nanocorps, un fragment d'anticorps fonctionnel de 12 à 15 kDa à partir de camélidés comprenant un échafaudage universel et trois boucles CDR flexibles (Figure 2)20, qui peuvent former une poche de liaison avec des tailles adaptables pour les épitopes de petites molécules21,22. Notamment, la sélection in vitro d'une bibliothèque de protéines combinatoires devrait être rentable et généralisable pour l'ingénierie CID parce que la même bibliothèque de haute qualité peut être appliquée à différents ligands.
Dans ce protocole et cette vidéo, nous nous concentrons sur la description de la sélection in vitro en deux étapes et de la validation des liants d'ancrage (figure 3A) et des liants de dimerisation (figure 3B) en examinant la bibliothèque combinatoire nanobody avec une diversité supérieure à 109 en utilisant le CBD comme cible, mais le protocole devrait s'appliquer à d'autres bibliothèques de protéines ou à des cibles à petites molécules. Le dépistage des liants CID prend habituellement de 6 à 10 semaines(figure 4).
Access restricted. Please log in or start a trial to view this content.
Protocole
1. Construction de la bibliothèque
- Utilisez une bibliothèque d'anticorps combinatoire synthétique à un seul domaine avec une diversité de 1,23 à 7,14 x 109, comme décrit précédemment19. Bien que ce protocole n'inclut pas la construction de bibliothèques, il peut être appliqué à d'autres bibliothèques de liants combinatoires.
2. Biotinylation de la cible de ligand ou du ligand
- Biotinylate le ligand sélectionné, par exemple, CBD et tétrahydrocannabinol (THC)19, via diverses stratégies de synthèse chimique, selon les sites de biotinylation appropriés d'une cible.
3. Ancrer le criblage de liant
- Début de la sélection
- Commencez chaque tour de sélection en inoculant une seule colonie à cellules TG1, fraîchement cultivée en 6 ml de 2YT à 37 oC et 250 tours par minute (rpm) à une absorption de 600 nm (OD600) de 0,5. Incuber les cellules sur la glace pour l'utilisation à l'étape 3.5.1.
- Sélection négative avec perles de streptavidin à la biotine
- Préparer les « perles de sélection négatives » en lavant 300 L de perles magnétiques recouvertes de streptavidin à l'aide d'un support de séparation magnétique, 3x avec 0,05 % de salin tampon tamponné par phosphate avec tampon Tween (PBST, 1 x PBS avec 0,05 % vol/vol Tween 20 %) et 2x avec 1 x PBS.
- Resuspendre les perles avec 1 ml de 1% de caséine dans 1 x PBS (pH 7,4), et saturer les perles en ajoutant 5x la capacité de liaison déclarée à l'aide de biotine. Incuber à température ambiante (RT) sur un rotateur pendant 1 h.
- Laver les perles 5x à l'aide de 0,05% PBST et 3x en utilisant 1 x PBS, pour un total de huit lavages.
- Ajouter 1013 particules de phage dans 1% de caséine/1% de BSA dans 1 x PBS (pH 7,4) et incuber à RT sur un rotateur pendant 1 h.
- Après l'incubation, recueillir le supernatant à utiliser à l'étape 3.3.6.
- Sélection positive avec perles de streptavidin à ligand biotinylated
- Préparer les « perles de sélection positives » en utilisant la demi-année du volume des perles utilisées pour les « perles de sélection négatives » suivant les étapes 3.2.1.
- Resuspendre les perles avec 1 mL 1% de caséine en 1 PBS, pH 7,4 et saturer les perles en ajoutant 5fois la pleine capacité de liaison calculée sur la base du manuel à l'aide du ligand biotinylated de choix. Incuber à RT sur un rotateur pendant 1 h.
- Laver les perles 5x à l'aide de 0,05% PBST et 3x en utilisant 1 x PBS, pour un total de huit lavages.
- Bloquer les perles avec 1 ml de 1% de caséine/1% BSA dans 1 x PBS (pH 7.4) et incuber à RT sur un rotateur pendant 1 h pour empêcher la liaison non spécifique entre les phages et les perles magnétiques enduites de streptavidin.
- Laver les perles magnétiques enduites de streptavidin 3x à l'aide de 0,05 % de PBST et une fois à l'aide de 1 x PBS, pour un total de quatre lavages.
- Resuspendre les perles magnétiques enduites de streptavidin à l'aide des phages non liés prélevés à partir de l'étape 3.2.5 et incuber à RT sur un rotateur pendant 1 h.
- Extraire le supernatant sans déranger les perles magnétiques. Enregistrer les phages non liés comme entrée, à utiliser à l'étape 3.5.1.
- Laver les perles 10x à l'aide de 0,05% PBST et 5x en utilisant 1 x PBS. Entre chaque trois lavages les transférer à un nouveau tube pour éviter les phages non spécifiquement liés aux parois du tube.
- Suppression de nanocorps phages
- Des phages liés à l'elute de façon compétitive en ajoutant 450 L du ligand non biotinylated,en utilisant une concentration dans la gamme de micromolaires (p. ex., 10 à 50 M) et en couvant à RT sur un rotateur pendant 30 min. La concentration de ligand séquestrée pour l'élution compétitive des phages liés dépend du KD souhaité du « liant d'ancrage ». Les concentrations de ligands peuvent être relativement élevées dans les rondes de sélection initiales, puis diminuer dans les rondes ultérieures.
- Recueillir supernatant et enregistrer les phages élutés comme sortie, à utiliser à l'étape 3.5.2.
- Titrations d'entrée/sortie et infection
- Pour la titration d'entrée, préparer 10fois dilutions en série dans 1 x PBS jusqu'à 109-plier avec le phage d'entrée de l'étape 3.3.7. Utilisez les dilutions en série de 107à 109 pour faire des infections en transférant 10 phage d'entrée ll de chaque dilution à 70 cellules TG1 ll (OD600 de 0,5). Incuber à 37 oC pendant 45 min, déposer les cellules TG1 infectées sur trois plats de 90 mm 2YT-agar contenant 100 'g/mL d'ampicilline et 2% (wt/vol) de glucose, et couver toute la nuit à 37 oC. À partir des plaques de nuit, l'entrée de phage peut être calculée comme suit :
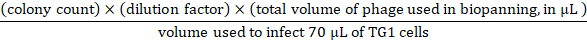
- Pour l'infection de sortie et la titration, transférez les phages élutés de l'étape 3.4.2 à 3 ml des cellules TG1 (OD600 de 0.5). Incuber dans un bain d'eau à 37 oC pendant 45 min. Préparer ensuite 10 fois les dilutions en série en 2YT jusqu'à 103fois, déposer chaque dilution sur des plats de 90 mm 2YT-agar et incuber toute la nuit à 37 oC. À partir des plaques de nuit, la production de phage peut être calculée comme suit :
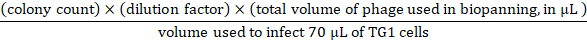
- Divisez les autres cellules TG1 infectées sur trois plaques de 150 mm 2YT-agar contenant 100 ampicilline de 100 g/mL et 2 % (wt/vol) de glucose. Incuber les assiettes pendant la nuit à 37 oC.
- Pour la titration d'entrée, préparer 10fois dilutions en série dans 1 x PBS jusqu'à 109-plier avec le phage d'entrée de l'étape 3.3.7. Utilisez les dilutions en série de 107à 109 pour faire des infections en transférant 10 phage d'entrée ll de chaque dilution à 70 cellules TG1 ll (OD600 de 0,5). Incuber à 37 oC pendant 45 min, déposer les cellules TG1 infectées sur trois plats de 90 mm 2YT-agar contenant 100 'g/mL d'ampicilline et 2% (wt/vol) de glucose, et couver toute la nuit à 37 oC. À partir des plaques de nuit, l'entrée de phage peut être calculée comme suit :
- Amplification et récupération des bibliothèques pour d'autres séries de sélection
- Ajouter 3 ml de 2YT par plaque, gratter avec un grattoir à cellules stériles et recueillir toutes les cellules dans un tube conique de 50 ml. Mélanger les cellules recueillies avec du glycérol stérile (concentration finale de 20 % wt/vol). Mesurer l'OD600 du mélange et faire des aliquots de stock de 3 à 5. Conserver à -80 oC pour un stockage à long terme.
- Pour le sauvetage de phage, diluer le mélange bactérien TG1 contenant du phagemid à l'aide de 25 ml de milieux 2YT complétés par 2 % de glucose et 100 'g/mL d'ampicilline à un OD600 de 0,1. Cellules de culture à 37 oC et 250 tr/min à un OD600 de 0,5.
- Superinfecter les cellules en ajoutant le phage d'aide CM13 à 5 x 109 pfu/mL et incuber à 37 oC et 250 tr/min pendant 45 min. Le phage d'aide CM13 fournit les protéines requises de manteau de phage pour l'assemblage des particules complètes de phage.
- Centrifuger la culture à 8 000 x g pendant 10 min pour enlever le glucose. Resuspendre les cellules à l'aide de 50 ml de 2YT médias complétés par 100 'g/mL ampicilline et 50 'g/mL kanamycin et incuber à 25 oC et 250 tr/min pendant la nuit.
- Centrifugeles les cellules de la culture de la nuit à 9 000 x g, 4 oC pendant 30 min. Transférer le supernatant dans un nouveau tube et précipiter les phages dans le supernatant à l'aide d'une solution PEG/NaCl de volume 1/5 (20 % wt/vol polyéthylène glycol-6 000 et 2,5 M NaCl). Mélanger délicatement et déposer sur la glace pendant 1 h.
- Recueillir les particules de phage par centrifugation à l'aide de 12 000 x g à 4 oC pendant 30 min. Resuspendre les granulés à l'aide de 1 ml de 1 x PBS, et transférer la suspension dans un tube de microcentrifuge. Centrifuger le tube à 20 000 x g et 4 oC pendant 10 min pour éliminer les bactéries résiduelles.
- Transférer le supernatant dans un nouveau tube de microcentrifuge sans déranger la pastille bactérienne. Utilisez une dilution de 1:100 pour mesurer l'absorption à 269 nm et 320 nm. Le nombre total de phages peut être calculé à l'aide de la formule suivante23:
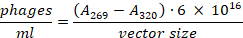
- Entreposez la bibliothèque de phages à 4 oC pour une utilisation à court terme ou à 25 % de glycérol à -80 oC pour un stockage à long terme.
- Répéter les tours de sélection (étapes 3.1-3.6) pour les 3 à 6 tours ou jusqu'à ce que l'enrichissement désiré soit observé (voir la section Résultats). Plaqueet choisir des clones uniques (section 4) afin de caractériser leur affinité et leur spécificité avec le ligand (sections 5 à 7).
4. Isolement de clone unique
- Pour isoler les clones individuels d'une sous-bibliothèque enrichie, préparez 10 fois les dilutions en série des cellules TG1 infectées par le phage (étape 3.5.2). Dilutions en série de plaques sur des plats de 90 mm 2YT-agar contenant 100 'g/mL d'ampicilline et 2% (wt/vol) de glucose et incuber à 37 oC pendant la nuit.
- Dans les assiettes de nuit, choisissez des colonies simples dans 250 oL de supports 2YT complétés par une ampicilline de 100 g/mL par puits dans des plaques stériles de puits profonds et de croissance à 37 oC pendant la nuit.
- À partir des cultures de la nuit, inoculez 10 l à 500 l de supports 2YT frais complétés par une ampicilline de 100 g/mL.
- Cultivez les cellules à un OD600 de 0,5, ajoutez le phage d'aide CM13 à 5 x 109 pfu/mL et incubez à 37 oC et 250 tr/min pendant 45 min.
- Ajouter 500 l de supports 2YT complétés par une ampicilline de 100 g/mL et 50 'g/mL de kanamycine. Incuber à 25 oC et 250 tr/min pendant la nuit.
- Centrifuger les plaques de puits profonds des cultures de nuit à 3 000 x g pendant 10 min. Recueillir le supernatant contenant les particules de phage sans déranger la granule cellulaire.
- Les particules de phage peuvent être utilisées pour ELISA pour déterminer la spécificité des clones sélectionnés au ligand. La biotine ou un homologue structurel de la cible peut être utilisé comme un contrôle négatif.
5. Validation du liant d'ancrage par ELISA
- Enduisez 96 plaques ELISA de puits à l'aide de 100 l de streptavidin de 5 g/mL dans un tampon de revêtement (100 mM de tampon carbonate, pH à 8,6) à 4 oC pendant la nuit.
- Laver les plaques ELISA 3x à l'aide de 0,05 % de PBST et ajouter 100 L de 1 cible biotinylated à la cible. Ajouter 100 L de 1 m de biotine ou d'homologue cible aux puits de contrôle. Incuber à RT pendant 1 h.
- Laver les plaques 5x à l'aide de 0,05 % de PBST et bloquer la liaison non spécifique en ajoutant 300 OL de 1 % de caséine en 1 x PBS. Incuber à RT pendant 1 h.
- Laver les plaques ELISA 3x à l'aide de 0,05%-PBST et ajouter le supernatant de phage purifié. Incuber pour 1 h à RT.
- Laver les plaques ELISA 10x à l'aide de 0,05 % de PBST et ajouter 100 peroxidase de raifort L (HRP)-M13 anticorps protéiques en couches principales (1:10 000 dilution avec 1 x PBS avec 1% de caséine). Incuber à RT pendant 1 h.
- Laver les plaques ELISA 3x à l'aide de 0,05 % de PBST et ajouter un substrat de tétramethylbenzidine (TMB) de 100 ll. Incuber pendant 10 min ou jusqu'à ce qu'un changement de couleur visible soit observé. Arrêtez la réaction en ajoutant 100 oL de 1 M HCl. Lisez la plaque à 450 nm sur un spectrophotomètre.
- Pour l'expression et la purification des protéines, choisissez les clones présentant une grande affinité et spécificité pour la cible (voir Discussion).
6. Expression, purification et biotinylation protéinées
- Comme indiqué précédemment19, sous-clone rapponnable clones sélectionnés de la section 5 et express comme C-terminal Avi-marqué et his-tagged nanobodies.
- Exprimer des nanocorps sélectionnés dans le périplasme des cellules E. coli WK6 (généralement en culture 1 L), libérer par choc osmotique et purifier à l'aide d'une colonne nickel-NTA (voir Tableau des matériaux).
- Échanger tampon avec une colonne de dessalement (1 x PBS avec 5% de glycérol; voir Tableau des matériaux).
- Nanobodies biotinylate à l'aide d'un kit commercial (voir Tableau des matériaux) pour une utilisation ultérieure.
7. Caractérisation du liant d'ancrage par BLI
- Analyser l'affinité et la cinétique de liaison des liants d'ancrage sélectionnés en immobilisant 200 nM de liants d'ancrage biotinylated sur des biocapteurs de streptavidin (voir Tableau des matériaux) avec tampon d'analyse de liaison (1 x PBS (pH - 7,4), 0,05% Tween 20, 0,2% BSA, 3% méthanol).
- Calculer les constantes de dissociation (KD) des interactions liant-ligand d'ancrage par analyse à l'état stable à l'aide d'un logiciel d'analyse de données (voir Tableau des matériaux). Les valeurs KD obtenues vont généralement du micromolaire à un et à deux chiffres.
8. Dépistage du classeur de dimerisation
REMARQUE : Le criblage biopanning des « liants de dimerisation » est semblable à celui des liants d'ancre, excepté deux étapes critiques : 1) les liants de dimerisation sont choisis utilisant un liant d'ancre biotinylated choisi et le complexe de liant-ligand d'ancre pour le négatif et des sélections positives, respectivement. 2) Pendant l'étape d'élution, 100 mM de triéthylamine est utilisé pour élifier des phages sélectionnés positivement qui n'étaient liés qu'au complexe de la cible de liant d'ancre- ligand. La solution de triméthylamine de 100 mM (pH à 11,5) est utilisée pour éliminer les clones positifs en perturbant les interactions protéiques.
- Début de la sélection
- Commencez chaque tour de sélection en inoculant une seule colonie de cellules TG1, fraîchement cultivée sur un support minimal, en 6 mL 2YT à 37 oC et 250 tr/min à un OD600 de 0,5. Incuber les cellules sur la glace.
- Enlèvement de nanocorps sélectionnés négativement
- Préparer le « tube de soustraction » en utilisant 400 L de perles magnétiques recouvertes de streptavidin et suivre l'étape 3.2. Cependant, au lieu de saturer avec de la biotine, ajoutez 5 fois la pleine capacité de liaison calculée à l'aide du liant d'ancrage biotinylated sélectionné et enregistrez les phages non liés à utiliser à l'étape 8.3.3.
- Sélection de nanocorps sélectionnés positivement
- Préparer le « tube de capture » en utilisant la demi-année du volume de perles magnétiques recouvertes de streptavidin utilisé pour le « tube de soustraction » et en suivant les étapes 3.3.2 à 3.3.3. Cependant, au lieu de saturer avec le ligand biotinylated, ajoutez cinq fois la pleine capacité de liaison calculée à l'aide du liant d'ancrage biotinylatedsélectionné.
- Pour former le complexe liant-ligand d'ancrage pour la sélection positive de liant de dimerisation, ajoutez une concentration assez élevée de ligand non biotinylated. Cela permettra à la plupart des liants d'ancrage liés à la streptavidin de former le complexe lié au ligand.
- Suivez les étapes 3.3.3 à 3.3.8, en utilisant les phages non liés tirés du « tube de soustraction ».
- Suppression de nanocorps sélectionnés positivement
- Éluter les phages liés au complexe liant-ligand de l'ancre en ajoutant 450 l de triéthylamine de 100 mM et en couvant à RT sur un rotateur pendant 10 min.
- Recueillir les phages élucés de façon compétitive et suivre les étapes 3.4.1 à 3.4.2.
- D'autres séries de sélection de classeurs de dimerisation
- Suivez les étapes 3.5 et 3.6 pour amplifier et récupérer la bibliothèque afin d'effectuer d'autres tours de sélection. Répétez les rondes de sélection pendant 3 à 6 tours ou jusqu'à ce que l'enrichissement désiré soit observé. Plaqueet choisir des clones uniques (voir à la section 4) afin de caractériser leur affinité et leur spécificité par la cible.
9. Caractérisation du liant de dimerisation par ELISA
- Suivez les étapes de la section 4 pour isoler les clones individuels pour la caractérisation via ELISA.
- Pour tester l'affinité des candidats du liant de dimerisation au complexe liant-ligand de l'ancre, enrober la plaque cible ELISA à l'aide d'un liant d'ancrage biotinylated de 100 nM. Après l'incubation pendant 1 h, ajouter 1 m de la cible ligand pour former le complexe liant-ligand de l'ancre.
- La plaque de commande doit être enduite à l'aide du liant d'ancre biotinylated seul pour filtrer les clones qui peuvent également se lier au liant d'ancrage libre. Ajouter 100 l de liant d'ancre biotinylated de 100 nM et incuber à RT pendant 1 h.
- Suivez les sections 5.3-5.7.
10. Caractérisation du liant de dimerisation par BLI
- L'affinité et la cinétique de liaison des liants de dimerisation pour le liant d'ancrage - complexe de ligand peuvent être analysées en immobilisant les liants de dimerisation biotinylated sur les biocapteurs de streptavidin (SA) avec le tampon d'analyse de liaison et puis analysés avec 1 liant d'ancre pré-équilibrated avec des dilutions sérielles du ligand. Le KD, kon, et koff des interactions peuvent être calculés en utilisant notre méthode signalée19.
Access restricted. Please log in or start a trial to view this content.
Résultats
Nous décrivons la sélection in vitro en deux étapes et la validation des liants d'ancrage et de dimerisation en examinant la bibliothèque combinatoire nanobody avec une diversité supérieure à 109 en utilisant CBD comme cible. Il est important d'évaluer l'enrichissement du phage biopanning au cours des cycles successifs de sélection des liants d'ancrage et de dimerisation. Les résultats typiques d'enrichissement après 4 à 6 tours de sélection, comme le montre la figure 5,
Access restricted. Please log in or start a trial to view this content.
Discussion
Il est essentiel de choisir les concentrations correctes de bibliothèques de phage d'entrée pour différentes séries de biopanning. Nous avons généralement commencé à partir d'une bibliothèque d'entrée de 12101013 particules de phage avec une diversité de '109, permettant '100 à 1.000 copies de chaque clone de phage pour être présenté dans le résultat de traction vers le bas. Si la concentration de phages dans un test contraignant est trop élevée ou faible, la probabilit?...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Un brevet provisoire relatif à ce travail a été déposé par l'Université de Washington.
Remerciements
Ce travail a été soutenu par le University of Washington Innovation Award (à L.G.), une subvention des National Institutes of Health des États-Unis (1R35GM128918 à L.G.), et un fonds de démarrage de l'Université de Washington (à L.G.). H.J. a reçu le soutien d'une bourse de premier cycle de la Washington Research Foundation. K.W. a été soutenu par une bourse de premier cycle de l'Université de Washington Institute for Protein Design.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 1-Step Ultra TMB ELISA substrate solution | Thermo Fisher Scientific | 34029 | |
| Agar | Thermo Fisher Scientific | BP1423-2 | |
| Amicon Ultra-15 Centrifugal Filter unit (3 kDa cutoff) | Millipore | UFC900324 | |
| Ampicillin | Thermo Fisher Scientific | BP1760-25 | |
| Bio-Rad Protein Assay Kit II | Bio-Rad | 5000002 | |
| BirA biotin-protein ligase standard reaction kit | Avidity | BirA500 | |
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A2153-50G | |
| Casein | Sigma-Aldrich | C7078-1KG | |
| CM13 Helper phage | Antibody Design Labs | PH020L | |
| D-(+)-Glucose monohydrate | Alfa Aesar | A11090 | |
| Dynabeads M-280 Streptavidin | Thermo Fisher Scientific | 11205D | |
| DynaMag-2 Magnet | Thermo Fisher Scientific | 12321D | |
| EDTA | Thermo Fisher Scientific | BP120-1 | |
| Fast DNA Ladder | New England Biolabs | N3238S | |
| FastDigest BglI | Thermo Fisher Scientific | FD0074 | |
| Glycerol | Thermo Fisher Scientific | BP229-1 | |
| HiLoad 16/600 Superdex 200 pg | GE Healthcare | 28989335 | |
| HiPrep 26/10 Desalting Column | GE Healthcare | 17508701 | |
| HisTrap-FF-1ml | GE Healthcare | 11000458 | |
| Imidazole | Alfa Aesar | 161-0718 | |
| IPTG | Thermo Fisher Scientific | 34060 | |
| Kanamycin | Thermo Fisher Scientific | BP906-5 | |
| M13 Major Coat Protein Antibody | Santa Cruz Biotechnology | sc-53004 | |
| NaCl | Sigma-Aldrich | S3014-500G | |
| NanoDrop 2000/2000c Spectrophotometers | Thermo Fisher Scientific | ND-2000 | |
| Nunc 96-Well Polypropylene DeepWell Storage Plates | Thermo Fisher Scientific | 260251 | |
| Nunc MaxiSorp | Thermo Fisher Scientific | 44-2404-21 | |
| Octet RED96 | ForteBio | N/A | |
| pADL-23c Phagemid Vector | Antibody Design Labs | PD0111 | |
| PEG-6000 | Sigma-Aldrich | 81260-1KG | |
| Platinum SuperFi DNA Polymerase | Invitrogen | 12351010 | |
| PureLink PCR Purification Kit | Thermo Fisher Scientific | K310001 | |
| QIAprep Spin M13 Kit | Qiagen | 27704 | |
| Recovery Medium | Lucigen | 80026-1 | |
| SpectraMax Plus 384 | Molecular Devices | N/A | |
| Sucrose | Sigma-Aldrich | S0389-1KG | |
| Super Streptavidin (SSA) Biosensors | ForteBio | 18-5057 | |
| Superdex 75 increase 10/300 GL Column | GE Healthcare | 28-9909-44 | |
| T4 DNA Ligase | Thermo Fisher Scientific | 15224-025 | |
| TG1 Electrocompetent Cells | Lucigen | 60502-1 | |
| Triethylamine | Sigma-Aldrich | 471283-100mL | |
| Trizma Base | Sigma-Aldrich | T1503 | |
| Tryptone | Thermo Fisher Scientific | BP9726-5 | |
| Tween 20 | Thermo Fisher Scientific | BP337-500 | |
| Yeast Extract | Thermo Fisher Scientific | BP1422-2 | |
| Zeba Spin Desalting Column | Thermo Fisher Scientific | 89882 |
Références
- Stanton, B. Z., Chory, E. J., Crabtree, G. R. Chemically induced proximity in biology and medicine. Science. 359 (6380), (2018).
- Wu, C. Y., Roybal, K. T., Puchner, E. M., Onuffer, J., Lim, W. A. Remote control of therapeutic T cells through a small molecule-gated chimeric receptor. Science. 350 (6258), (2015).
- Straathof, K. C., et al. An inducible caspase 9 safety switch for T-cell therapy. Blood. 105 (11), 4247-4254 (2005).
- Di Stasi, A., et al. Inducible apoptosis as a safety switch for adoptive cell therapy. The New England Journal of Medicine. 365 (18), 1673-1683 (2011).
- Mank, M., et al. A FRET-based calcium biosensor with fast signal kinetics and high fluorescence change. Biophysical Journal. 90 (5), 1790-1796 (2006).
- Nagai, T., Sawano, A., Park, E. S., Miyawaki, A. Circularly permuted green fluorescent proteins engineered to sense Ca2+. Proceedings of the National Academy of Sciences of the United States of America. 98 (6), 3197-3202 (2001).
- Hunter, M. M., Margolies, M. N., Ju, A., Haber, E. High-affinity monoclonal antibodies to the cardiac glycoside, digoxin. Journal of Immunology. 129 (3), 1165-1172 (1982).
- Bradbury, A. R. M., Sidhu, S., Dubel, S., McCafferty, J. Beyond natural antibodies: the power of in vitro display technologies. Nature Biotechnology. 29 (3), 245-254 (2011).
- Chen, G., et al. Isolation of high-affinity ligand-binding proteins by periplasmic expression with cytometric screening (PECS). Nature. Biotechnology. 19 (6), 537-542 (2001).
- Tinberg, C. E., et al. Computational design of ligand-binding proteins with high affinity and selectivity. Nature. 501 (7466), 212-216 (2013).
- Spencer, D. M., Wandless, T. J., Schreiber, S. L., Crabtree, G. R. Controlling signal transduction with synthetic ligands. Science. 262 (5136), 1019-1024 (1993).
- Ho, S. N., Biggar, S. R., Spencer, D. M., Schreiber, S. L., Crabtree, G. R. Dimeric ligands define a role for transcriptional activation domains in reinitiation. Nature. 382 (6594), 822-826 (1996).
- Belshaw, P. J., Ho, S. N., Crabtree, G. R., Schreiber, S. L. Controlling protein association and subcellular localization with a synthetic ligand that induces heterodimerization of proteins. Proceedings of the National Academy of Sciences of the United States of America. 93 (10), 4604-4607 (1996).
- Farrar, M. A., AlberolaIla, J., Perlmutter, R. M. Activation of the Raf-1 kinase cascade by coumermycin-induced dimerization. Nature. 383 (6596), 178-181 (1996).
- Erhart, D., et al. Chemical Development of Intracellular Protein Heterodimerizers. Chemistry & Biology. 20 (4), 549-557 (2013).
- Ballister, E. R., Aonbangkhen, C., Mayo, A. M., Lampson, M. A., Chenoweth, D. M. Localized light-induced protein dimerization in living cells using a photocaged dimerizer. Nature Communications. 17 (5), 5475(2014).
- Hill, Z. B., Martinko, A. J., Nguyen, D. P., Wells, J. A. Human antibody-based chemically induced dimerizers for cell therapeutic applications. Nature Chemical Biology. 14 (2), 112-117 (2018).
- Foight, G. W., et al. Multi-input chemical control of protein dimerization for programming graded cellular responses. Nature Biotechnology. 37 (10), 1209-1216 (2019).
- Kang, S., et al. COMBINES-CID: An efficient method for de novo engineering of highly specific chemically induced protein dimerization systems. Journal of the American Chemical Society. 141 (28), 10948-10952 (2019).
- Muyldermans, S. Nanobodies: natural single-domain antibodies. Annual Review of Biochemistry. 82, 775-797 (2013).
- Fanning, S. W., Horn, J. R. An anti-hapten camelid antibody reveals a cryptic binding site with significant energetic contributions from a nonhypervariable loop. Protein Science. 20 (7), 1196-1207 (2011).
- Zavrtanik, U., Luken, J., Loris, R., Lah, J., Hadzi, S. Structural basis of epitope recognition by heavy-chain camelid antibodies. Journal of Molecular Biology. 430 (21), 4369-4386 (2018).
- Denhardt, D. T., Dressler, D., Ray, D. S. The Single-Stranded DNA Phages. , 605-625 (1978).
- Virnekas, B., et al. Trinucleotide phosphoramidites: ideal reagents for the synthesis of mixed oligonucleotides for random mutagenesis. Nucleic Acids Research. 22 (25), 5600-5607 (1994).
- Gu, L., et al. Multiplex single-molecule interaction profiling of DNA-barcoded proteins. Nature. 515 (7528), 554-557 (2014).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon