É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Criando sistemas de dimerização de proteínas quimicamente induzidas altamente específicos pela seleção de fagos de uma biblioteca de anticorpos de domínio único combinatorial
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Criar sistemas quimicamente induzidos da dimerização da proteína com afinidade e especificidade desejadas para toda a ligand pequena da molécula dada teria muitas aplicações biológicas da detecção e do atuação. Aqui, descrevemos um método eficiente e generalizável para a engenharia de novo de sistemas de dimerização quimicamente induzidos através da seleção manada de uma biblioteca de anticorpos combinatorial de domínio combinatório exibida por fago.
Resumo
Eventos de imersão de proteínas que ocorrem apenas na presença de um ligand de molécula pequena permitem o desenvolvimento de biossensores de pequenas moléculas para a dissecação e manipulação de vias biológicas. Atualmente, apenas um número limitado de sistemas de dimerização quimicamente induzida (CID) existe e engenharia de novos com sensibilidade desejada e seletividade para ligantes específicos de pequenas moléculas continua a ser um desafio no campo da engenharia de proteínas. Nós aqui descrevemos um método de triagem de alta produtividade, combin binders-enabled seleição de CID (COMBINES-CID), para a engenharia de novo de sistemas CID aplicável a uma grande variedade de ligantes. Este método usa a seleção em duas etapas de uma biblioteca de nanobody combinatória exibida por fago para obter 1) "pastas de âncora" que primeiro se ligam a um ligand de interesse e, em seguida, 2) "pastas de imersão" que só se ligam a complexos de ligand de pasta de ancoragem. Para selecionar pastas de âncora, uma biblioteca combinatória de mais de 109 nanobodies randomizados de complementaridade (CDR) é rastreada com um ligande biotinylated e os hits são validados com o ligand não rotulado pela interferometria de camada biológica (BLI). Para obter pastas de imersão, a biblioteca nanobody é rastreada com complexos de ligand escora como alvos para triagem positiva e as pastas de âncora sem limites para triagem negativa. Combines-CID é amplamente aplicável para selecionar pastas CID com outras imunoglobulina, não imunoglobulina, ou andaimes computacionalmente concebidos para criar biossensores para a detecção in vitro e in vivo de drogas, metabólitos, moléculas de sinalização, etc.
Introdução
Os sistemas CID, nos quais duas proteínas se dinamizarapenas na presença de um ligand de molécula pequena (Figura 1),oferecem ferramentas versáteis para dissecar e manipular vias metabólicas, sinalização e outras vias biológicas1. Eles demonstraram o potencial na atuação biológica, como a ativação de células T controladas por drogas2 e a apoptose3,4,para melhorar a segurança e eficácia da terapia de células T adotivas. Além disso, eles fornecem uma nova metodologia para a detecção in vivo ou in vitro de alvos de pequenas moléculas. Por exemplo, as proteínas CID podem ser geneticamente fundidas com sistemas de repórter de fluorescência (por exemplo, transferência de energia de ressonância fluorescência (FRET)5 e proteínas fluorescentes permutadas circularmente)6 para medições in vivo em tempo real, ou servir como reagentes de afinidade para ensaios imunosordobrados ligados a enzimas sanduíches (ELISA).
Apesar de suas amplas aplicações, a criação de novos sistemas CID que podem ser controlados por um dado ligand de pequenas moléculas tem grandes desafios. Métodos estabelecidos de engenharia de pasta de proteínas, incluindo imunização animal7,seleção in vitro8,9,e design computacional deproteína10 podem gerar proteínas de ligação que funcionam através de interações binárias de proteína-ligand. No entanto, esses métodos têm dificuldades em criar um complexo cid ternary induzido por ligand. Alguns métodos criam CID ligando quimicamente dois ligantes que se ligam independentemente às mesmas ou diferentes proteínas11,12,13,14,15,16 ou dependem da seleção de proteínas de pasta, como anticorpos que visam complexos pré-existentes de pequenas moléculas e proteínas17,18,e, portanto, têm uma escolha limitada de ligantes.
Recentemente desenvolvemos um método de bin dersbin ders-e nabled de CID (COMBINES-CID) método para engenharia de novo de sistemas CID19. Este método pode obter a alta especificidade da dimerização induzida por ligand (por exemplo, uma constante de dissociação da pasta de escorização âncora, KD (sem ligand)/KD (com ligand) > 1.000). A especificidade da imersão é alcançada usando pastas de âncora com locais de ligação flexíveis que podem introduzir mudanças conformais sobre a ligação, fornecendo uma base para a seleção de pastas conformaçãoseletivas que reconhecem apenas pastas de âncora ligadas. Demonstramos uma prova de princípio criando heterodimers induzidos por canabidiol (CBD) de nanobodies, um fragmento de anticorpo funcional de 12-15 kDa de camelid compreendendo um andaime universal e três loops cdr flexíveis (Figura 2)20, que podem formar um bolso de ligação com tamanhos adaptáveis para epitopes de pequenas moléculas21,22. Notavelmente, a seleção in vitro de uma biblioteca de proteína combinatória deve ser rentável e generalizável para a engenharia CID porque a mesma biblioteca de alta qualidade pode ser aplicada a diferentes ligantes.
Neste protocolo e vídeo, nos concentramos em descrever a seleção de duas etapas in vitro e validação de âncora(Figura 3A)e pastas de imersão(Figura 3B),selecionando a biblioteca nanobody combinatória com uma diversidade superior a 109 usando CBD como alvo, mas o protocolo deve ser aplicável a outras bibliotecas de proteínas ou alvos de pequenas moléculas. A triagem de pastas CID geralmente leva 6-10 semanas(Figura 4).
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Construção da biblioteca
- Use uma biblioteca de anticorpos de domínio único combinatório sintético com uma diversidade de ~1.23-7.14 x 109, como descrito anteriormente19. Embora este protocolo não inclua a construção de bibliotecas, ele pode ser aplicado a outras bibliotecas de pasta combinatória.
2. Biotinylation do alvo ou do ligand do ligand
- Biotinylate o ligand selecionado, por exemplo, CBD e tetrahidrocanabinol (THC)19, através de várias estratégias de síntese química, dependendo dos locais de biotinylação adequados de um alvo.
3. Escou-se a seleção da pasta da escor
- Início da seleção
- Comece cada rodada de seleção inoculando uma única colônia de células TG1, cultivada na hora em 6 mL de 2YT a 37 °C e 250 revoluções por minuto (rpm) para uma absorção de 600 nm (OD600) de ~ 0,5. Incubar as células no gelo para o uso na etapa 3.5.1.
- Seleção negativa com contas de streptavidin biotina-bound
- Prepare as "contas de seleção negativa" lavando 300 μL de contas magnéticas revestidas de streptavidin usando um rack de separação magnética, 3x com sisina tampão de fosfato de 0,05% com tampão de pré-tween (PBST, 1 x PBS com 0,05% vol/vol Tween 20%) e 2x com 1 x PBS.
- Resuspenda as contas com 1 mL de 1% de casenina em 1 x PBS (pH = 7,4), e saqueie as contas adicionando 5x a capacidade de ligação relatada usando biotina. Incubar à temperatura ambiente (RT) em um rotador por 1 h.
- Lave as contas 5x usando 0,05% PBST e 3x usando 1 x PBS, para um total de oito lavares.
- Adicionar ~ 1013 partículas de fago em 1% casein/1% BSA em 1 x PBS (pH = 7,4) e incubar em RT em um rotador por 1 h.
- Após a incubação, recolher o supernatant para ser usado na etapa 3.3.6.
- Seleção positiva com contas de streptavidin biotinylated ligand-bound
- Prepare as "contas de seleção positivas" usando 1/2 o volume das contas usadas para as "contas de seleção negativas" seguindo os passos 3.2.1.
- Resuspenda as contas com 1 mL 1% casein em 1 × PBS, pH 7.4 e saturar as contas, adicionando 5x a capacidade de ligação total calculada com base no manual usando o ligand biotinylated de escolha. Incubar em RT em um rotator para 1 h.
- Lave as contas 5x usando 0,05% PBST e 3x usando 1 x PBS, para um total de oito lavares.
- Bloqueie as contas com 1 mL de 1% de caseina/1% BSA em 1 x PBS (pH = 7,4) e incubar no RT em um rotador por 1 h para evitar a ligação inespecífica entre os fagos e as contas magnéticas revestidas de streptavidina.
- Lave as contas magnéticas revestidas de streptavidin 3x usando 0,05% PBST e uma vez usando 1 x PBS, para um total de quatro lavações.
- Resuspenda as contas magnéticas revestidas de streptavidina usando os fagos não vinculados retirados do passo 3.2.5 e incubam na RT em um rotador por 1 h.
- Extraia o supernatant sem perturbar os grânulos magnéticos. Salve os fagos não vinculados como entrada, para ser usado na etapa 3.5.1.
- Lave as contas 10x usando 0,05% PBST e 5x usando 1 x PBS. Entre cada três lavagens transferi-los para um novo tubo para evitar fagos inespecificamente ligados às paredes do tubo.
- Lubrificação de nanobodies phage-indicados
- Os fagos vinculados competitivamente elute, adicionando 450 μL do ligand não-biotinylado, usando uma concentração na faixa micromolar (por exemplo, 10-50 μM) e incubação em RT em um rotador por 30 min. A concentração de ligand selecionada para a lusão competitiva de fagos encadernados depende do KD desejado da "pasta de âncora". As concentrações de ligand podem ser relativamente elevadas em círculos iniciais da seleção e diminuídas então em uns círculos mais atrasados.
- Coletar supernatant e salvar os fagos eluted como saída, para ser usado na etapa 3.5.2.
- Instimas/saída de titrations e infecção
- Para a titulação de entrada, prepare diluições em série 10x em 1 x PBS até 109- dobre com o fago de entrada do passo 3.3.7. Use as 107-109 diluições em série para fazer infecções transferindo 10 μL phage de entrada de cada diluição para 70 μL Células TG1 (OD600 de ~0,5). Incubar a 37 °C por 45 min, placa as células TG1 infectadas em três 90 mm 2YT-ágar pratos contendo 100 μg/mL ampicilina e 2% (wt /vol) glicose, e incubar durante a noite em 37 °C. A partir das placas durante a noite, a entrada de fagos pode ser calculada da seguinte forma:
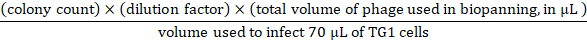
- Para infecção e titration da saída, transfira os fagos eluted da etapa 3.4.2 a 3 mL de pilhas TG1 (OD600 de ~0.5). Incubar em um banho de água em 37 °C por 45 min. Em seguida, prepare diluições em série 10x em 2YT até 103vezes, placa de cada diluição em 90 mm 2YT-ágar pratos, e incubar durante a noite em 37 °C. A partir das placas durante a noite, a saída do fago pode ser calculada da seguinte forma:
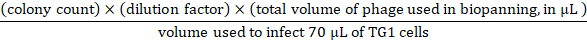
- Divida as células TG1 infectadas restantes em três placas de 2YT-ágar de 150 mm contendo 100 μg/mL ampicilina e 2% (wt/vol) glicose. Incubate placas durante a noite em 37 °C.
- Para a titulação de entrada, prepare diluições em série 10x em 1 x PBS até 109- dobre com o fago de entrada do passo 3.3.7. Use as 107-109 diluições em série para fazer infecções transferindo 10 μL phage de entrada de cada diluição para 70 μL Células TG1 (OD600 de ~0,5). Incubar a 37 °C por 45 min, placa as células TG1 infectadas em três 90 mm 2YT-ágar pratos contendo 100 μg/mL ampicilina e 2% (wt /vol) glicose, e incubar durante a noite em 37 °C. A partir das placas durante a noite, a entrada de fagos pode ser calculada da seguinte forma:
- Amplificação e recuperação da biblioteca para novas rodadas de seleção
- Adicione 3 mL de 2YT por placa, raspe com um raspador de células estéreis e colete todas as células em um tubo cônico de 50 mL. Misture as células coletadas com glicerol estéril (20% wt/vol concentração final). Medir o OD600 da mistura e fazer 3-5 alíquotas de ações. Guarde a -80°C para armazenamento a longo prazo.
- Para resgate de fagos, diluir o phagemid contendo mistura bacteriana TG1 usando 25 mL de mídia 2YT complementado com glicose de 2% e 100 μg/mL ampicilina para um OD600 de ~ 0,1. Células culturais a 37 °C e 250 rpm para um OD600 de ~0,5.
- Superinfectar as células, adicionando phage ajudante CM13 em 5 x 109 pfu/mL e incubar em 37 °C e 250 rpm para 45 min. O fago do ajudante CM13 fornece proteínas exigidas do revestimento do fago para o conjunto de partículas completas do fago.
- Centrífuga a cultura em 8.000 x g por 10 min para remover a glicose. Resuspenda as células usando 50 mL de mídia 2YT complementadas com 100 μg/mL ampicilina e 50 μg/mL kanamicina e incubada a 25 °C e 250 rpm durante a noite.
- Centrífuga as células da cultura durante a noite em 9.000 x g, 4 °C para 30 min. Transferência supernatant para um novo tubo e precipitar fagos no supernatant usando 1/5 volume PEG / Solução NaCl (20% wt / vol polietileno glicol-6.000 e 2,5 M NaCl). Misture suavemente e coloque no gelo por 1 h.
- Colete partículas de fago por centrífuga usando 12.000 x g a 4 °C por 30 min. Resuspende as pelotas usando 1 mL de 1 x PBS e transfira a suspensão para um tubo de microcentrífuga. Centrífuga do tubo a 20.000 x g e 4 °C por 10 min para remover bactérias residuais.
- Transfira o supernatant a um tubo novo do microcentrífuga sem perturbar a pelota bacteriana. Use uma diluição de 1:100 para medir a absorção em 269 nm e 320 nm. O número total de fagos pode ser calculado usando a seguinte fórmula23:
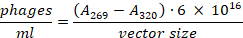
- Armazenar biblioteca de fagos a 4 °C para uso a curto prazo ou com 25% glicerol a -80 °C para armazenamento a longo prazo.
- Repetir rodadas de seleção (passos 3,1-3,6) para 3-6 rodadas ou até que o enriquecimento desejado é observado (consulte a seção resultados). Placa e escolher clones individuais (seção 4), a fim de caracterizar a sua afinidade e especificidade para o ligante (seções 5-7).
4. Isolamento de clones únicos
- Para isolar clones individuais de uma subbiblioteca enriquecida, prepare diluições em série 10x das células TG1 infectadas por fago (passo 3.5.2). Diluições em série de placas em pratos de 90 mm 2YT-ágar contendo 100 μg/mL ampicilina e 2% (wt/vol) glicose e incubação a 37 °C durante a noite.
- A partir das placas durante a noite, escolha colônias individuais em 250 μL de mídia 2YT complementada com 100 μg/mL ampicilina por poço em placas estéreis de poços profundos e cresça a 37 °C durante a noite.
- Das culturas durante a noite, inocular 10 μL em 500 μL de mídia 2YT fresco complementado com 100 μg/mL ampicilina.
- Crescer as células para um OD600 de ~ 0,5, adicionar phage ajudante CM13 em 5 x 109 pfu/mL e incubar em 37 °C e 250 rpm para 45 min.
- Adicione 500 μL de 2YT mídia complementada com 100 μg/mL ampicilina e 50 μg/mL kanamicina. Incubar a 25 °C e 250 rpm durante a noite.
- Centrífugaas as placas de poço profundo das culturas durante a noite em 3.000 x g por 10 min. Coletar o supernatant contendo as partículas de fago sem perturbar a pelota celular.
- As partículas de fago podem ser usadas para elisa para determinar a especificidade dos clones selecionados para o ligante. Biotina ou homolog estrutural do alvo pode ser usado como um controle negativo.
5. Validação da pasta da escora por ELISA
- Casaco 96 placas ELISA bem usando 100 μL de 5 μg/mL streptavidin em tampão de revestimento (tampão de carbonato de 100 mM, pH = 8,6) a 4 °C durante a noite.
- Lave as placas ELISA 3x usando 0,05% PBST e adicione 100 μL de 1 μM alvo biotinylated para os poços-alvo. Adicione 100 μL de 1 μM biotina ou homologe de destino aos poços de controle. Incubar em RT por 1 h.
- Lave as placas 5x usando 0,05% PBST e bloqueie a ligação inespecífica adicionando 300 μL de 1% de casenina em 1 x PBS. Incubar em RT por 1 h.
- Lave as placas ELISA 3x usando 0,05%-PBST e adicione o supernatant fago purificado. Incubar por 1 h em RT.
- Lave as placas ELISA 10x usando 0,05% PBST e adicione 100 μL peroxidase de rábano (HRP)-M13 anticorpo de proteína de revestimento principal (1:10,000 diluição com 1 x PBS com 1% caseina). Incubar em RT por 1 h.
- Lave as placas ELISA 3x usando 0,05% PBST e adicione 100 μL tetramethylbenzidina (TMB) substrato. Incubar por 10 min ou até que uma mudança de cor visível é observada. Pare a reação adicionando 100 μL de 1 M HCl. Leia a placa em 450 nm em um espectrômetro.
- Para expressão e purificação de proteínas, escolha os clones que mostram alta afinidade e especificidade para o alvo (ver Discussão).
6. Expressão, purificação e biotinyminação proteica
- Como relatado anteriormente19, clones subclone selecionados da seção 5 e expresso como C-terminal Avi-marcados e seus nanobodies marcados.
- Expresse nanobodies selecionados no periplasm de pilhas de E. coli WK6 (tipicamente na cultura de 1 L), liberação pelo choque osmótico, e purify usando uma coluna do níquel-NTA (veja tabela dos materiais).
- Buffer de câmbio com uma coluna de salga (1 x PBS com 5% glicerol; ver Tabela de Materiais).
- Nanobodies biotinylate usando um jogo comercial (veja a tabela dos materiais)para um uso mais adicional.
7. Caracterização da pasta da escora por BLI
- Analise a afinidade e a cinética vinculantes dos pastas de âncora selecionados imobilizando pastas de âncora biotinylated de 200 nM em biossensores de streptavidina (ver Tabela de Materiais)com buffer de ensaio vinculativo (1 x PBS (pH = 7,4), 0,05% Tween 20, 0,2% BSA, 3% metanol).
- Calcule as constantes de dissociação(KD)de interações de ancoragem binder-ligand por análise de estado estável usando software de análise de dados (ver Tabela de Materiais). Os valores obtidos de KD variam tipicamente do micromolar de um a dois dígitos.
8. Triagem de pasta de imersão
NOTA: A triagem biopanning de "pastas de imersão" é semelhante à de pastas âncora, com exceção de dois passos críticos: 1) Pastas de imersão são selecionados usando um fichário âncora biotinylated selecionados e o complexo de pasta-ligand âncora para o negativo e seleções positivas, respectivamente. 2) Durante a etapa de elução, 100 mM trietilamina é usado para elute fagos positivamente selecionados que só foram vinculados ao complexo alvo âncora binder-ligand. A solução de trimetilamina de 100 mM (pH = 11,5) é usada para elute clones positivos, interrompendo as interações proteicas.
- Início da seleção
- Comece cada rodada de seleção inoculando uma única colônia de células TG1, cultivada na hora em uma mídia mínima, em 6 mL 2YT a 37 °C e 250 rpm para um OD600 de ~ 0,5. Incubate células no gelo.
- Remoção de nanobodies negativamente selecionados
- Prepare o "tubo de subtração" usando 400 μL de contas magnéticas revestidas de streptavidin a seguir o passo 3.2. No entanto, em vez de saturar com biotina, adicione 5x a capacidade de ligação total calculada usando o fichário de âncora biotinylated selecionado e salve os fagos não vinculados para serem usados na etapa 8.3.3.
- Seleção de nanobodies positivamente selecionados
- Prepare o "tubo de captura" usando 1/2 o volume de contas magnéticas revestidas de streptavidin usado para o "tubo de subtração" e seguindo os passos 3,3,2 a 3,3,3. No entanto, em vez de saturar com o ligand biotinylated, adicione cinco vezes a capacidade de ligação total calculada usando o fichário de âncora biotinylatedselecionado.
- Para formar o complexo de ligand escora para a seleção positiva da pasta de imersão, adicione uma concentração alta o suficiente de ligand não-biotinylated. Isso permitirá que a maioria dos ancorativos ligados à streptavidin a forma do complexo ligado à ligand.
- Siga os passos 3.3.3 a 3.3.8, usando os fagos não vinculados retirados do "tubo de subtração".
- Lubrificação de nanobodies positivamente selecionados
- Elute os fagos ligados ao complexo de ligand escora, adicionando 450 μL de trietilamina de 100 mM, e incubando em RT em um rotador por 10 min.
- Colete os fagos competitivos e siga os passos 3,4,1 a 3,4,2.
- Outras rodadas de seleção de pasta de imersão
- Siga os passos 3,5 e 3,6 para amplificar e recuperar a biblioteca, a fim de realizar novas rodadas de seleção. Repita rodadas de seleção para 3-6 rodadas ou até que o enriquecimento desejado seja observado. Placa e escolher clones individuais (referem-se à seção 4), a fim de caracterizar a sua afinidade e especificidade para o alvo.
9. Caracterização da pasta de imersão por ELISA
- Siga os passos na seção 4 para isolar clones individuais para caracterização via ELISA.
- Para testar a afinidade de candidatos à pasta de imersão ao complexo de ligand de ancoragem, cubra a placa-alvo ELISA usando 100 μL de 100 nM ficglateta de ancoragem. Após a incubação para 1 h, adicione 1 μM do alvo do ligand para dar forma ao complexo do binder-ligand da escora.
- A placa de controle deve ser revestida usando a pasta de âncora biotinylated sozinho para selecionar os clones que podem igualmente ligar à pasta livre da escora. Adicione 100 μL de 100 nM fichário âncora biotinylated e incubar em RT por 1 h.
- Siga as seções 5,3-5,7.
10. Caracterização da pasta de imersão por BLI
- A afinidade e a cinética vinculante dos fichários de imersão para o complexo de ligand de ancoragem podem ser analisados pela imobilização de fichálatas de imersão biotinylated em biossensores de streptavidin (SA) com o buffer de ensaio de ligação e, em seguida, ensaiocom 1 μM ancoragem binder pré-equilibrado com diluições em série do ligand. O KD, kon,e kfora das interações podem ser calculados usando o nosso método relatado19.
Access restricted. Please log in or start a trial to view this content.
Resultados
Descrevemos a seleção in vitro em duas etapas e a validação de pastas de âncora e imersão, selecionando a biblioteca de nanobody combinatória com uma diversidade superior a 109 usando cbd como alvo. Avaliar o enriquecimento da biopanning do phage durante os círculos sucessivos da seleção para ligações da escora e do dimerization é importante. Resultados típicos de enriquecimento após 4-6 rodadas de seleção, como mostrado na Figura 5 são uma boa indicação de que...
Access restricted. Please log in or start a trial to view this content.
Discussão
É fundamental escolher as concentrações corretas de bibliotecas de fagos de entrada para diferentes rodadas de biopanning. Nós normalmente começou a partir de uma biblioteca de entrada de ~ 1012-1013 partículas de fago com uma diversidade >109, permitindo ~ 100-1.000 cópias de cada clone fago para ser apresentado no ensaio pull-down. Se a concentração de fagos em um ensaio vinculativo for muito alta ou baixa, a probabilidade de ligação inespecífica ou perda de clones positivos...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Uma patente provisória relacionada a este trabalho foi arquivada pela Universidade de Washington.
Agradecimentos
Este trabalho foi apoiado pelo Prêmio de Inovação da Universidade de Washington (para a L.G.), uma doação dos Institutos Nacionais de Saúde dos EUA (1R35GM128918 para a L.G.), e um fundo de startups da Universidade de Washington (para l.G.). H.J. foi apoiado por uma bolsa de graduação da Washington Research Foundation. K.W. foi apoiado por uma bolsa de graduação do Instituto de Design de Proteínas da Universidade de Washington.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| 1-Step Ultra TMB ELISA substrate solution | Thermo Fisher Scientific | 34029 | |
| Agar | Thermo Fisher Scientific | BP1423-2 | |
| Amicon Ultra-15 Centrifugal Filter unit (3 kDa cutoff) | Millipore | UFC900324 | |
| Ampicillin | Thermo Fisher Scientific | BP1760-25 | |
| Bio-Rad Protein Assay Kit II | Bio-Rad | 5000002 | |
| BirA biotin-protein ligase standard reaction kit | Avidity | BirA500 | |
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A2153-50G | |
| Casein | Sigma-Aldrich | C7078-1KG | |
| CM13 Helper phage | Antibody Design Labs | PH020L | |
| D-(+)-Glucose monohydrate | Alfa Aesar | A11090 | |
| Dynabeads M-280 Streptavidin | Thermo Fisher Scientific | 11205D | |
| DynaMag-2 Magnet | Thermo Fisher Scientific | 12321D | |
| EDTA | Thermo Fisher Scientific | BP120-1 | |
| Fast DNA Ladder | New England Biolabs | N3238S | |
| FastDigest BglI | Thermo Fisher Scientific | FD0074 | |
| Glycerol | Thermo Fisher Scientific | BP229-1 | |
| HiLoad 16/600 Superdex 200 pg | GE Healthcare | 28989335 | |
| HiPrep 26/10 Desalting Column | GE Healthcare | 17508701 | |
| HisTrap-FF-1ml | GE Healthcare | 11000458 | |
| Imidazole | Alfa Aesar | 161-0718 | |
| IPTG | Thermo Fisher Scientific | 34060 | |
| Kanamycin | Thermo Fisher Scientific | BP906-5 | |
| M13 Major Coat Protein Antibody | Santa Cruz Biotechnology | sc-53004 | |
| NaCl | Sigma-Aldrich | S3014-500G | |
| NanoDrop 2000/2000c Spectrophotometers | Thermo Fisher Scientific | ND-2000 | |
| Nunc 96-Well Polypropylene DeepWell Storage Plates | Thermo Fisher Scientific | 260251 | |
| Nunc MaxiSorp | Thermo Fisher Scientific | 44-2404-21 | |
| Octet RED96 | ForteBio | N/A | |
| pADL-23c Phagemid Vector | Antibody Design Labs | PD0111 | |
| PEG-6000 | Sigma-Aldrich | 81260-1KG | |
| Platinum SuperFi DNA Polymerase | Invitrogen | 12351010 | |
| PureLink PCR Purification Kit | Thermo Fisher Scientific | K310001 | |
| QIAprep Spin M13 Kit | Qiagen | 27704 | |
| Recovery Medium | Lucigen | 80026-1 | |
| SpectraMax Plus 384 | Molecular Devices | N/A | |
| Sucrose | Sigma-Aldrich | S0389-1KG | |
| Super Streptavidin (SSA) Biosensors | ForteBio | 18-5057 | |
| Superdex 75 increase 10/300 GL Column | GE Healthcare | 28-9909-44 | |
| T4 DNA Ligase | Thermo Fisher Scientific | 15224-025 | |
| TG1 Electrocompetent Cells | Lucigen | 60502-1 | |
| Triethylamine | Sigma-Aldrich | 471283-100mL | |
| Trizma Base | Sigma-Aldrich | T1503 | |
| Tryptone | Thermo Fisher Scientific | BP9726-5 | |
| Tween 20 | Thermo Fisher Scientific | BP337-500 | |
| Yeast Extract | Thermo Fisher Scientific | BP1422-2 | |
| Zeba Spin Desalting Column | Thermo Fisher Scientific | 89882 |
Referências
- Stanton, B. Z., Chory, E. J., Crabtree, G. R. Chemically induced proximity in biology and medicine. Science. 359 (6380), (2018).
- Wu, C. Y., Roybal, K. T., Puchner, E. M., Onuffer, J., Lim, W. A. Remote control of therapeutic T cells through a small molecule-gated chimeric receptor. Science. 350 (6258), (2015).
- Straathof, K. C., et al. An inducible caspase 9 safety switch for T-cell therapy. Blood. 105 (11), 4247-4254 (2005).
- Di Stasi, A., et al. Inducible apoptosis as a safety switch for adoptive cell therapy. The New England Journal of Medicine. 365 (18), 1673-1683 (2011).
- Mank, M., et al. A FRET-based calcium biosensor with fast signal kinetics and high fluorescence change. Biophysical Journal. 90 (5), 1790-1796 (2006).
- Nagai, T., Sawano, A., Park, E. S., Miyawaki, A. Circularly permuted green fluorescent proteins engineered to sense Ca2+. Proceedings of the National Academy of Sciences of the United States of America. 98 (6), 3197-3202 (2001).
- Hunter, M. M., Margolies, M. N., Ju, A., Haber, E. High-affinity monoclonal antibodies to the cardiac glycoside, digoxin. Journal of Immunology. 129 (3), 1165-1172 (1982).
- Bradbury, A. R. M., Sidhu, S., Dubel, S., McCafferty, J. Beyond natural antibodies: the power of in vitro display technologies. Nature Biotechnology. 29 (3), 245-254 (2011).
- Chen, G., et al. Isolation of high-affinity ligand-binding proteins by periplasmic expression with cytometric screening (PECS). Nature. Biotechnology. 19 (6), 537-542 (2001).
- Tinberg, C. E., et al. Computational design of ligand-binding proteins with high affinity and selectivity. Nature. 501 (7466), 212-216 (2013).
- Spencer, D. M., Wandless, T. J., Schreiber, S. L., Crabtree, G. R. Controlling signal transduction with synthetic ligands. Science. 262 (5136), 1019-1024 (1993).
- Ho, S. N., Biggar, S. R., Spencer, D. M., Schreiber, S. L., Crabtree, G. R. Dimeric ligands define a role for transcriptional activation domains in reinitiation. Nature. 382 (6594), 822-826 (1996).
- Belshaw, P. J., Ho, S. N., Crabtree, G. R., Schreiber, S. L. Controlling protein association and subcellular localization with a synthetic ligand that induces heterodimerization of proteins. Proceedings of the National Academy of Sciences of the United States of America. 93 (10), 4604-4607 (1996).
- Farrar, M. A., AlberolaIla, J., Perlmutter, R. M. Activation of the Raf-1 kinase cascade by coumermycin-induced dimerization. Nature. 383 (6596), 178-181 (1996).
- Erhart, D., et al. Chemical Development of Intracellular Protein Heterodimerizers. Chemistry & Biology. 20 (4), 549-557 (2013).
- Ballister, E. R., Aonbangkhen, C., Mayo, A. M., Lampson, M. A., Chenoweth, D. M. Localized light-induced protein dimerization in living cells using a photocaged dimerizer. Nature Communications. 17 (5), 5475(2014).
- Hill, Z. B., Martinko, A. J., Nguyen, D. P., Wells, J. A. Human antibody-based chemically induced dimerizers for cell therapeutic applications. Nature Chemical Biology. 14 (2), 112-117 (2018).
- Foight, G. W., et al. Multi-input chemical control of protein dimerization for programming graded cellular responses. Nature Biotechnology. 37 (10), 1209-1216 (2019).
- Kang, S., et al. COMBINES-CID: An efficient method for de novo engineering of highly specific chemically induced protein dimerization systems. Journal of the American Chemical Society. 141 (28), 10948-10952 (2019).
- Muyldermans, S. Nanobodies: natural single-domain antibodies. Annual Review of Biochemistry. 82, 775-797 (2013).
- Fanning, S. W., Horn, J. R. An anti-hapten camelid antibody reveals a cryptic binding site with significant energetic contributions from a nonhypervariable loop. Protein Science. 20 (7), 1196-1207 (2011).
- Zavrtanik, U., Luken, J., Loris, R., Lah, J., Hadzi, S. Structural basis of epitope recognition by heavy-chain camelid antibodies. Journal of Molecular Biology. 430 (21), 4369-4386 (2018).
- Denhardt, D. T., Dressler, D., Ray, D. S. The Single-Stranded DNA Phages. , 605-625 (1978).
- Virnekas, B., et al. Trinucleotide phosphoramidites: ideal reagents for the synthesis of mixed oligonucleotides for random mutagenesis. Nucleic Acids Research. 22 (25), 5600-5607 (1994).
- Gu, L., et al. Multiplex single-molecule interaction profiling of DNA-barcoded proteins. Nature. 515 (7528), 554-557 (2014).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados