A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
ב Vivo CRISPR / Cas9 הקרנה בו זמנית להעריך תפקוד גנים בעור העכבר וחלל הפה
In This Article
Summary
כאן אנו מתארים מתודולוגיית הקרנה מהירה וישירה vivo CRISPR /Cas9 באמצעות אולטרסאונד מונחה זריקות lentiviral עוברי הרחם כדי להעריך בו זמנית פונקציות של מספר גנים בעור וחלל הפה של עכברים immunocompetent.
Abstract
מודלים של עכברים מהונדסים גנטית (GEMM) סייעו בהערכת תפקוד הגנים, מידול מחלות אנושיות, ושימשו מודל פרה-קולינרי להערכת אפיקים טיפוליים. עם זאת, האופי עתיר הזמן, העבודה והעלות שלהם מגביל את התועלת שלהם לניתוח שיטתי של תפקוד הגנים. ההתקדמות האחרונה בטכנולוגיות לעריכת גנום מתגברת על מגבלות אלה ומאפשרת יצירה מהירה של הפרעות גנים ספציפיות ישירות בתוך איברי עכבר ספציפיים באופן מרובה פיקסלים ומהיר. כאן, אנו מתארים שיטה מבוססת CRISPR /Cas9 (מקובצים באופן קבוע Interspaced קצר Palindromic חוזר) כדי ליצור אלפי שיבוטים נוקאאוט גנים בתוך האפיתל של העור חלל הפה של עכברים, ולספק פרוטוקול המפרט את השלבים הדרושים כדי לבצע מסך ישיר vivo CRISPR עבור גנים מדכאי גידול. גישה זו יכולה להיות מיושמת על איברים אחרים או טכנולוגיות CRISPR/Cas9 אחרות כגון CRISPR-הפעלה או CRISPR-inactivation ללמוד את הפונקציה הביולוגית של גנים במהלך הומאוסטזיס רקמה או בהגדרות מחלה שונות.
Introduction
אחד האתגרים בחקר הסרטן בעידן הפוסט-גנומי הוא לכרות את הכמות העצומה של נתוני הגנום עבור מוטציות גנטיות סיבתיות ולזהות צמתים ברשת הגנים שניתן לסמן אותם כמטרה טיפולית. בעוד ניתוחים ביואינפורמטיים סייעו מאוד לקראת מטרות אלה, הקמת מודלים יעילים במבחנה וב-vivo היא תנאי מוקדם לפענוח המורכבות של מערכות ביולוגיות ומדינות מחלה ולאפשר פיתוח תרופות. בעוד מודלים קונבנציונליים של עכבר מהונדס שימשו בהרחבה למחקרים גנטיים של סרטן vivo, האופי שלהם עלות, זמן- ועבודה אינטנסיבית אסרה במידה רבה על ניתוח שיטתי של מאות גנים סרטניים putative נפרם על ידי גנומיקה מודרנית. כדי להתגבר על צוואר בקבוק זה, שילבנו אולטרסאונד הוקמה בעבר מונחה במתודולוגיית הזרקת הרחם1,2 עם CRISPR / Cas9 (מקובצים באופן קבוע Interspaced קצר Palindromic חוזר) טכנולוגיית עריכתגנים 3 בו זמנית לגרום וללמוד מוטציות אובדן תפקוד של מאות גנים בעור חלל הפה של עכבר אחד.
המתודולוגיה המתוארת כאן משתמשת בזריקות מונחות אולטרסאונד של lentiviruses מהונדסים לתוך חלל מי השפיר של עוברי עכבר חיים ביום העוברי E9.5. המטען העדני המכיל רכיבי CRISPR/Cas9 מתמר את האקטודרם בעל השכבות היחידות, אשר מאוחר יותר מעורר את האפיתל של העור וחלל הפה. העור מורכב מאפידרמיס חיצוני, ואחריו קרום מרתף ודרמיס. אפידרמיס הוא אפיתל מרובד המורכב משכבה פנימית בסיסית, השומרת על קשר עם קרום המרתף ויש לה יכולת רבייה ותא גזע. שכבת הבסיס מעוררת את השכבות המובחנות לעיל כגון שכבות קרנית ספיניות, פרטניות ושכבות2,4. מחקרי מעקב שושלת מראים כי שיטה ישירה זו ב- vivo CRISPR/Cas9 מבצעת מניפולציה גנטית בתאי גזע שוכני רקמות בתוך שכבת הבסיס הנמשכת לאורך כל הבגרות. כמו lentivirus ניתן titrated כדי לתמר את E9.5 פני השטח ectoderm בצפיפות שיבוט, שיטה זו יכולה לשמש כדי ליצור עכברי פסיפס מחסה אלפי שיבוטים נוק אאוט גן דיסקרטי. לאחר מכן ניתן להשתמש ברצף הדור הבא כדי לנתח את ההשפעה של אבלציה של גנים בתיווך CRISPR/Cas9 בתוך שיבוטים אלה באופן מרובה ערכים5.
לאחרונה השתמשנו בשיטה זו כדי להעריך את הפונקציה של 484 גנים המראים מוטציות חוזרות ונשנות בקרצינומה של תאי קשקש הראש והצוואר האנושיים (HNSCC)5. HNSCC הוא סרטן הרסני עם שיעור תמותה גבוה של 40-50% וזה הסרטןהשישי בשכיחותו בעולם6. HNSCC להתעורר רירית רירית של דרכי הנשימה העליונות או חלל הפה קשורים עם צריכת טבק ואלכוהול או זיהום וירוס הפפילומה האנושי (HPV). קרצינומה של תאי קשקש עוריים (SCC) הם גידולים בעור ומייצגים את הסרטן השני בשכיחותו בבני אדם7. SCC עורית ו HNSCC דומים מאוד מבחינה היסטולוגית מולקולרית, עם אחוז גבוה של מקרים המציגים שינוי TP53, PIK3CA, NOTCH1, ו HRAS8. אמנם יש רק קומץ גנים שעברו מוטציה בתדירות גבוהה, אך ישנם מאות גנים שנמצאו שעברו מוטציה בתדירות נמוכה (< 5%), תופעה הידועה בכינויה התפלגות הזנב הארוך. מכיוון שרוב הגנים ארוכי הזנב חסרים אימות ביולוגי או קליני, השתמשנו בטכנולוגיית ההקרנה in vivo CRISPR זו כדי מודל של אובדן תפקוד של גנים אלה בעכברים נוטים לגידולים עם מוטציות רגישות ב- p53, Pik3ca או Hras וזיהינו מספר גנים מדכאי גידולים חדשניים המשתפים פעולה עם p53, Pik3ca או Hras כדי לעורר התפתחות הגידול5.
כאן, אנו מתארים פרוטוקול מפורט כדי ליצור ספריות sgRNA lentiviral CRISPR sgRNA מרובות ולבצע CRISPR / Cas9 גנים לדפוק את המסכים באקטודרם משטח העכבר. שים לב, מתודולוגיה זו יכולה להיות מותאמת לשלב טכנולוגיות אחרות מניפולציה גנים כגון הפעלת CRISPR (CRISPRa) ו CRISPR איון (CRISPRi) או שונה כדי למקד מערכות איברים אחרות בעכבר ללמוד פונקציות גנים.
Protocol
פרוטוקול זה אושר ובוצע בהתאם ל- IACUC של אוניברסיטת טורונטו.
1. עיצוב ושיבוט של ספריות CRISPR במאגר
- בחר 4-5 sgRNAs המכוונים לגנים של עכברים בעלי עניין ממשאבים כגון מעצב sgRNA של המכון הרחב (https://portals.broadinstitute.org/gpp/public/analysis-tools/sgrna-design) או שרת CHOPCHOP (https://chopchop.cbu.uib.no). בחר מספר שווה של sgRNAs שאינם מיקוד מן למשל, Sanjana et al.9 כדי ליצור בגודל שווה שאינו מיקוד פקדים gRNA הספריה.
- בעת בניית ספריות sgRNA, ודא שיש כיסוי מספיק עבור כל sgRNA במערכת האיברים הממוקדת. עבור עור העכבר, האפידרמיס ב E9.5 היא שכבה אחת המכילה ~ 150,000 תאים, שרובם יש קיבולת תאי גזע4. אם התמרה עדנטי ויראלי תוצאות 15-20% זיהומיות, רק 18,000-24,000 תאים יועברו ב E9.5. התאם את קנה המידה של הניסוי בהתאם.
- לשיבוט והגברה של ספריות sgRNA אלה, הוסיפו אתרי הגבלת אנזימים של BsmBI וכן רצפי דנ"א מחייבים פריימר לסוף sgRNA בגובה 5 ו-3 אינץ' והזמינו את האוליגוס באורך מלא כשבב אוליגונוקלאוטיד(איור 1A).
- לספריות מרובות-ערכים שונות בשבב אחד, הוסף רצפי פריימר ספציפיים לספריה.
- באמצעות זוגות פריימר המתאימים, להגביר כל ספרייה בנפרד משבב אוליגו בריכה. בצינור PCR, לערבב 25 μL של 2x פולימראז מאסטר לערבב, 20 μL של DNase / RNase מים חינם, 5 ng של DNA שבב אוליגו, 2.5 μL של פריימר קדימה המתאים ו 2.5 μL של פריימר הפוך מתאים. השתמש 12-15 מחזורים להגביר עם 98 מעלות צלזיוס denaturation, חישול 63-67 מעלות צלזיוס, 72 °C (72 °F) הרחבת פרמטרים עבור כל מחזור במכונת PCR.
- הפעל מוצר PCR על ג'ל agarose 2.5% ולטהר ~ 100 bp PCR המוצר באמצעות ערכת ניקוי DNA ג'ל.
- הכינו את פלסמיד עמוד השדרה.
- תקציר 5 מיקרוגרם של Cre-recombinase המכיל pLKO-Cre ממולא v3 פלסמיד עם 2 μL של BsmBI ב 50 μL תגובה לערבב במשך 1 שעות ב 55 מעלות צלזיוס, ואחריו 1 μL של תפירה phosphatase אלקליין במשך 45 דקות ב 37 מעלות צלזיוס על פי הוראות היצרן.
הערה: Cre-recombinase המכיל pLKO-Cre ממולא v3 פלסמיד הוא פלסמיד מבוסס lenti-ויראלי המכיל אנזים Cre-recombinase כדי להסיר קלטת Lox-Stop-Lox בתאי עכבר ויש לו גם מקדם U6 כדי לנהוג הביטוי של sgRNA ו tracrRNA. גרסה עם Cas9 וללא Cas9 ניתן להשתמש וזמין על #158030 Addgene #158031. - הפעל את ה-DNA מתעכל על 1% ג'ל agarose ולטהר את הלהקה וקטור 7 kb ליניארי באמצעות ערכת ניקוי DNA ג'ל. שימו לב, רצועת ממולאים של 2 kb צריכה להיות גלויה גם היא ומצביעה על תקציר מוצלח.
- תקציר 5 מיקרוגרם של Cre-recombinase המכיל pLKO-Cre ממולא v3 פלסמיד עם 2 μL של BsmBI ב 50 μL תגובה לערבב במשך 1 שעות ב 55 מעלות צלזיוס, ואחריו 1 μL של תפירה phosphatase אלקליין במשך 45 דקות ב 37 מעלות צלזיוס על פי הוראות היצרן.
- הגדר את תגובת הקשירה כדי ליצור ספריית פלסמיד sgRNA.
- מערבבים 1 מיקרוגרם של וקטור מטוהר ו 30 ng של PCR מטוהרים להוסיף עם 2 μL של BsmBI, 5 μL של ליגאז DNA T4, 10 μM ATP ו 1x מאגר ספציפי BsmBI. דגירה תערובת קשירה לילה ב 37 °C (69 °F). השתמש בצינור נוסף המכיל את כל החומרים לעיל למעט תוספת PCR כפקד שלילי.
- למחרת בבוקר, לטהר את תערובת קשירה באמצעות ערכת ניקוי אוליגו ו elute ב 7 μL של מים חינם RNase / DNase.
- אלקטרופורט הספרייה לתאים מוסמכים.
- הוסף 2 μL של ספריית sgRNA eluted או תערובת קשירת בקרה שלילית ל 25 μL של תאים אלקטרו-תחרותיים מופשרים בקובטות מצוננות מראש (1.0 מ"מ) על קרח. אלקטרופורט בעקבות פרוטוקול היצרן (10 μF, 600 אוהם, 1800 וולט). כדי cuvette להוסיף 975 μL של מדיום התאוששות (או בינוני SOC) בתוך 10 s של הדופק.
- להעביר תאים electroporated לצינור תרבות דגירה במשך שעה ב 37 °C (69 °F) באינקובטור טלטול חיידקי ב 300 סל"ד.
- הערך את יעילות הטרנספורמציה ואת כיסוי הספרייה לכל sgRNA.
- הכן דילול פי 100 על ידי העברת 10 μL של תגובת הטרנספורמציה המכילה תאים electroporated עם ספריית sgRNA או קשירת שליטה שלילית ל 990 μL של התאוששות בינוני ומערבבים היטב.
- צלחת 10 μL של תערובת טרנספורמציה מדוללת על צלחת אגר 10 ס"מ מחממת מראש + אמפילין (100 מיקרוגרם / L). התוצאה היא דילול של פי 10,000 של המשתנים ולהשתמש בצלחת זו כדי לחשב את יעילות הטרנספורמציה. בצע דגירה של הצלחות ב 30 מעלות צלזיוס עבור 14-16 שעות.
- צלחת שאר תגובת השינוי על ידי הפצת 100 μL של תאים התאושש על כל צלחת של סך של 10 מראש מחומם LB + צלחות אגר ampicillin. דגירה הצלחות עבור 14-16 שעות ב 30 מעלות צלזיוס. שימו לב, צמיחה ב-30°C ממזערת את ההסתגרות בין שתי החזרות ארוכות הטווח בתוך הפלסמיד הנגיפי.
- כדי להעריך את הצלחת השיבוט, חשב את יעילות הטרנספורמציה. לספור את מספר המושבות על לוח הדילול המכיל transformants מספריית sgRNA או קשירת בקרה שלילית (10 ס"מ לוחות, שלב 1.8.2). תערובת שליטה שלילית לא צריכה להיות כל או רק מעט מאוד מושבות. כדי להשיג את המספר הכולל של המושבות, הכפל את מספר המושבות ב-10,000.
הערה: המספר הכולל של המושבות שנספר מייצג כיסוי ספריה שאמור להיות מינימום של מושבות 200x לכל sgRNA.- ודא שלספריה עם 2000 sgRNAs יהיו לפחות 400,000 מושבות. במקרה שאין מספיק מושבות, לחזור ולהגדיר יותר electroporation.
- בקרת איכות: מלוח הדילול, בחרו 20 מושבות והוסיפו כל מושבה לצינור תרבות בודד המכיל 3 מ"ל של מדיה LB + אפיקילין. דגירה כל 20 צינורות לילה ב 30 מעלות צלזיוס רועד ב 250 סל"ד. לטהר את ה-DNA פלסמיד באמצעות ערכת הכנה מיני על פי הוראות היצרן ורצף סנגר כל 20 דגימות DNA פלסמיד כדי לאמת כל דגימה יש רצף sgRNA שונה באמצעות פריימר U6 (5 '-GAG GGC CTA TTT CTT ATG ATG ATT CC-3').
- לקצור את המושבות מכל צלחת 15 ס"מ.
- הוסף 7 מ"ל של מדיום LB, ולאחר מכן לגרד את המושבות את צלחת LB אגר עם מפזר תאים. השתמש פיפטה 10 מ"ל להעביר את התאים לתוך בקבוק חרוט סטרילי 2 L. יש לחזור על הפעולה עבור כל הצלחות וחיידקי הבריכה בבקבוקון 2 L. דגירה הבקבוק במשך 2-3 שעות רועד ב 30 מעלות צלזיוס. צנטריפוגה התרבות ולאסוף את גלולה.
- לטהר דנ"א פלסמיד באמצעות ערכת טיהור מקסי-פלסמיד.
- בקרת איכות נוספת: השתמש ב- 1 ננוגרם של ספריית sgRNA בהכנה למקסי והפעל תגובת PCR באמצעות פריימרים לריצוף הדור הבא בהתאם להוראות היצרן. ניתן לאמת את הייצוג של כל sgRNAs בספריה על ידי פלטפורמת רצף עמוק.
2. ייצור של לנטיוירוס טיטר גבוה מתאים התמרכוציה vivo
הערה: בצע את כל השלבים בסעיף זה של הפרוטוקול במתקן BSL2+ בארון בטיחות ביולוגי מסוג A2 מסוג Class II. 293FT ובמיוחד תאי האריזה של 293NT מאפשרים ייצור וירוסים גבוה יותר. השתמש בתאי מעבר נמוך (
- הכינו תאי אריזה ויראליים.
- ביום 1, צלחת HEK293T תאים על 6 פולי-L-ליצין מצופה 15 ס"מ צלחות ב 35% מפגש במדיה צמיחה (DMEM + 10% FBS + 1% פניצילין-סטרפטומיצין פתרון אנטיביוטי (w / v)). דגירה תאים לילה ב 37 °C (69 °F), 5% CO2.
- לאחר התאים המצולפים הם 70-80% confluent להתפשט באופן שווה, להחליף את מדיה הצמיחה עם 25 מ"ל של סרום מדיה DMEM ללא אנטיביוטיקה.
- הוסף מדיה לאט לצדדים של צלופשים בעת האכלה, כמו HEK293T ו 293NT תאים יכולים להתנתק בקלות מפלסטיק תרבות הרקמות
- הכינו תערובת טרנס-פליטה.
- הוסף 65 מיקרוגרם של פלסמידים אריזה lentiviral psPAX2, 43 מיקרוגרם של VSV-G הבעת מעטפה pMD2.G, 65 מיקרוגרם של פלסמיד ספריית sgRNA ו 240 μL של 1 מ"ג / מ"ל פוליאתילנימין (PEI) ל 6 מ"ל של DMEM ומערבולת.
הערה: השתמש תמיד בבקבוק DMEM שלא נפתח או נפתח לאחרונה עבור ה- transfection, מכיוון שה- pH בשלב זה הוא קריטי, ו- DMEM הופך לבסיסי יותר עם הזמן לאחר הפתיחה. - דגירה במשך 15 דקות בטמפרטורת החדר. תערובת זו של transfection מספיקה לתאים המצופה על 6 x 15 ס"מ צלחות עם 870 ס"מ2 שטח פנים. התאם את תמהיל ההעתקה בהתאם לדרישות הניסוי.
- הוסף 65 מיקרוגרם של פלסמידים אריזה lentiviral psPAX2, 43 מיקרוגרם של VSV-G הבעת מעטפה pMD2.G, 65 מיקרוגרם של פלסמיד ספריית sgRNA ו 240 μL של 1 מ"ג / מ"ל פוליאתילנימין (PEI) ל 6 מ"ל של DMEM ומערבולת.
- הדבקה של תאי אריזה ויראליים.
- לאחר 15 דקות של דגירה, להחדיר כל אחד 15 ס"מ לוחות על ידי הוספת 1 מ"ל של תערובת transfection dropwise לאורך כל הצלחת. דגירה ב 37 °C (69 °F), 5% CO2 עבור 8 שעות.
- לאחר הדגירה, להסיר את המדיה ללא סרום המכיל את תערובת transfection ולהוסיף 30 מ"ל של מדיה תרבות רגילה צלחות 15 ס"מ ותרבות עבור 48 שעות ב 37 °C (69 °F), 5% CO2.
- לאחר 48 שעות, לאסוף את supernatant ויראלי לסנן דרך 0.45 מיקרומטר PVDF מסנן. שמור 1 מ"ל של supernatant ויראלי ולאחסן ב -80 מעלות צלזיוס לבקרת איכות ו titering ויראלי.
- לרכז את supernatant ויראלי על ידי צנטריפוגה שיפוע סוכרוז בצנטריפוגה במהירות גבוהה באמצעות ראש רוטור SW28.
- צינורות ultracentrifuge ראשוניים על ידי שטיפתם עם 70% אתנול, ואחריו 3 שטיפות עם PBS. באופן שווה להפיץ כ 30 מ"ל של supernatant ויראלי לתוך כל צינור 6-ultracentrifuge.
- בעדינות פיפטה לתחתית של כל צינור 4 מ"ל של 20% פתרון סוכרוז (20 גרם של סוכרוז ב 100 מ"ל של PBS). הנח את ששת צינורות ultracentrifuge לתוך ששת דליי SW28 נדנדה ולאזן במדויק כל זוג מנוגד של דליים על ידי הוספה או הסרה של supernatant ויראלי.
- צנטריפוגה ויראלי supernatant ב 4 °C (69 °F) עבור לפחות 2 שעות ועד 4 שעות ב 80,000 x גרם ו 4 °C (69 °F). לאחר הצנטריפוגה נעשה, בזהירות להשליך את supernatant.
- מסננים את supernatant הנותרים על ידי הצבת צינורות ultracentrifuge במהופך על מגבת נייר רכה סטרילית לפחות 2 דקות ולנגב את כל טיפות של supernatant בתוך הצינור באמצעות מגבת נייר רכה כדי להיפטר מכל מדיום שיורית. גלולה לבנה אפרפרה קטנה עשויה להיות גלויה בתחתית הצינור.
- הוסף 20-25 μL קר, טרי PBS לכל צינור. צינורות חותם עם סרט פרפין וצינורות דגירה זקוף על קרח עם רעידות עדינות על שייקר פלטפורמה מסלולית במשך ~ 2 שעות.
- באמצעות פיפטה 20 μL, בזהירות לעקור ו resuspend גלולה של הצינור הראשון, נזהר לא ליצור בועות. להעביר את המדיום לצינור הבא ולחזור על התהליך עד כל כדורי הנגיף כבר נעק ושולב לתוך צינור אחד של כ 120-150 נפח μL. דגירה זה השעיה ויראלית גבוהה טיטר לעוד ~ 2 שעות על קרח עם רעידות עדינות.
- מעבירים את ההשעיה הנגיפית לצינור מיקרוצנטריפוגה של 1.5 מ"ל ומסובבים את הצינור ב-4 מעלות צלזיוס בצנטריפוגה בקירור וקירור מראש של השולחן ב-12,000 x גרם למשך 2 דקות. Aliquot supernatant ויראלי ברור בזהירות כמו 10 μL aliquots בצינורות נפרדים 0.2 מ"ל ולאחסן ב -80 מעלות צלזיוס. להשליך את גלולה בתחתית הצינור כמו זה יהיה לסתום את המחט במהלך זריקות עובריות.
- טיטרציה של ספריית CRISPR לנטיוירוס במאגר
הערה: ההשעיה נגיפית וכתוצאה מכך צריך להניב ריכוז כ 2,000 פי ואת פתרון הנגיף וכתוצאה מכך צריך titer ויראלי של 107-109, וזה מספיק טוב עבור יותר מ 100 E9.5 ניתוחים המתוארים להלן.- זרע R26-לוקס-STOP-לוקס-tdTomato עכבר פיברובלסטים עובריים (MEFs) או R26-Lox-STOP-Lox-tdTomato קרטינוציטים ראשוניים או כל קו תאים קר-כתב אחר לתוך צלחת 2 x 6 באר.
- ברגע שהתא מגיע ~ 40% מפגש, התאים מוכנים להעברה. באותו זמן, לקבוע את מספר התאים בתוך באר אחת כדי לאפשר חישוב של titer ויראלי מאוחר יותר. לשנות את המדיה של תאי Cre-כתב למדיה צמיחה בתוספת פוליברן 10 מיקרוגרם / מ"ל (= הקסדימתרין ברומיד).
- לדלל 1 μL של וירוס מרוכז עם 2 מ"ל של מדיה צמיחה. הוסף 0, 10, 50, 200 μL של השעיה ויראלית מדוללת ו 50, 200 μL של וירוס לא מרוכז משלב 2.4 ומערבבים צלחות ודגרת במהלך הלילה ב 37 °C (69 °F), 5% CO2.
- הסר מדיית צמיחה המכילה וירוס למחרת, לשטוף תאים עם PBS ולהחליף עם מדיה צמיחה נורמלית. העל-טבעי בשלב זה עדיין נחשב לפסולת ויראלית ויש להשליך אותו בהתאם לתקנות BSL2 המוסדיות.
- כ 36-48 שעות לאחר התמרה, להעריך שילוב מחדש בתיווך Cre של קלטת לוקס-STOP-לוקס וביטוי של tdTomato על ידי FACS. חשב טיטר ויראלי. חשב יחידות יוצרות מושבה (cfu)/mL באמצעות הנוסחה הבאה:
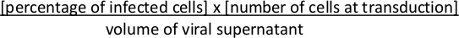
הערה: השוואה בין ההשעיה הנגיפית המרוכזת עם ההשעיה הנגיפית הבלתי מעורערת שלה תאפשר להעריך את ההצלחה של (1) ייצור ויראלי כמו גם (2) ריכוז ויראלי. לחלופין, ניתן להעריך את הטיטר הוויראלי באמצעות שיטת qRT-PCR10. ייצור בקנה מידה גדול וריכוז של lentivirus יכול להתבצע גם באמצעות ריכוז דו שלבי עם ריכוז מחסנית ואחריו ultracentrifugation כפי שתואר קודםלכן 2.
3. ניתוח מונחה אולטרה קול והזרקה
הערה: טכנולוגיה זו הותאמה מ4,11. Microinjection המיועד להעתקה של אפיתל פני השטח חייב להתבצע ביום העוברי E9.5, כאשר אקטודרם פני השטח מורכב משכבה אחת ולפני היווצרות של periderm החל מ- E10, אשר ימנע התמרה של שכבה בסיסית זו. רצוי להגדיר עכברים ביום שישי, כך היום הראשון האפשרי עם E9.5 עוברים הוא יום שני הבא. השתמשו בעכברי Rosa26-Lox-STOP-LOX-Cas9-GFP (מעבדת ג'קסון #024857) ליעילות אופטימלית של CRISPR/Cas912.
- הגדר עכברים זכר ונקבה המתאימים עבור הריונות בזמן 10 ימים לפני תאריך הניתוח הרצוי.
- הכנס-בדוק עכברים במהלך הימים הבאים כדי לזהות עכברים שעלולים להיות בהריון. אשר את ההריון יום לפני הניתוח המתוכנן (כלומר, E8.5) באמצעות אולטרסאונד.
- מכינים את צלחת הפטרי המותאמת.
- באמצעות סרט הדבקה דביק דו צדדי, להדביק את קרום הסיליקון על החלק התחתון של צלחת פטרי מכסה את הפתח העגול. בעזרת מספריים חדות, חותכים פתח אליפטי של 2x10 מ"מ בקרום הסיליקון.
- הכנת מערכת המיקרו-ג'ינג'ים
- הכן מחטי הזרקה מ נימי זכוכית עבים באמצעות משיכה micropipette עם ההגדרות הבאות: לחץ = 200; חום = 769; משוך = 0; מהירות = 140; זמן = 100.
- באמצעות מספריים בסדר לחתוך את קצה המחט ברמה שבה קוטרו הוא ~ 30 מיקרומטר. משפשפים את המחט באמצעות מחדד מחט ל 20 ° על צלחת שוחקת בדרגה עדינה עם הרטבה רגילה במשך 20 דקות.
- לעקר את מחט microinjection על ידי דחיפת 70% אתנול באמצעות מזרק 10 מ"ל עם מחט 26 G1/2. באמצעות מזרק 10 מ"ל ומחט 26 G1/2 טעון בשמן מינרלי, למלא את מחט microinjection. ודא כי אין בועות במחט microinjection.
- הר מחט microinjector על micromanipulator על פי הוראות היצרן.
- טען את המחט עם ספריית sgRNA ויראלית.
- פיפטה 10 μL של השעיה ויראלית על parafilm קבוע על משטח שטוח.
- באמצעות מיקרומניפולטור, לגרש את השמן המינרלי. נוכחות של נפח קטן של שמן במחט ימנע lentivirus מליצור קשר עם הבוכנה. לטעון על 4-5 μL השעיה ויראלית על ידי שאיפה איטית ללא כל בועות אוויר.
- הרדמה עכבר בהריון באמצעות תא אינדוקציה isoflurane. הגדר את וסת החמצן ל 1 L / min ו isoflurane מאדה ל 2%. כדי לקבוע אם החיה מורדמת, בדוק לפי קמצוץ בוהן לאחר ~ 3-5 דקות בתא אינדוקציה איזופלורן.
- מניחים את הצד הגחוני של בעלי החיים המרדים על שלב הניתוח המחומם (40 מעלות צלזיוס) ומחילים קלטות ניתוח כדי להחזיק את הכפות במקום עם צינור ההרדמה של חרוט האף לאספקה מתמדת של isoflurane וחמצן עד להשלמת הניתוח.
- אשר E9.5 הריון אם זה לא נעשה ב E8.5.
- החל כמות קטנה (~ 1/2 כפית) של קרם להסרת שיער על הבטן ולהפיץ אותו על פני שטח 3 x 3 ס"מ באמצעות מוליך כותנה קצה.
- לאחר 2-3 דקות, להסיר בעדינות את השמנת עם גזה או רקמה ולנגב את האזור נקי באמצעות PBS ו 70% אתנול.
- באמצעות בדיקה אולטרסאונד וג'ל, לבדוק את העכבר בהריון ליום עוברי E9.5. יש לציין, זה יכול להיעשות גם יום קודם לכן ב E8.5 כדי לחסוך זמן ביום הניתוח בפועל.
- הסר ג'ל אולטרסאונד ולנגב אזור נקי באמצעות BPS ולאחר מכן 70% אתנול לחטא.
- להזריק 0.02 סמ"ק של משכך כאבים Buprenorphine תת עורית בסכר העכבר.
- לחשוף בניתוח את הרחם
- באמצעות מלקחיים ומספריים סטריליים, לבצע חתך אנכי ~ 2 ס"מ לתוך העור של העכבר.
- שימוש במלקחיים קהים מפרידים את העור סביב החתך מהציצי הצפק.
- באמצעות מלקחיים ומספריים סטריליים, לחתוך דרך הצפק.
- בעזרת מלקחיים קהים, מוציאים בעדינות את קרן הרחם השמאלית והימנית וסופרים את העוברים.
- הכנס מחדש את רוב קרני הרחם אך השאר את הקצה הדיסטלי של קרן רחם אחת המכילה 3 עוברים חשופים.
- באמצעות מלקחיים קהים, בעדינות לדחוף ולמשוך את הרחם עם 3 עוברים דרך הפתח בקרום סיליקון של צלחת פטרי שונה.
- לייצב את צלחת פטרי באמצעות קוביות של חימר דוגמנות.
- לייצב את הרחם עם שלושת העוברים בתוך צלחת פטרי באמצעות תבנית סיליקון. משטחים את תקע הסיליקון בצד העוברים באמצעות סכין חדה.
- מלאו את צלחת הפטרי ב-PBS סטרילי. לשטוף את קרום הסיליקון על החלק התחתון של צלחת פטרי עם הבטן של הסכר בהריון ובכך למנוע כל דליפה.
- להעביר את ראש אולטרסאונד לתוך צלחת פטרי ~ 0.5 ס"מ על גבי העובר העליון ולהתאים את הבמה, כך חלל מי השפיר הופך גלוי בבירור בתצוגת אולטרסאונד.
- ליישר את המחט מזרק בזהירות לתוך צלחת פטרי שונה. באמצעות micromanipulator, למקם את קצה המחט בתוך ~ 5 מ"מ של העובר העליון. ואז לעבור את המחט קדימה ואחורה ולעבור לתוך המטוס שבו הקצה שלה מופיע הבהיר ביותר בתמונת אולטרסאונד.
- באמצעות micromanipulator לדחוף את המחט דרך קיר הרחם לתוך חלל מי השפיר.
- להזריק lentivirus המכיל ספריית sgRNA.
- מראש לתכנת את המזרק ל 62 nL ומהירות הזרקה איטית. המיקרו-אינג'קטור יכול להיות מתוכנת להזריק כמויות ספציפיות במהירות איטית או מהירה.
- לחץ על כפתור הזרק 8 פעמים עבור סך של 496 nL לכל עובר. כוונן את עוצמת הקול לצרכים הנדרשים ולטיטר ויראלי. טיטר ויראלי של 1 x 108 pfu / mL ו 496 נפח הזרקת nL יגרום כ 30% זיהומיות.
- חזור על אותו הליך עבור שני עוברים אחרים.
- הרם את ראש האולטרסאונד והסר את תקע הסיליקון.
- בעדינות לדחוף את 3 עוברים לתוך הבטן של העכבר באמצעות החלפת כותנה סטרילית.
- משוך בעדינות את 3 העוברים הבאים וחזור על הליך ההזרקה עד להזרקת מספר העוברים הרצוי.
הערה: לא יעלה על 30 דקות של הרדמה כמו סכרים בהריון נוטים הרבה יותר להפיל את העוברים שלהם מעבר לזמן זה. מנתח מנוסה יכול להזריק עד 12 עוברים בתוך 30 דקות ניתוח.
- להרים את ראש אולטרסאונד, להסיר את micromanipulator עם המחט, לשאוף את PBS ולהסיר את צלחת פטרי. באמצעות מגבונים סטריליים, לספוג כל PBS שאולי הצטבר בחלל הבטן.
- סגור את החתך הצפק באמצעות תפרים נספגים. השתמש בשני סיכות כדי לסגור את החתך בעור הבטן.
הערה: בעת ביצוע אולטרה-קול מונחה בניתוחי הרחם, יש לנקוט בזהירות מירבית כדי לשמור על סביבה נקייה, לשמור על עקרות ככל האפשר ולהימנע מכל מזהמים אפשריים. אם יותר מניתוח אחד חייב להיעשות באותו יום, חשוב לעקר את מכשירי הניתוח באמצעות מעקר חרוזים ולנקות מכשירים אחרים שנתקלים ברקמת עכבר מנוקה עם אתנול. זה מגדיל מאוד את ההישרדות הגבוהה יותר של העוברים לאחר הניתוח. שיעור ההישרדות של שיטת הניתוח המתוארת כאן הוא באופן עקבי בין 80-100%. - טיפול לאחר הניתוח.
- אפשר לנקבה ההרה להתאושש בכלוב מחומם ולהתבונן במשך 15-30 דקות.
- בדוק תעלת הנרתיק עבור דם, שהוא סימן מוקדם של הפלה וסיבוכים. להקלה על הכאב לאחר הניתוח, יש לתת את משכך הכאבים פעמיים ביום במשך יומיים לאחר הניתוח.
- זהה עוברים שעברו עירוי/תינוק Rosa26-Lox-STOP-Lox-Cas9-GFP עכברים באמצעות מיקרוסקופ ניתוח פלואורסצנטי, פנס חלבון פלואורסצנטי ומשקפי סינון או על ידי genotyping עבור חלקיקים ויראליים משולבים באוזן או בזנב קליפים.
הערה: עכברים מזוהים בצורה הטובה ביותר באמצעות מיקרוסקופ פלואורסצנטי עד יום לאחר הלידה 3 לפני השיער מתחיל לגדול. - כדי להבטיח כיסוי מספיק עבור ספריית CRISPR, חשב כיסוי בהתבסס על הפרמטרים הבאים: ectoderm משטח העכבר E9.5 מורכב ~ 150,000 תאים; transduction של ~ 20% תוצאות שיעור זיהום כפול מינימלי (~ 1/10 תאים נגועים כפולים)4,5,11; ב 20% זיהומיות, לכל עובר יש 30,000 תאים נגועים. ודא שלפחות 100 תאים בודדים עוברים טרנס-רנ"א נתון. לדוגמה, מאגר של 2000 sgRNAs דורש 2 x 105 תאים או 6-7 בעלי חיים.
4. הליך רצף עמוק
- בנקודת קצה ניסיונית כגון היווצרות גידול או כל פנוטיפ אחר, להקריב את העכבר לקצור את הרקמה של עניין. השתמש בערכת מיצוי DNA של דם ורקמות כדי לטהר דנ"א גנומי בהתאם לפרוטוקול היצרן. לפני תחילת מיצוי DNA מגידולים, לנקות את ספסל העבודה עם DNA למחוק ולעבוד הרחק מאזור המעבדה שבו פלסמידים מטופלים.
הערה: חשוב ליצור דוגמת ייחוס כדי לאפשר חישוב שינויים בקיפול sgRNA לאורך זמן. עוברים שעברו עירוי 3 ימים לאחר ההדבקה או תאים שעברו חילוף (ראה שלב 2. 6) יכולים לשמש כמדגם הפניה לקביעת ייצוג sgRNA בספריה המקורית. - השתמש 100 ng – 1.5 מיקרוגרם DNA גנומי כתבנית בתגובת PCR 50 μL טרום הגברה 1 באמצעות פריימרים החיצוניים המפורטים בטבלה 1 ואת תוכנית PCR המתואר בטבלה 2. הגדר מספיק תגובה כדי להניב כיסוי רצוי.
הערה: הגדר את תגובת PCR barcoding באזור ייעודי נפרד של המעבדה או מכסה המנוע של תרבית הרקמות כי הוא ניקה ביסודיות על ידי DNA למחוק כדי למנוע זיהום. הפעל שליטה שלילית עבור כל לוח PCR עם מים כתבנית כדי לוודא שאין זיהום. - לאגור את כל התגובה PCR 1 ולהפעיל 20 μL על 1.5% בקרת agarose ג'ל כדי לאמת הגברה מוצלחת של הלהקה 600 bp.
- השתמש ב- 5 μL של תגובת PCR1 במאגר כתבנית בתגובת PCR 2 של 50 μL באמצעות שילוב פריימר ברקוד ייחודי המפורט בטבלה 3 ותוכנית PCR המתוארת בטבלה 4. הפעל 50 μL של PCR 2 תגובה על 2% ג'ל agarose לטהר את הלהקה 200bp באמצעות ערכת מיצוי ג'ל לפי הוראות היצרן.
הערה: שלב טרום ההגברה נדרש רק להגברה של רקמה טרנסיבית מורכבת. אם הגברה של גידול יחיד שככל הנראה התפתחה בצורת תא שיבוט ובכך מכילה רק sgRNA אחד, ניתן לדלג על PCR1 טרום הגברה ולהמשיך מיד עם PCR2. - לכמת את ריכוז הדנ"א באמצעות כימות פלואורומטרי ונשלח לריצוף עמוק למתקן הרצף (למשל, מיליון קריאות לכל גידול).
- השתמש ב- Bowtie v1.2.3, המאפשר יישורים לא מנוצלים בלבד, ישר קריאות רצף לספריית sgRNA13. צור אינדקס עניבת פרפר באמצעות פקודת בניית עניבת פרפר הזן רצפי sgRNA בפורמט fasta. מפה את הקריאות באמצעות הפקודה עניבת פרפר עם אפשרויות -m 2 -v 1, המאפשרת מקסימום שני אי-התאמות במהלך יישור קריאה ומבטלת קריאות המיישרות יותר מרצף sgRNA ספריה אחד. בדרך כלל, יותר מ- 80% מהקריאות מיושרות תחת פרמטרים אלה.
- השתמש בפקודה MAGeCK Count כדי להשיג טבלת ספירת קריאה באמצעות הקובץ המיושר כקלט14. טבלת ספירת sgRNA יכולה לשמש לניתוח במורד הזרם כגון, קביעת sgRNA דיפרנציאלי (MAGeCK) ומציאת גנים חיוניים (בייגל).
תוצאות
איור 1A מציג את העיצוב של האוליגונוקלאוטידים עבור multiplexing מספר ספריות CRISPR מותאמות אישית באופן חסכוני בשבב אוליגו יחיד של 12k או 92k. לאחר בחירת sgRNAs (צבע כחול מקודד), האוליגונוקלאוטידים מעוצבים עם אתרי הגבלה (BsmBI בצבע כתום) וזוגות פריימר PCR ספציפיים לספריה (מקודדים בצבע ירוק). ניתן...
Discussion
עריכת הגנום CRISPR/Cas9 הייתה בשימוש נרחב במחקרי הפריה חוץ גופית וב-vivo כדי לחקור תפקודי גנים ותהליכים תאיים. רוב מחקרי vivo משתמשים בתאים ערוכים בגן CRISPR/Cas9 המושתלים במודל של בעלי חיים (allograft או xenograft). אמנם זהו כלי רב עוצמה לחקר גנטיקה סרטן ותפקודים הסלולר, זה עדיין חסר microenvironment רקמה מקורית ועלול לע...
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
עבודה זו נתמכה על ידי מענק פרויקט מהמכון הקנדי לחקר הבריאות (CIHR 365252), קרן קרמביל וקרן המחקר של אונטריו סבב מצוינות מחקר 8 (RE08-065). סמפת קומאר לוגנאתן הוא מקבל מלגת האגודה הקנדית למלחמה בסרטן (BC-F-16#31919).
Materials
| Name | Company | Catalog Number | Comments |
| 0.45 micron filter | Sigma | S2HVU02RE | |
| 12k or 92k oligo chip | Customarray Inc. (Genscript) | ||
| 15 cm cell culture plates | Corning | ||
| 293FT | Invitrogen | R70007 | |
| 293NT | Systems Biosciences | LV900A-1 | |
| Alkaline phosphatase | NEB | M0290L | |
| Amplicillin | Fisher Scientific | BP1760-25 | |
| ATP | NEB | 9804S | |
| BsmBI | NEB | R0580L | |
| Chromic gut suture | Covidien | ||
| Deep sequencing (Next-Seq or Hi-Seq) | Illumina | ||
| DNA-cleanup kit | Zymo Research | D4008 | |
| DNAesy Blood and Tissue DNA extraction kit | Qiagen | 69506 | |
| Endura electrocompetent cells | Lucigen | 60242-1 | |
| Glass Capillaries | Drummond | 3-000-203-G/X | |
| HEK293T cells | ATCC | CRL-3216 | |
| High-Speed Centrifuge | Beckman Coulter | MLS-50 | |
| LB Agar | Wisent Technologies | 800-011-LG | |
| Micropipette puller | Sutter Instrument | P97 | |
| Mineral oil | Sigma | M5904 | |
| Mini-prep plasmid Kit | Frogga Bio | PDH300 | |
| Mouse oxygen anaesthesia system | Visual Sonics | ||
| Nanoject II micromanipulator | Drummond | ||
| NEBuffer 3.1 (Buffer for BsmBI) | NEB | R0580L | |
| Needle sharpener | Sutter Instrument | BV-10 | |
| Oligo cleanup kit | Zymo research | D4060 | |
| PAGE purified illumina sequencing primer | IDT DNA | ||
| PEI (polyethyleneimine) | Sigma | 408727-100ML | |
| Permoplast modeling clay | |||
| Petridish with central opening | Visual Sonics | ||
| pMD2.G | Addgene | 12259 | |
| psPAX2 | Addgene | 12260 | |
| Q5 Polymerase 2x Master mix | NEB | M0494L | |
| Qubit Fluorometric Quantification | Invitrogen | Q33327 | |
| Semicircular Silicone plug | Corning | ||
| Silicone membrane | Visual Sonics | ||
| T4 DNA ligase | NEB | M0202L | |
| Ultra-centrifuge tubes | Beckman Coulter | 344058 | |
| Vevo2000 ultrasound system | Visual Sonics |
References
- Beronja, S., Fuchs, E. RNAi-mediated gene function analysis in skin. Methods in Molecular Biology. 961, 351-361 (2013).
- Beronja, S., Livshits, G., Williams, S., Fuchs, E. Rapid functional dissection of genetic networks via tissue-specific transduction and RNAi in mouse embryos. Nature Medicine. 16, 821-827 (2010).
- Shalem, O., et al. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science. 343, 84-87 (2014).
- Beronja, S., et al. RNAi screens in mice identify physiological regulators of oncogenic growth. Nature. 501, 185-190 (2013).
- Loganathan, S. K., et al. Rare driver mutations in head and neck squamous cell carcinomas converge on NOTCH signaling. Science. 367, 1264-1269 (2020).
- Leemans, C. R., Snijders, P. J. F., Brakenhoff, R. H. The molecular landscape of head and neck cancer. Nature Reviews Cancer. 18, 269-282 (2018).
- Green, A. C., Olsen, C. M. Cutaneous squamous cell carcinoma: an epidemiological review. British Journal of Dermatology. 177, 373-381 (2017).
- Campbell, J. D., et al. pathway network, and immunologic features distinguishing squamous carcinomas. Cell Reports. 23, 194-212 (2018).
- Sanjana, N. E., Shalem, O., Zhang, F. Improved vectors and genome-wide libraries for CRISPR screening. Nature Methods. 11, 783-784 (2014).
- Geraerts, M., Willems, S., Baekelandt, V., Debyser, Z., Gijsbers, R. Comparison of lentiviral vector titration methods. BMC Biotechnology. 6, 34 (2006).
- Schramek, D., et al. Direct in vivo RNAi screen unveils myosin IIa as a tumor suppressor of squamous cell carcinomas. Science. 343, 309-313 (2014).
- Platt, R. J., et al. CRISPR-Cas9 knockin mice for genome editing and cancer modeling. Cell. 159, 440-455 (2014).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10, 25 (2009).
- Li, W., et al. MAGeCK enables robust identification of essential genes from genome-scale CRISPR/Cas9 knockout screens. Genome Biology. 15, 554 (2014).
- Winters, I. P., Murray, C. W., Winslow, M. M. Towards quantitative and multiplexed in vivo functional cancer genomics. Nature Reviews Genetics. 19, 741-755 (2018).
- Rogers, Z. N., et al. Mapping the in vivo fitness landscape of lung adenocarcinoma tumor suppression in mice. Nature Genetics. 50, 483-486 (2018).
- Chow, R. D., et al. AAV-mediated direct in vivo CRISPR screen identifies functional suppressors in glioblastoma. Nature Neuroscience. 20, 1329-1341 (2017).
- Wang, G., et al. Mapping a functional cancer genome atlas of tumor suppressors in mouse liver using AAV-CRISPR–mediated direct in vivo screening. Science Advances. 4, 5508 (2018).
- Chan, K., Tong, A. H. Y., Brown, K. R., Mero, P., Moffat, J. Pooled CRISPR-based genetic screens in mammalian cells. Journal of Visualized Experiments. (151), e59780 (2019).
- Hart, T., et al. High-resolution CRISPR screens reveal fitness genes and genotype-specific cancer liabilities. Cell. 163, 1515-1526 (2015).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved