È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Screening CRISPR/Cas9 in vivo per valutare contemporaneamente la funzione genica nella pelle del topo e nella cavità orale
In questo articolo
Riepilogo
Qui descriviamo una metodologia di screening CRISPR/Cas9 in vivo rapida e diretta utilizzando iniezioni lentivirali embrionali guidate da ultrasuoni in utero per valutare contemporaneamente le funzioni di diversi geni nella pelle e nella cavità orale dei topi immunocompetenti.
Abstract
I modelli di topo geneticamente modificati (GEMM) sono stati fondamentali per valutare la funzione genica, modellare le malattie umane e servire come modello preclinico per valutare le vie terapeutiche. Tuttavia, la loro natura ad alta intensità di tempo, manodopera e costi limita la loro utilità per l'analisi sistematica della funzione genica. I recenti progressi nelle tecnologie di editing del genoma superano tali limitazioni e consentono la rapida generazione di specifiche perturbazioni geniche direttamente all'interno di specifici organi del topo in modo multiplexato e rapido. Qui, descriviamo un metodo basato su CRISPR / Cas9 (Clustered Regularly Interspaced Short Palindromic Repeats) per generare migliaia di cloni gene knock-out all'interno dell'epitelio della pelle e della cavità orale dei topi e fornire un protocollo che dettaglia i passaggi necessari per eseguire uno schermo CRISPR in vivo diretto per i geni soppressori del tumore. Questo approccio può essere applicato ad altri organi o ad altre tecnologie CRISPR/Cas9 come l'attivazione CRISPR o l'inattivazione CRISPR per studiare la funzione biologica dei geni durante l'omeostasi tissutale o in vari contesti di malattia.
Introduzione
Una delle sfide per la ricerca sul cancro nell'era post-genomica è estrarre la grande quantità di dati genomici per le mutazioni geniche causali e identificare i nodi nella rete genica che possono essere mirati terapeuticamente. Mentre le analisi bioinformatiche hanno contribuito immensamente a raggiungere questi obiettivi, stabilire modelli efficienti in vitro e in vivo è un prerequisito per decifrare la complessità dei sistemi biologici e degli stati delle malattie e per consentire lo sviluppo di farmaci. Mentre i modelli convenzionali di topi transgenici sono stati ampiamente utilizzati per studi di genetica del cancro in vivo, la loro natura ad alta intensità di costi, tempo e lavoro ha ampiamente vietato l'analisi sistematica delle centinaia di geni del cancro putativo non rivelato dalla genomica moderna. Per superare questo collo di bottiglia, abbiamo combinato una tecnologia di editing genica guidata da ultrasuoni precedentemente stabilita nella metodologia di iniezione utero1,2 con una tecnologia di editing genico CRISPR / Cas9 (Clustered Regularly Interspaced Short Palindromic Repeats)3 per indurre e studiare simultaneamente mutazioni di perdita di funzione di centinaia di geni nella pelle e nella cavità orale di un singolo topo.
La metodologia qui descritta utilizza iniezioni guidate da ultrasuoni di lentivirus ingegnerizzati nella cavità amniotica degli embrioni di topo viventi al giorno embrionale E9.5. Il carico lentivirale contenente componenti CRISPR/Cas9 trasduce l'ectoderma superficiale monostrato, che successivamente dà origine all'epitelio della pelle e della cavità orale. La pelle è composta da un'epidermide esterna, seguita da membrana basale e derma. L'epidermide è un epitelio stratificato composto da uno strato interno basale, che mantiene il contatto con la membrana basale e ha capacità proliferativa e cellulare. Lo strato basale dà origine agli strati differenziati sopra come strati spinosi, granulari e strato corneo2,4. Studi di tracciamento del lignaggio mostrano che questo metodo CRISPR/Cas9 in vivo diretto manipola geneticamente le cellule staminali residenti nei tessuti all'interno dello strato basale che persistono per tutta l'età adulta. Poiché il lentivirus può essere titolato per trasdurre l'ectoderma superficiale E9.5 a densità clonale, questo metodo può essere utilizzato per generare topi mosaico che ospitano migliaia di cloni discreti a knock-out genico. Il sequenziamento di nuova generazione può quindi essere utilizzato per analizzare l'effetto dell'ablazione genica mediata da CRISPR /Cas9 all'interno di tali cloni in modomultiplexato 5.
Recentemente abbiamo utilizzato questo metodo per valutare la funzione di 484 geni che mostrano mutazioni ricorrenti nel carcinoma a cellule squamose della testa e del collo umano (HNSCC)5. L'HNSCC è un cancro devastante con un alto tasso di mortalità del 40-50% ed è il6 ° cancro più comune in tutto ilmondo 6. L'HNSCC si presenta nei rivestimenti mucosi delle vie aeree superiori o della cavità orale e sono associati al consumo di tabacco e alcol o all'infezione da papillomavirus umano (HPV). Il carcinoma a cellule squamose cutanee (SCC) sono tumori della pelle e rappresentano il secondo tumore più comune nell'uomo7. Cutaneous SCC e HNSCC sono istologicamente e molecolarmente molto simili, con un'alta percentuale di casi che presentano alterazioni in TP53, PIK3CA, NOTCH1 e HRAS8. Mentre ci sono solo una manciata di geni mutati ad alta frequenza, ci sono centinaia di geni trovati mutati a bassa frequenza (< 5%), un fenomeno comunemente indicato come distribuzione a coda lunga. Poiché la maggior parte dei geni a coda lunga manca di convalida biologica o clinica, abbiamo utilizzato questa tecnologia di screening CRISPR in vivo per modellare la perdita di funzione di questi geni nei topi soggetti a tumore con mutazioni sensibilizzanti in p53, Pik3ca o Hras e identificato diversi nuovi geni soppressori del tumore che collaborano con p53, Pik3ca o Hras per innescare lo sviluppo tumorale5.
Qui, descriviamo un protocollo dettagliato per generare librerie di sgRNA CRISPR lentivirale sgRNA multiplexato ed eseguire schermi di knock-out genico CRISPR / Cas9 nell'ectoderma della superficie del mouse. Da notare che questa metodologia può essere adattata per incorporare altre tecnologie di manipolazione genica come l'attivazione CRISPR (CRISPRa) e l'inattivazione CRISPR (CRISPRi) o modificata per indirizzare altri sistemi di organi nel topo per studiare le funzioni geniche.
Protocollo
Questo protocollo è stato approvato ed eseguito in conformità con la IACUC dell'Università di Toronto.
1. Progettazione e clonazione di librerie CRISPR in pool
- Seleziona 4-5 sgRNA mirati ai geni del mouse di interesse da risorse come il progettista di sgRNA del Broad Institute (https://portals.broadinstitute.org/gpp/public/analysis-tools/sgrna-design) o il server CHOPCHOP (https://chopchop.cbu.uib.no). Selezionare un numero uguale di sgRNA non mirati, ad esempio Sanjana e altri9, per generare una libreria di gRNA di controlli non mirati di dimensioni uguali.
- Durante la costruzione delle librerie di sgRNA, assicurarsi che ci sia una copertura sufficiente per ogni sgRNA nel sistema di organi mirato. Per la pelle del topo, l'epidermide di E9.5 è un singolo strato che contiene ~ 150.000 cellule, la maggior parte delle quali hanno capacità di cellulestaminali 4. Se la trasduzione lenti-virale si traduce in un'infettività del 15-20%, solo 18.000-24.000 cellule saranno trasdotto a E9,5. Ridimensionare l'esperimento di conseguenza.
- Per la clonazione e l'amplificazione di queste librerie di sgRNA, aggiungere siti di restrizione enzimatica BsmBI e sequenze di DNA leganti primer a 5' e 3' estremità dello sgRNA e ordinare gli oligo a lunghezza intera risultanti come chip oligonucleotide pooled (Figura 1A).
- Per multiplexare librerie diverse su un chip, aggiungere sequenze primer specifiche della libreria.
- Utilizzando le coppie di primer appropriate, amplificare ogni libreria separatamente dal chip oligo in pool. In un tubo PCR, mescolare 25 μL di miscela master di polimerasi 2x, 20 μL di acqua libera di DNasi/RNasi, 5 ng di DNA di chip oligo, 2,5 μL di primer in avanti appropriato e 2,5 μL di primer inverso appropriato. Utilizzare 12-15 cicli e amplificare con denaturazione di 98 °C, ricottura a 63-67 °C, parametri di estensione di 72 °C per ogni ciclo nella macchina PCR.
- Eseguire il prodotto PCR su un gel di agarosio al 2,5% e purificare il prodotto PCR ~ 100 bp utilizzando un kit di pulizia del DNA in gel.
- Preparare il plasmide dorsale.
- Digerire 5 μg della Cre-ricombinasi contenente pLKO-Cre stuffer v3 plasmide con 2 μL di BsmBI in miscela di reazione 50 μL per 1 h a 55 °C, seguita da 1 μL di incubazione di fosfatasi alcalina per 45 min a 37 °C secondo le istruzioni del produttore.
NOTA: La cre-ricombinasi contenente pLKO-Cre stuffer v3 plasmide è un plasmide a base lenti virale che contiene enzima Cre-ricombinasi per rimuovere la cassetta Lox-Stop-Lox nelle cellule del topo e ha anche un promotore U6 per guidare l'espressione di sgRNA e tracrRNA. Una versione con Cas9 e senza Cas9 può essere utilizzata e disponibile su Addgene #158030 e #158031. - Eseguire il DNA digerito su un gel di agarosio all'1% e purificare la banda vettoriale linearizzata da 7 kb utilizzando un kit di pulizia del DNA in gel. Da notare che dovrebbe essere visibile anche una banda stuffer da 2 kb che indica un digest di successo.
- Digerire 5 μg della Cre-ricombinasi contenente pLKO-Cre stuffer v3 plasmide con 2 μL di BsmBI in miscela di reazione 50 μL per 1 h a 55 °C, seguita da 1 μL di incubazione di fosfatasi alcalina per 45 min a 37 °C secondo le istruzioni del produttore.
- Impostare la reazione di legatura per generare una libreria plasmide sgRNA.
- Mescolare 1 μg di vettore purificato e 30 ng di inserto PCR purificato con 2 μL di BsmBI, 5 μL di T4 DNA ligasi, 10 μM ATP e 1x buffer specifico per BsmBI. Incubare il mix di legatura durante la notte a 37 °C. Utilizzare un tubo aggiuntivo contenente tutti i materiali di cui sopra ad eccezione dell'inserto PCR come controllo negativo.
- Il giorno dopo, purificare il mix di liga utilizzando il kit di pulizia oligo ed elute in 7 μL di acqua libera di RNasi / DNasi.
- Elettroporare la libreria in celle competenti.
- Aggiungere 2 μL di libreria di sgRNA eluita o miscela di legatura di controllo negativo a 25 μL di celle elettrocompetenti scongelate in cumvette pre-refrigerate (1,0 mm) sul ghiaccio. Elettroporato seguendo il protocollo del produttore (10 μF, 600 Ohm, 1800 Volt). Alla cuvetta aggiungere 975 μL di mezzo di recupero (o mezzo SOC) entro 10 s dall'impulso.
- Trasferire le cellule elettroporate in un tubo di coltura e incubare per un'ora a 37 °C in un incubatore di scuotimento batterico a 300 giri/min.
- Stimare l'efficienza di trasformazione e la copertura della libreria per sgRNA.
- Preparare una diluizione di 100 volte trasferendo 10 μL della reazione di trasformazione contenente cellule elettroporate con libreria di sgRNA o legatura di controllo negativo a 990 μL di Recupero Medio e mescolare bene.
- La piastra 10 μL della miscela di trasformazione diluita su una piastra di agar preri warmed da 10 cm LB + ampicillina (100 μg/L). Ciò si traduce in una diluizione di 10.000 volte dei trasformatori e utilizzare questa piastra per calcolare l'efficienza di trasformazione. Eseguire l'incubazione delle piastre a 30 °C per 14-16 ore.
- Placcare il resto della reazione di trasformazione diffondendo 100 μL di cellule recuperate su ogni piastra per un totale di 10 piastre di agar preri warmed da 15 cm LB + ampicillina. Incubare le piastre per 14-16 ore a 30 °C. Da notare che la crescita a 30 °C riduce al minimo la ricombinazione tra le due ripetizioni a terminale lungo all'interno del plasmide virale.
- Per valutare il successo della clonazione, calcolare l'efficienza di trasformazione. Contare il numero di colonie sulla piastra di diluizione contenente trasformatori della libreria di sgRNA o legatura di controllo negativo (piastre di 10 cm, passo 1.8.2). Il mix di controllo negativo non dovrebbe avere colonie o solo pochissime. Per ottenere il numero totale di colonie, moltiplicare il numero di colonie per 10.000.
NOTA: Il numero totale contato di colonie rappresenta una copertura della biblioteca che dovrebbe essere di almeno 200 colonie per sgRNA.- Assicurati che una libreria con 2000 sgRNA abbia almeno 400.000 colonie. Nel caso in cui non ci siano abbastanza colonie, ripeti e imposta più elettroporazione.
- Controllo qualità: Dalla piastra di diluizione, raccogliere 20 colonie e aggiungere ogni colonia a un singolo tubo di coltura contenente 3 ml di supporti LB + ampicillina. Incubare tutti i 20 tubi durante la notte a 30 °C tremando a 250 giri/min. Purificare il DNA plasmide utilizzando il kit di mini-preparazione secondo le istruzioni del produttore e la sequenza Sanger tutti i 20 campioni di DNA plasmide per verificare che ogni campione abbia una sequenza di sgRNA diversa utilizzando il primer U6 (5'-GAG GGC CTT CTT ATG ATT CC-3').
- Raccogli le colonie da ogni piatto di 15 cm.
- Aggiungere 7 mL di mezzo LB, quindi raschiare le colonie dalla piastra LB Agar con uno spargitore di cellule. Utilizzare una pipetta da 10 ml per trasferire le cellule in un pallone conico sterile da 2 L. Ripetere per tutti i piatti e i batteri della piscina nel pallone da 2 L. Incubare il pallone per tremare 2-3 ore a 30 °C. Centrifugare la coltura e raccogliere il pellet.
- Purificare il DNA plasmide utilizzando un kit di purificazione maxi-plasmide.
- Controllo di qualità aggiuntivo: utilizzare 1 ng di DNA plasmide della libreria di sgRNA maxi-preparazione ed eseguire una reazione PCR utilizzando primer di sequenziamento di nuova generazione secondo le istruzioni del produttore. La rappresentazione di tutti gli sgRNA nella libreria può essere verificata tramite una piattaforma di sequenziamento profondo.
2. Produzione di lentivirus ad alto formicolio adatto alla trasduzione in vivo
NOTA: eseguire tutti i passaggi descritti in questa sezione del protocollo in una struttura BSL2+ in un cabinet di biosicurezza di classe II, tipo A2. 293FT e in particolare le celle di imballaggio 293NT consentono una maggiore produzione di virus. Utilizzare celle a passaggio basso (
- Preparare cellule di imballaggio virali.
- Il primo giorno, le cellule hek293T a piastre di piastra HEK293T su 6 piastre di poli-L-lisina rivestite di 15 cm al 35% di confluenza nei mezzi di crescita (DMEM + 10% FBS + 1% di soluzione antibiotica penicillina-streptomicina (w/v)). Incubare le cellule durante la notte a 37 °C, 5% CO2.
- Una volta che le cellule placcate sono confluenti al 70-80% e distribuite uniformemente, sostituire i mezzi di crescita con 25 mL di siero e mezzi DMEM senza antibiotici.
- Aggiungere lentamente i supporti ai lati dei contenitori durante l'alimentazione, poiché le cellule HEK293T e 293NT possono staccarsi facilmente dalla plastica di coltura tissutale
- Preparare il mix di trasfezione.
- Aggiungere 65 μg di plasmidi di imballaggio lentivirale psPAX2, 43 μg di VSV-G esprimendo busta plasmide pMD2.G, 65 μg di plasmide libreria sgRNA e 240 μL di 1 mg/mL polietileneimina (PEI) a 6 mL di DMEM e vortice.
NOTA: utilizzare sempre una bottiglia di DMEM non aperta o aperta molto di recente per la trasfezione, poiché il pH in questo passaggio è fondamentale e DMEM diventa più semplice nel tempo una volta aperto. - Incubare per 15 minuti a temperatura ambiente. Questa miscela di trasfezione è sufficiente per le celle placcate su piastre da 6 x 15 cm con superficie di 870 cm2. Scalare la miscela di trasfezione in base alle esigenze dell'esperimento.
- Aggiungere 65 μg di plasmidi di imballaggio lentivirale psPAX2, 43 μg di VSV-G esprimendo busta plasmide pMD2.G, 65 μg di plasmide libreria sgRNA e 240 μL di 1 mg/mL polietileneimina (PEI) a 6 mL di DMEM e vortice.
- Trasfezione di cellule virali da imballaggio.
- Dopo 15 minuti di incubazione, trasfeziona ciascuna delle piastre di 15 cm aggiungendo 1 mL della miscela di trasfezione dropwise in tutta la piastra. Incubare a 37 °C, 5% CO2 per 8 h.
- Dopo l'incubazione, rimuovere il supporto privo di siero contenente la miscela di trasfezione e aggiungere 30 mL di mezzi di coltura regolari alle piastre e alla coltura di 15 cm per 48 ore a 37 °C, 5% CO2.
- Dopo 48 ore, raccogliere il supernatante virale e filtrare attraverso il filtro PVDF da 0,45 μm. Conservare 1 mL del supernatante virale e conservare a -80 °C per il controllo qualità e la titolazione virale.
- Concentrare il supernatante virale mediante centrifugazione a gradiente di saccarosio in centrifuga ad alta velocità utilizzando la testa del rotore SW28.
- Tubi prime ultracentrifuge lavandoli con il 70% di etanolo, seguiti da 3 risciacqui con PBS. Distribuire uniformemente circa 30 ml del supernatante virale in ciascuno dei tubi a 6 ultracentrifuge.
- Pipettare delicatamente sul fondo di ciascun tubo 4 ml della soluzione di saccarosio al 20% (20 g di saccarosio in 100 mL di PBS). Posizionare i sei tubi ultracentrifugo nei sei secchi oscillanti SW28 e bilanciare con precisione ogni coppia opposta di secchi aggiungendo o rimuovendo il supernatante virale.
- Centrifugare il supernatante virale a 4 °C per almeno 2 ore e fino a 4 ore a 80.000 x g e 4 °C. Una volta eseguita la centrifugazione, scartare con cura il supernatante.
- Scolare il supernatante rimanente posizionando tubi ultracentrifugo capovolti su un tovagliolo di carta morbida sterile per almeno 2 minuti e pulire eventuali goccioline di supernatante all'interno del tubo utilizzando un tovagliolo di carta morbido per sbarazzarsi di qualsiasi mezzo residuo. Un piccolo pellet bianco grigiastro potrebbe essere visibile nella parte inferiore del tubo.
- Aggiungere pbs fresco e freddo da 20-25 μL ad ogni tubo. Sigillare i tubi con pellicola di paraffina e incubare i tubi in posizione verticale sul ghiaccio con un delicato scuotimento su uno shaker orbitale per ~ 2 ore.
- Utilizzando una pipetta da 20 μL, spodesare e rimescolare con cura il pellet del primo tubo, facendo attenzione a non formare bolle. Trasferire il mezzo al tubo successivo e ripetere il processo fino a quando tutti i pellet del virus non sono stati slogati e combinati in un unico tubo di circa 120-150 μL di volume. Incubare questa sospensione virale ad alto ribaltamento per altri ~ 2 h sul ghiaccio con delicati scuotimenti.
- Trasferire la sospensione virale in un tubo di microcentrifugo da 1,5 ml e ruotare il tubo a 4 °C in una centrifuga da tavolo refrigerata e pre-raffreddata a 12.000 x g per 2 min. Aliquotare attentamente il supernatante virale chiaro come aliquote da 10 μL in tubi separati da 0,2 ml e conservare a -80 °C. Scartare il pellet nella parte inferiore del tubo in quanto ciò ostruirà l'ago durante le iniezioni embrionali.
- Titolazione della libreria di lentivirus CRISPR in pool
NOTA: La sospensione virale risultante deve produrre una concentrazione approssimativa di 2.000 volte e la soluzione virale risultante e deve avere un titolo virale di10 7-10 9, che è abbastanza buono per oltre 100 interventi chirurgici E9.5 descritti di seguito.- Semi R26-Lox-STOP-Lox-td Fibroblasti embrionali di topo di pomodoro (MEF) o R26-Lox-STOP-Lox-tdCheratinociti primari del pomodoro o qualsiasi altra linea cellulare Cre-reporter in una piastra del pozzo 2 x 6.
- Una volta che la cellula raggiunge ~ 40% di confluenza, le cellule sono pronte per la trasduzione. A quel tempo, determinare il numero di cellule all'interno di un pozzo per consentire il calcolo del ttarettare virale in seguito. Cambiare i supporti delle cellule cre-reporter in mezzi di crescita integrati con polibrene 10 μg/mL (=bromuro di esadimetalina).
- Diluire 1 μL di virus concentrato con 2 mL di mezzi di crescita. Aggiungere 0, 10, 50, 200 μL di sospensione virale diluita e 50, 200 μL di virus non concentrato dal passaggio 2.4 e mescolare le piastre e incubare durante la notte a 37 °C, 5% CO2.
- Rimuovere i mezzi di crescita contenenti virus il giorno successivo, lavare le cellule con PBS e sostituirli con normali mezzi di crescita. Il supernatante a questo punto è ancora considerato uno spreco virale e deve essere smaltito in conformità con le normative istituzionali BSL2.
- Circa 36-48 ore dopo la trasduzione, valutare la ricombinazione mediata da Cre della cassetta Lox-STOP-Lox e l'espressione di tdTomato da parte di FACS. Calcola il titer virale. Calcolare le unità di formazione delle colonie (cfu)/mL utilizzando la seguente formula:
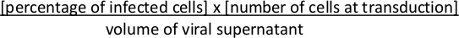
NOTA: Il confronto tra la sospensione virale concentrata con il suo supernatante virale non concentrato consentirà di stimare il successo della (1) produzione virale e (2) della concentrazione virale. In alternativa, il titer virale può essere stimato utilizzando il metodo qRT-PCR10. La produzione e la concentrazione su larga scala di lentivirus possono anche essere eseguite utilizzando una concentrazione in due punti con una concentrazione di cartucce seguita da ultracentrifugazione comedescritto in precedenza 2.
3. Chirurgia e iniezione guidate ultra-sonore
NOTA: Questa tecnologia è stata adattatada 4,11. La microiniezione orientata alla trasduzione dell'epitelio superficiale deve essere eseguita al giorno embrionale E9.5, quando l'ectoderma superficiale è costituito da un singolo strato e prima della formazione del periderm a partire dall'E10, il che impedirebbe la trasduzione di questo strato basale. Preferibilmente impostare i topi il venerdì, in modo che il primo giorno possibile con embrioni E9.5 sia il lunedì successivo. Utilizzare topi Rosa26-Lox-STOP-LOX-Cas9-GFP (Jackson Laboratory #024857) per un'efficienza CRISPR/Cas9ottimale 12.
- Impostare topi maschi e femmine appropriati per gravidanze a tempo 10 giorni prima della data di intervento desiderata.
- Collegare i topi nei giorni successivi per identificare topi potenzialmente gravidi. Confermare la gravidanza il giorno prima dell'intervento chirurgico pianificato (cioè E8.5) utilizzando gli ultrasuoni.
- Preparare la piastra di Petri modificata.
- Utilizzando il nastro adesivo a doppia parte, incollare la membrana in silicone sul fondo della piastra di Petri che copre l'apertura circolare. Utilizzando forbici affilate, tagliare un'apertura ovale di 2 x 10 mm nella membrana in silicone.
- Preparazione del sistema di microiniezione
- Preparare aghi di iniezione da un capillare di vetro a parete spessa utilizzando un estrattore di micropipette con le seguenti impostazioni: Pressione = 200; Calore = 769; Pull = 0; Velocità = 140; Tempo = 100.
- Uso di forbici fini per tagliare la punta dell'ago al livello in cui il suo diametro è ~ 30 μm. Smussare l'ago utilizzando affilatore ad ago a 20° su una piastra abrasiva di qualità fine con bagnatura regolare per 20 minuti.
- Sterilizzare l'ago di microiniezione spingendo il 70% di etanolo utilizzando una siringa da 10 ml con un ago da 26 G1/2. Utilizzando una siringa da 10 ml e un ago da 26 G1/2 caricato con olio minerale, riempire l'ago di microiniezione. Assicurarsi che non ci siano bolle nell'ago di microiniezione.
- Montare l'ago del microiniettore sul micromanipolatore secondo le istruzioni del produttore.
- Caricare l'ago con la libreria di sgRNA virale.
- Pipetta 10 μL di sospensione virale su un parafilm fissato su una superficie piana.
- Usando il micromanipolatore, espellere l'olio minerale. La presenza di un piccolo volume di olio nell'ago impedirà al lentivirus di contattare il pistone. Caricare circa 4-5 μL di sospensione virale per aspirazione lenta senza bolle d'aria.
- Anestetizzare il topo incinta usando la camera di induzione dell'isoflurane. Impostare il regolatore di ossigeno su 1 L/min e vaporizzatore di isoflurane al 2%. Per determinare se l'animale è anestetizzato, controllare con il dito del piedi dopo ~ 3-5 minuti nella camera di induzione dell'isoflurane.
- Posizionare il lato ventrale animale anestetizzato sullo stadio di chirurgia riscaldata (40 °C) e applicare nastri chirurgici per tenere le zampe in posizione con il tubo di anestesia naso-cono per un apporto costante di isoflurane e ossigeno fino al completamento dell'intervento chirurgico.
- Confermare la gravidanza E9.5 se non è stata eseguita all'E8.5.
- Applicare una piccola quantità (~ 1/2 cucchiaino) di una crema per la depilazione sull'addome e stenderla su un'area di 3 x 3 cm utilizzando un applicatore con punta di cotone.
- Dopo 2-3 minuti, rimuovere delicatamente la crema con garza o tessuto e pulire l'area utilizzando PBS e 70% di etanolo.
- Utilizzando la sonda ad ultrasuoni e il gel, controllare il topo incinta per il giorno embrionale E9.5. Naturalmente, questo può essere fatto anche il giorno prima all'E8.5 per risparmiare tempo nel giorno dell'intervento chirurgico effettivo.
- Rimuovere il gel ad ultrasuoni e pulire l'area utilizzando BPS e quindi il 70% di etanolo per disinfettare.
- Iniettare 0,02 cc di Buprenorfina analgesica per via sottocutanea nella diga del topo.
- Esporre chirurgicamente l'utero
- Usando forcep e forbici sterili, fai un'incisione verticale di ~ 2 cm nella pelle del topo.
- L'uso di forcep smussate separa la pelle intorno all'incisione dal peritoneo.
- Usando forcep sterili e forbici, tagliare il peritoneo.
- Usando le forcep smussate, estrarre delicatamente il corno dell'utero sinistro e destro e contare gli embrioni.
- Reinserimento la maggior parte delle corna uterine ma lasciare esposta l'estremità distale di un corno uterino contenente 3 embrioni.
- Usando le forcep smussate, spingere e tirare delicatamente l'utero con i 3 embrioni attraverso l'apertura nella membrana siliconica della piastra di Petri modificata.
- Stabilizzare la piastra di Petri utilizzando cubetti di argilla modellante.
- Stabilizzare l'utero con i tre embrioni all'interno della piastra di Petri utilizzando uno stampo in silicone. Appiattire il tappo di silicone sul lato degli embrioni usando un coltello affilato.
- Riempire la piastra di Petri con PBS sterile. Lavare la membrana di silicio sul fondo della piastra di Petri con la pancia della diga incinta evitando così eventuali perdite.
- Spostare la testa ad ultrasuoni nella piastra di Petri ~ 0,5 cm in cima all'embrione superiore e regolare lo stadio in modo che la cavità amniotica diventi chiaramente visibile nella vista ad ultrasuoni.
- Allineare accuratamente l'ago dell'iniettore nella piastra di Petri modificata. Usando il micromanipolatore, posizionare la punta dell'ago entro ~ 5 mm dall'embrione superiore. Quindi attivare o disattivare l'ago avanti e indietro e spostarsi nel piano dove la sua punta appare più luminosa nell'immagine ad ultrasuoni.
- Usando il micromanipolatore spingere l'ago attraverso la parete uterina nella cavità amniotica.
- Iniettare lentivirus contenente libreria di sgRNA.
- Pre-programmare l'iniettore a 62 nL e rallentare la velocità di iniezione. Il microiniettore può essere programmato per iniettare volumi specifici a una velocità lenta o veloce.
- Premere il pulsante Inietta 8 volte per un totale di 496 nL per embrione. Regolare il volume in base alle esigenze richieste e al titer virale. Un titer virale di 1 x 108 pfu/mL e 496 nL volume di iniezione si tradurrà in circa il 30% di infettività.
- Ripetere la stessa procedura per altri due embrioni.
- Sollevare la testa ad ultrasuoni e rimuovere la spina in silicone.
- Spingere delicatamente i 3 embrioni nell'addome del topo usando uno scambio di cotone sterile.
- Estrarre delicatamente i prossimi 3 embrioni e ripetere la procedura di iniezione fino a quando non viene iniettato il numero desiderato di embrioni.
NOTA: Non superare i 30 minuti di anestesia poiché le dighe gravide hanno molte più probabilità di abortire i loro embrioni oltre quel tempo. Un chirurgo esperto può iniettare fino a 12 embrioni entro 30 minuti di intervento chirurgico.
- Sollevare la testa ad ultrasuoni, rimuovere il micromanipolatore con l'ago, aspirare il PBS e rimuovere la piastra di petri. Utilizzando salviette sterili, assorbire qualsiasi PBS che potrebbe aver accumulato nella cavità addominale.
- Chiudere l'incisione peritoneale utilizzando suture assorbibili. Utilizzare due graffette per chiudere l'incisione nella pelle addominale.
NOTA: Durante l'esecuzione di interventi chirurgici ultra-sonori in utero, si deve fare la massima attenzione per mantenere un ambiente pulito, mantenere la sterilità il più possibile ed evitare eventuali contaminanti. Se più di un intervento chirurgico deve essere eseguito lo stesso giorno, è importante sterilizzare gli strumenti di dissezione utilizzando uno sterilizzatore di perline e pulire altri apparecchi che incontrano tessuto di topo pulito con etanolo. Ciò aumenta notevolmente la maggiore sopravvivenza degli embrioni dopo l'intervento chirurgico. Il tasso di sopravvivenza per il metodo chirurgico qui descritto è costantemente compreso tra l'80 e il 100%. - Cure post-operatorie.
- Lasciare che la femmina incinta si riprenda in una gabbia riscaldata e osservi per 15-30 minuti.
- Controlla il canale vaginale per il sangue, che è un primo segno di aborto e complicazioni. Per alleviare il dolore postoperatorio, somministrare l'analgesico due volte al giorno per due giorni dopo l'intervento chirurgico.
- Identificare embrioni trasdotti/neonati rosa26-Lox-STOP-Lox-Cas9-GFP utilizzando un microscopio sezionante fluorescente, una torcia proteica fluorescente e occhiali filtranti o genotipizzazione per particelle virali integrate in clip dell'orecchio o della coda.
NOTA: I topi sono meglio identificati usando il microscopio fluorescente fino al giorno 3 postnatale prima che i capelli inizino a crescere. - Per garantire una copertura sufficiente per la libreria CRISPR, calcolare la copertura in base ai seguenti parametri: ectoderma della superficie del mouse E9.5 è costituito da ~ 150.000 celle; la trasduzione di ~20% si traduce in un tasso minimo di doppia infezione (~1/10 doppia cellula infetta)4,5,11; con il 20% di infettività, ogni embrione ha 30.000 cellule infette. Assicurarsi che almeno 100 singole cellule siano trasdotto con un dato sgRNA. Ad esempio, un pool di 2000 sgRNA richiede 2 x 105 cellule o 6-7 animali.
4. Procedura di sequenziamento profondo
- Su endpoint sperimentali come la formazione tumorale o qualsiasi altro fenotipo, sacrificare il topo e raccogliere il tessuto di interesse. Utilizzare il kit di estrazione del DNA del sangue e dei tessuti per purificare il DNA genomico seguendo il protocollo del produttore. Prima di iniziare l'estrazione del DNA dai tumori, pulire il banco di lavoro con la cancellazione del DNA e lavorare lontano dall'area di laboratorio in cui vengono gestiti i plasmidi.
NOTA: È importante generare un campione di riferimento per consentire il calcolo delle variazioni di piegatura dello sgRNA nel tempo. Gli embrioni trasdotti 3 giorni dopo l'infezione o le cellule trasdutte (vedi fase 2. 6) possono servire come campione di riferimento per determinare la rappresentazione dello sgRNA nella libreria originale. - Utilizzare il DNA genomico da 100 ng a 1,5 μg come modello in una reazione PCR 1 di pre-amplificazione da 50 μL utilizzando i primer esterni elencati nella tabella 1 e il programma PCR delineato nella tabella 2. Impostare una reazione sufficiente per ottenere la copertura desiderata.
NOTA: Impostare la reazione PCR della decodifica a barre in un'area dedicata separata del laboratorio o in una cappa di coltura tissutale che viene pulita accuratamente dalla cancellazione del DNA per evitare qualsiasi contaminazione. Eseguire un controllo negativo per ogni piastra PCR con acqua come modello per assicurarsi che non vi sia contaminazione. - Mettere in comune tutta la reazione PCR 1 ed eseguire 20 μL su un gel di agarosio di controllo dell'1,5% per verificare l'amplificazione riuscita di una banda di 600 bp.
- Utilizzare 5 μL della reazione PCR1 in pool come modello in una reazione di 50 μL di PCR 2 utilizzando una combinazione di primer con codice a barre univoca elencata nella tabella 3 e nel programma PCR delineato nella tabella 4. Eseguire 50 μL di reazione PCR 2 su un gel di agarosio al 2% e purificare la banda da 200 bp utilizzando un kit di estrazione del gel secondo le istruzioni del produttore.
NOTA: La fase di pre-amplificazione è necessaria solo per l'amplificazione di un tessuto trasdotto complesso. Se amplificando un singolo tumore che presumibilmente si è sviluppato forma una cellula clonale e quindi contiene un solo sgRNA, si può saltare la pre-amplificazione PCR1 e procedere immediatamente con PCR2. - Quantificare la concentrazione di DNA utilizzando la quantificazione fluorometrica e inviare il sequenziamento profondo alla struttura di sequenziamento (ad esempio, 1 milione di letture per tumore).
- Utilizzare Bowtie v1.2.3, che consente solo allineamenti non ancorati, allineare le letture sequenziate alla libreria sgRNA13. Create un indice bowtie utilizzando il comando bowtie-build che immette le sequenze di sgRNA in formato fasta. Mappare le letture utilizzando il comando bowtie con le opzioni -m 2 -v 1, che consente al massimo due disallineamenti durante l'allineamento di lettura e scarta le letture che allineano più di una sequenza di sgRNA della libreria. In genere, più dell'80% delle letture si allinea sotto questi parametri.
- Utilizzare il comando Conteggio MAGeCK per ottenere la tabella di conteggio delle lette utilizzando il file allineato come input14. La tabella del conteggio degli sgRNA può essere utilizzata per l'analisi a valle come, determinare gli sgRNA differenziali (MAGeCK) e trovare geni essenziali (BAGEL).
Risultati
La figura 1A mostra la progettazione degli oligonucleotidi per il multiplexing di diverse librerie CRISPR personalizzate in modo conveniente in un singolo chip oligo da 12k o 92k. Una volta selezionati gli sgRNA (codificati a colori blu), gli oligonucleotidi sono progettati con siti di restrizione (BsmBI di colore arancione) e coppie di primer PCR specifiche della libreria (codificate a colori verdi). Diverse librerie possono essere progettate utilizzando una combinazione unica di coppie di ...
Discussione
L'editing del genoma CRISPR/Cas9 è stato ampiamente utilizzato in studi in vitro e in vivo per studiare le funzioni geniche e i processi cellulari. La maggior parte degli studi in vivo utilizza cellule modificate dal gene CRISPR/Cas9 innestate in un modello animale (allografto o xenograft). Sebbene questo sia un potente strumento per studiare la genetica del cancro e le funzioni cellulari, manca ancora del microambiente dei tessuti nativi e potrebbe provocare ferite e / o risposte immunitarie.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto da una sovvenzione di progetto del Canadian Institute of Health Research (CIHR 365252), della Krembil Foundation e dell'Ontario Research Fund Research Excellence Round 8 (RE08-065). Sampath Kumar Loganathan è il destinatario di una borsa di studio della Canadian Cancer Society (BC-F-16#31919).
Materiali
| Name | Company | Catalog Number | Comments |
| 0.45 micron filter | Sigma | S2HVU02RE | |
| 12k or 92k oligo chip | Customarray Inc. (Genscript) | ||
| 15 cm cell culture plates | Corning | ||
| 293FT | Invitrogen | R70007 | |
| 293NT | Systems Biosciences | LV900A-1 | |
| Alkaline phosphatase | NEB | M0290L | |
| Amplicillin | Fisher Scientific | BP1760-25 | |
| ATP | NEB | 9804S | |
| BsmBI | NEB | R0580L | |
| Chromic gut suture | Covidien | ||
| Deep sequencing (Next-Seq or Hi-Seq) | Illumina | ||
| DNA-cleanup kit | Zymo Research | D4008 | |
| DNAesy Blood and Tissue DNA extraction kit | Qiagen | 69506 | |
| Endura electrocompetent cells | Lucigen | 60242-1 | |
| Glass Capillaries | Drummond | 3-000-203-G/X | |
| HEK293T cells | ATCC | CRL-3216 | |
| High-Speed Centrifuge | Beckman Coulter | MLS-50 | |
| LB Agar | Wisent Technologies | 800-011-LG | |
| Micropipette puller | Sutter Instrument | P97 | |
| Mineral oil | Sigma | M5904 | |
| Mini-prep plasmid Kit | Frogga Bio | PDH300 | |
| Mouse oxygen anaesthesia system | Visual Sonics | ||
| Nanoject II micromanipulator | Drummond | ||
| NEBuffer 3.1 (Buffer for BsmBI) | NEB | R0580L | |
| Needle sharpener | Sutter Instrument | BV-10 | |
| Oligo cleanup kit | Zymo research | D4060 | |
| PAGE purified illumina sequencing primer | IDT DNA | ||
| PEI (polyethyleneimine) | Sigma | 408727-100ML | |
| Permoplast modeling clay | |||
| Petridish with central opening | Visual Sonics | ||
| pMD2.G | Addgene | 12259 | |
| psPAX2 | Addgene | 12260 | |
| Q5 Polymerase 2x Master mix | NEB | M0494L | |
| Qubit Fluorometric Quantification | Invitrogen | Q33327 | |
| Semicircular Silicone plug | Corning | ||
| Silicone membrane | Visual Sonics | ||
| T4 DNA ligase | NEB | M0202L | |
| Ultra-centrifuge tubes | Beckman Coulter | 344058 | |
| Vevo2000 ultrasound system | Visual Sonics |
Riferimenti
- Beronja, S., Fuchs, E. RNAi-mediated gene function analysis in skin. Methods in Molecular Biology. 961, 351-361 (2013).
- Beronja, S., Livshits, G., Williams, S., Fuchs, E. Rapid functional dissection of genetic networks via tissue-specific transduction and RNAi in mouse embryos. Nature Medicine. 16, 821-827 (2010).
- Shalem, O., et al. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science. 343, 84-87 (2014).
- Beronja, S., et al. RNAi screens in mice identify physiological regulators of oncogenic growth. Nature. 501, 185-190 (2013).
- Loganathan, S. K., et al. Rare driver mutations in head and neck squamous cell carcinomas converge on NOTCH signaling. Science. 367, 1264-1269 (2020).
- Leemans, C. R., Snijders, P. J. F., Brakenhoff, R. H. The molecular landscape of head and neck cancer. Nature Reviews Cancer. 18, 269-282 (2018).
- Green, A. C., Olsen, C. M. Cutaneous squamous cell carcinoma: an epidemiological review. British Journal of Dermatology. 177, 373-381 (2017).
- Campbell, J. D., et al. pathway network, and immunologic features distinguishing squamous carcinomas. Cell Reports. 23, 194-212 (2018).
- Sanjana, N. E., Shalem, O., Zhang, F. Improved vectors and genome-wide libraries for CRISPR screening. Nature Methods. 11, 783-784 (2014).
- Geraerts, M., Willems, S., Baekelandt, V., Debyser, Z., Gijsbers, R. Comparison of lentiviral vector titration methods. BMC Biotechnology. 6, 34 (2006).
- Schramek, D., et al. Direct in vivo RNAi screen unveils myosin IIa as a tumor suppressor of squamous cell carcinomas. Science. 343, 309-313 (2014).
- Platt, R. J., et al. CRISPR-Cas9 knockin mice for genome editing and cancer modeling. Cell. 159, 440-455 (2014).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10, 25 (2009).
- Li, W., et al. MAGeCK enables robust identification of essential genes from genome-scale CRISPR/Cas9 knockout screens. Genome Biology. 15, 554 (2014).
- Winters, I. P., Murray, C. W., Winslow, M. M. Towards quantitative and multiplexed in vivo functional cancer genomics. Nature Reviews Genetics. 19, 741-755 (2018).
- Rogers, Z. N., et al. Mapping the in vivo fitness landscape of lung adenocarcinoma tumor suppression in mice. Nature Genetics. 50, 483-486 (2018).
- Chow, R. D., et al. AAV-mediated direct in vivo CRISPR screen identifies functional suppressors in glioblastoma. Nature Neuroscience. 20, 1329-1341 (2017).
- Wang, G., et al. Mapping a functional cancer genome atlas of tumor suppressors in mouse liver using AAV-CRISPR–mediated direct in vivo screening. Science Advances. 4, 5508 (2018).
- Chan, K., Tong, A. H. Y., Brown, K. R., Mero, P., Moffat, J. Pooled CRISPR-based genetic screens in mammalian cells. Journal of Visualized Experiments. (151), e59780 (2019).
- Hart, T., et al. High-resolution CRISPR screens reveal fitness genes and genotype-specific cancer liabilities. Cell. 163, 1515-1526 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon