Method Article
מתן יעיל של הפרעת RNA דרך הפה (RNAi) ליתושים אנופלס גמביה בוגרים
In This Article
Summary
מתן אוראלי של dsRNA המיוצר על ידי חיידקים, שיטת אספקה להפרעות RNA (RNAi) המשמשת באופן שגרתי ב - Caenorhabditis elegans, יושם כאן בהצלחה על יתושים בוגרים. השיטה שלנו מאפשרת מחקרים חזקים בגנטיקה הפוכה ומחקרי וקטורים חוסמי העברה ללא שימוש בהזרקה.
Abstract
הפרעת RNA הייתה כלי בשימוש רב לניתוח גנטי הפוך במשך שני עשורים. אצל יתושים בוגרים, מתן RNA דו-גדילי (dsRNA) הושג בעיקר באמצעות הזרקה, הדורשת זמן משמעותי ואינה מתאימה ליישומי שדה. כדי להתגבר על מגבלות אלה, כאן אנו מציגים שיטה יעילה יותר להפעלה חזקה של RNAi על ידי אספקה דרך הפה של dsRNA לגמביה אנופלס בוגרת. dsRNA ארוכים יוצרו בזן Escherichia coli HT115 (DE3), ותרחיף מרוכז של חיידקים המכילים dsRNA מומתים בחום ב-10% סוכרוז הוצע על כדורי צמר גפן ad-libitum ליתושים בוגרים. כדורי צמר גפן הוחלפו כל יומיים למשך הטיפול. שימוש בשיטה זו כדי להתמקד בדאבלסקס (גן המעורב בהתמיינות בין המינים) או בראש מזלג (המקודד גורם שעתוק של בלוטת הרוק) הביא לביטוי מופחת של גני מטרה ו/או לאות אימונופלואורסצנציה של חלבון, כפי שנמדד על ידי PCR כמותי בזמן אמת (qRT-PCR) או מיקרוסקופיה קונפוקלית פלואורסצנטית, בהתאמה. נצפו גם פגמים במורפולוגיה של בלוטת הרוק. שיטה גמישה מאוד זו, ידידותית למשתמש, בעלות נמוכה וחסכונית בזמן של אספקת dsRNA יכולה להיות ישימה באופן נרחב לגני מטרה החשובים לפיזיולוגיה של וקטור חרקים ומעבר לה.
Introduction
מחלות רבות מועברות על ידי יתושים, מה שהופך את המחקר של פיזיולוגיה יתושים וגנטיקה למשימה חשובה. השימוש ב-RNAi באורגניזמים אלה בלט ב-20 השנים האחרונות ואיפשר אפיון תפקודי של גנים רבים של יתושים 1,2,3,4,5. הטכניקה הנפוצה ביותר להעברת dsRNA הייתה microinjection, אשר יש את החסרונות כי זה יכול לפצוע את היתושים דורש זמן ומאמץ משמעותי. שיטות אספקה דרך הפה עבור RNAi נבדקו, אך בעיקר בשלב הזחל של היתושים 6,7,8,9. אספקה דרך הפה של dsRNA אצל יתושים בוגרים לא נחקרה במלואה ויכולה להיות כלי שימושי לחקר הביולוגיה הווקטורית והבקרה הווקטורית.
מלריה מועברת על ידי יתושי אנופלס כאשר יתוש נקבה נגועה לוקחת ארוחת דם מפונדקאי לא נגוע ומזריקה רוק המכיל טפילי מלריה10. כדי שבסופו של דבר יועבר ברוק של יתוש, הטפיל חייב להתגבר על מכשולים רבים, כולל התחמקות ממערכת החיסון של היתושים, חציית מחסום המידגוט ופלישה לבלוטות הרוק11. ארכיטקטורת בלוטת הרוק של יתושים (SG) היא המפתח לפלישת הטפילים וארכיטקטורה זו נשלטת הן על ידי גורמי שעתוק מרכזיים המבטאים בלוטת הרוק והן על ידי גורמים קובעים של דימורפיזם מיני. מספר גורמי שעתוק שמורים מאוד נדרשים לצורך אפיון תאי ותחזוקה הומאוסטטית של בלוטות הרוק ולייצור והפרשת חלבוני רוק הפועלים בהזנתדם 12,13,14. ראש מזלג (Fkh) הוא גורם שעתוק סליל מכונף המתפקד כרגולטור עיקרי של מבנה ותפקוד SG של חרקים (בהתבסס על מחקרים בזבובי פירות ובעש תולעי המשי)15,16,17,18,19,20. ב-Drosophila SGs, Fkh מתפקד עם Sage, גורם שעתוק סליל-לולאה-סליל בסיסי ספציפי ל-SG (bHLH), כדי לקדם את הישרדות ה-SG ואת ייצור הרוק19. מווסת שותף חשוב וחיובי של ייצור הרוק בדרוזופילה הוא CrebA, גורם שעתוק רוכסן לאוצין שנחקר היטב, אשר מווסת את הביטוי של גנים של מסלול הפרשה 21,22,23. יש גם מידה חזקה של התמיינות מורפולוגית בבלוטות הרוק הנשיות שככל הנראה ממלאת תפקיד מפתח, לא רק בהזנת דם אלא גם ביכולתם של טפילים לפלוש לרקמה זו24.
לרבים מהגנים המעורבים בקביעת הישרדות בלוטת הרוק, המבנה, הפיזיולוגיה והדימורפיזם המיני יש פרופילי ביטוי מרחביים מורכבים 25,26,27, ושיטות המסירה המסורתיות של dsRNA להשראת RNAi אינן תמיד יעילות בהתמקדות בגנים מסוג זה או ברקמות אחרות. עם זאת, אספקה דרך הפה של dsRNA בשלב הזחל Aedes aegypti ו- An. gambiae יתושים שימשה בהצלחה כדי להשתיק את הצורה הספציפית לנקבה של הגן dsx 9,28. מחקרים קודמים שהשתמשו ב-dsRNA בבלוטות הרוק של היתושים מצאו שלמרות שנדרשו כמויות גדולות של dsRNA, אפקט ההשתקה היה ארוך יחסית (לפחות 13 יום)29. כאן נבדקה היכולת של זן E. coli HT115 (DE3) המבטא dsRNA ספציפי לרצף עבור dsx, fkh או CrebA לגרום להשתקת RNAi של גנים אלה אצל יתושים נקבות בוגרות. מתן אוראלי של dsRNA גרם להפלת גנים ב- An. gambiae, עם הפחתה ברורה ברמות mRNA ועם פנוטיפים התואמים את אובדן התפקוד של גנים אלה. לפיכך, סביר להניח שגישה זו תעבוד כדי להפיל את תפקודם של מגוון גנים של בלוטות הרוק.
Protocol
1. שיבוט dsRNA לתוך וקטור ביטוי E. coli
- בחר את רצף גני המטרה כדי להכניס לווקטור המתאים לביטוי dsRNA. אחזר את ערכי הביטויים מתוך Vectorbase.org באמצעות השיטה הבאה.
- חפש גן בעל עניין (לדוגמה, טבלה 1) בתיבת החיפוש של דף הבית.
- בדף הגן שנוצר, נווט אל 8. סעיף תמלול .
- חפשו את הניסויים הרלוונטיים המפורטים בביטוי גנים RNA-seq ו-microarray.
- תמלל ערכים בעלי עניין לתוכנת הגיליון האלקטרוני וצור טבלת נתונים.
- בחר פלסמיד זמין מסחרית עם לפחות מקדם T7 אחד לשימוש. אם לפלסמיד שנבחר יש רק מקדם T7 אחד (כפי שעושים רוב הפלסמידים המסחריים), כלול מקדם T7 שני בפריימר ההפוך שישמש להגברת ה-dsDNA עבור הגן המעניין.
הערה: ניתן לבחור את רצף ה- dsRNA עבור גני המטרה באמצעות יישום האינטרנט E-RNAi לתכנון ריאגנטים RNAi30. ניתן לתכנן dsRNA ארוך (כ-400 bp) או dsRNA קצר שיער (shRNA) על סמך רצפי גנים ספציפיים. רצפים אלה צריכים להיות מוגברים ומרוצפים לאישור זהות לפני השיבוט. אזורי הגנים הנבחרים, הפלסמידים והמקדמים ששימשו במחקר זה מפורטים בקובץ משלים 1. - בצע שיבוט על פי הליך פשוט של שלב אחד שתואר קודם לכן 9,31. לשם כך, לטהר את מוצר ה-PCR ולפגוע בדנ"א הפלסמידי הליניארי. השתמש בתוצר של הקשירה לטרנספורמציה של הלם חום של תאי E. coli 32 מוכשרים. בחר את התאים שעברו שינוי צורה באמצעות סינון כחול/לבן. אשר את כיוון ההוספה באמצעות PCR של פריימר T7 ואשר את הרצף באמצעות פריימרים M13.
הערה: ניתן להשתמש בהקרנות לבנות/כחולות כאשר הפלסמיד שנבחר לטרנספורמציה נושא את הגן lacZ המקודד β-גלקטוזידאז. מושבות לבנות צריכות להכיל את התוספת הרצויה בתוך lacZ וניתן לבחור אותן כדי לאשר עוד יותר את הנוכחות והכיוון של רצף המטרה33. - טהרו את הפלסמיד מהטרנספורמציה הראשונה והשתמשו בו כדי לשנות את E. coli HT115 (DE3) המוכשר כפי שתואר קודם לכן34. לאחר אישור כי פלסמיד עם התוסף נמצא ב E. coli HT115 (DE3) המוסמך, לעשות גליצרול מניות של חיידקים לשימוש יחיד.
הערה: יש לרכוש או להכין dsRNA בקרה מתאים שאינו קשור לשימוש בכל ניסוי. במקרה זה, נעשה שימוש ברצף של הגן הלא קשור aintegumenta (נמלה) מ - Arabidopsis thaliana .
2. הכנת חיידקים מומתי חום המבטאים dsRNA
- לגדל תרבית ממושבת חיידקים אחת של זן E. coli HT115 (DE3) המכילה את ה-dsRNA המבטאת פלסמיד ב-50 מ"ל של ציר לוריא (LB) המכיל 100 מיקרוגרם/מ"ל של אמפיצילין ו-12.5 מיקרוגרם/מ"ל של טטרציקלין, על שייקר פלטפורמה (180 סל"ד) ב-37 מעלות צלזיוס למשך 12 שעות.
- דיללו את תרבית החיידקים (1:1000) ל-2x שמרי טריפטון (2x YT) המכילים 100 מיקרוגרם/מ"ל של אמפיצילין ו-12.5 מיקרוגרם/מ"ל של טטרציקלין.
- לגרום לייצור dsRNA על ידי הוספת 40 μM (ריכוז סופי) איזופרופיל β-D-1-thiogalactopyranoside (IPTG).
- כאשר התאים מגיעים ל- O.D.600 = 0.4, בערך לאחר 2 שעות של אינדוקציה ב-37 מעלות צלזיוס עם תסיסה ב-180 סל"ד, הכינו תרחיף מרוכז של חיידקים מומתים בחום כפי שתואר על ידי Taracena et al 9. גלול את התאים על ידי צנטריפוגה (4000 x g, 4 °C , 10 דקות) ושטיפת תאים בנפח אחד של מאגר נתרן פוספט (PBS).
- סובבו שוב באותם תנאים, השעו מחדש ב-PBS ל-1/100 מהנפח ההתחלתי, והניחו ב-70 מעלות צלזיוס למשך שעה אחת.
- הכינו 400 μL aliquots של החיידקים המומתים בחום ואחסנו את האליקוטים האלה בטמפרטורה של -20 מעלות צלזיוס עד לשימוש נוסף (אין לאחסן במשך יותר משבוע). תרחיף זה של חיידקים מומתי חום מכיל את ה-dsRNA הספציפי לניסויי ה-RNAi. בצע הליך זה הן עבור חיידקי ה-dsRNA של גן המטרה והן עבור בקרת ה-dsRNA שאינה קשורה לשימוש בכל ניסוי.
3. האכלת יתושים בחיידקים מומתי חום המבטאים dsRNA
- להפשיר אליקוט אחד של dsRNA (HT115 (DE3) תרחיף חיידקים) ולערבב עם 1.6 מ"ל של תמיסת סוכר של 12% המכילה 0.2% מתילפרבן.
- השרו כדור צמר גפן קטן בתמיסה זו והניחו את כדור הכותנה הספוג בתוך כלוב המכיל יתושים בני 5 ימים. יש לוודא שהיתושים ניזונים מתמיסה זו, והם קולטים בו זמנית גם את הסוכר וגם את החיידקים המכילים dsRNA.
- החליפו את כדור הצמר גפן המושרה בתמיסת סוכר dsRNA כל יומיים במשך 8 ימים רצופים.
- שמור על כלובי יתושים בתנאים קבועים, כלומר, 27 מעלות צלזיוס ו -80% לחות יחסית עם פוטופריוד של 12 שעות: 12 שעות: פוטו-אופן כהה, המופרד על ידי שחר של 30 דקות ותקופת דמדומים של 30 דקות.
4. בדיקת רמות ביטוי גנים ממוקדות
- מרדימים את היתושים באופן קר על ידי הנחת המיכל על קרח למשך דקה או עד שהיתושים מפסיקים לזוז. לאחר שהיתושים מורדמים, הניחו אותם על משטח קר כדי לבודד את הנקבות לצורך כריתה.
- מרססים 70% אתנול על היתושים ומניחים אותם על משטח זכוכית עם PBS. בעזרת זוג מלקחיים, יש לאבטח את ראש היתוש ביציבות ולמשוך את בית החזה באיטיות רבה, מה שמאפשר לבלוטות הרוק להשתחרר לתוך ה-PBS.
- שמור את בלוטות הרוק ב- PBS קר כקרח עד ש-10 אנשים נותחו. מאגר עשרה SGs למיצוי RNA בשיטת guanidinium thiocyanate-phenol-chloroform. השעו את גלולת ה-RNA ב-30 μL של מים נטולי RNase.
- השתמש ב-1 μL aliquot של ה-RNA שחולץ מה-SG בשלב הקודם, כדי לקרוא את הספיגה ב-260 וב-280 ננומטר ולחשב את ריכוז ה-RNA של כל דגימה על-ידי הכפלת גורם הדילול. יחס של 260/280 של ~2.0 מצביע על RNA באיכות טובה.
- השתמש ב-1 מיקרוגרם של הרנ"א המטוהר כדי לסנתז דנ"א משלים (cDNA) באמצעות ערכת שעתוק הפוכה מסחרית.
- בצע דילול של 1:10 של ה- cDNA כדי להכין תגובת RT-PCR על פי המלצות היצרן. עבור כל דגימה, הכינו תגובה לגן המטרה ובמקביל, הגדירו תגובה עם הגן משק הבית (HK). הגדר כל תגובה גנטית במשולש טכני כדי למנוע את ההשפעה של וריאציה אקראית מהשיטה.
הערה: כאן, הגן An. gambiae ריבוזומלי S7 (GeneBank: L20837.1) ואקטין (VectorBase: AGAP000651) שימשו כגנים HK. - השתמש בכל הפריימרים בריכוז סופי של 300 ננומטר, בעקבות האינדיקציות של היצרן הירוק-SYBR. הגבר עם תנאי PCR סטנדרטיים: 95 מעלות צלזיוס למשך 10 דקות, ואחריו 40 מחזורים של 15 שניות ב-95 מעלות צלזיוס ו-60 מעלות צלזיוס ב-60 מעלות צלזיוס.
הערה: כדי לכמת את ביטוי הגנים, נעשה שימוש בשיטת דלתא-דלתא-Ct (ΔΔCt). דלתא Ct (ΔCt) הוא ההבדל בין ה-Ct של גן המטרה לבין ה-Ct של גן משק הבית. ΔΔCt הוא ההבדל בין ΔCt של קבוצת הניסוי לבין ΔCt של קבוצת הביקורת35.
5. הערכה פנוטיפית: האכלה מוצלחת בדם
- כדי להעריך את היכולת להזין דם, קבעו קבוצות של 15 נקבות יתושים שטופלו במטרה ובקרת dsRNA על כלובים קטנים (קוטר 12 ס"מ) והרעיבו אותם במשך 4 שעות.
- באמצעות אמבטיית מים מסתובבת המוגדרת ל-37 מעלות צלזיוס, מאכילי יתושי זכוכית (קוטר 24 מ"מ) וקרום פרפילם, מציעים דם כבשים מושחת ליתושים.
הערה: ניתן לרכוש דם מספק מסחרי אשר שואב אותו באופן אספטי מבעלי חיים בריאים ותורמים ממוצא אמריקאי ודפיבריינטים באופן ידני ללא נוגדי קרישה או תוספים. - על ידי תצפית ישירה, ספירה ותיעוד של מספר ניסיונות הבדיקה להשיג בהצלחה ארוחת דם מחמש הנקבות הראשונות כדי להשתלב באופן מלא בכל קבוצה.
הערה: כדי למנוע שינויים מטבוליים משמעותיים אצל היתושים, שעלולים להפריע למשאבי האנרגיה המשפיעים על התנהגות מחפשי הדם, הרעב נשמר למינימום (4 שעות). כתוצאה מכך, לא כל יתוש היה מחפש בשקיקה את ארוחת הדם והגבלנו את מספר הנקבות הלכודות לחמש (שליש מהסך הכולל של כל קבוצה), כדי להפחית את ההשפעה של משתני זמן כגון חשיפה לריח אנושי, שינוי טמפרטורה בין התאים לבין משטחי ההזנה וכו '.
6. הערכה פנוטיפית: מורפולוגיה של בלוטות הרוק והורדת ויסות של חלבונים רלוונטיים
- יש לבודד רקמה טרייה ב-1x מי מלח עם מאגר פוספט (PBS) כמתואר בשלב 4.2 ולתקן באצטון קר כקרח במשך 90 שניות. יש לשטוף מספר פעמים ב-1x PBS לאחר הסרת האצטון. דגירה עם נוגדנים ראשוניים לילה ב 4 °C עם antiserum (ראה טבלת חומרים) מדולל ל 1x PBS.
הערה: ראה טבלת חומרים לזיהוי הנוגדנים העיקריים המשמשים לחלבוני רוק (חלבון אנופלס נגד טסיות דם, AAPP; Mucin 2, MUC2), גורמי שעתוק SG (ראש מזלג, fkh; מרווה, מרווה; תגובה מחזורית-AMP חלבון קושר אלמנט A, CrebA), וסמן של שלפוחיות הפרשה (Rab11). נוגדנים אלה משמשים כקריאות עבור צורה ותפקוד של SG. עם זאת, כל נוגדן המתאים לאימונופלואורסצנציה צריך להיות מתאים לפרוטוקול זה. - יש לשטוף ב-1x PBS מספר פעמים. הוסיפו נוגדנים משניים (פלואורסצנטיים) המדוללים ב-1x PBS, ודגרו בחושך בטמפרטורת החדר במשך שעתיים. הוסף כל נוגדן [כגון 4′,6-דיאמידינו-2-פנילינדול (DAPI; דנ"א), נבט חיטה אגלוטינין (WGA; עבור כיטין), פאלואידין (עבור F-אקטין), ו/או נילוס אדום (עבור שומנים)] 30 דקות לפני תום הדגירה של 2 שעות.
- יש לשטוף שלוש פעמים ב-1x PBS. לאחר מכן, הרכיבו את הרקמות ב-100% גליצרול על מגלשת מיקרוסקופ סטנדרטית עם כיסוי בעובי 1 מ"מ ואחסנו בטמפרטורה של -20 מעלות צלזיוס עד להדמיה באמצעות מיקרוסקופ קונפוקלי פלואורסצנטי.
הערה: כדי לקבל נתונים כמותיים, הגדרות ההדמיה חייבות להיות קבועות. כאן נכללו רק תמונות הקרנה בעוצמה מרבית דרך כל הנפח התלת-ממדי של הרקמה, וכל כימות התמונה נורמל בין טיפולים (במסגרת ניסוי) המבוססים על אות DAPI בשרידי רקמות שאינם SG (גוף שומן, קוטיקול או ראש) שנמצאים גם הם על השקופית.
תוצאות
כדי להתחיל, נתוני ביטוי מיקרו-מערך מ-VectorBase שימשו לסריקת מטרות פוטנציאליות בשלבי התפתחות36,37 כדי לקבוע את מצב הביטוי של כל הגנים הרלוונטיים למחקר הנוכחי (טבלה 1). כצפוי, כל גני המטרה שבחרנו הראו ביטוי ב-SGs בוגרים. רמות ה-aapp וה-sage היו גבוהות במיוחד (טבלה 1). כמו כן, יש לציין את רמות הביטוי הגבוהות של f-Agdsx אצל נקבה בוגרת SGs9.
מקטעים ספציפיים מכל גן הוערכו לשימוש כ-dsRNA באמצעות יישום האינטרנט E-RNAi לתכנון ריאגנטים RNAi30. כ-400 bp אזורים המכילים רצפים ייחודיים לכל גן מטרה שוכפלו לאחר מכן (איור 1A), הפכו לזני החיידקים המתאימים, ושימשו להכנת תרחיפים של חיידקים מומתי חום, אשר הונעו לייצר dsRNA. יתושים בוגרים הוזנו במשך 8 ימים על כדורי הצמר גפן ספוגי סוכרוז המכילים את ההשעיות החיידקיות של dsRNA עבור f-Agdsx,fkh או נמלה (ההדברה השלילית שאינה קשורה).
לצורך ניתוח האכלת RNAi של יתושים נקבות, נקבע לראשונה אם האכלות f-Agdsx או fkh dsRNA גרמו להשתקת גנים. הפחתה של 98.8% (±2.1) ברמות התעתיק של fkh נצפתה בקבוצה שניזונה מ-fkh-dsRNA (איור 1B), מה שמצביע על כך שה-dsRNA הפחית ביעילות רבה את שפע תעתיקי ה-fkh ב-SGs. באופן מפתיע, רמות ה-mRNA של fkh הופחתו ב-82.0% (±18.9) אצל היתושים שטופלו ב-dsRNA עבור f-Agdsx, שהיו בעלי הפחתה של 89.86% (±4.48) של f-Agdsx , מה שמרמז על כך ש-fkh יכול להיות מטרה ל-F-Dsx בבלוטת הרוק. במקביל לירידה המשמעותית ברמות הביטוי של fkh , יתושי ה-fkh-knockdown הראו עלייה משמעותית במספר ניסיונות הבדיקה הדרושים להזנת דם. היתושים האלה הפגינו, בממוצע, פי חמישה יותר ניסיונות האכלה מאשר קבוצת הביקורת או ה-f-Agdsx dsRNA שניזונו מיתושים כדי להיות מלאים בדם (איור 1C). זה הוביל לשאלה אם טיפולי ה-RNAi של ה-fkh גרמו לשינויים בלוקליזציה ו/או בהפצה של מווסתי שעתוק מרכזיים (SG TFs Sage ו-CrebA) (איור 2), חלבונים מופרשים (AAPP ומוצין) (איור 3) ומכונות הפרשה [אדום הנילוס (שומנים) ו-Rab11 (בועיות הפרשה)] (איור 4). חשוב לציין כי נצפו הבדלים משמעותיים בעוצמת הכתם באזורי אונה שונים, אונות ו-SGs בודדים.
כפי שנחזה, רמות המרווה והכתמת ה-CrebA הופחתו במידה ניכרת בכל אונות ה-SG בעקבות fkh RNAi (איור 2B) בהשוואה לבקרת נמלים RNAi (איור 2A). הפחתות הן בערכי העוצמה המרבית הגבוהה ביותר (קווים מקווקווים אדומים ותוויות מספריות) והן בערכי העוצמה המרבית הנמוכה ביותר (קווים מקווקווים כחולים ותוויות מספריות) בפרופילי סריקת קווים הציעו הפחתה באזורים של אות גבוה ונמוך בתוך הרקמה (איורים 2A,B). נתונים אלה מצביעים על כך ש- An. gambiae fkh RNAi יעיל וכי fkh מווסת את הייצור ו / או היציבות של SG TFs Sage ו- CrebA ב- An. gambiae, בדומה לקשר הגנטי שלהם ב- Drosophila SGs 19,38,39.
כאשר בוחנים חלבונים בעלי מרכיבים גבוהים ברוק, הרמות של חלבון אנטי-טסיות אנופלס (AAPP)40,41 הופחתו בכל שלוש אונות ה-SG לאחר fkh RNAi, בהשוואה לטיפול ב-RNAi בקרה (איור 3A,B; ירוק). מצד שני, לא נצפו שינויים ברמות של מוצין (איור 3A,B; סגול). נתונים אלה מצביעים על כך ש-Fkh תורם באופן שונה לביטוי של גנים שונים של חלבוני רוק.
לבסוף נצפו שני סמני הפרשה (איורים 4A,B): Rab11 (שלפוחיות הקשורות לאנדוזומים למיחזור אפי)42 ואדום הנילוס (ליפידים). פלואורסצנציה מופחתת של Rab11 נצפתה באונות צידיות דיסטליות (DL) לאחר טיפול ב-fkh RNAi (איור 4A v לעומת 4B v; ירוק). עם זאת, אות Rab11 מוגבר באונות המדיאליות (M) והפרוקסימליות לרוחב (PL) (איור 4A vii, ix לעומת 4B vii, ix; ירוק) התרחש גם הוא. לא נצפה הבדל ניכר באות אדום הנילוס (איורים 4A,B; סגול) לאחר fkh RNAi בהשוואה לטיפול ב-RNAi הבקרה. נתונים אלה מצביעים על כך שהפחתת fkh עשויה לשנות חלק מפעולות מנגנון ההפרשה באופן מורכב השונה בין אונות SG.
| מערך נתונים: | גולצב | ניירה אוביידו | ניירה אוביידו | אופה | אופה | אופה | אופה | ||
| סמל הגן | פונקציה | מזהה AGAP | עובר (25 שעות) | זחלי L3 | L3 SG | נקבה בוגרת גוף (3 ימים) | זכר בוגר גוף (3 ימים) | נקבה בוגרת SG (3 ימים) | זכר בוגר SG (3 ימים) |
| AAPP | חלבון רוק | AGAP009974 | 3.92 | 4.38 | 4.33 | 3.81 | 2.46 | 11.92 | 2.69 |
| CrebA | גורם txn | AGAP001464 | 6.28 | 5.22 | 5.92 | 2.99 | 2.96 | 3.27 | 3.13 |
| " | גורם txn | AGAP011038 | 4.50 | 4.46 | 5.23 | 2.96 | 2.86 | 3.05 | 2.88 |
| dsx | גורם txn | AGAP004050 | 4.91 | 5.39 | 5.55 | 3.72 | 4.00 | 4.57 | 4.01 |
| fkh | גורם txn | AGAP001671 | 5.18 | 4.67 | 5.25 | 2.99 | 3.09 | 3.21 | 3.05 |
| MUC2 | חלבון רוק | AGAP012020 | 4.59 | 5.53 | 5.63 | 2.96 | 3.07 | 3.08 | 3.26 |
| ראב11 | סחר בשלפוחית | AGAP004559 | 10.21 | 7.47 | 8.60 | 4.90 | 3.79 | 3.38 | 2.96 |
| מרווה | גורם txn | AGAP013335 | 5.32 | 5.96 | 8.89 | 3.40 | 3.33 | 7.37 | 7.23 |
טבלה 1: פרופילי ביטוי מיקרו-מערך ממוצעים של log2 עבור An. גמביה גנים מעניינים. מוצגים שמות גנים, קטגוריה פונקציונלית, מזהי Vectorbase (AGAP) ונתוני ביטוי מיקרו-מערך ממוצעים של log2 שנאספו מ- Vectorbase. נתונים אלה מצביעים על כך שהגנים המעניינים שלנו (המעורבים בביולוגיה של תאי בלוטת הרוק (SG) ובהפרשתם) באים לידי ביטוי ומועשרים בשלב זחלי 3 (L3) וב-SGs בוגרים, בהשוואה לאנשים שלמים.
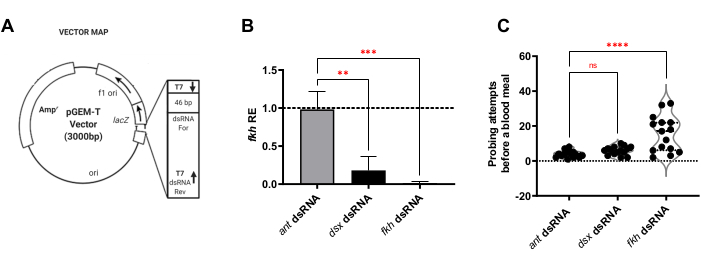
איור 1: f-Agdsx ו-fkh knockdown במבוגרים An. gambiae מפחית את רמות ה-mRNA של fkh ב-SGs ומשפיע על היכולת הנשית להזין דם. (A) תמונה מייצגת של עיצוב הפלסמיד המשמש לייצור dsRNA במתודולוגיה זו. רצף מקדם T7 השני מתווסף לפלסמיד על ידי הכללתו בפריימר 3' המשמש להגברת התוסף לשיבוט לתוך הפלסמיד pGEMT. לאחר מכן הפלסמיד הופך לחיידקי E. coli HT115 (DE3) ותמיסת האכלה עשויה מתרחיף של חיידקים מושרים המומתים בחום ב-10% מי סוכר. (B) בעלי חיים שניזונו מתמיסת האכלה של dsRNA עבור f-Agdsx או fkh, הראו רמות נמוכות משמעותית של תעתיקי fkh (ANOVA חד-כיווני עם השוואות מרובות; n=15). עם זאת, רק הקבוצה שניזונה מ-fkh dsRNA (C) הראתה הבדל משמעותי במספר ניסיונות הנשיכה הדרושים לרכישת ארוחת דם. יתושים בקבוצה זו היו זקוקים, בממוצע, לפי חמישה ממספר ניסיונות הבדיקה כדי להשיג ארוחת דם מוצלחת מהנדרש על ידי קבוצת הבקרה או קבוצות ההזנה dsx-dsRNA (ANOVA חד-כיוונית עם השוואות מרובות; n=15). פסי שגיאה מציינים את שגיאת התקן של הממוצע (SEM). כל ניסוי נערך בשלושה שכפולים ביולוגיים נפרדים. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
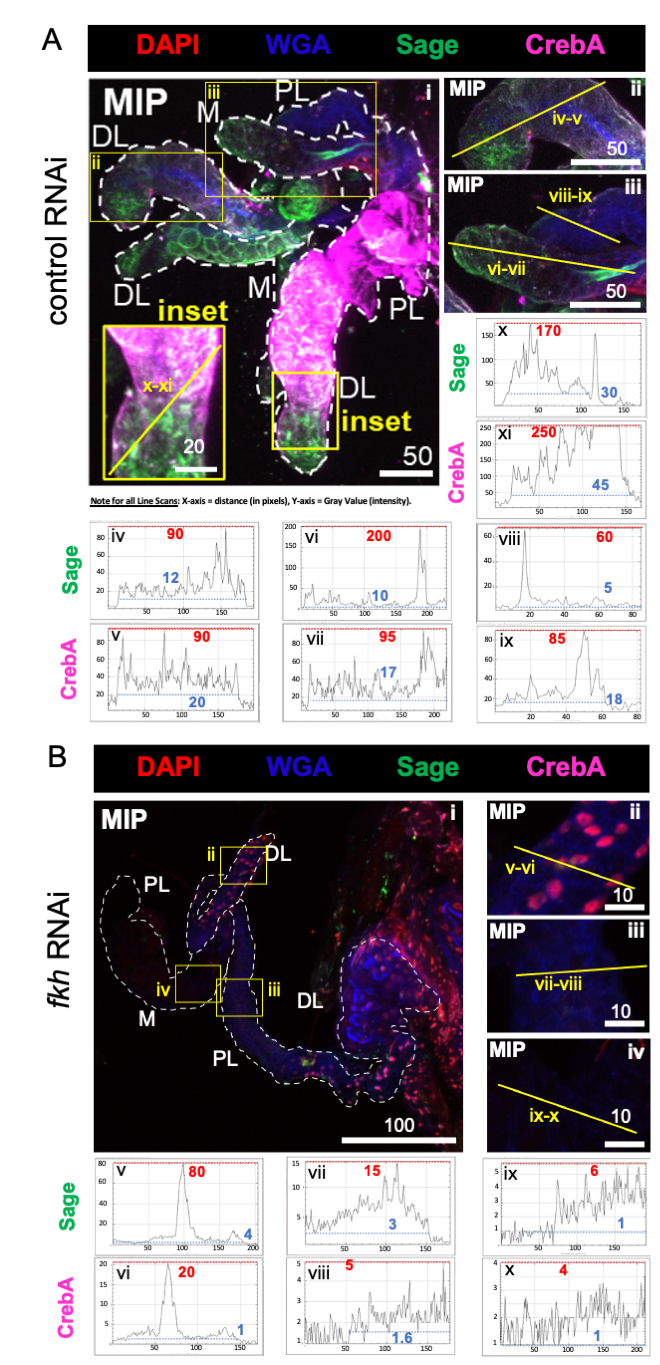
איור 2: תקיעת fkh בבלוטות הרוק של An. gambiae בוגרת מפחיתה את רמות גורם השעתוק של SG. מוצגות תמונות מייצגות מהיום ה-13 של הנקבה הבוגרת An. gambiae SGs לאחר 8 ימים (ימים 5-13) של חשיפה פומית ל-(A) בקרת dsRNA שאינה קשורה (נמלה) או (B) dsRNA המכוונת לראש המזלג SG TF (fkh, AGAP001671) ב-10% סוכרוז מוכתם בצבעים DAPI (DNA; אדום), שכותרתו אגלוטינין של נבט חיטה (WGA, chitin/ O-GlcNAcylation; כחול), אנטי-סרה נגד SG TFs Sage (ירוק) ו-CrebA (סגול). אורכי סרגלי קנה המידה המוצגים הם מיקרונים. SGs (i) מתוארים עם מקפים לבנים. קווים צהובים בתמונות אונה מוגדלת (של האזורים המוקפים בקופסאות צהובות, ומסומנים כ"משובצים") מציינים היכן נערכו סריקות הקו של עוצמת האות. עוצמות הערוצים הירוקות והסגולות המתאימות לסריקות קו עבור כל אונה מוגדלת משורטטות (תמיד משמאל לימין ב-SG) בגרפים שמתחת לתמונות; ציר X = מרחק (בפיקסלים) וציר Y = יחידה אפורה (עוצמת פיקסלים). הטווח הדינמי של עוצמת הפיקסלים מתוחם בקווים מנוקדים אדומים (מרביים) וכחולים (מינימום) והערכים המתאימים מוצגים בכל גרף. MIP = הקרנה תלת-ממדית בעוצמה מרבית דרך כל עומק ה-SG. DL: אונה צידית דיסטלית; M: אונה מדיאלית; PL: אונה צידית פרוקסימלית; SD: צינור רוק. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
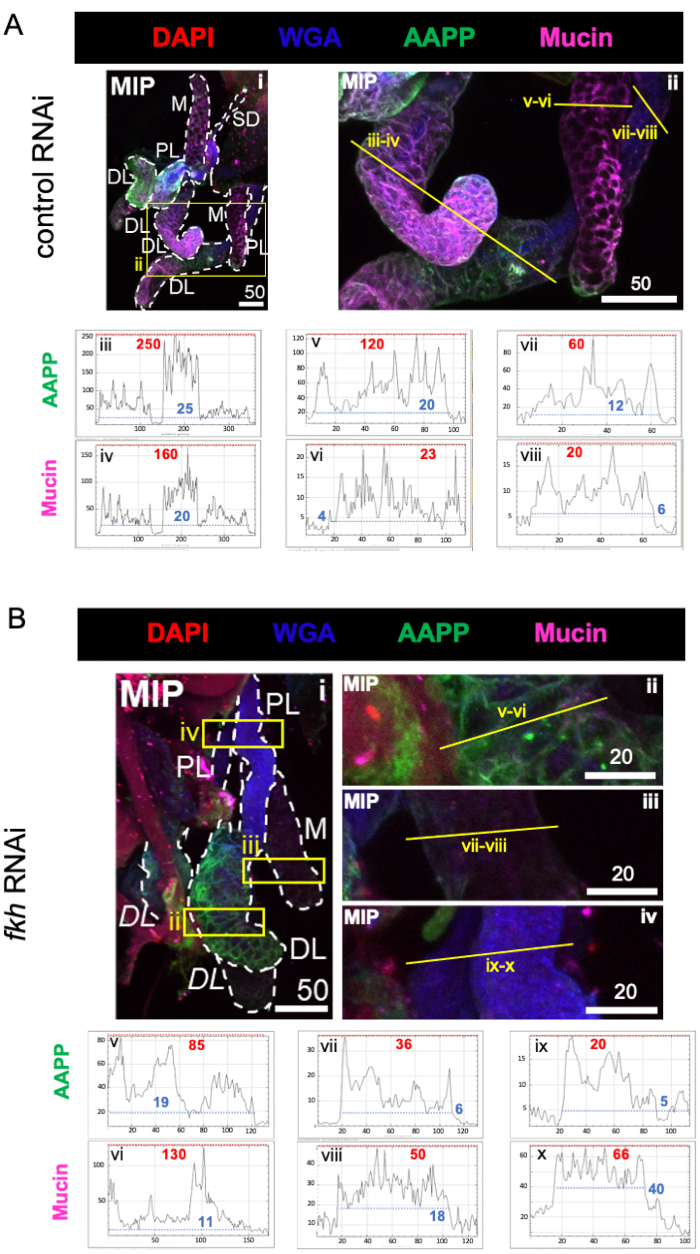
איור 3: התרסקות fkh בבלוטות הרוק של An. gambiae למבוגרים מפחיתה את רמות החלבון המופרשות על ידי SG. מוצגות תמונות מייצגות מהיום ה-13 של הנקבה הבוגרת An. gambiae SGs לאחר 8 ימים (ימים 5-13) של חשיפה אוראלית ל-(A) בקרת dsRNA שאינה קשורה (נמלה), או (B) dsRNA המכוון לראש המזלג SG TF (fkh,AGAP001671) ב-10% סוכרוז מוכתם בצבעים DAPI (DNA; אדום), שכותרתו אגלוטינין של נבט חיטה (WGA, chitin/ O-GlcNAcylation; כחול), וחלבוני הרוק AAPP (ירוק) ומוצין (MUC2, סגול). אורכי סרגלי קנה המידה המוצגים הם מיקרונים. SGs (i) מתוארים עם מקפים לבנים. קווים צהובים בתמונות אונה מוגדלת (של האזורים המוקפים בקופסאות צהובות) מציינים היכן נערכו סריקות הקו של עוצמת האות. עוצמות הערוצים הירוקות והסגולות המתאימות לסריקות קו עבור כל אונה משורטטות (תמיד משמאל לימין ב-SG) בגרפים שמתחת לתמונות; ציר X = מרחק (בפיקסלים) וציר Y = יחידה אפורה (עוצמת פיקסלים). הטווח הדינמי של עוצמת הפיקסלים מופרד בקווים מקווקווים אדומים (מרביים) וכחולים (מינימום) והערכים המתאימים מוצגים בכל גרף. MIP = הקרנה תלת-ממדית בעוצמה מרבית דרך כל עומק ה-SG. DL: אונה צידית דיסטלית; M: אונה מדיאלית; PL: אונה צידית פרוקסימלית; SD: צינור רוק. תוויות "DL" נטויות (Bi) מציינות שני אזורים גלויים של אותה אונת DL. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
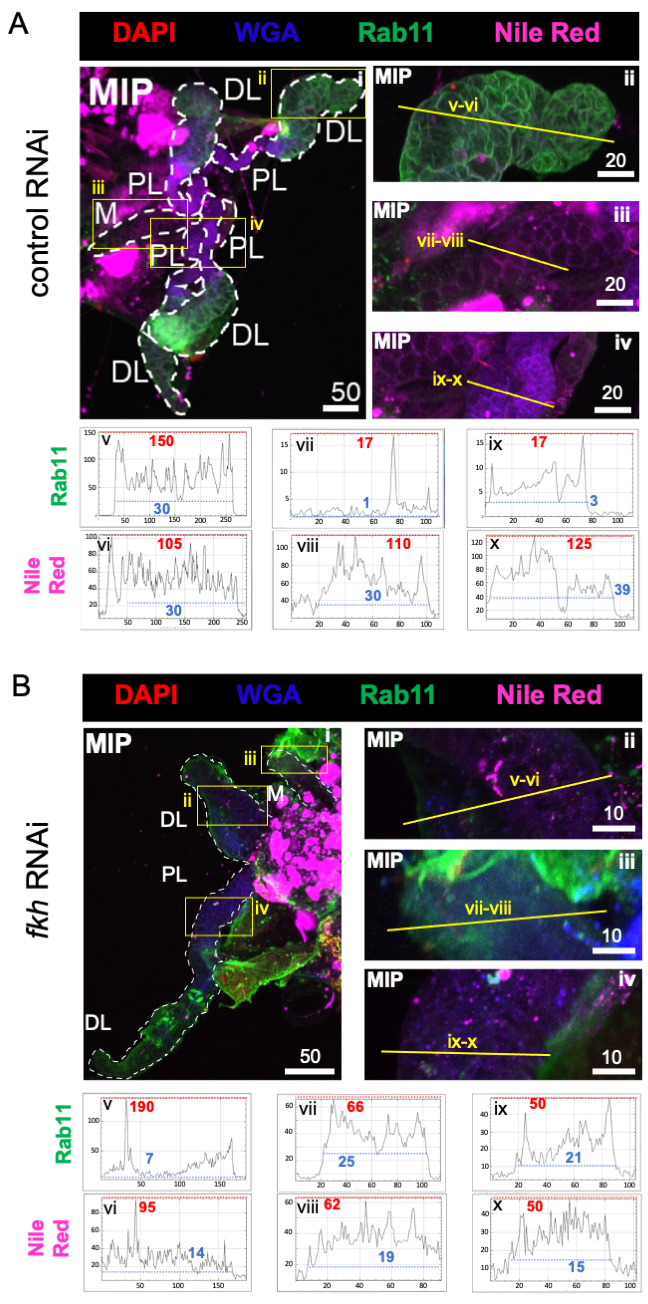
איור 4: נפילת fkh בבלוטות הרוק הבוגרות של An. gambiae מפחיתה את סמני הפרשת ה-SG. מוצגות תמונות מייצגות מהיום ה-13 של הנקבה הבוגרת An. gambiae SGs לאחר 8 ימים (ימים 5-13) של חשיפה פומית ל-(A) בקרת dsRNA שאינה קשורה (נמלה), או (B) dsRNA המכוון לראש המזלג SG TF (fkh, AGAP001671) ב-10% סוכרוז מוכתם בצבעים DAPI (DNA; אדום), שכותרתו אגלוטינין של נבט חיטה (WGA, chitin/ O-GlcNAcylation; כחול), אדום הנילוס (ליפידים; סגול), ואנטיסרה כנגד סמן שלפוחית האנדוזום הממחזרת Rab11 (ירוק). אורכי סרגלי קנה המידה המוצגים הם מיקרונים. SGs (i) מתוארים עם מקפים לבנים. קווים צהובים בתמונות אונה מוגדלת (של האזורים המוקפים בקופסאות צהובות) מציינים היכן נערכו סריקות הקו של עוצמת האות. עוצמות הערוצים הירוקות והסגולות המתאימות לסריקות קו עבור כל אונה משורטטות (תמיד משמאל לימין ב-SG) בגרפים שמתחת לתמונות; ציר X = מרחק (בפיקסלים) וציר Y = יחידה אפורה (עוצמת פיקסלים). הטווח הדינמי של עוצמת הפיקסלים מופרד בקווים מקווקווים אדומים (מרביים) וכחולים (מינימום) והערכים המתאימים מוצגים בכל גרף. MIP = הקרנה תלת-ממדית בעוצמה מרבית דרך כל עומק ה-SG. DL: אונה צידית דיסטלית; M: אונה מדיאלית; PL: אונה צידית פרוקסימלית; SD: צינור רוק. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
קובץ משלים 1. אנא לחץ כאן כדי להוריד קובץ זה.
Discussion
ליכולת להעביר ביעילות dsRNA ליתושי An. gambiae על ידי האכלה דרך הפה יש השלכות רחבות על מחקרים על ביולוגיה וקטורית הן במעבדה והן בשטח. Microinjection כבר מזמן מקובל כדרך המועדפת של אספקת כימיקלים, נוגדנים, RNAi, ואסטרטגיות שינוי גנטיות יתושים43,44. ניתן להימנע מהתוצאה של מניפולציה פיזית משמעותית, נזק לתאים ומתח על ידי שימוש בלידה דרך הפה, שעשויה להתאים גם ליישומים בקנה מידה גדול או בשטח. עבודות קודמות הציעו כי RNAi פועל בכל מקום בתוך יתושבוגר בודד 29, ומאפשר השפעות בכל הרקמות, כולל בלוטות הרוק. על ידי האכלת יתושים במספרים גדולים של E. coli המבטאים dsRNA, המתעכלים באופן אסינכרוני לאורך פרק זמן ארוך, ניתן להשיג חשיפה עקבית ואחידה ל-RNAi על פני כל הפרטים בכלוב. שיטה זו מאפשרת להאכיל מספר רב של יתושים ולנתח את השונות הפוטנציאלית של הפנוטיפים המתקבלים בהתאם לגן המטרה. עם זאת, אחד השיקולים החשובים הוא האפשרות של הפצה הטרוגנית של החיידקים, ומכאן dsRNA, בסיבי הכותנה. 400 מיקרול' של חיידקים המשמשים מדי יום להזנת יתושים מסוכר יכילו כ-≤4.6 מיקרוגרם של dsRNA, כפי שתואר וחושב קודם לכן9, אך כמות ה-dsRNA שנבלעה על ידי כל יתוש לא נקבעה בנפרד. אם בניית מבני dsRNA הופכת לשגרה, פרוטוקול טיפול פשוט זה מאפשר הטמעה מהירה של טכניקה זו על ידי כל חוקר יתושים. א-פריורי, זמן ההוצאה במהלך הטיפול (30 דקות ביום) הוא טריוויאלי בהשוואה לזמן שלוקח ללמוד וליישם מיקרו-איניג'ינג בגדלים דומים של דגימות.
האכלת dsRNA משמשת באופן שגרתי למחקרי גנטיקה הפוכה באורגניזם המודל Caenorhabditis elegans45. רמת שימוש כבדה זו מדגישה את הערך של גישת הלידה האוראלית. בניית ספרייה כלל-גנומית של An. gambiae ב-E. coli שעברה טרנספורמציה, בדומה לזו הקיימת ב-C. elegans46,47, תאפשר בדיקה גנטית הפוכה מהירה אצל יתושים בקנה מידה מוגבר. עם זאת, חשוב לציין כי יעילות השיטה תלויה במידה רבה ברמות האנדוגניות של התעתיק ואם הביטוי אינו מוגבל לרקמת המטרה אלא מבוטא באופן רחב יותר 4,8,44. בנוסף, ישנן עדויות לכך שכמה קוטלי חרקים יכולים לגרום להימנעות התנהגותית מיתושים48, והאכלה בחיידקים שעלולים לגרום להם לתופעות לוואי עלולה לעורר דפוסים דומים של הימנעות. בסביבה המבוקרת של המעבדה, שבה ליתושים לא היה מקור מזון חלופי, לא הייתה להם ברירה להימנע ממי הסוכר עם E. coli והצורך במקור מזין כנראה יגבר על האינסטינקט להימנע מהחיידקים. עם זאת, יש לשקול זאת אם האסטרטגיה נועדה לשמש בהגדרות פחות מבוקרות.
ייתכן שניתן יהיה לכוון למספר גנים בו-זמנית (באמצעות מבנה אחד, מספר מבנים או תערובת של חיידקים מבודדים שעברו טרנספורמציה), אך נדרשים מחקרים נוספים כדי להעריך את היעילות. שיקול חשוב נוסף לנקודה זו הוא הערכה של השפעות אפשריות מחוץ למטרה או סינרגטיות בעת שימוש ביעדים בודדים או מרובים. הקמת גנים וקבוצות בקרה מתאימים היא חלק חשוב בתכנון הניסוי. יתר על כן, מפתה לשער כי גישה זו יכולה לשמש כדי להתמקד בפתוגנים או וירוסים אחרים49. עבודה קודמת לקראת אינדוקציה של RNAi ביתושים בוצעה בתנאים שבהם הריאגנט הוזרק ישירות, ולכן E. coli לא היו נוכחים. ה - E. coli עשוי לספק תא מגן המאפשר שחרור איטי יותר של dsRNA לאורך זמן, ומבטיח שהחשיפה תהיה פחות או יותר רציפה לאורך תקופה ארוכה בהרבה29.
לבסוף, תוצאות אלה מראות כי ההשפעות של טכניקה זו ניתנות לכוונון על ידי התאמת מסגרת הזמן (אורך ויום ההתחלה) של החשיפה והכמות של E. coli בשימוש. תכונה זו אפשרה לנו לחקור את הפונקציות של גנים חיוניים (dsx ו - fkh) על ידי זיהוי תנאי נפילה אופטימליים על ידי ניסוי וטעייה. זה משפר מאוד את הסבירות שניתן לחקור גנים ממוקדים בעלי עניין באמצעות טכניקה זו.
לסיכום, נמצא כי מסירה דרך הפה של RNAi ליתושים בוגרים יכולה להיות פשוטה, רב-תכליתית וגישה רבת עוצמה לחקר תפקוד הגנים של היתושים וליצירת כלים חדשניים ובלתי ניתנים להדברה וקטורית של מחלות המועברות על ידי יתושים.
Disclosures
המחברים מדווחים כי אין להם ניגודי עניינים לחשוף.
Acknowledgements
המחברים מבקשים להודות לצוות ולמדענים בענף האנטומולוגיה ובמחלקה למחלות טפיליות ומלריה ב-CDC, ולבריאן טריג ומישל צ'יו על הסיוע בהכנת חיידקים ב-JHU ו/או על דיונים מועילים בעבודה זו. אנו מודים ל-JHMRI Insectary ולמנהל כריס קיזיטו על הגישה והגידול של יתושי An. gambiae . אנו מודים ל-Wei Huang (JHSPH) על הסיוע בהשגת פלסמידים PJet GFP ו-pPB47 GFP לשימוש במחקר זה. המימון לעבודה זו ניתן על ידי: NIH R21AI153588 (ל- DJA), מלגת פוסט-דוקטורט של מכון ג'ונס הופקינס לחקר המלריה (ל- MW); ועל ידי מענק מקרן Good Ventures ופרויקט הפילנתרופיה הפתוחה לקרן CDC שכותרתה תמיכה בשמירת בהקפאה ודיכוי התפתחות נשית של יתושים כדי לסייע במחקר על מלריה, פרויקט פילנתרופיה פתוחה, 2017. אנו מעריכים מאוד את הסיוע של צוות מתקן המיקרוסקופ JHU ואת התמיכה הרלוונטית במענק NIH עבור המיקרוסקופ שבו נעשה שימוש (מענק NIH #: S10OD016374). הממצאים והמסקנות בכתב יד זה הם של המחברים ואינם מייצגים בהכרח את דעותיו של ה-CDC. השימוש בשמות מסחריים הוא לצורך זיהוי בלבד ואינו מרמז על תמיכה על ידי המרכזים לבקרת מחלות ומניעתן, שירות בריאות הציבור או מחלקת הבריאות ושירותי האנוש של ארה"ב.
Materials
| Name | Company | Catalog Number | Comments |
| 1 Kb Plus DNA Ladder | Thermo Fisher Scientific | 10787018 | |
| 2x Yeast Extract Tryptone (2xYT) Medium | BD Difco | DF0440-17 | |
| AAPP | n/a | n/a | Antisera. 1:50 dilution (rabbit); gift from Fabrizio Lombardo |
| AccuStart II PCR Supermix | Quantabio | 95137-100 | |
| Agarose | Millipore Sigma | A9539 | |
| Ampicillin | Millipore Sigma | A5354 | |
| Anopheles gambiae G3 | BioDefense and Emerging Infections (BEI) Malaria Research and Reference Reagent Resource Center (MR4) | MRA-112 | |
| BugDorm | BioQuip | 1452 | |
| Centrifuge 5810R | Eppendorf | P022628181 | |
| CrebA | DSHB | CrebA Rbt-PC | Antisera. 1:50 dilution (rabbit); generated by the Andrew Lab |
| Damiens diet | BioServ | ||
| DAPI | Life Technologies | n/a | 4′,6-diamidino-2-phenylindole; 1:200 dilution. |
| Defibrinated sheep blood | HemoStat | DSB050 | |
| Escherichia coli HT115 (DE3) | |||
| Ethidium bromide | Millipore Sigma | E7637 | |
| High-Capacity cDNA Reverse Transcription Kit | Thermo Fisher Scientific | 4368814 | |
| Isopropyl β-D-1-thiogalactopyranoside | Millipore Sigma | I5502 | |
| JM109 Competent cells | Promega | L2005 | |
| Luria Broth Media | Thermo Fisher Scientific | 10855001 | |
| Mucin 2 | Proteintech | Muc2; 27 675-1-AP | Antisera. 1:100 dilution (mouse). |
| Nanodrop 2000 | Thermo Fisher Scientific | ||
| Nile Red | Sigma | n/a | Lipid dye; 1:50 dilution. |
| Owl EasyCast B2 Mini Gel Horizontal Electrophoresis | Thermo Fisher Scientific | Model B2 | |
| pGEMT easy | Promega | A3600 | |
| Power SYBR-green PCR master MIX | Applied Biosystems | 4367659 | |
| PureLink PCR purification kit | Thermo Fisher Scientific | K31001 | |
| QuantaStudio 6 | Applied Biosystems | ||
| QuantStudio6 Real Time PCR System | Applied Biosystems | ||
| Rab11 | n/a | n/a | Antisera. 1:100 dilution (rabbit); generated by the Andrew Lab |
| Rh-WGA | Vector Labs | n/a | Rhodamine-conjugated wheat germ agglutinin (chitin, O-GlcNAcylation dye); 1:40 dilution |
| Sage | n/a | n/a | Antisera. 1:50 dilution (rat); generated by the Andrew Lab |
| T4 DNA ligase | Promega | M1801 | |
| Tetracycline | Millipore Sigma | 87128 | |
| Trizol | Thermo Fisher Scientific | 15596018 | |
| Zeiss LSM700 fluorescence confocal microscope | Zeiss | ||
| ANTIBODIES | |||
| Chicken anti-Rat IgG (H+L), Alexa Fluor 647 | Thermo Fisher Scientific | A21472 | |
| Goat anti-Mouse IgG (H+L), Alexa Fluor 647 | Thermo Fisher Scientific | A28181 | |
| IgG (H+L) Goat anti-Rabbit, Alexa Fluor 488 | Thermo Fisher Scientific | A27034 | |
| Rabbit anti-Goat IgG (H+L), Alexa Fluor 488 | Thermo Fisher Scientific | A27012 | |
| PRIMERS | |||
| ACT-2f: TACAACTCGATCATGAAGTGCGA | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| ACT-3r: CCCGGGTACATGGTGGTACCGC CGGA | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| FKH_RNAi_F: GCCGACTTATGCTTAGCCCA | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| FKH_RNAi_R: TAGCCGTCAATTCCTCCTGC | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| newDSX-f: AGAGGGCGGGGAAATTCTAGT | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| newDSX-r: GGGCTTGTGGCAGTACGAATA | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| S7qf1: AGAACCAGCAGACCACCATC | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| S7qr1: GCTGCAAACTTCGGCTATTC | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
References
- Hoa, N. T., Keene, K. M., Olson, K. E., Zheng, L. Characterization of RNA interference in an Anopheles gambiae cell line. Insect Biochemistry and Molecular Biology. 33, 949-957 (2003).
- Caplen, N., Zheng, Z., Falgout, B., Morgan, R. Inhibition of viral gene expression and replication in mosquito cells by dsRNA-triggered RNA interference | Elsevier enhanced reader. Molecular Therapy. 6, 243-251 (2002).
- Brown, A. E., Catteruccia, F. Toward silencing the burden of malaria: progress and prospects for RNAi-based approaches. BioTechniques. , 38-44 (2006).
- Airs, P. M., Bartholomay, L. C. RNA interference for mosquito and mosquito-borne disease control. Insects. 8, (2017).
- Blandin, S., et al. Reverse genetics in the mosquito Anopheles gambiae: targeted disruption of the Defensin gene. EMBO Reports. 3 (9), 852-856 (2002).
- Garver, L., Dimopoulos, G. Protocol for RNAi assays in adult mosquitoes (A. gambiae). Journal of Visualized Experiments: JoVE. (5), e230 (2007).
- Whyard, S., et al. Silencing the buzz: a new approach to population suppression of mosquitoes by feeding larvae double-stranded RNAs. Parasites & Vectors. 8, 96 (2015).
- Wiltshire, R. M., Duman-Scheel, M. Advances in oral RNAi for disease vector mosquito research and control. Current Opinion in Insect Science. 40, 18-23 (2020).
- Taracena, M. L., Hunt, C. M., Benedict, M. Q., Pennington, P. M., Dotson, E. M. Downregulation of female doublesex expression by oral-mediated RNA interference reduces number and fitness of Anopheles gambiae adult females. Parasites & Vectors. 12, 170 (2019).
- Grassi, B. Studi di uno zoologo sulla malaria. Real Accademia dei Lincei. 3, 229 (1901).
- Smith, R. C., Jacobs-lorena, M. Plasmodium - Mosquito interactions: A tale of roadblocks and detours. Advances in Insect Physiology. 39, (2010).
- Das, S., et al. Transcriptomic and functional analysis of the Anopheles gambiae salivary gland in relation to blood feeding. BMC Genomics. 11, 1-14 (2010).
- Francischetti, I. M. B., Valenzuela, J. G., Pham, V. M., Garfield, M. K., Ribeiro, J. M. C. Toward a catalog for the transcripts and proteins (sialome) from the salivary gland of the malaria vector Anopheles gambiae. Journal of Experimental Biology. 205, 2429-2451 (2002).
- Henderson, K. D., Isaac, D. D., Andrew, D. J. Cell fate specification in thedrosophila salivary gland: The integration of homeotic gene function with the DPP signaling cascade. Developmental Biology. 205, 10-21 (1999).
- Mach, V., Ohno, K., Kokubo, H., Suzuki, Y. The Drosophila fork head factor directly controls larval salivary gland-specific expression of the glue protein gene Sgs3. Nucleic Acids Research. 24 (12), 2387-2394 (1996).
- Weiserova, M., et al. Mini-Mu transposition of bacterial genes on the transmissible plasmid. Folia Microbiologica. 32 (5), 368-375 (1987).
- Abrams, E. W., Mihoulides, W. K., Andrew, D. J. Fork head and Sage maintain a uniform and patent salivary gland lumen through regulation of two downstream target genes, PH4αSG1 and PH4αSG2. Development. 133, 3517-3527 (2006).
- Myat, M. M., Isaac, P. P., Andrew, D. J. Early genes required for salivary gland fate determination and morphogenesis in Drosophila melanogaster. Advances in Dental Research. 14, 89-98 (2000).
- Fox, R. M., Vaishnavi, A., Maruyama, R., Andrew, D. J. Organ-specific gene expression: the bHLH protein Sage provides tissue specificity to Drosophila FoxA. Development of Cell Biology. 140, 2160-2171 (2013).
- Maruyama, R., Grevengoed, E., Stempniewicz, P., Andrew, D. J. Genome-wide analysis reveals a major role in cell fate maintenance and an unexpected role in endoreduplication for the Drosophila FoxA gene fork head. PLOS ONE. 6, 20901 (2011).
- Johnson, D. M., et al. CrebA increases secretory capacity through direct transcriptional regulation of the secretory machinery, a subset of secretory cargo, and other key regulators. Traffic. 21, 560-577 (2020).
- Fox, R. M., Hanlon, C. D., Andrew, D. J. The CrebA/Creb3-like transcription factors are major and direct regulators of secretory capacity. Journal of Cell Biology. 191, 479-492 (2010).
- Abrams, E. W., Andrew, D. J. CrebA regulates secretory activity in the Drosophila salivary gland and epidermis. Development. 132, 2743-2758 (2005).
- Wells, M. B., Andrew, D. J. Anopheles salivary gland architecture shapes plasmodium sporozoite availability for transmission. mBio. 10 (4), 01238 (2019).
- Pei-Wen, L., Xiao-Cong, L., Jin-Bao, G., Yan, L., Xiao-Guang, C. Molecular cloning, characterization and expression analysis of sex determiantion gene doublesex from Anopheles gambiae (Diptera: Culicidae). Acta Entomologica Sinica. 58 (2), 122-131 (2015).
- Scali, C., Catteruccia, F., Li, Q., Crisanti, A. Identification of sex-specific transcripts of the Anopheles gambiae doublesex gene. The Journal of Experimental Biology. 208, 3701-3709 (2005).
- Price, D. C., Egizi, A., Fonseca, D. M. Characterization of the doublesex gene within the Culex pipiens complex suggests regulatory plasticity at the base of the mosquito sex determination cascade. BMC Evolutionary Biology. 15, 1-13 (2015).
- Mysore, K., et al. siRNA-mediated silencing of doublesex during female development of the dengue vector mosquito Aedes aegypti. PLoS Neglected Tropical Diseases. 9, 1-21 (2015).
- Boisson, B., et al. Gene silencing in mosquito salivary glands by RNAi. FEBS Letters. 580, 1988-1992 (2006).
- Horn, T., Boutros, M. E-RNAi: a web application for the multi-species design of RNAi reagents-2010 update. Nucleic Acids Research. 38, 332-339 (2010).
- Taracena, M. L., et al. Genetically modifying the insect gut microbiota to control chagas disease vectors through systemic RNAi. PLoS Neglected Tropical Diseases. 9, (2015).
- Sambrook, J., Fritsch, E. F., Maniatis, T. . Molecular Cloning: A Laboratory Manual. , (1989).
- Ullmann, A., Jacob, F., Monod, J. Characterization by in vitro complementation of a peptide corresponding to an operator-proximal segment of the β-galactosidase structural gene of Escherichia coli. Journal of Molecular Biology. 24, 339-343 (1967).
- Timmons, L. Bacteria-mediated RNAi-General outline. Carnegie Institution of Washington. , (2000).
- Pfaffl, M. W. Relative quantification. Real-time PCR. , 63-82 (2004).
- Neira-Oviedo, M., et al. The RNA-Seq approach to studying the expression of mosquito mitochondrial genes. Insect Molecular Biology. 20, 141-152 (2011).
- Baker, D. A., et al. A comprehensive gene expression atlas of sex- and tissue-specificity in the malaria vector, Anopheles gambiae. BMC Genomics. 12, (2011).
- Loganathan, R., Hoon, J., Wells, M. B., Andrew, D. J. Secrets of secretion - How studies of the Drosophila salivary gland have informed our understanding of the cellular networks underlying secretory organ form and function. Cellular Networks in Development. , 143 (2021).
- Chung, S., Hanlon, C. D., Andrew, D. J. Building and specializing epithelial tubular organs: The Drosophila salivary gland as a model system for revealing how epithelial organs are specified, form and specialize. Wiley Interdisciplinary Reviews: Developmental Biology. 3, 281-300 (2014).
- Yoshida, S., et al. Inhibition of collagen-induced platelet aggregation by anopheline antiplatelet protein, a saliva protein from a malaria vector mosquito. Blood. 111, 2007-2014 (2008).
- Wells, M. B., Villamor, J., Andrew, D. J. Salivary gland maturation and duct formation in the African malaria mosquito Anopheles gambiae. Scientific Reports. 7 (1), 601 (2017).
- Takahashi, S., et al. Rab11 regulates exocytosis of recycling vesicles at the plasma membrane. Journal of Cell Science. 125, 4049-4057 (2012).
- Catteruccia, F., Levashina, E. A. RNAi in the malaria vector, Anopheles gambiae. Methods in Molecular Biology. 555, 63-75 (2009).
- Balakrishna Pillai, A., et al. RNA interference in mosquito: understanding immune responses, double-stranded RNA delivery systems and potential applications in vector control. Insect Molecular Biology. 26, 127-139 (2017).
- Fire, A., et al. Potent and specific genetic interference by double-stranded RNA in caenorhabditis elegans. Nature. 391, 806-811 (1998).
- Kamath, R. S., Ahringer, J. Genome-wide RNAi screening in Caenorhabditis elegans. Methods. 30, 313-321 (2003).
- Kamath, R. S., et al. Systematic functional analysis of the Caenorhabditis elegans genome using RNAi. Nature. 421, 231-237 (2003).
- Carrasco, D., et al. Behavioural adaptations of mosquito vectors to insecticide control. Current Opinion in Insect Science. 34, 48-54 (2019).
- Magalhaes, T., et al. Induction of RNA interference to block Zika virus replication and transmission in the mosquito Aedes aegypti. Insect Biochemistry and Molecular Biology. , 111 (2019).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved