Method Article
Somministrazione efficace di interferenza orale dell'RNA (RNAi) alle zanzare Anopheles gambiae adulte
In questo articolo
Riepilogo
La somministrazione orale di dsRNA prodotto dai batteri, un metodo di consegna per l'interferenza dell'RNA (RNAi) che viene regolarmente utilizzato in Caenorhabditis elegans, è stato applicato con successo qui alle zanzare adulte. Il nostro metodo consente robusti studi di genetica inversa e studi vettoriali che bloccano la trasmissione senza l'uso dell'iniezione.
Abstract
L'interferenza dell'RNA è stata uno strumento molto utilizzato per l'analisi genetica inversa per due decenni. Nelle zanzare adulte, la somministrazione di RNA a doppio filamento (dsRNA) è stata effettuata principalmente tramite iniezione, che richiede un tempo significativo e non è adatta per applicazioni sul campo. Per superare queste limitazioni, qui presentiamo un metodo più efficiente per una robusta attivazione dell'RNAi mediante somministrazione orale di dsRNA ad Anopheles gambiae adulto. I dsRNA lunghi sono stati prodotti nel ceppo di Escherichia coli HT115 (DE3) e una sospensione concentrata di batteri contenenti dsRNA uccisi termicamente nel 10% di saccarosio è stata offerta su batuffoli di cotone ad-libitum alle zanzare adulte. I batuffoli di cotone sono stati sostituiti ogni 2 giorni per tutta la durata del trattamento. L'uso di questo metodo per colpire il doublesex (un gene coinvolto nella differenziazione sessuale) o la testa della forcella (che codifica per un fattore di trascrizione della ghiandola salivare) ha portato a una riduzione dell'espressione genica bersaglio e / o del segnale di immunofluorescenza proteica, come misurato rispettivamente dalla PCR quantitativa in tempo reale (qRT-PCR) o dalla microscopia confocale a fluorescenza. Sono stati osservati anche difetti nella morfologia delle ghiandole salivari. Questo metodo altamente flessibile, intuitivo, a basso costo ed efficiente in termini di tempo per la somministrazione di dsRNA potrebbe essere ampiamente applicabile ai geni bersaglio importanti per la fisiologia degli insetti vettori e oltre.
Introduzione
Molte malattie sono trasmesse dalle zanzare, rendendo lo studio della fisiologia e della genetica delle zanzare un'impresa importante. L'uso di RNAi in questi organismi è stato prominente negli ultimi 20 anni e ha permesso la caratterizzazione funzionale di molti geni delle zanzare 1,2,3,4,5. La tecnica più comunemente usata per la somministrazione di dsRNA è stata la microiniezione, che ha gli svantaggi che può ferire le zanzare e richiede tempo e sforzi significativi. Sono stati testati metodi di consegna orale per RNAi, ma principalmente nello stadio larvale delle zanzare 6,7,8,9. La somministrazione orale di dsRNA nelle zanzare adulte non è stata completamente esplorata e potrebbe essere uno strumento utile per lo studio della biologia vettoriale e del controllo dei vettori.
La malaria viene trasmessa dalle zanzare Anopheles quando una zanzara femmina infetta prende un pasto di sangue da un ospite non infetto e inietta saliva contenente parassiti malarici10. Per essere infine trasmesso nella saliva di una zanzara, il parassita deve superare molti ostacoli, tra cui l'elusione del sistema immunitario della zanzara, l'attraversamento della barriera del midgut e l'invasione delle ghiandole salivari11. L'architettura della ghiandola salivare della zanzara (SG) è la chiave per l'invasione dei parassiti e tale architettura è controllata sia dai fattori di trascrizione chiave espressi dalla ghiandola salivare, sia dai determinanti del dimorfismo sessuale. Diversi fattori di trascrizione altamente conservati sono necessari per la specificazione cellulare e il mantenimento omeostatico delle ghiandole salivari e per la produzione e la secrezione di proteine salivari che funzionano nell'alimentazione del sangue 12,13,14. La testa della forcella (Fkh) è un fattore di trascrizione dell'elica alata che funziona come uno dei principali regolatori della struttura e della funzione SG degli insetti (sulla base di studi sui moscerini della frutta e sulla falena del baco da seta)15,16,17,18,19,20. Nei Drosophila SGs, Fkh funziona con Sage, un fattore di trascrizione di base elica-loop-elica (bHLH) specifico per SG, per promuovere la sopravvivenza SG e la produzione di saliva19. Un importante co-regolatore positivo della produzione di saliva in Drosophila è CrebA, un fattore di trascrizione della cerniera di leucina ben studiato che sovraregola l'espressione dei geni della via secretoria 21,22,23. C'è anche un forte grado di differenziazione morfologica nelle ghiandole salivari femminili che probabilmente svolge un ruolo chiave, non solo nell'alimentazione del sangue, ma anche nella capacità dei parassiti di invadere questo tessuto24.
Molti dei geni coinvolti nel determinare la sopravvivenza, la struttura, la fisiologia e il dimorfismo sessuale delle ghiandole salivari hanno complessi profili di espressione spaziotemporale 25,26,27 e i metodi tradizionali di consegna del dsRNA per indurre l'RNAi non sono sempre efficaci nel colpire questi tipi di geni in questo o in altri tessuti. Tuttavia, la somministrazione orale di dsRNA nello stadio larvale Aedes aegypti e An. gambiae mosquitoes è stata utilizzata con successo per silenziare la forma femminile specifica del gene dsx 9,28. Studi precedenti che utilizzavano dsRNA nelle ghiandole salivari delle zanzare hanno rilevato che, sebbene fossero necessarie grandi quantità di dsRNA, l'effetto silenziante era relativamente duraturo (almeno 13 giorni)29. Qui, è stata testata la capacità del ceppo di E. coli HT115 (DE3) ucciso dal calore che esprime dsRNA specifico della sequenza per dsx, fkh o CrebA di indurre il silenziamento RNAi di questi geni nelle zanzare femmine adulte. La somministrazione orale di knockdown genico indotto da dsRNA in An. gambiae, con chiare riduzioni dei livelli di mRNA e con fenotipi coerenti con la perdita di funzione di questi geni. Pertanto, questo approccio probabilmente funzionerà per abbattere la funzione di una varietà di geni delle ghiandole salivari.
Protocollo
1. Clonazione di dsRNA nel vettore di espressione di E. coli
- Selezionare la sequenza del gene bersaglio da inserire in un vettore appropriato per l'espressione del dsRNA. Recuperate i valori delle espressioni da Vectorbase.org utilizzando il metodo seguente.
- Cerca un gene di interesse (ad esempio, Tabella 1) nella casella di ricerca della home page.
- Nella pagina del gene risultante, vai all'8. Sezione di trascrittomica .
- Cerca gli esperimenti di espressione genica RNA-seq e microarray rilevanti elencati.
- Trascrivi i valori di interesse nel software del foglio di calcolo e crea una tabella di dati.
- Selezionare un plasmide disponibile in commercio con almeno un promotore T7 da utilizzare. Se il plasmide selezionato ha un solo promotore T7 (come fanno la maggior parte dei plasmidi commerciali), includere un secondo promotore T7 nel primer inverso da utilizzare per l'amplificazione del dsDNA per il gene di interesse.
NOTA: La sequenza dsRNA per i geni bersaglio può essere selezionata utilizzando l'applicazione web E-RNAi per la progettazione dei reagenti RNAi30. Sia il dsRNA lungo (circa 400 bp) che il dsRNA a forcina corta (shRNA) possono essere progettati sulla base di specifiche sequenze geniche. Queste sequenze dovrebbero essere amplificate e sequenziate per la conferma dell'identità prima della clonazione. Le regioni geniche, i plasmidi e i promotori selezionati utilizzati in questo studio sono elencati nel file supplementare 1. - Eseguire la clonazione secondo una semplice procedura in un'unica fase descritta in precedenza 9,31. A tale scopo, purificare il prodotto PCR e legare al DNA plasmidico linearizzato. Utilizzare il prodotto della legatura per la trasformazione da shock termico delle cellule competenti di E. coli 32. Selezionare le celle trasformate tramite screening blu/bianco. Confermare l'orientamento dell'inserto utilizzando una PCR con primer T7 e confermare la sequenza utilizzando primer M13.
NOTA: Gli screening bianco/blu possono essere utilizzati quando il plasmide selezionato per la trasformazione trasporta il gene lacZ che codifica per β-galattosidasi. Le colonie bianche devono contenere l'inserto desiderato all'interno del lacZ e possono essere selezionate per confermare ulteriormente la presenza e l'orientamento della sequenza target33. - Purificare il plasmide dalla prima trasformazione e utilizzarlo per trasformare il competente E. coli HT115 (DE3) come precedentemente descritto34. Dopo la conferma che il plasmide con l'inserto è presente nel competente E. coli HT115 (DE3), fare scorte di glicerolo di batteri monouso.
NOTA: Un appropriato dsRNA di controllo non correlato deve essere acquisito o preparato per l'uso in ogni esperimento. In questo caso, viene utilizzata la sequenza per il gene non correlato aintegumenta (formica) da Arabidopsis thaliana .
2. Preparazione di batteri uccisi dal calore che esprimono dsRNA
- Coltivare una coltura da una singola colonia batterica di E. coli ceppo HT115 (DE3) contenente il dsRNA che esprime il plasmide in 50 mL di Luria Brodo (LB) contenente 100 μg/mL di ampicillina e 12,5 μg/mL di tetraciclina, su uno scuotitore di piattaforma (180 rpm) a 37 °C per 12 h.
- Diluire la coltura batterica (1:1000) in 2s mezzi di lievito triptone (2x YT) contenenti 100 μg/mL di ampicillina e 12,5 μg/mL di tetraciclina.
- Indurre la produzione di dsRNA aggiungendo 40 μM (concentrazione finale) di isopropil β-D-1-tiogalattopoliranoside (IPTG).
- Quando le cellule raggiungono un O.D.600 = 0,4, approssimativamente dopo 2 ore di induzione a 37 °C con agitazione a 180 rpm, preparare una sospensione concentrata di batteri uccisi dal calore come descritto da Taracena et al 9. Pellet le celle per centrifugazione (4000 x g, 4 °C, 10 min) e lavare le cellule in un volume di tampone fosfato di sodio (PBS).
- Ruotare di nuovo nelle stesse condizioni, sospendere nuovamente in PBS a 1/100 del volume iniziale e posizionare a 70 °C per 1 ora.
- Effettuare aliquote da 400 μL dei batteri uccisi termicamente e conservare queste aliquote a -20 °C fino a nuovo utilizzo (non conservare per più di una settimana). Questa sospensione di batteri uccisi dal calore contiene il dsRNA specifico per gli esperimenti RNAi. Eseguire questa procedura sia per il gene bersaglio dsRNA-batteri che per il controllo dsRNA non correlato da utilizzare in ogni esperimento.
3. Nutrire le zanzare con batteri uccisi dal calore che esprimono dsRNA
- Scongelare un'aliquota di dsRNA (sospensione batterica HT115 (DE3)) e mescolare con 1,6 mL di soluzione zuccherina al 12% contenente lo 0,2% di metilparabene.
- Immergere un piccolo batuffolo di cotone in questa soluzione e posizionare il batuffolo di cotone imbevuto all'interno di una gabbia contenente zanzare di 5 giorni. Assicurarsi che le zanzare si nutrano di questa soluzione, raccogliendo contemporaneamente sia lo zucchero che i batteri contenenti dsRNA.
- Cambiare il batuffolo di cotone imbevuto di soluzione di dsRNA-zucchero a giorni alterni per 8 giorni consecutivi.
- Mantenere le gabbie per zanzare in condizioni costanti, cioè 27 °C e 80% di umidità relativa con un fotoperiodo di 12 h:12 h luce: fotociclo scuro, separato da un periodo di 30 minuti all'alba e 30 minuti al tramonto.
4. Livelli di espressione genica target del test
- Anestetizzare a freddo le zanzare mettendo il contenitore sul ghiaccio per un minuto o fino a quando le zanzare smettono di muoversi. Una volta che le zanzare sono anestetizzate, posizionarle su una superficie fredda per isolare le femmine per la dissezione.
- Spruzzare il 70% di etanolo alle zanzare e posizionarle su una superficie di vetro con PBS. Con un paio di pinze, fissare la testa della zanzara e tirare il torace molto lentamente, consentendo alle ghiandole salivari di essere rilasciate nel PBS.
- Mantenere le ghiandole salivari in PBS ghiacciato fino a quando 10 individui sono stati sezionati. Pool Ten SG per l'estrazione dell'RNA utilizzando il metodo guanidinium tiocianato-fenolo-cloroformio. Sospendere il pellet di RNA in 30 μL di acqua priva di RNasi.
- Utilizzare 1 μL di aliquota dell'RNA estratto dal SG nella fase precedente, per leggere l'assorbanza a 260 e 280 nm e calcolare la concentrazione di RNA di ciascun campione moltiplicando per il fattore di diluizione. Un rapporto 260/280 di ~ 2,0 indica un RNA di buona qualità.
- Utilizzare 1 μg di RNA purificato per sintetizzare dna complementare (cDNA) utilizzando un kit commerciale di trascrizione inversa.
- Effettuare una diluizione 1:10 del cDNA per preparare una reazione RT-PCR secondo le raccomandazioni del produttore. Per ogni campione, preparare una reazione per il gene bersaglio e, in parallelo, impostare una reazione con il gene housekeeping (HK). Impostare ogni reazione genica in un triplicetto tecnico per eliminare l'impatto della variazione casuale dal metodo.
NOTA: Qui, il gene S7 ribosomiale An. gambiae (GeneBank: L20837.1) e l'actina (VectorBase: AGAP000651) sono stati utilizzati come geni HK. - Utilizzare tutti i primer ad una concentrazione finale di 300 nM, seguendo le indicazioni del produttore SYBR-green. Amplificare con condizioni PCR standard: 95 °C per 10 min, seguiti da 40 cicli di 15 s a 95 °C e 60 s a 60 °C.
NOTA: Per quantificare l'espressione genica, viene utilizzato il metodo delta-delta-Ct (ΔΔCt). Delta Ct (ΔCt) è la differenza tra la Tc del gene bersaglio e la Ct del gene housekeeping. ΔΔCt è la differenza tra il ΔCt del gruppo sperimentale e il ΔCt del gruppo di controllo35.
5. Valutazione fenotipica: alimentazione sanguigna di successo
- Per valutare la capacità di nutrirsi per il sangue, impostare gruppi di 15 zanzare femmine trattate con dsRNA bersaglio e controllo su piccole gabbie (diametro 12 cm) e farli morire di fame per 4 ore.
- Utilizzando un bagno d'acqua circolante impostato a 37 °C, gli alimentatori di zanzare in vetro (diametro 24 mm) e la membrana parafilm, offrono sangue di pecora defibrinato alle zanzare.
NOTA: il sangue può essere acquistato da un venditore commerciale che lo preleva in modo asettico da animali sani e donatori di origine statunitense e defibrinati manualmente senza anticoagulanti o additivi. - Con l'osservazione diretta, conta e registra il numero di tentativi di sondaggio per acquisire con successo un pasto di sangue dalle prime cinque femmine a diventare completamente ingorgate in ciascun gruppo.
NOTA: Per evitare cambiamenti metabolici significativi nelle zanzare, che potrebbero interferire con le risorse energetiche influenzando il comportamento di ricerca del sangue, la fame è stata ridotta al minimo (4 ore). Di conseguenza, non tutte le zanzare cercherebbero avidamente la farina di sangue e abbiamo limitato il conteggio delle femmine ingorgate a cinque (un terzo del totale di ciascun gruppo), per ridurre l'effetto di variabili temporali come l'esposizione all'odore umano, il cambiamento di temperatura tra le camere e le superfici di alimentazione, ecc.
6. Valutazione fenotipica: morfologia delle ghiandole salivari e down-regolazione delle proteine rilevanti
- Isolare il tessuto fresco in 1x soluzione salina tamponata con fosfato (PBS) come descritto nel passaggio 4.2 e fissare in acetone ghiacciato per 90 s. Risciacquare più volte in 1x PBS dopo aver rimosso l'acetone. Incubare con anticorpi primari durante la notte a 4 °C con antisiero (vedere Tabella dei materiali) diluito in 1x PBS.
NOTA: Vedere Tabella dei materiali per l'identificazione degli anticorpi primari utilizzati per le proteine della saliva (proteina antipiastrinica Anopheles , AAPP; Mucina 2, MUC2), fattori di trascrizione SG (Fork Head, fkh; Salvia, salvia; Proteina legante l'elemento di risposta ciclico-AMP A, CrebA) e un marcatore di vescicole secretorie (Rab11). Questi anticorpi sono usati come letture per la forma e la funzione SG. Tuttavia, qualsiasi anticorpo adatto per l'immunofluorescenza dovrebbe essere adatto a questo protocollo. - Lavare in 1x PBS più volte. Aggiungere anticorpi secondari (fluorescenti) diluiti in 1x PBS e incubare al buio a temperatura ambiente per 2 ore. Aggiungere qualsiasi controcolorazione [come 4′,6-diamidino-2-fenilindolo (DAPI; DNA), agglutinina di germe di grano (WGA; per chitina), falloidina (per F-actina) e/o rosso Nilo (per i lipidi)] 30 minuti prima della fine dell'incubazione di 2 ore.
- Lavare tre volte in 1x PBS. Quindi, montare i tessuti in glicerolo al 100% su un vetrino per microscopio standard con un coperchio di 1 mm di spessore e conservare a -20 °C fino all'imaging utilizzando un microscopio confocale a fluorescenza.
NOTA: per ottenere dati quantitativi, le impostazioni di imaging devono essere mantenute costanti. Qui, sono state incluse solo immagini di proiezione di massima intensità attraverso l'intero volume 3D del tessuto e tutta la quantificazione dell'immagine è stata normalizzata tra i trattamenti (all'interno di un esperimento) in base al segnale DAPI in resti di tessuto non SG (corpo grasso, cuticola o testa) presenti anche sul vetrino.
Risultati
Per iniziare, i dati di espressione di microarray di VectorBase sono stati utilizzati per scansionare potenziali bersagli attraverso le fasi di sviluppo 36,37 per determinare lo stato di espressione di tutti i geni rilevanti per lo studio corrente (Tabella 1). Come previsto, tutti i geni bersaglio scelti hanno mostrato espressione in SG adulti. I livelli di aapp e salvia erano particolarmente alti (Tabella 1). Degni di nota sono stati anche gli alti livelli di espressione di f-Agdsx nelle SGs9 di sesso femminile adulta.
Segmenti specifici di ciascun gene sono stati valutati per l'uso come dsRNA utilizzando l'applicazione web E-RNAi per la progettazione di reagenti RNAi30. Le regioni ~ 400 bp contenenti sequenze uniche per ciascun gene bersaglio sono state quindi clonate (Figura 1A), trasformate nei ceppi batterici appropriati e utilizzate per preparare sospensioni di batteri uccisi dal calore, che sono stati indotti a produrre dsRNA. Le zanzare adulte sono state alimentate per 8 giorni sui batuffoli di cotone imbevuti di saccarosio contenenti le sospensioni batteriche di dsRNA per f-Agdsx, fkh o formica (il controllo negativo non correlato).
Per l'analisi dell'alimentazione RNAi delle zanzare femmine, è stato prima determinato se f-Agdsx o fkh dsRNA-feedings inducessero il silenziamento genico. Una riduzione del 98,8% (±2,1) dei livelli di trascrizione fkh è stata osservata nel gruppo alimentato con fkh-dsRNA (Figura 1B), indicando che il dsRNA ha ridotto molto efficacemente l'abbondanza di trascritti fkh nei SG. Sorprendentemente, i livelli di fkh mRNA sono stati ridotti dell'82,0% (±18,9) nelle zanzare trattate con dsRNA per f-Agdsx, che avevano un 89,86% (±4,48) di riduzione di f-Agdsx , suggerendo che fkh potrebbe essere un bersaglio di F-Dsx nella ghiandola salivare. In concomitanza con la significativa riduzione dei livelli di espressione di fkh, le zanzare fkh-knockdown hanno mostrato un aumento significativo del numero di tentativi di sondaggio necessari per l'alimentazione del sangue. Queste zanzare hanno mostrato, in media, cinque volte più tentativi di alimentazione rispetto al gruppo di controllo o alle zanzare alimentate con f-Agdsx dsRNA per essere completamente ingorgate di sangue (Figura 1C). Ciò ha portato a chiedersi se i trattamenti RNAi knockdown fkh abbiano causato cambiamenti nella localizzazione e / o distribuzione dei regolatori trascrizionali chiave (SG TF Sage e CrebA) (Figura 2), proteine secrete (AAPP e mucina) (Figura 3) e macchinari secretori [Nile Red (lipidi) e Rab11 (vescicole secretorie)] (Figura 4). È importante sottolineare che sono state osservate differenze sostanziali nell'intensità della colorazione in diverse regioni del lobo, lobi e singoli SG.
Come previsto, i livelli di colorazione di salvia e CrebA sono stati marcatamente ridotti in tutti i lobi SG dopo fkh RNAi (Figura 2B) rispetto all'RNAi di controllo delle formiche (Figura 2A). Le riduzioni sia dei valori di intensità massima più alti (linee tratteggiate rosse ed etichette numeriche) che dei valori di intensità massima più bassi (linee tratteggiate blu ed etichette numeriche) nei profili di scansione delle linee hanno suggerito riduzioni nelle aree di segnale sia alto che basso all'interno del tessuto (Figure 2A, B). Questi dati suggeriscono che An. gambiae fkh RNAi è efficace e che fkh regola la produzione e/o la stabilità dei TF SG Sage e CrebA in An. gambiae, analogamente alla loro relazione genetica in Drosophila SGs 19,38,39.
Quando si considerano proteine salivari molto abbondanti, i livelli di proteina antipiastrinica Anopheles (AAPP)40,41 sono stati ridotti in tutti e tre i lobi SG dopo fkh RNAi, rispetto al trattamento con RNAi di controllo (Figura 3A,B; verde). D'altra parte, non sono stati osservati cambiamenti nei livelli di mucina (Figura 3A, B; viola). Questi dati suggeriscono che Fkh contribuisce in modo diverso all'espressione di diversi geni della proteina della saliva.
Infine, sono stati osservati due marcatori di secrezione (Figure 4A,B): Rab11 (vescicole associate a endosomi di riciclo apicale)42 e Rosso Nilo (lipidi). Una ridotta fluorescenza di Rab11 è stata osservata nei lobi laterali distali (DL) dopo il trattamento con fkh RNAi (Figura 4A v vs. 4B v; verde). Tuttavia, si è verificato anche un aumento del segnale Rab11 nei lobi mediali (M) e laterali prossimali (PL) (Figura 4A vii, ix vs 4B vii, ix; verde). Nessuna differenza percepibile è stata osservata nel segnale rosso del Nilo (Figure 4A,B; viola) dopo fkh RNAi rispetto al trattamento con RNAi di controllo. Questi dati suggeriscono che la riduzione di fkh può alterare l'azione di alcuni macchinari secretori in un modo complesso che differisce tra i lobi SG.
| Dataset: | Goltsev · | Neira Oviedo | Neira Oviedo | Fornaio | Fornaio | Fornaio | Fornaio | ||
| simbolo del gene | funzione | AGAP ID | embrione (25 ore) | Larve L3 | L3 SG | femmina adulta corpo (3 giorni) | maschio adulto corpo (3 giorni) | femmina adulta SG (3 giorni) | maschio adulto SG (3 giorni) |
| AAPP · | proteine della saliva | AGAP009974 | 3.92 | 4.38 | 4.33 | 3.81 | 2.46 | 11.92 | 2.69 |
| CrebA · | fattore txn | AGAP001464 · | 6.28 | 5.22 | 5.92 | 2.99 | 2.96 | 3.27 | 3.13 |
| " | fattore txn | AGAP011038 | 4.50 | 4.46 | 5.23 | 2.96 | 2.86 | 3.05 | 2.88 |
| Dsx | fattore txn | AGAP004050 | 4.91 | 5.39 | 5.55 | 3.72 | 4.00 | 4.57 | 4.01 |
| fkh | fattore txn | AGAP001671 · | 5.18 | 4.67 | 5.25 | 2.99 | 3.09 | 3.21 | 3.05 |
| MUC2 · | proteine della saliva | AGAP012020 | 4.59 | 5.53 | 5.63 | 2.96 | 3.07 | 3.08 | 3.26 |
| Rab11 | traffico vescicolare | AGAP004559 | 10.21 | 7.47 | 8.60 | 4.90 | 3.79 | 3.38 | 2.96 |
| Saggio | fattore txn | AGAP013335 | 5.32 | 5.96 | 8.89 | 3.40 | 3.33 | 7.37 | 7.23 |
Tabella 1: Profili medi di espressione microarray log2 per An. gambiae geni di interesse. Sono mostrati i nomi dei geni, la categoria funzionale, gli identificatori Vectorbase (AGAP) e i dati di espressione del microarray log2 medio raccolti da Vectorbase. Questi dati indicano che i nostri geni di interesse (coinvolti nella biologia cellulare e nella secrezione delle ghiandole salivari (SG)) sono espressi e arricchiti nello stadio larvale 3 (L3) e negli SG adulti, rispetto a individui interi.
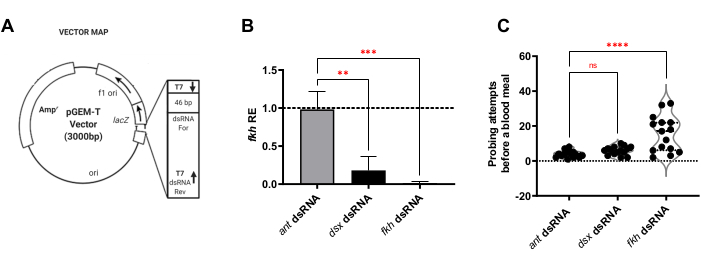
Figura 1: f-Agdsx e fkh knockdown in An. gambiae adulto riduce i livelli di fkh mRNA negli SG e influenza la capacità femminile di nutrirsi per il sangue. (A) Immagine rappresentativa del disegno del plasmide utilizzato per la produzione di dsRNA in questa metodologia. La seconda sequenza del promotore T7 viene aggiunta al plasmide includendola nel primer 3' utilizzato per amplificare l'inserto da clonare nel plasmide pGEMT. Il plasmide viene quindi trasformato in batteri E. coli HT115 (DE3) e una soluzione di alimentazione è costituita da una sospensione di batteri indotti uccisi dal calore in acqua zuccherata al 10%. (B) Gli animali nutriti con una soluzione di alimentazione a dsRNA per f-Agdsx o fkh, hanno mostrato livelli significativamente più bassi di trascritti fkh (ANOVA unidirezionale con confronti multipli; n = 15). Tuttavia, solo il gruppo alimentato con fkh dsRNA (C) ha mostrato una differenza significativa nel numero di tentativi di morso necessari per acquisire un pasto di sangue. Le zanzare di questo gruppo avevano bisogno, in media, di cinque volte il numero di tentativi di sondaggio per ottenere un pasto di sangue di successo rispetto a quello richiesto dal controllo o dai gruppi alimentati con dsx-dsRNA (ANOVA unidirezionale con confronti multipli; n = 15). Le barre di errore indicano l'errore standard della media (SEM). Ogni esperimento è stato condotto in tre repliche biologiche separate. Fare clic qui per visualizzare una versione più grande di questa figura.
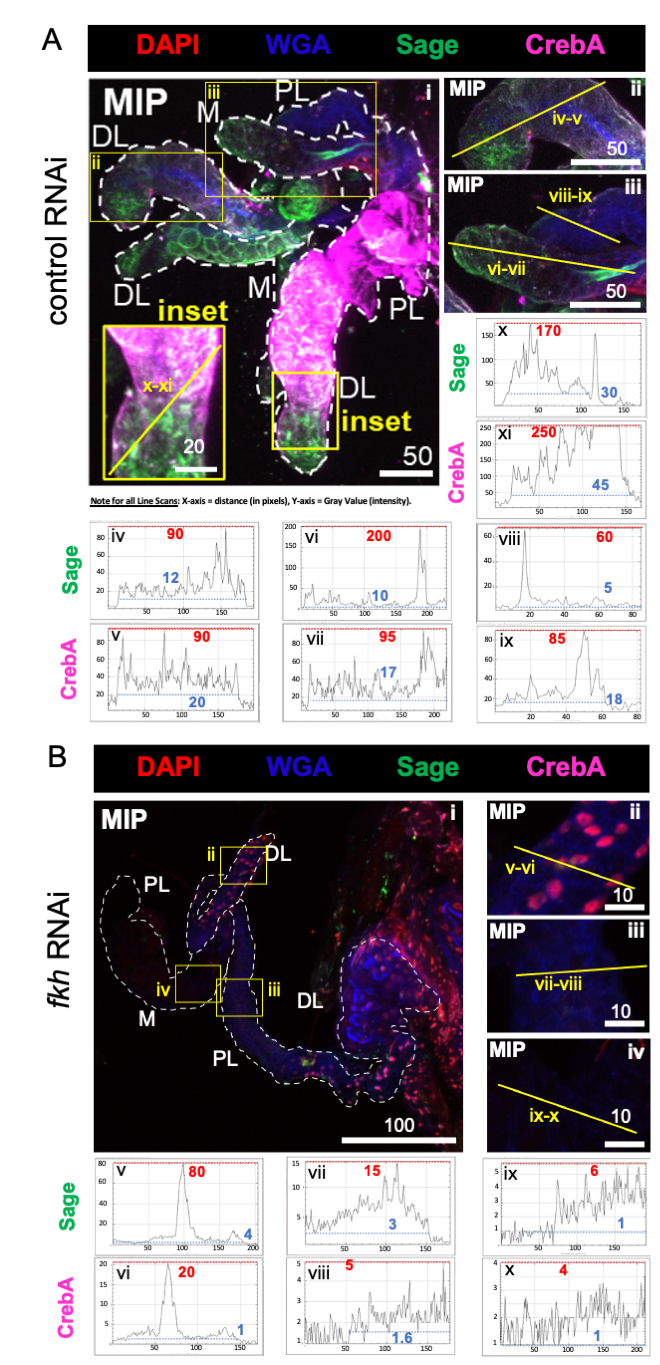
Figura 2: fkh knockdown nelle ghiandole salivari an. gambiae adulte riduce i livelli del fattore di trascrizione SG. Sono mostrate immagini rappresentative del giorno 13 femmina adulta An. gambiae SG dopo 8 giorni (giorni 5-13) di esposizione orale a (A) controllo dsRNA non correlato (formica) o (B) dsRNA mirato alla testa della forcella SG TF (fkh, AGAP001671) in 10% di saccarosio colorato con i coloranti DAPI (DNA; rosso), agglutinina di germe di grano etichettato (WGA, chitina / O-GlcNAcilazione; blu), antisieri contro i TF SG Sage (verde) e CrebA (viola). Le lunghezze delle barre di scala mostrate sono micron. Gli SG (i) sono delineati con trattini bianchi. Le linee gialle nelle immagini del lobo ingrandita (delle regioni racchiuse da caselle gialle ed etichettate "inset") indicano dove sono state condotte le scansioni di linea dell'intensità del segnale. Le intensità dei canali verdi e viola corrispondenti alle scansioni delle linee per ciascun lobo ingrandito sono tracciate (sempre da sinistra a destra nel SG) nei grafici sotto le immagini; Asse X = distanza (in pixel) e asse Y = unità grigia (intensità pixel). La gamma dinamica dell'intensità dei pixel è delimitata da linee tratteggiate rosse (massimo) e blu (minimo) e i valori corrispondenti sono mostrati su ciascun grafico. MIP = proiezione 3D di massima intensità attraverso l'intera profondità SG. DL: lobo laterale distale; M: lobo mediale; PL: lobo laterale prossimale; SD: dotto salivare. Fare clic qui per visualizzare una versione più grande di questa figura.
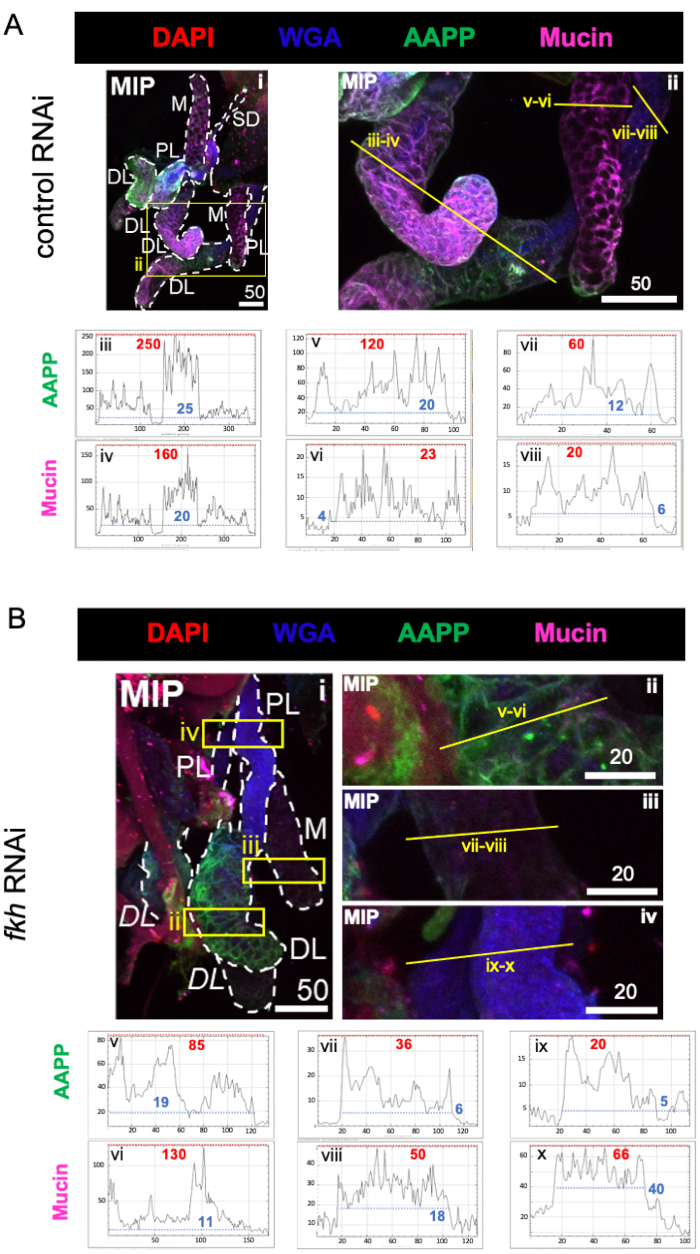
Figura 3: l'abbattimento di fkh nelle ghiandole salivari di An. gambiae adulte riduce i livelli di proteine secrete SG. Sono mostrate immagini rappresentative del giorno 13 femmina adulta An. gambiae SG dopo 8 giorni (giorni 5-13) di esposizione orale a (A) controllo dsRNA non correlato (formica), o (B) dsRNA mirato alla testa della forcella SG TF (fkh, AGAP001671) in 10% di saccarosio colorato con i coloranti DAPI (DNA; rosso), agglutinina del germe di grano etichettato (WGA, chitina / O-GlcNAcilazione; blu), e le proteine della saliva AAPP (verde) e Muucin (MUC2, viola). Le lunghezze delle barre di scala mostrate sono micron. Gli SG (i) sono delineati con trattini bianchi. Le linee gialle nelle immagini del lobo ingrandita (delle regioni racchiuse da caselle gialle) indicano dove sono state condotte le scansioni di linea dell'intensità del segnale. Le intensità dei canali verdi e viola corrispondenti alle scansioni delle linee per ciascun lobo sono tracciate (sempre da sinistra a destra nel SG) nei grafici sotto le immagini; Asse X = distanza (in pixel) e asse Y = unità grigia (intensità pixel). La gamma dinamica dell'intensità dei pixel è delimitata da linee tratteggiate rosse (massimo) e blu (minimo) e i valori corrispondenti sono mostrati su ciascun grafico. MIP = proiezione 3D di massima intensità attraverso l'intera profondità SG. DL: lobo laterale distale; M: lobo mediale; PL: lobo laterale prossimale; SD: dotto salivare. Le etichette "DL" in corsivo (Bi) indicano due regioni visibili dello stesso lobo DL. Fare clic qui per visualizzare una versione più grande di questa figura.
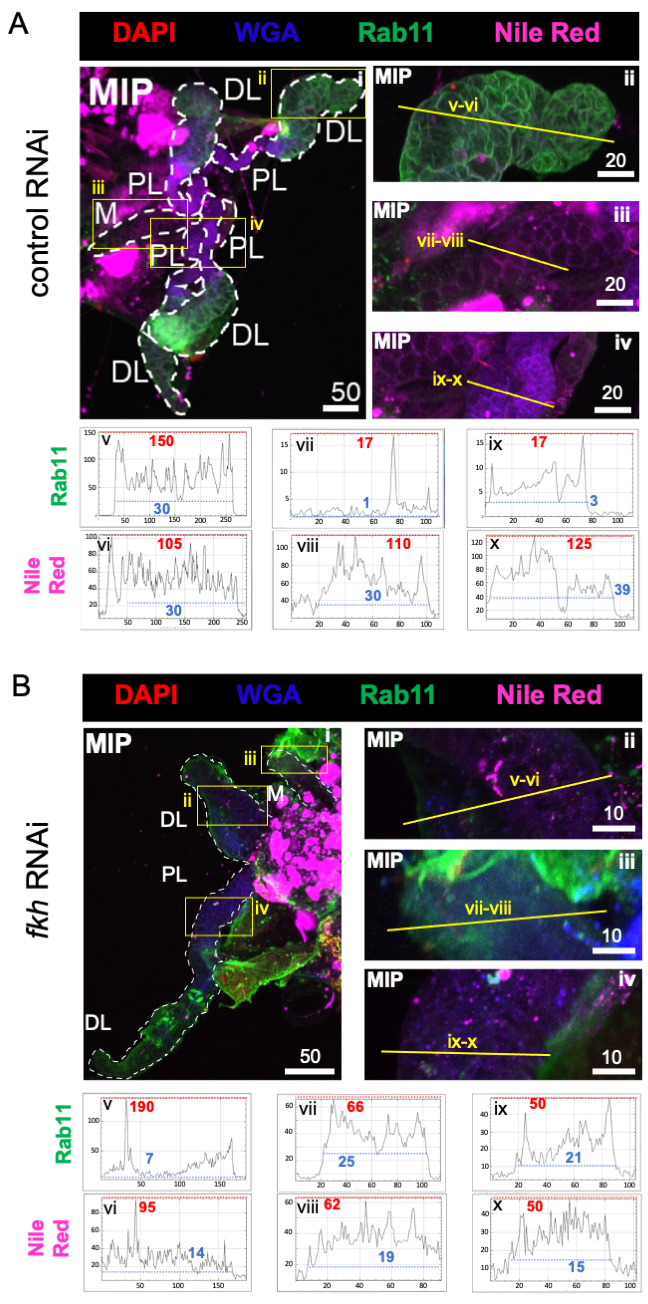
Figura 4: l'abbattimento di fkh nelle ghiandole salivari di An. gambiae adulte riduce i marcatori di secrezione SG. Sono mostrate immagini rappresentative del giorno 13 femmina adulta An. gambiae SG dopo 8 giorni (giorni 5-13) di esposizione orale a (A) controllo dsRNA non correlato (formica), o (B) dsRNA mirato alla testa della forcella SG TF (fkh, AGAP001671) in 10% di saccarosio colorato con i coloranti DAPI (DNA; rosso), agglutinina del germe di grano etichettato (WGA, chitina / O-GlcNAcilazione; blu), Rosso Nilo (lipidi; viola) e antisieri contro il marcatore di vescicole endosomitiche riciclate Rab11 (verde). Le lunghezze delle barre di scala mostrate sono micron. Gli SG (i) sono delineati con trattini bianchi. Le linee gialle nelle immagini del lobo ingrandita (delle regioni racchiuse da caselle gialle) indicano dove sono state condotte le scansioni di linea dell'intensità del segnale. Le intensità dei canali verdi e viola corrispondenti alle scansioni delle linee per ciascun lobo sono tracciate (sempre da sinistra a destra nel SG) nei grafici sotto le immagini; Asse X = distanza (in pixel) e asse Y = unità grigia (intensità pixel). La gamma dinamica dell'intensità dei pixel è delimitata da linee tratteggiate rosse (massimo) e blu (minimo) e i valori corrispondenti sono mostrati su ciascun grafico. MIP = proiezione 3D di massima intensità attraverso l'intera profondità SG. DL: lobo laterale distale; M: lobo mediale; PL: lobo laterale prossimale; SD: dotto salivare. Fare clic qui per visualizzare una versione più grande di questa figura.
Fascicolo complementare 1. Fare clic qui per scaricare questo file.
Discussione
La capacità di fornire efficacemente dsRNA alle zanzare An. gambiae mediante alimentazione orale ha ampie implicazioni per gli studi di biologia vettoriale sia in laboratorio che sul campo. La microiniezione è stata a lungo accettata come la modalità preferita di somministrazione di sostanze chimiche, anticorpi, RNAi e strategie di modificazione genetica nelle zanzare43,44. La conseguenza di una sostanziale manipolazione fisica, danni cellulari e stress può essere evitata con l'uso della somministrazione orale, che potrebbe anche essere potenzialmente adatta per applicazioni su larga scala o sul campo. Lavori precedenti hanno suggerito che l'RNAi agisce ubiquitariamente all'interno di una singola zanzara adulta29, consentendo effetti in tutti i tessuti, comprese le ghiandole salivari. Alimentando le zanzare con un gran numero di E. coli che esprimono dsRNA che vengono digeriti in modo asincrono per un lungo periodo di tempo, si può potenzialmente ottenere un'esposizione coerente e uniforme all'RNAi in tutti gli individui in una gabbia. Questo metodo consente di nutrire un gran numero di zanzare e analizzare la potenziale variabilità dei fenotipi risultanti a seconda del gene bersaglio. Tuttavia, una considerazione importante è la possibilità di distribuzione eterogenea dei batteri, e quindi del dsRNA, nella fibra di cotone. I 400 μL di batteri utilizzati quotidianamente per l'alimentazione con zucchero delle zanzare conterrebbero circa ≤ 4,6 μg di dsRNA, come descritto e calcolato in precedenza9, ma la quantità di dsRNA ingerita da ciascuna zanzara non è stata determinata individualmente. Se la costruzione di costrutti dsRNA diventa di routine, questo semplice protocollo di trattamento consente una rapida assimilazione di questa tecnica da parte di qualsiasi ricercatore di zanzare. A priori, il dispendio di tempo durante il trattamento (30 min al giorno) è banale rispetto al tempo impiegato per imparare e applicare la microiniezione a campioni di dimensioni simili.
L'alimentazione di dsRNA viene abitualmente utilizzata per studi di genetica inversa nell'organismo modello Caenorhabditis elegans45. Questo pesante livello di utilizzo sottolinea il valore dell'approccio di consegna orale. La costruzione di una libreria a livello di genoma di An. gambiae in E. coli trasformato, simile a quella che esiste in C. elegans46,47, consentirebbe un rapido screening genetico inverso nelle zanzare su scala aumentata. Tuttavia, è importante notare che l'efficacia del metodo dipende in larga misura dai livelli endogeni di trascrizione e se l'espressione non è limitata al tessuto bersaglio ma espressa più ampiamente 4,8,44. Inoltre, ci sono prove che alcuni insetticidi potrebbero indurre l'evitamento comportamentale dalle zanzare48 e l'alimentazione con batteri che potenzialmente inducono effetti avversi in essi potrebbe innescare modelli simili di evitamento. Nell'ambiente controllato del laboratorio, dove le zanzare non avevano una fonte di cibo alternativa, non avevano la possibilità di evitare l'acqua zuccherata con E. coli e la necessità di una fonte nutriente avrebbe probabilmente prevalso sull'istinto di evitare i batteri. Tuttavia, questo dovrebbe essere considerato se la strategia fosse destinata ad essere utilizzata in ambienti meno controllati.
Potrebbe essere possibile indirizzare più geni contemporaneamente (usando un costrutto, più costrutti o una miscela di isolati batterici trasformati), ma sono necessari ulteriori studi per valutare l'efficacia. Un'altra considerazione importante a questo punto è la valutazione di possibili effetti off-target o sinergici quando si utilizzano bersagli singoli o multipli. La creazione di geni e gruppi di controllo appropriati è una parte importante del disegno sperimentale. Inoltre, si è tentati di ipotizzare che questo approccio potrebbe essere utilizzato per colpire altri agenti patogeni o virus49. Il lavoro precedente verso l'induzione dell'RNAi nelle zanzare è stato eseguito in condizioni in cui il reagente è stato iniettato direttamente, quindi E. coli non era presente. L'E. coli può fornire un compartimento protettivo che consente il rilascio più lento di dsRNA nel tempo, assicurando che l'esposizione sia più o meno continua per un periodo molto più lungo29.
Infine, questi risultati mostrano che gli effetti di questa tecnica sono sintonizzabili regolando il lasso di tempo (lunghezza e giorno di inizio) dell'esposizione e la quantità di E. coli utilizzata. Questa caratteristica ci ha permesso di studiare le funzioni dei geni essenziali (dsx e fkh) identificando condizioni ottimali di abbattimento per tentativi ed errori. Ciò aumenta notevolmente la probabilità che i geni bersaglio di interesse possano essere studiati utilizzando questa tecnica.
In sintesi, è stato riscontrato che la somministrazione orale di RNAi alle zanzare adulte può essere semplice, versatile e un approccio potente allo studio della funzione del gene della zanzara e per la creazione di strumenti nuovi e malleabili per il controllo vettoriale delle malattie trasmesse dalle zanzare.
Divulgazioni
Gli autori riferiscono di non avere conflitti di interesse da divulgare.
Riconoscimenti
Gli autori desiderano ringraziare lo staff e gli scienziati all'interno del ramo di entomologia e della divisione di malattie parassitarie e malaria presso il CDC, e Brian Trigg e Michelle Chiu per l'assistenza con la preparazione dei batteri presso JHU e / o utili discussioni su questo lavoro. Ringraziamo l'insetto JHMRI e il manager Chris Kizito per l'accesso e l'allevamento delle zanzare An. gambiae . Ringraziamo Wei Huang (JHSPH) per l'assistenza nell'ottenere plasmidi PJet GFP e pPB47 GFP per l'uso in questo studio. Il finanziamento per questo lavoro è stato fornito da: NIH R21AI153588 (a DJA), una johns Hopkins Malaria Research Institute Postdoctoral Fellowship (a MW); e da una sovvenzione della Good Ventures Foundation e dell'Open Philanthropy Project alla CDC Foundation intitolata Support cryopreservation and suppression of female development in mosquitoes to assist research for malaria, Open Philanthropy Project, 2017. Apprezziamo profondamente l'assistenza del personale della JHU Microscope Facility e il supporto della sovvenzione NIH applicabile per il microscopio utilizzato (NIH Grant #: S10OD016374). I risultati e le conclusioni di questo manoscritto sono quelli degli autori e non rappresentano necessariamente le opinioni del CDC. L'uso dei nomi commerciali è solo per l'identificazione e non implica l'approvazione da parte dei Centers for Disease Control and Prevention, del Servizio sanitario pubblico o del Dipartimento della salute e dei servizi umani degli Stati Uniti.
Materiali
| Name | Company | Catalog Number | Comments |
| 1 Kb Plus DNA Ladder | Thermo Fisher Scientific | 10787018 | |
| 2x Yeast Extract Tryptone (2xYT) Medium | BD Difco | DF0440-17 | |
| AAPP | n/a | n/a | Antisera. 1:50 dilution (rabbit); gift from Fabrizio Lombardo |
| AccuStart II PCR Supermix | Quantabio | 95137-100 | |
| Agarose | Millipore Sigma | A9539 | |
| Ampicillin | Millipore Sigma | A5354 | |
| Anopheles gambiae G3 | BioDefense and Emerging Infections (BEI) Malaria Research and Reference Reagent Resource Center (MR4) | MRA-112 | |
| BugDorm | BioQuip | 1452 | |
| Centrifuge 5810R | Eppendorf | P022628181 | |
| CrebA | DSHB | CrebA Rbt-PC | Antisera. 1:50 dilution (rabbit); generated by the Andrew Lab |
| Damiens diet | BioServ | ||
| DAPI | Life Technologies | n/a | 4′,6-diamidino-2-phenylindole; 1:200 dilution. |
| Defibrinated sheep blood | HemoStat | DSB050 | |
| Escherichia coli HT115 (DE3) | |||
| Ethidium bromide | Millipore Sigma | E7637 | |
| High-Capacity cDNA Reverse Transcription Kit | Thermo Fisher Scientific | 4368814 | |
| Isopropyl β-D-1-thiogalactopyranoside | Millipore Sigma | I5502 | |
| JM109 Competent cells | Promega | L2005 | |
| Luria Broth Media | Thermo Fisher Scientific | 10855001 | |
| Mucin 2 | Proteintech | Muc2; 27 675-1-AP | Antisera. 1:100 dilution (mouse). |
| Nanodrop 2000 | Thermo Fisher Scientific | ||
| Nile Red | Sigma | n/a | Lipid dye; 1:50 dilution. |
| Owl EasyCast B2 Mini Gel Horizontal Electrophoresis | Thermo Fisher Scientific | Model B2 | |
| pGEMT easy | Promega | A3600 | |
| Power SYBR-green PCR master MIX | Applied Biosystems | 4367659 | |
| PureLink PCR purification kit | Thermo Fisher Scientific | K31001 | |
| QuantaStudio 6 | Applied Biosystems | ||
| QuantStudio6 Real Time PCR System | Applied Biosystems | ||
| Rab11 | n/a | n/a | Antisera. 1:100 dilution (rabbit); generated by the Andrew Lab |
| Rh-WGA | Vector Labs | n/a | Rhodamine-conjugated wheat germ agglutinin (chitin, O-GlcNAcylation dye); 1:40 dilution |
| Sage | n/a | n/a | Antisera. 1:50 dilution (rat); generated by the Andrew Lab |
| T4 DNA ligase | Promega | M1801 | |
| Tetracycline | Millipore Sigma | 87128 | |
| Trizol | Thermo Fisher Scientific | 15596018 | |
| Zeiss LSM700 fluorescence confocal microscope | Zeiss | ||
| ANTIBODIES | |||
| Chicken anti-Rat IgG (H+L), Alexa Fluor 647 | Thermo Fisher Scientific | A21472 | |
| Goat anti-Mouse IgG (H+L), Alexa Fluor 647 | Thermo Fisher Scientific | A28181 | |
| IgG (H+L) Goat anti-Rabbit, Alexa Fluor 488 | Thermo Fisher Scientific | A27034 | |
| Rabbit anti-Goat IgG (H+L), Alexa Fluor 488 | Thermo Fisher Scientific | A27012 | |
| PRIMERS | |||
| ACT-2f: TACAACTCGATCATGAAGTGCGA | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| ACT-3r: CCCGGGTACATGGTGGTACCGC CGGA | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| FKH_RNAi_F: GCCGACTTATGCTTAGCCCA | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| FKH_RNAi_R: TAGCCGTCAATTCCTCCTGC | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| newDSX-f: AGAGGGCGGGGAAATTCTAGT | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| newDSX-r: GGGCTTGTGGCAGTACGAATA | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| S7qf1: AGAACCAGCAGACCACCATC | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| S7qr1: GCTGCAAACTTCGGCTATTC | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
Riferimenti
- Hoa, N. T., Keene, K. M., Olson, K. E., Zheng, L. Characterization of RNA interference in an Anopheles gambiae cell line. Insect Biochemistry and Molecular Biology. 33, 949-957 (2003).
- Caplen, N., Zheng, Z., Falgout, B., Morgan, R. Inhibition of viral gene expression and replication in mosquito cells by dsRNA-triggered RNA interference | Elsevier enhanced reader. Molecular Therapy. 6, 243-251 (2002).
- Brown, A. E., Catteruccia, F. Toward silencing the burden of malaria: progress and prospects for RNAi-based approaches. BioTechniques. , 38-44 (2006).
- Airs, P. M., Bartholomay, L. C. RNA interference for mosquito and mosquito-borne disease control. Insects. 8, (2017).
- Blandin, S., et al. Reverse genetics in the mosquito Anopheles gambiae: targeted disruption of the Defensin gene. EMBO Reports. 3 (9), 852-856 (2002).
- Garver, L., Dimopoulos, G. Protocol for RNAi assays in adult mosquitoes (A. gambiae). Journal of Visualized Experiments: JoVE. (5), e230 (2007).
- Whyard, S., et al. Silencing the buzz: a new approach to population suppression of mosquitoes by feeding larvae double-stranded RNAs. Parasites & Vectors. 8, 96 (2015).
- Wiltshire, R. M., Duman-Scheel, M. Advances in oral RNAi for disease vector mosquito research and control. Current Opinion in Insect Science. 40, 18-23 (2020).
- Taracena, M. L., Hunt, C. M., Benedict, M. Q., Pennington, P. M., Dotson, E. M. Downregulation of female doublesex expression by oral-mediated RNA interference reduces number and fitness of Anopheles gambiae adult females. Parasites & Vectors. 12, 170 (2019).
- Grassi, B. Studi di uno zoologo sulla malaria. Real Accademia dei Lincei. 3, 229 (1901).
- Smith, R. C., Jacobs-lorena, M. Plasmodium - Mosquito interactions: A tale of roadblocks and detours. Advances in Insect Physiology. 39, (2010).
- Das, S., et al. Transcriptomic and functional analysis of the Anopheles gambiae salivary gland in relation to blood feeding. BMC Genomics. 11, 1-14 (2010).
- Francischetti, I. M. B., Valenzuela, J. G., Pham, V. M., Garfield, M. K., Ribeiro, J. M. C. Toward a catalog for the transcripts and proteins (sialome) from the salivary gland of the malaria vector Anopheles gambiae. Journal of Experimental Biology. 205, 2429-2451 (2002).
- Henderson, K. D., Isaac, D. D., Andrew, D. J. Cell fate specification in thedrosophila salivary gland: The integration of homeotic gene function with the DPP signaling cascade. Developmental Biology. 205, 10-21 (1999).
- Mach, V., Ohno, K., Kokubo, H., Suzuki, Y. The Drosophila fork head factor directly controls larval salivary gland-specific expression of the glue protein gene Sgs3. Nucleic Acids Research. 24 (12), 2387-2394 (1996).
- Weiserova, M., et al. Mini-Mu transposition of bacterial genes on the transmissible plasmid. Folia Microbiologica. 32 (5), 368-375 (1987).
- Abrams, E. W., Mihoulides, W. K., Andrew, D. J. Fork head and Sage maintain a uniform and patent salivary gland lumen through regulation of two downstream target genes, PH4αSG1 and PH4αSG2. Development. 133, 3517-3527 (2006).
- Myat, M. M., Isaac, P. P., Andrew, D. J. Early genes required for salivary gland fate determination and morphogenesis in Drosophila melanogaster. Advances in Dental Research. 14, 89-98 (2000).
- Fox, R. M., Vaishnavi, A., Maruyama, R., Andrew, D. J. Organ-specific gene expression: the bHLH protein Sage provides tissue specificity to Drosophila FoxA. Development of Cell Biology. 140, 2160-2171 (2013).
- Maruyama, R., Grevengoed, E., Stempniewicz, P., Andrew, D. J. Genome-wide analysis reveals a major role in cell fate maintenance and an unexpected role in endoreduplication for the Drosophila FoxA gene fork head. PLOS ONE. 6, 20901 (2011).
- Johnson, D. M., et al. CrebA increases secretory capacity through direct transcriptional regulation of the secretory machinery, a subset of secretory cargo, and other key regulators. Traffic. 21, 560-577 (2020).
- Fox, R. M., Hanlon, C. D., Andrew, D. J. The CrebA/Creb3-like transcription factors are major and direct regulators of secretory capacity. Journal of Cell Biology. 191, 479-492 (2010).
- Abrams, E. W., Andrew, D. J. CrebA regulates secretory activity in the Drosophila salivary gland and epidermis. Development. 132, 2743-2758 (2005).
- Wells, M. B., Andrew, D. J. Anopheles salivary gland architecture shapes plasmodium sporozoite availability for transmission. mBio. 10 (4), 01238 (2019).
- Pei-Wen, L., Xiao-Cong, L., Jin-Bao, G., Yan, L., Xiao-Guang, C. Molecular cloning, characterization and expression analysis of sex determiantion gene doublesex from Anopheles gambiae (Diptera: Culicidae). Acta Entomologica Sinica. 58 (2), 122-131 (2015).
- Scali, C., Catteruccia, F., Li, Q., Crisanti, A. Identification of sex-specific transcripts of the Anopheles gambiae doublesex gene. The Journal of Experimental Biology. 208, 3701-3709 (2005).
- Price, D. C., Egizi, A., Fonseca, D. M. Characterization of the doublesex gene within the Culex pipiens complex suggests regulatory plasticity at the base of the mosquito sex determination cascade. BMC Evolutionary Biology. 15, 1-13 (2015).
- Mysore, K., et al. siRNA-mediated silencing of doublesex during female development of the dengue vector mosquito Aedes aegypti. PLoS Neglected Tropical Diseases. 9, 1-21 (2015).
- Boisson, B., et al. Gene silencing in mosquito salivary glands by RNAi. FEBS Letters. 580, 1988-1992 (2006).
- Horn, T., Boutros, M. E-RNAi: a web application for the multi-species design of RNAi reagents-2010 update. Nucleic Acids Research. 38, 332-339 (2010).
- Taracena, M. L., et al. Genetically modifying the insect gut microbiota to control chagas disease vectors through systemic RNAi. PLoS Neglected Tropical Diseases. 9, (2015).
- Sambrook, J., Fritsch, E. F., Maniatis, T. . Molecular Cloning: A Laboratory Manual. , (1989).
- Ullmann, A., Jacob, F., Monod, J. Characterization by in vitro complementation of a peptide corresponding to an operator-proximal segment of the β-galactosidase structural gene of Escherichia coli. Journal of Molecular Biology. 24, 339-343 (1967).
- Timmons, L. Bacteria-mediated RNAi-General outline. Carnegie Institution of Washington. , (2000).
- Pfaffl, M. W. Relative quantification. Real-time PCR. , 63-82 (2004).
- Neira-Oviedo, M., et al. The RNA-Seq approach to studying the expression of mosquito mitochondrial genes. Insect Molecular Biology. 20, 141-152 (2011).
- Baker, D. A., et al. A comprehensive gene expression atlas of sex- and tissue-specificity in the malaria vector, Anopheles gambiae. BMC Genomics. 12, (2011).
- Loganathan, R., Hoon, J., Wells, M. B., Andrew, D. J. Secrets of secretion - How studies of the Drosophila salivary gland have informed our understanding of the cellular networks underlying secretory organ form and function. Cellular Networks in Development. , 143 (2021).
- Chung, S., Hanlon, C. D., Andrew, D. J. Building and specializing epithelial tubular organs: The Drosophila salivary gland as a model system for revealing how epithelial organs are specified, form and specialize. Wiley Interdisciplinary Reviews: Developmental Biology. 3, 281-300 (2014).
- Yoshida, S., et al. Inhibition of collagen-induced platelet aggregation by anopheline antiplatelet protein, a saliva protein from a malaria vector mosquito. Blood. 111, 2007-2014 (2008).
- Wells, M. B., Villamor, J., Andrew, D. J. Salivary gland maturation and duct formation in the African malaria mosquito Anopheles gambiae. Scientific Reports. 7 (1), 601 (2017).
- Takahashi, S., et al. Rab11 regulates exocytosis of recycling vesicles at the plasma membrane. Journal of Cell Science. 125, 4049-4057 (2012).
- Catteruccia, F., Levashina, E. A. RNAi in the malaria vector, Anopheles gambiae. Methods in Molecular Biology. 555, 63-75 (2009).
- Balakrishna Pillai, A., et al. RNA interference in mosquito: understanding immune responses, double-stranded RNA delivery systems and potential applications in vector control. Insect Molecular Biology. 26, 127-139 (2017).
- Fire, A., et al. Potent and specific genetic interference by double-stranded RNA in caenorhabditis elegans. Nature. 391, 806-811 (1998).
- Kamath, R. S., Ahringer, J. Genome-wide RNAi screening in Caenorhabditis elegans. Methods. 30, 313-321 (2003).
- Kamath, R. S., et al. Systematic functional analysis of the Caenorhabditis elegans genome using RNAi. Nature. 421, 231-237 (2003).
- Carrasco, D., et al. Behavioural adaptations of mosquito vectors to insecticide control. Current Opinion in Insect Science. 34, 48-54 (2019).
- Magalhaes, T., et al. Induction of RNA interference to block Zika virus replication and transmission in the mosquito Aedes aegypti. Insect Biochemistry and Molecular Biology. , 111 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon