Method Article
Administración efectiva de ARN oral de interferencia (ARNi) a mosquitos adultos Anopheles gambiae
En este artículo
Resumen
La administración oral de dsRNA producido por bacterias, un método de administración para la interferencia de ARN (RNAi) que se usa rutinariamente en Caenorhabditis elegans, se aplicó con éxito aquí a mosquitos adultos. Nuestro método permite estudios robustos de genética inversa y estudios de vectores que bloquean la transmisión sin el uso de inyección.
Resumen
La interferencia de ARN ha sido una herramienta muy utilizada para el análisis genético inverso durante dos décadas. En mosquitos adultos, la administración de ARN de doble cadena (dsRNA) se ha logrado principalmente a través de la inyección, que requiere un tiempo significativo y no es adecuada para aplicaciones de campo. Para superar estas limitaciones, aquí presentamos un método más eficiente para la activación robusta de ARNi mediante la administración oral de dsRNA a Anopheles gambiae adulto. Se produjeron dsRNAs largos en la cepa HT115 (DE3) de Escherichia coli , y se ofreció una suspensión concentrada de bacterias que contienen dsRNA muertas por calor en sacarosa al 10% en bolas de algodón ad-libitum a mosquitos adultos. Las bolas de algodón se reemplazaron cada 2 días durante la duración del tratamiento. El uso de este método para atacar el doble sexo (un gen involucrado en la diferenciación sexual) o la cabeza de horquilla (que codifica un factor de transcripción de la glándula salival) dio como resultado una reducción de la expresión génica objetivo y / o la señal de inmunofluorescencia de proteínas, medida por PCR cuantitativa en tiempo real (qRT-PCR) o microscopía confocal de fluorescencia, respectivamente. También se observaron defectos en la morfología de las glándulas salivales. Este método altamente flexible, fácil de usar, de bajo costo y eficiente en el tiempo de administración de dsRNA podría ser ampliamente aplicable a genes objetivo importantes para la fisiología de los insectos vectores y más allá.
Introducción
Muchas enfermedades son transmitidas por mosquitos, por lo que el estudio de la fisiología y la genética de los mosquitos es una tarea importante. El uso de ARNi en estos organismos ha sido prominente en los últimos 20 años y ha permitido la caracterización funcional de muchos genes de mosquitos 1,2,3,4,5. La técnica más utilizada para la administración de dsRNA ha sido la microinyección, que tiene los inconvenientes de que puede dañar a los mosquitos y requiere mucho tiempo y esfuerzo. Se han probado métodos de administración oral para el ARNi, pero principalmente en la etapa larvaria de los mosquitos 6,7,8,9. La administración oral de dsRNA en mosquitos adultos no se ha explorado completamente y podría ser una herramienta útil para el estudio de la biología y el control de vectores.
La malaria es transmitida por los mosquitos Anopheles cuando un mosquito hembra infectado toma una comida de sangre de un huésped no infectado e inyecta saliva que contiene parásitos de la malaria10. Para transmitirse en última instancia en la saliva de un mosquito, el parásito debe superar muchos obstáculos, incluida la evasión del sistema inmunológico del mosquito, la travesía de la barrera del intestino medio y la invasión de las glándulas salivales11. La arquitectura de las glándulas salivales de los mosquitos (SG) es clave para la invasión de parásitos y esa arquitectura está controlada tanto por factores clave de transcripción expresados por las glándulas salivales como por los determinantes del dimorfismo sexual. Se requieren varios factores de transcripción altamente conservados para la especificación celular y el mantenimiento homeostático de las glándulas salivales y para la producción y secreción de proteínas salivales que funcionan en la alimentación sanguínea 12,13,14. La cabeza de horquilla (Fkh) es un factor de transcripción de hélice alada que funciona como un importante regulador de la estructura y función sg del insecto (basado en estudios en moscas de la fruta y la polilla del gusano de seda)15,16,17,18,19,20. En los SG de Drosophila, Fkh funciona con Sage, un factor de transcripción básico de hélice-bucle-hélice (bHLH) específico de SG, para promover la supervivencia de SG y la producción de saliva19. Un co-regulador importante y positivo de la producción de saliva en Drosophila es CrebA, un factor de transcripción de cremallera de leucina bien estudiado que regula al alza la expresión de los genes de la vía secretora 21,22,23. También existe un fuerte grado de diferenciación morfológica en las glándulas salivales femeninas que probablemente juega un papel clave, no solo en la alimentación sanguínea sino también en la capacidad de los parásitos para invadir este tejido24.
Muchos de los genes involucrados en la determinación de la supervivencia, la estructura, la fisiología y el dimorfismo sexual de las glándulas salivales tienen perfiles complejos de expresión espaciotemporal 25,26,27, y los métodos tradicionales de administración de dsRNA para inducir ARNi no siempre son eficientes para dirigirse a este tipo de genes en este u otros tejidos. Sin embargo, la administración oral de dsRNA en los mosquitos Aedes aegypti y An. gambiae en etapa larvaria se ha utilizado con éxito para silenciar la forma específica de la hembra del gen dsx 9,28. Estudios previos que utilizaron dsRNA en glándulas salivales de mosquitos encontraron que, aunque se requerían grandes cantidades de dsRNA, el efecto silenciador fue relativamente duradero (al menos 13 días)29. Aquí, se probó la capacidad de la cepa de E. coli HT115 (DE3) muerta por calor que expresa dsRNA específico de secuencia para dsx, fkh o CrebA para inducir el silenciamiento de RNAi de estos genes en mosquitos hembra adultos. La administración oral de dsRNA indujo la eliminación de genes en An. gambiae, con claras reducciones en los niveles de ARNm y con fenotipos consistentes con la pérdida de función de estos genes. Por lo tanto, este enfoque probablemente funcionará para derribar la función de una variedad de genes de las glándulas salivales.
Protocolo
1. Clonación de dsRNA en vector de expresión de E. coli
- Seleccione la secuencia de genes diana para insertarla en un vector apropiado para la expresión de dsRNA. Recupere los valores de expresión de Vectorbase.org mediante el método siguiente.
- Busque un gen de interés (por ejemplo, la Tabla 1) en el cuadro de búsqueda de la página de inicio.
- En la página del gen resultante, navegue hasta el 8. Sección de transcriptómica .
- Busque los experimentos de expresión génica de ARN-seq y microarray relevantes enumerados.
- Transcriba valores de interés en el software de hoja de cálculo y crea una tabla de datos.
- Seleccione un plásmido disponible comercialmente con al menos un promotor T7 para ser utilizado. Si el plásmido seleccionado tiene solo un promotor T7 (como la mayoría de los plásmidos comerciales), incluya un segundo promotor T7 en el cebador inverso que se utilizará para la amplificación del dsDNA para el gen de interés.
NOTA: La secuencia de dsRNA para los genes diana se puede seleccionar utilizando la aplicación web E-RNAi para el diseño de reactivos de ARNi30. Se puede diseñar dsRNA largo (aproximadamente 400 pb) o dsRNA de horquilla corta (shRNA) en función de secuencias genéticas específicas. Estas secuencias deben amplificarse y secuenciarse para la confirmación de la identidad antes de la clonación. Las regiones genéticas, plásmidos y promotores seleccionados utilizados en este estudio se enumeran en el Archivo Suplementario 1. - Realizar la clonación de acuerdo con un procedimiento simple de un solo paso descrito anteriormente 9,31. Para este propósito, purificar el producto de PCR y ligar al ADN plásmido linealizado. Utilice el producto de la ligadura para la transformación por choque térmico de células competentes de E. coli 32. Seleccione las celdas transformadas a través de la detección azul/blanca. Confirme la orientación del inserto mediante una PCR de imprimación T7 y confirme la secuencia utilizando cebadores M13.
NOTA: Los exámenes blancos/ azules se pueden usar cuando el plásmido seleccionado para la transformación lleva el gen lacZ que codifica para la β-galactosidasa. Las colonias blancas deben contener el inserto deseado dentro de la lacZ y se pueden seleccionar para confirmar aún más la presencia y orientación de la secuencia objetivo33. - Purificar el plásmido desde la primera transformación y utilizarlo para transformar E. coli HT115 (DE3) competente como se describió anteriormente34. Después de la confirmación de que el plásmido con el inserto está presente en la E. coli HT115 (DE3) competente, haga reservas de glicerol de bacterias para un solo uso.
NOTA: Se debe adquirir o preparar un dsRNA de control no relacionado apropiado para su uso en cada experimento. En este caso, se utiliza la secuencia para el gen no relacionado aintegumenta (hormiga) de Arabidopsis thaliana .
2. Preparación de bacterias muertas por calor que expresan dsRNA
- Cultivar un cultivo a partir de una sola colonia bacteriana de la cepa HT115 (DE3) de E. coli que contenga el plásmido que expresa dsRNA en 50 ml de caldo de Luria (LB) que contenga 100 μg/ml de ampicilina y 12,5 μg/ml de tetraciclina, en un agitador de plataforma (180 rpm) a 37 °C durante 12 h.
- Diluir el cultivo bacteriano (1:1000) en 2x medios de triptona de levadura (2x YT) que contengan 100 μg/ml de ampicilina y 12,5 μg/ml de tetraciclina.
- Inducir la producción de dsRNA mediante la adición de 40 μM (concentración final) de isopropil β-D-1-tiogalactopiranósido (IPTG).
- Cuando las células alcanzan un O.D.600 = 0.4, aproximadamente después de 2 h de inducción a 37 °C con agitación a 180 rpm, preparar una suspensión concentrada de bacterias muertas por calor como lo describen Taracena et al 9. Granular las células por centrifugación (4000 x g, 4 °C, 10 min) y lavar las células en un volumen de tampón de fosfato de sodio (PBS).
- Girar de nuevo en las mismas condiciones, volver a suspender en PBS a 1/100 del volumen inicial, y colocar a 70 °C durante 1 h.
- Haga alícuotas de 400 μL de las bacterias muertas por el calor y guarde estas alícuotas a -20 ° C hasta su uso posterior (no almacene durante más de una semana). Esta suspensión de bacterias muertas por calor contiene el dsRNA específico para los experimentos de ARNi. Lleve a cabo este procedimiento tanto para las bacterias dsRNA del gen objetivo como para el control de dsRNA no relacionado que se utilizará en cada experimento.
3. Alimentar a los mosquitos con bacterias muertas por el calor que expresan dsRNA
- Descongelar una alícuota de dsRNA (suspensión de bacterias HT115 (DE3)) y mezclar con 1,6 ml de solución de azúcar al 12% que contiene 0,2% de metilparabeno.
- Remoje una pequeña bola de algodón en esta solución y coloque la bola de algodón empapada dentro de una jaula que contenga mosquitos de 5 días de edad. Asegúrese de que los mosquitos se alimenten de esta solución, recogiendo tanto el azúcar como las bacterias que contienen dsRNA simultáneamente.
- Cambie la bola de algodón empapada en solución de dsRNA-azúcar cada dos días durante 8 días consecutivos.
- Mantenga las jaulas de mosquitos en condiciones constantes, es decir, 27 ° C y 80% de humedad relativa con un fotoperíodo de 12 h: 12 h de luz: fotociclo oscuro, separado por un período de 30 minutos al amanecer y 30 minutos al atardecer.
4. Niveles de expresión génica objetivo del ensayo
- Anestesia en frío a los mosquitos colocando el recipiente sobre hielo durante un minuto o hasta que los mosquitos dejen de moverse. Una vez que los mosquitos estén anestesiados, colóquelos en una superficie fría para aislar a las hembras para su disección.
- Rocíe etanol al 70% a los mosquitos y colóquelos en una superficie de vidrio con PBS. Con un par de fórceps, asegure la cabeza del mosquito firme y tire del tórax muy lentamente, permitiendo que las glándulas salivales se liberen en el PBS.
- Mantenga las glándulas salivales en PBS helado hasta que 10 individuos hayan sido diseccionados. Pool Ten SG para la extracción de ARN utilizando el método de tiocianato de guanidinio-fenol-cloroformo. Suspenda el gránulo de ARN en 30 μL de agua libre de RNasa.
- Utilice 1 μL alícuota del ARN extraído del SG en el paso anterior, para leer la absorbancia a 260 y 280 nm y calcular la concentración de ARN de cada muestra multiplicando por el factor de dilución. Una relación 260/280 de ~2.0 indica ARN de buena calidad.
- Utilice 1 μg del ARN purificado para sintetizar ADN complementario (ADNc) utilizando un kit de transcripción inversa comercial.
- Haga una dilución 1:10 del ADNc para preparar una reacción rt-PCR de acuerdo con las recomendaciones del fabricante. Para cada muestra, prepare una reacción para el gen objetivo y, en paralelo, configure una reacción con el gen de limpieza (HK). Establezca cada reacción genética en un triplicado técnico para eliminar el impacto de la variación aleatoria del método.
NOTA: Aquí, el gen S7 ribosómico de An. gambiae (Banco de germoplasma: L20837.1) y la actina (VectorBase: AGAP000651) se han utilizado como genes HK. - Utilice todos los cebadores a una concentración final de 300 nM, siguiendo las indicaciones del fabricante SYBR-green. Amplificar con condiciones estándar de PCR: 95 °C durante 10 min, seguido de 40 ciclos de 15 s a 95 °C y 60 s a 60 °C.
NOTA: Para cuantificar la expresión génica, se utiliza el método delta-delta-Ct (ΔΔCt). Delta Ct (ΔCt) es la diferencia entre el Ct del gen objetivo y el Ct del gen de limpieza. ΔΔCt es la diferencia entre el ΔCt del grupo experimental y el ΔCt del grupo control35.
5. Evaluación fenotípica: alimentación sanguínea exitosa
- Para evaluar la capacidad de alimentación sanguínea, establezca grupos de 15 mosquitos hembra tratados con dsRNA objetivo y de control en jaulas pequeñas (12 cm de diámetro) y muédalos de hambre durante 4 h.
- Usando un baño de agua circulante a 37 ° C, los comederos de vidrio para mosquitos (24 mm de diámetro) y la membrana de parafilm, ofrecen sangre de oveja desfibrinada a los mosquitos.
NOTA: La sangre se puede adquirir de un proveedor comercial que la extrae asépticamente de animales sanos y donantes de origen estadounidense y desfibrina manualmente sin anticoagulantes ni aditivos. - Mediante la observación directa, cuente y registre el número de intentos de sondeo para adquirir con éxito una comida de sangre de las primeras cinco hembras para que se congestione completamente en cada grupo.
NOTA: Para evitar cambios metabólicos significativos en los mosquitos, que podrían interferir con los recursos energéticos que afectan el comportamiento de búsqueda de sangre, la inanición se mantuvo al mínimo (4 h). Como resultado, no todos los mosquitos buscarían ávidamente la comida de sangre y limitamos el recuento de las hembras congestionadas a cinco (un tercio del total de cada grupo), para reducir el efecto de variables de tiempo como la exposición al olor humano, el cambio de temperatura entre las cámaras y las superficies de alimentación, etc.
6. Evaluación fenotípica: morfología de las glándulas salivales y regulación a la baja de las proteínas relevantes
- Aislar el tejido fresco en 1x solución salina tamponada con fosfato (PBS) como se describe en el paso 4.2 y fijar en acetona helada durante 90 s. Enjuague varias veces en 1x PBS después de quitar la acetona. Incubar con anticuerpos primarios durante la noche a 4 °C con antisuero (ver Tabla de Materiales) diluido en 1x PBS.
NOTA: Ver Tabla de Materiales para la identificación de los anticuerpos primarios utilizados para las proteínas de la saliva (Proteína antiplaquetaria Anopheles , AAPP; Mucina 2, MUC2), factores de transcripción SG (Fork Head, fkh; Sabio, sabio; Proteína de unión a elementos de respuesta cíclica-AMP A, CrebA), y un marcador de vesículas secretoras (Rab11). Estos anticuerpos se utilizan como lecturas para la forma y función de SG. Sin embargo, cualquier anticuerpo adecuado para la inmunofluorescencia debe ser adecuado para este protocolo. - Lavar en 1x PBS varias veces. Agregue anticuerpos secundarios (fluorescentes) diluidos en 1x PBS e incube en la oscuridad a temperatura ambiente durante 2 h. Agregue cualquier contratinción [como 4′,6-diamidino-2-fenilindol (DAPI; ADN), aglutinina de germen de trigo (WGA; para quitina), faloidina (para F-actina) y/o rojo del Nilo (para lípidos)] 30 min antes del final de la incubación de 2 h.
- Lavar tres veces en 1x PBS. Luego, monte los tejidos en glicerol al 100% en un portaobjetos de microscopio estándar con un capa de 1 mm de grosor y guárdelo a -20 ° C hasta obtener imágenes con un microscopio confocal de fluorescencia.
NOTA: Para obtener datos cuantitativos, la configuración de imágenes debe mantenerse constante. Aquí, solo se incluyeron imágenes de proyección de máxima intensidad a través de todo el volumen 3D del tejido, y toda la cuantificación de la imagen se normalizó entre los tratamientos (dentro de un experimento) en función de la señal DAPI en restos de tejido no SG (cuerpo graso, cutícula o cabeza) también presentes en la diapositiva.
Resultados
Para comenzar, los datos de expresión de microarrays de VectorBase se utilizaron para escanear objetivos potenciales en las etapas de desarrollo 36,37 para determinar el estado de expresión de todos los genes relevantes para el estudio actual (Tabla 1). Como era de esperar, todos nuestros genes diana elegidos mostraron expresión en SG adultos. Los niveles de aapp y salvia fueron particularmente altos (Tabla 1). También fueron de destacar los altos niveles de expresión de f-Agdsx en mujeres adultas SGs9.
Se evaluaron segmentos específicos de cada gen para su uso como dsRNA utilizando la aplicación web E-RNAi para el diseño de reactivos de ARNi30. Las regiones de ~ 400 pb que contienen secuencias únicas para cada gen objetivo se clonaron (Figura 1A), se transformaron en las cepas bacterianas apropiadas y se utilizaron para preparar suspensiones de bacterias muertas por calor, que fueron inducidas a producir dsRNA. Los mosquitos adultos fueron alimentados durante 8 días con las bolas de algodón empapadas en sacarosa que contenían las suspensiones bacterianas de dsRNA para f-Agdsx, fkh u hormiga (el control negativo no relacionado).
Para el análisis de la alimentación por ARNi de mosquitos hembra, primero se determinó si la alimentación con f-Agdsx o fkh dsRNA indujo el silenciamiento génico. Se observó una reducción del 98,8% (±2,1) en los niveles de transcripción de fkh en el grupo alimentado con fkh-dsRNA (Figura 1B), lo que indica que el dsRNA redujo de manera muy efectiva la abundancia de transcripciones de fkh en SGs. Sorprendentemente, los niveles de fkh mRNA se redujeron en un 82,0% (±18,9) en los mosquitos tratados con dsRNA para f-Agdsx, que tuvieron un 89,86% (±4,48) de reducción de f-Agdsx , sugiriendo que fkh podría ser un objetivo de F-Dsx en la glándula salival. Concomitantemente con la reducción significativa en los niveles de expresión de fkh, los mosquitos fkh-knockdown exhibieron un aumento significativo en el número de intentos de sondeo necesarios para alimentarse con sangre. Estos mosquitos exhibieron, en promedio, cinco veces más intentos de alimentación que el grupo de control o f-Agdsx dsRNA alimentados con mosquitos para estar completamente llenos de sangre (Figura 1C). Esto llevó a preguntarse si los tratamientos de ARNi de derribo de fkh causaron cambios en la localización y / o distribución de reguladores transcripcionales clave (SG TF Sage y CrebA) (Figura 2), proteínas secretadas (AAPP y mucina) (Figura 3) y maquinaria secretora [Rojo del Nilo (lípidos) y Rab11 (vesículas secretoras)] (Figura 4). Es importante destacar que se observaron diferencias sustanciales en la intensidad de la tinción en diferentes regiones de lóbulos, lóbulos y SG individuales.
Como se predijo, los niveles de tinción de salvia y CrebA se redujeron notablemente en todos los lóbulos SG después del ARNi fkh (Figura 2B) en comparación con el ARNi de control de hormigas (Figura 2A). Las reducciones tanto en los valores de intensidad máxima más altos (líneas discontinuas rojas y etiquetas numéricas) como en los valores de intensidad máxima más bajos (líneas discontinuas azules y etiquetas numéricas) en los perfiles de escaneo de línea sugirieron reducciones en áreas de señal alta y baja dentro del tejido (Figuras 2A, B). Estos datos sugieren que An. gambiae fkh RNAi es eficaz y que fkh regula la producción y/o estabilidad de los SG TF Sage y CrebA en An. gambiae, análogo a su relación genética en Drosophila SGs 19,38,39.
Al considerar proteínas altamente abundantes que componen saliva, los niveles de proteína antiplaquetaria Anopheles (AAPP)40,41 se redujeron en los tres lóbulos SG después del ARNi fkh, en comparación con el tratamiento con ARNi control (Figura 3A, B; verde). Por otro lado, no se observaron cambios en los niveles de Mucina (Figura 3A,B; púrpura). Estos datos sugieren que Fkh contribuye de manera diferente a la expresión de diferentes genes de proteínas de saliva.
Finalmente, se observaron dos marcadores de secreción (Figuras 4A,B): Rab11 (vesículas asociadas a endosomas de reciclaje apical)42 y Rojo del Nilo (lípidos). Se observó una reducción de la fluorescencia de Rab11 en los lóbulos laterales distales (DL) después del tratamiento con arNi fkh (Figura 4A v vs. 4B v; verde). Sin embargo, también se produjo un aumento de la señal de Rab11 en los lóbulos medial (M) y lateral proximal (PL) (Figura 4A vii, ix vs. 4B vii, ix; verde). No se observaron diferencias perceptibles en la señal roja del Nilo (Figuras 4A, B; púrpura) después del ARNi fkh en comparación con el tratamiento con ARNi control. Estos datos sugieren que la reducción de fkh puede alterar alguna acción de la maquinaria secretora de una manera compleja que difiere entre los lóbulos SG.
| Conjunto de datos: | Goltsev | Neira Oviedo | Neira Oviedo | Pastelero | Pastelero | Pastelero | Pastelero | ||
| símbolo del gen | función | AGAP ID | embrión (25 hr.) | Larvas L3 | L3 SG | hembra adulta cuerpo (3 días) | macho adulto cuerpo (3 días) | hembra adulta SG (3 días) | macho adulto SG (3 días) |
| AAPP | proteína de saliva | AGAP009974 | 3.92 | 4.38 | 4.33 | 3.81 | 2.46 | 11.92 | 2.69 |
| CrebA | Factor txn | AGAP001464 | 6.28 | 5.22 | 5.92 | 2.99 | 2.96 | 3.27 | 3.13 |
| " | Factor txn | AGAP011038 | 4.50 | 4.46 | 5.23 | 2.96 | 2.86 | 3.05 | 2.88 |
| Dsx | Factor txn | AGAP004050 | 4.91 | 5.39 | 5.55 | 3.72 | 4.00 | 4.57 | 4.01 |
| fkh | Factor txn | AGAP001671 | 5.18 | 4.67 | 5.25 | 2.99 | 3.09 | 3.21 | 3.05 |
| MUC2 | proteína de saliva | AGAP012020 | 4.59 | 5.53 | 5.63 | 2.96 | 3.07 | 3.08 | 3.26 |
| Rab11 | tráfico vesicular | AGAP004559 | 10.21 | 7.47 | 8.60 | 4.90 | 3.79 | 3.38 | 2.96 |
| salvia | Factor txn | AGAP013335 | 5.32 | 5.96 | 8.89 | 3.40 | 3.33 | 7.37 | 7.23 |
Tabla 1: Perfiles de expresión de microarrays log2 medios para An. Gambiae genes de interés. Se muestran los nombres de genes, la categoría funcional, los identificadores de Vectorbase (AGAP) y los datos de expresión de microarrays log2 promedio recopilados de Vectorbase. Estos datos indican que nuestros genes de interés (implicados en la biología celular de las glándulas salivales (SG) y la secreción) se expresan y enriquecen en el estadio larvario 3 (L3) y en los SG adultos, en comparación con individuos enteros.
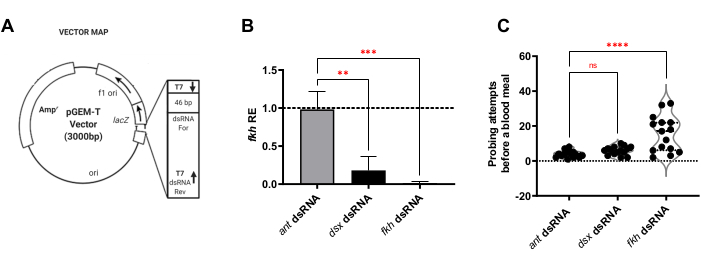
Figura 1: f-Agdsx y fkh knockdown en adultos An. gambiae reduce los niveles de fkh mRNA en los SG y afecta la capacidad femenina para alimentarse con sangre. (A) Imagen representativa del diseño del plásmido utilizado para la producción de dsRNA en esta metodología. La segunda secuencia promotora de T7 se agrega al plásmido incluyéndolo en el cebador de 3' utilizado para amplificar el inserto que se clonará en el plásmido pGEMT. El plásmido se transforma en la bacteria E. coli HT115 (DE3) y una solución de alimentación está hecha de una suspensión de bacterias inducidas que matan el calor en agua azucarada al 10%. (B) Los animales alimentados con una solución de alimentación de dsRNA para f-Agdsx o fkh, mostraron niveles significativamente más bajos de transcripciones de fkh (ANOVA unidireccional con comparaciones múltiples; n = 15). Sin embargo, solo el grupo alimentado con fkh dsRNA (C) mostró una diferencia significativa en el número de intentos de mordedura necesarios para adquirir una comida de sangre. Los mosquitos en este grupo necesitaron, en promedio, cinco veces el número de intentos de sondeo para obtener una comida de sangre exitosa que la que necesitaban los grupos de control o los grupos alimentados con dsx-dsRNA (ANOVA unidireccional con comparaciones múltiples; n = 15). Las barras de error indican el error estándar de la media (SEM). Cada experimento se llevó a cabo en tres réplicas biológicas separadas. Haga clic aquí para ver una versión más grande de esta figura.
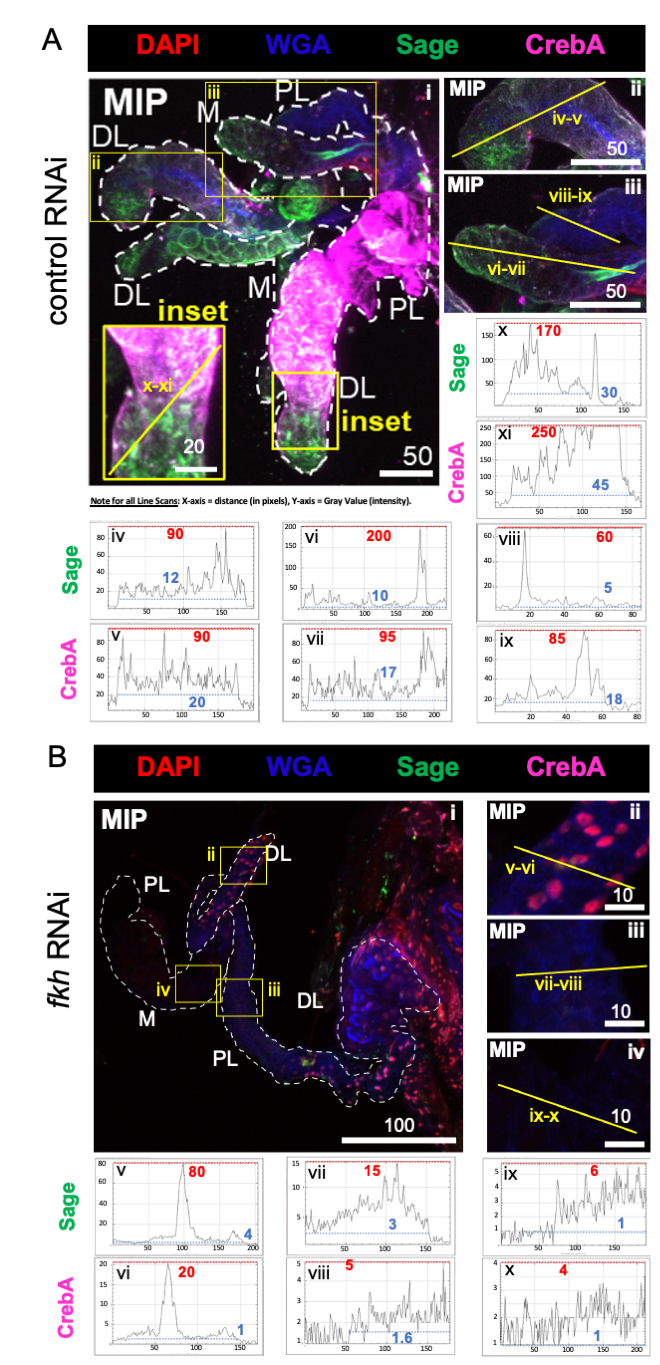
Figura 2: el derribo de fkh en las glándulas salivales de An. gambiae adulto reduce los niveles de factor de transcripción SG. Se muestran imágenes representativas de an. gambiae SG de an. gambiae hembra adulta del día 13 después de 8 días (días 5-13) de exposición oral a (A) no relacionado con el control de dsRNA (hormiga) o (B) dsRNA dirigido a la cabeza de la horquilla SG TF (fkh, AGAP001671) en sacarosa al 10% teñida con los colorantes DAPI (ADN; rojo), aglutinina de germen de trigo marcado (WGA, quitina / O-GlcNAcilación; azul), antisueros contra los SG TF Sage (verde) y CrebA (morado). Las longitudes de barra de escala que se muestran son micras. Los SG (i) están delineados con guiones blancos. Las líneas amarillas en las imágenes del lóbulo con zoom (de las regiones encerradas por cajas amarillas y etiquetadas como "recuadro") indican dónde se realizaron los escaneos de línea de intensidad de señal. Las intensidades de canal verde y púrpura correspondientes a los escaneos de línea para cada lóbulo ampliado se trazan (siempre de izquierda a derecha en el SG) en los gráficos debajo de las imágenes; Eje X = distancia (en píxeles) y eje Y = unidad gris (intensidad de píxeles). El rango dinámico de la intensidad de píxeles está delimitado por líneas punteadas rojas (máximas) y azules (mínimas) y los valores correspondientes se muestran en cada gráfico. MIP = proyección 3D de máxima intensidad a través de toda la profundidad SG. DL: lóbulo lateral distal; M: lóbulo medial; PL: lóbulo lateral proximal; SD: conducto salival. Haga clic aquí para ver una versión más grande de esta figura.
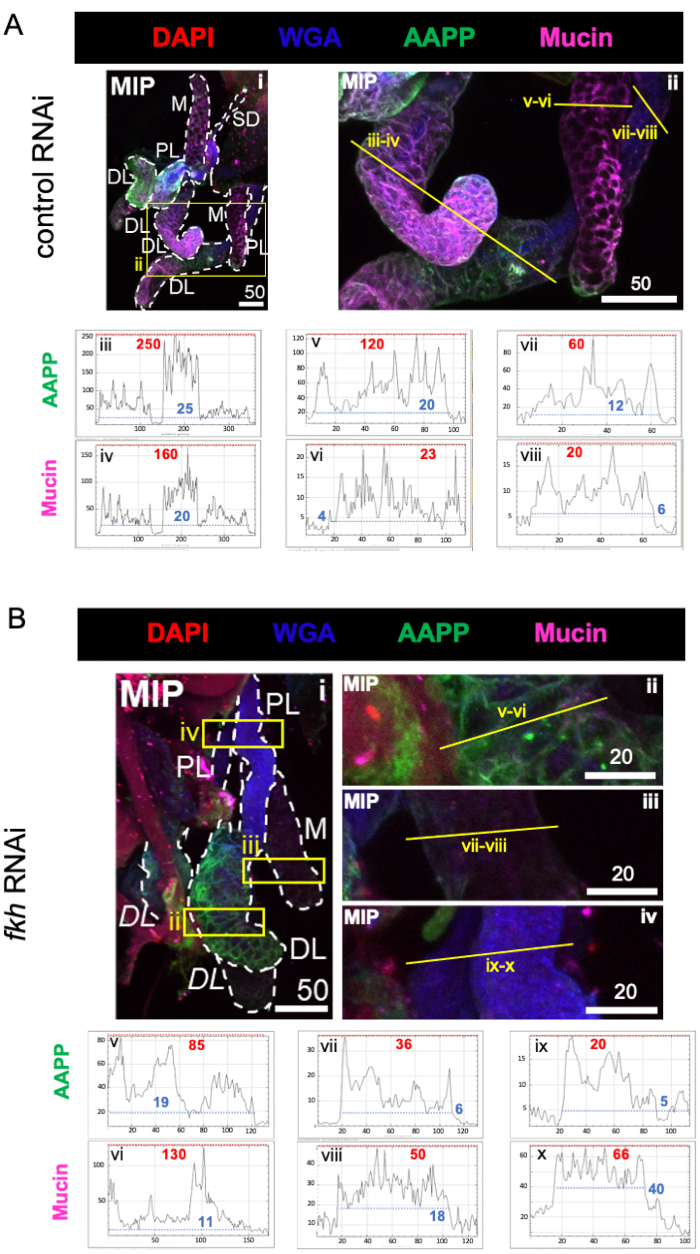
Figura 3: el derribo de fkh en las glándulas salivales adultas de An. gambiae reduce los niveles de proteína secretada por SG. Se muestran imágenes representativas de an. gambiae SG de an. gambiae hembra adulta del día 13 después de 8 días (días 5-13) de exposición oral a (A) control de dsRNA no relacionado (hormiga) o (B) dsRNA dirigido a la cabeza de horquilla SG TF (fkh, AGAP001671) en sacarosa al 10% teñida con los colorantes DAPI (ADN; rojo), aglutinina de germen de trigo marcado (WGA, quitina / O-GlcNAcylation; azul), y las proteínas salivales AAPP (verde) y Mucina (MUC2, púrpura). Las longitudes de barra de escala que se muestran son micras. Los SG (i) están delineados con guiones blancos. Las líneas amarillas en las imágenes de lóbulos con zoom (de las regiones encerradas por cuadros amarillos) indican dónde se realizaron los escaneos de línea de intensidad de señal. Las intensidades de canal verde y púrpura correspondientes a escaneos de línea para cada lóbulo se trazan (siempre de izquierda a derecha en el SG) en los gráficos debajo de las imágenes; Eje X = distancia (en píxeles) y eje Y = unidad gris (intensidad de píxeles). El rango dinámico de la intensidad de píxeles está delimitado por líneas discontinuas rojas (máximas) y azules (mínimas) y los valores correspondientes se muestran en cada gráfico. MIP = proyección 3D de máxima intensidad a través de toda la profundidad SG. DL: lóbulo lateral distal; M: lóbulo medial; PL: lóbulo lateral proximal; SD: conducto salival. Las etiquetas "DL" en cursiva (Bi) indican dos regiones visibles del mismo lóbulo DL. Haga clic aquí para ver una versión más grande de esta figura.
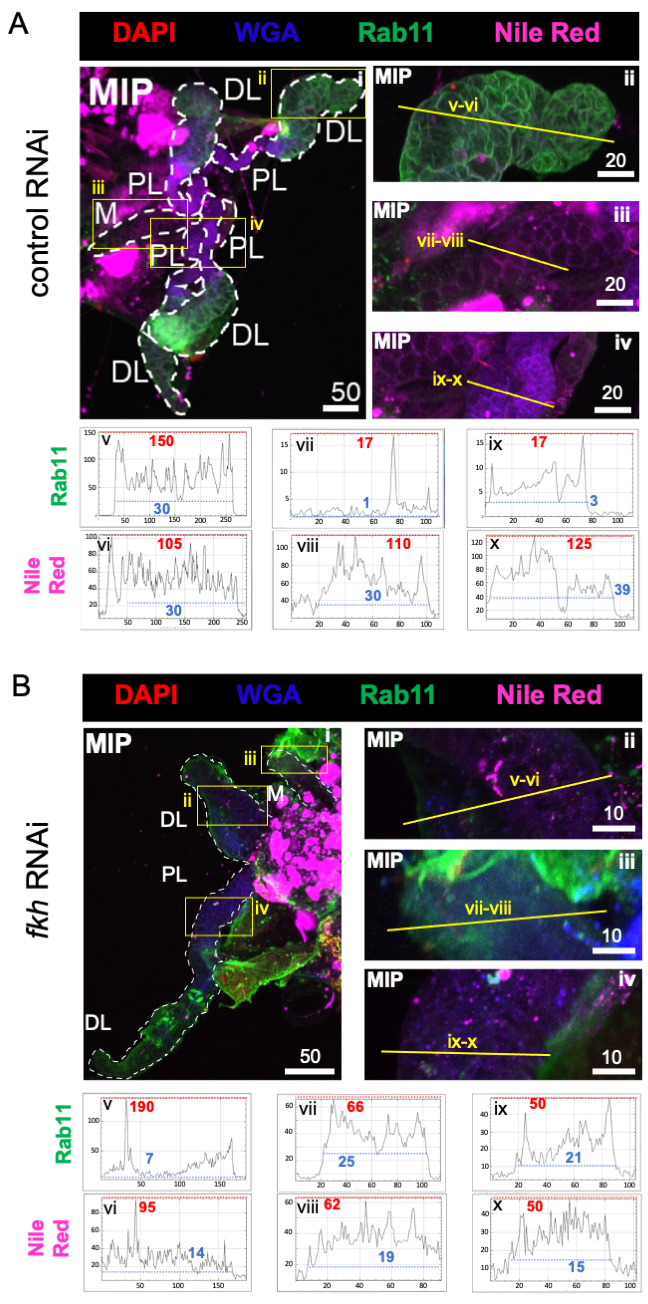
Figura 4: el derribo de fkh en glándulas salivales adultas de An. gambiae reduce los marcadores de secreción de SG. Se muestran imágenes representativas de an. gambiae SGs de an. gambiae hembra adulta del día 13 después de 8 días (días 5-13) de exposición oral a (A) control de dsRNA no relacionado (hormiga) o (B) dsRNA dirigido a la cabeza de horquilla SG TF (fkh, AGAP001671) en sacarosa al 10% teñida con los colorantes DAPI (ADN; rojo), aglutinina de germen de trigo marcado (WGA, quitina / O-GlcNAcilación; azul), Rojo del Nilo (lípidos; púrpura), y antisueros contra el marcador de vesícula endosómica de reciclaje Rab11 (verde). Las longitudes de barra de escala que se muestran son micras. Los SG (i) están delineados con guiones blancos. Las líneas amarillas en las imágenes de lóbulos con zoom (de las regiones encerradas por cuadros amarillos) indican dónde se realizaron los escaneos de línea de intensidad de señal. Las intensidades de canal verde y púrpura correspondientes a los escaneos de línea para cada lóbulo se trazan (siempre de izquierda a derecha en el SG) en los gráficos debajo de las imágenes; Eje X = distancia (en píxeles) y eje Y = unidad gris (intensidad de píxeles). El rango dinámico de la intensidad de píxeles está delimitado por líneas discontinuas rojas (máximas) y azules (mínimas) y los valores correspondientes se muestran en cada gráfico. MIP = proyección 3D de máxima intensidad a través de toda la profundidad SG. DL: lóbulo lateral distal; M: lóbulo medial; PL: lóbulo lateral proximal; SD: conducto salival. Haga clic aquí para ver una versión más grande de esta figura.
Expediente complementario 1. Haga clic aquí para descargar este archivo.
Discusión
La capacidad de administrar dsRNA de manera efectiva a los mosquitos An. gambiae mediante alimentación oral tiene amplias implicaciones para los estudios de biología de vectores tanto en el laboratorio como en el campo. La microinyección ha sido aceptada durante mucho tiempo como el modo preferido de administración de productos químicos, anticuerpos, ARNi y estrategias de modificación genética en mosquitos43,44. La consecuencia de la manipulación física sustancial, el daño celular y el estrés se pueden evitar mediante el uso de la administración oral, que también podría ser potencialmente adecuada para aplicaciones a gran escala o de campo. Trabajos anteriores han sugerido que el ARNi actúa de manera ubicua dentro de un mosquito adulto individual29, lo que permite efectos en todos los tejidos, incluidas las glándulas salivales. Al alimentar a los mosquitos con un gran número de E. coli que expresan dsRNA que se digieren de forma asíncrona durante un largo período de tiempo, se puede lograr una exposición consistente y uniforme al ARNi en todos los individuos en una jaula. Este método permite alimentar a un gran número de mosquitos y analizar la variabilidad potencial de los fenotipos resultantes dependiendo del gen objetivo. Sin embargo, una consideración importante es la posibilidad de una distribución heterogénea de las bacterias, y por lo tanto del dsRNA, en la fibra de algodón. Los 400 μL de bacterias utilizados diariamente para la alimentación con azúcar de los mosquitos contendrían aproximadamente ≤4,6 μg de dsRNA, como se describió y calculó previamente9, pero la cantidad de dsRNA ingerida por cada mosquito no se determinó individualmente. Si la construcción de construcciones de dsRNA se convierte en rutina, este protocolo de tratamiento simple permite una rápida asimilación de esta técnica por parte de cualquier investigador de mosquitos. A priori, el gasto de tiempo durante el tratamiento (30 min por día) es trivial en comparación con el tiempo necesario para aprender y aplicar la microinyección a tamaños de muestra similares.
La alimentación con dsRNA se utiliza habitualmente para estudios de genética inversa en el organismo modelo Caenorhabditis elegans45. Este alto nivel de uso subraya el valor del enfoque de administración oral. La construcción de una biblioteca de an. gambiae para todo el genoma en E. coli transformada, similar a la que existe en C. elegans46,47, permitiría un rápido cribado genético inverso en mosquitos a una escala mayor. Sin embargo, es importante tener en cuenta que la eficiencia del método depende en gran medida de los niveles endógenos de transcripción y si la expresión no se limita al tejido diana sino que se expresa de manera más amplia 4,8,44. Además, hay evidencia de que algunos insecticidas podrían inducir la evitación conductual de los mosquitos48, y la alimentación con bacterias que potencialmente inducen efectos adversos en ellos podría desencadenar patrones similares de evitación. En el entorno controlado del laboratorio, donde los mosquitos no tenían una fuente de alimento alternativa, no tenían la opción de evitar el agua azucarada con E. coli y la necesidad de una fuente nutritiva probablemente anularía el instinto de evitar las bacterias. Sin embargo, esto debe considerarse si la estrategia estuviera destinada a ser utilizada en entornos menos controlados.
Puede ser posible dirigirse a múltiples genes simultáneamente (utilizando una construcción, múltiples construcciones o una mezcla de aislados bacterianos transformados), pero se necesitan más estudios para evaluar la efectividad. Otra consideración importante para este punto es la evaluación de posibles efectos fuera del objetivo o sinérgicos cuando se utilizan objetivos únicos o múltiples. El establecimiento de genes y grupos de control apropiados es una parte importante del diseño experimental. Además, es tentador especular que este enfoque podría usarse para atacar otros patógenos o virus49. El trabajo previo hacia la inducción de ARNi en mosquitos se realizó en condiciones en las que el reactivo se inyectó directamente, por lo que E. coli no estaba presente. La E. coli puede proporcionar un compartimento protector que permite la liberación más lenta de dsRNA con el tiempo, asegurando que la exposición sea más o menos continua durante un período mucho más largo29.
Finalmente, estos resultados muestran que los efectos de esta técnica son ajustables ajustando el marco de tiempo (duración y día de inicio) de la exposición y la cantidad de E. coli utilizada. Esta característica nos permitió estudiar las funciones de los genes esenciales (dsx y fkh) mediante la identificación de condiciones óptimas de derribo por ensayo y error. Esto aumenta en gran medida la probabilidad de que los genes objetivo de interés puedan investigarse utilizando esta técnica.
En resumen, se encontró que la administración oral de ARNi a mosquitos adultos puede ser simple, versátil y un enfoque poderoso para estudiar la función de los genes de los mosquitos y para la creación de herramientas novedosas y maleables para el control vectorial de enfermedades transmitidas por mosquitos.
Divulgaciones
Los autores informan que no tienen conflictos de intereses que revelar.
Agradecimientos
Los autores desean agradecer al personal y a los científicos de la Subdivisión de Entomología y la División de Enfermedades Parasitarias y Malaria de los CDC, y a Brian Trigg y Michelle Chiu por su asistencia con la preparación de bacterias en JHU y / o discusiones útiles de este trabajo. Agradecemos al insectario y gerente de JHMRI, Chris Kizito, por el acceso y la cría de mosquitos An. gambiae . Agradecemos a Wei Huang (JHSPH) por su ayuda en la obtención de plásmidos PJet GFP y pPB47 GFP para su uso en este estudio. El financiamiento para este trabajo fue proporcionado por: NIH R21AI153588 (a DJA), una beca postdoctoral del Instituto de Investigación de la Malaria de Johns Hopkins (a MW); y por una subvención de la Fundación Good Ventures y el Proyecto de Filantropía Abierta a la Fundación CDC titulada Apoyo a la criopreservación y supresión del desarrollo femenino en mosquitos para ayudar a la investigación de la malaria, Open Philanthropy Project, 2017. Agradecemos profundamente la asistencia del personal de JHU Microscope Facility y el apoyo de subvención de los NIH aplicable para el microscopio utilizado (NIH Grant #: S10OD016374). Los hallazgos y conclusiones de este manuscrito son los de los autores y no representan necesariamente los puntos de vista de los CDC. El uso de nombres comerciales es solo para identificación y no implica la aprobación de los Centros para el Control y la Prevención de Enfermedades, el Servicio de Salud Pública o el Departamento de Salud y Servicios Humanos de los Estados Unidos.
Materiales
| Name | Company | Catalog Number | Comments |
| 1 Kb Plus DNA Ladder | Thermo Fisher Scientific | 10787018 | |
| 2x Yeast Extract Tryptone (2xYT) Medium | BD Difco | DF0440-17 | |
| AAPP | n/a | n/a | Antisera. 1:50 dilution (rabbit); gift from Fabrizio Lombardo |
| AccuStart II PCR Supermix | Quantabio | 95137-100 | |
| Agarose | Millipore Sigma | A9539 | |
| Ampicillin | Millipore Sigma | A5354 | |
| Anopheles gambiae G3 | BioDefense and Emerging Infections (BEI) Malaria Research and Reference Reagent Resource Center (MR4) | MRA-112 | |
| BugDorm | BioQuip | 1452 | |
| Centrifuge 5810R | Eppendorf | P022628181 | |
| CrebA | DSHB | CrebA Rbt-PC | Antisera. 1:50 dilution (rabbit); generated by the Andrew Lab |
| Damiens diet | BioServ | ||
| DAPI | Life Technologies | n/a | 4′,6-diamidino-2-phenylindole; 1:200 dilution. |
| Defibrinated sheep blood | HemoStat | DSB050 | |
| Escherichia coli HT115 (DE3) | |||
| Ethidium bromide | Millipore Sigma | E7637 | |
| High-Capacity cDNA Reverse Transcription Kit | Thermo Fisher Scientific | 4368814 | |
| Isopropyl β-D-1-thiogalactopyranoside | Millipore Sigma | I5502 | |
| JM109 Competent cells | Promega | L2005 | |
| Luria Broth Media | Thermo Fisher Scientific | 10855001 | |
| Mucin 2 | Proteintech | Muc2; 27 675-1-AP | Antisera. 1:100 dilution (mouse). |
| Nanodrop 2000 | Thermo Fisher Scientific | ||
| Nile Red | Sigma | n/a | Lipid dye; 1:50 dilution. |
| Owl EasyCast B2 Mini Gel Horizontal Electrophoresis | Thermo Fisher Scientific | Model B2 | |
| pGEMT easy | Promega | A3600 | |
| Power SYBR-green PCR master MIX | Applied Biosystems | 4367659 | |
| PureLink PCR purification kit | Thermo Fisher Scientific | K31001 | |
| QuantaStudio 6 | Applied Biosystems | ||
| QuantStudio6 Real Time PCR System | Applied Biosystems | ||
| Rab11 | n/a | n/a | Antisera. 1:100 dilution (rabbit); generated by the Andrew Lab |
| Rh-WGA | Vector Labs | n/a | Rhodamine-conjugated wheat germ agglutinin (chitin, O-GlcNAcylation dye); 1:40 dilution |
| Sage | n/a | n/a | Antisera. 1:50 dilution (rat); generated by the Andrew Lab |
| T4 DNA ligase | Promega | M1801 | |
| Tetracycline | Millipore Sigma | 87128 | |
| Trizol | Thermo Fisher Scientific | 15596018 | |
| Zeiss LSM700 fluorescence confocal microscope | Zeiss | ||
| ANTIBODIES | |||
| Chicken anti-Rat IgG (H+L), Alexa Fluor 647 | Thermo Fisher Scientific | A21472 | |
| Goat anti-Mouse IgG (H+L), Alexa Fluor 647 | Thermo Fisher Scientific | A28181 | |
| IgG (H+L) Goat anti-Rabbit, Alexa Fluor 488 | Thermo Fisher Scientific | A27034 | |
| Rabbit anti-Goat IgG (H+L), Alexa Fluor 488 | Thermo Fisher Scientific | A27012 | |
| PRIMERS | |||
| ACT-2f: TACAACTCGATCATGAAGTGCGA | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| ACT-3r: CCCGGGTACATGGTGGTACCGC CGGA | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| FKH_RNAi_F: GCCGACTTATGCTTAGCCCA | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| FKH_RNAi_R: TAGCCGTCAATTCCTCCTGC | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| newDSX-f: AGAGGGCGGGGAAATTCTAGT | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| newDSX-r: GGGCTTGTGGCAGTACGAATA | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| S7qf1: AGAACCAGCAGACCACCATC | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| S7qr1: GCTGCAAACTTCGGCTATTC | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
Referencias
- Hoa, N. T., Keene, K. M., Olson, K. E., Zheng, L. Characterization of RNA interference in an Anopheles gambiae cell line. Insect Biochemistry and Molecular Biology. 33, 949-957 (2003).
- Caplen, N., Zheng, Z., Falgout, B., Morgan, R. Inhibition of viral gene expression and replication in mosquito cells by dsRNA-triggered RNA interference | Elsevier enhanced reader. Molecular Therapy. 6, 243-251 (2002).
- Brown, A. E., Catteruccia, F. Toward silencing the burden of malaria: progress and prospects for RNAi-based approaches. BioTechniques. , 38-44 (2006).
- Airs, P. M., Bartholomay, L. C. RNA interference for mosquito and mosquito-borne disease control. Insects. 8, (2017).
- Blandin, S., et al. Reverse genetics in the mosquito Anopheles gambiae: targeted disruption of the Defensin gene. EMBO Reports. 3 (9), 852-856 (2002).
- Garver, L., Dimopoulos, G. Protocol for RNAi assays in adult mosquitoes (A. gambiae). Journal of Visualized Experiments: JoVE. (5), e230 (2007).
- Whyard, S., et al. Silencing the buzz: a new approach to population suppression of mosquitoes by feeding larvae double-stranded RNAs. Parasites & Vectors. 8, 96 (2015).
- Wiltshire, R. M., Duman-Scheel, M. Advances in oral RNAi for disease vector mosquito research and control. Current Opinion in Insect Science. 40, 18-23 (2020).
- Taracena, M. L., Hunt, C. M., Benedict, M. Q., Pennington, P. M., Dotson, E. M. Downregulation of female doublesex expression by oral-mediated RNA interference reduces number and fitness of Anopheles gambiae adult females. Parasites & Vectors. 12, 170 (2019).
- Grassi, B. Studi di uno zoologo sulla malaria. Real Accademia dei Lincei. 3, 229 (1901).
- Smith, R. C., Jacobs-lorena, M. Plasmodium - Mosquito interactions: A tale of roadblocks and detours. Advances in Insect Physiology. 39, (2010).
- Das, S., et al. Transcriptomic and functional analysis of the Anopheles gambiae salivary gland in relation to blood feeding. BMC Genomics. 11, 1-14 (2010).
- Francischetti, I. M. B., Valenzuela, J. G., Pham, V. M., Garfield, M. K., Ribeiro, J. M. C. Toward a catalog for the transcripts and proteins (sialome) from the salivary gland of the malaria vector Anopheles gambiae. Journal of Experimental Biology. 205, 2429-2451 (2002).
- Henderson, K. D., Isaac, D. D., Andrew, D. J. Cell fate specification in thedrosophila salivary gland: The integration of homeotic gene function with the DPP signaling cascade. Developmental Biology. 205, 10-21 (1999).
- Mach, V., Ohno, K., Kokubo, H., Suzuki, Y. The Drosophila fork head factor directly controls larval salivary gland-specific expression of the glue protein gene Sgs3. Nucleic Acids Research. 24 (12), 2387-2394 (1996).
- Weiserova, M., et al. Mini-Mu transposition of bacterial genes on the transmissible plasmid. Folia Microbiologica. 32 (5), 368-375 (1987).
- Abrams, E. W., Mihoulides, W. K., Andrew, D. J. Fork head and Sage maintain a uniform and patent salivary gland lumen through regulation of two downstream target genes, PH4αSG1 and PH4αSG2. Development. 133, 3517-3527 (2006).
- Myat, M. M., Isaac, P. P., Andrew, D. J. Early genes required for salivary gland fate determination and morphogenesis in Drosophila melanogaster. Advances in Dental Research. 14, 89-98 (2000).
- Fox, R. M., Vaishnavi, A., Maruyama, R., Andrew, D. J. Organ-specific gene expression: the bHLH protein Sage provides tissue specificity to Drosophila FoxA. Development of Cell Biology. 140, 2160-2171 (2013).
- Maruyama, R., Grevengoed, E., Stempniewicz, P., Andrew, D. J. Genome-wide analysis reveals a major role in cell fate maintenance and an unexpected role in endoreduplication for the Drosophila FoxA gene fork head. PLOS ONE. 6, 20901 (2011).
- Johnson, D. M., et al. CrebA increases secretory capacity through direct transcriptional regulation of the secretory machinery, a subset of secretory cargo, and other key regulators. Traffic. 21, 560-577 (2020).
- Fox, R. M., Hanlon, C. D., Andrew, D. J. The CrebA/Creb3-like transcription factors are major and direct regulators of secretory capacity. Journal of Cell Biology. 191, 479-492 (2010).
- Abrams, E. W., Andrew, D. J. CrebA regulates secretory activity in the Drosophila salivary gland and epidermis. Development. 132, 2743-2758 (2005).
- Wells, M. B., Andrew, D. J. Anopheles salivary gland architecture shapes plasmodium sporozoite availability for transmission. mBio. 10 (4), 01238 (2019).
- Pei-Wen, L., Xiao-Cong, L., Jin-Bao, G., Yan, L., Xiao-Guang, C. Molecular cloning, characterization and expression analysis of sex determiantion gene doublesex from Anopheles gambiae (Diptera: Culicidae). Acta Entomologica Sinica. 58 (2), 122-131 (2015).
- Scali, C., Catteruccia, F., Li, Q., Crisanti, A. Identification of sex-specific transcripts of the Anopheles gambiae doublesex gene. The Journal of Experimental Biology. 208, 3701-3709 (2005).
- Price, D. C., Egizi, A., Fonseca, D. M. Characterization of the doublesex gene within the Culex pipiens complex suggests regulatory plasticity at the base of the mosquito sex determination cascade. BMC Evolutionary Biology. 15, 1-13 (2015).
- Mysore, K., et al. siRNA-mediated silencing of doublesex during female development of the dengue vector mosquito Aedes aegypti. PLoS Neglected Tropical Diseases. 9, 1-21 (2015).
- Boisson, B., et al. Gene silencing in mosquito salivary glands by RNAi. FEBS Letters. 580, 1988-1992 (2006).
- Horn, T., Boutros, M. E-RNAi: a web application for the multi-species design of RNAi reagents-2010 update. Nucleic Acids Research. 38, 332-339 (2010).
- Taracena, M. L., et al. Genetically modifying the insect gut microbiota to control chagas disease vectors through systemic RNAi. PLoS Neglected Tropical Diseases. 9, (2015).
- Sambrook, J., Fritsch, E. F., Maniatis, T. . Molecular Cloning: A Laboratory Manual. , (1989).
- Ullmann, A., Jacob, F., Monod, J. Characterization by in vitro complementation of a peptide corresponding to an operator-proximal segment of the β-galactosidase structural gene of Escherichia coli. Journal of Molecular Biology. 24, 339-343 (1967).
- Timmons, L. Bacteria-mediated RNAi-General outline. Carnegie Institution of Washington. , (2000).
- Pfaffl, M. W. Relative quantification. Real-time PCR. , 63-82 (2004).
- Neira-Oviedo, M., et al. The RNA-Seq approach to studying the expression of mosquito mitochondrial genes. Insect Molecular Biology. 20, 141-152 (2011).
- Baker, D. A., et al. A comprehensive gene expression atlas of sex- and tissue-specificity in the malaria vector, Anopheles gambiae. BMC Genomics. 12, (2011).
- Loganathan, R., Hoon, J., Wells, M. B., Andrew, D. J. Secrets of secretion - How studies of the Drosophila salivary gland have informed our understanding of the cellular networks underlying secretory organ form and function. Cellular Networks in Development. , 143 (2021).
- Chung, S., Hanlon, C. D., Andrew, D. J. Building and specializing epithelial tubular organs: The Drosophila salivary gland as a model system for revealing how epithelial organs are specified, form and specialize. Wiley Interdisciplinary Reviews: Developmental Biology. 3, 281-300 (2014).
- Yoshida, S., et al. Inhibition of collagen-induced platelet aggregation by anopheline antiplatelet protein, a saliva protein from a malaria vector mosquito. Blood. 111, 2007-2014 (2008).
- Wells, M. B., Villamor, J., Andrew, D. J. Salivary gland maturation and duct formation in the African malaria mosquito Anopheles gambiae. Scientific Reports. 7 (1), 601 (2017).
- Takahashi, S., et al. Rab11 regulates exocytosis of recycling vesicles at the plasma membrane. Journal of Cell Science. 125, 4049-4057 (2012).
- Catteruccia, F., Levashina, E. A. RNAi in the malaria vector, Anopheles gambiae. Methods in Molecular Biology. 555, 63-75 (2009).
- Balakrishna Pillai, A., et al. RNA interference in mosquito: understanding immune responses, double-stranded RNA delivery systems and potential applications in vector control. Insect Molecular Biology. 26, 127-139 (2017).
- Fire, A., et al. Potent and specific genetic interference by double-stranded RNA in caenorhabditis elegans. Nature. 391, 806-811 (1998).
- Kamath, R. S., Ahringer, J. Genome-wide RNAi screening in Caenorhabditis elegans. Methods. 30, 313-321 (2003).
- Kamath, R. S., et al. Systematic functional analysis of the Caenorhabditis elegans genome using RNAi. Nature. 421, 231-237 (2003).
- Carrasco, D., et al. Behavioural adaptations of mosquito vectors to insecticide control. Current Opinion in Insect Science. 34, 48-54 (2019).
- Magalhaes, T., et al. Induction of RNA interference to block Zika virus replication and transmission in the mosquito Aedes aegypti. Insect Biochemistry and Molecular Biology. , 111 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados