A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
סימולציה מבוססת מבנה ודגימה של תנועות חלבון פקטור שעתוק לאורך דנ"א מצעד בקנה מידה אטומי לדיפוזיה גסה-גרגרית
* These authors contributed equally
In This Article
Summary
מטרת פרוטוקול זה היא לחשוף דינמיקה מבנית של דיפוזיה חד-ממדית של חלבון לאורך דנ"א, תוך שימוש בחלבון מקדם שעתוק צמחי WRKY כמערכת מופתית. לשם כך יושמו הן סימולציות של דינמיקה מולקולרית אטומיסטית והן של דינמיקה מולקולרית גסה, יחד עם דגימות חישוביות נרחבות.
Abstract
החלקה חד-ממדית (1-D) של חלבון גורם שעתוק (TF) לאורך הדנ"א חיונית להפצה מקלה של ה-TF כדי לאתר את אתר הדנ"א של המטרה לצורך ויסות גנטי. זיהוי רזולוציה של זוג בסיסים (bp) של ה-TF מחליק או דורך על הדנ"א הוא עדיין מאתגר מבחינה ניסיונית. לאחרונה ביצענו סימולציות של דינמיקה מולקולרית של כל האטום (MD) הלוכדות דריכה ספונטנית של 1-bp של חלבון TF קטן בתחום WRKY לאורך הדנ"א. בהתבסס על נתיב הדריכה WRKY של 10 מיקרו-μs שהתקבל מהדמיות כאלה, הפרוטוקול כאן מראה כיצד לבצע דגימות קונפורמיות נרחבות יותר של מערכות TF-DNA, על ידי בניית מודל מצב מרקוב (MSM) עבור דריכה של חלבון 1-bp, עם מספרים שונים של מצבי מיקרו ומאקרו שנבדקו לבניית MSM. על מנת לבחון חיפוש דיפוזיה תהליכי 1-D של חלבון ה-TF יחד עם דנ"א עם בסיס מבני, הפרוטוקול מראה עוד כיצד לבצע סימולציות MD גסות-גרגרים (CG) כדי לדגום דינמיקה ארוכת טווח של המערכת. מודלים וסימולציות כאלה של CG שימושיים במיוחד כדי לחשוף את ההשפעות האלקטרוסטטיות של חלבון-דנ"א על תנועות הדיפוזיה התהליכיות של חלבון ה-TF מעל עשרות מיקרו-שניות, בהשוואה לתת-מיקרו-שניות לתנועות דריכה של חלבוני מיקרו-שניות שנחשפו מהדמיות הכל-אטומיות.
Introduction
גורמי שעתוק (TF) מחפשים את דנ"א המטרה כדי לקשור ולווסת שעתוק גנים ופעילויות קשורות1. מלבד הדיפוזיה התלת-ממדית (התלת-ממדית), הוצע כי הדיפוזיה הקלה של TF חיונית לחיפוש דנ"א של מטרה, שבו החלבונים יכולים גם להחליק או לקפוץ לאורך דנ"א חד-ממדי (1D), או לקפוץ עם העברה בין-סגמנטלית על הדנ"א 2,3,4,4,5,6,7.
במחקר שנערך לאחרונה, ערכנו עשרות סימולציות של מיקרו-שניות (μs) של דינמיקה מולקולרית של שיווי משקל אטומי (MD) על TF צמחי - חלבון תחום WRKY על ה-DNA8. דריכה שלמה של 1 bp של WRKY על דנ"א פולי-A בתוך מיקרו-שניות נלכדה. נצפו התנועות של החלבון לאורך חריץ הדנ"א וקשרי המימן (HBs) דינמיקת שבירת-רפורמה. בעוד שמסלול כזה מייצג נתיב אחד שנדגם, עדיין חסר נוף כולל של דריכה חלבונית. כאן אנו מראים כיצד להרחיב דגימות חישוביות סביב נתיב דריכת החלבון שנלכד בתחילה עם מודל מצב מרקוב (MSM) שנבנה, אשר יושמו באופן נרחב להדמיית מגוון מערכות ביומולקולריות הכוללות שינויים קונפורמיים משמעותיים והפרדת קנה מידה של זמן 9,10,11,12,13,13,14,15,16, 17,18,19. המטרה היא לחשוף את ההרכב הקונפורמי והמצבים המטא-יציבים של דיפוזיית חלבון ה-TF לאורך הדנ"א לצעד מחזורי אחד.
בעוד שסימולציית MD לעיל חושפת רזולוציה אטומית של תנועות החלבון עבור 1 bp על הדנ"א, הדינמיקה המבנית של דיפוזיה תהליכית ארוכת שנים של TF לאורך DNA באותה רזולוציה גבוהה כמעט ואינה נגישה. עם זאת, ביצוע סימולציות MD גסות-גרגרים (CG) ברמת השאריות הוא נגיש מבחינה טכנית. ניתן להרחיב למעשה את סולם הזמן של סימולציית CG לעשרות או מאות פעמים יותר מהסימולציות האטומיות 20,21,22,23,24,25,26,27,28,29. כאן, אנו מראים את סימולציות ה- CG שנערכו על ידי הטמעת תוכנת CafeMol שפותחה על ידי מעבדת טקדה30.
בפרוטוקול הנוכחי, אנו מציגים תחילה את ההדמיות האטומיות של החלבון בתחום WRKY לאורך דנ"א פולי-A ואת בניית ה-MSM, המתמקדות בדגימת תנועות הדריכה של החלבון במשך 1 bp בלבד לאורך הדנ"א. לאחר מכן אנו מציגים את המידול וההדמיות של CG של אותה מערכת חלבון-דנ"א, אשר מרחיבים את הדגימה החישובית לדיפוזיה התהליכית של חלבונים על פני עשרות bps לאורך הדנ"א.
כאן, אנו משתמשים בתוכנת GROMACS 31,32,33 כדי לבצע סימולציות MD וב-MSMbuilder34 כדי לבנות את ה-MSM עבור תמונות קונפורמיות שנדגמו, כמו גם כדי להשתמש ב-VMD35 כדי להמחיש את הביו-מולקולות. הפרוטוקול דורש שהמשתמש יוכל להתקין וליישם את התוכנה לעיל. ההתקנה והיישום של תוכנת CafeMol30 נחוצה לאחר מכן לביצוע סימולציות CG MD. ניתוחים נוספים של המסלולים וההדמיה מתבצעים גם ב- VMD.
Protocol
1. בניית מודל מצב מרקוב (MSM) מהדמיות MD אטומיות
- מסלול דריכה ספונטני של חלבונים ואיסוף מבנים ראשוניים
- השתמש במסלול MD של 10-μs שהושג בעבר ב-10-μs all-atomMD 8 כדי לחלץ 10000 פריימים באופן שווה מנתיב דריכה "קדימה" של 1 bp (כלומר, מסגרת אחת עבור כל ננו-שניה). המספר הכולל של המסגרות צריך להיות גדול מספיק כדי לכלול את כל הקונפורמציות הייצוגיות.
- הכן את נתיב המעבר עם 10000 מסגרות ב- VMD על-ידי לחיצה על קובץ > שמור קואורדינטות, הקלד חלבון או גרעין בתיבה אטומים נבחרים ובחר מסגרות בתיבה מסגרות, לחץ על שמור כדי לקבל את המסגרות הדרושות.
הערה: מסלול סימולציה של 10 μs all-atom MD שהושג בעבר (נקרא כאן "מסלול דריכה קדימה" עבור WRKY דריכה למרחק של 1-bp על דנ"א הומוגני של פולי-A8 של 34 bp שימש כנתיב הראשוני לשיגור דגימות קונפורמיות נוספות. עם זאת, שים לב שברוב הפרקטיקות נבנה נתיב ראשוני, על ידי ביצוע סימולציות MD מנווטות או ממוקדות, או יישום שיטות כלליות ליצירת נתיב וכו'.36,37,38,38,39. - יישור הציר הארוך של דנ"א הייחוס (מהמבנה הגבישי) לציר ה-x, והגדר את מרכז המסה הראשוני (COM) של הדנ"א המלא של 34 bp במקור מרחב הקואורדינטות לנוחות של ניתוח נתונים נוסף. לשם כך, לחץ על הרחבות > Tk Console ב- VMD והקלד בחלון הפקודה של מסוף Tk:
מקור סיבוב.tcl
את סקריפט tcl ניתן למצוא בקובץ משלים 3. - לאחר מכן חישבו את השורש-הממוצע-ריבוע-מרחק (RMSD) של עמוד השדרה של החלבון על-ידי יישור הדנ"א המרכזי של 10 bp (A 14 עד 23 ו-T 14' עד 23') לזה מהמבנה הגבישי40, וה-RMSD מייצג מדדים גיאומטריים של המערכות (ראו איור 1A). עשה זאת על ידי לחיצה על VMD > הרחבות > ניתוח > כלי מסלול RMSD וסוג גרעין ושאריות 14 עד 23 ו- 46 עד 55 בתיבת בחירת האטום, לחץ על ישר ולאחר מכן על תיבת RMSD כדי לחשב את ערכי RMSD.
- חישוב דרגת הסיבוב של החלבון סביב DNA Θ(t) במישור y-z ב-MATLAB על-ידי הקלדת הפקודה
rad2deg(atan(z/y))
כאשר המיקום הזוויתי הראשוני מוגדר כ- Θ(0)=0, כפי שנערך בעבר8. - הקלד את הפקודה הבאה ב- MATLAB41 כדי להשתמש בשיטות K-means 42,43,44 וסווג את 10000 המבנים ל- 25 אשכולות על-ידי הקלדה:
[idx, C]=kmeans( X, 25)
כאן X היא מטריצה דו-ממדית של RMSD וזווית סיבובית של WRKY על הדנ"א. אסוף את המבנים של 25 מרכזי אשכולות אלה לסימולציות MD נוספות.
הערה: מאחר שהחלבון RMSD שנדגם ביחס לדנ"א מכסה טווח של כ-25 Å, אנו בוחרים ב-25 אשכולות שבהם יש צביר אחד לכל אנגסטרום.
- ביצוע סבב 1st של סימולציות MD והגדרות הסימולציה
- בנה מערכות אטומיסטיות עבור 25 המבנים על ידי שימוש בתוכנת GROMACS 5.1.232 תחת שדה כוח parmbsc145 ובאמצעות קובץ buildsystem.sh מקובץ משלים 2 במעטפת.
- בצע סימולציות MD של 60-ns עבור 25 מערכות אלה תחת אנסמבל NPT עם שלב זמן של 2 fs על ידי הקלדת הפקודה הבאה במעטפת:
gmx_mpi grompp -f md.mdp -c npt.gro -p topol.top -o md.tpr
gmx_mpi mdrun -deffnm md
- אשכולות 1סנט מסלולי MD עגולים
- הסר את 10 ה- ns הראשונים של כל מסלול סימולציה על-ידי הקלדת פגז:
gmx_mpi trjcat -f md.xtc -b 10000 -e 600000 -o newtraj.xtc
ולאסוף קונפורמציות ממסלולים של 25 × 50 ns עבור אשכולות כדי להכין את מבני הקלט לדגימות הנרחבות יותר הבאות (2 סימולציות MD בסיבובהשני ).
הערה: כדי להפחית את ההשפעה מהנתיב הראשוני ולאפשר שיווי משקל מקומי, הוסרו 10-ns מהתקופה הראשונית של הסימולציות. - בחר זוגות מרחקים בין חלבון לדנ"א כפרמטרים של קלט עבור היטל ניתוח רכיבים שאינם תלויים בזמן (tICA)46,47,48. השתמש בפקודה make_ndx ב- GROMACS כדי לעשות זאת:
gmx_mpi make_ndx -f קלט.pdb -o index.ndx
הערה: כאן נבחרו אטומי החלבון CA והאטומים הכבדים (NH1, NH2, OH, NZ, NE2, ND2) של שאריות Y119, K122, K125, R131, Y133, Q146, K144, R135, W116, R117, Y134, K118, Q121 שיכולים ליצור קשרי מימן (HBs) עם נוקלאוטיד הדנ"א, אשר מתחברים עם אטומי O1P O2P ו-N6 של נוקלאוטיד הדנ"א (A14-20, ט19-23). חומצות האמינו שנבחרו יכולות ליצור HBs יציבים או גשרי מלח עם DNA. - העתק את אינדקס האטום שנבחר לעיל מקובץ index.ndx לקובץ טקסט חדש (אינדקס.dat). קבל את המידע הזוגי בין אטומים אלה על ידי סקריפט הפיתון מקובץ משלים 1 generate_atom_indices.py והקלד:
python2.6 אינדקס generate_atom_indices.py.dat > AtomIndices.txt
זה יוצר את 415 זוגות המרחקים בין חלבון לדנ"א. - חשב את 415 זוגות המרחקים מכל מסלול על-ידי הקלדת הפקודה הבאה בחלון הפקודה של MSMbuilder:
msmb AtomPairsFeaturizer -out pair_features --pair_indices AtomIndices.txt --הפניות מובילות.pdb --trjs "מסלולים/*.xtc" --pair_features טרנספורמציה --צעד 5 - בצע tICA כדי להקטין את ממד הנתונים על שני הרכיבים הראשונים שאינם תלויים בזמן (tICs) או וקטורים על ידי הקלדה:
msmb tICA -i .. /tica_rc_a/tmp/ -o tica_results --n_components 2 --lag_time 10 --גמא 0.05 -t tica_results.h5
הערה: tICA היא שיטת הפחתת ממדים המחשבת את eigenvalue של מטריצת המתאם המפגרת בזמן כדי לקבוע את דרגות החופש המרגיעות האיטיות ביותר של מערכת הסימולציה על ידי המשוואה:
המתאם המפגרת בזמן כדי לקבוע את דרגות החופש המרגיעות האיטיות ביותר של מערכת הסימולציה על ידי המשוואה: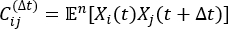
כאשר Xi(t) הוא הערך של קואורדינטת התגובה i-th בזמן t, ו- Xj(t+Δt) הוא הערך של קואורדינטת התגובה j-th בזמן t+Δt.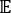 הוא ערך הציפייה של המכפלה של מסלולי הסימולציה הכוללים Xi(t) ו-Xj(t + Δt). הכיוונים לאורך דרגות החופש המרגיעות האיטיות ביותר תואמים את ה- eigenvalues הגדולים ביותר של מטריצת
הוא ערך הציפייה של המכפלה של מסלולי הסימולציה הכוללים Xi(t) ו-Xj(t + Δt). הכיוונים לאורך דרגות החופש המרגיעות האיטיות ביותר תואמים את ה- eigenvalues הגדולים ביותר של מטריצת  המתאם המפגרת בזמן לעיל . כאן, נראה כי 2 tICs הם קבוצה מינימלית להבחנה בין שלושה מקרוסטטים בבניית MSM שלנו (טופלה מאוחר יותר). ניתן גם לחשב את מטריצה כללית ריילי מניין (GMRQ) ציון49, למשל, כדי לחקור קבוצה אופטימלית של רכיבים לשימוש.
המתאם המפגרת בזמן לעיל . כאן, נראה כי 2 tICs הם קבוצה מינימלית להבחנה בין שלושה מקרוסטטים בבניית MSM שלנו (טופלה מאוחר יותר). ניתן גם לחשב את מטריצה כללית ריילי מניין (GMRQ) ציון49, למשל, כדי לחקור קבוצה אופטימלית של רכיבים לשימוש. - השתמש בפקודה ב- MSMbuilder כדי לקבץ את מערכי הנתונים הצפויים ל- 100 אשכולות בשיטת K-center43,44 (ראה איור 1B):
msmb KCenters -i ./tica_results.h5 -o kcenters_output -t kcenters_output --n_clusters 100.
בחר את המבנה המרכזי של כל אשכול כמבנה ההתחלתי של הסיבובהשני של סימולציות MD. שמור על מידע הסימולציה של 100 המבנים המדומים, כולל מיקומים, טמפרטורות, לחצים וכו ', למעט המהירויות.
הערה: לאחר הסיבוב הראשון של 25 סימולציות, הזיכרון של הנתיב הראשוני הופחת, כך שאנו מייצרים יותר אשכולות, למשל, 100 אשכולות, בסיבוב השני, כדי להרחיב באופן משמעותי את הדגימות הקונפורמיות.
- הסר את 10 ה- ns הראשונים של כל מסלול סימולציה על-ידי הקלדת פגז:
- ביצוע סימולציות MD נרחבות בסיבובהשני
- בצע סימולציות MD של 60-ns החל מ-100 המבנים הראשוניים הללו לאחר הטלת מהירויות ראשוניות אקראיות על כל האטומים. הוסף את המהירויות הראשוניות האקראיות על ידי הפעלת יצירת המהירות בקובץ mdp, כלומר, שינוי הקובץ md.mdp gen_vel = לא gen_vel = כן.
- הסר את 10 ה- ns הראשונים של כל סימולציה כמתואר בשלב 1.3.1, אסוף 2,500,000 תמונות מ- 100 × 50 ns מסלולים באופן שווה כדי לבנות את ה- MSM.
הערה: שים לב שבבניית המקרוסטטים המאוחרת יותר, נמצאו מספר קטן של מצבים מחוץ לנתיב עם אוכלוסייה נמוכה במיוחד (~0.2%, בתחתית מישור X-Θ). מצבים אלה מחוץ לנתיב מסווגים כמקרו-מדינה אחת כאשר המספר הכולל של מקרו-סטטים מוגדר כ-3 עד 6 (איור 2B). מכיוון שמקרוסטייט בעל אוכלוסייה כה נמוכה כולל רק 3 מסלולים, שהוסרו בסופו של דבר, התוצאות המוצגות בפרוטוקול זה אכן התקבלו מ-97 × מסלולים של 50 ns, עם סך של 2,425,000 פריימים או תמונות.
- אשכולות מסלולי MD בסיבובהשני
- ביצוע tICA עבור מסלולי הסיבובהשני כפי שנעשה בעבר. הקלד ב- MSMbuilder:
msmb tICA -i .. /tica_rc_a/tmp/ -o tica_results --n_components 2 --lag_time 10 --גמא 0.05 -t tica_results.h5 - חישב את ציר הזמן המשתמע כדי לאמת פרמטרים עבור מספרי זמן השהיית המתאם Δt ומיקרוסטטים (ראו איור 1C),
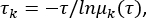
כאשר τ מייצג את זמן ההשהיה המשמש לבניית מטריצת הסתברות המעבר (TPM); μk(τ) מייצג את kth eigenvalue של ה- TPM תחת זמן השהיה של τ. השתמש בסקריפט python מקובץ משלים 1 עבור פייתון זה BuildMSMsAsVaryLagTime.py -d .. / -f .. /trajlist_num -i 50 -m 1000 -t 10 -n 20 -s 500. - שנה את מספר τ ו- microstates של זמן ההשהיה על-ידי שינוי הפרמטרים שבהם נעשה שימוש לעיל:
פייתון BuildMSMsAsVaryLagTime.py -d .. / -f .. /trajlist_num -i 50 -m 1000 -t 5 10 20 30 40 -n 20 -s 20 200 400 500 800 2000
הערה: המערכת נחשבת למרקוביאנית כאשר עקומות ציר הזמן המשתמעות מתחילות להתיישר עם הפרדת קנה המידה של הזמן. לאחר מכן, בחר את ה- Dt כזמן השהיית המתאם, ואת τ את זמן ההשהיה שבו ציר הזמן המשתמע מתחיל להתיישר כדי לבנות MSM. - בהתאם לכך, בחר מספר גדול יחסית (אך לא גדול מדי) של מצבים, N = 500, וזמן עיכוב מתאם קצר יחסית Δt = 10 ns. נמצא כי זמן ההשהיה הוא τ =10 ns לבניית MSM.
- סווג את ההתאמות ל-500 אשכולות (ראו איור 1D) באמצעות הפקודה:
msmb KCenters -i ./tica_results.h5 -o kcenters_output -t kcenters_output --n_clusters 500
- ביצוע tICA עבור מסלולי הסיבובהשני כפי שנעשה בעבר. הקלד ב- MSMbuilder:
- בניית MSM
- רכזו את 500 המיקרוסטטים ל-3-6 מקרוסטטים כדי לגלות את מספר המאקרוסטטים המתאימים ביותר על פי אלגוריתם PCCA+50 ב-MSMbuilder, על ידי שימוש בכתב הפיתון בקובץ משלים 1 msm_lumping_usingPCCAplus.py. זהה רשת קינטית מופחתת של מודלים לשינויים הקונפורמיים החיוניים ביותר של ביו-מולקולות, על-ידי בניית מספר קטן של מקרו-סטטים, כלומר על קיבוץ קינטי של מאות מיקרוסטטים כמתואר מתחתל-17,51.
- ממפה את הקונפורמציות הגבוהות ל-X (תנועת החלבון לאורך ציר ארוך הדנ"א) ואת זווית הסיבוב של החלבון לאורך הדנ"א עבור כל מקרו-סטאט כמתואר בשלבים 1.1.3 ו-1.1.4 (לדוגמה, אין מצב עם אוכלוסייה נמוכה מדי < 1%; ראו איור 2C). לאחר מכן מצא את 3 המקרוסטטים המייצגים בצורה הטובה ביותר את המערכת (איור 1E) . ראו איור 2D לקבלת תמונות של תנועת החלבון לאורך הדנ"א וזווית סיבוב החלבון סביב הדנ"א.
הערה: בעבודה קודמת שיצרה את נתיב הדריכה קדימה של חלבון ספונטני של 10 מיקרו-μs, ערכנו בנוסף סימולציות MD של שיווי משקל של 5 x 4 מיקרו-μs כדי להרחיב באופן מתון את הדגימות. הראינו את המיפוי של הנתיב הקדמי המקורי (ראו איור 2A משמאל) ומסלולי דגימה נוספים של 4 מיקרו-מיקרו-μs בנתיב קדימה שנערכו בעבר (ראו איור 2A מימין)8. מוצג המיפוי של 100 × 50 ns המקוריים (ראו איור 2B משמאל)8 ומסלולים של 97 × 50 ns ששימשו בעבודה זו (ראו איור 2B מימין).
- חישוב זמני המעבר הראשונים הממוצעים (MFPT)
- בצע חמישה מסלולים של 10 אלפיות השנייה מונטה קרלו (MC) בהתבסס על ה- TPM של MSM של 500 מיקרו-מדינה כאשר זמן ההשהיה של 10 ns מוגדר כשלב הזמן של MC. חשב את MFPT52 בין כל זוג של מקרוסטטים (איור 3) על ידי כתב הפיתון בקובץ משלים 1 פייתון פיתון mfpt_msm3.py.
- חשב את השגיאה הממוצעת והסטנדרטית של ה- MFPT באמצעות קובץ bash בקובץ משלים 2, הקלד:
sh mfpt_analysis.bash
2. ביצוע סימולציה של גרגירים גסים (CG) כדי לדגום דינמיקה ארוכת טווח
- בצע סימולציות CG באמצעות תוכנת CafeMol 3.030. ראה את הגדרות הסימולציה של CG שצוינו בקובץ תצורת הקלט עם סיומת .inp, כולל מבני קלט, פרמטרי סימולציה, קבצי פלט וכו '. הקלד את הפקודה הבאה במסוף כדי להפעיל את סימולציית CG:
cafemol XXX.inp - ציין את הבלוקים הבאים בקובץ הקלט, כאשר כל בלוק מתחיל בתווית < and ending with >>>>.
- הגדר בלוק שמות קבצים (נדרש) כדי לציין את ספריות העבודה ואת נתיב אחסון קבצי הקלט/פלט. הקלד את הדברים הבאים עבור בלוק שמות הקבצים עבור סימולציות אלה:
<<<< שמות קבצים
נתיב = XXXXX (נתיב עבודה)
filename = wrky (שמות קבצי הפלט)
פלט psf pdb סרט dcd rst
path_pdb = XXXXX (נתיב מבנה מקורי של קלט)
path_ini = XXXXX (נתיב מבנה ראשוני של קלט)
path_natinfo = XXXXX (נתיב קובץ מידע מקורי)
path_para = XXXXX (נתיב קבצי פרמטרים)
>>>>
הערה: מכיוון שמודלGo-model 53 משמש במידול CG, כלומר, החלבון יהיה מוטה לקונפורמציה המקורית, ולכן יש להגדיר את המבנה הממודל כקומפונמציה המקורית. כאן, המבנה הגבישי של הקלט הוגדר כקונפורמינציה המקומית. - הגדר את בלוק בקרת המשימה (הנדרש) להגדרת מצב הריצה של הסימולציות. הקלד את הפקודה הבאה:
<<<< job_cntl
i_run_mode = 2 (= 2 סימולציית הטמפרטורה הקבועה)
i_simulate_type = 1 (=1 דינמיקת לנגווין)
i_initial_state = 2 (=2 פירושו שהתצורה הראשונית היא תצורה מקורית)
>>>>
בחר את סימולציות הדינמיקה של לנגווין בטמפרטורה קבועה. - הגדר את בלוק היחידה והמצב (נדרש) להגדרת המידע עבור מבני קלט. הקלד את הפקודה הבאה:
<<<< unit_and_state
i_seq_read_style = 1 (=1 פירושו לקרוא רצפים מקובץ PDB)
i_go_native_read_style = 1 (=1 פירושו שהמבנה המקורי הוא מקובץ PDB)
1 חלבון חלבון.pdb (molecular_type native_structure יחידה ומצב)
2-3 DNA DNA.pdb (molecular_type native_structure יחידה ומדינה)
>>>>
הערה: יש צורך בקבצי מבנה הקלט הראשוניים (חלבון.pdb ודנ"א.pdb כאן). המבנים כתובים בפורמט pdb. יש צורך כאן בשני קבצי PDB: האחד הוא קובץ מבנה החלבון המכיל את קואורדינטות האטום הכבדות של WRKY (יחידה 1), והשני הוא הקואורדינטות של דנ"א דו-גדילי (ds) של 200 bp (יחידה 2-3). החלבון ממוקם בתחילה במרחק של 15 Å מהדנ"א. - הגדר את בלוק פונקציית האנרגיה (הנדרש) המוגדר בבלוק energy_function. הקלד את הפקודה הבאה:
<<<< energy_function
מקומי(1) L_GO
מקומי(2-3) L_DNA2
NLOCAL(1/1) GO EXV ELE
NLOCAL(2-3/2-3) ELE DNA
NLOCAL(1/2-3) EXV ELE
i_use_atom_protein = 0
i_use_atom_dna = 0
i_para_from_ninfo = 1
i_triple_angle_term = 2
>>>>
הערה: בהדמיות CG, החלבון מגורען גס על ידי מודל Go53 כאשר כל חומצת אמינו מיוצגת על ידי חלקיק CG הממוקם במיקום Cα שלו. הקונפורמציה של החלבון תהיה מוטה אז לכיוון המבנה הטבעי, או המבנה הגבישי כאן, תחת פוטנציאל Go (איור 4A משמאל). הדנ"א מתואר על ידי מודל 3SPN.254, שבו כל נוקלאוטיד מיוצג על ידי 3 חלקיקי CG S, P, N, המתאימים לסוכר, פוספט ובסיס חנקני, בהתאמה (איור 4A מימין). האינטראקציות האלקטרוסטטיות וה-vdW נחשבות בין שרשראות שונות. האינטראקציות האלקטרוסטטיות בין חלבון לדנ"א בסימולציית CG משוערות על ידי פוטנציאל דביי-הוקל55. האנרגיה הדוחה של vdW לובשת את אותה צורה כמו במודל Go. - הגדר את בלוק md_information (נדרש) להגדרת מידע הסימולציה. הקלד את הפקודה הבאה:
<<<< md_information
n_step_sim = 1
n_tstep(1) = 5000000000
tstep_size = 0.1
n_step_save = 1000
n_step_neighbor = 100
i_com_zeroing = 0
i_no_trans_rot = 0
tempk = 300.0
n_seed = -1
>>>>
n_tstep הוא שלב הסימולציה. הגדר את tstep_size כאורך הזמן של כל שלב MD, כל שלב זמן של CG Cafemol הוא בערך 200 fs30, כך שכל צעד MD כאן הוא 200 × 0.1 fs באופן עקרוני. עדכן את רשימת השכנים כל 100 שלבי MD (n_step_neighbor = 100). הגדר את טמפרטורת הסימולציה ל- 300 K. שלוט בטמפרטורה על ידי שימוש באלגוריתם Verlet מסוג מהירות לעדכון מבנה החלבון עם התרמוסטט של ברנדסן56.
הערה: n_step_sim הוא מספר האגן של הפוטנציאל המבוסס על מודל Go, או המספר המינימלי המקומי של עקומת האנרגיה. פוטנציאל של ריבוי אגנים מאפשר לקונפורמציה של חלבונים להיות מוטה לקונפורמציות שונות, כך שקומפונמציית חלבונים יכולה להשתנות ממינימום מקומי אחד למשנהו. כאן נעשה שימוש רק במודל Go של אגן יחיד, שמשמעותו רק קונפורמציה מוטה אחת (מבנה גבישי) לחלבון בסימולציות. בינתיים, מכיוון שאין אינטראקציית קשרי מימן בין חלבון לדנ"א וכו' במודלים בהקשר של CG, ניתן לדגום את התנועות המולקולריות אפילו מהר יותר, כלומר > פי 10 מאשר בסימולציות האטומיות. - הגדר בלוק אלקטרוסטטי (נדרש רק כאשר נעשה שימוש באינטראקציה אלקטרוסטטית) מכיוון שהאינטראקציה האלקטרוסטטית נחשבת בין שרשראות שונות, לכן השתמש בבלוק זה כדי להגדיר את הפרמטרים לאינטראקציה אלקטרוסטטית על ידי הקלדה:
<<<< אלקטרוסטטי
cutoff_ele = 10.0
ionic_strength = 0.15
>>>>
הגדר את אורך דביי באינטראקציה האלקטרוסטטית ל- 10 Å, המתאים לתנאי הפתרון. הגדר את העוצמה היונית ל-0.15 מ', כמו במצב הפיזיולוגי.
- הגדר בלוק שמות קבצים (נדרש) כדי לציין את ספריות העבודה ואת נתיב אחסון קבצי הקלט/פלט. הקלד את הדברים הבאים עבור בלוק שמות הקבצים עבור סימולציות אלה:
תוצאות
הזזה מצומדת לסיבוב או דריכה אחת של bp של WRKY מבניית MSM
כל קונפורמציות החלבון בדנ"א ממופות לתנועת האורך X ולזווית הסיבוב של החלבון COM לאורך הדנ"א (ראו איור 3A). הצימוד הליניארי של שתי מעלות אלה מצביע על דריכה מצומדת סיבוב של חלבון התחום WRKY על הדנ"א. ניתן לחלק את הקונפורמצ...
Discussion
עבודה זו עוסקת באופן שבו ניתן לבצע סימולציה חישובית מבוססת מבנה ודגימות כדי לחשוף גורם שעתוק או חלבון TF הנע לאורך הדנ"א, לא רק בפירוט האטומי של דריכה, אלא גם בדיפוזיה התהליכית, החיונית לדיפוזיה הקלה של TF בחיפוש מטרות הדנ"א. כדי לעשות זאת, נבנה לראשונה מודל מצב מרקוב או MSM של חלבון קטן בתחום TF W...
Disclosures
לכותבים אין ניגוד עניינים.
Acknowledgements
עבודה זו נתמכה על ידי מענק NSFC #11775016 ו #11635002. JY נתמך על ידי CMCF של UCI באמצעות NSF DMS 1763272 וקרן סימונס מענק #594598 וקרן סטארט-אפ מ- UCI. בע"מ נתמכה על ידי קרן מדעי הטבע של שנגחאי #20ZR1425400 #21JC1403100. אנו מכירים גם בתמיכה החישובית של מרכז המחקר המדעי החישובי בבייג'ינג (CSRC).
Materials
| Name | Company | Catalog Number | Comments |
| CafeMol | Kyoto University | coarse-grained (CG) simulations | |
| GROMACS | University of Groningen Royal Institute of Technology Uppsala University | molecular dynamics simulations software | |
| Matlab | MathWorks | Numerical calculation software | |
| MSMbuilder | Stanford University | build MSM | |
| VMD | UNIVERSITY OF ILLINOIS AT URBANA-CHAMPAIGN | molecular visualization program |
References
- Latchman, D. S. Transcription factors: an overview. The International Journal of Biochemistry & Cell Biology. 29 (12), 1305-1312 (1997).
- Berg, O. G., von Hippel, P. H. Selection of DNA binding sites by regulatory proteins. Statistical-mechanical theory and application to operators and promoters. Journal of Molecular Biology. 193 (4), 723-750 (1987).
- von Hippel, P. H., Berg, O. G. Facilitated target location in biological systems. The Journal of Biological Chemistry. 264 (2), 675-678 (1989).
- Halford, S. E., Marko, J. F. How do site-specific DNA-binding proteins find their targets. Nucleic Acids Research. 32 (10), 3040-3052 (2004).
- Slusky, M., Mirny, L. A. Kinetics of protein-DNA interaction: facilitated target location in sequence-dependent potential. Biophysical Journal. 87 (6), 4021-4035 (2004).
- Bauer, M., Metzler, R. Generalized facilitated diffusion model for DNA-binding proteins with search and recognition states. Biophysical Journal. 102 (10), 2321-2330 (2012).
- Shvets, A. A., Kochugaeva, M. P., Kolomeisky, A. B. Mechanisms of Protein Search for Targets on DNA: Theoretical Insights. Molecules. 23 (9), 2106 (2018).
- Dai, L., Xu, Y., Du, Z., Su, X. D., Yu, J. Revealing atomic-scale molecular diffusion of a plant-transcription factor WRKY domain protein along DNA. Proceedings of the National Academy of Sciences of the United States of America. 118 (23), 2102621118 (2021).
- Chodera, J. D., Singhal, N., Pande, V. S., Dill, K. A., Swope, W. C. Automatic discovery of metastable states for the construction of Markov models of macromolecular conformational dynamics. The Journal of Chemical Physics. 126 (15), 155101 (2007).
- Pan, A. C., Roux, B. Building Markov state models along pathways to determine free energies and rates of transitions. The Journal of Chemical Physics. 129 (6), 064107 (2008).
- Bowman, G. R., Huang, X., Pande, V. S. Using generalized ensemble simulations and Markov state models to identify conformational states. Methods. 49 (2), 197-201 (2009).
- Prinz, J. H., et al. Markov models of molecular kinetics: Generation and validation. The Journal of chemical physics. 134 (17), 174105 (2011).
- Chodera, J. D., Noé, F. Markov state models of biomolecular conformational dynamics. Current Opinion in Structural Biology. 25, 135-144 (2014).
- Malmstrom, R. D., Lee, C. T., Van Wart, A. T., Amaro, R. E. On the Application of Molecular-Dynamics Based Markov State Models to Functional Proteins. Journal of Chemical Theory and Computation. 10 (7), 2648-2657 (2014).
- Husic, B. E., Pande, V. S. Markov State Models: From an Art to a Science. Journal of the American Chemical Society. 140 (7), 2386-2396 (2018).
- Sittel, F., Stock, G. Perspective: Identification of collective variables and metastable states of protein dynamics. The Journal of chemical physics. 149 (15), 150901 (2018).
- Wang, W., Cao, S., Zhu, L., Huang, X. Constructing Markov State Models to elucidate the functional conformational changes of complex biomolecules. WIREs Computational Molecular Science. 8, 1343 (2018).
- Peng, S., et al. Target search and recognition mechanisms of glycosylase AlkD revealed by scanning FRET-FCS and Markov state models. Proceedings of the National Academy of Sciences of the United States of America. 117 (36), 21889-21895 (2020).
- Tian, J., Wang, L., Da, L. T. Atomic resolution of short-range sliding dynamics of thymine DNA glycosylase along DNA minor-groove for lesion recognition. Nucleic Acids Research. 49 (3), 1278-1293 (2021).
- Chu, J. -. W., Izveko, S., Voth, G. The multiscale challenge for biomolecular systems: coarse-grained modeling. Molecular Simulation. 32 (3-4), 211-218 (2006).
- Marrink, S. J., Risselada, H. J., Yefimov, S., Tieleman, D. P., De Vries, A. H. The MARTINI force field: coarse grained model for biomolecular simulations. The Journal of Physical Chemistry B. 111 (27), 7812-7824 (2007).
- Givaty, O., Levy, Y. Protein sliding along DNA: dynamics and structural characterization. Journal of Molecular Biology. 385 (4), 1087-1097 (2009).
- Khazanov, N., Levy, Y. Sliding of p53 along DNA can be modulated by its oligomeric state and by cross-talks between its constituent domains. Journal of Molecular Biology. 408 (2), 335-355 (2011).
- Riniker, S., Allison, J. R., van Gunsteren, W. F. On developing coarse-grained models for biomolecular simulation: a review. Physical Chemistry Chemical Physics : PCCP. 14 (36), 12423-12430 (2012).
- Kmiecik, S., et al. Coarse-Grained Protein Models and Their Applications. Chemical Reviews. 116 (14), 7898-7936 (2006).
- Bhattacherjee, A., Krepel, D., Levy, Y. Coarse-grained models for studying protein diffusion along DNA. WIREs Computational Molecular Science. 6, 515-531 (2016).
- Wang, J., et al. Machine Learning of Coarse-Grained Molecular Dynamics Force Fields. ACS Central Science. 5 (5), 755-767 (2019).
- Joshi, S. Y., Deshmukh, S. A. A review of advancements in coarse-grained molecular dynamics simulations. Molecular Simulation. 47 (10-11), 786-803 (2021).
- Bigman, L. S., Greenblatt, H. M., Levy, Y. What Are the Molecular Requirements for Protein Sliding along DNA. The Journal of Physical Chemistry B. 125 (12), 3119-3131 (2021).
- Kenzaki, H., et al. CafeMol: A Coarse-Grained Biomolecular Simulator for Simulating Proteins at Work. Journal of Chemical Theory and Computation. 7 (6), 1979-1989 (2011).
- Berendsen, H. J. C., vander Spoel, D., van Drunen, R. GROMACS: a message-passing parallel molecular dynamics implementation. Computer Physics Communications. 91 (1-3), 43-56 (1995).
- vander Spoel, D., et al. GROMACS: fast, flexible, and free. Journal of Computational Chemistry. 26 (16), 1701-1718 (2005).
- Abraham, M. J., et al. GROMACS: High performance molecular simulations through multi-level parallelism from laptops to supercomputers. SoftwareX. 1-2, 19-25 (2015).
- Harrigan, M. P., et al. MSMBuilder: Statistical Models for Biomolecular Dynamics. Biophysical journal. 112 (1), 10-15 (2017).
- Humphrey, W., Dalke, A., Schulten, K. VMD: visual molecular dynamics. Journal of Molecular Graphics. 14 (1), 33-38 (1996).
- Izrailev, S., et al. Steered Molecular Dynamics. Computational Molecular Dynamics: Challenges, Methods, Ideas. 4, 39-65 (1999).
- Schlitter, J., Engels, M., Krüger, P. Targeted molecular dynamics: a new approach for searching pathways of conformational transitions. Journal of Molecular Graphics. 12 (2), 84-89 (1994).
- Maragliano, L., Fischer, A., Vanden-Eijnden, E., Ciccotti, G. String method in collective variables: minimum free energy paths and isocommittor surfaces. The Journal of Chemical Physics. 125 (2), 24106 (2006).
- Weiss, D. R., Levitt, M. Can morphing methods predict intermediate structures. Journal of Molecular Biology. 385 (2), 665-674 (2009).
- Xu, Y. P., Xu, H., Wang, B., Su, X. D. Crystal structures of N-terminal WRKY transcription factors and DNA complexes. Protein. 11 (3), 208-213 (2020).
- Higham, D. J., Higham, N. J. MATLAB guide. Society for Industrial and Applied Mathematics. , (2016).
- Hartigan, J. A., Wong, M. A. Algorithm AS 136: A K-Means Clustering Algorithm. Journal of the Royal Statistical Society. Series C (Applied Statistics). 28 (1), 100-108 (1979).
- Gonzalez, T. F. Clustering to minimize the maximum intercluster distance. Theoretical Computer Science. 38, 293-306 (1985).
- Zhao, Y., Sheong, F. K., Sun, J., Sander, P., Huang, X. A fast parallel clustering algorithm for molecular simulation trajectories. Journal of Computational Chemistry. 34 (2), 95-104 (2013).
- Ivani, I., et al. Parmbsc1: a refined force field for DNA simulations. Nature Methods. 13 (1), 55-58 (2016).
- Naritomi, Y., Fuchigami, S. Slow dynamics of a protein backbone in molecular dynamics simulation revealed by time-structure based independent component analysis. The Journal of Chemical Physics. 139 (21), 215102 (2013).
- Naritomi, Y., Fuchigami, S. Slow dynamics in protein fluctuations revealed by time-structure based independent component analysis: the case of domain motions. The Journal of Chemical Physics. 134 (6), 065101 (2011).
- Pérez-Hernández, G., Paul, F., Giorgino, T., De Fabritiis, G., Noé, F. Identification of slow molecular order parameters for Markov model construction. The Journal of Chemical Physics. 139 (1), 015102 (2013).
- McGibbon, R. T., Pande, V. S. Variational cross-validation of slow dynamical modes in molecular kinetics. The Journal of Chemical Physics. 142 (12), 124105 (2015).
- Deuflhard, P., Weber, M. Robust Perron cluster analysis in conformation dynamics. Linear Algebra and its Applications. 398, 161-184 (2005).
- Silva, D. A., et al. Millisecond dynamics of RNA polymerase II translocation at atomic resolution. Proceedings of the National Academy of Sciences of the United States of America. 111 (21), 7665-7670 (2014).
- Swope, W. C., Pitera, J. W., Suits, F. Describing Protein Folding Kinetics by Molecular Dynamics Simulations. 1. Theory. The Journal of Physical Chemistry B. 108 (21), 6571-6581 (2004).
- Clementi, C., Nymeyer, H., Onuchic, J. N. Topological and energetic factors: what determines the structural details of the transition state ensemble and "en-route" intermediates for protein folding? An investigation for small globular proteins. Journal of molecular biology. 298 (5), 937-953 (2000).
- Hinckley, D. M., Freeman, G. S., Whitmer, J. K., De Pablo, J. J. An experimentally-informed coarse-grained 3-Site-Per-Nucleotide model of DNA: structure, thermodynamics, and dynamics of hybridization. The Journal of chemical physics. 139 (14), 144903 (2013).
- Debye, P., Huckel, E. The theory of the electrolyte II-The border law for electrical conductivity. Physikalische Zeitschrift. 24, 305-325 (1923).
- Berendsen, H. J., Postma, J. V., van Gunsteren, W. F., DiNola, A., Haak, J. R. Molecular dynamics with coupling to an external bath. The Journal of Chemical Physics. 81, 3684-3690 (1984).
- Bowman, G. R. Improved coarse-graining of Markov state models via explicit consideration of statistical uncertainty. The Journal of Chemical Physics. 137 (13), 134111 (2012).
- Jain, A., Stock, G. Identifying metastable states of folding proteins. Journal of Chemical Theory and Computation. 8 (10), 3810-3819 (2012).
- Röblitz, S., Weber, M. Fuzzy spectral clustering by PCCA+: application to Markov state models and data classification. Advances in Data Analysis and Classification. 7, 147-179 (2013).
- Mardt, A., Pasquali, L., Wu, H., Noé, F. VAMPnets for deep learning of molecular kinetics. Nature Communications. 9 (1), 5 (2018).
- Wang, W., Liang, T., Sheong, F. K., Fan, X., Huang, X. An efficient Bayesian kinetic lumping algorithm to identify metastable conformational states via Gibbs sampling. The Journal of Chemical Physics. 149 (7), 072337 (2018).
- Chen, W., Sidky, H., Ferguson, A. L. Nonlinear discovery of slow molecular modes using state-free reversible VAMPnets. The Journal of Chemical Physics. 150 (21), 214114 (2019).
- Gu, H., et al. RPnet: a reverse-projection-based neural network for coarse-graining metastable conformational states for protein dynamics. Physical Chemistry Chemical Physics :PCCP. 24 (3), 1462-1474 (2022).
- Lane, T. J., Bowman, G. R., Beauchamp, K., Voelz, V. A., Pande, V. S. Markov state model reveals folding and functional dynamics in ultra-long MD trajectories. Journal of the American Chemical Society. 133 (45), 18413-18419 (2011).
- Konovalov, K. A., Unarta, I. C., Cao, S., Goonetilleke, E. C., Huang, X. Markov State Models to Study the Functional Dynamics of Proteins in the Wake of Machine Learning. JACS Au. 1 (9), 1330-1341 (2021).
- Cao, S., Montoya-Castillo, A., Wang, W., Markland, T. E., Huang, X. On the advantages of exploiting memory in Markov state models for biomolecular dynamics. The Journal of Chemical Physics. 153 (1), 014105 (2020).
- Brandani, G. B., Takada, S. Chromatin remodelers couple inchworm motion with twist-defect formation to slide nucleosomal DNA. PLoS Computational Biology. 14 (11), 1006512 (2018).
- Tan, C., Terakawa, T., Takada, S. Dynamic Coupling among Protein Binding, Sliding, and DNA Bending Revealed by Molecular Dynamics. Journal of the American Chemical Society. 138 (27), 8512-8522 (2016).
- Terakawa, T., Takada, S. p53 dynamics upon response element recognition explored by molecular simulations. Scientific reports. 5, 17107 (2015).
- Brandani, G. B., Niina, T., Tan, C., Takada, S. DNA sliding in nucleosomes via twist defect propagation revealed by molecular simulations. Nucleic Acids Research. 46 (6), 2788-2801 (2018).
- Knotts, T. A., Rathore, N., Schwartz, D. C., de Pablo, J. J. A coarse grain model for DNA. The Journal of Chemical Physics. 126 (8), 084901 (2007).
- Freeman, G. S., Hinckley, D. M., Lequieu, J. P., Whitmer, J. K., de Pablo, J. J. Coarse-grained modeling of DNA curvature. The Journal of Chemical Physics. 141 (16), 165103 (2014).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved