Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Структурно-ориентированное моделирование и отбор проб движения белка транскрипционного фактора вдоль ДНК от ступенчатого шага атомного масштаба до крупнозернистой диффузии
* Эти авторы внесли равный вклад
В этой статье
Резюме
Целью данного протокола является выявление структурной динамики одномерной диффузии белка по ДНК с использованием в качестве образцовой системы растительного транскрипционного фактора WRKY доменного белка. Для этого было реализовано как атомистическое, так и крупнозернистое моделирование молекулярной динамики, а также обширные вычислительные выборки.
Аннотация
Одномерное (1-D) скольжение белка транскрипционного фактора (TF) вдоль ДНК необходимо для облегченной диффузии TF для обнаружения целевого участка ДНК для генетической регуляции. Обнаружение разрешения пары оснований (bp) tf, скользящего или наступающего на ДНК, все еще экспериментально сложно. Недавно мы выполнили моделирование молекулярной динамики всех атомов (MD), фиксирующее спонтанное 1-bp степпинг небольшого белка TF домена WRKY вдоль ДНК. Основываясь на 10 мкс ступенчатого пути WRKY, полученном в результате такого моделирования, протокол здесь показывает, как проводить более обширные конформационные выборки систем TF-ДНК путем построения модели состояния Маркова (MSM) для степпинга белка 1-bp с различным количеством микро- и макро-состояний, протестированных для построения MSM. Чтобы исследовать процессный 1-D диффузионный поиск белка TF вместе со структурной основой, протокол дополнительно показывает, как проводить крупнозернистое (CG) MD-моделирование для выборки долгосрочной масштабной динамики системы. Такое CG-моделирование и симуляции особенно полезны для выявления электростатического воздействия белка-ДНК на процессивные диффузионные движения белка TF выше десятков микросекунд по сравнению с субмикросекундами до микросекунд ступенчатыми движениями белка, выявленными в результате моделирования всех атомов.
Введение
Транскрипционные факторы (TF) ищут целевую ДНК для связывания и регулирования транскрипции генов и связанных с ней действий1. Помимо трехмерной (3D) диффузии, было предложено, чтобы облегченная диффузия TF была необходима для поиска целевой ДНК, в котором белки также могут скользить или прыгать по одномерной (1D) ДНК или прыгать с межсегментальным переносом на ДНК 2,3,4,5,6,7.
В недавнем исследовании мы провели десятки микросекунд (мкс) моделирования молекулярной динамики всеатомного равновесия (MD) на растительном TF - белке домена WRKY на ДНК8. Был зафиксирован полный шаг WRKY на поли-А ДНК в течение микросекунд. Наблюдались движения белка вдоль канавки ДНК и динамика прорыва-риформинга водородных связей (HBs). Хотя такая траектория представляет собой один отобранный путь, общий ландшафт степпинга белка все еще отсутствует. Здесь мы покажем, как расширить вычислительные выборки вокруг первоначально захваченного пути шага белка с помощью построенной модели состояния Маркова (MSM), которая была широко реализована для моделирования различных биомолекулярных систем, включающих существенные конформационные изменения и разделение по шкале времени 9,10,11,12,13,14,15,16, 17,18,19. Цель состоит в том, чтобы выявить конформационный ансамбль и метастабильные состояния диффузии белка TF вдоль ДНК за одну циклическую стадию.
В то время как приведенное выше моделирование MD показывает атомное разрешение движений белка для 1 bp на ДНК, структурная динамика длительной процессивной диффузии TF вдоль ДНК с тем же высоким разрешением едва ли доступна. Однако проведение моделирования крупнозернистых (CG) MD на уровне остатков технически доступно. Шкала времени моделирования CG может быть эффективно расширена до десятков или сотен раз дольше, чем атомное моделирование 20,21,22,23,24,25,26,27,28,29. Здесь мы показываем компьютерное моделирование, проведенное с помощью программного обеспечения CafeMol, разработанного Takada lab30.
В текущем протоколе мы представляем атомное моделирование белка домена WRKY вдоль поли-А ДНК и построение МСМ, которые сосредоточены на выборке шаговых движений белка только для 1 bp вдоль ДНК. Затем мы представляем CG-моделирование и симуляции той же системы белка-ДНК, которые расширяют вычислительную выборку до процессной диффузии белка на десятки бит в секунду по ДНК.
Здесь мы используем программное обеспечение GROMACS 31,32,33 для проведения MD-моделирования и MSMbuilder34 для построения MSM для выборочных конформационных снимков, а также используем VMD 35 для визуализации биомолекул. Протокол требует, чтобы пользователь мог установить и реализовать программное обеспечение, указанное выше. Установка и внедрение программного обеспечения CafeMol30 затем необходимо для проведения моделирования CG MD. Дальнейший анализ траекторий и визуализация также проводятся в VMD.
протокол
1. Построение модели состояния Маркова (МСМ) на основе атомного моделирования MD
- Спонтанный белковый ступенчатый путь и сбор начальных структур
- Используйте ранее полученную 10-мкс полноатомную траекторию MD8 для равномерного извлечения 10000 кадров из «прямого» ступенчатого пути 1-bp (т.е. одного кадра на каждую наносекунду). Общее количество кадров должно быть достаточно большим, чтобы включать все репрезентативные конформации.
- Подготовьте путь перехода с 10000 кадрами в VMD, щелкнув Файл > Сохранить координаты, введите белок или нуклеину в поле Выбранные атомы и выберите кадры в поле Кадры, нажмите кнопку Сохранить , чтобы получить необходимые кадры.
ПРИМЕЧАНИЕ: В качестве начального пути для запуска дальнейших конформационных выборок использовалась ранее полученная 10-мкс траектория моделирования MD (называемая здесь «траекторией шага вперед») для WRKY, шагающего на расстояние 1-bp на однородной поли-A ДНК8 с 34 bp. Обратите внимание, что в большинстве практик, однако, начальный путь строится путем выполнения управляемого или целевого моделирования MD или реализации общих методов генерации путей и т. Д. 36,37,38,39. - Выровнять длинную ось эталонной ДНК (от кристаллической структуры) к оси x и установить начальный центр масс (COM) полной 34-bp ДНК в начале координатного пространства для удобства дальнейшего анализа данных. Для этого щелкните Расширения > Tk Console в VMD и введите в командном окне консоли Tk:
источник rotate.tcl
Скрипт tcl можно найти в дополнительном файле 3. - Затем вычислите среднеквадратичное расстояние (RMSD) белковой основы, выровняв центральную ДНК 10 bp (A от 14 до 23 и T 14' до 23') к кристаллической структуре40, и RMSD представляют геометрические меры систем (см. Рисунок 1A). Для этого щелкните VMD > Расширения > Анализ > инструмент траектории RMSD и введите нуклеичные и остатки от 14 до 23 и 46 до 55 в поле выбора атома, нажмите кнопку Выровнять , а затем поле RMSD, чтобы вычислить значения RMSD.
- Рассчитайте степень вращения белка вокруг ДНК Θ(t) на плоскости y-z в MATLAB, набрав команду
rad2deg(atan(z/y))
с начальным угловым позиционированием, определенным как Θ(0)=0, как проводилось ранее8. - Введите следующую команду в MATLAB41, чтобы использовать методы K-средних 42,43,44 и классифицируйте 10000 структур в 25 кластеров, введя:
[idx, C]=kmeans( X, 25)
здесь X представляет собой 2D-матрицу RMSD и угла вращения WRKY на ДНК. Соберите структуры этих 25 кластерных центров для дальнейшего моделирования MD.
ПРИМЕЧАНИЕ: Поскольку белок RMSD, отобранный относительно ДНК, охватывает диапазон около 25 Å, мы выбираем 25 кластеров, чтобы иметь один кластер на ангстрем.
- Проведение1-го раунда MD симуляций и настроек моделирования
- Построение атомистических систем для 25 структур с помощью программного обеспечения GROMACS 5.1.232 под силовым полемparmbsc1 45 и с помощью файла buildsystem.sh из дополнительного файла 2 в оболочке.
- Проведите моделирование 60-ns MD для этих 25 систем в рамках ансамбля ДНЯО с временным шагом 2 fs, набрав следующую команду в оболочке:
gmx_mpi grompp -f md.mdp -c npt.gro -p topol.top -o md.tpr
gmx_mpi mdrun -deffnm md
- Кластеризация 1ул круговые траектории MD
- Удалите первые 10 нс каждой траектории моделирования, введя в оболочку:
gmx_mpi trjcat -f md.xtc -b 10000 -e 600000 -o newtraj.xtc
и собирать конформации из траекторий 25 × 50 нс для кластеризации для подготовки входных структур для последующих более обширных выборок (моделирование MD2-го раунда).
ПРИМЕЧАНИЕ: Чтобы уменьшить воздействие от начального пути и обеспечить локальное равновесие, 10-нс начального периода моделирования были удалены. - Выберите пары расстояний между белком и ДНК в качестве входных параметров для независимого от времени компонентного анализа (tICA)46,47,48 проекции. Используйте команду make_ndx в GROMACS для этого:
gmx_mpi make_ndx -f вход.pdb -o index.ndx
ПРИМЕЧАНИЕ: Здесь были выбраны атомы белка CA и тяжелые атомы (NH1, NH2, OH, NZ, NE2, ND2) остатка Y119, K122, K125, R131, Y133, Q146, K144, R135, W116, R117, Y134, K118, Q121, которые могут образовывать водородные связи (HBs) с нуклеотидом ДНК, которые спариваются с атомами O1P O2P и N6 нуклеотида ДНК (A14-20, Т19-23). Выбранные аминокислоты могут образовывать либо стабильные HBs, либо солевые мостики с ДНК. - Скопируйте выбранный выше индекс атома из файла index.ndx в новый текстовый файл (index.dat). Получите информацию о паре между этими атомами с помощью скрипта python из дополнительного файла 1 generate_atom_indices.py и введите:
индекс generate_atom_indices.py python2.6.dat > AtomIndices.txt
Это генерирует 415 пар расстояний между белком и ДНК. - Рассчитайте 415 пар расстояний от каждой траектории, введя следующую команду в командном окне MSMbuilder:
msmb AtomPairsFeaturizer -out pair_features --pair_indices AtomIndices.txt --top references.pdb --trjs "trajectories/*.xtc" --transform pair_features --stride 5 - Проведите tICA для уменьшения размерности данных на первые 2 независимых от времени компонента (tIC) или векторов, набрав:
msmb tICA -i .. /tica_rc_a/tmp/ -o tica_results --n_components 2 --lag_time 10 --gamma 0.05 -t tica_results.h5
ПРИМЕЧАНИЕ: tICA - это метод уменьшения размерности, который вычисляет собственное значение матрицы корреляции с задержкой во времени для определения самых медленных расслабляющих степеней свободы системы моделирования по уравнению:
корреляции с задержкой во времени для определения самых медленных расслабляющих степеней свободы системы моделирования по уравнению: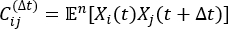
где Xi(t) — значение i-й координаты реакции в момент времени t, а Xj(t+Δt) — значение координаты j-й реакции в момент времени t+Δt.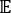 — ожидаемое значение произведения Xi(t) и Xj(t + Δt) общих траекторий моделирования. Направления вдоль самых медленных расслабляющих степеней свободы соответствуют наибольшим собственным значениям приведенной выше матрицы
— ожидаемое значение произведения Xi(t) и Xj(t + Δt) общих траекторий моделирования. Направления вдоль самых медленных расслабляющих степеней свободы соответствуют наибольшим собственным значениям приведенной выше матрицы  корреляции с задержкой во времени. Здесь 2 tIC кажутся минимальным набором для дифференциации трех макросостояний в нашей конструкции MSM (рассмотренной позже). Можно также вычислить обобщенную матрицу коэффициента Рэлея (GMRQ) с оценкой49, например, для изучения оптимального набора компонентов, которые будут использоваться.
корреляции с задержкой во времени. Здесь 2 tIC кажутся минимальным набором для дифференциации трех макросостояний в нашей конструкции MSM (рассмотренной позже). Можно также вычислить обобщенную матрицу коэффициента Рэлея (GMRQ) с оценкой49, например, для изучения оптимального набора компонентов, которые будут использоваться. - Используйте команду в MSMbuilder для кластеризации проецируемых наборов данных в 100 кластеров методом K-center43,44 (см. рисунок 1B):
msmb KCenters -i ./tica_results.h5 -o kcenters_output -t kcenters_output --n_clusters 100.
Выберите центральную структуру каждого кластера в качестве исходной структуры для2-го раунда моделирования MD. Поддерживайте информацию моделирования моделируемых 100 структур, включая положения, температуры, давления и т. Д., За исключением скоростей.
ПРИМЕЧАНИЕ: После первого раунда из 25 симуляций память начального пути была уменьшена, поэтому мы генерируем больше кластеров, например, 100 кластеров, во втором раунде, чтобы существенно расширить конформационные выборки.
- Удалите первые 10 нс каждой траектории моделирования, введя в оболочку:
- Проведение2-го раунда обширного моделирования MD
- Проведите 60-нс MD моделирование, начиная с этих 100 начальных структур после наложения случайных начальных скоростей на все атомы. Сложите случайные начальные скорости, включив генерацию скоростей в mdp-файле, т.е. изменив файл md.mdp gen_vel = no на gen_vel = yes.
- Удалите первые 10 нс каждого моделирования, как описано в шаге 1.3.1, соберите 2 500 000 снимков со 100 × 50 нс траекторий равномерно, чтобы построить МСМ.
ПРИМЕЧАНИЕ: Обратите внимание, что в более поздней конструкции макросостояний было обнаружено небольшое количество внепутных состояний с особенно низкой популяцией (~0,2%, на дне плоскости X-Θ). Эти внедорожные состояния классифицируются как одно макросостояние, когда общее число макросостояний установлено как от 3 до 6 (рисунок 2B). Поскольку такое низкое популяционное макросостояние включает в себя только 3 траектории, которые были удалены в конце концов, результаты, показанные в этом протоколе, были получены действительно из 97 × 50 траекторий ns, в общей сложности 2 425 000 кадров или снимков.
- Кластеризация траекторий2-го раунда MD
- Проведите tICA для траекторий2-го раунда, как это было сделано ранее. Тип в MSMbuilder:
msmb tICA -i .. /tica_rc_a/tmp/ -o tica_results --n_components 2 --lag_time 10 --gamma 0.05 -t tica_results.h5 - Вычислите подразумеваемую временную шкалу для проверки параметров времени задержки корреляции Δt и чисел микросостояний (см. Рисунок 1С),
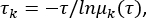
где τ представляет время задержки, используемое для построения матрицы вероятности перехода (TPM); μk(τ) представляет собственное значение kth доверенного платформенного модуля при времени задержки τ. Используйте скрипт python из дополнительного файла 1 для этого python BuildMSMsAsVaryLagTime.py -d .. / -ф.. /trajlist_num -i 50 -м 1000 -т 10 -н 20 -с 500. - Измените время задержки τ и число микросостояний, изменив параметры, используемые выше:
python BuildMSMsAsVaryLagTime.py -d .. / -ф.. /trajlist_num -i 50 -м 1000 -т 5 10 20 30 40 -н 20 -с 20 200 400 500 800 2000
ПРИМЕЧАНИЕ: Система считается марковской, когда подразумеваемые кривые временной шкалы начинают выравниваться с разделением по шкале времени. Затем выберите Dt в качестве времени задержки корреляции и τ время задержки, когда подразумеваемая шкала времени начинает выравниваться для построения MSM. - Соответственно, выбирайте сравнительно большое (но не слишком большое) число состояний N = 500 и сравнительно короткое время задержки корреляции Δt = 10 нс. Было обнаружено, что время задержки составляет τ = 10 нс для построения MSM.
- Классифицируйте конформации на 500 кластеров (см. рисунок 1D) с помощью команды:
MSMB KCenters -i ./tica_results.h5 -o kcenters_output -t kcenters_output --n_clusters 500
- Проведите tICA для траекторий2-го раунда, как это было сделано ранее. Тип в MSMbuilder:
- Строительство МСМ
- Объедините 500 микросостояний в 3–6 макросостояний, чтобы узнать количество макросостояний, которые лучше всего подходят в соответствии с алгоритмом PCCA+50 в MSMbuilder, используя скрипт python в дополнительном файле 1 python msm_lumping_usingPCCAplus.py. Идентифицировать уменьшенную кинетическую сеть моделей для наиболее существенных конформационных изменений биомолекул путем построения небольшого числа макросостояний, т.е. при кинетическом объединении сотен микросостояний, как описано ниже17,51.
- Сопоставьте многомерные конформации с X (движение белка вдоль длинной оси ДНК) и углом вращения белка вдоль ДНК для каждого макросостояния, как описано на этапах 1.1.3 и 1.1.4 (например, ни одно состояние со слишком низкой популяцией < 1%; см. Рисунок 2C). Затем найдите 3 макросостояния, которые лучше всего представляют систему (рисунок 1E). Смотрите рисунок 2D для снимков движения белка вдоль ДНК и угла вращения белка вокруг ДНК.
ПРИМЕЧАНИЕ: В предыдущей работе, генерирующей 10 мкс спонтанного шага белка вперед, мы дополнительно провели равновесное моделирование MD 5 x 4 мкс для умеренного расширения выборок. Мы показали отображение исходного прямого пути (см. Рисунок 2А слева) и дальнейших траекторий отбора проб 4 мкс на прямом пути, проведенном ранее (см. Рисунок 2А справа)8. Показано отображение исходных траекторий 100 × 50 нс (см. Рисунок 2B слева)8 и траекторий 97 × 50 нс, используемых в этой работе (см. Рисунок 2B справа).
- Расчет среднего времени первого прохождения (MFPT)
- Проведите пять 10-мс траекторий Монте-Карло (MC) на основе TPM МСМ 500 микросостояний с временем задержки 10 нс, установленным в качестве временного шага MC. Рассчитайте MFPT52 между каждой парой макросостояний (рисунок 3) скриптом python в дополнительном файле 1 python python mfpt_msm3.py.
- Рассчитайте среднюю и стандартную ошибку MFPT с помощью bash-файла в дополнительном файле 2, введите:
sh mfpt_analysis.bash
2. Проведение крупнозернистого (CG) моделирования для выборки долгосрочной динамики
- Проводите компьютерное моделирование с помощью программного обеспечения CafeMol 3.030. Просмотрите параметры моделирования CG, указанные во входном конфигурационном файле с расширением .inp, включая входные структуры, параметры моделирования, выходные файлы и т. Д. Введите следующую команду на терминале, чтобы запустить CG-моделирование:
кафемол XXX.inp - Укажите следующие блоки во входном файле, причем каждый блок начинается с метки < and ending with >>>>.
- Задайте блок имен файлов (обязательно), чтобы указать рабочие каталоги и путь к хранилищу входных/выходных файлов. Введите следующее для блока имен файлов для этих симуляций:
<<<< имена файлов
путь = XXXXX (рабочий путь)
filename = wrky (имена выходных файлов)
ВЫХОД PSF pdb фильм dcd rst
path_pdb = XXXXX (входной путь к собственной структуре)
path_ini = XXXXX (входной начальный путь к структуре)
path_natinfo = XXXXX (путь к собственному информационному файлу)
path_para = XXXXX (путь к файлам параметров)
>>>>
ПРИМЕЧАНИЕ: Поскольку Go-модель53 используется в моделировании CG, т.е. белок будет смещен к нативной конформации, поэтому необходимо установить смоделированную структуру как нативную конформацию. Здесь входная кристаллическая структура была установлена как нативная конформация. - Установите блок управления заданиями (обязательно) для определения режима выполнения симуляций. Введите следующую команду:
<<<< job_cntl
i_run_mode = 2 (= 2 моделирование постоянной температуры)
i_simulate_type = 1 (=1 динамика Ланжевена)
i_initial_state = 2 (=2 означает, что начальная конфигурация является собственной конфигурацией)
>>>>
Выберите постоянную температуру моделирования динамики Ланжевена. - Задайте блок единицы измерения и состояния (обязательно) для определения информации для входных структур. Введите следующую команду:
<<<< unit_and_state
i_seq_read_style = 1 (=1 означает последовательности чтения из PDB-файла)
i_go_native_read_style = 1 (=1 означает, что собственная структура взята из PDB-файла)
1 белок.pdb (единица molecular_type native_structure)
2-3 ДНК ДНК.pdb (единица и состояние molecular_type native_structure)
>>>>
ПРИМЕЧАНИЕ: Необходимы исходные файлы входной структуры (белок.pdb и ДНК.pdb здесь). Структуры написаны в формате pdb. Здесь необходимы два файла pdb: один - файл структуры белка, содержащий координаты тяжелых атомов WRKY (блок 1), а другой - координаты 200-bp двухцепочечной (ds) ДНК (блок 2-3). Белок первоначально помещается на расстоянии 15 Å от ДНК. - Задайте блок функций энергии (обязательный), определенный в блоке energy_function. Введите следующую команду:
<<<< energy_function
МЕСТНЫЙ(1) L_GO
МЕСТНЫЕ(2-3) L_DNA2
NLOCAL(1/1) GO EXV ELE
NLOCAL(2-3/2-3) ELE ДНК
NLOCAL(1/2-3) EXV ELE
i_use_atom_protein = 0
i_use_atom_dna = 0
i_para_from_ninfo = 1
i_triple_angle_term = 2
>>>>
ПРИМЕЧАНИЕ: В моделировании CG белок крупнозернистый по Go-модели53 , причем каждая аминокислота представлена частицей CG, помещенной в положение Cα. Конформация белка будет смещена в сторону нативной структуры, или кристаллической структуры здесь, под потенциалом Go (рисунок 4A слева). ДНК описана моделью54 3SPN.2, в которой каждый нуклеотид представлен 3 частицами CG S, P, N, которые соответствуют сахару, фосфату и азотистому основанию соответственно (рисунок 4A справа). Рассмотрены электростатические и vdW взаимодействия между различными цепями. Электростатические взаимодействия между белком и ДНК в моделировании CG аппроксимируются потенциалом Дебая-Хюккеля55. Энергия отталкивания vdW принимает ту же форму, что и в модели Go. - Установите блок md_information (обязательно) для определения информации моделирования. Введите следующую команду:
<<<< md_information
n_step_sim = 1
n_tstep(1) = 5000000000
tstep_size = 0.1
n_step_save = 1000
n_step_neighbor = 100
i_com_zeroing = 0
i_no_trans_rot = 0
tempk = 300.0
n_seed = -1
>>>>
Самым n_tstep является этап моделирования. Установите tstep_size как продолжительность времени каждого шага MD, каждый шаг времени CG Cafemol составляет около 200 fs30, поэтому каждый шаг MD здесь составляет 200 × 0,1 fs в принципе. Обновляйте список соседей каждые 100 шагов MD (n_step_neighbor = 100). Установите температуру моделирования на 300 К. Контролируйте температуру, используя алгоритм Верле скоростного типа для обновления структуры белка с термостатом Берендсена56.
ПРИМЕЧАНИЕ: N_step_sim является номер бассейна потенциала, основанного на модели Го, или локальное минимальное число кривой энергии. Потенциал нескольких бассейнов позволяет конформации белка смещаться к различным конформациям, так что конформация белка может изменяться от одного локального минимума к другому. Здесь используется только модель Go с одним бассейном, что означает только одну смещенную конформацию (кристаллическую структуру) для белка в моделировании. Между тем, поскольку в контексте CG нет взаимодействия между белком и ДНК водородными связями и т. Д., Молекулярные движения могут быть отобраны еще быстрее, то есть > в 10 раз, чем в атомном моделировании. - Установка электростатического блока (требуется только при использовании электростатического взаимодействия) в качестве электростатического взаимодействия рассматривается между различными цепями, поэтому используйте этот блок для определения параметров электростатического взаимодействия путем типизации:
<<<< электростатический
cutoff_ele = 10.0
ionic_strength = 0.15
>>>>
Установите длину Дебая в электростатическом взаимодействии равным 10 Å, что соответствует условию раствора. Установите ионную силу на 0,15 М, как при физиологическом состоянии.
- Задайте блок имен файлов (обязательно), чтобы указать рабочие каталоги и путь к хранилищу входных/выходных файлов. Введите следующее для блока имен файлов для этих симуляций:
Результаты
Скольжение с вращением или шаг WRKY 1 bp от конструкции MSM
Все конформации белка на ДНК сопоставляются с продольным движением X и углом вращения белка COM вдоль ДНК (см. Рисунок 3A). Линейная связь этих двух степеней указывает на вращательную связь белка домена WRKY на Д...
Обсуждение
В этой работе рассматривается, как проводить структурное вычислительное моделирование и выборку, чтобы выявить транскрипционный фактор или белок TF, движущийся вдоль ДНК, не только при атомной детализации шага, но и при процессивной диффузии, которая необходима для облегченной диффузи...
Раскрытие информации
У авторов нет конфликта интересов.
Благодарности
Эта работа была поддержана грантами NSFC #11775016 и #11635002. JY был поддержан CMCF UCI через NSF DMS 1763272 и грантОм Фонда Саймонса No 594598 и стартовым фондом от UCI. LTD была поддержана Фондом естественных наук Шанхайского #20ZR1425400 и #21JC1403100. Мы также признаем вычислительную поддержку со стороны Пекинского исследовательского центра вычислительной науки (CSRC).
Материалы
| Name | Company | Catalog Number | Comments |
| CafeMol | Kyoto University | coarse-grained (CG) simulations | |
| GROMACS | University of Groningen Royal Institute of Technology Uppsala University | molecular dynamics simulations software | |
| Matlab | MathWorks | Numerical calculation software | |
| MSMbuilder | Stanford University | build MSM | |
| VMD | UNIVERSITY OF ILLINOIS AT URBANA-CHAMPAIGN | molecular visualization program |
Ссылки
- Latchman, D. S. Transcription factors: an overview. The International Journal of Biochemistry & Cell Biology. 29 (12), 1305-1312 (1997).
- Berg, O. G., von Hippel, P. H. Selection of DNA binding sites by regulatory proteins. Statistical-mechanical theory and application to operators and promoters. Journal of Molecular Biology. 193 (4), 723-750 (1987).
- von Hippel, P. H., Berg, O. G. Facilitated target location in biological systems. The Journal of Biological Chemistry. 264 (2), 675-678 (1989).
- Halford, S. E., Marko, J. F. How do site-specific DNA-binding proteins find their targets. Nucleic Acids Research. 32 (10), 3040-3052 (2004).
- Slusky, M., Mirny, L. A. Kinetics of protein-DNA interaction: facilitated target location in sequence-dependent potential. Biophysical Journal. 87 (6), 4021-4035 (2004).
- Bauer, M., Metzler, R. Generalized facilitated diffusion model for DNA-binding proteins with search and recognition states. Biophysical Journal. 102 (10), 2321-2330 (2012).
- Shvets, A. A., Kochugaeva, M. P., Kolomeisky, A. B. Mechanisms of Protein Search for Targets on DNA: Theoretical Insights. Molecules. 23 (9), 2106 (2018).
- Dai, L., Xu, Y., Du, Z., Su, X. D., Yu, J. Revealing atomic-scale molecular diffusion of a plant-transcription factor WRKY domain protein along DNA. Proceedings of the National Academy of Sciences of the United States of America. 118 (23), 2102621118 (2021).
- Chodera, J. D., Singhal, N., Pande, V. S., Dill, K. A., Swope, W. C. Automatic discovery of metastable states for the construction of Markov models of macromolecular conformational dynamics. The Journal of Chemical Physics. 126 (15), 155101 (2007).
- Pan, A. C., Roux, B. Building Markov state models along pathways to determine free energies and rates of transitions. The Journal of Chemical Physics. 129 (6), 064107 (2008).
- Bowman, G. R., Huang, X., Pande, V. S. Using generalized ensemble simulations and Markov state models to identify conformational states. Methods. 49 (2), 197-201 (2009).
- Prinz, J. H., et al. Markov models of molecular kinetics: Generation and validation. The Journal of chemical physics. 134 (17), 174105 (2011).
- Chodera, J. D., Noé, F. Markov state models of biomolecular conformational dynamics. Current Opinion in Structural Biology. 25, 135-144 (2014).
- Malmstrom, R. D., Lee, C. T., Van Wart, A. T., Amaro, R. E. On the Application of Molecular-Dynamics Based Markov State Models to Functional Proteins. Journal of Chemical Theory and Computation. 10 (7), 2648-2657 (2014).
- Husic, B. E., Pande, V. S. Markov State Models: From an Art to a Science. Journal of the American Chemical Society. 140 (7), 2386-2396 (2018).
- Sittel, F., Stock, G. Perspective: Identification of collective variables and metastable states of protein dynamics. The Journal of chemical physics. 149 (15), 150901 (2018).
- Wang, W., Cao, S., Zhu, L., Huang, X. Constructing Markov State Models to elucidate the functional conformational changes of complex biomolecules. WIREs Computational Molecular Science. 8, 1343 (2018).
- Peng, S., et al. Target search and recognition mechanisms of glycosylase AlkD revealed by scanning FRET-FCS and Markov state models. Proceedings of the National Academy of Sciences of the United States of America. 117 (36), 21889-21895 (2020).
- Tian, J., Wang, L., Da, L. T. Atomic resolution of short-range sliding dynamics of thymine DNA glycosylase along DNA minor-groove for lesion recognition. Nucleic Acids Research. 49 (3), 1278-1293 (2021).
- Chu, J. -. W., Izveko, S., Voth, G. The multiscale challenge for biomolecular systems: coarse-grained modeling. Molecular Simulation. 32 (3-4), 211-218 (2006).
- Marrink, S. J., Risselada, H. J., Yefimov, S., Tieleman, D. P., De Vries, A. H. The MARTINI force field: coarse grained model for biomolecular simulations. The Journal of Physical Chemistry B. 111 (27), 7812-7824 (2007).
- Givaty, O., Levy, Y. Protein sliding along DNA: dynamics and structural characterization. Journal of Molecular Biology. 385 (4), 1087-1097 (2009).
- Khazanov, N., Levy, Y. Sliding of p53 along DNA can be modulated by its oligomeric state and by cross-talks between its constituent domains. Journal of Molecular Biology. 408 (2), 335-355 (2011).
- Riniker, S., Allison, J. R., van Gunsteren, W. F. On developing coarse-grained models for biomolecular simulation: a review. Physical Chemistry Chemical Physics : PCCP. 14 (36), 12423-12430 (2012).
- Kmiecik, S., et al. Coarse-Grained Protein Models and Their Applications. Chemical Reviews. 116 (14), 7898-7936 (2006).
- Bhattacherjee, A., Krepel, D., Levy, Y. Coarse-grained models for studying protein diffusion along DNA. WIREs Computational Molecular Science. 6, 515-531 (2016).
- Wang, J., et al. Machine Learning of Coarse-Grained Molecular Dynamics Force Fields. ACS Central Science. 5 (5), 755-767 (2019).
- Joshi, S. Y., Deshmukh, S. A. A review of advancements in coarse-grained molecular dynamics simulations. Molecular Simulation. 47 (10-11), 786-803 (2021).
- Bigman, L. S., Greenblatt, H. M., Levy, Y. What Are the Molecular Requirements for Protein Sliding along DNA. The Journal of Physical Chemistry B. 125 (12), 3119-3131 (2021).
- Kenzaki, H., et al. CafeMol: A Coarse-Grained Biomolecular Simulator for Simulating Proteins at Work. Journal of Chemical Theory and Computation. 7 (6), 1979-1989 (2011).
- Berendsen, H. J. C., vander Spoel, D., van Drunen, R. GROMACS: a message-passing parallel molecular dynamics implementation. Computer Physics Communications. 91 (1-3), 43-56 (1995).
- vander Spoel, D., et al. GROMACS: fast, flexible, and free. Journal of Computational Chemistry. 26 (16), 1701-1718 (2005).
- Abraham, M. J., et al. GROMACS: High performance molecular simulations through multi-level parallelism from laptops to supercomputers. SoftwareX. 1-2, 19-25 (2015).
- Harrigan, M. P., et al. MSMBuilder: Statistical Models for Biomolecular Dynamics. Biophysical journal. 112 (1), 10-15 (2017).
- Humphrey, W., Dalke, A., Schulten, K. VMD: visual molecular dynamics. Journal of Molecular Graphics. 14 (1), 33-38 (1996).
- Izrailev, S., et al. Steered Molecular Dynamics. Computational Molecular Dynamics: Challenges, Methods, Ideas. 4, 39-65 (1999).
- Schlitter, J., Engels, M., Krüger, P. Targeted molecular dynamics: a new approach for searching pathways of conformational transitions. Journal of Molecular Graphics. 12 (2), 84-89 (1994).
- Maragliano, L., Fischer, A., Vanden-Eijnden, E., Ciccotti, G. String method in collective variables: minimum free energy paths and isocommittor surfaces. The Journal of Chemical Physics. 125 (2), 24106 (2006).
- Weiss, D. R., Levitt, M. Can morphing methods predict intermediate structures. Journal of Molecular Biology. 385 (2), 665-674 (2009).
- Xu, Y. P., Xu, H., Wang, B., Su, X. D. Crystal structures of N-terminal WRKY transcription factors and DNA complexes. Protein. 11 (3), 208-213 (2020).
- Higham, D. J., Higham, N. J. MATLAB guide. Society for Industrial and Applied Mathematics. , (2016).
- Hartigan, J. A., Wong, M. A. Algorithm AS 136: A K-Means Clustering Algorithm. Journal of the Royal Statistical Society. Series C (Applied Statistics). 28 (1), 100-108 (1979).
- Gonzalez, T. F. Clustering to minimize the maximum intercluster distance. Theoretical Computer Science. 38, 293-306 (1985).
- Zhao, Y., Sheong, F. K., Sun, J., Sander, P., Huang, X. A fast parallel clustering algorithm for molecular simulation trajectories. Journal of Computational Chemistry. 34 (2), 95-104 (2013).
- Ivani, I., et al. Parmbsc1: a refined force field for DNA simulations. Nature Methods. 13 (1), 55-58 (2016).
- Naritomi, Y., Fuchigami, S. Slow dynamics of a protein backbone in molecular dynamics simulation revealed by time-structure based independent component analysis. The Journal of Chemical Physics. 139 (21), 215102 (2013).
- Naritomi, Y., Fuchigami, S. Slow dynamics in protein fluctuations revealed by time-structure based independent component analysis: the case of domain motions. The Journal of Chemical Physics. 134 (6), 065101 (2011).
- Pérez-Hernández, G., Paul, F., Giorgino, T., De Fabritiis, G., Noé, F. Identification of slow molecular order parameters for Markov model construction. The Journal of Chemical Physics. 139 (1), 015102 (2013).
- McGibbon, R. T., Pande, V. S. Variational cross-validation of slow dynamical modes in molecular kinetics. The Journal of Chemical Physics. 142 (12), 124105 (2015).
- Deuflhard, P., Weber, M. Robust Perron cluster analysis in conformation dynamics. Linear Algebra and its Applications. 398, 161-184 (2005).
- Silva, D. A., et al. Millisecond dynamics of RNA polymerase II translocation at atomic resolution. Proceedings of the National Academy of Sciences of the United States of America. 111 (21), 7665-7670 (2014).
- Swope, W. C., Pitera, J. W., Suits, F. Describing Protein Folding Kinetics by Molecular Dynamics Simulations. 1. Theory. The Journal of Physical Chemistry B. 108 (21), 6571-6581 (2004).
- Clementi, C., Nymeyer, H., Onuchic, J. N. Topological and energetic factors: what determines the structural details of the transition state ensemble and "en-route" intermediates for protein folding? An investigation for small globular proteins. Journal of molecular biology. 298 (5), 937-953 (2000).
- Hinckley, D. M., Freeman, G. S., Whitmer, J. K., De Pablo, J. J. An experimentally-informed coarse-grained 3-Site-Per-Nucleotide model of DNA: structure, thermodynamics, and dynamics of hybridization. The Journal of chemical physics. 139 (14), 144903 (2013).
- Debye, P., Huckel, E. The theory of the electrolyte II-The border law for electrical conductivity. Physikalische Zeitschrift. 24, 305-325 (1923).
- Berendsen, H. J., Postma, J. V., van Gunsteren, W. F., DiNola, A., Haak, J. R. Molecular dynamics with coupling to an external bath. The Journal of Chemical Physics. 81, 3684-3690 (1984).
- Bowman, G. R. Improved coarse-graining of Markov state models via explicit consideration of statistical uncertainty. The Journal of Chemical Physics. 137 (13), 134111 (2012).
- Jain, A., Stock, G. Identifying metastable states of folding proteins. Journal of Chemical Theory and Computation. 8 (10), 3810-3819 (2012).
- Röblitz, S., Weber, M. Fuzzy spectral clustering by PCCA+: application to Markov state models and data classification. Advances in Data Analysis and Classification. 7, 147-179 (2013).
- Mardt, A., Pasquali, L., Wu, H., Noé, F. VAMPnets for deep learning of molecular kinetics. Nature Communications. 9 (1), 5 (2018).
- Wang, W., Liang, T., Sheong, F. K., Fan, X., Huang, X. An efficient Bayesian kinetic lumping algorithm to identify metastable conformational states via Gibbs sampling. The Journal of Chemical Physics. 149 (7), 072337 (2018).
- Chen, W., Sidky, H., Ferguson, A. L. Nonlinear discovery of slow molecular modes using state-free reversible VAMPnets. The Journal of Chemical Physics. 150 (21), 214114 (2019).
- Gu, H., et al. RPnet: a reverse-projection-based neural network for coarse-graining metastable conformational states for protein dynamics. Physical Chemistry Chemical Physics :PCCP. 24 (3), 1462-1474 (2022).
- Lane, T. J., Bowman, G. R., Beauchamp, K., Voelz, V. A., Pande, V. S. Markov state model reveals folding and functional dynamics in ultra-long MD trajectories. Journal of the American Chemical Society. 133 (45), 18413-18419 (2011).
- Konovalov, K. A., Unarta, I. C., Cao, S., Goonetilleke, E. C., Huang, X. Markov State Models to Study the Functional Dynamics of Proteins in the Wake of Machine Learning. JACS Au. 1 (9), 1330-1341 (2021).
- Cao, S., Montoya-Castillo, A., Wang, W., Markland, T. E., Huang, X. On the advantages of exploiting memory in Markov state models for biomolecular dynamics. The Journal of Chemical Physics. 153 (1), 014105 (2020).
- Brandani, G. B., Takada, S. Chromatin remodelers couple inchworm motion with twist-defect formation to slide nucleosomal DNA. PLoS Computational Biology. 14 (11), 1006512 (2018).
- Tan, C., Terakawa, T., Takada, S. Dynamic Coupling among Protein Binding, Sliding, and DNA Bending Revealed by Molecular Dynamics. Journal of the American Chemical Society. 138 (27), 8512-8522 (2016).
- Terakawa, T., Takada, S. p53 dynamics upon response element recognition explored by molecular simulations. Scientific reports. 5, 17107 (2015).
- Brandani, G. B., Niina, T., Tan, C., Takada, S. DNA sliding in nucleosomes via twist defect propagation revealed by molecular simulations. Nucleic Acids Research. 46 (6), 2788-2801 (2018).
- Knotts, T. A., Rathore, N., Schwartz, D. C., de Pablo, J. J. A coarse grain model for DNA. The Journal of Chemical Physics. 126 (8), 084901 (2007).
- Freeman, G. S., Hinckley, D. M., Lequieu, J. P., Whitmer, J. K., de Pablo, J. J. Coarse-grained modeling of DNA curvature. The Journal of Chemical Physics. 141 (16), 165103 (2014).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены