Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Simulation et échantillonnage basés sur la structure des mouvements des protéines du facteur de transcription le long de l’ADN, du passage à l’échelle atomique à la diffusion à grain grossier
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
L’objectif de ce protocole est de révéler la dynamique structurelle de la diffusion unidimensionnelle des protéines le long de l’ADN, en utilisant une protéine du domaine WRKY du facteur de transcription végétale comme système exemplaire. Pour ce faire, des simulations de dynamique moléculaire atomistique et à gros grains ainsi que des échantillonnages informatiques approfondis ont été mis en œuvre.
Résumé
Le glissement unidimensionnel (1-D) de la protéine du facteur de transcription (TF) le long de l’ADN est essentiel pour faciliter la diffusion du TF afin de localiser le site d’ADN cible pour la régulation génétique. La détection de la résolution de la paire de bases (pb) du glissement tf ou du pas sur l’ADN est encore expérimentalement difficile. Nous avons récemment effectué des simulations de dynamique moléculaire (MD) entièrement atomiques capturant un pas spontané de 1 pb d’une petite protéine TF de domaine WRKY le long de l’ADN. Basé sur le chemin de pas WRKY de 10 μs obtenu à partir de telles simulations, le protocole montre ici comment effectuer des échantillonnages conformationnels plus étendus des systèmes TF-ADN, en construisant le modèle d’état de Markov (MSM) pour le pas de protéine 1-bp, avec différents nombres de micro- et macro-états testés pour la construction MSM. Afin d’examiner la recherche par diffusion 1D processive de la protéine TF le long de l’ADN avec la base structurelle, le protocole montre en outre comment mener des simulations MD à grains grossiers (CG) pour échantillonner la dynamique à longue échelle du système. De telles modélisations et simulations CG sont particulièrement utiles pour révéler les impacts électrostatiques protéine-ADN sur les mouvements de diffusion processifs de la protéine TF au-dessus de dizaines de microsecondes, par rapport aux mouvements de pas de protéines submicrosecondes à microsecondes révélés par les simulations de tous les atomes.
Introduction
Les facteurs de transcription (TF) recherchent l’ADN cible pour lier et réguler la transcription des gènes et les activités connexes1. Mis à part la diffusion tridimensionnelle (3D), la diffusion facilitée de TF a été suggérée comme étant essentielle pour la recherche d’ADN cible, dans laquelle les protéines peuvent également glisser ou sauter le long de l’ADN unidimensionnel (1D), ou sauter avec transfert intersegmental sur l’ADN 2,3,4,5,6,7.
Dans une étude récente, nous avons mené des dizaines de microsecondes (μs) de simulations de dynamique moléculaire d’équilibre (MD) d’équilibre tout atome sur une plante TF - la protéine du domaine WRKY sur l’ADN8. Un pas complet de 1 bp de WRKY sur l’ADN poly-A en quelques microsecondes a été capturé. Les mouvements de la protéine le long du sillon de l’ADN et la dynamique de rupture-reformage des liaisons hydrogène (HB) ont été observés. Bien qu’une telle trajectoire représente un chemin échantillonné, un paysage global de pas de protéines fait encore défaut. Ici, nous montrons comment étendre les échantillonnages informatiques autour de la voie d’étape protéique initialement capturée avec le modèle d’état de Markov (MSM) construit, qui ont été largement mis en œuvre pour simuler une variété de systèmes biomoléculaires impliquant des changements conformationnels substantiels et une séparation à l’échelle de temps 9,10,11,12,13,14,15,16, 17,18,19. Le but est de révéler l’ensemble conformationnel et les états méta-stables de la diffusion de la protéine TF le long de l’ADN pour une étape cyclique.
Alors que la simulation MD ci-dessus révèle la résolution atomique des mouvements des protéines pour 1 pb sur l’ADN, la dynamique structurelle de la diffusion processive à long terme du TF le long de l’ADN à la même haute résolution est difficilement accessible. La réalisation de simulations de DM à grains grossiers (CG) au niveau des résidus est toutefois techniquement accessible. L’échelle de temps de simulation CG peut être effectivement étendue à des dizaines ou des centaines de fois plus que les simulations atomiques 20,21,22,23,24,25,26,27,28,29. Ici, nous montrons les simulations CG réalisées en implémentant le logiciel CafeMol développé par Takada lab30.
Dans le protocole actuel, nous présentons d’abord les simulations atomiques de la protéine du domaine WRKY le long de l’ADN poly-A et la construction MSM, qui se concentrent sur l’échantillonnage des mouvements de pas de la protéine pour seulement 1 pb le long de l’ADN. Ensuite, nous présentons la modélisation CG et les simulations du même système protéine-ADN, qui étendent l’échantillonnage informatique à la diffusion processive protéique sur des dizaines de bps le long de l’ADN.
Ici, nous utilisons le logiciel GROMACS31,32,33 pour effectuer des simulations MD et MSMbuilder34 pour construire le MSM pour des instantanés conformationnels échantillonnés, ainsi que pour utiliser VMD35 pour visualiser les biomolécules. Le protocole exige que l’utilisateur puisse installer et implémenter le logiciel ci-dessus. L’installation et la mise en œuvre du logiciel CafeMol30 sont alors nécessaires pour réaliser les simulations CG MD. D’autres analyses des trajectoires et de la visualisation sont également effectuées dans VMD.
Protocole
1. Construction du modèle d’état de Markov (MSM) à partir de simulations md atomiques
- Voie spontanée de pas protéique et collecte des structures initiales
- Utilisez une trajectoire MD8 de 10 μs entièrement atome obtenue précédemment pour extraire 10000 images uniformément d’un chemin de pas « vers l’avant » de 1 bp (c’est-à-dire une trame pour chaque nanoseconde). Le nombre total de trames doit être suffisamment grand pour inclure toutes les conformations représentatives.
- Préparez le chemin de transition avec 10000 images dans VMD en cliquant sur Fichier > Enregistrer les coordonnées, tapez protéine ou nucléique dans la zone Atomes sélectionnés et choisissez des images dans la zone Cadres, cliquez sur Enregistrer pour obtenir les images nécessaires.
REMARQUE: Une trajectoire de simulation MD de 10 μs tout atome précédemment obtenue (appelée « trajectoire de pas en avant » ici) pour wrky marchant à 1 bp sur un ADN poly-A homogène de 34 pb8 a été utilisée comme voie initiale pour lancer d’autres échantillonnages conformationnels. Notez que dans la plupart des pratiques, cependant, un chemin initial est construit, en effectuant des simulations MD pilotées ou ciblées, ou en mettant en œuvre des méthodes générales de génération de chemins, etc. 36,37,38,39. - Alignez l’axe long de l’ADN de référence (de la structure cristalline) sur l’axe des x et définissez le centre de masse initial (COM) de l’ADN complet de 34 pb à l’origine de l’espace de coordonnées pour faciliter une analyse plus approfondie des données. Pour ce faire, cliquez sur Extensions > console Tk dans VMD, puis tapez dans la fenêtre de commande de la console Tk :
source rotate.tcl
Le script tcl se trouve dans le fichier supplémentaire 3. - Calculez ensuite la distance quadratique moyenne (RMSD) de l’épine dorsale protéique en alignant l’ADN central de 10 bp (A 14 à 23 et T 14' à 23') à celui de la structure cristalline40, et le RMSD représente des mesures géométriques des systèmes (voir figure 1A). Pour ce faire, cliquez sur VMD > Extensions > Analysis >'outil de trajectoire RMSD et tapez les noyaux et les résidus 14 à 23 et 46 à 55 dans la zone de sélection d’atomes, cliquez sur Aligner , puis sur RMSD pour calculer les valeurs RMSD.
- Calculer le degré de rotation de la protéine autour de l’ADN Θ(t) sur le plan y-z dans MATLAB en tapant la commande
rad2deg(atan(z/y))
avec le positionnement angulaire initial défini comme Θ(0)=0, comme conduit précédemment8. - Tapez la commande suivante dans MATLAB41 pour utiliser les méthodes K-means 42,43,44 et classez les 10000 structures en 25 clusters en tapant :
[idx, C]=kmeans( X, 25)
ici X est une matrice 2D de RMSD et d’angle de rotation de WRKY sur l’ADN. Rassemblez les structures de ces 25 centres de cluster pour d’autres simulations MD.
REMARQUE: Étant donné que la protéine RMSD échantillonnée par rapport à l’ADN couvre une plage d’environ 25 Å, nous choisissons 25 grappes pour avoir une grappe par angstrom.
- Réalisation dela 1 ère série de simulations MD et des paramètres de simulation
- Construisez des systèmes atomistiques pour les 25 structures en utilisant le logiciel GROMACS 5.1.232 sous le champ de force parmbsc145 et en utilisant le fichier buildsystem.sh du fichier supplémentaire 2 dans le shell.
- Effectuez des simulations MD 60-ns pour ces 25 systèmes sous ensemble NPT avec un pas de temps de 2 fs en tapant la commande suivante dans shell :
gmx_mpi grompp -f md.mdp -c npt.gro -p topol.top -o md.tpr
gmx_mpi mdrun -deffnm md
- Regroupement du 1St trajectoires autour de MD
- Supprimez les 10 premières ns de chaque trajectoire de simulation en tapant shell :
gmx_mpi trjcat -f md.xtc -b 10000 -e 600000 -o newtraj.xtc
et recueillir les conformations des trajectoires de 25 × 50 ns pour le regroupement afin de préparer les structures d’entrée pour les échantillonnages ultérieurs plus étendus (simulations MD 2ème tour).
REMARQUE: Pour réduire l’impact de la trajectoire initiale et permettre l’équilibrage local, 10-ns de la période initiale de simulations ont été supprimés. - Choisissez des paires de distance entre la protéine et l’ADN comme paramètres d’entrée pour la projection de l’analyse en composantes indépendante du temps (tICA)46,47,48. Utilisez la commande make_ndx dans GROMACS pour ce faire :
gmx_mpi make_ndx entrée -f.pdb -o index.ndx
REMARQUE: Ici, les atomes de protéine CA et les atomes lourds (NH1, NH2, OH, NZ, NE2, ND2) du résidu Y119, K122, K125, R131, Y133, Q146, K144, R135, W116, R117, Y134, K118, Q121 qui peuvent former des liaisons hydrogène (HB) avec le nucléotide d’ADN ont été sélectionnés, qui s’associent aux atomes O1P O2P et N6 du nucléotide d’ADN (A14-20, T19-23). Les acides aminés sélectionnés peuvent former des HB stables ou des ponts salins avec l’ADN. - Copiez l’index atom sélectionné ci-dessus à partir du fichier index.ndx dans un nouveau fichier texte (index.dat). Obtenez les informations de paire entre ces atomes par le script python à partir du fichier supplémentaire 1 generate_atom_indices.py et tapez:
Index generate_atom_indices.py python2.6.dat > AtomIndices.txt
Cela génère les 415 paires de distance entre la protéine et l’ADN. - Calculez les 415 paires de distance de chaque trajectoire en tapant la commande suivante dans la fenêtre de commande MSMbuilder :
msmb AtomPairsFeaturizer -out pair_features --pair_indices AtomIndices.txt --top references.pdb --trjs « trajectories/*.xtc » --transformed pair_features --stride 5 - Effectuer tICA pour réduire la dimension des données sur les 2 premiers composants ou vecteurs indépendants du temps (tIC) en tapant :
msmb tICA -i .. /tica_rc_a/tmp/ -o tica_results --n_components 2 --lag_time 10 --gamma 0,05 -t tica_results,h5
REMARQUE: tICA est une méthode de réduction de dimension qui calcule la valeur propre de la matrice de corrélation décalée dans le temps pour déterminer les degrés de liberté de relaxation les plus lents du système de simulation par l’équation:
de corrélation décalée dans le temps pour déterminer les degrés de liberté de relaxation les plus lents du système de simulation par l’équation: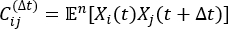
où Xi(t) est la valeur de la i-ième coordonnée de réaction au temps t, et Xj(t+Δt) est la valeur de la j-ième coordonnée de réaction au temps t+Δt.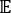 est la valeur attendue du produit des trajectoires de simulation globales Xi(t) et Xj(t + Δt). Les directions le long des degrés de liberté de relaxation les plus lents correspondent aux valeurs propres les plus grandes de la matrice
est la valeur attendue du produit des trajectoires de simulation globales Xi(t) et Xj(t + Δt). Les directions le long des degrés de liberté de relaxation les plus lents correspondent aux valeurs propres les plus grandes de la matrice  de corrélation décalée dans le temps ci-dessus. Ici, 2 tIC semblent être un ensemble minimal pour différencier trois macro-états sur notre construction MSM (abordé plus tard). On peut également calculer le score de quotient de Rayleigh à matrice généralisée (GMRQ)49, par exemple, pour explorer un ensemble optimal de composants à utiliser.
de corrélation décalée dans le temps ci-dessus. Ici, 2 tIC semblent être un ensemble minimal pour différencier trois macro-états sur notre construction MSM (abordé plus tard). On peut également calculer le score de quotient de Rayleigh à matrice généralisée (GMRQ)49, par exemple, pour explorer un ensemble optimal de composants à utiliser. - Utilisez la commande dans MSMbuilder pour regrouper les jeux de données projetés en 100 clusters par la méthode K-center43,44 (voir Figure 1B) :
msmb KCenters -i ./tica_results.h5 -o kcenters_output -t kcenters_output --n_clusters 100.
Sélectionnez la structure centrale de chaque cluster comme structure initiale pour la 2ème série de simulations MD. Conservez les informations de simulation des 100 structures simulées, y compris les positions, les températures, les pressions, etc., à l’exception des vitesses.
REMARQUE: Après la première série de 25 simulations, la mémoire du chemin initial a été réduite, de sorte que nous générons plus de grappes, par exemple 100 grappes, dans la deuxième ronde, pour élargir considérablement les échantillons conformationnels.
- Supprimez les 10 premières ns de chaque trajectoire de simulation en tapant shell :
- Réalisation des simulations approfondies du2 e tour de MD
- Effectuez des simulations MD 60-ns à partir de ces 100 structures initiales après avoir imposé des vitesses initiales aléatoires à tous les atomes. Ajoutez les vitesses initiales aléatoires en activant la génération de vitesse dans le fichier mdp, c’est-à-dire en changeant le fichier md.mdp gen_vel = no en gen_vel = yes.
- Supprimez les 10 premiers ns de chaque simulation comme décrit à l’étape 1.3.1, collectez 2 500 000 instantanés des trajectoires de 100 × 50 ns uniformément pour construire le MSM.
REMARQUE: Notez que dans la construction ultérieure des macro-états, un petit nombre d’états hors trajectoire avec une population particulièrement faible (~ 0,2%, au bas du plan X-Θ) ont été trouvés. Ces états hors chemin sont classés comme un macro-état lorsque le nombre total de macro-états est défini sur 3 à 6 (Figure 2B). Étant donné qu’un macro-état de population aussi faible ne comprend que 3 trajectoires, qui ont été supprimées à la fin, les résultats montrés dans ce protocole ont en effet été obtenus à partir de 97 × 50 ns trajectoires, avec un total de 2 425 000 images ou instantanés.
- Regroupement des 2èmes trajectoires autour de MD
- Effectuer tICA pour les trajectoires du 2e tour comme fait précédemment. Tapez MSMbuilder:
msmb tICA -i .. /tica_rc_a/tmp/ -o tica_results --n_components 2 --lag_time 10 --gamma 0,05 -t tica_results,h5 - Calculer l’échelle de temps implicite pour valider les paramètres du temps de retard de corrélation Δt et les nombres de micro-états (voir Figure 1C),
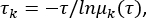
où τ représente le temps de latence utilisé pour construire la matrice de probabilité de transition (TPM); μk(τ) représente la kème valeur propre du TPM sous un temps de décalage de τ. Utilisez le script python du fichier supplémentaire 1 pour ce python BuildMSMsAsVaryLagTime.py -d .. / -f .. /trajlist_num -i 50 -m 1000 -t 10 -n 20 -s 500. - Variez le temps de latence τ et le nombre de micro-états en modifiant les paramètres utilisés ci-dessus :
python BuildMSMsAsVaryLagTime.py -d .. / -f .. /trajlist_num -i 50 -m 1000 -t 5 10 20 30 40 -n 20 -s 20 200 400 500 800 2000
REMARQUE: Le système est considéré comme markovien lorsque les courbes d’échelle de temps implicites commencent à se stabiliser avec la séparation d’échelle de temps. Ensuite, choisissez le Dt comme temps de retard de corrélation, et le τ le temps de décalage où l’échelle de temps implicite commence à se stabiliser pour construire MSM. - En conséquence, choisissez un nombre relativement grand (mais pas trop grand) d’états, N = 500, et un temps de retard de corrélation relativement court Δt = 10 ns. Le temps de latence s’est avéré être τ = 10 ns pour construire MSM.
- Classez les conformations en 500 clusters (voir Figure 1D) à l’aide de la commande :
msmb KCenters -i ./tica_results.h5 -o kcenters_output -t kcenters_output --n_clusters 500
- Effectuer tICA pour les trajectoires du 2e tour comme fait précédemment. Tapez MSMbuilder:
- Construction de VSS
- Regroupez les 500 micro-états en 3 à 6 macro-états pour déterminer le nombre de macro-états qui conviennent le mieux selon l’algorithme PCCA+50 dans MSMbuilder, en utilisant le script python dans le fichier supplémentaire 1 python msm_lumping_usingPCCAplus.py. Identifier un réseau cinétique réduit de modèles pour les changements conformationnels les plus essentiels des biomolécules, en construisant un petit nombre de macro-états, c’est-à-dire en regroupant cinétiquement des centaines de micro-états comme décrit ci-dessous17,51.
- Cartographier les conformations de haute dimension au X (mouvement de la protéine le long de l’axe long de l’ADN) et l’angle de rotation de la protéine le long de l’ADN pour chaque macro-état, comme décrit aux étapes 1.1.3 et 1.1.4 (par exemple, aucun état avec une population trop faible < 1%; voir la figure 2C). Trouvez ensuite les 3 macro-états qui représentent le mieux le système (Figure 1E). Voir la figure 2D pour des instantanés du mouvement des protéines le long de l’ADN et de l’angle de rotation des protéines autour de l’ADN.
REMARQUE: Dans des travaux antérieurs générant la voie de pas en avant de la protéine spontanée de 10 μs, nous avons également effectué des simulations MD d’équilibre de 5 x 4 μs pour élargir modérément les échantillons. Nous avons montré la cartographie de la trajectoire avant d’origine (voir la figure 2A à gauche) et d’autres trajectoires d’échantillonnage de 4 μs sur la trajectoire avant effectuée précédemment (voir la figure 2A à droite)8. La cartographie des trajectoires originales de 100 × 50 ns (voir figure 2B à gauche)8 et des trajectoires de 97 × 50 ns utilisées dans ce travail est montrée (voir la figure 2B à droite).
- Calcul des temps moyens de premier passage (MFPT)
- Effectuez cinq trajectoires Monte Carlo (MC) de 10 ms basées sur le TPM du MSM à 500 micro-états avec le temps de latence de 10 ns défini comme pas de temps de MC. Calculez MFPT52 entre chaque paire de macro-états (Figure 3) par le script python dans le fichier supplémentaire 1 python mfpt_msm3.py.
- Calculez l’erreur moyenne et l’erreur type du MFPT à l’aide du fichier bash dans le fichier supplémentaire 2, tapez :
sh mfpt_analysis.bash
2. Effectuer une simulation à gros grains (CG) pour échantillonner la dynamique à long terme
- Effectuez des simulations CG à l’aide du logiciel CafeMol 3.030. Consultez les paramètres de simulation CG spécifiés dans le fichier de configuration d’entrée avec une extension .inp, y compris les structures d’entrée, les paramètres de simulation, les fichiers de sortie, etc. Tapez la commande suivante sur le terminal pour exécuter la simulation CG :
cafemol XXX.inp - Spécifiez les blocs suivants dans le fichier d’entrée, chaque bloc commençant par l’étiquette < and ending with >>>>.
- Définissez le bloc des noms de fichiers (obligatoire) pour spécifier les répertoires de travail et le chemin d’accès au magasin de fichiers d’entrée/sortie. Tapez ce qui suit pour le bloc de noms de fichiers pour ces simulations :
<<<< noms de fichiers
chemin = XXXXX (chemin d’accès)
filename = wrky (les noms des fichiers de sortie)
SORTIE psf pdb film dcd rst
path_pdb = XXXXX (chemin de structure natif d’entrée)
path_ini = XXXXX (chemin de structure initial d’entrée)
path_natinfo = XXXXX (chemin d’accès au fichier d’informations natif)
path_para = XXXXX (chemin des fichiers de paramètres)
>>>>
REMARQUE: Comme le modèle Go53 est utilisé dans la modélisation CG, c’est-à-dire que la protéine sera biaisée vers la conformation native, il faut donc définir la structure modélisée comme conformation native. Ici, la structure cristalline d’entrée a été définie comme la conformation native. - Définissez le bloc de contrôle de tâche (obligatoire) pour définir le mode d’exécution des simulations. Tapez la commande suivante :
<<<< job_cntl
i_run_mode = 2 (= 2 la simulation de température constante)
i_simulate_type = 1 (=1 dynamique de Langevin)
i_initial_state = 2 (=2 signifie que la configuration initiale est une configuration native)
>>>>
Sélectionnez les simulations de dynamique Langevin à température constante. - Définissez l’unité et le bloc d’état (obligatoire) pour définir les informations pour les structures d’entrée. Tapez la commande suivante :
<<<< unit_and_state
i_seq_read_style = 1 (=1 signifie des séquences de lecture à partir du fichier PDB)
i_go_native_read_style = 1 (=1 signifie que la structure native provient du fichier PDB)
1 protéine .pdb (unité et état molecular_type native_structure)
2-3 ADN ADN.pdb (unité et état molecular_type native_structure)
>>>>
REMARQUE: Les fichiers de structure d’entrée initiaux (protéine.pdb et ADN.pdb ici) sont nécessaires. Les structures sont écrites au format pdb. Deux fichiers pdb sont nécessaires ici: l’un est le fichier de structure protéique contenant les coordonnées des atomes lourds de WRKY (unité 1), et l’autre est les coordonnées de l’ADN double brin (ds) de 200 bp (unité 2-3). La protéine est initialement placée à 15 Å de l’ADN. - Définissez le bloc de fonction d’énergie (obligatoire) défini dans le bloc energy_function. Tapez la commande suivante :
<<<< energy_function
LOCAL(1) L_GO
LOCAL(2-3) L_DNA2
NLOCAL(1/1) ALLER EXV ELE
ADN NLOCAL(2-3/2-3) ELE
NLOCAL(1/2-3) EXV ELE
i_use_atom_protein = 0
i_use_atom_dna = 0
i_para_from_ninfo = 1
i_triple_angle_term = 2
>>>>
REMARQUE: Dans les simulations CG, la protéine est grossièrement grainée par le modèle Go53 avec chaque acide aminé représenté par une particule CG placée à sa position Cα. La conformation protéique sera alors biaisée vers la structure native, ou structure cristalline ici, sous le potentiel Go (Figure 4A à gauche). L’ADN est décrit par le modèle 3SPN.254, dans lequel chaque nucléotide est représenté par 3 particules CG S, P, N, qui correspondent respectivement au sucre, au phosphate et à la base azotée (Figure 4A à droite). Les interactions électrostatiques et vdW sont considérées entre différentes chaînes. Les interactions électrostatiques entre la protéine et l’ADN dans la simulation CG sont approchées par le potentiel de Debye-Hückel55. L’énergie répulsive vdW prend la même forme que dans le modèle Go. - Définissez le bloc md_information (obligatoire) pour définir les informations de simulation. Tapez la commande suivante :
<<<< md_information
n_step_sim = 1
n_tstep(1) = 500000000
tstep_size = 0,1
n_step_save = 1000
n_step_neighbor = 100
i_com_zeroing = 0
i_no_trans_rot = 0
tempk = 300,0
n_seed = -1
>>>>
La n_tstep est l’étape de simulation. Définissez la tstep_size comme la durée de chaque étape MD, chaque pas de temps CG Cafemol est d’environ 200 fs30, donc chaque étape MD ici est de 200 × 0,1 fs en principe. Mettez à jour la liste des voisins toutes les 100 étapes MD (n_step_neighbor = 100). Réglez la température de simulation sur 300 K. Contrôlez la température en utilisant l’algorithme Verlet de type vitesse pour mettre à jour la structure des protéines avec le thermostat Berendsen56.
REMARQUE : Le n_step_sim est le numéro de bassin du potentiel basé sur le modèle Go, ou le nombre minimal local de la courbe d’énergie. Un potentiel à plusieurs bassins permet à la conformation des protéines d’être biaisée en différentes conformations, de sorte que la conformation des protéines peut passer d’un minimum local à un autre. Ici, seul le modèle Go à bassin unique est utilisé, ce qui signifie une seule conformation biaisée (structure cristalline) pour les protéines dans les simulations. Pendant ce temps, comme il n’y a pas d’interaction de liaison hydrogène protéine-ADN, etc. modélisée dans le contexte CG, les mouvements moléculaires peuvent être échantillonnés encore plus rapidement, c’est-à-dire > 10 fois que dans les simulations atomiques. - Définissez le bloc électrostatique (requis uniquement lorsque l’interaction électrostatique est utilisée) car l’interaction électrostatique est considérée entre différentes chaînes, utilisez donc ce bloc pour définir les paramètres de l’interaction électrostatique en tapant:
<<<< électrostatique
cutoff_ele = 10,0
ionic_strength = 0,15
>>>>
Réglez la longueur de Debye dans l’interaction électrostatique à 10 Å, correspondant à l’état de la solution. Réglez la force ionique à 0,15 M, comme à l’état physiologique.
- Définissez le bloc des noms de fichiers (obligatoire) pour spécifier les répertoires de travail et le chemin d’accès au magasin de fichiers d’entrée/sortie. Tapez ce qui suit pour le bloc de noms de fichiers pour ces simulations :
Résultats
Glissement couplé à la rotation ou pas de 1 bp de WRKY de la construction MSM
Toutes les conformations protéiques sur l’ADN sont mappées au mouvement longitudinal X et à l’angle de rotation de la protéine COM le long de l’ADN (voir la figure 3A). Le couplage linéaire de ces deux degrés indique un pas couplé à la rotation de la protéine du domaine WRKY sur l’ADN. Les conformations peuvent être regroupées en 3 macro-états (S1, S2 et S3) dans le MSM. Le...
Discussion
Ce travail porte sur la façon de mener des simulations et des échantillonnages informatiques basés sur la structure pour révéler un facteur de transcription ou une protéine TF se déplaçant le long de l’ADN, non seulement au détail atomique du pas, mais aussi dans la diffusion processive, ce qui est essentiel pour la diffusion facilitée de TF dans la recherche de cible d’ADN. Pour ce faire, le modèle d’état de Markov ou MSM d’une petite protéine du domaine TF WRKY marchant pour 1-bp le long de l’ADN...
Déclarations de divulgation
Les auteurs n’ont pas de conflit d’intérêts.
Remerciements
Ce travail a été soutenu par les subventions #11775016 et #11635002 de la NSFC. JY a été soutenu par le CMCF de l’UCI via NSF DMS 1763272 et la subvention de la Fondation Simons #594598 et fonds de démarrage de l’UCI. LTD a été soutenu par la Fondation des sciences naturelles de Shanghai #20ZR1425400 et #21JC1403100. Nous reconnaissons également le soutien informatique du Beijing Computational Science Research Center (CSRC).
matériels
| Name | Company | Catalog Number | Comments |
| CafeMol | Kyoto University | coarse-grained (CG) simulations | |
| GROMACS | University of Groningen Royal Institute of Technology Uppsala University | molecular dynamics simulations software | |
| Matlab | MathWorks | Numerical calculation software | |
| MSMbuilder | Stanford University | build MSM | |
| VMD | UNIVERSITY OF ILLINOIS AT URBANA-CHAMPAIGN | molecular visualization program |
Références
- Latchman, D. S. Transcription factors: an overview. The International Journal of Biochemistry & Cell Biology. 29 (12), 1305-1312 (1997).
- Berg, O. G., von Hippel, P. H. Selection of DNA binding sites by regulatory proteins. Statistical-mechanical theory and application to operators and promoters. Journal of Molecular Biology. 193 (4), 723-750 (1987).
- von Hippel, P. H., Berg, O. G. Facilitated target location in biological systems. The Journal of Biological Chemistry. 264 (2), 675-678 (1989).
- Halford, S. E., Marko, J. F. How do site-specific DNA-binding proteins find their targets. Nucleic Acids Research. 32 (10), 3040-3052 (2004).
- Slusky, M., Mirny, L. A. Kinetics of protein-DNA interaction: facilitated target location in sequence-dependent potential. Biophysical Journal. 87 (6), 4021-4035 (2004).
- Bauer, M., Metzler, R. Generalized facilitated diffusion model for DNA-binding proteins with search and recognition states. Biophysical Journal. 102 (10), 2321-2330 (2012).
- Shvets, A. A., Kochugaeva, M. P., Kolomeisky, A. B. Mechanisms of Protein Search for Targets on DNA: Theoretical Insights. Molecules. 23 (9), 2106 (2018).
- Dai, L., Xu, Y., Du, Z., Su, X. D., Yu, J. Revealing atomic-scale molecular diffusion of a plant-transcription factor WRKY domain protein along DNA. Proceedings of the National Academy of Sciences of the United States of America. 118 (23), 2102621118 (2021).
- Chodera, J. D., Singhal, N., Pande, V. S., Dill, K. A., Swope, W. C. Automatic discovery of metastable states for the construction of Markov models of macromolecular conformational dynamics. The Journal of Chemical Physics. 126 (15), 155101 (2007).
- Pan, A. C., Roux, B. Building Markov state models along pathways to determine free energies and rates of transitions. The Journal of Chemical Physics. 129 (6), 064107 (2008).
- Bowman, G. R., Huang, X., Pande, V. S. Using generalized ensemble simulations and Markov state models to identify conformational states. Methods. 49 (2), 197-201 (2009).
- Prinz, J. H., et al. Markov models of molecular kinetics: Generation and validation. The Journal of chemical physics. 134 (17), 174105 (2011).
- Chodera, J. D., Noé, F. Markov state models of biomolecular conformational dynamics. Current Opinion in Structural Biology. 25, 135-144 (2014).
- Malmstrom, R. D., Lee, C. T., Van Wart, A. T., Amaro, R. E. On the Application of Molecular-Dynamics Based Markov State Models to Functional Proteins. Journal of Chemical Theory and Computation. 10 (7), 2648-2657 (2014).
- Husic, B. E., Pande, V. S. Markov State Models: From an Art to a Science. Journal of the American Chemical Society. 140 (7), 2386-2396 (2018).
- Sittel, F., Stock, G. Perspective: Identification of collective variables and metastable states of protein dynamics. The Journal of chemical physics. 149 (15), 150901 (2018).
- Wang, W., Cao, S., Zhu, L., Huang, X. Constructing Markov State Models to elucidate the functional conformational changes of complex biomolecules. WIREs Computational Molecular Science. 8, 1343 (2018).
- Peng, S., et al. Target search and recognition mechanisms of glycosylase AlkD revealed by scanning FRET-FCS and Markov state models. Proceedings of the National Academy of Sciences of the United States of America. 117 (36), 21889-21895 (2020).
- Tian, J., Wang, L., Da, L. T. Atomic resolution of short-range sliding dynamics of thymine DNA glycosylase along DNA minor-groove for lesion recognition. Nucleic Acids Research. 49 (3), 1278-1293 (2021).
- Chu, J. -. W., Izveko, S., Voth, G. The multiscale challenge for biomolecular systems: coarse-grained modeling. Molecular Simulation. 32 (3-4), 211-218 (2006).
- Marrink, S. J., Risselada, H. J., Yefimov, S., Tieleman, D. P., De Vries, A. H. The MARTINI force field: coarse grained model for biomolecular simulations. The Journal of Physical Chemistry B. 111 (27), 7812-7824 (2007).
- Givaty, O., Levy, Y. Protein sliding along DNA: dynamics and structural characterization. Journal of Molecular Biology. 385 (4), 1087-1097 (2009).
- Khazanov, N., Levy, Y. Sliding of p53 along DNA can be modulated by its oligomeric state and by cross-talks between its constituent domains. Journal of Molecular Biology. 408 (2), 335-355 (2011).
- Riniker, S., Allison, J. R., van Gunsteren, W. F. On developing coarse-grained models for biomolecular simulation: a review. Physical Chemistry Chemical Physics : PCCP. 14 (36), 12423-12430 (2012).
- Kmiecik, S., et al. Coarse-Grained Protein Models and Their Applications. Chemical Reviews. 116 (14), 7898-7936 (2006).
- Bhattacherjee, A., Krepel, D., Levy, Y. Coarse-grained models for studying protein diffusion along DNA. WIREs Computational Molecular Science. 6, 515-531 (2016).
- Wang, J., et al. Machine Learning of Coarse-Grained Molecular Dynamics Force Fields. ACS Central Science. 5 (5), 755-767 (2019).
- Joshi, S. Y., Deshmukh, S. A. A review of advancements in coarse-grained molecular dynamics simulations. Molecular Simulation. 47 (10-11), 786-803 (2021).
- Bigman, L. S., Greenblatt, H. M., Levy, Y. What Are the Molecular Requirements for Protein Sliding along DNA. The Journal of Physical Chemistry B. 125 (12), 3119-3131 (2021).
- Kenzaki, H., et al. CafeMol: A Coarse-Grained Biomolecular Simulator for Simulating Proteins at Work. Journal of Chemical Theory and Computation. 7 (6), 1979-1989 (2011).
- Berendsen, H. J. C., vander Spoel, D., van Drunen, R. GROMACS: a message-passing parallel molecular dynamics implementation. Computer Physics Communications. 91 (1-3), 43-56 (1995).
- vander Spoel, D., et al. GROMACS: fast, flexible, and free. Journal of Computational Chemistry. 26 (16), 1701-1718 (2005).
- Abraham, M. J., et al. GROMACS: High performance molecular simulations through multi-level parallelism from laptops to supercomputers. SoftwareX. 1-2, 19-25 (2015).
- Harrigan, M. P., et al. MSMBuilder: Statistical Models for Biomolecular Dynamics. Biophysical journal. 112 (1), 10-15 (2017).
- Humphrey, W., Dalke, A., Schulten, K. VMD: visual molecular dynamics. Journal of Molecular Graphics. 14 (1), 33-38 (1996).
- Izrailev, S., et al. Steered Molecular Dynamics. Computational Molecular Dynamics: Challenges, Methods, Ideas. 4, 39-65 (1999).
- Schlitter, J., Engels, M., Krüger, P. Targeted molecular dynamics: a new approach for searching pathways of conformational transitions. Journal of Molecular Graphics. 12 (2), 84-89 (1994).
- Maragliano, L., Fischer, A., Vanden-Eijnden, E., Ciccotti, G. String method in collective variables: minimum free energy paths and isocommittor surfaces. The Journal of Chemical Physics. 125 (2), 24106 (2006).
- Weiss, D. R., Levitt, M. Can morphing methods predict intermediate structures. Journal of Molecular Biology. 385 (2), 665-674 (2009).
- Xu, Y. P., Xu, H., Wang, B., Su, X. D. Crystal structures of N-terminal WRKY transcription factors and DNA complexes. Protein. 11 (3), 208-213 (2020).
- Higham, D. J., Higham, N. J. MATLAB guide. Society for Industrial and Applied Mathematics. , (2016).
- Hartigan, J. A., Wong, M. A. Algorithm AS 136: A K-Means Clustering Algorithm. Journal of the Royal Statistical Society. Series C (Applied Statistics). 28 (1), 100-108 (1979).
- Gonzalez, T. F. Clustering to minimize the maximum intercluster distance. Theoretical Computer Science. 38, 293-306 (1985).
- Zhao, Y., Sheong, F. K., Sun, J., Sander, P., Huang, X. A fast parallel clustering algorithm for molecular simulation trajectories. Journal of Computational Chemistry. 34 (2), 95-104 (2013).
- Ivani, I., et al. Parmbsc1: a refined force field for DNA simulations. Nature Methods. 13 (1), 55-58 (2016).
- Naritomi, Y., Fuchigami, S. Slow dynamics of a protein backbone in molecular dynamics simulation revealed by time-structure based independent component analysis. The Journal of Chemical Physics. 139 (21), 215102 (2013).
- Naritomi, Y., Fuchigami, S. Slow dynamics in protein fluctuations revealed by time-structure based independent component analysis: the case of domain motions. The Journal of Chemical Physics. 134 (6), 065101 (2011).
- Pérez-Hernández, G., Paul, F., Giorgino, T., De Fabritiis, G., Noé, F. Identification of slow molecular order parameters for Markov model construction. The Journal of Chemical Physics. 139 (1), 015102 (2013).
- McGibbon, R. T., Pande, V. S. Variational cross-validation of slow dynamical modes in molecular kinetics. The Journal of Chemical Physics. 142 (12), 124105 (2015).
- Deuflhard, P., Weber, M. Robust Perron cluster analysis in conformation dynamics. Linear Algebra and its Applications. 398, 161-184 (2005).
- Silva, D. A., et al. Millisecond dynamics of RNA polymerase II translocation at atomic resolution. Proceedings of the National Academy of Sciences of the United States of America. 111 (21), 7665-7670 (2014).
- Swope, W. C., Pitera, J. W., Suits, F. Describing Protein Folding Kinetics by Molecular Dynamics Simulations. 1. Theory. The Journal of Physical Chemistry B. 108 (21), 6571-6581 (2004).
- Clementi, C., Nymeyer, H., Onuchic, J. N. Topological and energetic factors: what determines the structural details of the transition state ensemble and "en-route" intermediates for protein folding? An investigation for small globular proteins. Journal of molecular biology. 298 (5), 937-953 (2000).
- Hinckley, D. M., Freeman, G. S., Whitmer, J. K., De Pablo, J. J. An experimentally-informed coarse-grained 3-Site-Per-Nucleotide model of DNA: structure, thermodynamics, and dynamics of hybridization. The Journal of chemical physics. 139 (14), 144903 (2013).
- Debye, P., Huckel, E. The theory of the electrolyte II-The border law for electrical conductivity. Physikalische Zeitschrift. 24, 305-325 (1923).
- Berendsen, H. J., Postma, J. V., van Gunsteren, W. F., DiNola, A., Haak, J. R. Molecular dynamics with coupling to an external bath. The Journal of Chemical Physics. 81, 3684-3690 (1984).
- Bowman, G. R. Improved coarse-graining of Markov state models via explicit consideration of statistical uncertainty. The Journal of Chemical Physics. 137 (13), 134111 (2012).
- Jain, A., Stock, G. Identifying metastable states of folding proteins. Journal of Chemical Theory and Computation. 8 (10), 3810-3819 (2012).
- Röblitz, S., Weber, M. Fuzzy spectral clustering by PCCA+: application to Markov state models and data classification. Advances in Data Analysis and Classification. 7, 147-179 (2013).
- Mardt, A., Pasquali, L., Wu, H., Noé, F. VAMPnets for deep learning of molecular kinetics. Nature Communications. 9 (1), 5 (2018).
- Wang, W., Liang, T., Sheong, F. K., Fan, X., Huang, X. An efficient Bayesian kinetic lumping algorithm to identify metastable conformational states via Gibbs sampling. The Journal of Chemical Physics. 149 (7), 072337 (2018).
- Chen, W., Sidky, H., Ferguson, A. L. Nonlinear discovery of slow molecular modes using state-free reversible VAMPnets. The Journal of Chemical Physics. 150 (21), 214114 (2019).
- Gu, H., et al. RPnet: a reverse-projection-based neural network for coarse-graining metastable conformational states for protein dynamics. Physical Chemistry Chemical Physics :PCCP. 24 (3), 1462-1474 (2022).
- Lane, T. J., Bowman, G. R., Beauchamp, K., Voelz, V. A., Pande, V. S. Markov state model reveals folding and functional dynamics in ultra-long MD trajectories. Journal of the American Chemical Society. 133 (45), 18413-18419 (2011).
- Konovalov, K. A., Unarta, I. C., Cao, S., Goonetilleke, E. C., Huang, X. Markov State Models to Study the Functional Dynamics of Proteins in the Wake of Machine Learning. JACS Au. 1 (9), 1330-1341 (2021).
- Cao, S., Montoya-Castillo, A., Wang, W., Markland, T. E., Huang, X. On the advantages of exploiting memory in Markov state models for biomolecular dynamics. The Journal of Chemical Physics. 153 (1), 014105 (2020).
- Brandani, G. B., Takada, S. Chromatin remodelers couple inchworm motion with twist-defect formation to slide nucleosomal DNA. PLoS Computational Biology. 14 (11), 1006512 (2018).
- Tan, C., Terakawa, T., Takada, S. Dynamic Coupling among Protein Binding, Sliding, and DNA Bending Revealed by Molecular Dynamics. Journal of the American Chemical Society. 138 (27), 8512-8522 (2016).
- Terakawa, T., Takada, S. p53 dynamics upon response element recognition explored by molecular simulations. Scientific reports. 5, 17107 (2015).
- Brandani, G. B., Niina, T., Tan, C., Takada, S. DNA sliding in nucleosomes via twist defect propagation revealed by molecular simulations. Nucleic Acids Research. 46 (6), 2788-2801 (2018).
- Knotts, T. A., Rathore, N., Schwartz, D. C., de Pablo, J. J. A coarse grain model for DNA. The Journal of Chemical Physics. 126 (8), 084901 (2007).
- Freeman, G. S., Hinckley, D. M., Lequieu, J. P., Whitmer, J. K., de Pablo, J. J. Coarse-grained modeling of DNA curvature. The Journal of Chemical Physics. 141 (16), 165103 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon