A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
חקר האינטראקציה הביומולקולרית בין המלווה המולקולרי Hsp90 לבין חלבון הלקוח שלו Kinase Cdc37 באמצעות טכנולוגיית ביוסנסינג בעלת אפקט שדה
In This Article
Summary
ביוסנסינג של אפקט שדה (FEB) היא טכניקה נטולת תוויות לאיתור אינטראקציות ביומולקולריות. הוא מודד את הזרם החשמלי דרך הביו-סנסור של הגרפן שאליו משותקות מטרות הקשירה. טכנולוגיית FEB שימשה להערכת אינטראקציות ביומולקולריות בין Hsp90 ל-Cdc37 וזוהתה אינטראקציה חזקה בין שני החלבונים.
Abstract
אינטראקציות ביומולקולריות ממלאות תפקידים רב-תכליתיים בתהליכים תאיים רבים על-ידי ויסות ותיאום של אירועים ביולוגיים רלוונטיים מבחינה תפקודית. ביו-מולקולות כגון חלבונים, פחמימות, ויטמינים, חומצות שומן, חומצות גרעין ואנזימים הם אבני בניין בסיסיות של יצורים חיים; הם מתכנסים לרשתות מורכבות במערכות ביולוגיות כדי לסנכרן מספר עצום של אירועי חיים. חלבונים בדרך כלל משתמשים ברשתות אינטראקציה מורכבות כדי לבצע את תפקידיהם; לפיכך חובה להעריך אינטראקציות כאלה כדי לפענח את חשיבותן בתאים הן ברמת התא והן ברמת האורגניזם. לקראת מטרה זו, אנו מציגים טכנולוגיה המתפתחת במהירות, ביוסנסינג בעל אפקט שדה (FEB), כדי לקבוע אינטראקציות ביומולקולריות ספציפיות. FEB היא טכניקת זיהוי ביומולקולרית, נטולת תוויות ואמינה לקביעת אינטראקציות ספציפיות ומשתמשת בביו-סנסורים מבוססי אלקטרוניקה באיכות גבוהה. טכנולוגיית FEB יכולה לנטר אינטראקציות בטווח הננו-מולארי בשל הננו-חומרים הניתנים להתאמה ביולוגית המשמשים על פני השטח של הביו-סנסור שלה. כהוכחת היתכנות, הוברה האינטראקציה בין חלבון-חלבון (PPI) בין חלבון הלם חום 90 (Hsp90) לבין מחזור חלוקת התא 37 (Cdc37). Hsp90 הוא מלווה מולקולרי תלוי ATP הממלא תפקיד חיוני בקיפול, יציבות, התבגרות ובקרת איכות של חלבונים רבים, ובכך מווסת פונקציות תאיות חיוניות מרובות. Cdc37 נחשב למלווה מולקולרי ספציפי לחלבון קינאז, מכיוון שהוא מזהה ומגייס באופן ספציפי קינאזות חלבונים ל-Hsp90 כדי לווסת את מסלולי התמרת האותות במורד הזרם. ככזה, Cdc37 נחשב למלווה משותף של Hsp90. מסלול המלווה-קינאז (קומפלקס Hsp90/Cdc37) מופעל יתר על המידה בממאירות מרובה המקדמת את צמיחת התאים; לכן, זהו יעד פוטנציאלי לטיפול בסרטן. המחקר הנוכחי מדגים את היעילות של טכנולוגיית FEB באמצעות מערכת המודל Hsp90/Cdc37. FEB זיהה PPI חזק בין שני החלבונים (ערכי KD של 0.014 μM, 0.053 μM ו-0.072 μM בשלושה ניסויים בלתי תלויים). לסיכום, FEB היא פלטפורמת זיהוי PPI ללא תווית וחסכונית, המציעה מדידות מהירות ומדויקות.
Introduction
אינטראקציות ביומולקולריות:
חלבונים הם חלקים חיוניים של אורגניזמים ומשתתפים במסלולים מולקולריים רבים כגון חילוף חומרים של תאים, מבנה התא, איתות תאים, תגובות חיסוניות, הידבקות תאים ועוד. בעוד שחלבונים מסוימים מבצעים את תפקידם באופן עצמאי, רוב החלבונים מקיימים אינטראקציה עם חלבונים אחרים באמצעות ממשק מקשר כדי לתאם פעילות ביולוגית תקינה1.
ניתן לסווג אינטראקציות ביומולקולריות בעיקר על סמך המאפיינים המבניים והתפקודיים המובהקים של חלבונים המעורבים2, למשל, בהתבסס על משטחי החלבון, היציבות המורכבת או ההתמדה של אינטראקציות3. זיהוי חלבונים חיוניים ותפקידם באינטראקציות ביומולקולריות הוא חיוני להבנת מנגנונים ביוכימיים ברמה המולקולרית4. נכון לעכשיו, ישנן גישות שונות לזיהוי אינטראקציות אלה5: in vitro6, בסיליקו7, בתאים חיים8, ex vivo9, ו in vivo10 כאשר לכל אחד מהם יש חוזקות וחולשות משלו.
מבחני in vivo מבוצעים תוך שימוש בחיה כולה ככלי ניסיוני11, ומבחניה- ex vivo מבוצעים על תמציות רקמה או על איברים שלמים (למשל, לב, מוח, כבד) בסביבה חיצונית מבוקרת על ידי מתן שינויים מינימליים בתנאים טבעיים. היישום הנפוץ ביותר של מחקרי in vivo ו - ex vivo הוא להעריך את הפרמקוקינטיקה, הפרמקודינמיקה והשפעות הרעילות של חומרים פרמקולוגיים פוטנציאליים לפני ניסויים בבני אדם על ידי הבטחת הבטיחות והיעילות הכוללות שלהם12.
ניתן לזהות אינטראקציות ביומולקולריות גם בתוך תאים חיים. הדמיית תאים חיים מאפשרת לנו לצפות באינטראקציות דינמיות בזמן שהם מבצעים את התגובות של מסלול ביוכימי מסוים13. יתר על כן, טכניקות זיהוי, כגון ביולומינסנציה או העברת אנרגיית תהודה פלואורסצנטית, יכולות לספק מידע על היכן ומתי אינטראקציות אלה מתרחשות בתוך תא14. אף על פי שזיהוי בתאים חיים מציע פרטים חיוניים, מתודולוגיות זיהוי אלה מסתמכות על אופטיקה ותוויות, שייתכן שאינן משקפות את הביולוגיה המקורית; הם גם פחות מבוקרים משיטות במבחנה ודורשים מומחיות מיוחדת כדי לבצע15.
השיטות החישוביות של in silico משמשות בעיקר לסינון בקנה מידה גדול של מולקולות מטרה לפני הניסויים במבחנה . שיטות חיזוי חישוביות, מסדי נתונים מבוססי מחשב, עגינה מולקולרית, יחסי מבנה-פעילות כמותיים וגישות סימולציה אחרות של דינמיקה מולקולרית הם בין המבוססים היטב בכלי סיליקו 16. בהשוואה לטכניקות ניסיוניות מייגעות, הכלים בסיליקו יכולים בקלות לבצע תחזיות עם רגישות גבוהה, אך עם דיוק מופחת בביצועי החיזוי17.
בדיקות במבחנה מבוצעות עם מיקרואורגניזמים או מולקולות ביולוגיות מחוץ להקשר הביולוגי הסטנדרטי שלהם. הצגת אינטראקציות ביומולקולריות באמצעות שיטות במבחנה היא קריטית להבנת תפקודי החלבון והביולוגיה שמאחורי הרשת המורכבת של תפקוד התאים. מתודולוגיית הבדיקה המועדפת נבחרת על פי התכונות הפנימיות של החלבון, הערכים הקינטיים והאופן והעוצמה של האינטראקציות18,19.
האינטראקציה Hsp90/Cdc37:
מסלול המלווה-קינאז, המחבר בין Hsp90 ל-Cdc37, הוא מטרה טיפולית מבטיחה בביולוגיה של הגידול20. Hsp90 ממלא תפקיד מרכזי בבקרת מחזור התא, בהרכבת חלבונים, בהישרדות התאים ובמסלולי איתות. חלבונים המסתמכים על Hsp90 לצורך תפקודם מועברים ל-Hsp90 לצורך קומפלקסציה באמצעות מלווה משותף, כגון Cdc37. קומפלקס Hsp90/Cdc37 שולט בקיפול של רוב החלבון קינאזות ומשמש כמרכז לשלל רשתות איתותתוך-תאיות 21. זוהי מטרה אנטי-סרטנית מבטיחה בשל הביטוי המוגבה שלה בממאירות שונות, כולל לוקמיה מיאלובלסטית חריפה, מיאלומה נפוצה וקרצינומה הפטוצלולרית22,23.
טכניקות נפוצות לזיהוי אינטראקציה ביומולקולרית במבחנה
Co-immunoprecipitation (co-IP) היא טכניקה המסתמכת על ספציפיות אנטיגן-נוגדנים כדי לזהות אינטראקציות רלוונטיות ביולוגית24. החיסרון העיקרי של שיטה זו הוא חוסר היכולת שלה לזהות אינטראקציות בעלות זיקה נמוכה וערכים קינטיים24. שיטות ביופיזיות כגון קלורימטריה איזותרמית של טיטרציה (ITC), תהודת פלסמון פני השטח (SPR), אינטרפרומטריה ביו-שכבתית (BLI) וטכנולוגיית FEB עדיפות לקביעת הערכים הקינטיים.
ITC היא שיטת זיהוי ביו-פיזיקלית המבוססת על קביעת אנרגיית קשירת יחד עם ניתוח תרמודינמיקה מלא לאפיון אינטראקציות ביומולקולריות25. היתרון העיקרי של ITC הוא שהוא אינו דורש כל תיוג או קיבוע של חלבון המטרה. הקשיים העיקריים שבהם נתקל ITC הם הריכוז הגבוה של חלבון המטרה הנדרש לניסוי אחד והקושי לנתח קומפלקסים לא קוולנטיים עקב אנתלפיות קישור קטנות26. גם SPR וגם BLI הן טכניקות ביופיזיות נטולות תוויות המסתמכות על אימוביליזציה של מולקולת המטרה על משטח החיישנים, ולאחר מכן זריקות של האנליט מעל המטרה המשותקת27,28. ב- SPR, שינויים במקדם השבירה במהלך אינטראקציות ביומולקולריות נמדדים27; ב-BLI, ההתאבכות באור המוחזר נרשמת בזמן אמת כשינוי באורך הגל כפונקציה של הזמן28. גם SPR וגם BLI חולקים יתרונות משותפים של הצעת יכולות ספציפיות, רגישות וזיהוי גבוהות29. בשתי השיטות, חלבון המטרה משותק על משטחי ביוסנסור, ולכן, ייתכן שיש אובדן מסוים של הקונפורמציה הטבעית של המטרה, מה שמקשה על הבחנה בין אינטראקציות ספציפיות לעומת לא ספציפיות30. BLI משתמשת בביו-סנסורים חד-פעמיים חד-פעמיים יקרים כדי לשתק את המטרה, ולכן היא טכניקה יקרה31. בהשוואה לכלי זיהוי ביומולקולריים מבוססים אלה, טכנולוגיית FEB מציעה פלטפורמה אמינה ונטולת תוויות על ידי שימוש בריכוזי ננומולארים נמוכים לזיהוי ביומולקולרי בזמן אמת עם אפיון קינטי. טכנולוגיית FEB גם מתגברת על האתגרים המבעבעים העומדים בפני ITC והיא חסכונית יותר בהשוואה ל- SPR או BLI.
ביוסנסורים מבוססי טרנזיסטור בעל אפקט שדה (FET) הוא תחום מתפתח לאיתור אינטראקציות ביומולקולריות על ידי הצעת יישומים ביו-רפואיים מגוונים. במערכת FET, מטרות משותקות לשבבי הביו-סנסור ואינטראקציות מזוהות על ידי שינויים בהולכה32. התכונה הייחודית שיש לקחת בחשבון בפיתוח ביוסנסור אלקטרוני יעיל היא התכונות הפיזיקוכימיות כגון האופי המוליך למחצה והיציבות הכימית של חומר הציפוי המשמש לייצור משטח החיישן33. חומרים קונבנציונליים כמו סיליקון המשמש ל-FET הגבילו את הרגישות של חיישנים משום שהם דורשים שכבות תחמוצת דחוקות בין תעלת הטרנזיסטור לבין סביבה מסוימת לתפקוד תקיןשל 34. יתר על כן, טרנזיסטורים של סיליקון רגישים לסביבות מלח גבוהות, ובכך מקשים על מדידת אינטראקציות ביולוגיות בסביבתם הטבעית. הביוסנסור מבוסס הגרפן מוצג כחלופה מכיוון שהוא מציע יציבות כימית מעולה ושדה חשמלי. מאחר שגרפן הוא שכבה אטומית יחידה של פחמן, הוא גם רגיש ביותר כמוליכה למחצה וגם תואם כימית לתמיסות ביולוגיות; שתי התכונות הללו רצויות כדי ליצור ביוסנסורים אלקטרוניים תואמים35. פוטנציאל ההעמסה האולטרה-גבוה המדהים של ביו-מולקולות המוצע על ידי ביוסנסורים מצופים גרפן מוביל לפיתוח טכנולוגיית FEB מבוססת-גרפן.
עקרון טכנולוגיית FEB: FEB היא טכניקת זיהוי ביומולקולרית נטולת תוויות המודדת את הזרם החשמלי דרך הביו-סנסור של הגרפן שאליו משותקות מטרות הקישור. אינטראקציות בין החלבון המשותק לבין האנליט גורמות לשינויים בזרם המנוטרים בזמן אמת, ומאפשרים מדידות קינטיות מדויקות36.
מכשור: מערכת FEB כוללת שבב חיישן טרנזיסטור בעל אפקט שדה גרפן (gFET) וקורא אלקטרוני שמפעיל מתח קבוע לאורך כל הניסוי (איור 1). האנליט מיושם בתמיסה על חלבון המטרה המשותק על פני השטח של הביו-סנסור. כאשר מתרחשת אינטראקציה, שינוי בזרם נמדד ומתועד בזמן אמת. ככל שריכוז האנליטים עולה, גם החלק של האנליט המאוגד יגדל, מה שיגרום לחילופים גבוהים יותר בזרם. באמצעות תוכנת הניתוח האוטומטית המסופקת עם המכשיר (טבלת החומרים), I-Response נמדדת ומתועדת במונחים של יחידות ביוסנסינג (BU)37. I-Response מוגדר כשינוי בזרם (I) באמצעות שבב הביו-סנסור הנמדד בזמן אמת על סמך האינטראקציה של המטרה המשותקת עם האנליט. תוכנת הניתוח האוטומטית FEB יכולה לנתח הן את ה-I-Response והן את ה-C-Response לאירועי אינטראקציה דינמיים, כאשר ה-C-Response מתעד את השינויים בקיבוליות (C). הווריאציות הן ב-I-Response והן ב-C-Response מתאימות ישירות לשבריר של האנליט המאוגד וניתן לנתח אותן עוד יותר כדי ליצור ערכי KD . העדפת ברירת המחדל של תוכנת הניתוח האוטומטית היא I-Response.
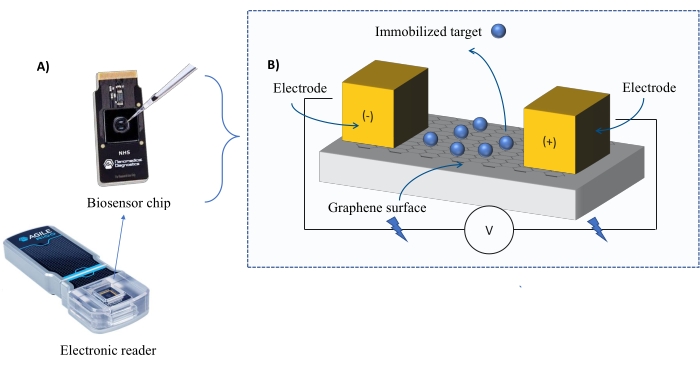
איור 1: סקירה כללית של מערך הניסוי. (B) סקירה כללית של רכיבי השבב. השבב מחובר לשתי אלקטרודות המספקות זרם למערכת. פני השטח של השבב מכוסים בגרפן, שכאשר הוא מופעל יכול לקשור את המטרה. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
מתודולוגיה:
בתחילה, שבב הביוסנסור המופעל מוכנס למכשיר FEB (איור 1) ולאחר מכן ביצוע השלבים המתוארים להלן: (1) כיול: (1) כיול: הניסוי מתחיל בכיול המערכת באמצעות 1x מי מלח עם מאגר פוספט (PBS; pH = 7.4) כדי ליצור את תגובת שיווי המשקל הבסיסית. (2) שיוך: האנליט מוצג בשבב, וה-I-Response מנוטר עד להגשמת רוויית הקשירה. (3) דיסוציאציה: האנליט מנותק באמצעות 1x PBS. (4) התחדשות: שרידי האנליט מוסרים באמצעות 1x PBS. (5) כביסה: בסך הכל חמש שטיפות מבוצעות באמצעות 1x PBS להסרה יסודית של האנליטים הכבולים והלא מאוגדים מהשבב.
ניתוח:
ניתוח הנתונים מתבצע באמצעות התוכנה האוטומטית לחלוטין המסופקת עם המכשיר. תוכנת הניתוח האוטומטית יוצרת עלילת היל פיט עם ערך KD . עלילת ההתאמה של היל מתארת את הקשר של אנליט לחלבון המטרה כפונקציה של ריכוזי אנליטים. הריכוז שבו מושגת תגובה חצי מקסימלית הוא פרופורציונלי לערך KD . ערך KD נמוך מייצג זיקת קשירה גבוהה ולהיפך.
כדי לאמת את הנתונים המתקבלים מניסוי FEB, תגובות I מופקות מכל נקודת קריאה עבור כל ריכוז אנאליטים באמצעות תוכנת סקירת/ייצוא הנתונים וניתן לייצא אותן לתוכנות ניתוח סטטיסטיות אחרות (ראה טבלת חומרים) כפי שיוסבר להלן.
Access restricted. Please log in or start a trial to view this content.
Protocol
הערה: החלבונים הרקומביננטיים ששימשו במחקר זה, Hsp90 ו-Cdc37, התקבלו באופן מסחרי (ראו טבלת חומרים).
1. הפעלת שבב
הערה: כל החומרים שיש להשתמש בהם בניסוי מפורטים בטבלת החומרים. סנן את כל הפתרונות המוכנים באמצעות מסנן סטרילי של 0.2 מיקרומטר.
- הכן תמיסת קרבודימיד (EDC) של 1-אתיל-3-(3-דימתילאמינו) על-ידי הוספת 2 מ"ג של EDC ל-2.5 מ"ל של 1 M 2-(N-morpholino) חומצה אתאנית סולפונית (MES) (pH = 6.0) בצינור של 15 מ"ל. הכן תמיסות N-Hydroxysulfosuccinimide (sulfo-NHS) על ידי הוספת 6 מ"ג של סולפו-NHS ל-2.5 מ"ל של 1 M MES buffer (pH = 6.0) בצינור נפרד של 15 מ"ל. Aliquot 50 μL של כל תמיסה בצינורות עצמאיים ואחסנו בטמפרטורה של -20 מעלות צלזיוס לשימוש עתידי.
- מערבבים נפחים שווים של תמיסת EDC sand sulfo-NHS (50 μL של EDC + 50 μL של sulfo-NHS) על ידי צנרת למעלה ולמטה (אין מערבולת).
הערה: יש להשתמש בתמיסה המעורבת של EDC/sulfo-NHS תוך 30 דקות כדי לשמור על קישור צולב יעיל לתפקוד נכון של השבבים. - מניחים את שבב הביו-סנסור (5.7 ס"מ x 2.4 ס"מ; ראו טבלת חומרים) המסופק על ידי החברה בצלחת פטרי מזכוכית עם מכסה מותאם. כל שלבי הפונקציונליזציה הכרוכים בהפעלת שבבים מוצעים להיעשות בתוך צלחת פטרי. החל 50 μL של 1 M MES buffer (pH = 6.0) על שבב ביוסנסור, דגירה למשך דקה אחת בטמפרטורת החדר, ולאחר מכן שאף את המאגר.
- החל 50 μL של פתרון EDC/sulfo-NHS באופן מיידי על שבב החיישן. כסו את צלחת הפטרי ודגרו במשך 15 דקות בטמפרטורת החדר.שאפו לתמיסת EDC/sulfo-NHS מהשבב.
- לשטוף את השבב פעם אחת עם 50 μL של 1 M MES buffer (pH = 6.0); שאפו את מאגר ה-MES.
2. מיקוד אימוביליזציה של חלבונים
- יש לשטוף את השבב 2x עם 50 μL של 1x PBS (pH = 7.4). שאפו את ה-PBS מהשבב והוסיפו את מולקולת המטרה, Hsp90 (50 μL; 500 ננומטר).
הערה: אי-התאמה של מאגר עלולה להרוס את הניסוי כולו; לפיכך, לפני הניסוי, חשוב לוודא כי מולקולת המטרה נמצאת באותו מאגר כמו זה המשמש לכיול (למשל, 1x PBS (pH = 7.4)). במידת הצורך, בצע חילופי חיץ על ידי דיאליזה לילית לפני הניסוי. בניסוי זה, דיאליזה לילית בוצעה עבור שני החלבונים הרקומביננטיים, Hsp90 ו- Cdc37, כנגד 1x PBS (pH = 7.4; ראה טבלת חומרים) עם חילופי חיץ מתאימים ב- 4 °C . ריכוז חומר המטרה (Hsp90 במקרה זה) עשוי להשתנות בהתאם לפרוטוקולים ניסיוניים שונים ולאופיים של חומרי המטרה (חלבון/פפטיד/ליגנדות). - מכסים את צלחת הפטרי מזכוכית ודוגרים למשך 30 דקות בטמפרטורת החדר. שאפו את התמיסה המכילה את מולקולת המטרה ושטפו פי 3 ב-50 מיקרול'ל של 1x PBS (pH = 7.4). שאפו את תמיסת 1x PBS (pH = 7.4) מהשבב.
- הוסיפו 50 μL של תמיסת Quench 1 (3.9 mM amino-PEG5-alcohol ב-1x PBS (pH = 7.4)) לתמיסת ה-Chip. כסו את צלחת הפטרי מזכוכית ודגרו למשך 15 דקות בטמפרטורת החדר. שאפו לפתרון Quench 1 מהשבב.
- הוסף 50 μL של תמיסת Quench 2 (1 M אתנולמין (pH = 8.5)) לשבב. מכסים את צלחת הפטרי הזכוכית ודגרו במשך 15 דקות בטמפרטורת החדר. שאפו לתמיסת Quench 2 מהשבב ושטפו את השבב 5x באמצעות 50 μL של 1x PBS, והשאירו את טיפת ה-PBS האחרונה על החיישן.
3. הכנת דוגמאות אנליטה
- הכן סדרת דילול אנאליטים עבור Cdc37 בטווח הריכוזים הרצוי. עבור הניסוי הראשון נעשה שימוש בריכוזים הבאים: 25 ננומטר, 50 ננומטר, 100 ננומטר, 200 ננומטר, 400 ננומטר, 800 ננומטר, 1,000 ננומטר, 2,000 ננומטר, 3,000 ננומטר ו-5,000 ננומטר. עבור הניסוי השני, נעשה שימוש בקבוצה שונה של ריכוזים הנעים בין 0.4 ננומטר ל-200 ננומטר.
- תכנן שהניסוי יכלול לפחות שמונה ריכוזי אנליטים שונים כדי לקבל ערך KD אמין. הכינו את הדילולים השונים של חלבון האנליט באותו מאגר כמו זה המשמש לכיול וחלבון מטרה; הנה זה 1x PBS (pH = 7.4).
4. טעינת שבב הביוסנסור המופעל להתקן FEB
הערה: התקן FEB מורכב מקורא הכולל חיוויים של תאורת LED ומחסנית להכנסת שבב הביוסנסור.
- לאחר אימוביליזציה של חלבון מטרה, הכנס את השבב המופעל למחסנית של המכשיר, המחובר באמצעות USB למחשב. לאחר החדרת השבב, תוצג בקורא נורית LED ירוקה המציינת שהתקן ה-FEB מוכן לניסוי. התקן את התוכנה האוטומטית (ראה טבלת חומרים) המסופקת על ידי החברה במחשב, שאליו מחובר מכשיר ה- FEB, כדי לנטר את הניסוי צעד אחר צעד כמתואר להלן.
5. הפעל את הניסוי
- לחץ על מודול הפעל ניסוי בתוכנה האוטומטית ובחר 10 נקודות עם התחדשות או כל פרוטוקול רצוי אחר. מלא את הפרטים הבאים: שם המפעיל, שם הניסוי, תאריך (לדוגמה, יאנה, Hsp90 + Cdc37, 14.03.2021); מאגר התחדשות (למשל, מאגר PBS); מטרה משותקת (למשל, Hsp90); analyte in solution (למשל, Cdc37). ראה איור משלים S1 לקבלת פרטים.
- לחץ על לחצן התחל את הניסוי המוצג בתוכנה ופעל לפי ההוראות המוצגות על-ידי התוכנה האוטומטית כמתואר להלן.
הערה: התוכנה אוטומטית לחלוטין, ידידותית למשתמש ומנחה את המשתמש לאורך הניסוי צעד אחר צעד. חלון קופץ יופיע על המסך עם הוראות להמשיך הלאה בכל שלב בניסוי. התוכנה תספק הוראות לכל שלב שחוזר על עצמו ברצף החל מכיול, אסוציאציה אנליטית, דיסוציאציה, התחדשות ושטיפה (5x) עבור כל ריכוז אנליט לאורך הניסוי. - בצע כיול מכשירים. לשם כך, שאפו את פתרון ה-PBS הנותר מהשבב והחלו 50 μL של מאגר כיול (1x PBS; pH = 7.4). לחץ על כפתור המשך והמתן 5 דקות עד ששלב הכיול יסתיים. התוכנה מציגה את נקודת הקצה שנקבעה עבור שלב הכיול (5 דקות) עם אזעקת אזהרה למעקב.
- לאחר מכן, בצע אסוציאציה אנאליטית. לשם כך, שאפו את מאגר הכיול מהשבב והחלו 50 μL של ריכוז האנאליטים הנמוך ביותר (25 ננומטר של Cdc37). לחץ על המשך כפתור והמתן 5 דקות עד לסיום שלב האסוציאציה. התוכנה מציגה את נקודת הקצה של שלב השיוך (5 דקות) עם אזעקת אזהרה להמשך.
- בצע דיסוציאציה אנאליטית. לשם כך, שאפו את תמיסת האנליט מהשבב והחלו 50 μL של מאגר הדיסוציאציה (1x PBS; pH = 7.4). לחץ על כפתור המשך והמתן 5 דקות עד לסיום משך שלב הדיסוציאציה (5 דקות). התוכנה מציגה את נקודת הקצה לשלב הדיסוציאציה (5 דקות) עם אזעקת אזהרה למעקב.
- לאחר מכן, בצע התחדשות שבבים. שאפו את תמיסת הדיסוציאציה מהשבב והחלו 50 μL של מאגר התחדשות (1x PBS; pH = 7.4). לחץ על לחצן המשך והמתן 30 שניות עד לסיום שלב ההתחדשות (30 שניות). התוכנה מציגה את נקודת הקצה לשלב ההתחדשות (30 שניות) עם אזעקת אזהרה למעקב.
- לבסוף, לשטוף את השבב. שאפו את תמיסת ההתחדשות מהשבב והחלו 50 μL של מאגר שטיפה (1x PBS; pH = 7.4) על השבב. לשאוף את הפתרון מן השבב ולחזור על זה 5x. השאירו את הטיפה האחרונה של מאגר הכביסה על השבב ולחצו על כפתור המשך והמתינו 30 שניות עד שמשך שלב הכביסה יסתיים בתצוגת התוכנה.
הערה: התוכנה מציגה את נקודת הקצה של שלב הכביסה (30 שניות) עם אזעקת אזהרה כדי להמשיך במחזור הבא של הניסוי. - חזור על השלבים עבור כל ריכוז אנאליט שבו נעשה שימוש; חמשת השלבים של כיול, אסוציאציה אנליטית, דיסוציאציה, התחדשות ושטיפה (5x) מהווים מחזור אחד. עבור הניסוי שהוצג כאן, ביצענו 10 מחזורים עבור 10 ריכוזי אנאליטים (שנעו בין 25 ננומטר ל-5,000 ננומטר או 0.4 ננומטר עד 200 ננומטר; איור 2).
6. ניתוח
- לחץ על לחצן ניתוח שנראה בחלק העליון של תוכנת הניתוח האוטומטית בסוף הניסוי. יופיע חלון תצוגה המכיל את כל נקודות הניסוי. בחלון, ודא שריכוזי האנליטים המשמשים לפרוטוקול שנקבע נכונים.
- לחץ על לחצן הפעל ניתוח כדי ליצור את ערך KD באופן אוטומטי. התוכנה יוצרת עלילת היל fit על ידי התוויית ריכוזי האנאליטים כנגד תגובות ה-I המתאימות שמהן מחושב קבוע הדיסוציאציה בשיווי משקל, ערך KD .
- ייצא את הנתונים הגולמיים לניתוח בתוכנות ניתוח סטטיסטיות אחרות באמצעות תוכנת סקירת/ייצוא הנתונים כמתואר להלן.
- העתק את קובץ R1R שנוצר באופן אוטומטי בסוף הניסוי (לדוגמה, Hsp90 + Cdc37 14.03.2021) לתיקיה חדשה בשולחן העבודה. פתח את תוכנת סקירת/ייצוא הנתונים שסופקה על-ידי החברה (ראה איור משלים S2A).
- לחץ על עיבוד נתונים > עיבוד קבצי R1R > אישור במסך הבית של תוכנת סקירת /ייצוא הנתונים.
- בחר את התיקיה המכילה קבצי R1R שנוצרו בשולחן העבודה בשלב 6.3.1. ולחץ על כפתור אישור . פעולה זו יוצרת עותק של נתוני R1R המקוריים, כדי לסקור ולערוך את הנתונים מבלי לעקוף את הקובץ המקורי.
- לחץ על סמל עיבוד הנתונים המוצג במסך הבית של תוכנת סקירת/ייצוא הנתונים. לחץ על טען קבצי R1R מעובדים > אישור. בחר את אותה תיקיה שנוצרה בשלב 6.3.1 המכילה קבצי R1R מעובדים. לחץ על אישור. בשלב זה, התיקיה המכילה את קבצי הניסוי מוכנה לבדיקה.
- במסך הבית של תוכנת סקירת/ייצוא נתונים, לחץ על ניתוח נתונים. בחר כיול > כיול קבצי R1R ערוכים > אישור (ראה איור משלים S2B). שלב זה מכייל את כל נקודות הנתונים בהתאם לשלב הכיול הראשון, כדי ליצור קו בסיס.
- במסך הבית של תוכנת סקירת/ייצוא נתונים, לחץ על ניתוח נתונים. בחר סקור וערוך קבצי R1R > אישור. סקור את נקודות הנתונים, מחק נקודות או הוסף שלבים באמצעות לחצן מחק/הוסף שלב . ודא שכל השלבים נמצאים במקום הנכון, לדוגמה, שלב הכיול נמצא בקו הבסיס, שלב השיוך נמצא בשיא (ראה איור משלים S3) ולאחר מכן לחץ על לחצן שמור כדי לשמור את כל השינויים שבוצעו.
- במסך הבית של תוכנת סקירת/ייצוא נתונים, לחץ על ניתוח נתונים. בחר נתח/תרשים נתונים, בחר את השלבים לייצוא (ראה איור משלים S4). הוסף כל שלב שיוך (לדוגמה, שלב 2, שלב 7 וכן הלאה) לאחר חיסור שלב הכיול (לדוגמה, שלב 1, שלב 6 וכן הלאה) לרשימת הייצוא לפני ייצוא הנתונים.
- לחץ על ייצוא נתונים אלה. התוכנה מייצרת קובץ גיליון אלקטרוני המכיל את ה- I-Response עבור כל נקודת נתונים של ריכוז אנאליט מכל טרנזיסטור (אם כל הטרנזיסטורים פועלים, יהיו לנו שלושה ערכי I-response שונים עבור כל נקודת ריכוז). השתמש בקובץ גיליון אלקטרוני זה כדי לנתח עוד יותר את הנתונים בתוכנה הסטטיסטית.
- פתח את תוכנת הניתוח הסטטיסטי. צור טבלת XY עם שלושה ערכי Y; הקש צור. הנתונים עבור טבלה זו (ערכי X, Y) מועתקים מקובץ הגיליון האלקטרוני שנוצר בשלב 6.3.8. ציר ה-x מתאים לריכוז האנליט (בהתאם לריכוז שבו נעשה שימוש), ושלושת ערכי ה-Y תואמים את ה-I-Responses, שהתקבלו מתוכנת סקירת/ייצוא הנתונים שסופקה על ידי החברה.
- לחץ על נתח נתונים אלה. בחר ניתוחי XY > רגרסיה לא ליניארית (התאמת עקומה) > רוויית קשירה > אתר אחד > סה"כ. התוכנה תנתח את הנתונים באופן אוטומטי, תיצור את הערך KD ותיצור גרף של נקודות הנתונים.
Access restricted. Please log in or start a trial to view this content.
תוצאות
תוצאות מניסוי 1:
חלבון המטרה Hsp90 (500 ננומטר) היה משותק לשבב בעקבות פרוטוקול אימוביליזציה של המטרה כפי שתואר לעיל. עבור הניסוי הראשון הוכנו 10 ריכוזים של חלבון האנאליטים, Cdc37, שנעו בין 25 ננומטר ל-5,000 ננומטר, בהתבסס על הנתונים הזמינים בספרות (ראו טבלה 1).
ניתן לעק...
Access restricted. Please log in or start a trial to view this content.
Discussion
במחקר זה הוערכה ההיתכנות של שימוש בטכנולוגיית FEB (גישת אפיון קינטי בזמן אמת) כדי לקבוע את האינטראקציה הביומולקולרית בין Hsp90 ל-Cdc37. ניסוי החקר הראשוני (הניסוי הראשון) הציע כי בחירת ריכוזי האנליטים הנכונים היא חלק קריטי בניסוי וכי יש לתכנן את הניסוי על ידי הכללת נקודות ריכוז מעל ומתחת לערך K...
Access restricted. Please log in or start a trial to view this content.
Disclosures
המחברים מצהירים על היעדר ניגודי עניינים, כספיים או אחרים.
Acknowledgements
מחקר זה נתמך על ידי מענק מהקרן הדו-לאומית למדע (BSF) ל- S.K.S. ו- N.Q.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| Automated analysis software | Agile plus software, Cardea (Nanomed) | NA CAS number: NA | Referred to in the text as the automated analysis software supplied with the instrument. Generates automated analysis. |
| COOH-BPU (Biosensing Processing Unit) | Agile plus software, Cardea (Nanomed) | NA CAS number: NA | biosensor chip |
| Data review software | Datalign 1.0, Cardea (Nanomed) | NA CAS number: NA | Referred to as the supplied data review software in the text. Supplied with the instrument and allows to review and export the information data points. |
| Dialysis bag | CelluSep, Membrane filtration products | T2-10-15 CAS number: NA | T2 tubings (6,000-8,000 MWCO), (10 mm fw, 6.4mm Ø, 0.32ml/cm, 15m) |
| EDC (1-Ethyl-3-(3-dimethylamino propyl) carbodiimide) | Cardea (Nanomed) | EDC160322-02 CAS number: 25952-53-8 | White powder |
| ITC (Isothermal titration calorimetry) system | Microcal-PEAQ-ITC (Malvern, United Kingdom) | NA CAS number: NA | |
| MES (2-(N-morpholino) ethane sulfonic acid) buffer | Merck | M3671-50G CAS number: 4432-31-9 | White powder |
| NHS (N-Hydroxysulfosuccinimide) chips | Cardea (Nanomed) | NA CAS number: NA | Graphene-based chip |
| PBS (Phosphate-buffered saline) X 10 | Bio-Lab | 001623237500 CAS number: 7758-11-4 | Liquid transparent solution |
| Pipete | Thermo Scientific | 11855231 CAS number: NA | Finnpipette F3 5-50 µL, yellow |
| Quench 1 (3.9 mM amino-PEG5-alcohol in 1 X PBS) | Cardea (Nanomed) | 0105-001-002-001 CAS number: NA | Liquid, transparent solution |
| Quench 2 (1 M ethanolamine (pH=8.5)) | Cardea (Nanomed) | 0105-001-003-001 CAS number: NA | Liquid, transparent solution |
| Recombinant protein Cdc37 | Abcam | ab256157 CAS number: NA | |
| Recombinant protein Hsp90 beta | Abcam | ab80033 CAS number: NA | |
| Spreadsheet | Excel, Microsoft office | NA CAS number: NA | |
| Statistical software | GraphPad, Prism | NA CAS number: NA | Referred to as the other statistical software. Sigma plot, phyton or other statistical programes may also be used |
| Sulfo-NHS | Cardea (Nanomed) | NHS160321-07 CAS number: 106627-54-7 | White powder |
| Tips | Alex red | LC 1093-800-000 CAS number: NA | Tip 1-200 µl, in bulk, 1,000 pcs |
References
- Tuncbag, N., Gursoy, A., Guney, E., Nussinov, R., Keskin, O. Architectures and functional coverage of protein-protein interfaces. Journal of Molecular Biology. 381 (3), 785-802 (2008).
- Berggård, T., Linse, S., James, P. Methods for the detection and analysis of protein–protein interactions. Proteomics. 7 (16), 2833-2842 (2007).
- Magliery, T. J., et al. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: Scope and mechanism. Journal of the American Chemical Society. 127 (1), 146-157 (2005).
- Xing, S., Wallmeroth, N., Berendzen, K. W., Grefen, C. Techniques for the analysis of protein-protein interactions in vivo. Plant Physiology. 171 (2), 727-758 (2016).
- Nguyen, T. N., Goodrich, J. A. Protein-protein interaction assays: Eliminating false positive interactions. Nature Methods. 3 (2), 135-139 (2006).
- Fernández-Suárez, M., Chen, T. S., Ting, A. Y. Protein-protein interaction detection in vitro and in cells by proximity biotinylation. Journal of the American Chemical Society. 130 (29), 9251-9253 (2008).
- Jiang, M., Niu, C., Cao, J., Ni, D. -A., Chu, Z. In silico-prediction of protein–protein interactions network about MAPKs and PP2Cs reveals a novel docking site variants in Brachypodium distachyon. Scientific Reports. 8 (1), 15083(2018).
- Yazawa, M., Sadaghiani, A. M., Hsueh, B., Dolmetsch, R. E. Induction of protein-protein interactions in live cells using light. Nature Biotechnology. 27 (10), 941-945 (2009).
- Wang, W., Goodman, M. T. Antioxidant property of dietary phenolic agents in a human LDL-oxidation ex vivo model: Interaction of protein binding activity. Nutrition Research. 19 (2), 191-202 (1999).
- Xing, S., Wallmeroth, N., Berendzen, K. W., Grefen, C. Techniques for the analysis of protein-protein interactions in vivo. Plant Physiology. 171 (2), 727-758 (2016).
- Qvit, N., Disatnik, M. -H., Sho, E., Mochly-Rosen, D. Selective phosphorylation inhibitor of delta protein kinase C–Pyruvate dehydrogenase kinase protein–protein interactions: Application for myocardial injury in vivo. Journal of the American Chemical Society. 138 (24), 7626-7635 (2016).
- Alam, M. N., Bristi, N. J., Rafiquzzaman, M. Review on in vivo and in vitro methods evaluation of antioxidant activity. Saudi Pharmaceutical Journal. 21 (2), 143-152 (2013).
- Paulmurugan, R., Gambhir, S. S. Novel fusion protein approach for efficient high-throughput screening of small molecule–mediating protein-protein interactions in cells and living animals. Cancer Research. 65 (16), 7413-7420 (2005).
- Boute, N., Jockers, R., Issad, T. The use of resonance energy transfer in high-throughput screening: BRET versus FRET. Trends in Pharmacological Sciences. 23 (8), 351-354 (2002).
- Deriziotis, P., Graham, S. A., Estruch, S. B., Fisher, S. E. Investigating protein-protein interactions in live cells using bioluminescence resonance energy transfer. Journal of Visualized Experiments: JoVE. (87), e51438(2014).
- Ekins, S., Mestres, J., Testa, B. In silico pharmacology for drug discovery: Methods for virtual ligand screening and profiling. British Journal of Pharmacology. 152 (1), 9-20 (2007).
- Valerio, L. G. Application of advanced in silico methods for predictive modeling and information integration. Expert Opinion on Drug Metabolism & Toxicology. 8 (4), 395-398 (2012).
- Piehler, J. New methodologies for measuring protein interactions in vivo and in vitro. Current Opinion in Structural Biology. 15 (1), 4-14 (2005).
- Ideker, T., Sharan, R. Protein networks in disease. Genome Research. 18 (4), 644-652 (2008).
- Lu, H., et al. Recent advances in the development of protein–protein interactions modulators: mechanisms and clinical trials. Signal Transduction and Targeted Therapy. 5 (1), 213(2020).
- Jarosz, D. Hsp90: A global regulator of the genotype-to-phenotype map in cancers. Advances in Cancer Research. 129, 225-247 (2016).
- Johnson, V. A., Singh, E. K., Nazarova, L. A., Alexander, L. D., McAlpine, S. R. Macrocyclic inhibitors of Hsp90. Current Topics in Medicinal Chemistry. 10 (14), 1380-1402 (2010).
- Mahalingam, D., et al. Targeting HSP90 for cancer therapy. British Journal of Cancer. 100 (10), 1523-1529 (2009).
- Stewart, A., Fisher, R. A. Co-Immunoprecipitation: Isolation of protein signaling complexes from native tissues. Methods in Cell Biology. 112, 33-54 (2012).
- Pierce, M. M., Raman, C. S., Nall, B. T. Isothermal titration calorimetry of protein-protein interactions. Methods. 19 (2), San Diego, Calif. 213-221 (1999).
- Paketurytė, V., et al. Inhibitor binding to carbonic anhydrases by isothermal titration calorimetry. Carbonic Anhydrase as Drug Target. , Springer International Publishing. 79-95 (2019).
- Grote, J., Dankbar, N., Gedig, E., Koenig, S. Surface plasmon resonance/mass spectrometry interface. Analytical Chemistry. 77 (4), 1157-1162 (2005).
- Kumaraswamy, S., Tobias, R. Label-free kinetic analysis of an antibody–antigen interaction using biolayer interferometry. Methods in Molecular Biology. , 165-182 (2015).
- Wallner, J., Lhota, G., Jeschek, D., Mader, A., Vorauer-Uhl, K. Application of bio-layer interferometry for the analysis of protein/liposome interactions. Journal of Pharmaceutical and Biomedical Analysis. 72, 150-154 (2013).
- Singh, A. N., Ramadan, K., Singh, S. Experimental methods to study the kinetics of protein–protein interactions. Advances in Protein Molecular and Structural Biology Methods. , 115-124 (2022).
- Frenzel, D., Willbold, D. Kinetic titration series with biolayer interferometry. PLoS One. 9 (9), 106882(2014).
- Vu, C. -A., Chen, W. -Y. Field-effect transistor biosensors for biomedical applications: Recent advances and future prospects. Sensors. 19 (19), 4214(2019).
- Bergveld, P. A critical evaluation of direct electrical protein detection methods. Biosensors & Bioelectronics. 6 (1), 55-72 (1991).
- Lowe, B. M., Sun, K., Zeimpekis, I., Skylaris, C. K., Green, N. G. Field-effect sensors – from pH sensing to biosensing: sensitivity enhancement using streptavidin–biotin as a model system. The Analyst. 142 (22), 4173-4200 (2017).
- Goldsmith, B. R., et al. Digital biosensing by foundry-fabricated graphene sensors. Scientific Reports. 9 (1), 434(2019).
- Afsahi, S., et al. Novel graphene-based biosensor for early detection of Zika virus infection. Biosensors and Bioelectronics. 100, 85-88 (2018).
- Afsahi, S. J., et al. Towards novel graphene-enabled diagnostic assays with improved signal-to-noise ratio. MRS Advances. 2 (60), 3733-3739 (2017).
- Roe, S. M., et al. The mechanism of Hsp90 regulation by the protein kinase-specific cochaperone p50cdc37. Cell. 116 (1), 87-98 (2004).
- Gaiser, A. M., Kretzschmar, A., Richter, K. Cdc37-Hsp90 complexes are responsive to nucleotide-induced conformational changes and binding of further cofactors. The Journal of Biological Chemistry. 285 (52), 40921-40932 (2010).
- Popescu, A. I., Găzdaru, D. M., Chilom, C. G., Bacalum, M. Biophysical interactions: Their paramount importance for life. Romanian Reports in Physics. 65 (3), 1063-1077 (2013).
- Surya, S., Abhilash, J., Geethanandan, K., Sadasivan, C., Haridas, M. A profile of protein-protein interaction: Crystal structure of a lectin-lectin complex. International Journal of Biological Macromolecules. 87, 529-536 (2016).
- Velazquez-Campoy, A., Freire, E. ITC in the post-genomic era...? Priceless. Biophysical Chemistry. 115 (23), 115-124 (2005).
- Concepcion, J., et al. Label-free detection of biomolecular interactions using bioLayer interferometry for kinetic characterization. Combinatorial Chemistry & High Throughput Screening. 12 (8), 791-800 (2009).
- Helmerhorst, E., Chandler, D. J., Nussio, M., Mamotte, C. D. Real-time and label-free bio-sensing of molecular interactions by surface plasmon resonance: A laboratory medicine perspective. The Clinical Biochemist. Reviews. 33 (4), 161-173 (2012).
- Jacob, N. T., et al. Synthetic molecules for disruption of the MYC protein-protein interface. Bioorganic & Medicinal Chemistry. 26 (14), 4234-4239 (2018).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved