É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Explorando a interação biomolecular entre a acompanhante molecular Hsp90 e sua proteína cliente Kinase Cdc37 usando tecnologia de biosensão de efeito de campo
Neste Artigo
Resumo
A biosensação de efeito de campo (FEB) é uma técnica livre de rótulos para detectar interações biomoleculares. Ele mede a corrente elétrica através do biosensor de grafeno ao qual os alvos de ligação são imobilizados. A tecnologia FEB foi utilizada para avaliar interações biomoleculares entre Hsp90 e Cdc37 e foi detectada uma forte interação entre as duas proteínas.
Resumo
As interações biomoleculares desempenham papéis versáteis em inúmeros processos celulares, regulando e coordenando eventos biológicos funcionalmente relevantes. Biomoléculas como proteínas, carboidratos, vitaminas, ácidos graxos, ácidos nucleicos e enzimas são blocos fundamentais de construção de seres vivos; eles se reúnem em redes complexas em biossistemas para sincronizar uma miríade de eventos de vida. As proteínas normalmente utilizam redes interativas complexas para realizar suas funções; por isso é obrigatório avaliar tais interações para desvendar sua importância nas células tanto em níveis celulares quanto de organismos. Em direção a esse objetivo, introduzimos uma tecnologia emergente em rápida inovação, biosensação de efeito de campo (FEB), para determinar interações biomoleculares específicas. FEB é uma técnica de detecção biomolecular sem rótulos e confiável para determinar interações específicas e usa biosensores de alta qualidade à base de eletrônicos. A tecnologia FEB pode monitorar interações na faixa de nanomolar devido aos nanomateriais biocompatíveis usados em sua superfície biosensor. Como prova de conceito, elucidada a interação proteína-proteína (PPI) entre a proteína de choque térmico 90 (Hsp90) e o ciclo de divisão celular 37 (Cdc37). Hsp90 é um acompanhante molecular dependente de ATP que desempenha um papel essencial no controle de dobra, estabilidade, maturação e qualidade de muitas proteínas, regulando assim múltiplas funções celulares vitais. O Cdc37 é considerado um acompanhante molecular específico da quinase, pois reconhece e recruta especificamente quinases proteicas para Hsp90 para regular suas vias de transdução de sinal a jusante. Como tal, o Cdc37 é considerado co-acompanhante do Hsp90. A via acompanhante-quinase (complexo Hsp90/Cdc37) é hiperativada em múltiplas malignidades que promovem o crescimento celular; portanto, é um alvo potencial para a terapia do câncer. O presente estudo demonstra a eficiência da tecnologia FEB utilizando o sistema modelo Hsp90/Cdc37. A FEB detectou um PPI forte entre as duas proteínas (valores KD de 0,014 μM, 0,053 μM e 0,072 μM em três experimentos independentes). Em resumo, a FEB é uma plataforma de detecção de PPI sem rótulos e econômica, que oferece medições rápidas e precisas.
Introdução
Interações biomoleculares:
As proteínas são partes essenciais dos organismos e participam de inúmeras vias moleculares, como metabolismo celular, estrutura celular, sinalização celular, respostas imunes, adesão celular e muito mais. Enquanto algumas proteínas executam suas funções de forma independente, a maioria das proteínas interagem com outras proteínas usando uma interface de ligação para coordenar a atividade biológica adequada1.
As interações biomoleculares podem ser classificadas principalmente com base nas distintas características estruturais e funcionais das proteínas envolvidas2, por exemplo, com base nas superfícies proteicas, na estabilidade complexa ou na persistência das interações3. Identificar proteínas essenciais e seus papéis nas interações biomoleculares é vital para a compreensão de mecanismos bioquímicos no nível molecular4. Atualmente, existem várias abordagens para detectar essas interações5: in vitro6, no silico7, nas células vivas8, ex vivo9, e in vivo10 com cada um tendo seus próprios pontos fortes e fracos.
Os ensaios in vivo são realizados utilizando todo o animal como ferramenta experimental11, e os ensaiost he ex vivo são realizados em extratos de tecido ou órgãos inteiros (por exemplo, coração, cérebro, fígado) em um ambiente externo controlado, proporcionando alterações mínimas em condições naturais. A aplicação mais comum de estudos in vivo e ex vivo é avaliar a farmacocinética, a farmacodinâmica e os efeitos de toxicidade de potenciais agentes farmacológicos antes dos testes em humanos, garantindo sua segurança e eficácia globais12.
Interações biomoleculares também podem ser detectadas dentro de células vivas. As células vivas de imagem nos permitem observar interações dinâmicas enquanto executam as reações de uma determinada via bioquímica13. Além disso, técnicas de detecção, como bioluminescência ou transferência de ressonância fluorescência, podem fornecer informações sobre onde e quando essas interações ocorrem dentro da célula14. Embora a detecção em células vivas ofereça detalhes cruciais, essas metodologias de detecção dependem de óptica e rótulos, que podem não refletir a biologia nativa; eles também são menos controlados do que os métodos in vitro e requerem especialização especializada para realizar15.
Os métodos computacionais em silico são usados principalmente para a triagem em larga escala de moléculas-alvo antes dos experimentos in vitro . Métodos de previsão computacional, bancos de dados baseados em computador, acoplamento molecular, relações de estrutura-atividade quantitativa e outras abordagens de simulação de dinâmica molecular estão entre os bem estabelecidos nas ferramentas silico 16. Em comparação com técnicas experimentais laboriosas, as ferramentas em silico podem facilmente fazer previsões com alta sensibilidade, mas com menor precisão no desempenho preditivo17.
Ensaios in vitro são realizados com microrganismos ou moléculas biológicas fora de seu contexto biológico padrão. Retratar interações biomoleculares através de métodos in vitro é fundamental para entender as funções proteicas e a biologia por trás da complexa rede de funcionamento celular. A metodologia de ensaio preferencial é escolhida de acordo com as propriedades intrínsecas da proteína, valores cinéticos e o modo e intensidade das interações18,19.
A interação Hsp90/Cdc37:
A via acompanhante-quinase, que conecta Hsp90 e Cdc37, é um alvo terapêutico promissor na biologia tumoral20. O Hsp90 desempenha um papel central no controle do ciclo celular, montagem de proteínas, sobrevivência celular e caminhos de sinalização. Proteínas que dependem do Hsp90 para suas funções são entregues ao Hsp90 para complexação através de um co-acompanhante, como o Cdc37. O complexo Hsp90/Cdc37 controla a dobra da maioria das quinases proteicas e serve como um hub para uma infinidade de redes de sinalização intracelular21. É um alvo anti-tumor promissor devido à sua expressão elevada em várias malignidades, incluindo leucemia mieloblástica aguda, mieloma múltiplo e carcinoma hepatocelular22,23.
Comumente usadas técnicas de detecção de interação biomolecular in vitro
A co-imunoprecipitação (co-IP) é uma técnica que depende da especificidade do anticorpo de antígeno para identificar interações biologicamente relevantes24. A principal desvantagem deste método é sua incapacidade de detectar interações de baixa afinidade e valores cinéticos24. Métodos biofísicos como calorimetria de titulação isotérmica (ITC), ressonância de plasmon superficial (SPR), interferometria biocamida (BLI) e tecnologia FEB são preferidos para determinar os valores cinéticos.
ITC é um método de detecção biofísica baseado na determinação da energia vinculante, juntamente com uma análise termodinâmica completa para caracterizar interações biomoleculares25. A principal vantagem do ITC é que ele não requer nenhuma rotulagem ou fixação da proteína alvo. As principais dificuldades encontradas pelo ITC são a alta concentração de proteína-alvo necessária para um experimento e a dificuldade em analisar complexos não covalentes devido a pequenas indeseçõesde ligação 26. Tanto o SPR quanto o BLI são técnicas biofísicas sem rótulos que dependem da imobilização da molécula alvo na superfície do sensor, seguidas de injeções subsequentes do analito sobre o alvo imobilizado27,28. Em SPR, são medidas alterações no índice refrativo durante interações biomoleculares27; em BLI, a interferência na luz refletida é registrada em tempo real como uma mudança no comprimento de onda em função do tempo28. Tanto o SPR quanto o BLI compartilham vantagens comuns de oferecer altas especificidades, sensibilidade e recursos de detecção29. Em ambos os métodos, a proteína alvo é imobilizada em superfícies biosensoras e, portanto, pode haver alguma perda da conformação nativa do alvo, o que dificulta a discriminação entre interações específicas versus não específicas30. A BLI usa biosensores descartáveis de fibra óptica caros para imobilizar o alvo e, portanto, é uma técnicacara 31. Em comparação com essas ferramentas bem estabelecidas de detecção biomolecular, a tecnologia FEB oferece uma plataforma confiável e livre de rótulos usando baixas concentrações de nanomolar para detecção biomolecular em tempo real com caracterização cinética. A tecnologia FEB também supera os desafios borbulhantes enfrentados no ITC e é mais econômica em comparação com a SPR ou BLI.
O biosensores baseados em transistor de efeito de campo (FET) é um campo emergente para detectar interações biomoleculares, oferecendo aplicações biomédicas variadas. No sistema FET, os alvos são imobilizados para os chips biosensor e as interações são detectadas por alterações na condutância32. A característica única a ser considerada no desenvolvimento de um biosensor eletrônico eficiente são as propriedades físico-químicas, como a natureza semi-condutora e a estabilidade química do material de revestimento usado para fabricar a superfície do sensor33. Materiais convencionais como o silício usado para fet limitaram a sensibilidade dos sensores porque requer camadas de óxido entre o canal do transistor e um ambiente específico para o bom funcionamento34. Além disso, os transistores de silício são sensíveis a ambientes de alto sal, dificultando a mensuração das interações biológicas em seu ambiente natural. O biosensor à base de grafeno é apresentado como uma alternativa, pois oferece excelente estabilidade química e campo elétrico. Uma vez que o grafeno é uma única camada atômica de carbono, é extremamente sensível como um semicondutor e quimicamente compatível com soluções biológicas; ambas as qualidades são desejáveis para gerar biosensores eletrônicos compatíveis35. O notável potencial de carregamento ultra-alto de biomoléculas oferecido por biosensores revestidos de grafeno leva ao desenvolvimento da tecnologia FEB baseada em grafeno.
Princípio da tecnologia FEB: FEB é uma técnica de detecção biomolecular sem rótulos que mede a corrente elétrica através do biosensor de grafeno ao qual os alvos de ligação são imobilizados. As interações entre a proteína imobilizada e o analito resultam em alterações na corrente que são monitoradas em tempo real, permitindo medições cinéticas precisas36.
Instrumentação: O sistema FEB compreende um chip de sensor transistor de efeito de campo de grafeno (gFET) e um leitor eletrônico que aplica uma tensão constante ao longo do experimento (Figura 1). O analito é aplicado em solução para a proteína alvo imobilizada na superfície do biosensor. Quando ocorre uma interação, uma alteração na corrente é medida e registrada em tempo real. À medida que a concentração de analito aumenta, a fração de analito ligado também aumentará, causando maiores alterações na correnteza. Utilizando o software de análise automatizada fornecido com o instrumento (Tabela de Materiais), o I-Response é medido e registrado em termos de unidades de biosensação (BU)37. A I-Resposta é definida como a alteração na corrente (I) através do chip biosensor medido em tempo real após a interação do alvo imobilizado com o analito. O software de análise automatizada FEB pode analisar tanto o I-Response quanto o C-Response para eventos de interação dinâmica, onde o C-Response registra as alterações na capacitância (C). As variações tanto na I-Resposta quanto na Resposta C correspondem diretamente à fração de analito vinculado e podem ser analisadas para gerar valores KD . A preferência padrão do software de análise automatizada é I-Response.
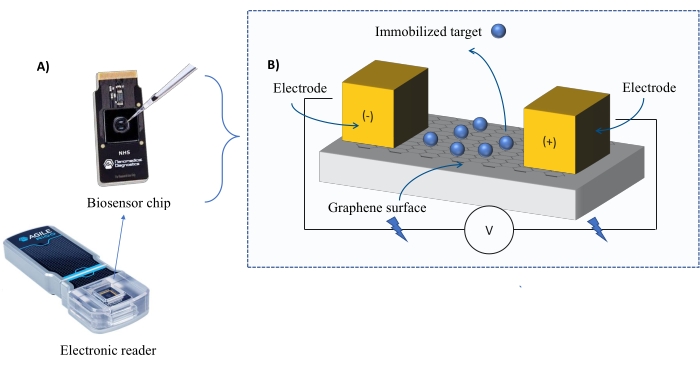
Figura 1: Visão geral da configuração experimental. (A) chip baseado em grafeno e um leitor eletrônico. (B) Uma visão geral dos componentes do chip. O chip está ligado a dois eletrodos que fornecem corrente ao sistema. A superfície do chip é coberta com grafeno, que quando ativado pode ligar o alvo. Clique aqui para ver uma versão maior desta figura.
Metodologia:
Inicialmente, o chip biosensor ativado é inserido no dispositivo FEB (Figura 1) seguido da execução das etapas abaixo: (1) Calibração: O experimento começa com a calibração do sistema usando 1x salina tamponada de fosfato (PBS; pH = 7,4) para criar a resposta de equilíbrio de linha de base. (2) Associação: O analito é introduzido no chip e o I-Response é monitorado até que a saturação de vinculação seja alcançada. (3) Dissociação: O analito é dissociado utilizando 1x PBS. (4) Regeneração: Os remanescentes do analito são removidos utilizando-se 1x PBS. (5) Lavagem: Um total de cinco lavagens são realizadas utilizando 1x PBS para a remoção completa dos analitos amarrados e desvinculados do chip.
Análise:
A análise de dados é realizada utilizando-se o software totalmente automatizado fornecido com o instrumento. O software de análise automatizada gera um gráfico de ajuste hill com um valor KD . O enredo de ajuste de Hill descreve a associação de um analito à proteína alvo em função de concentrações de analitos. A concentração na qual uma resposta semi-máxima é alcançada é proporcional ao valor KD . Um baixo valor KD representa alta afinidade vinculante e vice-versa.
Para validar os dados obtidos a partir do experimento FEB, as Respostas I são extraídas de cada ponto de leitura para cada concentração de analitos usando o software de revisão/exportação de dados e podem ser exportadas para outros softwares de análise estatística (ver Tabela de Materiais), conforme explicado abaixo.
Access restricted. Please log in or start a trial to view this content.
Protocolo
NOTA: As proteínas recombinantes utilizadas neste estudo, Hsp90 e Cdc37, foram obtidas comercialmente (ver Tabela de Materiais).
1. Ativação de chips
NOTA: Todos os materiais a serem utilizados no experimento estão listados na Tabela de Materiais. Filtre todas as soluções preparadas através de um filtro estéril de 0,2 μm.
- Prepare a solução de carbodiimídeo de 1-Ethyl-3-3-(3-dimethylamino propyl) (EDC) adicionando 2 mg de EDC a 2,5 mL de 1 M 2-(N-morpholino) tampão de ácido sulfônico de etano (MES) (pH = 6,0) em um tubo de 15 mL. Prepare as soluções N-Hydroxysulfosuccinimide (sulfo-NHS) adicionando 6 mg de sulfo-NHS a 2,5 mL de tampão mes de 1 M (pH = 6,0) em um tubo separado de 15 mL. Aliquot 50 μL de cada solução em tubos independentes e armazenar a -20 °C para uso futuro.
- Misture volumes iguais de solução EDC e sulfo-NHS (50 μL de EDC + 50 μL de sulfo-NHS) por pipetar para cima e para baixo (não vórtice).
NOTA: A solução mista de EDC/sulfo-NHS deve ser usada dentro de 30 minutos para manter uma ligação cruzada eficaz para uma funcionalidade adequada do chip. - Coloque o chip biosensor (5,7 cm x 2,4 cm; veja Tabela de Materiais) fornecido pela empresa em uma placa de vidro Petri com tampa equipada. Todas as etapas de funcionalização envolvidas na ativação do chip são sugeridas para serem feitas dentro da placa de Petri. Aplique 50 μL de 1 M de tampão MES (pH = 6,0) no chip biosensor, incubar por 1 min em temperatura ambiente e, em seguida, aspirar o buffer.
- Aplique 50 μL de solução EDC/sulfo-NHS imediatamente ao chip do sensor. Cubra a placa de Petri e incubar por 15 minutos em temperatura ambiente. Aspirar solução EDC/sulfo-NHS do chip.
- Enxágüe o chip uma vez com 50 μL de tampão MES de 1 M (pH = 6,0); aspirar o buffer MES.
2. Imobilização de proteínas-alvo
- Enxágüe o chip 2x com 50 μL de 1x PBS (pH = 7,4). Aspire o PBS do chip e adicione a molécula alvo, Hsp90 (50 μL; 500 nM).
NOTA: A incompatibilidade de buffer pode arruinar todo o experimento; portanto, antes do experimento, é importante ter certeza de que a molécula alvo está no mesmo buffer que a usada para calibração (por exemplo, 1x PBS (pH = 7,4)). Se necessário, realize uma troca de buffer por diálise durante a noite antes do experimento. Neste experimento, foi realizada diálise noturna para ambas as proteínas recombinantes, Hsp90 e Cdc37, contra 1x PBS (pH = 7,4; ver Tabela de Materiais) com troca de tampão adequada a 4 °C. A concentração do material alvo (Hsp90 neste caso) pode variar de acordo com diferentes protocolos experimentais e a natureza dos materiais-alvo (proteína/peptídeo/ligantes). - Cubra a placa de vidro Petri e incubar por 30 minutos em temperatura ambiente. Aspire a solução contendo a molécula alvo e enxágue 3x com 50 μL de 1x PBS (pH = 7,4). Aspire a solução 1x PBS (pH = 7,4) do chip.
- Adicione 50 μL de Súsia 1 (3,9 mM amino-PEG5-álcool em 1x PBS (pH = 7,4)) solução ao chip. Cubra a placa de petri de vidro e incubar por 15 minutos à temperatura ambiente. Aspire a solução Quench 1 do chip.
- Adicione 50 μL de Solução Quench 2 (1 M de etanolamina (pH = 8,5)) ao chip. Cubra a placa de petri de vidro e incubar por 15 minutos em temperatura ambiente. Aspirar a solução Quench 2 do chip e enxaguar o chip 5x usando 50 μL de 1x PBS, deixando a última gota pbs no sensor.
3. Preparando amostras de analito
- Prepare a série de diluição de analito para Cdc37 na faixa de concentração desejada. Para o primeiro experimento, foram utilizadas as seguintes concentrações: 25 nM, 50 nM, 100 nM, 200 nM, 400 nM, 800 nM, 1.000 nM, 2.000 nM, 3.000 nM e 5.000 nM. Para o segundo experimento, utilizou-se um conjunto diferente de concentrações que variavam de 0,4 nM a 200 nM.
- Projete o experimento para incluir pelo menos oito concentrações diferentes de analito para obter um valor KD confiável. Preparar as diferentes diluições da proteína de analito no mesmo tampão que a usada para calibração e proteína-alvo; aqui é 1x PBS (pH = 7,4).
4. Carregamento do chip biosensor ativado no dispositivo FEB
NOTA: O dispositivo FEB consiste em um leitor com indicações de luz LED e um cartucho para inserir o chip biosensor.
- Após a imobilização da proteína alvo, insira o chip ativado no cartucho do dispositivo, que é conectado via USB a um computador. Após a inserção do chip, uma luz LED verde será exibida no leitor indicando que o dispositivo FEB está pronto para o experimento. Instale o software automatizado (ver Tabela de Materiais) fornecido pela empresa no computador, ao qual o dispositivo FEB está conectado, para monitorar o experimento passo a passo conforme descrito abaixo.
5. Execute o experimento
- Pressione o módulo Executar Experimento no software automatizado e escolha 10 pontos com regeneração ou qualquer outro protocolo desejado. Preencha os seguintes detalhes: nome do operador, nome do experimento, data (por exemplo, Yana, Hsp90 + Cdc37, 14.03.2021); tampão de regeneração (por exemplo, tampão PBS); alvo imobilizado (por exemplo, Hsp90); analito em solução (por exemplo, Cdc37). Consulte a Figura Suplementar S1 para obter detalhes.
- Pressione o botão Iniciar o teste exibido no software e siga as instruções mostradas pelo software automatizado conforme descrito abaixo.
NOTA: O software é totalmente automatizado, fácil de usar e orienta o usuário durante todo o experimento passo a passo. Uma janela pop-up aparecerá na tela com instruções para prosseguir em cada etapa do experimento. O software fornecerá instruções para cada passo repetitivo consecutivamente da calibração, associação de analitos, dissociação, regeneração e lavagem (5x) para cada concentração de analitos ao longo do experimento. - Executar a calibração do instrumento. Para isso, aspire a solução PBS restante do chip e aplique 50 μL de tampão de calibração (1x PBS; pH = 7,4). Pressione o botão Continuar e aguarde por 5 minutos até que a etapa de calibração esteja concluída. O software exibe o ponto final determinado para a etapa de calibração (5 minutos) com um alarme de aviso para acompanhamento.
- Em seguida, faça uma associação de analitos. Para isso, aspire o tampão de calibração do chip e aplique 50 μL da menor concentração de analito (25 nM de Cdc37). Pressione o botão Continuar e aguarde por 5 minutos até que a etapa de associação esteja concluída. O software exibe o ponto final para a etapa de associação (5 min) com um alarme de aviso para prosseguir.
- Faça uma dissociação de analitos. Para isso, aspire a solução de analito do chip e aplique 50 μL do tampão de dissociação (1x PBS; pH = 7,4). Pressione o botão Continuar e aguarde por 5 minutos até que a duração da etapa de dissociação (5 min) esteja concluída. O software exibe o ponto final para a etapa de dissociação (5 min) com um alarme de aviso para acompanhamento.
- Em seguida, realize a regeneração de chips. Aspire a solução de dissociação do chip e aplique 50 μL de tampão de regeneração (1x PBS; pH = 7,4). Pressione o botão Continuar e aguarde por 30 s até que a duração da etapa de regeneração (30 s) esteja concluída. O software exibe o ponto final para a etapa de regeneração (30 s) com um alarme de aviso para acompanhamento.
- Finalmente, lave o chip. Aspire a solução de regeneração do chip e aplique 50 μL de tampão de lavagem (1x PBS; pH = 7,4) no chip. Aspire a solução do chip e repita este 5x. Deixe a última gota de tampão de lavagem no chip e pressione o botão Continuar e aguarde por 30 s até que a duração da etapa de lavagem seja concluída no visor do software.
NOTA: O software exibe o ponto final para a etapa de lavagem (30 s) com um alarme de aviso para prosseguir com o próximo ciclo do experimento. - Repita os passos para cada concentração de analito utilizada; as cinco etapas de calibração, associação de analitos, dissociação, regeneração e lavagem (5x) constituem um ciclo. Para o experimento aqui mostrado, realizamos 10 ciclos para 10 concentrações de analito (variando de 25 nM a 5.000 nM ou 0,4 nM a 200 nM; Figura 2).
6. Análise
- Pressione o botão Análise visto na parte superior do software de análise automatizada no final do experimento. Uma janela de exibição contendo todos os pontos experimentais aparecerá. Na janela, certifique-se de que as concentrações de analito utilizadas para o protocolo prescrito estão corretas.
- Pressione o botão Análise de execução para gerar automaticamente o valor KD . O software gera um gráfico de ajuste de Hill plotando as concentrações de analitos contra as respostas I correspondentes das quais a constante de dissociação em equilíbrio, valor KD , é calculada.
- Exporte os dados brutos para análise em outros softwares de análise estatística usando o software de revisão/exportação de dados descrito abaixo.
- Copie o arquivo R1R criado automaticamente no final do experimento (por exemplo, Hsp90 + Cdc37 14.03.2021) para uma nova pasta na área de trabalho. Abra o software de Análise/Exportação de Dados fornecido pela empresa (ver Figura Suplementar S2A).
- Clique em Arquivos > Process R1R > OK na tela inicial do software de revisão/exportação de dados.
- Selecione a pasta contendo arquivos R1R criados na área de trabalho na etapa 6.3.1. e pressione o botão OK . Isso cria uma cópia dos dados R1R originais, para revisar e editar os dados sem substituir o arquivo original.
- Pressione o ícone de processamento de dados visto na tela inicial do software de revisão/exportação de dados. Pressione os arquivos R1R processados de carga > OK. Selecione a mesma pasta criada na etapa 6.3.1 contendo arquivos R1R processados. Pressione ok. Nesta etapa, a pasta que contém os arquivos de experimento está pronta para ser revisada.
- Na tela inicial do software de revisão/exportação de dados, pressione a Análise de Dados. Selecione Calibração > Calibrar arquivos R1R editados > OK (ver Figura Suplementar S2B). Esta etapa calibra todos os pontos de dados de acordo com a primeira etapa de calibração, para criar uma linha de base.
- Na tela inicial do software de revisão/exportação de dados, pressione a Análise de Dados. Selecione 'Revisão e editar arquivos R1R > OK. Revise os pontos de dados, exclua pontos ou adicione etapas usando o botão Excluir/Adicionar Passo . Certifique-se de que todas as etapas estão no lugar correto, por exemplo, a etapa de calibração está na linha de base, a etapa de associação está no pico (ver Figura Suplementar S3) e, em seguida, pressione o botão Salvar para salvar todas as alterações feitas.
- Na tela inicial do software de revisão/exportação de dados, pressione a Análise de Dados. Selecione Analisar/Plot Data, escolha as etapas para exportar (consulte Figura Suplementar S4). Adicione cada etapa de associação (por exemplo, passo 2, passo 7 e assim por diante) depois de subtrair a etapa de calibração (por exemplo, passo 1, passo 6 e assim por diante) à lista de exportação antes de exportar os dados.
- Pressione exportar esses dados. O software gera um arquivo de planilha contendo o I-Response para cada ponto de dados de concentração analito de cada transistor (se todos os transistores estiverem trabalhando, teremos três valores diferentes de resposta I para cada ponto de concentração). Use este arquivo de planilha para analisar melhor os dados no software estatístico.
- Abra o software de análise estatística. Criar uma tabela XY com três valores Y; pressione Criar. Os dados desta tabela (valores X, Y) são copiados do arquivo de planilha criado na etapa 6.3.8. O eixo x corresponde à concentração de analito (dependendo da concentração utilizada), e os três valores Y correspondem às Respostas I, obtidas a partir do software de Análise/Exportação de Dados fornecido pela empresa.
- Pressione Analisar esses dados. Escolha análises XY > regressão não linear (ajuste de curva) > saturação de ligação > Um Local > Total. O software analisará automaticamente os dados, gerará o valor KD e criará um gráfico dos pontos de dados.
Access restricted. Please log in or start a trial to view this content.
Resultados
Resultados do experimento 1:
A proteína alvo Hsp90 (500 nM) foi imobilizada para o chip seguindo o protocolo de imobilização de alvo, conforme descrito acima. Para o primeiro experimento, foram elaboradas 10 concentrações da proteína analito, Cdc37, variando de 25 nM a 5.000 nM, com base nos dados disponíveis na literatura (ver Tabela 1).
As etapas do experimento podem ser monitoradas em tempo real, seguindo as alterações ocorridas na I-Resposta (<...
Access restricted. Please log in or start a trial to view this content.
Discussão
Neste estudo, foi avaliada a viabilidade do uso da tecnologia FEB (abordagem de caracterização cinética em tempo real) para determinar a interação biomolecular entre Hsp90 e Cdc37. O experimento exploratório inicial (primeiro experimento) sugeriu que a escolha das concentrações adequadas de analito é uma parte crítica do experimento e que o experimento deve ser projetado por incluir pontos de concentração acima e abaixo do valor KD , que foram previstos com base nos dados disponíveis na literatura...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não declaram conflitos de interesse, financeiros ou não.
Agradecimentos
Esta pesquisa foi apoiada por uma bolsa da Binational Science Foundation (BSF) para s.K.S. e N.Q.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Automated analysis software | Agile plus software, Cardea (Nanomed) | NA CAS number: NA | Referred to in the text as the automated analysis software supplied with the instrument. Generates automated analysis. |
| COOH-BPU (Biosensing Processing Unit) | Agile plus software, Cardea (Nanomed) | NA CAS number: NA | biosensor chip |
| Data review software | Datalign 1.0, Cardea (Nanomed) | NA CAS number: NA | Referred to as the supplied data review software in the text. Supplied with the instrument and allows to review and export the information data points. |
| Dialysis bag | CelluSep, Membrane filtration products | T2-10-15 CAS number: NA | T2 tubings (6,000-8,000 MWCO), (10 mm fw, 6.4mm Ø, 0.32ml/cm, 15m) |
| EDC (1-Ethyl-3-(3-dimethylamino propyl) carbodiimide) | Cardea (Nanomed) | EDC160322-02 CAS number: 25952-53-8 | White powder |
| ITC (Isothermal titration calorimetry) system | Microcal-PEAQ-ITC (Malvern, United Kingdom) | NA CAS number: NA | |
| MES (2-(N-morpholino) ethane sulfonic acid) buffer | Merck | M3671-50G CAS number: 4432-31-9 | White powder |
| NHS (N-Hydroxysulfosuccinimide) chips | Cardea (Nanomed) | NA CAS number: NA | Graphene-based chip |
| PBS (Phosphate-buffered saline) X 10 | Bio-Lab | 001623237500 CAS number: 7758-11-4 | Liquid transparent solution |
| Pipete | Thermo Scientific | 11855231 CAS number: NA | Finnpipette F3 5-50 µL, yellow |
| Quench 1 (3.9 mM amino-PEG5-alcohol in 1 X PBS) | Cardea (Nanomed) | 0105-001-002-001 CAS number: NA | Liquid, transparent solution |
| Quench 2 (1 M ethanolamine (pH=8.5)) | Cardea (Nanomed) | 0105-001-003-001 CAS number: NA | Liquid, transparent solution |
| Recombinant protein Cdc37 | Abcam | ab256157 CAS number: NA | |
| Recombinant protein Hsp90 beta | Abcam | ab80033 CAS number: NA | |
| Spreadsheet | Excel, Microsoft office | NA CAS number: NA | |
| Statistical software | GraphPad, Prism | NA CAS number: NA | Referred to as the other statistical software. Sigma plot, phyton or other statistical programes may also be used |
| Sulfo-NHS | Cardea (Nanomed) | NHS160321-07 CAS number: 106627-54-7 | White powder |
| Tips | Alex red | LC 1093-800-000 CAS number: NA | Tip 1-200 µl, in bulk, 1,000 pcs |
Referências
- Tuncbag, N., Gursoy, A., Guney, E., Nussinov, R., Keskin, O. Architectures and functional coverage of protein-protein interfaces. Journal of Molecular Biology. 381 (3), 785-802 (2008).
- Berggård, T., Linse, S., James, P. Methods for the detection and analysis of protein–protein interactions. Proteomics. 7 (16), 2833-2842 (2007).
- Magliery, T. J., et al. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: Scope and mechanism. Journal of the American Chemical Society. 127 (1), 146-157 (2005).
- Xing, S., Wallmeroth, N., Berendzen, K. W., Grefen, C. Techniques for the analysis of protein-protein interactions in vivo. Plant Physiology. 171 (2), 727-758 (2016).
- Nguyen, T. N., Goodrich, J. A. Protein-protein interaction assays: Eliminating false positive interactions. Nature Methods. 3 (2), 135-139 (2006).
- Fernández-Suárez, M., Chen, T. S., Ting, A. Y. Protein-protein interaction detection in vitro and in cells by proximity biotinylation. Journal of the American Chemical Society. 130 (29), 9251-9253 (2008).
- Jiang, M., Niu, C., Cao, J., Ni, D. -A., Chu, Z. In silico-prediction of protein–protein interactions network about MAPKs and PP2Cs reveals a novel docking site variants in Brachypodium distachyon. Scientific Reports. 8 (1), 15083(2018).
- Yazawa, M., Sadaghiani, A. M., Hsueh, B., Dolmetsch, R. E. Induction of protein-protein interactions in live cells using light. Nature Biotechnology. 27 (10), 941-945 (2009).
- Wang, W., Goodman, M. T. Antioxidant property of dietary phenolic agents in a human LDL-oxidation ex vivo model: Interaction of protein binding activity. Nutrition Research. 19 (2), 191-202 (1999).
- Xing, S., Wallmeroth, N., Berendzen, K. W., Grefen, C. Techniques for the analysis of protein-protein interactions in vivo. Plant Physiology. 171 (2), 727-758 (2016).
- Qvit, N., Disatnik, M. -H., Sho, E., Mochly-Rosen, D. Selective phosphorylation inhibitor of delta protein kinase C–Pyruvate dehydrogenase kinase protein–protein interactions: Application for myocardial injury in vivo. Journal of the American Chemical Society. 138 (24), 7626-7635 (2016).
- Alam, M. N., Bristi, N. J., Rafiquzzaman, M. Review on in vivo and in vitro methods evaluation of antioxidant activity. Saudi Pharmaceutical Journal. 21 (2), 143-152 (2013).
- Paulmurugan, R., Gambhir, S. S. Novel fusion protein approach for efficient high-throughput screening of small molecule–mediating protein-protein interactions in cells and living animals. Cancer Research. 65 (16), 7413-7420 (2005).
- Boute, N., Jockers, R., Issad, T. The use of resonance energy transfer in high-throughput screening: BRET versus FRET. Trends in Pharmacological Sciences. 23 (8), 351-354 (2002).
- Deriziotis, P., Graham, S. A., Estruch, S. B., Fisher, S. E. Investigating protein-protein interactions in live cells using bioluminescence resonance energy transfer. Journal of Visualized Experiments: JoVE. (87), e51438(2014).
- Ekins, S., Mestres, J., Testa, B. In silico pharmacology for drug discovery: Methods for virtual ligand screening and profiling. British Journal of Pharmacology. 152 (1), 9-20 (2007).
- Valerio, L. G. Application of advanced in silico methods for predictive modeling and information integration. Expert Opinion on Drug Metabolism & Toxicology. 8 (4), 395-398 (2012).
- Piehler, J. New methodologies for measuring protein interactions in vivo and in vitro. Current Opinion in Structural Biology. 15 (1), 4-14 (2005).
- Ideker, T., Sharan, R. Protein networks in disease. Genome Research. 18 (4), 644-652 (2008).
- Lu, H., et al. Recent advances in the development of protein–protein interactions modulators: mechanisms and clinical trials. Signal Transduction and Targeted Therapy. 5 (1), 213(2020).
- Jarosz, D. Hsp90: A global regulator of the genotype-to-phenotype map in cancers. Advances in Cancer Research. 129, 225-247 (2016).
- Johnson, V. A., Singh, E. K., Nazarova, L. A., Alexander, L. D., McAlpine, S. R. Macrocyclic inhibitors of Hsp90. Current Topics in Medicinal Chemistry. 10 (14), 1380-1402 (2010).
- Mahalingam, D., et al. Targeting HSP90 for cancer therapy. British Journal of Cancer. 100 (10), 1523-1529 (2009).
- Stewart, A., Fisher, R. A. Co-Immunoprecipitation: Isolation of protein signaling complexes from native tissues. Methods in Cell Biology. 112, 33-54 (2012).
- Pierce, M. M., Raman, C. S., Nall, B. T. Isothermal titration calorimetry of protein-protein interactions. Methods. 19 (2), San Diego, Calif. 213-221 (1999).
- Paketurytė, V., et al. Inhibitor binding to carbonic anhydrases by isothermal titration calorimetry. Carbonic Anhydrase as Drug Target. , Springer International Publishing. 79-95 (2019).
- Grote, J., Dankbar, N., Gedig, E., Koenig, S. Surface plasmon resonance/mass spectrometry interface. Analytical Chemistry. 77 (4), 1157-1162 (2005).
- Kumaraswamy, S., Tobias, R. Label-free kinetic analysis of an antibody–antigen interaction using biolayer interferometry. Methods in Molecular Biology. , 165-182 (2015).
- Wallner, J., Lhota, G., Jeschek, D., Mader, A., Vorauer-Uhl, K. Application of bio-layer interferometry for the analysis of protein/liposome interactions. Journal of Pharmaceutical and Biomedical Analysis. 72, 150-154 (2013).
- Singh, A. N., Ramadan, K., Singh, S. Experimental methods to study the kinetics of protein–protein interactions. Advances in Protein Molecular and Structural Biology Methods. , 115-124 (2022).
- Frenzel, D., Willbold, D. Kinetic titration series with biolayer interferometry. PLoS One. 9 (9), 106882(2014).
- Vu, C. -A., Chen, W. -Y. Field-effect transistor biosensors for biomedical applications: Recent advances and future prospects. Sensors. 19 (19), 4214(2019).
- Bergveld, P. A critical evaluation of direct electrical protein detection methods. Biosensors & Bioelectronics. 6 (1), 55-72 (1991).
- Lowe, B. M., Sun, K., Zeimpekis, I., Skylaris, C. K., Green, N. G. Field-effect sensors – from pH sensing to biosensing: sensitivity enhancement using streptavidin–biotin as a model system. The Analyst. 142 (22), 4173-4200 (2017).
- Goldsmith, B. R., et al. Digital biosensing by foundry-fabricated graphene sensors. Scientific Reports. 9 (1), 434(2019).
- Afsahi, S., et al. Novel graphene-based biosensor for early detection of Zika virus infection. Biosensors and Bioelectronics. 100, 85-88 (2018).
- Afsahi, S. J., et al. Towards novel graphene-enabled diagnostic assays with improved signal-to-noise ratio. MRS Advances. 2 (60), 3733-3739 (2017).
- Roe, S. M., et al. The mechanism of Hsp90 regulation by the protein kinase-specific cochaperone p50cdc37. Cell. 116 (1), 87-98 (2004).
- Gaiser, A. M., Kretzschmar, A., Richter, K. Cdc37-Hsp90 complexes are responsive to nucleotide-induced conformational changes and binding of further cofactors. The Journal of Biological Chemistry. 285 (52), 40921-40932 (2010).
- Popescu, A. I., Găzdaru, D. M., Chilom, C. G., Bacalum, M. Biophysical interactions: Their paramount importance for life. Romanian Reports in Physics. 65 (3), 1063-1077 (2013).
- Surya, S., Abhilash, J., Geethanandan, K., Sadasivan, C., Haridas, M. A profile of protein-protein interaction: Crystal structure of a lectin-lectin complex. International Journal of Biological Macromolecules. 87, 529-536 (2016).
- Velazquez-Campoy, A., Freire, E. ITC in the post-genomic era...? Priceless. Biophysical Chemistry. 115 (23), 115-124 (2005).
- Concepcion, J., et al. Label-free detection of biomolecular interactions using bioLayer interferometry for kinetic characterization. Combinatorial Chemistry & High Throughput Screening. 12 (8), 791-800 (2009).
- Helmerhorst, E., Chandler, D. J., Nussio, M., Mamotte, C. D. Real-time and label-free bio-sensing of molecular interactions by surface plasmon resonance: A laboratory medicine perspective. The Clinical Biochemist. Reviews. 33 (4), 161-173 (2012).
- Jacob, N. T., et al. Synthetic molecules for disruption of the MYC protein-protein interface. Bioorganic & Medicinal Chemistry. 26 (14), 4234-4239 (2018).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados