Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Exploration de l’interaction biomoléculaire entre le chaperon moléculaire Hsp90 et sa protéine cliente Kinase Cdc37 à l’aide de la technologie de biodétection à effet de champ
Dans cet article
Résumé
La biodétection à effet de champ (FEB) est une technique sans étiquette permettant de détecter les interactions biomoléculaires. Il mesure le courant électrique à travers le biocapteur de graphène auquel les cibles de liaison sont immobilisées. La technologie FEB a été utilisée pour évaluer les interactions biomoléculaires entre Hsp90 et Cdc37 et une forte interaction entre les deux protéines a été détectée.
Résumé
Les interactions biomoléculaires jouent un rôle polyvalent dans de nombreux processus cellulaires en régulant et en coordonnant des événements biologiques fonctionnellement pertinents. Les biomolécules telles que les protéines, les glucides, les vitamines, les acides gras, les acides nucléiques et les enzymes sont des éléments constitutifs fondamentaux des êtres vivants; ils s’assemblent en réseaux complexes dans les biosystèmes pour synchroniser une myriade d’événements de la vie. Les protéines utilisent généralement des réseaux d’interactomes complexes pour remplir leurs fonctions; il est donc obligatoire d’évaluer de telles interactions pour démêler leur importance dans les cellules aux niveaux cellulaire et de l’organisme. Pour atteindre cet objectif, nous introduisons une technologie en émergence rapide, la biodétection à effet de champ (FEB), pour déterminer des interactions biomoléculaires spécifiques. FEB est une technique de détection biomoléculaire fiable et sans étiquette de paillasse pour déterminer des interactions spécifiques et utilise des biocapteurs électroniques de haute qualité. La technologie FEB peut surveiller les interactions dans la gamme nanomolaire en raison des nanomatériaux biocompatibles utilisés sur sa surface de biocapteur. Comme preuve de concept, l’interaction protéine-protéine (IPP) entre la protéine de choc thermique 90 (Hsp90) et le cycle de division cellulaire 37 (Cdc37) a été élucidée. Hsp90 est un chaperon moléculaire dépendant de l’ATP qui joue un rôle essentiel dans le repliement, la stabilité, la maturation et le contrôle de la qualité de nombreuses protéines, régulant ainsi de multiples fonctions cellulaires vitales. Cdc37 est considéré comme un chaperon moléculaire spécifique à la protéine kinase, car il reconnaît et recrute spécifiquement les protéines kinases à Hsp90 pour réguler leurs voies de transduction du signal en aval. En tant que tel, Cdc37 est considéré comme un co-chaperon de Hsp90. La voie chaperon-kinase (complexe Hsp90/Cdc37) est hyper-activée dans de multiples tumeurs malignes favorisant la croissance cellulaire; par conséquent, c’est une cible potentielle pour le traitement du cancer. La présente étude démontre l’efficacité de la technologie FEB à l’aide du système modèle Hsp90/Cdc37. Feb a détecté un fort IPP entre les deux protéines (valeurs KD de 0,014 μM, 0,053 μM et 0,072 μM dans trois expériences indépendantes). En résumé, FEB est une plate-forme de détection PPI sans étiquette et rentable, qui offre des mesures rapides et précises.
Introduction
Interactions biomoléculaires :
Les protéines sont des parties essentielles des organismes et participent à de nombreuses voies moléculaires telles que le métabolisme cellulaire, la structure cellulaire, la signalisation cellulaire, les réponses immunitaires, l’adhésion cellulaire, etc. Alors que certaines protéines remplissent leur(s) fonction(s) indépendamment, la plupart des protéines interagissent avec d’autres protéines en utilisant une interface de liaison pour coordonner l’activité biologique appropriée1.
Les interactions biomoléculaires peuvent principalement être classées en fonction des caractéristiques structurelles et fonctionnelles distinctes des protéines impliquées2, par exemple, en fonction des surfaces protéiques, de la stabilité complexe ou de la persistance des interactions3. L’identification des protéines essentielles et de leurs rôles dans les interactions biomoléculaires est essentielle pour comprendre les mécanismes biochimiques au niveau moléculaire4. Actuellement, il existe différentes approches pour détecter ces interactions5 : in vitro6, in silico7, dans les cellules vivantes8, ex vivo9 et in vivo10, chacune ayant ses propres forces et faiblesses.
Les essais in vivo sont effectués en utilisant l’animal entier comme outil expérimental11, et les essaisex vivo sont effectués sur des extraits de tissus ou des organes entiers (par exemple, le cœur, le cerveau, le foie) dans un environnement externe contrôlé en fournissant des altérations minimales dans les conditions naturelles. L’application la plus courante des études in vivo et ex vivo consiste à évaluer les effets pharmacocinétiques, pharmacodynamiques et toxiques d’agents pharmacologiques potentiels avant les essais chez l’homme en s’assurant de leur innocuité et de leur efficacitéglobales 12.
Les interactions biomoléculaires peuvent également être détectées au sein des cellules vivantes. L’imagerie des cellules vivantes nous permet d’observer des interactions dynamiques lorsqu’elles exécutent les réactions d’une voie biochimique particulière13. De plus, les techniques de détection, telles que la bioluminescence ou le transfert d’énergie par résonance de fluorescence, peuvent fournir des informations sur l’endroit et le moment où ces interactions se produisent dans la cellule14. Bien que la détection dans les cellules vivantes offre des détails cruciaux, ces méthodologies de détection reposent sur l’optique et les étiquettes, qui peuvent ne pas refléter la biologie native; ils sont également moins contrôlés que les méthodes in vitro et nécessitent une expertise spécialisée pour en effectuer15.
Les méthodes de calcul in silico sont principalement utilisées pour le criblage à grande échelle des molécules cibles avant les expériences in vitro . Les méthodes de prédiction computationnelle, les bases de données informatisées, l’amarrage moléculaire, les relations quantitatives structure-activité et d’autres approches de simulation de la dynamique moléculaire font partie des outils in silico bien établis16. Comparés à des techniques expérimentales laborieuses, les outils in silico peuvent facilement faire des prédictions avec une sensibilité élevée, mais avec une précision réduite dans les performances prédictives17.
Les essais in vitro sont effectués avec des micro-organismes ou des molécules biologiques en dehors de leur contexte biologique standard. La représentation des interactions biomoléculaires par des méthodes in vitro est essentielle à la compréhension des fonctions des protéines et de la biologie derrière le réseau complexe du fonctionnement cellulaire. La méthodologie de dosage préférée est choisie en fonction des propriétés intrinsèques de la protéine, des valeurs cinétiques et du mode et de l’intensité des interactions18,19.
L’interaction Hsp90/Cdc37 :
La voie chaperon-kinase, reliant Hsp90 et Cdc37, est une cible thérapeutique prometteuse en biologie tumorale20. Hsp90 joue un rôle central dans le contrôle du cycle cellulaire, l’assemblage des protéines, la survie cellulaire et les voies de signalisation. Les protéines qui dépendent de Hsp90 pour leurs fonctions sont livrées à Hsp90 pour la complexation par un co-chaperon, tel que Cdc37. Le complexe Hsp90/Cdc37 contrôle le repliement de la plupart des protéines kinases et sert de plaque tournante pour une multitude de réseaux de signalisation intracellulaire21. C’est une cible antitumorale prometteuse en raison de son expression élevée dans diverses tumeurs malignes, y compris la leucémie myéloblastique aiguë, le myélome multiple et le carcinome hépatocellulaire22,23.
Techniques de détection d’interactions biomoléculaires in vitro couramment utilisées
La co-immunoprécipitation (co-IP) est une technique qui repose sur la spécificité antigène-anticorps pour identifier les interactions biologiquement pertinentes24. Le principal inconvénient de cette méthode est son incapacité à détecter les interactions de faible affinité et les valeurs cinétiques24. Les méthodes biophysiques telles que la calorimétrie de titrage isotherme (ITC), la résonance plasmonique de surface (SPR), l’interférométrie biocouche (BLI) et la technologie FEB sont préférées pour déterminer les valeurs cinétiques.
ITC est une méthode de détection biophysique basée sur la détermination de l’énergie de liaison ainsi qu’une analyse thermodynamique complète pour caractériser les interactions biomoléculaires25. Le principal avantage de l’ITC est qu’il ne nécessite aucun marquage ou fixation de la protéine cible. Les principales difficultés rencontrées par l’ITC sont la forte concentration de protéines cibles requise pour une expérience et la difficulté d’analyser les complexes non covalents en raison de petites enthalpies de liaison26. Spr et BLI sont des techniques biophysiques sans étiquette qui reposent sur l’immobilisation de la molécule cible à la surface du capteur, suivie d’injections ultérieures de l’analyte sur la cible immobilisée27,28. Dans la SPR, les altérations de l’indice de réfraction au cours des interactions biomoléculaires sont mesurées27; dans BLI, l’interférence dans la lumière réfléchie est enregistrée en temps réel sous forme de changement de longueur d’onde en fonction du temps28. SPR et BLI partagent tous deux des avantages communs en offrant des capacités de spécificité, de sensibilité et de détection élevées29. Dans les deux méthodes, la protéine cible est immobilisée sur les surfaces des biocapteurs et, par conséquent, il peut y avoir une certaine perte de la conformation native de la cible, ce qui rend difficile la distinction entre les interactions spécifiques et non spécifiques30. BLI utilise des biocapteurs à fibre optique jetables coûteux pour immobiliser la cible, et est donc une technique coûteuse31. Par rapport à ces outils de détection biomoléculaire bien établis, la technologie FEB offre une plate-forme fiable et sans étiquette en utilisant de faibles concentrations nanomolaires pour la détection biomoléculaire en temps réel avec caractérisation cinétique. La technologie FEB surmonte également les défis bouillonnants rencontrés dans l’ITC et est plus rentable que SPR ou BLI.
Les biocapteurs à transistor à effet de champ (FET) sont un domaine émergent pour la détection des interactions biomoléculaires en offrant des applications biomédicales variées. Dans le système FET, les cibles sont immobilisées sur les puces de biocapteur et les interactions sont détectées par des changements de conductance32. La caractéristique unique à prendre en compte dans le développement d’un biocapteur électronique efficace réside dans les propriétés physico-chimiques telles que la nature semi-conductrice et la stabilité chimique du matériau de revêtement utilisé pour fabriquer la surface du capteur33. Les matériaux conventionnels comme le silicium utilisé pour le FET ont limité la sensibilité des capteurs car ils nécessitent des couches d’oxyde prises en sandwich entre le canal du transistor et un environnement spécifique pour un bon fonctionnement34. De plus, les transistors en silicium sont sensibles aux environnements riches en sel, ce qui rend difficile la mesure des interactions biologiques dans leur environnement naturel. Le biocapteur à base de graphène est présenté comme une alternative car il offre une excellente stabilité chimique et un champ électrique. Étant donné que le graphène est une seule couche atomique de carbone, il est à la fois extrêmement sensible en tant que semi-conducteur et chimiquement compatible avec les solutions biologiques; ces deux qualités sont souhaitables pour générer des biocapteurs électroniques compatibles35. Le remarquable potentiel de charge ultra-élevé des biomolécules offert par les biocapteurs revêtus de graphène conduit au développement de la technologie FEB des biocapteurs à base de graphène.
Principe de la technologie FEB: FEB est une technique de détection biomoléculaire sans étiquette qui mesure le courant électrique à travers le biocapteur de graphène auquel les cibles de liaison sont immobilisées. Les interactions entre la protéine immobilisée et l’analyte entraînent des altérations du courant qui sont surveillées en temps réel, ce qui permet des mesures cinétiques précises36.
Instrumentation : Le système FEB comprend une puce de capteur à transistor à effet de champ en graphène (gFET) et un lecteur électronique qui applique une tension constante tout au long de l’expérience (Figure 1). L’analyte est appliqué en solution sur la protéine cible immobilisée à la surface du biocapteur. Lorsqu’une interaction se produit, une altération du courant est mesurée et enregistrée en temps réel. Au fur et à mesure que la concentration de l’analyte augmente, la fraction d’analyte lié augmentera également, provoquant des alternances plus élevées dans le courant. À l’aide du logiciel d’analyse automatisé fourni avec l’instrument (Table des matériaux), I-Response est mesurée et enregistrée en termes d’unités de biodétection (BU)37. I-Response est définie comme l’altération du courant (I) à travers la puce de biocapteur mesurée en temps réel lors de l’interaction de la cible immobilisée avec l’analyte. Le logiciel d’analyse automatisé FEB peut analyser à la fois la réponse I et la réponse C aux événements d’interaction dynamique, où la réponse C enregistre les altérations de la capacité (C). Les variations de la réponse I et de la réponse C correspondent directement à la fraction de l’analyte lié et peuvent être analysées plus en détail pour générer des valeurs KD . La préférence par défaut du logiciel d’analyse automatisée est I-Response.
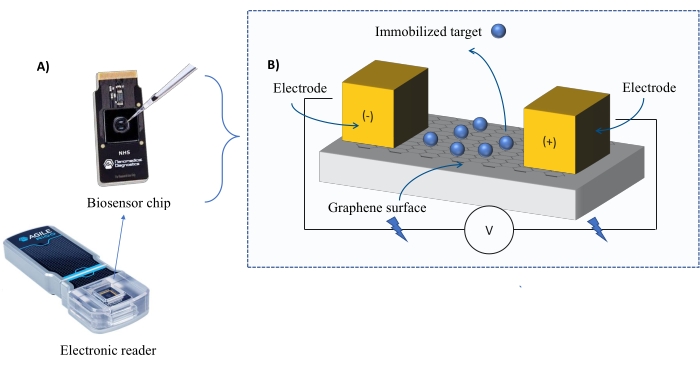
Figure 1: Vue d’ensemble de la configuration expérimentale. (A) Puce à base de graphène et lecteur électronique. (B) Une vue d’ensemble des composants de la puce. La puce est fixée à deux électrodes qui fournissent du courant au système. La surface de la puce est recouverte de graphène qui, lorsqu’il est activé, peut lier la cible. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Méthodologie:
Initialement, la puce de biocapteur activée est insérée dans le dispositif FEB (Figure 1) suivie de l’exécution des étapes décrites ci-dessous: (1) Étalonnage: L’expérience commence par l’étalonnage du système en utilisant 1x solution saline tamponnée au phosphate (PBS; pH = 7,4) pour créer la réponse d’équilibrage de base. (2) Association: L’analyte est introduit dans la puce et la réponse I est surveillée jusqu’à ce que la saturation de liaison soit atteinte. (3) Dissociation: L’analyte est dissocié à l’aide de 1x PBS. (4) Régénération: Les restes de l’analyte sont éliminés à l’aide de 1x PBS. (5) Lavage: Un total de cinq lavages sont effectués à l’aide de 1x PBS pour l’élimination complète des analytes liés et non liés de la puce.
Analyse:
L’analyse des données est effectuée à l’aide du logiciel entièrement automatisé fourni avec l’instrument. Le logiciel d’analyse automatisé génère un diagramme d’ajustement de Hill avec une valeur KD . Le diagramme d’ajustement de Hill décrit l’association d’un analyte à la protéine cible en fonction des concentrations d’analyte. La concentration à laquelle une réponse demi-maximale est obtenue est proportionnelle à la valeur KD . Une valeur KD faible représente une affinité de liaison élevée et vice versa.
Pour valider les données obtenues à partir de l’expérience FEB, les réponses I sont extraites de chaque point de lecture pour chaque concentration d’analyte à l’aide du logiciel d’examen/exportation des données et peuvent être exportées vers d’autres logiciels d’analyse statistique (voir tableau des matériaux) comme expliqué ci-dessous.
Access restricted. Please log in or start a trial to view this content.
Protocole
REMARQUE : Les protéines recombinantes utilisées dans cette étude, Hsp90 et Cdc37, ont été obtenues commercialement (voir tableau des matériaux).
1. Activation de la puce
REMARQUE : Tous les matériaux à utiliser dans l’expérience sont répertoriés dans le tableau des matériaux. Filtrer toutes les solutions préparées à l’aide d’un filtre stérile de 0,2 μm.
- Préparer une solution de 1-Éthyl-3-(3-diméthylaminopropyl)carbodiimure (EDC) en ajoutant 2 mg d’EDC à 2,5 mL de tampon d’acide éthane-sulfonique (MES) de 1 M2-(N-morpholino) éthane sulfonique (pH = 6,0) dans un tube de 15 mL. Préparer des solutions de N-Hydroxysulfosuccinimide (sulfo-NHS) en ajoutant 6 mg de sulfo-NHS à 2,5 mL de tampon MES de 1 M (pH = 6,0) dans un tube séparé de 15 mL. Aliquoter 50 μL de chaque solution dans des tubes indépendants et conserver à -20 °C pour une utilisation future.
- Mélanger des volumes égaux de solution de sulfo-NHS de sable EDC (50 μL d’EDC + 50 μL de sulfo-NHS) en pipetant de haut en bas (ne pas vortex).
REMARQUE: La solution mixte d’EDC / sulfo-NHS doit être utilisée dans les 30 minutes pour maintenir une réticulation efficace pour une fonctionnalisation appropriée de la puce. - Placez la puce de biocapteur (5,7 cm x 2,4 cm; voir Tableau des matériaux) fournie par l’entreprise dans une boîte de Petri en verre avec un couvercle intégré. Toutes les étapes de fonctionnalisation impliquées dans l’activation de la puce sont suggérées à faire dans la boîte de Pétri. Appliquer 50 μL de tampon MES de 1 M (pH = 6,0) sur la puce du biocapteur, incuber pendant 1 min à température ambiante, puis aspirer le tampon.
- Appliquer immédiatement 50 μL de solution EDC/sulfo-NHS sur la puce du capteur. Couvrir la boîte de Petri et incuber pendant 15 min à température ambiante. Aspirer la solution EDC/sulfo-NHS de la puce.
- Rincez la puce une fois avec 50 μL de tampon MES de 1 M (pH = 6,0); aspirer le tampon MES.
2. Immobilisation des protéines cibles
- Rincez la puce 2x avec 50 μL de 1x PBS (pH = 7,4). Aspirer le PBS de la puce et ajouter la molécule cible, Hsp90 (50 μL; 500 nM).
REMARQUE: L’incompatibilité de la mémoire tampon peut ruiner toute l’expérience; par conséquent, avant l’expérience, il est important de s’assurer que la molécule cible est dans le même tampon que celui utilisé pour l’étalonnage (par exemple, 1x PBS (pH = 7,4)). Si nécessaire, effectuez un échange tampon par dialyse de nuit avant l’expérience. Dans cette expérience, une dialyse de nuit a été effectuée pour les deux protéines recombinantes, Hsp90 et Cdc37, contre 1x PBS (pH = 7,4; voir tableau des matériaux) avec un échange tampon approprié à 4 ° C. La concentration du matériau cible (Hsp90 dans ce cas) peut varier selon différents protocoles expérimentaux et la nature des matériaux cibles (protéine/peptide/ligands). - Couvrir la boîte de Petri en verre et incuber pendant 30 min à température ambiante. Aspirer la solution contenant la molécule cible et rincer 3x avec 50 μL de 1x PBS (pH = 7,4). Aspirer la solution 1x PBS (pH = 7,4) de la puce.
- Ajouter 50 μL de Solution Quench 1 (3,9 mM d’amino-PEG5 dans 1x PBS (pH = 7,4)) à la puce. Couvrir la boîte de Petri en verre et incuber pendant 15 min à température ambiante. Aspirer la solution Quench 1 de la puce.
- Ajouter 50 μL de solution Dench 2 (1 M d’éthanolamine (pH = 8,5)) à la puce. Couvrir la boîte de Petri en verre et incuber pendant 15 min à température ambiante. Aspirer la solution Quench 2 de la puce et rincer la puce 5x en utilisant 50 μL de 1x PBS, en laissant la dernière gouttelette PBS sur le capteur.
3. Préparation d’échantillons d’analytes
- Préparer la série de dilution de l’analyte pour Cdc37 dans la plage de concentration souhaitée. Pour la première expérience, les concentrations suivantes ont été utilisées : 25 nM, 50 nM, 100 nM, 200 nM, 400 nM, 800 nM, 1 000 nM, 2 000 nM, 3 000 nM et 5 000 nM. Pour la deuxième expérience, un ensemble différent de concentrations allant de 0,4 nM à 200 nM a été utilisé.
- Concevez l’expérience pour inclure au moins huit concentrations d’analyte différentes afin d’obtenir une valeur KD fiable. Préparer les différentes dilutions de la protéine analytique dans le même tampon que celui utilisé pour l’étalonnage et la protéine cible; ici, c’est 1x PBS (pH = 7,4).
4. Chargement de la puce de biocapteur activée dans le dispositif FEB
REMARQUE: Le dispositif FEB se compose d’un lecteur doté d’indications lumineuses LED et d’une cartouche pour insérer la puce de biocapteur.
- Après l’immobilisation de la protéine cible, insérez la puce activée dans la cartouche de l’appareil, qui est connectée via USB à un ordinateur. Après l’insertion de la puce, une lumière LED verte s’affichera sur le lecteur indiquant que l’appareil FEB est prêt pour l’expérience. Installez le logiciel automatisé (voir Tableau des matériaux) fourni par l’entreprise sur l’ordinateur auquel l’appareil FEB est connecté pour surveiller l’expérience étape par étape comme décrit ci-dessous.
5. Exécutez l’expérience
- Appuyez sur le module Exécuter l’expérience du logiciel automatisé et choisissez 10 points avec régénération ou tout autre protocole souhaité. Remplissez les détails suivants: nom de l’opérateur, nom de l’expérience, date (par exemple, Yana, Hsp90 + Cdc37, 14.03.2021); tampon de régénération (p. ex., tampon PBS); cible immobilisée (p. ex., Hsp90); analyte en solution (p. ex. Cdc37). Voir la figure supplémentaire S1 pour plus de détails.
- Appuyez sur le bouton Commencer l’expérience affiché sur le logiciel et suivez les instructions indiquées par le logiciel automatisé comme décrit ci-dessous.
REMARQUE: Le logiciel est entièrement automatisé, convivial et guide l’utilisateur tout au long de l’expérience étape par étape. Une fenêtre pop-up apparaîtra à l’écran avec des instructions pour aller plus loin à chaque étape de l’expérience. Le logiciel fournira des instructions pour chaque étape répétitive consécutivement à partir de l’étalonnage, de l’association de l’analyte, de la dissociation, de la régénération et du lavage (5x) pour chaque concentration d’analyte tout au long de l’expérience. - Effectuez l’étalonnage de l’instrument. Pour ce faire, aspirez la solution de PBS restante de la puce et appliquez 50 μL de tampon d’étalonnage (1x PBS; pH = 7,4). Appuyez sur le bouton Continuer et attendez 5 minutes jusqu’à ce que l’étape d’étalonnage soit terminée. Le logiciel affiche le point de terminaison déterminé pour l’étape d’étalonnage (5 min) avec une alarme d’avertissement pour le suivi.
- Ensuite, effectuez une association d’analyte. Pour ce faire, aspirez le tampon d’étalonnage de la puce et appliquez 50 μL de la concentration d’analyte la plus faible (25 nM de Cdc37). Appuyez sur le bouton Continuer et attendez 5 minutes jusqu’à ce que l’étape d’association soit terminée. Le logiciel affiche le point de terminaison de l’étape d’association (5 min) avec une alarme d’avertissement pour continuer.
- Effectuer une dissociation d’analyte. Pour ce faire, aspirer la solution d’analyte de la puce et appliquer 50 μL du tampon de dissociation (1x PBS; pH = 7,4). Appuyez sur le bouton Continuer et attendez 5 min jusqu’à ce que la durée de l’étape de dissociation (5 min) soit terminée. Le logiciel affiche le point de terminaison de l’étape de dissociation (5 min) avec une alarme d’avertissement pour le suivi.
- Ensuite, effectuez la régénération de la puce. Aspirer la solution de dissociation de la puce et appliquer 50 μL de tampon de régénération (1x PBS; pH = 7,4). Appuyez sur le bouton Continuer et attendez 30 s jusqu’à ce que la durée de l’étape de régénération (30 s) soit terminée. Le logiciel affiche le point de terminaison pour l’étape de régénération (30 s) avec une alarme d’avertissement pour le suivi.
- Enfin, lavez la puce. Aspirer la solution de régénération de la puce et appliquer 50 μL de tampon de lavage (1x PBS; pH = 7,4) sur la puce. Aspirez la solution de la puce et répétez ce 5x. Laissez la dernière goutte de tampon de lavage sur la puce et appuyez sur le bouton Continuer et attendez 30 s jusqu’à ce que la durée de l’étape de lavage soit terminée dans l’écran du logiciel.
REMARQUE: Le logiciel affiche le point de terminaison de l’étape de lavage (30 s) avec une alarme d’avertissement pour passer au cycle suivant de l’expérience. - Répétez les étapes pour chaque concentration d’analyte utilisée; les cinq étapes d’étalonnage, d’association d’analytes, de dissociation, de régénération et de lavage (5x) constituent un cycle. Pour l’expérience présentée ici, nous avons effectué 10 cycles pour 10 concentrations d’analytes (allant de 25 nM à 5 000 nM ou de 0,4 nM à 200 nM; Graphique 2).
6. Analyse
- Appuyez sur le bouton Analyse situé en haut du logiciel d’analyse automatisée à la fin de l’expérience. Une fenêtre d’affichage contenant tous les points expérimentaux apparaîtra. Dans la fenêtre, assurez-vous que les concentrations d’analyte utilisées pour le protocole prescrit sont correctes.
- Appuyez sur le bouton Exécuter l’analyse pour générer automatiquement la valeur KD . Le logiciel génère un diagramme d’ajustement de Hill en traçant les concentrations d’analyte par rapport aux I-Réponses correspondantes à partir desquelles la constante de dissociation à l’équilibre, valeur KD , est calculée.
- Exportez les données brutes pour analyse dans d’autres logiciels d’analyse statistique à l’aide du logiciel de révision/exportation de données décrit ci-dessous.
- Copiez le fichier R1R créé automatiquement à la fin de l’expérience (par exemple, Hsp90 + Cdc37 14.03.2021) dans un nouveau dossier sur le bureau. Ouvrez le logiciel data review/export fourni par l’entreprise (voir la figure supplémentaire S2A).
- Cliquez sur Traitement des données > Traiter les fichiers R1R > OK sur l’écran d’accueil du logiciel de révision/exportation de données.
- Sélectionnez le dossier contenant les fichiers R1R créés sur le bureau à l’étape 6.3.1. et appuyez sur le bouton OK . Cela crée une copie des données R1R d’origine, pour examiner et modifier les données sans remplacer le fichier d’origine.
- Appuyez sur l’icône de traitement des données affichée sur l’écran d’accueil du logiciel de révision/exportation des données. Appuyez sur Charger les fichiers R1R traités > OK. Sélectionnez le même dossier créé à l’étape 6.3.1 contenant les fichiers R1R traités. Appuyez sur OK. À cette étape, le dossier contenant les fichiers d’expérience est prêt à être examiné.
- Sur l’écran d’accueil du logiciel de révision/exportation de données, appuyez sur Analyse des données. Sélectionnez Calibration > Calibrer les fichiers R1R modifiés > OK (voir figure supplémentaire S2B). Cette étape étalonne tous les points de données en fonction de la première étape d’étalonnage, pour créer une ligne de base.
- Sur l’écran d’accueil du logiciel de révision/exportation de données, appuyez sur Analyse des données. Sélectionnez Vérifier et modifier les fichiers R1R > OK. Passez en revue les points de données, supprimez des points ou ajoutez des étapes à l’aide du bouton Supprimer/Ajouter une étape . Assurez-vous que toutes les étapes sont au bon endroit, par exemple, l’étape d’étalonnage est sur la ligne de base, l’étape d’association est au sommet (voir figure supplémentaire S3), puis appuyez sur le bouton Enregistrer pour enregistrer toutes les modifications apportées.
- Sur l’écran d’accueil du logiciel de révision/exportation de données, appuyez sur Analyse des données. Sélectionnez Analyser/Tracer les données, choisissez les étapes à exporter (voir figure supplémentaire S4). Ajoutez chaque étape d’association (par exemple, étape 2, étape 7, etc.) après avoir soustrait l’étape d’étalonnage (par exemple, étape 1, étape 6, etc.) à la liste d’exportation avant d’exporter les données.
- Appuyez sur Exporter ces données. Le logiciel génère un fichier de feuille de calcul contenant la réponse I pour chaque point de données de concentration d’analyte de chaque transistor (si tous les transistors fonctionnent, nous aurons trois valeurs de réponse I différentes pour chaque point de concentration). Utilisez ce fichier de feuille de calcul pour analyser plus en détail les données dans le logiciel statistique.
- Ouvrez le logiciel d’analyse statistique. Créez une table XY avec trois valeurs Y ; appuyez sur Créer. Les données de cette table (valeurs X, Y) sont copiées à partir du fichier de feuille de calcul créé à l’étape 6.3.8. L’axe des x correspond à la concentration de l’analyte (selon la concentration utilisée), et les trois valeurs Y correspondent aux réponses I, obtenues à partir du logiciel d’examen/exportation de données fourni par l’entreprise.
- Appuyez sur Analyser ces données. Choisissez analyses XY > régression non linéaire (ajustement de la courbe) > saturation de liaison > un site > total. Le logiciel analysera automatiquement les données, générera la valeur KD et créera un graphique des points de données.
Access restricted. Please log in or start a trial to view this content.
Résultats
Résultats de l’expérience 1 :
La protéine cible Hsp90 (500 nM) a été immobilisée sur la puce en suivant le protocole d’immobilisation cible décrit ci-dessus. Pour la première expérience, 10 concentrations de la protéine analytique Cdc37, allant de 25 nM à 5 000 nM, ont été préparées sur la base des données disponibles dans la littérature (voir tableau 1).
Les étapes de l’expérience peuvent être surveillées en temps réel en suivan...
Access restricted. Please log in or start a trial to view this content.
Discussion
Dans cette étude, la faisabilité de l’utilisation de la technologie FEB (une approche de caractérisation cinétique en temps réel) a été évaluée pour déterminer l’interaction biomoléculaire entre Hsp90 et Cdc37. L’expérience exploratoire initiale (première expérience) suggérait que le choix des concentrations d’analyte appropriées est une partie essentielle de l’expérience et que l’expérience devrait être conçue en incluant des points de concentration au-dessus et au-dessous de la valeur K<...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts, financier ou autre.
Remerciements
Cette recherche a été soutenue par une subvention de la Binational Science Foundation (BSF) à S.K.S. et N.Q.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| Automated analysis software | Agile plus software, Cardea (Nanomed) | NA CAS number: NA | Referred to in the text as the automated analysis software supplied with the instrument. Generates automated analysis. |
| COOH-BPU (Biosensing Processing Unit) | Agile plus software, Cardea (Nanomed) | NA CAS number: NA | biosensor chip |
| Data review software | Datalign 1.0, Cardea (Nanomed) | NA CAS number: NA | Referred to as the supplied data review software in the text. Supplied with the instrument and allows to review and export the information data points. |
| Dialysis bag | CelluSep, Membrane filtration products | T2-10-15 CAS number: NA | T2 tubings (6,000-8,000 MWCO), (10 mm fw, 6.4mm Ø, 0.32ml/cm, 15m) |
| EDC (1-Ethyl-3-(3-dimethylamino propyl) carbodiimide) | Cardea (Nanomed) | EDC160322-02 CAS number: 25952-53-8 | White powder |
| ITC (Isothermal titration calorimetry) system | Microcal-PEAQ-ITC (Malvern, United Kingdom) | NA CAS number: NA | |
| MES (2-(N-morpholino) ethane sulfonic acid) buffer | Merck | M3671-50G CAS number: 4432-31-9 | White powder |
| NHS (N-Hydroxysulfosuccinimide) chips | Cardea (Nanomed) | NA CAS number: NA | Graphene-based chip |
| PBS (Phosphate-buffered saline) X 10 | Bio-Lab | 001623237500 CAS number: 7758-11-4 | Liquid transparent solution |
| Pipete | Thermo Scientific | 11855231 CAS number: NA | Finnpipette F3 5-50 µL, yellow |
| Quench 1 (3.9 mM amino-PEG5-alcohol in 1 X PBS) | Cardea (Nanomed) | 0105-001-002-001 CAS number: NA | Liquid, transparent solution |
| Quench 2 (1 M ethanolamine (pH=8.5)) | Cardea (Nanomed) | 0105-001-003-001 CAS number: NA | Liquid, transparent solution |
| Recombinant protein Cdc37 | Abcam | ab256157 CAS number: NA | |
| Recombinant protein Hsp90 beta | Abcam | ab80033 CAS number: NA | |
| Spreadsheet | Excel, Microsoft office | NA CAS number: NA | |
| Statistical software | GraphPad, Prism | NA CAS number: NA | Referred to as the other statistical software. Sigma plot, phyton or other statistical programes may also be used |
| Sulfo-NHS | Cardea (Nanomed) | NHS160321-07 CAS number: 106627-54-7 | White powder |
| Tips | Alex red | LC 1093-800-000 CAS number: NA | Tip 1-200 µl, in bulk, 1,000 pcs |
Références
- Tuncbag, N., Gursoy, A., Guney, E., Nussinov, R., Keskin, O. Architectures and functional coverage of protein-protein interfaces. Journal of Molecular Biology. 381 (3), 785-802 (2008).
- Berggård, T., Linse, S., James, P. Methods for the detection and analysis of protein–protein interactions. Proteomics. 7 (16), 2833-2842 (2007).
- Magliery, T. J., et al. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: Scope and mechanism. Journal of the American Chemical Society. 127 (1), 146-157 (2005).
- Xing, S., Wallmeroth, N., Berendzen, K. W., Grefen, C. Techniques for the analysis of protein-protein interactions in vivo. Plant Physiology. 171 (2), 727-758 (2016).
- Nguyen, T. N., Goodrich, J. A. Protein-protein interaction assays: Eliminating false positive interactions. Nature Methods. 3 (2), 135-139 (2006).
- Fernández-Suárez, M., Chen, T. S., Ting, A. Y. Protein-protein interaction detection in vitro and in cells by proximity biotinylation. Journal of the American Chemical Society. 130 (29), 9251-9253 (2008).
- Jiang, M., Niu, C., Cao, J., Ni, D. -A., Chu, Z. In silico-prediction of protein–protein interactions network about MAPKs and PP2Cs reveals a novel docking site variants in Brachypodium distachyon. Scientific Reports. 8 (1), 15083(2018).
- Yazawa, M., Sadaghiani, A. M., Hsueh, B., Dolmetsch, R. E. Induction of protein-protein interactions in live cells using light. Nature Biotechnology. 27 (10), 941-945 (2009).
- Wang, W., Goodman, M. T. Antioxidant property of dietary phenolic agents in a human LDL-oxidation ex vivo model: Interaction of protein binding activity. Nutrition Research. 19 (2), 191-202 (1999).
- Xing, S., Wallmeroth, N., Berendzen, K. W., Grefen, C. Techniques for the analysis of protein-protein interactions in vivo. Plant Physiology. 171 (2), 727-758 (2016).
- Qvit, N., Disatnik, M. -H., Sho, E., Mochly-Rosen, D. Selective phosphorylation inhibitor of delta protein kinase C–Pyruvate dehydrogenase kinase protein–protein interactions: Application for myocardial injury in vivo. Journal of the American Chemical Society. 138 (24), 7626-7635 (2016).
- Alam, M. N., Bristi, N. J., Rafiquzzaman, M. Review on in vivo and in vitro methods evaluation of antioxidant activity. Saudi Pharmaceutical Journal. 21 (2), 143-152 (2013).
- Paulmurugan, R., Gambhir, S. S. Novel fusion protein approach for efficient high-throughput screening of small molecule–mediating protein-protein interactions in cells and living animals. Cancer Research. 65 (16), 7413-7420 (2005).
- Boute, N., Jockers, R., Issad, T. The use of resonance energy transfer in high-throughput screening: BRET versus FRET. Trends in Pharmacological Sciences. 23 (8), 351-354 (2002).
- Deriziotis, P., Graham, S. A., Estruch, S. B., Fisher, S. E. Investigating protein-protein interactions in live cells using bioluminescence resonance energy transfer. Journal of Visualized Experiments: JoVE. (87), e51438(2014).
- Ekins, S., Mestres, J., Testa, B. In silico pharmacology for drug discovery: Methods for virtual ligand screening and profiling. British Journal of Pharmacology. 152 (1), 9-20 (2007).
- Valerio, L. G. Application of advanced in silico methods for predictive modeling and information integration. Expert Opinion on Drug Metabolism & Toxicology. 8 (4), 395-398 (2012).
- Piehler, J. New methodologies for measuring protein interactions in vivo and in vitro. Current Opinion in Structural Biology. 15 (1), 4-14 (2005).
- Ideker, T., Sharan, R. Protein networks in disease. Genome Research. 18 (4), 644-652 (2008).
- Lu, H., et al. Recent advances in the development of protein–protein interactions modulators: mechanisms and clinical trials. Signal Transduction and Targeted Therapy. 5 (1), 213(2020).
- Jarosz, D. Hsp90: A global regulator of the genotype-to-phenotype map in cancers. Advances in Cancer Research. 129, 225-247 (2016).
- Johnson, V. A., Singh, E. K., Nazarova, L. A., Alexander, L. D., McAlpine, S. R. Macrocyclic inhibitors of Hsp90. Current Topics in Medicinal Chemistry. 10 (14), 1380-1402 (2010).
- Mahalingam, D., et al. Targeting HSP90 for cancer therapy. British Journal of Cancer. 100 (10), 1523-1529 (2009).
- Stewart, A., Fisher, R. A. Co-Immunoprecipitation: Isolation of protein signaling complexes from native tissues. Methods in Cell Biology. 112, 33-54 (2012).
- Pierce, M. M., Raman, C. S., Nall, B. T. Isothermal titration calorimetry of protein-protein interactions. Methods. 19 (2), San Diego, Calif. 213-221 (1999).
- Paketurytė, V., et al. Inhibitor binding to carbonic anhydrases by isothermal titration calorimetry. Carbonic Anhydrase as Drug Target. , Springer International Publishing. 79-95 (2019).
- Grote, J., Dankbar, N., Gedig, E., Koenig, S. Surface plasmon resonance/mass spectrometry interface. Analytical Chemistry. 77 (4), 1157-1162 (2005).
- Kumaraswamy, S., Tobias, R. Label-free kinetic analysis of an antibody–antigen interaction using biolayer interferometry. Methods in Molecular Biology. , 165-182 (2015).
- Wallner, J., Lhota, G., Jeschek, D., Mader, A., Vorauer-Uhl, K. Application of bio-layer interferometry for the analysis of protein/liposome interactions. Journal of Pharmaceutical and Biomedical Analysis. 72, 150-154 (2013).
- Singh, A. N., Ramadan, K., Singh, S. Experimental methods to study the kinetics of protein–protein interactions. Advances in Protein Molecular and Structural Biology Methods. , 115-124 (2022).
- Frenzel, D., Willbold, D. Kinetic titration series with biolayer interferometry. PLoS One. 9 (9), 106882(2014).
- Vu, C. -A., Chen, W. -Y. Field-effect transistor biosensors for biomedical applications: Recent advances and future prospects. Sensors. 19 (19), 4214(2019).
- Bergveld, P. A critical evaluation of direct electrical protein detection methods. Biosensors & Bioelectronics. 6 (1), 55-72 (1991).
- Lowe, B. M., Sun, K., Zeimpekis, I., Skylaris, C. K., Green, N. G. Field-effect sensors – from pH sensing to biosensing: sensitivity enhancement using streptavidin–biotin as a model system. The Analyst. 142 (22), 4173-4200 (2017).
- Goldsmith, B. R., et al. Digital biosensing by foundry-fabricated graphene sensors. Scientific Reports. 9 (1), 434(2019).
- Afsahi, S., et al. Novel graphene-based biosensor for early detection of Zika virus infection. Biosensors and Bioelectronics. 100, 85-88 (2018).
- Afsahi, S. J., et al. Towards novel graphene-enabled diagnostic assays with improved signal-to-noise ratio. MRS Advances. 2 (60), 3733-3739 (2017).
- Roe, S. M., et al. The mechanism of Hsp90 regulation by the protein kinase-specific cochaperone p50cdc37. Cell. 116 (1), 87-98 (2004).
- Gaiser, A. M., Kretzschmar, A., Richter, K. Cdc37-Hsp90 complexes are responsive to nucleotide-induced conformational changes and binding of further cofactors. The Journal of Biological Chemistry. 285 (52), 40921-40932 (2010).
- Popescu, A. I., Găzdaru, D. M., Chilom, C. G., Bacalum, M. Biophysical interactions: Their paramount importance for life. Romanian Reports in Physics. 65 (3), 1063-1077 (2013).
- Surya, S., Abhilash, J., Geethanandan, K., Sadasivan, C., Haridas, M. A profile of protein-protein interaction: Crystal structure of a lectin-lectin complex. International Journal of Biological Macromolecules. 87, 529-536 (2016).
- Velazquez-Campoy, A., Freire, E. ITC in the post-genomic era...? Priceless. Biophysical Chemistry. 115 (23), 115-124 (2005).
- Concepcion, J., et al. Label-free detection of biomolecular interactions using bioLayer interferometry for kinetic characterization. Combinatorial Chemistry & High Throughput Screening. 12 (8), 791-800 (2009).
- Helmerhorst, E., Chandler, D. J., Nussio, M., Mamotte, C. D. Real-time and label-free bio-sensing of molecular interactions by surface plasmon resonance: A laboratory medicine perspective. The Clinical Biochemist. Reviews. 33 (4), 161-173 (2012).
- Jacob, N. T., et al. Synthetic molecules for disruption of the MYC protein-protein interface. Bioorganic & Medicinal Chemistry. 26 (14), 4234-4239 (2018).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon