A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
ספקטרוסקופיית נייטרונים ברזולוציה גבוהה לחקר דינמיקה של פיקו-שנייה-ננו-שנייה של חלבונים ומי לחות
In This Article
Summary
ספקטרוסקופיית פיזור אחורי של נייטרונים מציעה גישה לא הרסנית ונטולת תוויות לדינמיקה של ps-ns של חלבונים ומי ההידרציה שלהם. תהליך העבודה מוצג עם שני מחקרים על חלבוני עמילואיד: על הדינמיקה שנפתרה בזמן של ליזוזים במהלך צבירה ועל דינמיקת מי ההידרציה של טאו על היווצרות סיבים.
Abstract
פיזור נייטרונים מציע את האפשרות לחקור את הדינמיקה בתוך הדגימות עבור מגוון רחב של אנרגיות באופן לא הרסני וללא תיוג מלבד דאוטריום. בפרט, ספקטרוסקופיית פיזור אחורי של נייטרונים מתעדת את אותות הפיזור במספר זוויות פיזור בו זמנית ומתאימה היטב לחקר הדינמיקה של מערכות ביולוגיות בסקאלת הזמן ps-ns. על ידי שימוש ברכיבי D2O - ואולי גם רכיבי חיץ מפורקים - השיטה מאפשרת ניטור הן של דיפוזיה של מרכז המסה והן של תנועות עמוד השדרה והשרשרת הצידית (דינמיקה פנימית) של חלבונים במצב נוזלי.
בנוסף, ניתן לחקור את דינמיקת מי ההידרציה על ידי שימוש באבקות של חלבונים מחוררים עם H2O. מאמר זה מציג את תהליך העבודה שהופעל במכשיר IN16B במכון Laue-Langevin (ILL) כדי לחקור דינמיקה של חלבונים ומי הידרציה. הכנת דגימות תמיסה ודגימות אבקת חלבון hydrated באמצעות חילופי אדים מוסבר. הליך ניתוח הנתונים עבור דינמיקת מים של חלבונים והידרציה מתואר עבור סוגים שונים של מערכי נתונים (ספקטרום קוואזיאלסטי או סריקות חלונות קבועים) שניתן לקבל על ספקטרומטר פיזור אחורי של נייטרונים.
השיטה מודגמת באמצעות שני מחקרים שכללו חלבוני עמילואיד. הצבירה של ליזוזים לצברים כדוריים בגודל מיקרומטר - חלקיקים מסומנים - מתרחשת בתהליך בן שלב אחד על טווח המרחב והזמן שנבחן על IN16B, בעוד שהדינמיקה הפנימית נותרת ללא שינוי. יתר על כן, הדינמיקה של מי הידרציה של טאו נחקרה על אבקות לחות של חלבון perdeuterated. זה מראה כי תנועות תרגום של מים מופעלות על היווצרות של סיבי עמילואיד. לבסוף, נדונים צעדים קריטיים בפרוטוקול לגבי האופן שבו פיזור נייטרונים ממוקם ביחס לחקר הדינמיקה ביחס לשיטות ביופיזיקליות ניסיוניות אחרות.
Introduction
הנייטרון הוא חלקיק מסיבי וחסר מטען ששימש בהצלחה לאורך השנים לבדיקת דגימות בתחומים שונים, מפיזיקה בסיסית ועד ביולוגיה1. עבור יישומים ביולוגיים, פיזור נייטרונים בזווית קטנה, פיזור נייטרונים בלתי אלסטי, קריסטלוגרפיה ורפלקטומטריה של נייטרונים נמצאים בשימוש נרחב 2,3,4. פיזור נייטרונים קשיח מספק מדידה ממוצעת של הדינמיקה ללא צורך בתיוג ספציפי כשלעצמו, ואיכות אות שאינה תלויה בגודל או בחלבון5. המדידה יכולה להיעשות באמצעות סביבה מורכבת מאוד עבור החלבון הנחקר המחקה את התווך התוך-תאי, כגון ליזט חיידקי או אפילו in vivo 3,6,7. ניתן להשתמש במערכי ניסוי שונים כדי לחקור את הדינמיקה, כלומר i) גישה של זמן טיסה לדינמיקות sub-ps-ps, ii) גישה המעניקה פיזור לאחור לדינמיקות ps-ns, ו-iii) גישה נותנת ספין-הד לדינמיקה מ-ns למאות ns. פיזור אחורי של נייטרונים עושה שימוש בחוק בראג 2d sinθ = nλ, כאשר d הוא המרחק בין מישורים בגביש, θ זווית הפיזור, n סדר הפיזור ו-λ אורך הגל. השימוש בגבישים לפיזור לאחור לכיוון הגלאים מאפשר השגת רזולוציה גבוהה באנרגיה, בדרך כלל ~0.8 μeV. כדי למדוד את חילופי האנרגיה, משתמשים בכונן דופלר הנושא גביש בפיזור לאחור כדי להגדיר ולכוונן את אורך גל הנייטרונים הנכנס 8,9,10 (איור 1), או שניתן להשתמש במערך זמן טיסה במחיר של ירידה ברזולוציית האנרגיה 11.
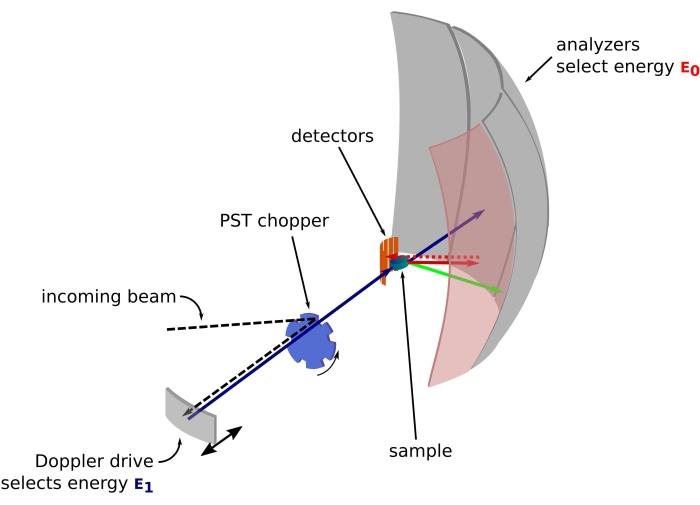
איור 1: שרטוט של ספקטרומטר פיזור לאחור של נייטרונים עם כונן דופלר. הקרן הנכנסת פוגעת במסוק טרנספורמציית חלל הפאזה (PST)42, המגדיל את השטף במיקום הדגימה. לאחר מכן הוא מפוזר לאחור לכיוון הדגימה על ידי כונן דופלר, אשר בוחר אנרגיה E1 (חץ ציאן). לאחר מכן הנייטרונים מפוזרים על ידי הדגימה (עם אנרגיות שונות המיוצגות על ידי צבע החיצים) והאנלייזרים, העשויים מגבישי Si 111, יפזרו לאחור נייטרונים רק עם אנרגיה ספציפית E0 (חיצים בצבע אדום כאן). לפיכך, העברת התנע q מתקבלת מהמיקום שזוהה של הנייטרון על מערך הגלאים, והעברת האנרגיה מתקבלת מההפרש E1- E0. זמן הטיסה הצפוי לפעימת הנייטרונים המופקת על ידי ה-PST משמש להשלכת האות מהנייטרונים המפוזרים ישירות לעבר צינורות הגלאי. קיצור: PST = טרנספורמציה של מרחב פאזה. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
עבור ספקטרוסקופיית פיזור לאחור, התרומה העיקרית לאות מדגימות עשירות בפרוטוני מימן, כגון חלבונים, מגיעה מפיזור לא קוהרנטי, שעבורו עוצמת הפיזור Sinc(q, ω) מוצגת על ידי Eq (1)12
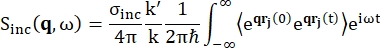 (1)
(1)
כאשר σinc הוא חתך הרוחב הלא קוהרנטי של היסוד הנחשב, k' הוא הנורמה של וקטור הגל המפוזר, k הנורמה של וקטור הגל הנכנס, q (= k - k') העברת התנע, r j(t) וקטור המיקום של אטום j בזמן t, ו-ω התדר המתאים למעבר האנרגיה בין הנייטרון הנכנס למערכת. הסוגריים הזוויתיים מציינים את ממוצע ההרכב. לפיכך, פיזור לא קוהרנטי בוחן את המתאם העצמי הממוצע של חלקיק בודד של מיקומי אטומים עם זמן ונותן את הדינמיקה העצמית הממוצעת על פני כל האטומים במערכת ומקורות זמן שונים (ממוצע אנסמבל). פונקציית הפיזור היא התמרת פורייה בזמן של פונקציית פיזור הביניים I(q, t), שניתן לראותה כהתמרת פורייה במרחב של פונקציית מתאם ואן הוב המוצגת על ידי Eq (2):
 (2)
(2)
כאשר ρ(r,t) היא צפיפות ההסתברות למציאת אטום במיקום r ובזמן t 13.
עבור תהליך דיפוזיה פיקיאנית, פונקציית הדיפוזיה העצמית נוצרת (ראו Eq (3)) לאחר התמרת פורייה כפולה בפונקציית פיזור המורכבת מלורנציאן של רוחב קו הנתון על ידי γ = Dq2.
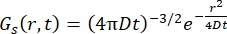 (3)
(3)
מודלים מתוחכמים יותר פותחו ונמצאו שימושיים כגון מודל דיפוזיית הקפיצה על ידי Singwi ו- Sjölander עבור ps-ns דינמיקה פנימית של חלבונים14 או מודל הסיבוב על ידי סירס עבור מי הידרציה15,16,17.
במכשיר פיזור אחורי של נייטרונים (NBS) IN16B8,9 ב-ILL, גרנובל, צרפת (איור משלים S1), מערך נפוץ עם חלבונים מורכב מגבישי Si 111 עבור האנלייזרים עם כונן דופלר לכוונון אורך הגל הנכנס (איור משלים S2A), ובכך נותן גישה לטווח העברת התנע ~0.2 Å-1 < q < ~2 Å-1 וטווח העברת אנרגיה של -30 μeV < 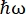 < 30 μeV - מתאים לטווחי זמן הנעים בין כמה ps לכמה ns ומרחקים של כמה Å. בנוסף, IN16B מציע את האפשרות לבצע סריקות גמישות ובלתי אלסטיות של חלונות קבועים (E/IFWS)10, הכוללות איסוף נתונים בהעברת אנרגיה קבועה. מכיוון שהשטף מוגבל בעבודה עם נייטרונים, E/IFWS מאפשר למקסם את השטף להעברת אנרגיה אחת, ובכך מקצר את זמן הרכישה הדרוש לקבלת יחס אות לרעש מספק. אפשרות עדכנית יותר היא מצב ספקטרומטר פיזור לאחור וזמן טיסה (BATS)11, המאפשר מדידה של מגוון רחב של העברות אנרגיה, (למשל, -150 μeV < <
< 30 μeV - מתאים לטווחי זמן הנעים בין כמה ps לכמה ns ומרחקים של כמה Å. בנוסף, IN16B מציע את האפשרות לבצע סריקות גמישות ובלתי אלסטיות של חלונות קבועים (E/IFWS)10, הכוללות איסוף נתונים בהעברת אנרגיה קבועה. מכיוון שהשטף מוגבל בעבודה עם נייטרונים, E/IFWS מאפשר למקסם את השטף להעברת אנרגיה אחת, ובכך מקצר את זמן הרכישה הדרוש לקבלת יחס אות לרעש מספק. אפשרות עדכנית יותר היא מצב ספקטרומטר פיזור לאחור וזמן טיסה (BATS)11, המאפשר מדידה של מגוון רחב של העברות אנרגיה, (למשל, -150 μeV < < 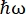 150 μeV), עם שטף גבוה יותר מאשר עם כונן דופלר, אך במחיר של רזולוציית אנרגיה נמוכה יותר (איור משלים S2B).
150 μeV), עם שטף גבוה יותר מאשר עם כונן דופלר, אך במחיר של רזולוציית אנרגיה נמוכה יותר (איור משלים S2B).
תכונה חשובה של פיזור נייטרונים היא שחתך הרוחב הלא קוהרנטי σinc הוא בעל ערך גבוה פי 40 עבור מימן מאשר עבור דאוטריום, והוא זניח עבור יסודות אחרים הנמצאים בדרך כלל בדגימות ביולוגיות. לכן, ניתן לחקור את הדינמיקה של חלבונים בסביבה נוזלית באמצעות מאגר deuterated, ומצב האבקה מאפשר לחקור דינמיקה פנימית של חלבונים עם אבקת חלבון מוקשה hydrated עם D 2 O, או המחקר של מים הידרציה עבוראבקת חלבון perdeuterated hydrated עם H2O. במצב נוזלי, פיזור אחורי של נייטרונים מאפשר בדרך כלל גישה בו זמנית לדיפוזיה העצמית של מרכז המסה של חלבונים (דיפוזיה מסוג פיקיאן) ולדינמיקה הפנימית שלהם. אלה האחרונים הם תנועות עמוד השדרה ושרשרת הצד המתוארות בדרך כלל על ידי מה שנקרא מודל דיפוזיה קפיצה או אחרים 3,18. באבקות חלבון מוקשה, דיפוזיית החלבון נעדרת ויש למדל רק דינמיקה פנימית. עבור מי הידרציה, התרומות של תנועות תרגומיות וסיבוביות של מולקולות מים מציגות תלות שונה בהעברת התנע q, המאפשרת את ההבחנה ביניהן בתהליך ניתוח הנתונים17.
מאמר זה מדגים את שיטת הפיזור לאחור של נייטרונים באמצעות מחקר של חלבונים שנמצאו מסוגלים להתפתח, להצטבר לצורה קנונית המורכבת מערימות של β-גדילים - מה שמכונה תבנית β צולבת19,20 - וליצור סיבים מוארכים. זהו מה שנקרא צבירה עמילואיד, אשר נחקר בהרחבה בשל תפקידו המרכזי בהפרעות נוירודגנרטיביות כגון אלצהיימר או פרקינסון21,22. המחקר של חלבוני העמילואיד מונע גם על ידי התפקיד הפונקציונלי שהם יכולים למלא 23,24 או הפוטנציאל הגבוה שלהם לפיתוח ביו-חומרים חדשים25. הדטרמיננטים הפיזיקוכימיים של צבירת העמילואיד עדיין אינם ברורים, ואין תיאוריה כללית של צבירת עמילואיד, למרות התקדמות עצומה במהלך השנים האחרונות21,26.
צבירת עמילואיד מרמזת על שינויים במבנה החלבון וביציבותו עם הזמן, שהמחקר שלהם מרמז באופן טבעי על דינמיקה, הקשורה ליציבות קונפורמציה של חלבונים, תפקוד חלבונים ונוף אנרגיית חלבון27. דינמיקה קשורה ישירות ליציבות של מצב מסוים באמצעות התרומה האנטרופית עבור התנועות המהירות ביותר28, ותפקוד החלבונים יכול להתקיים על ידי תנועות בסקאלות זמן שונות, החל מ-sub-ps עבור חלבונים רגישים לאור29 ועד ms עבור תנועות תחום, אשר ניתן להקל על ידי דינמיקה פיקו-שנייה-ננו-שנייה30.
יוצגו שתי דוגמאות לשימוש בספקטרוסקופיית פיזור לאחור של נייטרונים לחקר חלבוני עמילואיד, אחת במצב נוזלי לחקר דינמיקה של חלבונים ואחת במצב אבקה רוויית לחות לחקר דינמיקת מי הידרציה. הדוגמה הראשונה נוגעת לצבירה של ליזוזים לכדורים בגודל מיקרומטר (הנקראים חלקיקים) ואחריה בזמן אמת5, והשנייה השוואה של דינמיקת מים במצבים טבעיים ומצטברים של החלבון האנושי טאו31.
ליזוזים הוא אנזים המעורב בהגנה החיסונית ומורכב משאריות של 129 חומצות אמינו. ליזוזים יכול ליצור חלקיקים בחיץ מפורק ב pD של 10.5 ובטמפרטורה של 90 ° C. עם פיזור נייטרונים, הראינו כי התפתחות הזמן של מקדם הדיפוזיה של מרכז המסה של ליזוזים עוקבת אחר הקינטיקה המעריכית היחידה של תיאופלבין T פלואורסצנטי (בדיקה פלואורסצנטית המשמשת לניטור היווצרות תבניות β צולבות עמילואיד32), מה שמצביע על כך שהיווצרות מבני על חלקיקיים ותבניות β צולבות מתרחשות בשלב אחד עם אותו קצב. יתר על כן, הדינמיקה הפנימית נשארה קבועה לאורך כל תהליך הצבירה, אשר ניתן להסביר או על ידי שינוי קונפורמציה מהיר שלא ניתן לצפות בו על מכשירי NBS, או על ידי היעדר שינוי משמעותי באנרגיה הפנימית של החלבונים בעת הצבירה.
החלבון האנושי טאו הוא חלבון בעל הפרעה מהותית (IDP) המורכב מ-441 חומצות אמינו עבור מה שמכונה איזופורם 2N4R, אשר מעורב באופן בולט במחלת אלצהיימר33. באמצעות פיזור לאחור של נייטרונים על אבקות של חלבון טאו מפורר, הראינו כי דינמיקת מי הידרציה מוגברת במצב הסיבים, כאשר אוכלוסייה גבוהה יותר של מולקולות מים עוברות תנועות תרגומיות. התוצאה מצביעה על כך שעלייה באנטרופיית מי הידרציה עשויה להניע את פרפור העמילואיד של טאו.
Protocol
1. הכינו את החיץ המנוטרל לחלבונים במצב נוזלי
- ממיסים את כל רכיבי המאגר ב-D2O טהור.
- אם אלקטרודת ה- pH כוילתה ב- H2O, התאם את ה- pD בהתאם לנוסחה pD = pH + 0.4 באמצעות NaOD או DCl34.
הערה: השימוש ב-D 2O במקום ב-H2O עשוי להשפיע על מסיסות החלבון, וייתכן שיהיה צורך להתאים את תנאי החיץ (למשל, על ידי שינוי קל בריכוז המלח).
2. הכינו את האבקות H2O-hydrated של חלבון perdeuterated
- הכינו את מחזיק המדגם.
- נקו היטב מחזיק דגימת אלומיניום שטוח עם חותם חוט אינדיום וברגים עם מים ואתנול ותנו לו להתייבש.
הערה: נעשה שימוש במחזיק מדגם שטוח כך שניתן לפזר את האבקה באופן הומוגני על פני השטח. כמות האבקה צריכה להספיק כך שניתן יהיה לשמור עליה בין הקירות ולא תיפול כאשר מחזיק הדגימה ממוקם אנכית. - שקול את החלקים השונים של מחזיק הדגימה - תחתית, מכסה וחוט אינדיום - בנפרד על איזון מדויק.
- הניחו את אטם חוט האינדיום בקוטר 1 מ"מ בחריץ של החלק התחתון של מחזיק הדגימה, והשאירו חפיפה קטנה במקום שבו שני הקצוות מתחברים (איור 2A).
- הניחו כמות מתאימה של חלבון ליופילי (בדרך כלל ~ 100 מ"ג חלבון) כך שהוא ימלא את המשטח הפנימי של החלק התחתון של מחזיק הדגימה.
- נקו היטב מחזיק דגימת אלומיניום שטוח עם חותם חוט אינדיום וברגים עם מים ואתנול ותנו לו להתייבש.
- הוסיפו לחות לאבקת החלבון.
- הניחו את מחזיק הדגימה במייבש עם צלחת פטרי המכילה אבקת P2O5 למשך 24 שעות כדי לייבש לחלוטין את אבקת החלבון35 (איור 2B). שקלו את החלק התחתון היבש של מחזיק הדגימה המכיל את חותם האינדיום ואת האבקה היבשה כדי לקבל mיבש.
זהירות: אבקת P2O5 היא קורוזיבי מאוד. - הסר את P 2 O5 מן מייבש ולשים צלחת פטרי עם D2O בפנים. לשלוט על המסה של האבקה באופן קבוע כדי לבדוק את רמת הידרציה h = m hyd / m יבש שבו mhyd ו mיבש הם המסה של אבקתhydrated אבקה יבשה, בהתאמה.
הערה: עבור חלבונים הידרופוביים מאוד כגון אינסולין, ייתכן שיהיה צורך להגדיל את הטמפרטורה בתוך מייבש כדי לקבל לחץ אדים גבוה יותר ולהגיע לרמת ההידרציה הרצויה h. - חזור על שלבים 2.2.1 ו- 2.2.2 לפחות שלוש פעמים כדי להמיר כראוי את כל המימנים הניתנים להחלפה לדאוטרונים.
הערה: לחלופין, מחזורים של ייבוש בהקפאה והמסה ב-D2O טהור עשויים לשמש להחלפת H/D טובה יותר, בתנאי שהחלבון אינו מושפע מכך. - לחות את האבקה מעט מעל לרמה הרצויה, לתת לחלק התחתון של מחזיק הדגימה עם חוט אינדיום ואבקה hydrated להישאר על איזון דיוק, ולחכות המסה לרדת לאט לערך הרצוי כדי לקבל את היעד h (בדרך כלל 0.2-0.4 אם חלבון כדורי בגודל בינוני הוא להיות מכוסה על ידי שכבת הידרציה אחת שלמה).
- הניחו במהירות את המכסה על החלק התחתון וסגרו תחילה את מחזיק הדגימה עם ארבעה ברגים כדי לעצור את חילוף האדים (איור משלים S3A).
- הניחו והדקו את כל הברגים הנותרים עד שלא ייראה רווח בין החלק התחתון למכסה (איור משלים S3B).
- שקלו את מחזיק הדגימה האטום כדי לבדוק אם יש אובדן הידרציה פוטנציאלי כתוצאה מדליפות לאחר ניסוי הנייטרונים.
- הניחו את מחזיק הדגימה במייבש עם צלחת פטרי המכילה אבקת P2O5 למשך 24 שעות כדי לייבש לחלוטין את אבקת החלבון35 (איור 2B). שקלו את החלק התחתון היבש של מחזיק הדגימה המכיל את חותם האינדיום ואת האבקה היבשה כדי לקבל mיבש.
3. בצע את ניסוי פיזור הנייטרונים הלא קוהרנטי
- לדון ולבדוק שוב את תצורת המכשיר הדרוש לניסוי עם איש הקשר המקומי כמה שבועות לפני זמן הקרן שהוקצה.
- הכן את דגימת המצב הנוזלי.
- ממיסים את החלבון במאגר המפורק.
- לקבוע את נפח הנוזל המתאים שיש להכניס למחזיק הדגימה באמצעות מים (יש לוודא שאין הצפה כאשר מחזיק הדגימה סגור; איור 2C).
הערה: השלבים הבאים (3.3 ו-3.4) מתארים ניסוי שנערך על ספקטרומטר NBS IN16B ב-ILL8,9, תוך שימוש בתנור קריופורנס כסביבת דגימה. מערכת בקרת המכשירים תשתנה ממכשיר אחד למשנהו, אך עקרונות העבודה יישארו זהים.
- הכנס את הדגימה.
- יבשו היטב את מקל הדגימה (איור 2D), והוציאו את הדגימה הקודמת, אם קיימת, לאחר שבדקתם שמנת הקרינה המייננת נמוכה מ-100 μSv/h לפני הטיפול בחומר כלשהו (ב-ILL).
- הניחו את הדגימה, בדקו אם יש מרכוז נכון ביחס למרכז הקרן (איור משלים S4), והכניסו את מוט הדגימה לתנור ההקפאה (איור 2D). הפעל את משאבת הוואקום כדי להגיע לפחות מ 10-3 בר, ולשטוף את האוויר בתוך תנור ההקפאה על ידי חזרה על שלוש הפעמים הבאות: מלא את התנור בגז הליום עד שיגיע ללחץ אטמוספרי, והסר את הגז שוב באמצעות משאבת הוואקום.
הערה: במקרה של מחזיק מדגם שטוח, מחזיק הדגימה חייב להיות מכוון בזווית של 45° ביחס לקרן הנכנסת. טווח העברת התנע השימושי יכול להיות מופחת עקב ספיגה ופיזור על ידי התא. בולם נייטרונים חזק כגון קדמיום יכול לשמש כדי להסוות חלקים מסוימים של מחזיק הדגימה (למשל, ברגים, חלקים עבים). - הציגו קצת גז הליום בתנור ההקפאה כך שהלחץ הוא ~ 0.05 בר.
- רכישת נתונים (למשל, באמצעות NOMAD על IN16B ב- ILL, ההנחה היא שהמשתמש מעדיף טמפרטורה של 200 K לפני רכישת ספקטרום נויטרונים קוואזיאלסטי (QENS), ואז E/IFWS במהלך כבש טמפרטורה ל- 310 K ב- 0.5 K לדקה ולבסוף QENS ב- 310 K).
- באמצעות NOMAD, בכרטיסייה ביצוע, גרור ושחרר בקר FurnaceCryostat במשטח השיגור. כוונו את הטמפרטורה ל 200 K. השתמש במצב מהיר ופסק זמן של 30 דקות כך שלטמפרטורה יהיה זמן להתייצב. לחץ על סמל החצים המסתובבים כדי להפעיל אותו ברקע כך שניתן יהיה להשיג נתונים במהלך ירידת הטמפרטורה.
- גרור ושחרר את בקר IN16DopplerSettings , הגדר את פרופיל המהירות למהירות מדויקת שנקבעה על ידי Max ΔE, ערך של 0.00 μeV ו- 128 ערוצים כדי לקבל תצורת EFWS.
- גרור ושחרר בקר ספירה , מלא את השדה כתוביות בשם המאפשר זיהוי קל של הנתונים, והגדר 60 חזרות של סריקות של 30 שניות (איור משלים S5A).
- גרור ושחרר בקר IN16DopplerSettings , הגדר את פרופיל המהירות לסינוס שנקבע על ידי מהירות עם ערך של 4.5 m/s ו- 2,048 ערוצים כדי לקבל תצורת QENS.
- גרור ושחרר בקר ספירה עם 4 חזרות של סריקות של 30 דקות (איור משלים S5B).
- עבור רמפת הטמפרטורה, גרור ושחרר בקר FurnaceCryostat , הגדר את הטמפרטורה ל- 310 K, הגדר את Ramp ל - SetPoint עם Δ = 0.05 K ו- 6 שניות. השתמש בפסק זמן של 220 דקות (איור משלים S6A).
- השתמש בלולאה עם 65 חזרות. בפנים, הכנס בקר IN16DopplerSettings כמו בשלב 3.4.2, ואחריו ספירה אחת של 30 שניות. לאחר מכן, הכנס IN16DopplerSettings, כפי שתואר קודם לכן, אך באמצעות היסט אנרגיה של 1.5 μeV ו - 1,024 ערוצים ואחריו ספירה אחת של 3 דקות (איור משלים S6B).
- כדי לרכוש את ה-QENS האחרון ב-310K, גרור ושחרר את בקרי IN16DopplerSettings ו-Count שתצורתם הוגדרה כמתואר בשלבים 3.4.4 ו-3.4.5, בהתאמה.
- לחץ על לחצן התחל (משולש ימני בתחתית החלון) כדי להפעיל את הסקריפט.
הערה: כל ניסוי ידרוש רכישת נתוני כיול; כלומר, התא הריק לצורך תיקוני חיסור או בליעה, החיץ לבדו בטמפרטורות השונות המשמשות למדל את הרקע, ומדידה של ונדיום (או שווה ערך, הדגימה בטמפרטורה של 10 K ומטה) כדי לקבל את פונקציית הרזולוציה של המכשיר.
4. ניתוח נתונים - QENS
- ייבוא ערכת הנתונים באמצעות שיטת 'IN16B_QENS.process()' בתוכנת Python nPDyn v3.x36
>>>> מייבוא nPDyn.dataParsers IN16B_QENS
מדגם >>> = IN16B_QENS(
... <נתיב לקבצי נתונים>
... [detGroup=... פורמט>]
... ). תהליך()
מדגם >>> = sample.get_q_range(0.3, 1.8) - בצע תיקוני נתונים (אופציונלי) באמצעות הפקודות הבאות (עיין בתיעוד של nPDyn לקבלת מידע נוסף, איור 3):
#it מניח כי נתונים עבור תא ריק, ונדיום וחיץ
# יובאו כבר במערך נתונים שנקרא 'empty_cell', 'ונדיום',
# ו- 'buffer', בהתאמה.
# עבור חיסור תאים ריקים עם גורם שינוי קנה מידה
# (שגיאות מופצות באופן אוטומטי)
מדגם >>> = מדגם - 0.95 * empty_cell
# לתיקון באמצעות מקדם Paalman-Ping
# (בלעדי הדדית עם הדוגמה לעיל)
מדגם >>> = sample.absorptionCorrection(empty_cell)
# לנורמליזציה
מדגם >>> = sample.normalize(vanadium)
# לקשירה לאורך ציר נצפה
# נצפה הוא זמן הצבירה כאן
מדגם >>> = מדגם.bin(3, ציר=0) - התאם את נתוני הכיול. ניתן להתקין את מערך הנתונים - דגימות, תאים ריקים, מאגר מפורק (במידת הצורך) וונדיום באמצעות מודלים מובנים או מודל המוגדר על-ידי המשתמש (ראה תיעוד nPDyn):
>>> מייבוא nPDyn.models.builtins (
... דגםPVoigt,
... דגםמים,
... דגם מכוילD2O,
... )
# מודלים מובנים משתמשים בווקטור עמודה של התנע
# העברת ערכי Q
>>> q = vanadium.q[:, ללא]
# הוונדיום מצויד באמצעות פרופיל פסאודו-Voigt
>>> vanadium.fit(modelPVoigt(q)) - השתמש במודל המובנה עבור מי הידרציה שנקרא 'modelWater'. מודל זה קורא כפי שמוצג על-ידי Eq (4)17
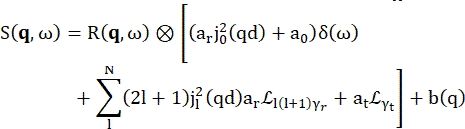 (4)
(4)
כאשר0,r ו-t הם סקלרים המסבירים את התרומה היחסית של אות אלסטי, תנועות סיבוב ותנועות תרגומיות, בהתאמה; j1(qd) היא פונקציית בסל כדורית מסדרl th, כאשר q היא העברת התנע; d מרחק O-H במולקולת המים; δ(ω) היא הדלתא של דיראק, אשר מוכפלת על ידי EISF כאן; N היא הסדר הגבוה ביותר של פונקציית בסל הכדורית המשמשת (בדרך כלל ~5);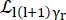 ו
ו 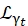 - הן תנועות סיבוב לורנציאניות ותנועות תרגומיות, בהתאמה; b(q) הוא מונח רקע שטוח. פונקציות בסל הכדוריות נותנות את התרומה היחסית של כל מצב תנע זוויתי של מולקולות המים, והמספר N נקבע על בסיס תחום q העברת התנע. במקרה של ספקטרומטר NBS טיפוסי, המונחים עד N=4 מסבירים כמעט לחלוטין את האות (איור משלים S7).
- הן תנועות סיבוב לורנציאניות ותנועות תרגומיות, בהתאמה; b(q) הוא מונח רקע שטוח. פונקציות בסל הכדוריות נותנות את התרומה היחסית של כל מצב תנע זוויתי של מולקולות המים, והמספר N נקבע על בסיס תחום q העברת התנע. במקרה של ספקטרומטר NBS טיפוסי, המונחים עד N=4 מסבירים כמעט לחלוטין את האות (איור משלים S7).
# כאן, משוואה 2 משמשת למי הידרציה
# פיתול עם פונקציית רזולוציה ותוספת של
# רקע D2O נעשה באופן אוטומטי עם
# סיפק ארגומנטים
>>> sample.fit(modelWater(q),
... res=ונדיום,
... bkgd=חיץ,
... volume_fraction_bkgd=0.95
... )
הערה: התרומות של תנועות סיבוביות ותרגומיות צריכות להיות מפותלות כדי להיות קפדניות לחלוטין. את הצלחתו של מודל תוספים יש לייחס לנוכחות אוכלוסיות שונות של מים על פני השטח החלבוני ולטווח האנרגיה המוגבל הנגיש. - השתמש באפשרויות הבאות כדי להתוות את הנתונים (איור 4):
>>> מ- nPDyn.plot import plot
>>> עלילה (מדגם)
5. ניתוח נתונים - רמפת טמפרטורה, סריקות חלון קבוע אלסטי (EFWS)
- השתמש בהליך דומה לסעיף 4 כדי לנרמל את נתוני הטמפרטורה לפי האות בטמפרטורה הנמוכה ביותר (בדרך כלל 10 K):
>>> מייבוא nPDyn.dataParsers IN16B_FWS
מדגם >>> = IN16B_FWS(
... <נתיב לקבצי נתונים>,
... detGroup=[detGroup=<קובץ קיבוץ מספרים שלמים או גלאי בתבנית XML>]
... ). תהליך()
# נורמליזציה עם 5 הנקודות הראשונות על הנצפה
ציר #, המתאים לטמפרטורה
מדגם >>> /= מדגם[:5].mean(0)
# טווח Q של העברת המומנטום המשמש כאן קטן יותר
# מכיוון שהמודל שבו נעשה שימוש תקף עבור q נמוך בלבד
מדגם >>> = sample.get_q_range(0.2, 0.8) - השתמש במודל גאוסיאני פשוט כדי להתחיל, שרוחבו נתון על ידי מה שמכונה תזוזה בריבוע ממוצע (MSD). בנה והתאם את המודל באמצעות הפקודות הבאות:
>>> לייבא numpy כ- NP
>>> מ- nPDyn.models ייבוא פרמטרים, מודל, רכיב
# a הוא גורם קנה מידה
>>> params = פרמטרים(
... a={'value': 1, 'bounds': (0, np.inf)},
... msd={'value': 1, 'bounds': (0, np.inf)}
... )
מודל >>> = מודל (פרמות)
>>> model.addComponent(Component(
... 'גאוסיאני',
... Lambda X, A, MSD: A * NP.EXP(-X ** 2 * MSD / 6)
... ))
>>> sample.fit(model, x=sample.q[:, ללא])
>>> עלילה (מדגם)
הערה: קירוב גאוס מתקיים תמיד עבור q2MSD << 1, אך ניתן להשתמש בטווח העברת תנע רחב יותר להשוואה יחסית בין דגימות. מודלים מתוחכמים יותר, החורגים מקירוב גאוס, פותחו37,38,39.
6. ניתוח נתונים - סריקות גמישות ולא אלסטיות עם חלונות קבועים (E/IFWS)
- בדומה לשלב 4, יבא את ערכת הנתונים אך באמצעות המחלקה 'IN16B_FWS':
>>> מייבוא nPDyn.dataParsers IN16B_FWS
מדגם >>> = IN16B_FWS(
... <נתיב לקבצי נתונים>
... [detGroup=<קובץ קיבוץ מספרים שלמים או גלאי בתבנית XML>]
... ). תהליך()
מדגם >>> = sample.get_q_range(0.3, 1.8) - התאם את נתוני הכיול ואת הנתונים לדוגמה.
- נתח את נתוני E/IFWS באמצעות MSD40 כללי או על ידי התייחסות אליהם כספקטרום QENS גס (שיש רק כמה נקודות נתונים על ציר האנרגיה). כאשר E/IFWS נתפס כ-QENS גס, מודלים המשמשים ל-QENS משמשים כדי להתאים את כל מערך הנתונים של E/IFWS בבת אחת (התאמה גלובלית של העברות אנרגיה והעברות מומנטום).
הערה: הפתרון האחרון - שימוש במודלים עבור QENS על נתוני E/IFWS - משמש כאן במקום שבו נכפית התלות בהעברת המומנטום של דיפוזיה של מרכז המסה ודינמיקה פנימית של חלבונים. - מודל דינמיקה של חלבונים בנוזלים באמצעות Eq (5) הבא ('modelProteinJumpDiff' ב- nPDyn):
 (5)
(5)
כאשר R(q,ω) היא פונקציית הרזולוציה; β סקלר בלתי תלוי עבור כל העברת תנע q; 0 הוא גורם המבנה האלסטי הלא קוהרנטי (EISF);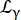 חשבונאות לורנציאנית לדיפוזיה של מרכז מסה עם רוחב נתון על ידי Eq (6);
חשבונאות לורנציאנית לדיפוזיה של מרכז מסה עם רוחב נתון על ידי Eq (6); 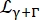 הוא לורנציאן הכולל דיפוזיה של מרכז מסה ותרומה בעקבות מודל דיפוזיית קפיצה14 המביא בחשבון דינמיקה פנימית (Eq (7);
הוא לורנציאן הכולל דיפוזיה של מרכז מסה ותרומה בעקבות מודל דיפוזיית קפיצה14 המביא בחשבון דינמיקה פנימית (Eq (7);  להיות האות המותאם מ D2O מחדש על ידי חלק נפח שלה בדגימה.
להיות האות המותאם מ D2O מחדש על ידי חלק נפח שלה בדגימה.
γ = Dsq2 (6)
Ds הוא מקדם הדיפוזיה העצמית.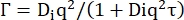 (7)
(7)
Di הוא מקדם הדיפוזיה הנראה לעין עבור דינמיקה פנימית ו-τ זמן הרפיה עבור תנועות דיפוזיות.
>>> מייבוא nPDyn.models.builtins (
... דגםPVoigt,
... modelProteinJumpDiff,
... דגם מכוילD2O,
... )
# מודלים מובנים משתמשים בווקטור עמודה של התנע
# העברת ערכי Q
>>> q = vanadium.q[:, ללא]
# הוונדיום מצויד באמצעות פרופיל פסאודו-Voigt
>>> vanadium.fit(modelPVoigt(q))
# עבור D2O טהור, דגם עם רוחב קו מכויל
# עבור טמפרטורות שונות כלול ב- nPDyn
>>> buffer.fit(modelCalibratedD2O(q, temp=363))
# כאן, משוואה 3 משמשת עבור דגימות נוזל
# פיתול עם פונקציית רזולוציה ותוספת של
# רקע D2O מתבצע באופן אוטומטי עם הארגומנטים שסופקו #
>>> sample.fit(modelProteinJumpDiff(q),
... res=ונדיום,
... bkgd=חיץ,
... volume_fraction_bkgd=0.95
... )
- נתח את נתוני E/IFWS באמצעות MSD40 כללי או על ידי התייחסות אליהם כספקטרום QENS גס (שיש רק כמה נקודות נתונים על ציר האנרגיה). כאשר E/IFWS נתפס כ-QENS גס, מודלים המשמשים ל-QENS משמשים כדי להתאים את כל מערך הנתונים של E/IFWS בבת אחת (התאמה גלובלית של העברות אנרגיה והעברות מומנטום).
- התווה את הנתונים המותאמים באמצעות:
>>> מ- nPDyn.plot import plot
>>> עלילה (מדגם)
תוצאות
הצבירה של ליזוזים לחלקיקים בוצעה ב 90 ° C עם ריכוז חלבון של 50 מ"ג / מ"ל בחיץ deuterated (0.1 M NaCl ב pD 10.5). היווצרות חלקיקים מופעלת על-ידי עליית הטמפרטורה ל-90°C ומתרחשת תוך 6 שעות (איור משלים S8). איסוף הנתונים בוצע ב-IN16B, כמתואר בפרוטוקול לעיל (הנתונים נאספים לצמיתות על ידי ה-ILL ונגישים ב-http://dx.doi.org/10....
Discussion
ספקטרוסקופיית נויטרונים היא השיטה היחידה המאפשרת לחקור את הדינמיקה הממוצעת של ps-ns של דגימות חלבון, ללא קשר לגודל החלבון או למורכבות התמיסה כאשר משתמשים בדאוטרציה6. באופן ספציפי, על ידי בדיקת דיפוזיה עצמית של מכלולי חלבונים בתמיסה, ניתן לקבוע באופן חד משמעי את הגודל ההידרודינ?...
Disclosures
למחברים אין ניגודי עניינים לחשוף.
Acknowledgements
המחברים אסירי תודה למיכאלה זמפוני במרכז יוליך למדעי הנייטרונים במרכז היינץ מאייר-לייבניץ, גרכינג, גרמניה, על חלק מניסויי פיזור הנייטרונים שנערכו במכשיר SPHERES. עבודה זו הפיקה תועלת מפעילויות קונסורציום מעבדת Deuteration Laboratory (DLAB) הממומן על ידי האיחוד האירופי תחת חוזים HPRI-2001-50065 ו- RII3-CT-2003-505925, ומפעילות במימון מועצת המחקר להנדסה ומדעי הפיזיקה בבריטניה (EPSRC) במסגרת Institut Laue Langevin EMBL DLAB תחת מענקים GR/R99393/01 ו- EP/C015452/1. תמיכה על ידי הנציבות האירופית במסגרת תוכנית המסגרת השביעית באמצעות פעולת המפתח: חיזוק אזור המחקר האירופי, תשתיות מחקר מוכרת [חוזה 226507 (NMI3)]. קווין פואנו וכריסטיאן בק מודים למשרד הפדרלי לחינוך ומחקר (BMBF, מענק מספר 05K19VTB) על מימון מלגות הפוסט-דוקטורט שלהם.
Materials
| Name | Company | Catalog Number | Comments |
| Aluminum sample holder | Not commercially available. Either the local contact on the instrument can provide them or they can be manufactured based on a technical drawing that can be provided by the local contact. | ||
| Deuterium chloride, 35 wt. % in D2O, ≥99 atom % D | Sigma-Aldrich | 543047 | |
| Deuterium oxide (D, 99.9%) | Eurisotop | DLM-4DR-PK | |
| Dow Corning high-vacuum silicone grease | Sigma-Aldrich | Z273554-1EA | |
| Ethanol 96%, EMSURE Reag. Ph Eur | Sigma-Aldrich | 1.5901 | |
| Glass dessicator | VWR | 75871-660 | |
| Glass dessicator plate, 140 mm | VWR | 89038-068 | |
| Indium wire, 1.0 mm (0.04 in) dia, Puratronic, 99.999% | Alfa Aesar | 00470.G1 | |
| Lysozyme from chicken egg white dialyzed, lyophilized, powder, ~100,000 U/mg | Sigma-Aldrich | 62970 | |
| nPDyn | v3.x | see github.com/kpounot/nPDyn, model functions fot fitting also included in the software | |
| OHAUS AX324 Adventurer balance, internal calibration | Dutscher | 92641 | |
| Phosphorus pentoxide, ReagentPlus, 99% | Sigma-Aldrich | 214701 | |
| Pipette ErgoOne 0.5-10 μL | Starlab | S7100-0510 | |
| Pipette ErgoOne 100-1,000 μL | Starlab | S7100-1000 | |
| Pipette ErgoOne 20-200 μL | Starlab | S7100-2200 | |
| Pipette tip TipOne 1,000 μL | Starlab | S1111-6001 | |
| Pipette tip TipOne 10 μL | Starlab | S1111-3200 | |
| Pipette tip TipOne 200 μL | Starlab | S1111-0206 | |
| Sodium deuteroxide solution, 40 wt. % in D2O, 99.5 atom % D | Sigma-Aldrich | 372072 |
References
- Jacrot, B. Des neutrons pour la science: Histoire de l'Institut Laue-Langevin. Des neutrons pour la science. EDP Sciences. , (2021).
- Mahieu, E., Gabel, F. Biological small-angle neutron scattering: recent results and development. Acta Crystallographica Section D. 74 (8), 715-726 (2018).
- Grimaldo, M., Roosen-Runge, F., Zhang, F., Schreiber, F., Seydel, T. Dynamics of proteins in solution. Quarterly Reviews of Biophysics. 52, 7 (2019).
- Martel, A., et al. Membrane permeation versus amyloidogenicity: A multitechnique study of islet amyloid polypeptide interaction with model membranes. Journal of the American Chemical Society. 139 (1), 137-148 (2017).
- Pounot, K., et al. Tracking internal and global diffusive dynamics during protein aggregation by high-resolution neutron spectroscopy. The Journal of Physical Chemistry Letters. 11 (15), 6299-6304 (2020).
- Grimaldo, M., et al. Protein short-time diffusion in a naturally crowded environment. The Journal of Physical Chemistry Letters. 10 (8), 1709-1715 (2019).
- Jasnin, M., Stadler, A., Tehei, M., Zaccai, G. Specific cellular water dynamics observed in vivo by neutron scattering and NMR. Physical Chemistry Chemical Physics. 12 (35), 10154-10160 (2010).
- Frick, B. The neutron backscattering spectrometer IN16 at ILL-high energy resolution with high intensity and excellent signal-to-noise ratio. Neutron News. 13 (2), 15-22 (2002).
- Frick, B., Mamontov, E., van Eijck, L., Seydel, T. Recent backscattering instrument developments at the ILL and SNS. Zeitschrift für Physikalische Chemie. 224 (1-2), 33-60 (2010).
- Frick, B., Combet, J., van Eijck, L. New possibilities with inelastic fixed window scans and linear motor Doppler drives on high resolution neutron backscattering spectrometers. Nuclear Instruments and Methods in Physics Research Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 669, 7-13 (2012).
- Appel, M., Frick, B., Magerl, A. A flexible high speed pulse chopper system for an inverted neutron time-of-flight option on backscattering spectrometers. Scientific Reports. 8 (1), 13580 (2018).
- Squires, G. L. . Introduction to the theory of thermal neutron scattering. , (1996).
- Singwi, K. S., Sjölander, A. Diffusive motions in water and cold neutron scattering. Physical Review. 119 (3), 863-871 (1960).
- Sears, V. F. Theory of cold neutron scattering by homonuclear diatomic liquids: i. free rotation. Canadian Journal of Physics. 44 (6), 1279-1297 (1966).
- Sears, V. F. Theory of cold neutron scattering by homonuclear liquid: ii. hindered rotation. Canadian Journal of Physics. 44 (6), 1299-1311 (1966).
- Schirò, G., et al. Translational diffusion of hydration water correlates with functional motions in folded and intrinsically disordered proteins. Nature Communications. 6, 6490 (2015).
- Grimaldo, M., et al. Hierarchical molecular dynamics of bovine serum albumin in concentrated aqueous solution below and above thermal denaturation. Physical Chemistry Chemical Physics. 17 (6), 4645-4655 (2015).
- Eanes, E. D., Glenner, G. G. X-ray diffraction studies on amyloid filaments. Journal of Histochemistry & Cytochemistry. 16 (11), 673-677 (1968).
- Bonar, L., Cohen, A. S., Skinner, M. M. Characterization of the Amyloid Fibril as a Cross-β Protein. Proceedings of the Society for Experimental Biology and Medicine. 131 (4), 1373-1375 (1969).
- Chiti, F., Dobson, C. M. Protein Misfolding, Amyloid Formation, and Human Disease: A Summary of Progress Over the Last Decade. Annual Review of Biochemistry. 86 (1), 27-68 (2017).
- Knowles, T. P. J., Vendruscolo, M., Dobson, C. M. The amyloid state and its association with protein misfolding diseases. Nature Reviews Molecular Cell Biology. 15 (6), 384-396 (2014).
- Maji, S. K., et al. Functional amyloids as natural storage of peptide hormones in pituitary secretory granules. Science. 325 (5938), 328-332 (2009).
- Li, J., et al. The RIP1/RIP3 necrosome forms a functional amyloid signaling complex required for programmed necrosis. Cell. 150 (2), 339-350 (2012).
- Knowles, T. P. J., Mezzenga, R. Amyloid fibrils as building blocks for natural and artificial functional materials. Advanced Materials. 28 (31), 6546-6561 (2016).
- Stephens, A. D., Kaminski Schierle, G. S. The role of water in amyloid aggregation kinetics. Current Opinion in Structural Biology. 58, 115-123 (2019).
- Adamcik, J., Mezzenga, R. Amyloid polymorphism in the protein folding and aggregation energy landscape. Angewandte Chemie International Edition. 57 (28), 8370-8382 (2018).
- Liu, Z., et al. Entropic contribution to enhanced thermal stability in the thermostable P450 CYP119. Proceedings of the National Academy of Sciences of the United States of America. 115 (43), 10049-10058 (2018).
- Coquelle, N., et al. Chromophore twisting in the excited state of a photoswitchable fluorescent protein captured by time-resolved serial femtosecond crystallography. Nature Chemistry. 10 (1), 31-37 (2018).
- Henzler-Wildman, K. A., et al. A hierarchy of timescales in protein dynamics is linked to enzyme catalysis. Nature. 450 (7171), 913-916 (2007).
- Fichou, Y., et al. Hydration water mobility is enhanced around tau amyloid fibers. Proceedings of the National Academy of Sciences of the United States of America. 112 (20), 6365-6370 (2015).
- Burns, J., Pennock, C. A., Stoward, P. J. The specificity of the staining of amyloid deposits with thioflavine T. The Journal of Pathology and Bacteriology. 94 (2), 337-344 (1967).
- Iqbal, K., Liu, F., Gong, C. -. X., Grundke-Iqbal, I. Tau in Alzheimer disease and related tauopathies. Current Alzheimer Research. 7 (8), 656-664 (2010).
- Krȩżel, A., Bal, W. A formula for correlating pKa values determined in D2O and H2O. Journal of Inorganic Biochemistry. 98 (1), 161-166 (2004).
- Dolman, M., Halling, P. J., Moore, B. D., Waldron, S. How dry are anhydrous enzymes? Measurement of residual and buried 18O-labeled water molecules using mass spectrometry. Biopolymers. 41 (3), 313-321 (1997).
- Pounot, K. kpounotnPDyn: v3.0.0. Zenodo. , (2021).
- Yi, Z., Miao, Y., Baudry, J., Jain, N., Smith, J. C. Derivation of mean-square displacements for protein dynamics from elastic incoherent neutron scattering. Journal of Physical Chemistry B. 116 (16), 5028-5036 (2012).
- Peters, J., Kneller, G. R. Motional heterogeneity in human acetylcholinesterase revealed by a non-Gaussian model for elastic incoherent neutron scattering. The Journal of Chemical Physics. 139 (16), 165102 (2013).
- Zeller, D., Telling, M. T. F., Zamponi, M., García Sakai, V., Peters, J. Analysis of elastic incoherent neutron scattering data beyond the Gaussian approximation. The Journal of Chemical Physics. 149 (23), 234908 (2018).
- Roosen-Runge, F., Seydel, T. A generalized mean-squared displacement from inelastic fixed window scans of incoherent neutron scattering as a model-free indicator of anomalous diffusion confinement. EPJ Web of Conferences. 83, 02015 (2015).
- Ortega, A., Amorós, D., García de la Torre, J. Prediction of hydrodynamic and other solution properties of rigid proteins from atomic- and residue-level models. Biophysical Journal. 101 (4), 892-898 (2011).
- Hennig, M., Frick, B., Seydel, T. IUCr Optimum velocity of a phase-space transformer for cold-neutron backscattering spectroscopy. Journal of Applied Crystallography. 44 (3), 467-472 (2011).
- Paalman, H. H., Pings, C. J. Numerical evaluation of X-ray absorption factors for cylindrical samples and annular sample cells. Journal of Applied Physics. 33 (8), 2635-2639 (1962).
- Ow, S. -. Y., Dunstan, D. E. The effect of concentration, temperature and stirring on hen egg white lysozyme amyloid formation. Soft Matter. 9 (40), 9692-9701 (2013).
- Tominaga, T., Sahara, M., Kawakita, Y., Nakagawa, H., Yamada, T. Evaluation of sample cell materials for aqueous solutions used in quasi-elastic neutron scattering measurements. Journal of Applied Crystallography. 54 (6), 1631-1640 (2021).
- Beck, C., et al. Following protein dynamics in real time during crystallization. Crystal Growth & Design. 19 (12), 7036-7045 (2019).
- Smith, A. A., Testori, E., Cadalbert, R., Meier, B. H., Ernst, M. Characterization of fibril dynamics on three timescales by solid-state NMR. Journal of Biomolecular NMR. 65 (3-4), 171-191 (2016).
- Wang, T., Jo, H., DeGrado, W. F., Hong, M. Water distribution, dynamics, and interactions with Alzheimer's β-amyloid fibrils investigated by solid-state NMR. Journal of the American Chemical Society. 139 (17), 6242-6252 (2017).
- Rezaei-Ghaleh, N., Giller, K., Becker, S., Zweckstetter, M. Effect of zinc dinding on β-amyloid structure and dynamics: Implications for Aβ aggregation. Biophysical Journal. 101 (5), 1202-1211 (2011).
- Vugmeyster, L., et al. Fast motions of key methyl groups in amyloid-β fibrils. Biophysical Journal. 111 (10), 2135-2148 (2016).
- Yang, X., Wang, B., Hoop, C. L., Williams, J. K., Baum, J. NMR unveils an N-terminal interaction interface on acetylated-α-synuclein monomers for recruitment to fibrils. Proceedings of the National Academy of Sciences of the United States of America. 118 (18), (2021).
- Tuttle, M. D., et al. Solid-state NMR structure of a pathogenic fibril of full-length human α-synuclein. Nature Structural & Molecular Biology. 23 (5), 409-415 (2016).
- Karamanos, T. K., Kalverda, A. P., Thompson, G. S., Radford, S. E. Mechanisms of amyloid formation revealed by solution NMR. Progress in Nuclear Magnetic Resonance Spectroscopy. 88-89, 86-104 (2015).
- Lai, Y. -. C., Kuo, Y. -. H., Chiang, Y. -. W. Identifying protein conformational dynamics using spin-label ESR. Chemistry - An Asian Journal. 14 (22), 3981-3991 (2019).
- Franck, J. M., Han, S. Overhauser dynamic nuclear polarization for the study of hydration dynamics, explained. Methods in Enzymology. 615, 131-175 (2019).
- Pavlova, A., et al. Protein structural and surface water rearrangement constitute major events in the earliest aggregation stages of tau. Proceedings of the National Academy of Sciences of the United States of America. 113 (2), 127-136 (2016).
- Lin, Y., et al. Liquid-liquid phase separation of tau driven by hydrophobic interaction facilitates fibrillization of tau. bioRxiv. , (2020).
- Decatur, S. M. Elucidation of residue-level structure and dynamics of polypeptides via isotope-edited infrared spectroscopy. Accounts of Chemical Research. 39 (3), 169-175 (2006).
- Chatani, E., Tsuchisaka, Y., Masuda, Y., Water Tsenkova, R. molecular system dynamics associated with amyloidogenic nucleation as revealed by real time near infrared spectroscopy and aquaphotomics. PLoS One. 9 (7), 101997 (2014).
- Goret, G., Aoun, B., Pellegrini, E. MDANSE: An interactive analysis environment for molecular dynamics simulations. Journal of Chemical Information and Modeling. 57 (1), 1-5 (2017).
- Fujiwara, S., et al. Internal dynamics of a protein that forms the amyloid fibrils observed by neutron scattering. Journal of the Physical Society of Japan. 82, (2013).
- Schiró, G., et al. Neutron scattering reveals enhanced protein dynamics in concanavalin a amyloid fibrils. Journal of Physical Chemistry Letters. 3 (8), 992-996 (2012).
- Pounot, K., et al. Zinc determines dynamical properties and aggregation kinetics of human insulin. Biophysical Journal. 120 (5), 886-898 (2021).
- Fujiwara, S., et al. Dynamic properties of human α-synuclein related to propensity to amyloid fibril formation. Journal of Molecular Biology. 431 (17), 3229-3245 (2019).
- Sanz, A., et al. High-pressure cell for simultaneous dielectric and neutron spectroscopy. Review of Scientific Instruments. 89 (2), 023904 (2018).
- Adams, M. A., et al. Simultaneous neutron scattering and Raman scattering. Applied Spectroscopy. 63 (7), 727-732 (2009).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved