È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Spettroscopia neutronica ad alta risoluzione per studiare la dinamica picosecondi-nanosecondi delle proteine e dell'acqua di idratazione
In questo articolo
Riepilogo
La spettroscopia di retrodiffusione neutronica offre un accesso non distruttivo e privo di etichette alla dinamica ps-ns delle proteine e della loro acqua di idratazione. Il flusso di lavoro è presentato con due studi sulle proteine amiloidi: sulla dinamica tempo-risolta del lisozima durante l'aggregazione e sulla dinamica dell'acqua di idratazione della tau sulla formazione delle fibre.
Abstract
Lo scattering neutronico offre la possibilità di sondare la dinamica all'interno dei campioni per una vasta gamma di energie in modo non distruttivo e senza etichettatura diversa dal deuterio. In particolare, la spettroscopia di retrodiffusione neutronica registra i segnali di scattering a più angoli di scattering simultaneamente ed è adatta per studiare la dinamica dei sistemi biologici sulla scala temporale ps-ns. Impiegando D2O-e possibilmente componenti tampone deuterati, il metodo consente il monitoraggio sia della diffusione del centro di massa che dei movimenti della spina dorsale e della catena laterale (dinamica interna) delle proteine allo stato liquido.
Inoltre, la dinamica dell'acqua di idratazione può essere studiata impiegando polveri di proteine perdeuterate idratate con H2O. Questo documento presenta il flusso di lavoro impiegato sullo strumento IN16B presso l'Institut Laue-Langevin (ILL) per studiare la dinamica delle proteine e dell'idratazione dell'acqua. Viene spiegata la preparazione di campioni di soluzione e campioni di polvere proteica idrata utilizzando lo scambio di vapore. La procedura di analisi dei dati per la dinamica delle proteine e dell'acqua di idratazione è descritta per diversi tipi di set di dati (spettri quasielastici o scansioni a finestra fissa) che possono essere ottenuti su uno spettrometro di retrodiffusione di neutroni.
Il metodo è illustrato con due studi che coinvolgono proteine amiloidi. L'aggregazione del lisozima in aggregati sferici di dimensioni μm - particolato indicato - è dimostrato che avviene in un processo in un'unica fase sull'intervallo di spazio e tempo sondato su IN16B, mentre la dinamica interna rimane invariata. Inoltre, la dinamica dell'acqua di idratazione della tau è stata studiata su polveri idratate di proteine perdeuterate. È dimostrato che i movimenti traslazionali dell'acqua si attivano sulla formazione di fibre amiloidi. Infine, vengono discussi i passaggi critici del protocollo su come lo scattering neutronico è posizionato per quanto riguarda lo studio della dinamica rispetto ad altri metodi biofisici sperimentali.
Introduzione
Il neutrone è una particella massiccia e priva di carica che è stata utilizzata con successo nel corso degli anni per sondare campioni in vari campi, dalla fisica fondamentale alla biologia1. Per le applicazioni biologiche, lo scattering neutronico a piccolo angolo, lo scattering neutronico anelastico, la cristallografia neutronica e la riflettometria sono ampiamente utilizzati 2,3,4. Lo scattering anelastico di neutroni fornisce una misura mediata dell'insieme della dinamica senza richiedere un'etichettatura specifica di per sé e una qualità del segnale che non dipende dalla dimensione o dalla proteina5. La misurazione può essere effettuata utilizzando un ambiente altamente complesso per la proteina in studio che imita il mezzo intracellulare, come un lisato batterico deuterato o anche in vivo 3,6,7. Diverse configurazioni sperimentali possono essere utilizzate per studiare le dinamiche, vale a dire i) l'accesso al tempo di volo alle dinamiche sub-ps-ps, ii) l'accesso alla dinamica ps-ns e iii) l'accesso alle dinamiche da ns a centinaia di ns. La retrodiffusione neutronica fa uso della legge di Bragg 2d sinθ = nλ, dove d è la distanza tra i piani in un cristallo, θ l'angolo di scattering, n l'ordine di scattering e λ la lunghezza d'onda. L'uso di cristalli per il backscattering verso i rivelatori consente di ottenere un'alta risoluzione in energia, tipicamente ~ 0,8 μeV. Per misurare lo scambio di energia, viene utilizzato un azionamento Doppler che trasporta un cristallo in retrodiffusione per definire e sintonizzare la lunghezza d'onda dei neutroni in arrivo 8,9,10 (Figura 1), oppure è possibile utilizzare una configurazione del tempo di volo al costo di una diminuzione della risoluzione energetica 11.
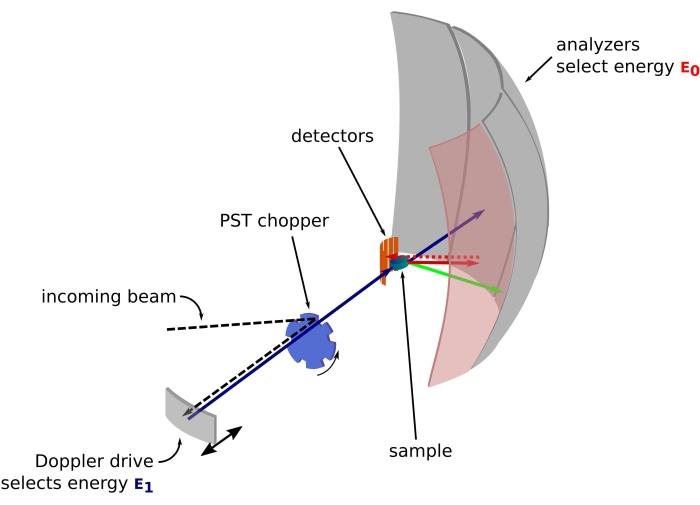
Figura 1: Schizzo di uno spettrometro di retrodiffusione di neutroni con un drive Doppler. Il fascio in entrata colpisce il chopper PST (Phase Space Transformation)42, che aumenta il flusso nella posizione del campione. Viene quindi retrodiffuso verso il campione dall'azionamento Doppler, che seleziona un'energia E1 (freccia ciano). I neutroni vengono poi dispersi dal campione (con energie diverse rappresentate dal colore delle frecce) e gli analizzatori, costituiti da cristalli di Si 111, disperderanno solo neutroni con una specifica energia E0 (qui frecce colorate di rosso). Quindi, il trasferimento di quantità di moto q è ottenuto dalla posizione rilevata del neutrone sull'array del rivelatore e il trasferimento di energia è ottenuto dalla differenza E1- E0. Il tempo di volo previsto per l'impulso neutronico prodotto dal PST viene utilizzato per scartare il segnale dai neutroni sparsi direttamente verso i tubi del rivelatore. Abbreviazione: PST = trasformazione dello spazio delle fasi. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Per la spettroscopia di retrodiffusione, il principale contributo al segnale da campioni ricchi di protoni di idrogeno, come le proteine, proviene dallo scattering incoerente, per il quale l'intensità di scattering Sinc(q, ω) è mostrata da Eq (1)12
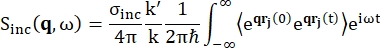 (1)
(1)
Dove σinc è la sezione trasversale incoerente dell'elemento considerato, k' è la norma del vettore d'onda diffuso, k la norma del vettore d'onda entrante, q (= k - k') il trasferimento di quantità di moto, r j(t) il vettore posizione dell'atomo j al tempo t e ω la frequenza corrispondente al trasferimento di energia tra il neutrone entrante e il sistema. Le parentesi angolari indicano la media dell'ensemble. Quindi, lo scattering incoerente sonda l'autocorrelazione delle singole particelle mediata dall'insieme delle posizioni degli atomi con il tempo e fornisce l'autodinamica mediata su tutti gli atomi nel sistema e le diverse origini temporali (media dell'insieme). La funzione di scattering è la trasformata di Fourier nel tempo della funzione di scattering intermedia I(q, t), che può essere vista come la trasformata di Fourier nello spazio della funzione di correlazione di van Hove mostrata da Eq (2):
 (2)
(2)
Dove ρ(r,t) è la densità di probabilità di trovare un atomo in posizione r e tempo t 13.
Per un processo di diffusione di Fick, la funzione di autodiffusione risulta (vedi Eq (3)) dopo una doppia trasformata di Fourier in una funzione di scattering consistente in un lorentziano di larghezza di linea data da γ = Dq2.
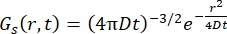 (3)
(3)
Sono stati sviluppati e trovati utili modelli più sofisticati come il modello di diffusione a salto di Singwi e Sjölander per la dinamica interna delle proteine ps-ns14 o il modello di rotazione di Sears per l'acqua di idratazione15,16,17.
Sullo strumento di retrodiffusione neutronica (NBS) IN16B 8,9 presso l'ILL, Grenoble, Francia (Figura supplementare S1), una configurazione comunemente usata con le proteine è costituita da cristalli di Si 111 per gli analizzatori con un drive Doppler per la regolazione della lunghezza d'onda in entrata (Figura supplementare S2A), dando così accesso all'intervallo di trasferimento della quantità di moto ~0,2 Å-1 < q < ~2 Å-1 e intervallo di trasferimento di energia di -30 μeV < 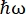 < 30 μeV, corrispondenti a scale temporali che vanno da pochi ps a pochi ns e distanze di pochi Å. Inoltre, IN16B offre la possibilità di eseguire scansioni elastiche e anelastiche a finestra fissa (E/IFWS)10, che includono l'acquisizione dei dati a un trasferimento di energia fisso. Poiché il flusso è limitato quando si lavora con neutroni, E/IFWS consente di massimizzare il flusso per un trasferimento di energia, riducendo così il tempo di acquisizione necessario per ottenere un soddisfacente rapporto segnale-rumore. Un'opzione più recente è il backscattering and time-of-flight spectrometer (BATS) mode11, che consente la misurazione di un'ampia gamma di trasferimenti di energia, (ad esempio, -150 μeV < <
< 30 μeV, corrispondenti a scale temporali che vanno da pochi ps a pochi ns e distanze di pochi Å. Inoltre, IN16B offre la possibilità di eseguire scansioni elastiche e anelastiche a finestra fissa (E/IFWS)10, che includono l'acquisizione dei dati a un trasferimento di energia fisso. Poiché il flusso è limitato quando si lavora con neutroni, E/IFWS consente di massimizzare il flusso per un trasferimento di energia, riducendo così il tempo di acquisizione necessario per ottenere un soddisfacente rapporto segnale-rumore. Un'opzione più recente è il backscattering and time-of-flight spectrometer (BATS) mode11, che consente la misurazione di un'ampia gamma di trasferimenti di energia, (ad esempio, -150 μeV < < 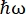 150 μeV), con un flusso più elevato rispetto al Doppler, ma al costo di una risoluzione energetica inferiore (Figura supplementare S2B).
150 μeV), con un flusso più elevato rispetto al Doppler, ma al costo di una risoluzione energetica inferiore (Figura supplementare S2B).
Una proprietà importante dello scattering neutronico è che la sezione d'urto incoerente σinc ha un valore 40 volte superiore per l'idrogeno che per il deuterio ed è trascurabile per altri elementi che si trovano comunemente nei campioni biologici. Pertanto, la dinamica delle proteine in un ambiente liquido può essere studiata utilizzando un tampone deuterato e lo stato della polvere consente lo studio della dinamica interna delle proteine con polvere proteica idrogenata idratata conD 2 O, o lo studio dell'acqua di idratazione per proteine perdeuterate in polvere idratate con H2O. Allo stato liquido, la retrodiffusione neutronica consente tipicamente l'accesso simultaneo all'autodiffusione del centro di massa delle proteine (diffusione di tipo Fickiano) e alla loro dinamica interna. Questi ultimi sono movimenti della spina dorsale e della catena laterale solitamente descritti dal cosiddetto modello di diffusione del salto o altri 3,18. Nelle polveri proteiche idrogenate, la diffusione proteica è assente e deve essere modellata solo la dinamica interna. Per l'acqua di idratazione, i contributi dei moti traslazionali e rotazionali delle molecole d'acqua presentano una diversa dipendenza dal trasferimento di quantità di moto q, che consente la loro distinzione nel processo di analisi dei dati17.
Questo articolo illustra il metodo di retrodiffusione neutronica con lo studio di proteine che sono risultate in grado di dispiegarsi, aggregarsi in una forma canonica costituita da pile di β-filamenti - il cosiddetto modello di β incrociato19,20 - e formare fibre allungate. Si tratta della cosiddetta aggregazione amiloide, ampiamente studiata per il suo ruolo centrale nelle malattie neurodegenerative come il morbo di Alzheimer o il Parkinson21,22. Lo studio delle proteine amiloidi è anche motivato dal ruolo funzionale che possono svolgere 23,24 o dal loro elevato potenziale per lo sviluppo di nuovi biomateriali25. I determinanti fisico-chimici dell'aggregazione amiloide rimangono poco chiari e non è disponibile alcuna teoria generale dell'aggregazione amiloide, nonostante gli enormi progressi compiuti negli ultimi anni21,26.
L'aggregazione amiloide implica cambiamenti nella struttura e nella stabilità delle proteine nel tempo, il cui studio implica naturalmente dinamiche, legate alla stabilità della conformazione delle proteine, alla funzione proteica e al panorama energetico delle proteine27. La dinamica è direttamente collegata alla stabilità di uno stato specifico attraverso il contributo entropico per i moti più veloci28, e la funzione proteica può essere sostenuta da movimenti su varie scale temporali da sub-ps per proteine sensibili alla luce29 a ms per movimenti di dominio, che possono essere facilitati dalla dinamica picosecondo-nanosecondo30.
Saranno presentati due esempi di utilizzo della spettroscopia di retrodiffusione neutronica per studiare le proteine amiloidi, uno allo stato liquido per studiare la dinamica delle proteine e uno allo stato di polvere idratata per studiare la dinamica dell'acqua di idratazione. Il primo esempio riguarda l'aggregazione del lisozima in sfere di dimensioni μm (chiamate particolato) seguite in tempo reale5, e il secondo un confronto della dinamica dell'acqua negli stati nativi e aggregati della proteina umana tau31.
Il lisozima è un enzima coinvolto nella difesa immunitaria ed è composto da 129 residui di aminoacidi. Il lisozima può formare particolato in tampone deuterato a pD di 10,5 e ad una temperatura di 90 °C. Con lo scattering neutronico, abbiamo dimostrato che l'evoluzione temporale del coefficiente di diffusione del centro di massa del lisozima segue la singola cinetica esponenziale della fluorescenza T della tioflavina (una sonda fluorescente utilizzata per monitorare la formazione di modelli di β incrociato amiloide32), indicando che le sovrastrutture particolate di formazione e i modelli di cross-β si verificano in un unico passaggio con la stessa velocità. Inoltre, la dinamica interna è rimasta costante durante tutto il processo di aggregazione, che può essere spiegato sia da un rapido cambiamento conformazionale che non può essere osservato sugli strumenti NBS, sia dall'assenza di cambiamenti significativi nell'energia interna della proteina dopo l'aggregazione.
La proteina umana tau è una proteina intrinsecamente disordinata (IDP) costituita da 441 aminoacidi per la cosiddetta isoforma 2N4R, che è particolarmente coinvolta nella malattia di Alzheimer33. Utilizzando la retrodiffusione neutronica su polveri di proteina tau perdeuterata, abbiamo dimostrato che la dinamica dell'acqua di idratazione è aumentata allo stato di fibra, con una maggiore popolazione di molecole d'acqua sottoposte a movimenti traslazionali. Il risultato suggerisce che un aumento dell'entropia dell'acqua di idratazione potrebbe guidare la fibrillazione amiloide della tau.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Preparare il tampone deuterato per le proteine allo stato liquido
- Sciogliere tutti i componenti del tampone in puro D2O.
- Se l'elettrodo pH è stato calibrato in H2O, regolare il pD secondo la formula pD = pH + 0,4 usando NaOD o DCl34.
NOTA: L'uso di D2 O invece di H2O potrebbe influire sulla solubilità delle proteine e potrebbe essere necessario adattare le condizioni tampone (ad esempio, con una leggera variazione della concentrazione salina).
2. Preparare le polveri H2O-idratate di proteine perdeuterate
- Preparare il portacampioni.
- Pulire accuratamente un portacampioni piatto in alluminio con la guarnizione del filo di indio e le viti con acqua ed etanolo e lasciarlo asciugare.
NOTA: viene utilizzato un portacampioni piatto in modo tale che la polvere possa essere distribuita in modo omogeneo sulla superficie. La quantità di polvere deve essere sufficiente in modo tale da poter essere mantenuta tra le pareti e non cadere quando il portacampioni viene posizionato verticalmente. - Pesare separatamente le diverse parti del portacampioni, il fondo, il coperchio e il filo di indio su una bilancia di precisione.
- Posizionare la guarnizione del filo di indio da 1 mm nella scanalatura della parte inferiore del portacampioni, lasciando una piccola sovrapposizione nel punto in cui le due estremità si uniscono (figura 2A).
- Posizionare una quantità appropriata di proteine liofilizzate (tipicamente ~ 100 mg di proteine) in modo tale da riempire la superficie interna della parte inferiore del portacampioni.
- Pulire accuratamente un portacampioni piatto in alluminio con la guarnizione del filo di indio e le viti con acqua ed etanolo e lasciarlo asciugare.
- Idratare la polvere proteica.
- Porre il portacampioni in un essiccatore con una capsula di Petri contenente polvere di P2O5 per 24 ore per asciugare completamente la polvere proteica35 (Figura 2B). Pesare la parte inferiore asciutta del portacampioni contenente il sigillo di indio e la polvere secca per ottenere masciutto.
ATTENZIONE: la polvere P2O5 è molto corrosiva. - Rimuovere il P 2 O5 dall'essiccatore e mettere una capsula di Petri con D2O all'interno. Controllare regolarmente la massa della polvere per controllare il livello di idratazione h = m hyd / m dry dove mhyd e mdry sono rispettivamente la massa della polvere idratata e della polvere secca.
NOTA: Per proteine altamente idrofobiche come l'insulina, potrebbe essere necessario aumentare la temperatura all'interno dell'essiccatore per ottenere una pressione di vapore più elevata e raggiungere il livello di idratazione desiderato h. - Ripetere i passaggi 2.2.1 e 2.2.2 almeno tre volte per convertire correttamente tutti gli idrogeni scambiabili in deuteroni.
NOTA: In alternativa, cicli di liofilizzazione e dissoluzione in D2O puro potrebbero essere utilizzati per un migliore scambio H/D a condizione che la proteina non ne sia influenzata. - Idratare la polvere leggermente al di sopra del livello desiderato, lasciare che la parte inferiore del supporto del campione con il filo di indio e la polvere idratata rimanga sulla bilancia di precisione e attendere che la massa diminuisca lentamente fino al valore desiderato per ottenere l'h target (tipicamente 0,2-0,4 se una proteina globulare di medie dimensioni deve essere coperta da uno strato di idratazione completo).
- Posizionare rapidamente il coperchio sulla parte inferiore e chiudere prima il portacampioni con quattro viti per interrompere lo scambio di vapore (Figura supplementare S3A).
- Posizionare e serrare tutte le viti rimanenti fino a quando non è visibile alcuno spazio tra la parte inferiore e il coperchio (Figura supplementare S3B).
- Pesare il portacampioni sigillato per verificare eventuali perdite di idratazione dovute a perdite dopo l'esperimento neutronico.
- Porre il portacampioni in un essiccatore con una capsula di Petri contenente polvere di P2O5 per 24 ore per asciugare completamente la polvere proteica35 (Figura 2B). Pesare la parte inferiore asciutta del portacampioni contenente il sigillo di indio e la polvere secca per ottenere masciutto.
3. Eseguire l'esperimento di scattering neutronico incoerente
- Discutere e ricontrollare la configurazione dello strumento necessario per l'esperimento con il contatto locale alcune settimane prima del beamtime assegnato.
- Preparare il campione allo stato liquido.
- Sciogliere la proteina nel tampone deuterato.
- Determinare il volume appropriato di liquido da inserire nel portacampioni usando acqua (assicurarsi che non vi sia trabocco quando il portacampioni è chiuso; Figura 2C).
NOTA: Le seguenti fasi (3.3 e 3.4) descrivono un esperimento condotto sullo spettrometro NBS IN16B all'ILL8,9, utilizzando una criofornace come ambiente campione. Il sistema di controllo dello strumento cambierà da uno strumento all'altro, ma i principi di funzionamento rimarranno gli stessi.
- Inserire l'esempio.
- Asciugare accuratamente lo stick del campione (Figura 2D) e rimuovere l'eventuale campione precedente dopo aver verificato che la dose di radiazioni ionizzanti sia inferiore a 100 μSv/h prima di maneggiare qualsiasi materiale (all'ILL).
- Posizionare il campione, verificare la corretta centratura rispetto al centro del fascio (Figura supplementare S4) e inserire il bastoncino del campione nella criofornace (Figura 2D). Accendere la pompa per vuoto per raggiungere meno di 10-3 bar e sciacquare l'aria all'interno della crioforno ripetendo le seguenti tre volte: riempire la criofornace con gas elio fino a raggiungere la pressione atmosferica e rimuovere nuovamente il gas utilizzando la pompa per vuoto.
NOTA: Nel caso di un portacampioni piatto, il portacampioni deve essere orientato con un angolo di 45° rispetto al fascio in entrata. L'intervallo di trasferimento del momento utile potrebbe essere ridotto a causa dell'assorbimento e della dispersione da parte della cellula. Un potente assorbitore di neutroni come il cadmio può essere utilizzato per mascherare alcune parti del portacampioni (ad esempio, viti, parti spesse). - Introdurre un po 'di gas elio nella criofornace in modo tale che la pressione sia ~ 0,05 bar.
- Acquisire dati (ad esempio, utilizzando NOMAD su IN16B all'ILL, si presume che l'utente preferisca una temperatura di 200 K prima di acquisire uno spettro di neutroni quasielastici (QENS), quindi E / IFWS durante una rampa di temperatura a 310 K a 0,5 K al minuto e infine un QENS a 310 K).
- Utilizzando NOMAD, nella scheda di esecuzione , trascinare e rilasciare un controller FurnaceCryostat nella piattaforma di lancio. Impostare la temperatura su 200 K. Utilizzare la modalità veloce e un timeout di 30 minuti in modo tale che la temperatura abbia il tempo di stabilizzarsi. Fare clic sull'icona delle frecce rotanti per eseguirlo in background in modo che i dati possano essere acquisiti durante la diminuzione della temperatura.
- Trascinare e rilasciare il controller IN16DopplerSettings , impostare il profilo di velocità su Velocità accurata impostata da Max ΔE, un valore di 0,00 μeV e 128 canali per ottenere una configurazione EFWS.
- Trascina e rilascia un controller Count , compila il campo Sottotitolo con un nome che consenta una facile identificazione dei dati e imposta 60 ripetizioni di scansioni di 30 s (Figura supplementare S5A).
- Trascinare e rilasciare un controller IN16DopplerSettings , impostare il profilo di velocità su Sine impostato da Speed con un valore di 4,5 m/s e 2.048 canali per ottenere una configurazione QENS.
- Trascina e rilascia un controller Count con 4 ripetizioni di scansioni di 30 minuti (Figura supplementare S5B).
- Per la rampa di temperatura, trascinare e rilasciare un controller FurnaceCryostat , impostare la temperatura su 310 K, impostare Ramp su SetPoint con Δ = 0,05 K e 6 s. Utilizzare un tempo di 220 minuti (figura supplementare S6A).
- Usa un ciclo for con 65 ripetizioni. All'interno, inserire un controller IN16DopplerSettings come nel passaggio 3.4.2, seguito da un singolo conteggio di 30 s. Successivamente, inserire IN16DopplerSettings, come descritto in precedenza, ma utilizzando un offset energetico di 1,5 μeV e 1.024 canali seguito da un singolo conteggio di 3 minuti (Figura supplementare S6B).
- Per acquisire l'ultimo QENS a 310 K, trascinare e rilasciare i controller IN16DopplerSettings e Count configurati come descritto rispettivamente nei passaggi 3.4.4 e 3.4.5.
- Premere il pulsante di avvio (triangolo rettangolo nella parte inferiore della finestra) per eseguire lo script.
NOTA: Ogni esperimento richiederà l'acquisizione di dati di calibrazione; cioè la cella vuota per le correzioni di sottrazione o assorbimento, il buffer da solo alle diverse temperature utilizzate per modellare lo sfondo e una misura di vanadio (o equivalentemente, il campione a una temperatura di 10 K o inferiore) per ottenere la funzione di risoluzione dello strumento.
4. Analisi dei dati - QENS
- Importare il set di dati utilizzando il metodo 'IN16B_QENS.process()' nel software Python nPDyn v3.x36
>>>> da nPDyn.dataParser importa IN16B_QENS
>>> esempio = IN16B_QENS(
...
... [detGroup=... formato>]
... ). processo()
>>> campione = sample.get_q_range(0,3; 1,8) - Eseguire correzioni dei dati (facoltativo) con i seguenti comandi (vedere la documentazione di nPDyn per ulteriori informazioni, Figura 3):
#it si presume che i dati per celle vuote, vanadio e buffer
# sono stati importati già nel set di dati chiamato 'empty_cell', 'vanadio',
# e 'buffer', rispettivamente.
# per la sottrazione di celle vuote con un fattore di scala
# (gli errori vengono propagati automaticamente)
>>> campione = campione - 0,95 * empty_cell
# per la correzione utilizzando il coefficiente di Paalman-Ping
# (mutuamente esclusivo con l'esempio sopra)
>>> sample = sample.absorptionCorrection(empty_cell)
# per la normalizzazione
>>> campione = sample.normalize(vanadio)
# per il binning lungo l'asse osservabile
# osservabile è il tempo di aggregazione qui
>>> campione = campione.bin(3; asse=0) - Adatta i dati di calibrazione. I campioni del set di dati, la cella vuota, il buffer deuterato (se necessario) e il vanadio possono essere montati utilizzando modelli integrati o un modello definito dall'utente (vedere la documentazione nPDyn):
>>> dall'importazione nPDyn.models.builtins (
... modelloPVoigt,
... modelloAcqua,
... modelloCalibratedD2O,
... )
# I modelli incorporati utilizzano un vettore colonna della quantità di moto
# Trasferisci valori Q
>>> q = vanadio.q[:, Nessuno]
# il vanadio è montato utilizzando un profilo pseudo-Voigt
>>> vanadium.fit(modelPVoigt(q)) - Utilizzare il modello integrato per l'acqua di idratazione chiamato "modelWater". Questo modello si legge come mostrato da Eq (4)17
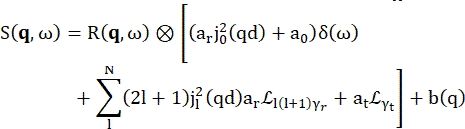 (4)
(4)
Dove a0, ar e at sono scalari che rappresentano il contributo relativo del segnale elastico, dei moti rotazionali e dei moti traslazionali, rispettivamente; j1(qd) è la funzione di Bessel sferica di ordinel-esimo , dove q è il trasferimento di quantità di moto; d la distanza O-H nella molecola d'acqua; δ(ω) è il delta di Dirac, che qui viene moltiplicato per l'EISF; N è l'ordine più alto della funzione di Bessel sferica utilizzata (tipicamente ~5);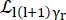 e
e 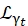 sono rispettivamente i moti rotazionali e traslazionali lorentziani; b(q) è un termine di fondo piatto. Le funzioni sferiche di Bessel danno il contributo relativo di ogni stato del momento angolare delle molecole d'acqua, e il numero N è determinato in base all'intervallo q di trasferimento della quantità di moto. Nel caso di un tipico spettrometro NBS, i termini fino a N = 4 spiegano quasi interamente il segnale (Figura supplementare S7).
sono rispettivamente i moti rotazionali e traslazionali lorentziani; b(q) è un termine di fondo piatto. Le funzioni sferiche di Bessel danno il contributo relativo di ogni stato del momento angolare delle molecole d'acqua, e il numero N è determinato in base all'intervallo q di trasferimento della quantità di moto. Nel caso di un tipico spettrometro NBS, i termini fino a N = 4 spiegano quasi interamente il segnale (Figura supplementare S7).
# Qui, l'equazione 2 è usata per l'acqua di idratazione
# convoluzione con funzione di risoluzione e aggiunta di
# Lo sfondo D2O viene eseguito automaticamente con il
# argomenti forniti
>>> sample.fit(modelWater(q),
... res=vanadio,
... bkgd=buffer,
... volume_fraction_bkgd=0,95
... )
NOTA: I contributi dei moti rotazionali e traslazionali dovrebbero essere contorti per essere perfettamente rigorosi. Il successo di un modello additivo è da attribuire alla presenza di popolazioni distinte di acqua sulla superficie proteica e alla limitata gamma di energia accessibile. - Utilizzare quanto segue per tracciare i dati (Figura 4):
>>> dal grafico di importazione nPDyn.plot
>>> trama(esempio)
5. Analisi dei dati - rampa di temperatura, scansioni elastiche a finestra fissa (EFWS)
- Utilizzare una procedura simile alla sezione 4 per normalizzare i dati della rampa di temperatura dal segnale alla temperatura più bassa (in genere 10 K):
>>> da nPDyn.dataParser importa IN16B_FWS
>>> esempio = IN16B_FWS(
... ,
... detGroup=[detGroup=]
... ). processo()
# normalizzazione con i primi 5 punti sull'osservabile
# asse, che corrispondono alla temperatura
>>> esempio /= sample[:5].mean(0)
# L'intervallo Q di trasferimento del momento utilizzato qui è più piccolo
# poiché il modello utilizzato è valido solo per Q basso
>>> campione = sample.get_q_range(0,2; 0,8) - Utilizzare un semplice modello gaussiano per iniziare, la cui larghezza è data dal cosiddetto spostamento quadratico medio (MSD). Compilate e adattate il modello utilizzando i seguenti comandi:
>>> importare numpy come np
>>> da nPDyn.models import Parametri, Modello, Componente
# a è un fattore di scala
>>> params = Parameters(
... a={'value': 1, 'bounds': (0, np.inf)},
... msd={'value': 1, 'bounds': (0, np.inf)}
... )
>>> model = Model(params)
>>> model.addComponent(Component(
... «gaussiano»,
... lambda x, a, msd: a * np.exp(-x ** 2 * msd / 6)
... ))
>>> sample.fit(model; x=sample.q[:; None])
>>> trama(esempio)
NOTA: L'approssimazione gaussiana vale sempre per q2MSD << 1, ma è possibile utilizzare un intervallo di trasferimento della quantità di moto più ampio per il confronto relativo tra campioni. Modelli più sofisticati, che vanno oltre l'approssimazione gaussiana, sono stati sviluppati37,38,39.
6. Analisi dei dati - scansioni elastiche e anelastiche a finestra fissa (E/IFWS)
- Analogamente al passaggio 4, importare il set di dati ma utilizzando la classe 'IN16B_FWS':
>>> da nPDyn.dataParser importa IN16B_FWS
>>> esempio = IN16B_FWS(
...
... [detGroup=]
... ). processo()
>>> campione = sample.get_q_range(0,3; 1,8) - Adattare i dati di calibrazione e i dati campione.
- Analizzare i dati E/IFWS utilizzando un MSD40 generalizzato o considerandoli come spettri QENS grossolani (con solo pochi punti dati sull'asse dell'energia). Quando E/IFWS è visto come QENS grossolano, i modelli utilizzati per QENS vengono utilizzati per adattare l'intero set di dati E/IFWS contemporaneamente (adattamento globale dei trasferimenti di energia e trasferimenti di quantità di moto).
NOTA: Quest'ultima soluzione - che utilizza modelli per QENS su dati E/IFWS - è usata qui dove viene imposta la dipendenza dal trasferimento di quantità di moto della diffusione del centro di massa e la dinamica interna delle proteine. - Modellare la dinamica delle proteine nei liquidi usando il seguente Eq (5) ('modelProteinJumpDiff' in nPDyn):
 (5)
(5)
Dove R(q,ω) è la funzione di risoluzione; β uno scalare indipendente per ogni trasferimento di quantità di moto q; a0 è il fattore di struttura elastico incoerente (EISF);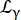 un spiegazione lorentziano della diffusione del centro di massa con una larghezza data da Eq (6);
un spiegazione lorentziano della diffusione del centro di massa con una larghezza data da Eq (6); 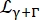 è un lorentziano che include la diffusione del centro di massa e un contributo seguendo il modello di salto-diffusione14 che tiene conto della dinamica interna (Eq (7);
è un lorentziano che include la diffusione del centro di massa e un contributo seguendo il modello di salto-diffusione14 che tiene conto della dinamica interna (Eq (7);  essendo il segnale adattato da D2O riscalato dalla sua frazione di volume nel campione.
essendo il segnale adattato da D2O riscalato dalla sua frazione di volume nel campione.
γ = Dsq2 (6)
Ds è il coefficiente di autodiffusione.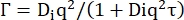 (7)
(7)
D i è il coefficiente di diffusione apparente per la dinamica interna e τ un tempo di rilassamento per i moti diffusivi.
>>> dall'importazione nPDyn.models.builtins (
... modelloPVoigt,
... modelProteinJumpDiff,
... modelloCalibratedD2O,
... )
# I modelli incorporati utilizzano un vettore colonna della quantità di moto
# Trasferisci valori Q
>>> q = vanadio.q[:, Nessuno]
# il vanadio è montato utilizzando un profilo pseudo-Voigt
>>> vanadium.fit(modelPVoigt(q))
# per puro D2O, un modello con larghezza di linea calibrata
# per temperature diverse è incluso in nPDyn
>>> buffer.fit(modelCalibratedD2O(q, temp=363))
# qui, l'equazione 3 viene utilizzata per i campioni liquidi
# convoluzione con funzione di risoluzione e aggiunta di
# Lo sfondo D2O viene eseguito automaticamente con gli argomenti # forniti
>>> sample.fit(modelProteinJumpDiff(q),
... res=vanadio,
... bkgd=buffer,
... volume_fraction_bkgd=0,95
... )
- Analizzare i dati E/IFWS utilizzando un MSD40 generalizzato o considerandoli come spettri QENS grossolani (con solo pochi punti dati sull'asse dell'energia). Quando E/IFWS è visto come QENS grossolano, i modelli utilizzati per QENS vengono utilizzati per adattare l'intero set di dati E/IFWS contemporaneamente (adattamento globale dei trasferimenti di energia e trasferimenti di quantità di moto).
- Tracciare i dati adattati utilizzando:
>>> dal grafico di importazione nPDyn.plot
>>> trama(esempio)
Access restricted. Please log in or start a trial to view this content.
Risultati
L'aggregazione del lisozima in particolato è stata effettuata a 90 °C con una concentrazione proteica di 50 mg/ml in un tampone deuterato (0,1 M NaCl a pD 10,5). La formazione di particolato è innescata dall'aumento della temperatura a 90 °C e avviene entro 6 ore (figura supplementare S8). L'acquisizione dei dati è stata eseguita su IN16B, come descritto nel protocollo sopra (i dati sono curati in modo permanente dall'ILL e accessibili a http://dx.doi.org/10.5291/ILL-DATA.8-04-811).
Access restricted. Please log in or start a trial to view this content.
Discussione
La spettroscopia neutronica è l'unico metodo che consente di sondare la dinamica ps-ns mediata dall'insieme di campioni proteici indipendentemente dalle dimensioni della proteina o dalla complessità della soluzione quando viene utilizzata la deuterazione6. In particolare, sondando l'autodiffusione di gruppi proteici in soluzione, la dimensione idrodinamica di tali assemblaggi può essere determinata in modo inequivocabile. Tuttavia, il metodo è comunemente limitato dal basso flusso di neutroni,...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Gli autori sono grati a Michaela Zamponi del Jülich Centre for Neutron Science presso l'Heinz Maier-Leibnitz Zentrum, Garching, Germania, per parte degli esperimenti di scattering neutronico condotti sullo strumento SPHERES. Questo lavoro ha beneficiato delle attività del consorzio Deuteration Laboratory (DLAB) finanziato dall'Unione Europea nell'ambito dei contratti HPRI-2001-50065 e RII3-CT-2003-505925, e dell'attività finanziata dal UK Engineering and Physical Sciences Research Council (EPSRC) all'interno dell'Institut Laue Langevin EMBL DLAB nell'ambito delle sovvenzioni GR/R99393/01 e EP/C015452/1. Il sostegno della Commissione europea nell'ambito del 7° programma quadro attraverso l'azione chiave: rafforzare lo Spazio europeo della ricerca, le infrastrutture di ricerca è riconosciuto [contratto 226507 (NMI3)]. Kevin Pounot e Christian Beck ringraziano il Ministero Federale dell'Istruzione e della Ricerca (BMBF, numero di sovvenzione 05K19VTB) per il finanziamento delle loro borse di studio post-dottorato.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Aluminum sample holder | Not commercially available. Either the local contact on the instrument can provide them or they can be manufactured based on a technical drawing that can be provided by the local contact. | ||
| Deuterium chloride, 35 wt. % in D2O, ≥99 atom % D | Sigma-Aldrich | 543047 | |
| Deuterium oxide (D, 99.9%) | Eurisotop | DLM-4DR-PK | |
| Dow Corning high-vacuum silicone grease | Sigma-Aldrich | Z273554-1EA | |
| Ethanol 96%, EMSURE Reag. Ph Eur | Sigma-Aldrich | 1.5901 | |
| Glass dessicator | VWR | 75871-660 | |
| Glass dessicator plate, 140 mm | VWR | 89038-068 | |
| Indium wire, 1.0 mm (0.04 in) dia, Puratronic, 99.999% | Alfa Aesar | 00470.G1 | |
| Lysozyme from chicken egg white dialyzed, lyophilized, powder, ~100,000 U/mg | Sigma-Aldrich | 62970 | |
| nPDyn | v3.x | see github.com/kpounot/nPDyn, model functions fot fitting also included in the software | |
| OHAUS AX324 Adventurer balance, internal calibration | Dutscher | 92641 | |
| Phosphorus pentoxide, ReagentPlus, 99% | Sigma-Aldrich | 214701 | |
| Pipette ErgoOne 0.5-10 μL | Starlab | S7100-0510 | |
| Pipette ErgoOne 100-1,000 μL | Starlab | S7100-1000 | |
| Pipette ErgoOne 20-200 μL | Starlab | S7100-2200 | |
| Pipette tip TipOne 1,000 μL | Starlab | S1111-6001 | |
| Pipette tip TipOne 10 μL | Starlab | S1111-3200 | |
| Pipette tip TipOne 200 μL | Starlab | S1111-0206 | |
| Sodium deuteroxide solution, 40 wt. % in D2O, 99.5 atom % D | Sigma-Aldrich | 372072 |
Riferimenti
- Jacrot, B. Des neutrons pour la science: Histoire de l'Institut Laue-Langevin. Des neutrons pour la science. EDP Sciences. , (2021).
- Mahieu, E., Gabel, F. Biological small-angle neutron scattering: recent results and development. Acta Crystallographica Section D. 74 (8), 715-726 (2018).
- Grimaldo, M., Roosen-Runge, F., Zhang, F., Schreiber, F., Seydel, T. Dynamics of proteins in solution. Quarterly Reviews of Biophysics. 52, 7(2019).
- Martel, A., et al. Membrane permeation versus amyloidogenicity: A multitechnique study of islet amyloid polypeptide interaction with model membranes. Journal of the American Chemical Society. 139 (1), 137-148 (2017).
- Pounot, K., et al. Tracking internal and global diffusive dynamics during protein aggregation by high-resolution neutron spectroscopy. The Journal of Physical Chemistry Letters. 11 (15), 6299-6304 (2020).
- Grimaldo, M., et al. Protein short-time diffusion in a naturally crowded environment. The Journal of Physical Chemistry Letters. 10 (8), 1709-1715 (2019).
- Jasnin, M., Stadler, A., Tehei, M., Zaccai, G. Specific cellular water dynamics observed in vivo by neutron scattering and NMR. Physical Chemistry Chemical Physics. 12 (35), 10154-10160 (2010).
- Frick, B. The neutron backscattering spectrometer IN16 at ILL-high energy resolution with high intensity and excellent signal-to-noise ratio. Neutron News. 13 (2), 15-22 (2002).
- Frick, B., Mamontov, E., van Eijck, L., Seydel, T. Recent backscattering instrument developments at the ILL and SNS. Zeitschrift für Physikalische Chemie. 224 (1-2), 33-60 (2010).
- Frick, B., Combet, J., van Eijck, L. New possibilities with inelastic fixed window scans and linear motor Doppler drives on high resolution neutron backscattering spectrometers. Nuclear Instruments and Methods in Physics Research Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 669, 7-13 (2012).
- Appel, M., Frick, B., Magerl, A. A flexible high speed pulse chopper system for an inverted neutron time-of-flight option on backscattering spectrometers. Scientific Reports. 8 (1), 13580(2018).
- Squires, G. L. Introduction to the theory of thermal neutron scattering. , Dover Publications. Mineola N.Y. (1996).
- Bee, M. Quasielastic neutron scattering. , Available from: http://inis.iaea.org/Search/search.aspx?orig_q=RN:20038756 (1988).
- Singwi, K. S., Sjölander, A. Diffusive motions in water and cold neutron scattering. Physical Review. 119 (3), 863-871 (1960).
- Sears, V. F. Theory of cold neutron scattering by homonuclear diatomic liquids: i. free rotation. Canadian Journal of Physics. 44 (6), 1279-1297 (1966).
- Sears, V. F. Theory of cold neutron scattering by homonuclear liquid: ii. hindered rotation. Canadian Journal of Physics. 44 (6), 1299-1311 (1966).
- Schirò, G., et al. Translational diffusion of hydration water correlates with functional motions in folded and intrinsically disordered proteins. Nature Communications. 6, 6490(2015).
- Grimaldo, M., et al. Hierarchical molecular dynamics of bovine serum albumin in concentrated aqueous solution below and above thermal denaturation. Physical Chemistry Chemical Physics. 17 (6), 4645-4655 (2015).
- Eanes, E. D., Glenner, G. G. X-ray diffraction studies on amyloid filaments. Journal of Histochemistry & Cytochemistry. 16 (11), 673-677 (1968).
- Bonar, L., Cohen, A. S., Skinner, M. M. Characterization of the Amyloid Fibril as a Cross-β Protein. Proceedings of the Society for Experimental Biology and Medicine. 131 (4), 1373-1375 (1969).
- Chiti, F., Dobson, C. M. Protein Misfolding, Amyloid Formation, and Human Disease: A Summary of Progress Over the Last Decade. Annual Review of Biochemistry. 86 (1), 27-68 (2017).
- Knowles, T. P. J., Vendruscolo, M., Dobson, C. M. The amyloid state and its association with protein misfolding diseases. Nature Reviews Molecular Cell Biology. 15 (6), 384-396 (2014).
- Maji, S. K., et al. Functional amyloids as natural storage of peptide hormones in pituitary secretory granules. Science. 325 (5938), 328-332 (2009).
- Li, J., et al. The RIP1/RIP3 necrosome forms a functional amyloid signaling complex required for programmed necrosis. Cell. 150 (2), 339-350 (2012).
- Knowles, T. P. J., Mezzenga, R. Amyloid fibrils as building blocks for natural and artificial functional materials. Advanced Materials. 28 (31), 6546-6561 (2016).
- Stephens, A. D., Kaminski Schierle, G. S. The role of water in amyloid aggregation kinetics. Current Opinion in Structural Biology. 58, 115-123 (2019).
- Adamcik, J., Mezzenga, R. Amyloid polymorphism in the protein folding and aggregation energy landscape. Angewandte Chemie International Edition. 57 (28), 8370-8382 (2018).
- Liu, Z., et al. Entropic contribution to enhanced thermal stability in the thermostable P450 CYP119. Proceedings of the National Academy of Sciences of the United States of America. 115 (43), 10049-10058 (2018).
- Coquelle, N., et al. Chromophore twisting in the excited state of a photoswitchable fluorescent protein captured by time-resolved serial femtosecond crystallography. Nature Chemistry. 10 (1), 31-37 (2018).
- Henzler-Wildman, K. A., et al. A hierarchy of timescales in protein dynamics is linked to enzyme catalysis. Nature. 450 (7171), 913-916 (2007).
- Fichou, Y., et al. Hydration water mobility is enhanced around tau amyloid fibers. Proceedings of the National Academy of Sciences of the United States of America. 112 (20), 6365-6370 (2015).
- Burns, J., Pennock, C. A., Stoward, P. J. The specificity of the staining of amyloid deposits with thioflavine T. The Journal of Pathology and Bacteriology. 94 (2), 337-344 (1967).
- Iqbal, K., Liu, F., Gong, C. -X., Grundke-Iqbal, I. Tau in Alzheimer disease and related tauopathies. Current Alzheimer Research. 7 (8), 656-664 (2010).
- Krȩżel, A., Bal, W. A formula for correlating pKa values determined in D2O and H2O. Journal of Inorganic Biochemistry. 98 (1), 161-166 (2004).
- Dolman, M., Halling, P. J., Moore, B. D., Waldron, S. How dry are anhydrous enzymes? Measurement of residual and buried 18O-labeled water molecules using mass spectrometry. Biopolymers. 41 (3), 313-321 (1997).
- Pounot, K. kpounotnPDyn: v3.0.0. Zenodo. , (2021).
- Yi, Z., Miao, Y., Baudry, J., Jain, N., Smith, J. C. Derivation of mean-square displacements for protein dynamics from elastic incoherent neutron scattering. Journal of Physical Chemistry B. 116 (16), 5028-5036 (2012).
- Peters, J., Kneller, G. R. Motional heterogeneity in human acetylcholinesterase revealed by a non-Gaussian model for elastic incoherent neutron scattering. The Journal of Chemical Physics. 139 (16), 165102(2013).
- Zeller, D., Telling, M. T. F., Zamponi, M., García Sakai, V., Peters, J. Analysis of elastic incoherent neutron scattering data beyond the Gaussian approximation. The Journal of Chemical Physics. 149 (23), 234908(2018).
- Roosen-Runge, F., Seydel, T. A generalized mean-squared displacement from inelastic fixed window scans of incoherent neutron scattering as a model-free indicator of anomalous diffusion confinement. EPJ Web of Conferences. 83, 02015(2015).
- Ortega, A., Amorós, D., García de la Torre, J. Prediction of hydrodynamic and other solution properties of rigid proteins from atomic- and residue-level models. Biophysical Journal. 101 (4), 892-898 (2011).
- Hennig, M., Frick, B., Seydel, T. IUCr Optimum velocity of a phase-space transformer for cold-neutron backscattering spectroscopy. Journal of Applied Crystallography. 44 (3), 467-472 (2011).
- Paalman, H. H., Pings, C. J. Numerical evaluation of X-ray absorption factors for cylindrical samples and annular sample cells. Journal of Applied Physics. 33 (8), 2635-2639 (1962).
- Ow, S. -Y., Dunstan, D. E. The effect of concentration, temperature and stirring on hen egg white lysozyme amyloid formation. Soft Matter. 9 (40), 9692-9701 (2013).
- Tominaga, T., Sahara, M., Kawakita, Y., Nakagawa, H., Yamada, T. Evaluation of sample cell materials for aqueous solutions used in quasi-elastic neutron scattering measurements. Journal of Applied Crystallography. 54 (6), 1631-1640 (2021).
- Beck, C., et al. Following protein dynamics in real time during crystallization. Crystal Growth & Design. 19 (12), 7036-7045 (2019).
- Smith, A. A., Testori, E., Cadalbert, R., Meier, B. H., Ernst, M. Characterization of fibril dynamics on three timescales by solid-state NMR. Journal of Biomolecular NMR. 65 (3-4), 171-191 (2016).
- Wang, T., Jo, H., DeGrado, W. F., Hong, M. Water distribution, dynamics, and interactions with Alzheimer's β-amyloid fibrils investigated by solid-state NMR. Journal of the American Chemical Society. 139 (17), 6242-6252 (2017).
- Rezaei-Ghaleh, N., Giller, K., Becker, S., Zweckstetter, M. Effect of zinc dinding on β-amyloid structure and dynamics: Implications for Aβ aggregation. Biophysical Journal. 101 (5), 1202-1211 (2011).
- Vugmeyster, L., et al. Fast motions of key methyl groups in amyloid-β fibrils. Biophysical Journal. 111 (10), 2135-2148 (2016).
- Yang, X., Wang, B., Hoop, C. L., Williams, J. K., Baum, J. NMR unveils an N-terminal interaction interface on acetylated-α-synuclein monomers for recruitment to fibrils. Proceedings of the National Academy of Sciences of the United States of America. 118 (18), (2021).
- Tuttle, M. D., et al. Solid-state NMR structure of a pathogenic fibril of full-length human α-synuclein. Nature Structural & Molecular Biology. 23 (5), 409-415 (2016).
- Karamanos, T. K., Kalverda, A. P., Thompson, G. S., Radford, S. E. Mechanisms of amyloid formation revealed by solution NMR. Progress in Nuclear Magnetic Resonance Spectroscopy. 88-89, 86-104 (2015).
- Lai, Y. -C., Kuo, Y. -H., Chiang, Y. -W. Identifying protein conformational dynamics using spin-label ESR. Chemistry - An Asian Journal. 14 (22), 3981-3991 (2019).
- Franck, J. M., Han, S. Overhauser dynamic nuclear polarization for the study of hydration dynamics, explained. Methods in Enzymology. 615, 131-175 (2019).
- Pavlova, A., et al. Protein structural and surface water rearrangement constitute major events in the earliest aggregation stages of tau. Proceedings of the National Academy of Sciences of the United States of America. 113 (2), 127-136 (2016).
- Lin, Y., et al. Liquid-liquid phase separation of tau driven by hydrophobic interaction facilitates fibrillization of tau. bioRxiv. , (2020).
- Decatur, S. M. Elucidation of residue-level structure and dynamics of polypeptides via isotope-edited infrared spectroscopy. Accounts of Chemical Research. 39 (3), 169-175 (2006).
- Chatani, E., Tsuchisaka, Y., Masuda, Y., Water Tsenkova, R. molecular system dynamics associated with amyloidogenic nucleation as revealed by real time near infrared spectroscopy and aquaphotomics. PLoS One. 9 (7), 101997(2014).
- Goret, G., Aoun, B., Pellegrini, E. MDANSE: An interactive analysis environment for molecular dynamics simulations. Journal of Chemical Information and Modeling. 57 (1), 1-5 (2017).
- Fujiwara, S., et al. Internal dynamics of a protein that forms the amyloid fibrils observed by neutron scattering. Journal of the Physical Society of Japan. 82, Suppl A (2013).
- Schiró, G., et al. Neutron scattering reveals enhanced protein dynamics in concanavalin a amyloid fibrils. Journal of Physical Chemistry Letters. 3 (8), 992-996 (2012).
- Pounot, K., et al. Zinc determines dynamical properties and aggregation kinetics of human insulin. Biophysical Journal. 120 (5), 886-898 (2021).
- Fujiwara, S., et al. Dynamic properties of human α-synuclein related to propensity to amyloid fibril formation. Journal of Molecular Biology. 431 (17), 3229-3245 (2019).
- Sanz, A., et al. High-pressure cell for simultaneous dielectric and neutron spectroscopy. Review of Scientific Instruments. 89 (2), 023904(2018).
- Adams, M. A., et al. Simultaneous neutron scattering and Raman scattering. Applied Spectroscopy. 63 (7), 727-732 (2009).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon