A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
טיהור הרטיקולום הסרקו-אנדופלזמי Ca2+-ATPase משריר הארנב
In This Article
Summary
פרוטוקול זה מתאר שיטת טיהור SERCA משופרת, הכוללת את הטרהלוז הדו-סוכר בשלב הצנטריפוגה הסופי. פחמימה זו מייצבת חלבונים בתנאים קשים. ה-SERCA המטוהר היה פעיל קטליטית והפגין טוהר גבוה, מה שהופך אותו למתאים למחקרים מבניים ופונקציונליים.
Abstract
חלק מה-ATPases מסוג P, כגון סרקו/רשתית אנדופלזמית Ca2+-ATPase (SERCA), הם חלבוני ממברנה גמישים מטבעם הדורשים תנאים פיזיקוכימיים ספציפיים במהלך הטיהור כדי להשיג אותם בטוהר ובאיכות מבנית גבוהה ובצורה פעילה קטליטית. הטרהלוז הדו-סוכר הוא מומס תואם המסונתז ומצטבר בריכוזים גבוהים בציטופלזמה של השמרים כדי לייצב את הממברנות והחלבונים. השימוש בטרהלוז כתוסף בפרוטוקול לטיהור קרום הפלזמה H+-ATPase מביא לתכשיר איכותי, שהמבנה ההקסמרי שלו מוצג בשיטות אנליטיות ביוכימיות. טרהלוז יכול, אם כן, לשמש כתוסף מייצב לטיהור חלבוני ממברנה (P-ATPases). פרוטוקול זה מתאר את שינוי הפרוטוקול הקלאסי לטיהור SERCA על ידי הכפפת SERCA לצנטריפוגה בשיפוע ריכוז טרהלוז. הכללת פחמימה זו הובילה לטיהור SERCA בצורה פעילה קטליטית עם טוהר גבוה וחשוב מכך, בצורה יציבה. אפיון ביוכימי חלקי של ה-SERCA המטוהר (SDS-PAGE, קינטיקה של אנזימים, תיוג FITC, ספקטרוסקופיה של דיכרואיזם מעגלי) הראה כי האנזים מתאים למחקרים פונקציונליים ומבניים. מוצע להשתמש בטרהלוז בפרוטוקול הטיהור של ATPases מסוג P וחלבוני ממברנה (וציטוזוליים) אחרים.
Introduction
חלבוני ממברנה/אנזימים הם מרכיבים ביולוגיים חיוניים של התאים מכיוון שהם ממלאים תפקידים קריטיים בתהליכים שונים 1,2,3. חלק מהפונקציות עשויות לכלול הובלה של יונים ומולקולות פנימה והחוצה מהתא/תאים פנימיים (אקטיבי/פסיבי), זיהוי תא-תא, קשירה בין-תאית, עיגון/התקשרות וחישה של הסביבה החיצונית באמצעות אינטגרציה עם מנגנון העברת האותות בתנאים פיזיקליים וכימיים רגילים וקשים (מלח גבוה, מים נמוכים, טמפרטורה גבוהה, עמידות לתרופות, וכו')3 לכן, קביעת המבנה התלת מימדי (תלת מימדי) של חלבוני ממברנה ו/או אנזימים הפכה להיות בעלת חשיבות רבה הן למחקר בסיסי והן למחקר יישומי 4,7,6. חשוב לציין, חלבונים/אנזימים ממברניים שימשו באופן נרחב כמטרות לגילוי תרופות (בין אם טבעיות או מתוכננות)7,8,9. כלומר, לחלבוני הממברנה יש חשיבות אינהרנטית בבריאות 9,10,11.
האופי ההידרופובי של חלבוני ממברנה/אנזימים הוא המאפיין הפיזיקוכימי המאתגר ביותר מבחינה טכנית עבור מעבדת הניסוי 12,13,14, על אחת כמה וכמה כאשר עובדים עם חלבוני ממברנה אינטגרליים אוליגומריים ו/או אינטגרליים מאוד15,16. בידוד כמויות מתאימות של חלבון/אנזים הממברנה באיכות הגבוהה ביותר האפשרית לבדיקות ניסיוניות פונקציונליות ומחקרים מבניים רצוי מאוד17. חלבוני ממברנה, בהיותם הידרופוביים מטבעם, קשים מאוד לטיהור, ובחירת חומר הניקוי היא בדרך כלל אחד הנושאים החשובים ביותר שיש לקחת בחשבון 17,18,19,20. בהקשר זה, שיטות המעבדה המשמשות לבידוד חלבוני ממברנה גורמות בדרך כלל למידה מסוימת של נזק לסידור המבני התלת-ממדי של החלבון21. חלק מהשיטות הללו כוללות שימוש ב-(א) סוניקציה (גלים קוליים בתדר/אנרגיה גבוהה), (ב) חומרי ניקוי מסיסים בחלבון (קשים, בינוניים או עדינים)22, (ג) לחצים גבוהים יחסית בכרומטוגרפיה של עמודות ואולטרה-צנטריפוגה במהירות גבוהה23, (ד) מולקולות משקעות, (ה) אנזימי עיכול ואחרים21. כל התהליכים הללו יכולים לתרום או להיות הגורם העיקרי לחוסר יציבות החלבון במהלך הטיהור21. מבחינה זו, נראה כי פרוטוקולים מסוימים עובדים טוב יחסית עבור חלבון ממברנה נתון. עם זאת, אופטימיזציה תמיד מתקבלת בברכה בעת שימוש במבחנים מודרניים חדשים או בשיטות הדורשות איכות גבוהה יותר של הכנת חלבון ממברנה כדי להשיג תוצאות משביעות רצון24. שלבי אופטימיזציה עשויים לכלול, אך אינם מוגבלים לשיפור עיצוב המבנה, מציאת תנאים אופטימליים לביטוי חלבון ממברנה, קביעת תנאי טיפול טובים יותר (כלומר, pH, טמפרטורה וכו'), מציאת חומר הניקוי התואם הטוב ביותר, התאמת שלבי טיהור כגון זמן סוניקציה, מהירות צנטריפוגה, ניסוח מחדש של פתרונות חיץ על ידי הוספת חומרים מייצבים וכו'.25,26,27. לכן, כל שינוי במתודולוגיית הטיהור שמוביל לעלייה באיכות (טוהר) ובפעילות של חלבון/אנזים הממברנה המטוהרת הוא חשוב.
במשפחת ATPase מסוג P, נראה כי קרום הפלזמה של השמרים H+-ATPase הוא אחד החברים הגמישים ביותר28,29. ה-H+-ATPase מאבד את פעילות ההידרוליזה של ה-ATP שלו עם התייבשות (ייבוש בהקפאה), הלם חום וכו'.28,29. השימוש בטרהלוז כמייצב חלבון נבדק בבידוד קרום הפלזמה H+-ATPase מהשמרים K. lactis30,31; תכשיר H+-ATPase שהתקבל היה בעל טוהר גבוה ופעיל קטליטי. חשוב לציין, זה איפשר לפתור את המצב האוליגומרי של האנזים בשיטות ביוכימיות, וחשף שהוא הקסמר ואושר מאוחר יותר על ידי מיקרוסקופ אלקטרוניםקריו 31,32,33,34,35. לכן, נראה סביר שהסידור התלת-ממדי העדין (תלת-ממדי) של חלבוני הממברנה יכול ללכת לאיבוד בתנאים קשים יחסית במהלך הטיהור36. אינאקטיבציה דו-פאזית קינטית נצפתה עבור H+-ATPase במהלך השבתה בתיווך חום29. בתאי שמרים, כמו באורגניזמים רבים אחרים, הטרהלוז הדו-סוכר מצטבר בריכוזים גבוהים בתנאי עקה סביבתית 37,38,39. טרהלוז שומר על שלמות הממברנה ותפקוד ההובלה על ידי ייצוב חלבונים (הן משובצים בממברנה והן ציטוזוליים) וממברנות התאים40,41. מנגנון הייצוב של טרהלוז נחקר בהרחבה על ידי מספר קבוצות ועל ידי המעבדה שלנו 42,43,44,45. ניסויים עם H+-ATPase ואנזימים אחרים הראו כי טרהלוז הוא מייצב החלבון היעיל ביותר מבין מונו-דו-סוכרים28,29. זה הוביל להכללתו בפרוטוקול הטיהור H+-ATPase30. לאחרונה, טרהלוז שימש גם לטיהור הרטיקולום הסרקופלזמי Ca2+-ATPase (SERCA) משריר העוויתות המהיר של ארנב עם תוצאות טובות בטוהר החלבון ובפעילות46. לכן, נראה כי טרהלוז הוא תוסף טוב ומתאים לטיהור ATPases מסוג P וכנראה חלבונים ממברניים וציטוזוליים אחרים.
עבור P-ATPases, קיומם של תחומים ציטופלזמיים מבניים הוא יתרון ניסיוני, במיוחד עבור מחקרי אינטראקציה בין סובסטרט/ליגנד47; קשירת ATP נחקרה בתחומי N רקומביננטיים טהורים ביותר 47,48,49, ובכך מבטלת את השיקולים הטכניים לטיהור אנזימי ממברנה שלמים 47,48,50, בין היתר51. למרבה הצער, חלק מהמחקרים הפונקציונליים (המרה קטליטית/אנרגיה) והמבניים (סידור תת-יחידות ואינטראקציה עם חלבונים אחרים) עדיין דורשים את כל ה-P-ATPase52,53. בהקשר זה, טיהור SERCA הושג על ידי מספר קבוצות מחקר 54,55,56,57,58. עם זאת, עדיין ניתן ליישם שיפורים46, למשל, הגדלת שלמות ה-ATPase המטוהר59, הימנעות מדנטורציה/שיבוש של קומפלקסי חלבון25, הגברת המסיסות ללא דנטורציה של חלבון הממברנה (כלומר, הימנעות מהיווצרות אגרגטים מקרומולקולריים)60, להוביל לתאימות טובה יותר עם מבחנים ושיטות אנליטיות אחרות במורד הזרם61 יתר על כן, מכיוון שאסטרטגיות ניסיוניות חדשות, תוספים, מעכבי אנזימים וכו' מופיעים בספרות המדעית 61,62,63,64,65,66, לפעמים יש צורך לבדוק אותם עם כל ה-P-ATPase. עבודה זו מתארת את הפרוטוקול לטיהור SERCA והשימוש בטרהלוז כתוסף לייצוב מבנה החלבון ופעילות ה-ATPase; כלומר, בנוסף להגברת איכות האנזים (המבני), טרהלוז מסייע במניעת אובדן מבנה ופעילות האנזים במהלך בידוד ואחסון האנזים, מה שעוזר לחסוך בחומר ביולוגי ובכך להפחית את מספר טיהורי האנזימים.
Protocol
כל ההליכים בבעלי חיים בוצעו בהתאם להנחיות בינלאומיות ומקומיות (NORMA Oficial Mexicana NOM-062-ZOO-1999) לטיפול בבעלי חיים במעבדות ניסוי 51,67,68. רקמת שריר התקבלה מ-Oryctolagus cuniculus מסוג בר מיחידה מקומית לטיפול בבעלי חיים (INE/CITES/DGVS-ZOO-E0055-SLP-98)46. וטרינר עם מומחיות בניהול חיות מעבדה ביצע את הדיסקציה והעיבוד הראשוניים של השרירים. פרטי הריאגנטים והציוד המשמש במחקר זה מפורטים בטבלת החומרים.
1. בידוד של רשתית סרקופלזמית משריר ארנב מהיר (תזמון, 7 - 9 שעות)
הערה: לפרטים על ההליך, עיין ב-Champeil et al.69.
- השג רקמת שריר מהירה (~70 גרם משתי הרגליים האחוריות לכל חיה) מ-Oryctolagus cuniculus מסוג בר.
הערה: נדרש זיהוי חזותי מוקדם של השרירים שיש לנתח70,71. - השרו את שריר העוויתות המהיר בבלנדר על ידי תלייתו בשלושה נפחים של 100 מ"מ של KCl, עם דקה אחת ודקה אחת במנוחה קרה כקרח, חזור על הפעולה שלוש פעמים72.
- הסר את פסולת הרקמה על ידי צנטריפוגה (1500 × גרם) בטמפרטורה של 4 מעלות צלזיוס למשך 5 דקות; צנטריפוגה שנייה (4200 × גרם) מבוצעת במידת הצורך.
- אסוף את הסופרנטנט והומוגניזציה שלו באמצעות מטחנת רקמות (10 משיכות במהירות נמוכה, כלומר 500-1,500 סל"ד)72,73. צנטריפוגה את ההומוגנאט (10,000 × גרם) בטמפרטורה של 4 מעלות צלזיוס למשך 15 דקות.
הערה: יש לנקוט בזהירות בעת הומוגניזציה, שכן שימוש במהירות גבוהה יחסית עלול לגרום לנזק למערכת ולמפעיל. - צנטריפוגה הסופרנטנט (המכיל את הרטיקולום הסרקופלזמי) (33,000 × גרם) בטמפרטורה של 4 מעלות צלזיוס למשך 120 דקות.
- השעו את הכדורים ב-40 מ"ל של 0.5 מ' סוכרוז וצנטריפוגה (12,000 × גרם) ב-4 מעלות צלזיוס למשך 15 דקות. יש לדלל את הסופרנטנט (0.6 מ' KCl ו-0.15 מ' סוכרוז) ולצנטריפוגה את התרחיף (34,000 × גרם) בטמפרטורה של 4 מעלות צלזיוס למשך 165 דקות.
- השעו את הגלולה המכילה את יריעות שלפוחיות הרטיקולום הסרקופלזמיות (SRVs) ב-0.3 מ' סוכרוז, 0.1 מ' KCl ו-5 מ"מ של Tris-HCl, pH 7.0. קבע את ריכוז החלבון על ידי שימוש במבחן Lowry74 ובאלבומין בסרום אנושי כתקן החלבון.
- יש להשתמש ב-SRVs (30-32 מ"ג/מ"ל) בנפח של 1 מ"ל. אחסן את דגימות ה-SRVs ב-72 מעלות צלזיוס להמשך עיבוד.
הערה: תפוקת החלבון היא 100-110 מ"ג SRV לבעל חיים.
2. טיהור SERCA (תזמון, 18 - 20 שעות)
הערה: לפרטים על ההליך, עיין ב-Rivera-Morán et al.46.
- יש לדלל את תרחיף ה-SRVs ל-2 מ"ג חלבון/מ"ל באמצעות 75 מ"מ קר כקרח של Tris-HCl, pH 7.2, 0.6 מ"מ של KCl, 6 מ"מ של EDTA, 1 מ"מ של EGTA ו-0.1% (w/v) דאוקסיכולאט (DOC). דגרו את מתלי ה-SRV על קרח למשך 10 דקות ותסיסה עדינה. צנטריפוגה מתלה ה-SRV (100,000 × גרם) ב-4 מעלות צלזיוס למשך 60 דקות.
הערה: יש להוסיף DOC טיפה אחר טיפה. - השעו את הכדורים ב-<3 מ"ל של 25 מ"מ של Tris-HCl, pH 7.5, 0.3 מ' של KCl, 45% גליצרול (v/v) ו-2 מ"מ EDTA. הומוגניזציה של המתלה והתאם לנפח סופי של 10 מ"ל. קבע את ריכוז החלבון ולאחר מכן התאם אותו ל-6.5 מ"ג/מ"ל. הוסף אזולקטין במינון של 5 מ"ג/מ"ל ו-0.85 (w/w) Zwittergent 3-14.
הערה: יש להוסיף אזולקטין ו-Zwittergent 3-14 טיפה אחר טיפה. - הומוגניזציה של המתלים והצנטריפוגה (100,000 × גרם) בטמפרטורה של 4 מעלות צלזיוס למשך 60 דקות. אוספים את הסופרנטנט ומדללים 1:2 עם 2 מ"מ של EGTA (pH 7.2).
- יוצקים את התרחיף בעדינות על שיפוע ריכוז טרהלוז לא רציף (45, 40, 35 ו-30% משקל/נפח) ב-10 מ"מ של Tris-HCl, pH 7.0, 1 מ"מ של EDTA, 0.1% דאוקסיכולט ו-1 מ"ג/מ"ל אזולקטין. צנטריפוגה (100,000 × גרם) בטמפרטורה של 4 מעלות צלזיוס למשך 14 שעות. אוספים את הגלולה השקופה והמעט צהבהבה שנוצרה בתחתית הצינורות.
הערה: יש לבצע הן את היווצרות שיפוע הטרהלוז והן את הוספת תרחיף הדגימה בטכניקות מתאימות. מומלץ לבצע צנטריפוגה למשך הלילה. - תלו בעדינות את הגלולה בנפח קטן (<1.5 מ"ל) של 25 מ"מ של Tris-HCl, pH 7.5, 0.3 של M KCl, 45% גליצרול ו-2 מ"מ של EDTA. קבע את ריכוז החלבון. התאם את ריכוז החלבון על ידי דילול ל-2 מ"ג/מ"ל. קח כמויות (~50 מיקרוליטר) של התרחיף ואחסן את ה-SERCA המטוהר ב-72 מעלות צלזיוס עד השימוש בו.
- בצע SDS-PAGE של SERCA72 המטוהר, ושל דגימות חלבון שנלקחו בשלבים שונים של טיהור. מכתים את הג'ל בכחול Coomassie72.
הערה: השימוש ב-0.6 M טרהלוז במקום גליצרול במאגר התרחיף מגביר את יציבות האנזים במהלך מחזורי האחסון וההקפאה-הפשרה 28,29.
3. בדיקה לפעילות ATPase (תזמון, 1 - 2 שעות)
- בדיקה מצומדת אנזימים: ATPase-Piruvate קינאז-לקטט דהידרוגנאז
- הכן את תערובת התגובה (1 מ"ל): 50 מ"מ MOPS (pH 7.0), 1 מ"מ EGTA, 80 מ"מ KCl, 5 מ"מ MgCl2, 3 מ"מ CaCl2, 5 מ"מ פוספואנולפירובאט, 250 מיקרומטר NADH. כלול ריכוזי ATP משתנים (0.001 עד 0.25 מ"מ).
- הומוגניזציה בזהירות של בדיקת התגובה על ידי מערבולת. דגרו את בדיקת התגובה ב-37 ◦C למשך 10 דקות. הוסיפו 0.9 U L-lactate dehydrogenase (LDH) ו-1.5 U pyruvate kinase (PK).
- התחל את תגובת ATPase על ידי תוספת של 10 מיקרוגרם של SERCA.
- קבע את היווצרות NADH. עקוב אחר השינוי בעוצמת הספיגה בכל שנייה במשך 600 שניות באורך גל (λ) של 340 ננומטר לאורך זמן באמצעות ספקטרופוטומטר עם מחזיק תא תרמוסטט.
הערה: מרווח הזמן למדידת הספיגה יכול להשתנות, אך נסה לרשום כמה שיותר נקודות נתונים כדי לצייר את הקו הישר הראשוני של תגובת ה-ATPase.
- קבע את קצב הידרוליזה של ATP
הערה: לפרטים על ההליך, עיין ב-Rivera-Morán et al.46.- חשב את ערך השיפוע (Δabs/min) של החלק הליניארי בכל עקומה שנוצרה. השתמש במקדם ההכחדה המולארי (ε) של NADH (λ = 6,220 M−1·cm−1) כדי לקבוע את פעילות ה-ATPase (μmoles ATP הידרוליזה/min.mg prot.)
- קבע את הפרמטרים הקינטיים Km ו - Vmax
- שרטט את נתוני המהירות מול ריכוז ה-ATP באמצעות תוכנת ניתוח נתונים וגרפים.
- התאם את הנתונים על ידי רגרסיה לא ליניארית למשוואת מיכאליס-מנטן (שווה ערך 1)46:
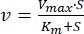
כאשר v היא המהירות, Vmax היא המהירות המקסימלית, [S] הוא ריכוז ה-ATP, ו-Km הוא קבוע מיכאליס-מנטן. - קבע Km ו - Vmax על ידי איטרציה.
הערה: הכללת DOC עשויה להגביר את תגובת קצב ה-ATPase; מומלץ לבדוק ריכוזים שונים של DOC כדי למצוא את האופטימלי.
4. בדיקת תיוג של SERCA עם פלואורסצאין איזותיוציאנט (FITC) (תזמון, 30 - 45 דקות, רק לתיוג FITC)
הערה: לפרטים על ההליך, עיין ב-54,75,76.
- השהה SERCA (20 מיקרוגרם) ב-50 מיקרוליטר (נפח סופי) של מאגר תיוג (100 מ"מ של KCl, 5 מ"מ MgCl2 ו-30 מ"מ של Tris-HCl, pH 8.9) המכיל 1 מ"מ של FITC. מערבבים על ידי מערבולת.
- דגרו את הדגימות בזמנים שונים (15 דקות, 10 דקות, 7 דקות, 5 דקות ו-2 דקות) בחושך ובטמפרטורת החדר.
- עצור את תגובת התיוג על ידי הוספת נפח אחד של מאגר עצירה קר כקרח (480 מ"מ סוכרוז ו-48 מ"מ MOPS, pH 7.0), המכיל ATP (5 מ"מ). דגירה על קרח במשך 5 דקות בחושך.
- הכפיף את ה-SERCA שכותרתו FITC ל-SDS-PAGE72. חשוף את הג'לים השקופים לאור UV (λ = 302 ננומטר). תצלום מתעד את התוצאה.
- מכתימים את הג'ל בכחול Coomassie. תצלום מתעד את התוצאה.
הערה: ביצוע תיוג FITC בחדר חשוך עשוי לשפר את איכות אות הקרינה.
5. ספקטרום דיכרואיזם מעגלי (CD) (תזמון, 30 דקות)
הערה: לפרטים על ההליך, עיין בסעיף11,77.
- השהה SERCA (3 מיקרומטר) ב-400 מיקרוליטר של מאגר פוספט של 10 מ"מ (pH 7.0) ב-25 מעלות צלזיוס.
- טען את הדגימה בתא באורך נתיב של 0.1 ס"מ. הגדר טווח λ של ספקטרום UV רחוק בין 190-260 ננומטר. הגדר את הרזולוציה הפנימית ואת רוחב הפס ל -1 ננומטר.
- הקלט את אות התקליטור ב-50 ננומטר/דקה ב-25 מעלות צלזיוס.
תוצאות
SDS-PAGE של SERCA בשלבים שונים של טיהור (איור 1). הג'ל המוכתם בכחול של Coomassie מראה את ההעשרה של רצועת החלבון SERCA (משקל מולקולרי לכאורה >100 kDa) ככל שפרוטוקול הטיהור מתקדם. רצועת החלבון המתאימה ל-SERCA מציגה טוהר של >90% לאחר צנטריפוגה בשיפוע ריכוז טרהלוז. תרשים של קצב הידרו?...
Discussion
רוב המולקולות והיונים אינם יכולים לחצות בחופשיות את קרומי התאים, למשל, פרוטון (H+) דורש טרנספורטר ממברנה בקרום הפלזמה של מגוון אורגניזמים ואברונים כגון מיטוכונדריה83,84. ממברנות התא הן סלקטיביות, והמולקולות והיונים שחוצים את קרומי התא...
Disclosures
המחבר מצהיר שאין לו אינטרסים כלכליים מתחרים.
Acknowledgements
המחבר מודה לעזרתם של אדמונדו מאטה-מוראלס בעריכת הסרטון, VM, ולנטין דה לה קרוז-טורס בטיהור ה-SRV, מיגל א. ריברה-מורן בטיהור וניתוח SERCA, וחואן ס. גונזלס-קסטרו, פרנקו א. חוארז, אלחנדרה נברז, ניקולס רושה-ויזואט וג'וסלין א. רמירז-אלונסו בהפקת וידאו. לא התקבלו כספים, מענקים או תמיכה אחרת לביצוע מחקר זה.
Materials
| Name | Company | Catalog Number | Comments |
| ATP | Sigma-Aldrich Corp | A2383 | |
| Azolectin from soybean | Sigma-Aldrich Corp | 44924 | |
| Benchtop UV transilluminator | Cole-Parmer | EW-97623-08 | Dual intensity High setting is ideal for analytical documentation. Low setting reduces photonicking or photobleaching of gel samples while doing preparative work. |
| CaCl2 • 2H2O | Sigma-Aldrich Corp | 223506 | |
| Coolpix B500 camera | Nikon Corp | S210 | |
| Coomassie brilliant blue G-250 | Bio-Rad | 1610406 | |
| Dodecyl maltoside | Sigma-Aldrich Corp | D4641 | |
| Ethylene glycol-bis(2-aminoethylether)-N,N,N′,N′-tetraacetic acid | Sigma-Aldrich Corp | E4378 | |
| Ethylenediaminetetraacetic acid tetrasodium salt dihydrate | Sigma-Aldrich Corp | E6511 | |
| Fluorescein isothiocyanate | Sigma-Aldrich Corp | F3651 | |
| KCl | JT Baker | 7447-40-7 | |
| MgCl2 • 6H2O | Sigma-Aldrich Corp | M9272 | |
| MOPS | Sigma-Aldrich Corp | M1254 | |
| NADH | Chem-Impex International Inc | 230 | |
| N-tetradecyl-N,N- dimethyl-3-ammonium-1-propanesulfonate | Sigma-Aldrich Corp | D0431 | |
| Phosphoenolpyruvate (PEP) | Chem-Impex International Inc | 9711 | |
| Rabbit muscle lactate dehydrogenase | Roche | 10003557103 | |
| Rabbit muscle pyruvate kinase | Sigma-Aldrich Corp | P1506 | |
| Sodium deoxycholate | Sigma-Aldrich Corp | D6750 | |
| Sodium dodecyl sulfate | Bio-Rad | 1610302 | |
| Spectropolarimeter | Jasco Corp. | Jasco J1500 | |
| Sucrose | Sigma-Aldrich Corp | 84100 | |
| Trehalose | Sigma-Aldrich Corp | T0167 | Dihydrate |
| Tris(hydroxymethyl)aminomethane hydrochloride | Sigma-Aldrich Corp | 857645 | |
| Ultracentrifuge | Beckman | Optima XPN | |
| UV/VIS spectrophotometer | Agilent Technologies | 8453 | The Agilent 8453 UV-Vis Spectrophotometer uses a photodiode array for simultaneous measurement of the complete ultra-violet to visible light spectrum |
| WiseStir HS- 30E | Daihan Scientific Co. | DH.WOS01010 | Ideal for all disperging and homogenizing applications, designed for tissue grinders. |
References
- Schuberth, C., Wedlich-Söldner, R. Building a patchwork - The yeast plasma membrane as model to study lateral domain formation. Biochim Biophys Acta Mol Cell Res. 1853 (4), 767-774 (2015).
- Douglas, L. M., Konopka, J. B. Fungal membrane organization: The eisosome concept. Annu Rev Microbiol. 68 (1), 377-393 (2014).
- Borrell, J. H., Domènech, &. #. 2. 1. 0. ;., Keough, K. M. W. . Membrane protein - Lipid interactions: Physics and Chemistry in the Bilayer. , (2016).
- Moraes, I., Evans, G., Sanchez-Weatherby, J., Newstead, S., Stewart, P. D. . The next generation in membrane protein structure determination. , (2016).
- White, S. H. Biophysical dissection of membrane proteins. Nature. 459 (7245), 344-346 (2009).
- Lacapère, J. -. J. Membrane protein structure determination. Methods Mol Biol. , 459 (1002).
- Palmgren, M. G. Plant plasma membrane H+-ATPases: Powerhouses for nutrient uptake. Annu Rev Plant Physiol Plant Mol Biol. 52 (1), 817-845 (2001).
- Yatime, L., et al. P-type ATPases as drug targets: Tools for medicine and science. Biochim Biophys Acta Bioenerg. 1787 (4), 207-220 (2009).
- Drews, J. Tetrahydrobiopterin biosynthesis as an off-target of sulfa drugs. Science. 287 (5460), 1960-1964 (2000).
- Xu, Z., Meshcheryakov, V. A., Poce, G., Chng, S. -. S. MmpL3 is the flippase for mycolic acids in mycobacteria. Proc Natl Acad Sci USA. 114 (30), 7993-7998 (2017).
- Siligardi, G., Hussain, R., Patching, S. G., Phillips-Jones, M. K. Ligand- and drug-binding studies of membrane proteins revealed through circular dichroism spectroscopy. Biochim Biophys Acta Biomembr. 1838 (1 PARTA), 34-42 (2014).
- Rawlings, A. E. Membrane proteins: Always an insoluble problem. Biochem Soc Trans. 44 (3), 790-795 (2016).
- Popot, J. -. L. . Membrane proteins in aqueous solutions. , 708 (2018).
- Palmgren, M. G., Sommarin, M., Ulvskov, P., Larsson, C. Effect of detergents on the H+-ATPase activity of inside-out and right-side-out plant plasma membrane vesicles. Biochim Biophys Acta Biomembr. 1021 (2), 133-140 (1990).
- McBride, Z., Chen, D., Reick, C., Xie, J., Szymanski, D. B. Global analysis of membrane-associated protein oligomerization using protein correlation profiling. Mol Cell Proteomics. 16 (11), 1972-1989 (2017).
- Yoneda, J. S., et al. Multimeric species in equilibrium in detergent-solubilized Na, K-ATPase. Int J Biol Macromol. 89, 238-245 (2016).
- Ohlendieck, K. Extraction of membrane proteins. Protein Purif Protocols. 59, 283-294 (2004).
- Champeil, P., et al. A robust method to screen detergents for membrane protein stabilization, revisited. Anal Biochem. 511, 31-35 (2016).
- Gimpl, K., Klement, J., Keller, S. Characterising protein/detergent complexes by triple-detection size-exclusion chromatography. Biol Proced Online. 18, 4 (2016).
- le Maire, M., et al. Gel chromatography and analytical ultracentrifugation to determine the extent of detergent binding and aggregation, and Stokes radius of membrane proteins using sarcoplasmic reticulum Ca2+-ATPase as an example. Nat Protoc. 3 (11), 1782-1795 (2008).
- Du, M., et al. Progress, applications, challenges and prospects of protein purification technology. Front Bioeng Biotechnol. 10, 1-26 (2022).
- Slotboom, D. J., Duurkens, R. H., Olieman, K., Erkens, G. B. Static light scattering to characterize membrane proteins in detergent solution. Methods. 46 (2), 73-82 (2008).
- Harding, S. E. Analytical ultracentrifugation as a matrix-free probe for the study of kinase related cellular and bacterial membrane proteins and glycans. Molecules. 26, 6080 (2021).
- Chen, Y. -. C., et al. Thermal stability, storage and release of proteins with tailored fit in silica. Sci Rep. 7 (1), 46568 (2017).
- Montigny, C. C., Arnou, B., Marchal, E., Champeil, P. Use of glycerol-containing media to study the intrinsic fluorescence properties of detergent-solubilized native or expressed SERCA1a. Biochemistry. 47 (46), 12159-12174 (2008).
- Simongini, M., Puglisi, A., Genovese, F., Hochkoeppler, A. Trehalose counteracts the dissociation of tetrameric rabbit lactate dehydrogenase induced by acidic pH conditions. Arch Biochem Biophys. 740, 109584 (2023).
- Shivanna, B. D., Rowe, E. S. Preservation of the native structure and function of Ca2+-ATPase from sarcoplasmic reticulum: Solubilization and reconstitution by new short-chain phospholipid detergent 1,2-diheptanoyl-sn-phosphatidylcholine. Biochem J. 325 (2), 533-542 (1997).
- Sampedro, J. G., Guerra, G., Pardo, J. P., Uribe, S. Trehalose-mediated protection of the plasma membrane H+-ATPase from Kluyveromyces lactis during freeze-drying and rehydration. Cryobiology. 37 (2), 131-138 (1998).
- Sampedro, J. G., Cortés, P., Muñoz-Clares, R. A., Fernández, A., Uribe, S. Thermal inactivation of the plasma membrane H+-ATPase from Kluyveromyces lactis. Protection by Trehalose. Biochim Biophys Acta Proteins Proteomics. 1544 (1-2), 64-73 (2001).
- Sampedro, J. G., et al. Fluorescence quenching by nucleotides of the plasma membrane H+-ATPase from Kluyveromyces lactis. Biochemistry. 46 (18), 5616-5622 (2007).
- Ruiz-Granados, Y. G., De La Cruz-Torres, V., Sampedro, J. G. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Zhao, P., Zhao, C., Chen, D., Yun, C., Li, H., Bai, L. Structure and activation mechanism of the hexameric plasma membrane H+-ATPase. Nat Commun. 12 (1), 6439 (2021).
- Auer, M., Scarborough, G. A., Kühlbrandt, W. Surface crystallization of the plasma membrane H+-ATPase on a carbon support film for electron crystallography. J Mol Biol. 287 (5), 961-968 (1999).
- Hennessey, J. P., Scarborough, G. A. Secondary structure of the Neurospora CRASSA plasma membrane H+-ATPase as estimated by circular dichroism. J Biol Chem. 263 (7), 3123-3130 (1988).
- Scarborough, G. A. Crystallization, structure and dynamics of the proton-translocating P-type ATPase. J Exp Biol. 203 (Pt 1), 147-154 (2000).
- Sumida, K. H., et al. Improving protein expression, stability, and function with proteinMPNN. J Am Chem Soc. 146 (3), 2054-2061 (2024).
- Eleutherio, E., Panek, A., De Mesquita, J. F., Trevisol, E., Magalhães, R. Revisiting yeast trehalose metabolism. Curr Genet. 61 (3), 263-274 (2015).
- Attfield, P. V. Trehalose accumulates in Saccharomyces cerevisiae during exposure to agents that induce heat shock response. FEBS Lett. 225 (1-2), 259-263 (1987).
- Marunde, M. R., et al. Improved tolerance to salt and water stress in Drosophila melanogaster cells conferred by late embryogenesis abundant protein. J Insect Physiol. 59 (4), 377-386 (2013).
- Crowe, J. H. Anhydrobiosis: An unsolved problem with applications in human welfare. Subcell Biochem. 71, 263-280 (2015).
- Crowe, J. H., Crowe, L. M., Chapman, D. Preservation of membranes in anhydrobiotic organisms: The role of trehalose. Science. 223 (4637), 701-703 (1984).
- Sampedro, J. G., Rivera-Moran, M. A., Uribe-Carvajal, S. Kramers' theory and the dependence of enzyme dynamics on trehalose-mediated viscosity. Catalysts. 10 (6), 1-19 (2020).
- Sampedro, J. G., Uribe, S. Trehalose-enzyme interactions result in structure stabilization and activity inhibition. The role of viscosity. Mol Cell Biochem. 256 - 257 (1-2), 319-327 (2004).
- Crowe, J. H. Trehalose as a "chemical chaperone": Fact and fantasy. Adv Exp Med Biol. 594, 143-158 (2007).
- Crowe, J. H., Carpenter, J. F., Crowe, L. M., Anchordoguy, T. J. Are freezing and dehydration similar stress vectors? A comparison of modes of interaction of stabilizing solutes with biomolecules. Cryobiology. 27 (3), 219-231 (1990).
- Rivera-Morán, M. A., Sampedro, J. G. Isolation of the sarcoplasmic reticulum Ca2+-ATPase from rabbit fast-twitch muscle. Methods Protoc. 6 (5), 102 (2023).
- Páez-Pérez, E. D., De La Cruz-Torres, V., Sampedro, J. G. Nucleotide binding in an engineered recombinant Ca2+-ATPase N-domain. Biochemistry. 55 (49), 6751-6765 (2016).
- Ramírez-Alonso, J. I., Sampedro, J. G. Effect of cations on ATP binding to the N-domain of Na+, K+-ATPase. J Fluoresc. , (2024).
- De la Cruz-Torres, V., Cataño, V., Olivo-Rodríguez, M., Sampedro, J. G. ANS Interacts with the Ca2+-ATPase nucleotide binding site. J Fluoresc. 30 (3), 483-496 (2020).
- Sampedro, J. G., Nájera, H., Uribe-Carvajal, S., Ruiz-Granados, Y. G. Mapping the ATP binding site in the plasma membrane H+-ATPase from Kluyveromyces lactis. J Fluoresc. 24 (6), 1849-1859 (2014).
- Kiani, A. K., et al. Ethical considerations regarding animal experimentation. J Prev Med Hyg. 63 (2 Suppl 3), E255-E266 (2022).
- Ruiz-Granados, Y., De La Cruz-Torres, V., Sampedro, J. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Wang, S., et al. Structural basis for sarcolipin's regulation of muscle thermogenesis by the sarcoplasmic reticulum Ca2+-ATPase. Sci Adv. 7 (48), eabi7154 (2021).
- Champeil, P., et al. ATP regulation of sarcoplasmic reticulum Ca2+-ATPase. Metal-free ATP and 8-bromo-ATP bind with high affinity to the catalytic site of phosphorylated ATPase and accelerate dephosphorylation. J Biol Chem. 263 (25), 12288-12294 (1988).
- Møller, J. V., Olesen, C. Preparation of Ca(2+)-ATPase1a enzyme from rabbit sarcoplasmic reticulum. Methods Mol Biol. 1377, 11-17 (2016).
- Wuytack, F., De Schutter, G., Casteels, R. Partial purification of (Ca2+ + Mg2+)-dependent ATPase from pig smooth muscle and reconstitution of an ATP-dependent Ca2+-transport system. Biochem J. 198 (2), 265-271 (1981).
- Mandal, A., et al. Solubilization, purification and reconstitution of Ca2+-ATPase from bovine pulmonary artery smooth muscle microsomes by different detergents: Preservation of native structure and function of the enzyme by DHPC. Biochim Biophys Acta Gen Subj. 1760 (1), 20-31 (2006).
- Inesi, G., Ma, H., Hua, S., Toyoshima, C. Characterization of Ca2+ ATPase residues involved in substrate and cation binding. Ann NY Acad Sci. 986 (1), 63-71 (2003).
- Van Winkle, W. B., Pitts, B. J., Entman, M. L. Rapid purification of canine cardiac sarcoplasmic reticulum Ca2+-ATPase. J Biol Chem. 253 (24), 8671-8673 (1978).
- Ruiz-Granados, Y. G., De La Cruz-Torres, V., Sampedro, J. G. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Teramoto, N., Sachinvala, N. D., Shibata, M. Trehalose and trehalose-based polymers for environmentally benign, biocompatible, and bioactive materials. Molecules. 13 (8), 1773-1816 (2008).
- Dao, T. T., et al. Demethoxycurcumin is a potent inhibitor of P-type ATPases from diverse kingdoms of life. PLOS ONE. 11 (9), e0163260 (2016).
- Bleeker, N. P., Cornea, R. L., Thomas, D. D., Xing, C. A novel SERCA inhibitor demonstrates synergy with classic SERCA inhibitors and targets multidrug-resistant AML. Mol Pharmaceutics. 10 (11), 4358-4366 (2013).
- Michelangeli, F., East, J. M. A diversity of SERCA Ca2+ pump inhibitors. Biochem Soc Trans. 39 (3), 789-797 (2011).
- Tadini-Buoninsegni, F., Smeazzetto, S., Gualdani, R., Moncelli, M. R. Drug interactions with the Ca2+-ATPase from Sarco(Endo)Plasmic Reticulum (SERCA). Front Mol Biosci. 5, 36 (2018).
- Clausen, J. D., et al. Elucidation of antimicrobial activity and mechanism of action by N-substituted carbazole derivatives. Bioorg Med Chem Lett. 27 (19), 4564-4570 (2017).
- Leary, S., et al. . AVMA guidelines for the euthanasia of animals: 2020 Edition, Members of the Panel on Euthanasia AVMA Staff Consultants. , (2020).
- de Aluja, A. S. Laboratory animals and official Mexican norms (NOM-062-ZOO-1999). Gac Med Mex. 138 (3), 295-298 (2002).
- Champeil, P., Buschlen-Boucly, S., Bastide, F., Gary-Bobo, C. Sarcoplasmic reticulum ATPase. Spin labeling detection of ligand-induced changes in the relative reactivities of certain sulfhydryl groups. J Biol Chem. 253 (4), 1179-1186 (1978).
- Skalec, A., Janeczek, M., Czerski, A. Anatomy and histology of the rabbit common calcanean tendon. J Vet Med Series C: Anat Histol Embryol. 48 (5), 466-475 (2019).
- Janes, L. E., Mioton, L. M., Fracol, M. E., Ko, J. H. An in vivo comparison: Novel mesh suture versus traditional suture-based repair in a rabbit tendon model. J Hand Surg Glob Online. 4 (1), 32-39 (2022).
- Bollag, D. M., Rozycki, M. D., Edelstein, S. J. . Protein Methods. , (1996).
- Gagné, F. Tissue preparation and subcellular fractionation techniques. Biochem Ecotoxicol. , 21-31 (2014).
- Lowry, O. H., Rosebrough, N. J., Farr, A. L., Randall, R. J. Protein measurement with the Folin phenol reagent. J Biol Chem. 193 (1), 265-275 (1951).
- Autry, J. M., Rubin, J. E., Svensson, B., Li, J. L., Thomas, D. D. Nucleotide activation of the Ca-ATPase. J Biol Chem. 287 (46), 39070-39082 (2012).
- Winters, D. L., Autry, J. M., Svensson, B., Thomas, D. D. Interdomain fluorescence resonance energy transfer in SERCA probed by cyan-fluorescent protein fused to the actuator domain. Biochemistry. 47 (14), 4246-4256 (2008).
- Miles, A. J., Wallace, B. A. Circular dichroism spectroscopy of membrane proteins. Chem Soc Rev. 14 (45), 1130-1135 (2016).
- Scofano, H. M., Vieyra, A., de Meis, L. Substrate regulation of the sarcoplasmic reticulum ATPase. Transient kinetic studies. J Biol Chem. 254 (20), 10227-10231 (1979).
- Yamamoto, T., Tonomura, Y. Reaction mechanism of the Ca++-dependent ATPase of sarcoplasmic reticulum from skeletal muscle: I. Kinetic studies. J Biochem. 62 (5), 558-575 (1967).
- Murphy, A. J. Affinity labeling of the active site of the Ca2+-ATPase of sarcoplasmic reticulum. Biochim Biophys Acta - Biomembr. 946 (1), 57-65 (1988).
- Csermely, P., Katopis, C., Wallace, B. A., Martonosi, A. The E1→E2 transition of Ca2+-transporting ATPase in sarcoplasmic reticulum occurs without major changes in secondary structure. A circular-dichroism study. Biochem J. 241 (3), 663-669 (1987).
- Abràmoff, M. D., Magalhães, P. J., Ram, S. J. Image processing with ImageJ. Biophotonics Int. 11 (7), 36-42 (2004).
- Scarborough, G. A. The plasma membrane proton-translocating ATPase. Cell Mol Life Sci. 57 (6), 871-883 (2000).
- Llopis, J., McCaffery, J. M., Miyawaki, A., Farquhar, M. G., Tsien, R. Y. Measurement of cytosolic, mitochondrial, and Golgi pH in single living cells with green fluorescent proteins. Proc Natl Acad Sci. 95 (12), 6803-6808 (1998).
- Kanai, R., Vilsen, B., Cornelius, F., Toyoshima, C. Crystal structures of Na+, K+-ATPase reveal the mechanism that converts the K+-bound form to Na+-bound form and opens and closes the cytoplasmic gate. FEBS Lett. 597 (15), 1957-1976 (2023).
- Palmgren, M. P-type ATPases: Many more enigmas left to solve. J Biol Chem. 299 (11), 105352 (2023).
- Frauenfelder, H. Ask not what physics can do for biology-Ask what biology can do for physics. Phys Biol. 11 (5), 053004 (2014).
- Frauenfelder, H. The physics of proteins. Annu Rev Biophys Biomol Struct. 36, (2010).
- Narhi, L. O. . Biophysics for Therapeutic Protein Development. , (2013).
- East, J. M. Purification of a membrane protein (Ca2+/Mg2+-ATPase) and its reconstitution into lipid vesicles. Methods Mol Biol. 27, 87-94 (1994).
- Lee, E. -. H., et al. Enhancement of enzyme activity and stability by poly(γ-glutamic acid). Polymer J. 42 (10), 818-822 (2010).
- Norrild, R. K., et al. Increasing protein stability by inferring substitution effects from high-throughput experiments. Cell Rep Methods. 2 (11), 100333 (2022).
- Song, Z., Zhang, Q., Wu, W., Pu, Z., Yu, H. Rational design of enzyme activity and enantioselectivity. Front Bioeng Biotechnol. 11, 1-14 (2023).
- Jidenko, M., Lenoir, G., Fuentes, J. M., le Maire, M., Jaxel, C. Expression in yeast and purification of a membrane protein, SERCA1a, using a biotinylated acceptor domain. Protein Expr Purif. 48 (1), 32-42 (2006).
- Hua, S., Ma, H., Lewis, D., Inesi, G., Toyoshima, C. Functional role of "N" (nucleotide) and "P" (phosphorylation) domain interactions in the sarcoplasmic reticulum (SERCA) ATPase. Biochemistry. 41 (7), 2264-2272 (2002).
- Pang, Y. -. H., Chen, J. -. W. Anisodamine causes the changes of structure and function in the transmembrane domain of the Ca2+-ATPase from sarcoplasmic reticulum. Biosci Biotechnol Biochem. 68 (1), 126-131 (2004).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved