É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Purificação do retículo sarco-endoplasmático Ca2+-ATPase do músculo de coelho
Neste Artigo
Resumo
Este protocolo descreve um método de purificação SERCA aprimorado, que inclui o dissacarídeo trealose na etapa final de centrifugação. Este carboidrato estabiliza as proteínas em condições adversas. O SERCA purificado foi cataliticamente ativo e apresentou alta pureza, tornando-o adequado para estudos estruturais e funcionais.
Resumo
Algumas ATPases do tipo P, como a sarco/retículo endoplasmático Ca2+-ATPase (SERCA), são proteínas de membrana inerentemente lábeis que requerem condições físico-químicas específicas durante a purificação para obtê-las com alta pureza e qualidade estrutural e em uma forma cataliticamente ativa. O dissacarídeo trealose é um soluto compatível que é sintetizado e acumulado em altas concentrações no citoplasma da levedura para estabilizar as membranas e proteínas. O uso da trealose como aditivo no protocolo de purificação da membrana plasmática H+-ATPase resulta em uma preparação de alta qualidade, cuja estrutura hexamérica é demonstrada por métodos analíticos bioquímicos. A trealose pode, portanto, ser usada como um aditivo estabilizador para a purificação de proteínas de membrana (P-ATPases). Este protocolo descreve a modificação do protocolo clássico para purificação do SERCA submetendo o SERCA à centrifugação em um gradiente de concentração de trealose. A inclusão deste carboidrato levou à purificação do SERCA em uma forma cataliticamente ativa com alta pureza e, mais importante, em uma forma estável. A caracterização bioquímica parcial do SERCA purificado (SDS-PAGE, cinética enzimática, marcação FITC, espectroscopia de dicroísmo circular) mostrou que a enzima é adequada para estudos funcionais e estruturais. Sugere-se o uso de trealose no protocolo de purificação de ATPases do tipo P e outras proteínas lábeis de membrana (e citosólicas).
Introdução
As proteínas/enzimas de membrana são componentes biológicos essenciais das células, pois desempenham papéis críticos em vários processos 1,2,3. Algumas das funções podem incluir o transporte de íons e moléculas para dentro e para fora da célula/compartimentos internos (ativo/passivo), reconhecimento célula-célula, ligação intercelular, ancoragem/fixação e detecção do ambiente externo por meio da integração com a maquinaria de transdução de sinal sob condições físicas e químicas normais e adversas (alto teor de sal, baixa água, alta temperatura, resistência a medicamentos, etc.)3 Portanto, a determinação da estrutura tridimensional (3D) de proteínas e/ou enzimas de membrana tornou-se de grande importância tanto para a pesquisa básica quanto para a aplicada 4,7,6. É importante ressaltar que as proteínas/enzimas de membrana têm sido amplamente utilizadas como alvos para a descoberta de medicamentos (sejam naturais ou por design)7,8,9. Ou seja, as proteínas de membrana têm uma importância inerente à saúde 9,10,11.
O caráter hidrofóbico das proteínas/enzimas de membrana é a propriedade físico-química tecnicamente mais desafiadora para o laboratório experimental 12,13,14, ainda mais quando se trabalha com proteínas de membrana integrais oligoméricas e/ou altamente lábeis 15,16. O isolamento de quantidades adequadas da proteína/enzima de membrana com a mais alta qualidade possível para ensaios experimentais funcionais e estudos estruturais é altamente desejável17. As proteínas de membrana, por serem inerentemente hidrofóbicas, são muito difíceis de purificar, e a escolha do detergente costuma ser uma das questões mais importantes a serem consideradas 17,18,19,20. Nesse sentido, os métodos laboratoriais usados para isolar proteínas de membrana normalmente causam algum grau de dano ao arranjo estrutural 3D da proteína21. Alguns desses métodos incluem o uso de (a) sonicação (ondas ultrassônicas de alta frequência/energia), (b) detergentes solubilizadores de proteínas (agressivos, médios ou suaves)22, (c) pressões relativamente altas em cromatografia em coluna e ultracentrifugação de alta velocidade23, (d) moléculas precipitantes, (e) enzimas digestivas e outros21. Todos esses processos podem contribuir ou ser a principal causa da desestabilização de proteínas durante a purificação21. A esse respeito, alguns protocolos parecem funcionar relativamente bem para uma determinada proteína de membrana. No entanto, a otimização é sempre bem-vinda ao usar novos ensaios modernos ou métodos que requerem uma maior qualidade de preparação de proteínas de membrana para obter resultados satisfatórios24. As etapas de otimização podem incluir, mas não estão limitadas a, melhorar o projeto da construção, encontrar condições ideais para a expressão da proteína da membrana, estabelecer melhores condições de manuseio (ou seja, pH, temperatura, etc.), encontrar o melhor detergente compatível, ajustar as etapas de purificação, como tempo de sonicação, velocidade de centrifugação, reformular soluções tampão adicionando agentes estabilizantes, etc.25,26,27. Portanto, qualquer alteração na metodologia de purificação que leve a um aumento na qualidade (pureza) e na atividade da proteína/enzima de membrana purificada é importante.
Na família ATPase do tipo P, a membrana plasmática da levedura H+-ATPase parece ser um dos membros mais lábeis28,29. A H+-ATPase perde sua atividade hidrolisante de ATP após desidratação (liofilização), choque térmico, etc.28,29. O uso da trealose como estabilizador proteico foi testado no isolamento da membrana plasmática H+-ATPase da levedura K. lactis30,31; a preparação de H+-ATPase obtida foi de alta pureza e cataliticamente ativa. É importante ressaltar que isso permitiu que o estado oligomérico da enzima fosse resolvido por métodos bioquímicos, revelando que era um hexâmero e posteriormente confirmado por microscopia crioeletrônica 31,32,33,34,35. Portanto, parece provável que o delicado arranjo tridimensional (3D) das proteínas de membrana possa ser perdido em condições relativamente adversas durante a purificação36. A cinética de inativação bifásica é observada para H+-ATPase durante a inativação mediada por calor29. Nas células de levedura, como em muitos outros organismos, o dissacarídeo trealose se acumula em altas concentrações sob condições de estresse ambiental 37,38,39. A trealose mantém a integridade da membrana e a função de transporte, estabilizando proteínas (tanto incorporadas na membrana quanto citosólicas) e membranas celulares40,41. O mecanismo estabilizador da trealose tem sido extensivamente estudado por vários grupos e por nosso laboratório 42,43,44,45. Experimentos com a H+-ATPase e outras enzimas mostraram que a trealose é o estabilizador proteico mais eficaz entre os mono e dissacarídeos28,29. Isso levou à sua inclusão no protocolo de purificação da H+-ATPase30. Recentemente, a trealose também tem sido usada na purificação do retículo sarcoplasmático Ca2+-ATPase (SERCA) do músculo de contração rápida de coelhos com bons resultados na pureza e atividade proteica46. Portanto, a trealose parece ser um aditivo bom e apropriado para a purificação de ATPases do tipo P e provavelmente outras proteínas de membrana e citosólicas.
Para as P-ATPases, a existência de domínios citoplasmáticos estruturais é uma vantagem experimental, especialmente para estudos de interação substrato/ligante47; A ligação ao ATP foi estudada em domínios N recombinantes altamente puros 47,48,49, eliminando assim as considerações técnicas para a purificação de enzimas de membrana inteiras 47,48,50, entre outras 51. Infelizmente, alguns estudos funcionais (conversão catalítica / energética) e estruturais (arranjo de subunidades e interação com outras proteínas) ainda requerem toda a P-ATPase 52,53. Nesse sentido, a purificação do SERCA tem sido alcançada por vários grupos de pesquisa 54,55,56,57,58. No entanto, melhorias ainda podem ser implementadas46, por exemplo, aumentando a integridade da ATPase purificada59, evitando a desnaturação / ruptura de proteínas de complexos proteicos25, aumentando a solubilização sem desnaturação da proteína de membrana (ou seja, evitando a formação de agregados macromoleculares) 60, levando a uma melhor compatibilidade com ensaios e outros métodos analíticos a jusante61 Além disso, à medida que novas estratégias experimentais, aditivos, inibidores enzimáticos, etc. aparecem na literatura científica 61,62,63,64,65,66, eles às vezes precisam ser testados com toda a P-ATPase. Este trabalho descreve o protocolo para a purificação do SERCA e o uso da trealose como aditivo para estabilizar a estrutura proteica e a atividade da ATPase; Ou seja, além de aumentar a qualidade da enzima (estrutural), a trealose ajuda a prevenir a perda de estrutura e atividade enzimática durante o isolamento e armazenamento da enzima, o que ajuda a economizar material biológico e, assim, reduzir o número de purificações enzimáticas.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Todos os procedimentos em animais foram realizados de acordo com as diretrizes internacionais e locais (NORMA Oficial Mexicana NOM-062-ZOO-1999) para o manejo de animais em laboratórios experimentais 51,67,68. O tecido muscular foi obtido de Oryctolagus cuniculus do tipo selvagem de uma unidade local de manejo de animais (INE/CITES/DGVS-ZOO-E0055-SLP-98)46. Um veterinário com experiência em manejo de animais de laboratório realizou a dissecção e processamento muscular inicial. Os detalhes dos reagentes e dos equipamentos utilizados neste estudo estão listados na Tabela de Materiais.
1. Isolamento do retículo sarcoplasmático do músculo de coelho de contração rápida (Timing, 7 - 9 h)
NOTA: Para detalhes sobre o procedimento, consulte Champeil et al.69.
- Obtenha tecido muscular de contração rápida (~ 70 g de ambas as patas traseiras por animal) de Oryctolagus cuniculus do tipo selvagem.
NOTA: É necessária a identificação visual prévia dos músculos a serem dissecados70,71. - Macerar o músculo de contração rápida no liquidificador, suspendendo-o em três volumes de 100 mM de KCl, com 1 min ligado e 1 min em repouso gelado, repetir três vezes72.
- Remover os restos de tecido por centrifugação (1500 × g) a 4 °C durante 5 min; Uma segunda centrifugação (4200 × g) é realizada, se necessário.
- Colete o sobrenadante e homogeneize-o usando um triturador de tecidos (10 golpes em baixa velocidade, ou seja, 500-1.500 rpm)72,73. Centrifugar o homogeneizado (10.000 × g) a 4 °C durante 15 min.
NOTA: Deve-se ter cuidado ao homogeneizar, pois o uso de uma velocidade relativamente alta pode resultar em danos ao sistema e ao operador. - Centrifugar o sobrenadante (contendo o retículo sarcoplasmático) (33.000 × g) a 4 °C durante 120 min.
- Suspender os pellets em 40 ml de sacarose 0,5 M e centrifugar (12.000 × g) a 4 °C durante 15 min. Diluir o sobrenadante (0,6 M de KCl e 0,15 M de sacarose) e centrifugar a suspensão (34.000 × g) a 4 °C durante 165 min.
- Suspenda o pellet contendo as folhas de vesículas do retículo sarcoplasmático (SRVs) em 0,3 M de sacarose, 0,1 M de KCl e 5 mM de Tris-HCl, pH 7,0. Determine a concentração de proteína usando o ensaio de Lowry74 e albumina sérica humana como padrão de proteína.
- Alíquota dos SRVs (30-32 mg/mL) em volume de 1 mL. Armazene as amostras de SRVs a -72 °C para processamento posterior.
NOTA: O rendimento proteico é de 100-110 mg SRVs por animal.
2. Purificação SERCA (Tempo, 18 - 20 h)
NOTA: Para detalhes sobre o procedimento, consulte Rivera-Morán et al.46.
- Dilua a suspensão de SRVs para 2 mg de proteína / mL usando 75 mM gelados de Tris-HCl, pH 7,2, 0,6 M de KCl, 6 mM de EDTA, 1 mM de EGTA e 0,1% (p / v) desoxicolato (DOC). Incube a suspensão SRVs no gelo por 10 min e agitação suave. Centrifugue a suspensão SRVs (100.000 × g) a 4 °C por 60 min.
NOTA: DOC deve ser adicionado gota a gota. - Suspenda os pellets em <3 mL de 25 mM de Tris-HCl, pH 7,5, 0,3 M de KCl, glicerol a 45% (v/v) e 2 mM de EDTA. Homogeneizar a suspensão e ajustar para um volume final de 10 ml. Determine a concentração de proteína e, em seguida, ajuste-a para 6,5 mg / mL. Adicionar azolectina a 5 mg/ml e Zwittergent 0,85 (p/p) 3-14.
NOTA: Azolectina e Zwittergent 3-14 devem ser adicionados gota a gota. - Homogeneizar a suspensão e centrifugar (100.000 × g) a 4 °C durante 60 min. Recolha o sobrenadante e dilua 1:2 com 2 mM de EGTA (pH 7,2).
- Despeje a suspensão suavemente em um gradiente de concentração de trealose descontínuo (45, 40, 35 e 30% p / v) em 10 mM de Tris-HCl, pH 7,0, 1 mM de EDTA, 0,1% de desoxicolato e 1 mg / mL de azolectina. Centrifugar (100.000 × g) a 4 °C durante 14 h. Recolha o pellet transparente e ligeiramente amarelado formado no fundo dos tubos.
NOTA: Tanto a formação do gradiente de trealose como a adição da suspensão da amostra devem ser realizadas utilizando técnicas adequadas. Recomenda-se centrifugação durante a noite. - Suspenda suavemente o pellet em um pequeno volume (<1,5 mL) de 25 mM de Tris-HCl, pH 7,5, 0,3 de M KCl, 45% de glicerol e 2 mM de EDTA. Determine a concentração de proteína. Ajustar a concentração de proteínas por diluição para 2 mg/ml. Pegue alíquotas (~ 50 μL) da suspensão e armazene o SERCA purificado a -72 ° C até seu uso.
- Execute SDS-PAGE do SERCA72 purificado e de amostras de proteínas colhidas em diferentes etapas da purificação. Manchar o gel com Coomassie Blue72.
NOTA: O uso de trealose 0,6 M em vez de glicerol no tampão de suspensão aumenta a estabilidade da enzima durante os ciclos de armazenamento e congelamento e descongelamento28,29.
3. Ensaio para atividade de ATPase (tempo, 1 - 2 h)
- Ensaio acoplado a enzimas: ATPase-Piruvato quinase-lactato desidrogenase
- Preparar a mistura de reacção (1 ml): 50 mM MOPS (pH 7,0), 1 mM EGTA, 80 mM KCl, 5 mM MgCl2, 3 mM CaCl2, 5 mM fosfoenolpiruvato, 250 μM NADH. Inclua concentrações variáveis de ATP (0,001 a 0,25 mM).
- Homogeneizar cuidadosamente o ensaio de reação por vórtice. Incube o ensaio de reação a 37 ◦C por 10 min. Adicione 0,9 U L-lactato desidrogenase (LDH) e 1,5 U piruvato quinase (PK).
- Inicie a reação ATPase pela adição de 10 μg de SERCA.
- Determine a formação de NADH. Acompanhe a mudança na intensidade da absorbância a cada segundo durante 600 s a um comprimento de onda (λ) de 340 nm ao longo do tempo usando um espectrofotômetro com um suporte de célula termostatizado.
NOTA: O intervalo de tempo para a medição da absorbância pode variar, mas tente registrar o maior número possível de pontos de dados para desenhar a linha reta inicial da reação ATPase.
- Determine a taxa de hidrólise de ATP
NOTA: Para detalhes sobre o procedimento, consulte Rivera-Morán et al.46.- Calcule o valor da inclinação (Δabs/min) da porção linear em cada curva formada. Use o coeficiente de extinção molar (ε) de NADH (λ = 6.220 M−1·cm−1) para determinar a atividade da ATPase (μmoles ATP hidrolisado/min.mg prot.)
- Determinar os parâmetros cinéticos Km e Vmáx
- Plote os dados de velocidade versus concentração de ATP usando software de análise de dados e gráficos.
- Ajuste os dados por regressão não linear à Equação de Michaelis-Menten (Eq. 1)46:
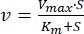
onde v é a velocidade, Vmax é a velocidade máxima, [S] é a concentração de ATP e Km é a constante de Michaelis-Menten. - Determine Km e Vmax por iteração.
NOTA: A inclusão de DOC pode aumentar a taxa de resposta da ATPase; recomenda-se testar diferentes concentrações de DOC para encontrar o ideal.
4. Ensaio de marcação de SERCA com isotiocianato de fluoresceína (FITC) (tempo, 30 - 45 min, apenas para marcação FITC)
NOTA: Para obter detalhes sobre o procedimento, consulte 54,75,76.
- Suspenda o SERCA (20 μg) em 50 μL (volume final) de tampão de marcação (100 mM de KCl, 5 mM de MgCl2 e 30 mM de Tris-HCl, pH 8,9) contendo 1 mM de FITC. Misture por vórtice.
- Incube as amostras em momentos diferentes (15 min, 10 min, 7 min, 5 min e 2 min) no escuro e em temperatura ambiente.
- Pare a reação de marcação adicionando 1 volume de tampão de parada gelado (480 mM de sacarose e 48 mM de MOPS, pH 7,0), contendo ATP (5 mM). Incube no gelo por 5 min no escuro.
- Sujeito o SERCA rotulado com FITC à SDS-PAGE72. Exponha os géis transparentes à luz UV (λ = 302 nm). Foto: documente o resultado.
- Manchar o gel com azul Coomassie. Foto: documente o resultado.
NOTA: Realizar a rotulagem FITC em uma sala escura pode melhorar a qualidade do sinal de fluorescência.
5. Espectros de dicroísmo circular (CD) (Tempo, 30 min)
NOTA: Para obter detalhes sobre o procedimento, consulte 11,77.
- Suspender SERCA (3 μM) em 400 μL de tampão fosfato 10 mM (pH 7,0) a 25 °C.
- Carregue a amostra em uma célula de 0,1 cm de comprimento de caminho. Defina a faixa λ dos espectros de UV distante entre 190-260 nm. Defina a resolução interna e a largura de banda para 1 nm.
- Registar o sinal de CD a 50 nm/min a 25 °C.
Access restricted. Please log in or start a trial to view this content.
Resultados
SDS-PAGE de SERCA em diferentes estágios de purificação (Figura 1). O gel corado com azul de Coomassie mostra o enriquecimento da banda da proteína SERCA (peso molecular aparente >100 kDa) à medida que o protocolo de purificação avança. A banda de proteína correspondente ao SERCA mostra uma pureza de >90% após centrifugação em um gradiente de concentração de trealose. Gráfico da taxa de hidrólise de ATP versus concentração de ATP (...
Access restricted. Please log in or start a trial to view this content.
Discussão
A maioria das moléculas e íons não pode atravessar livremente as membranas celulares, por exemplo, o próton (H+) requer um transportador de membrana na membrana plasmática de uma variedade de organismos e organelas, como as mitocôndrias83,84. As membranas celulares são seletivas e as moléculas e íons que atravessam as membranas celulares são diversos, portanto, vários tipos de proteínas de membrana podem se...
Access restricted. Please log in or start a trial to view this content.
Divulgações
O autor declara que não tem interesses financeiros concorrentes.
Agradecimentos
O autor agradece a ajuda de Edmundo Mata-Morales na edição do vídeo, VM, Valentín de la Cruz-Torres na purificação dos SRVs, Miguel A. Rivera-Moran na purificação e análise do SERCA e Juan C. Gonzalez-Castro, Franco E. Juarez, Alejandra Nevarez, Nicolas Rocha-Vizuet e Jocelin I. Ramírez-Alonso na produção do vídeo. Nenhum fundo, subsídio ou outro apoio foi recebido para conduzir este estudo.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| ATP | Sigma-Aldrich Corp | A2383 | |
| Azolectin from soybean | Sigma-Aldrich Corp | 44924 | |
| Benchtop UV transilluminator | Cole-Parmer | EW-97623-08 | Dual intensity High setting is ideal for analytical documentation. Low setting reduces photonicking or photobleaching of gel samples while doing preparative work. |
| CaCl2 • 2H2O | Sigma-Aldrich Corp | 223506 | |
| Coolpix B500 camera | Nikon Corp | S210 | |
| Coomassie brilliant blue G-250 | Bio-Rad | 1610406 | |
| Dodecyl maltoside | Sigma-Aldrich Corp | D4641 | |
| Ethylene glycol-bis(2-aminoethylether)-N,N,N′,N′-tetraacetic acid | Sigma-Aldrich Corp | E4378 | |
| Ethylenediaminetetraacetic acid tetrasodium salt dihydrate | Sigma-Aldrich Corp | E6511 | |
| Fluorescein isothiocyanate | Sigma-Aldrich Corp | F3651 | |
| KCl | JT Baker | 7447-40-7 | |
| MgCl2 • 6H2O | Sigma-Aldrich Corp | M9272 | |
| MOPS | Sigma-Aldrich Corp | M1254 | |
| NADH | Chem-Impex International Inc | 230 | |
| N-tetradecyl-N,N- dimethyl-3-ammonium-1-propanesulfonate | Sigma-Aldrich Corp | D0431 | |
| Phosphoenolpyruvate (PEP) | Chem-Impex International Inc | 9711 | |
| Rabbit muscle lactate dehydrogenase | Roche | 10003557103 | |
| Rabbit muscle pyruvate kinase | Sigma-Aldrich Corp | P1506 | |
| Sodium deoxycholate | Sigma-Aldrich Corp | D6750 | |
| Sodium dodecyl sulfate | Bio-Rad | 1610302 | |
| Spectropolarimeter | Jasco Corp. | Jasco J1500 | |
| Sucrose | Sigma-Aldrich Corp | 84100 | |
| Trehalose | Sigma-Aldrich Corp | T0167 | Dihydrate |
| Tris(hydroxymethyl)aminomethane hydrochloride | Sigma-Aldrich Corp | 857645 | |
| Ultracentrifuge | Beckman | Optima XPN | |
| UV/VIS spectrophotometer | Agilent Technologies | 8453 | The Agilent 8453 UV-Vis Spectrophotometer uses a photodiode array for simultaneous measurement of the complete ultra-violet to visible light spectrum |
| WiseStir HS- 30E | Daihan Scientific Co. | DH.WOS01010 | Ideal for all disperging and homogenizing applications, designed for tissue grinders. |
Referências
- Schuberth, C., Wedlich-Söldner, R. Building a patchwork - The yeast plasma membrane as model to study lateral domain formation. Biochim Biophys Acta Mol Cell Res. 1853 (4), 767-774 (2015).
- Douglas, L. M., Konopka, J. B. Fungal membrane organization: The eisosome concept. Annu Rev Microbiol. 68 (1), 377-393 (2014).
- Borrell, J. H., Domènech, Ò, Keough, K. M. W. Membrane protein - Lipid interactions: Physics and Chemistry in the Bilayer. , Springer International Publishing. (2016).
- Moraes, I., Evans, G., Sanchez-Weatherby, J., Newstead, S., Stewart, P. D. The next generation in membrane protein structure determination. , Springer International Publishing. (2016).
- White, S. H. Biophysical dissection of membrane proteins. Nature. 459 (7245), 344-346 (2009).
- Lacapère, J. -J. Membrane protein structure determination. Methods Mol Biol. , Humana Press. 459(1002).
- Palmgren, M. G. Plant plasma membrane H+-ATPases: Powerhouses for nutrient uptake. Annu Rev Plant Physiol Plant Mol Biol. 52 (1), 817-845 (2001).
- Yatime, L., et al. P-type ATPases as drug targets: Tools for medicine and science. Biochim Biophys Acta Bioenerg. 1787 (4), 207-220 (2009).
- Drews, J. Tetrahydrobiopterin biosynthesis as an off-target of sulfa drugs. Science. 287 (5460), 1960-1964 (2000).
- Xu, Z., Meshcheryakov, V. A., Poce, G., Chng, S. -S. MmpL3 is the flippase for mycolic acids in mycobacteria. Proc Natl Acad Sci USA. 114 (30), 7993-7998 (2017).
- Siligardi, G., Hussain, R., Patching, S. G., Phillips-Jones, M. K. Ligand- and drug-binding studies of membrane proteins revealed through circular dichroism spectroscopy. Biochim Biophys Acta Biomembr. 1838 (1 PARTA), 34-42 (2014).
- Rawlings, A. E. Membrane proteins: Always an insoluble problem. Biochem Soc Trans. 44 (3), 790-795 (2016).
- Popot, J. -L. Membrane proteins in aqueous solutions. , Springer International Publishing. 708(2018).
- Palmgren, M. G., Sommarin, M., Ulvskov, P., Larsson, C. Effect of detergents on the H+-ATPase activity of inside-out and right-side-out plant plasma membrane vesicles. Biochim Biophys Acta Biomembr. 1021 (2), 133-140 (1990).
- McBride, Z., Chen, D., Reick, C., Xie, J., Szymanski, D. B. Global analysis of membrane-associated protein oligomerization using protein correlation profiling. Mol Cell Proteomics. 16 (11), 1972-1989 (2017).
- Yoneda, J. S., et al. Multimeric species in equilibrium in detergent-solubilized Na, K-ATPase. Int J Biol Macromol. 89, 238-245 (2016).
- Ohlendieck, K. Extraction of membrane proteins. Protein Purif Protocols. 59, 283-294 (2004).
- Champeil, P., et al. A robust method to screen detergents for membrane protein stabilization, revisited. Anal Biochem. 511, 31-35 (2016).
- Gimpl, K., Klement, J., Keller, S. Characterising protein/detergent complexes by triple-detection size-exclusion chromatography. Biol Proced Online. 18, 4(2016).
- le Maire, M., et al. Gel chromatography and analytical ultracentrifugation to determine the extent of detergent binding and aggregation, and Stokes radius of membrane proteins using sarcoplasmic reticulum Ca2+-ATPase as an example. Nat Protoc. 3 (11), 1782-1795 (2008).
- Du, M., et al. Progress, applications, challenges and prospects of protein purification technology. Front Bioeng Biotechnol. 10, 1-26 (2022).
- Slotboom, D. J., Duurkens, R. H., Olieman, K., Erkens, G. B. Static light scattering to characterize membrane proteins in detergent solution. Methods. 46 (2), 73-82 (2008).
- Harding, S. E. Analytical ultracentrifugation as a matrix-free probe for the study of kinase related cellular and bacterial membrane proteins and glycans. Molecules. 26, 6080(2021).
- Chen, Y. -C., et al. Thermal stability, storage and release of proteins with tailored fit in silica. Sci Rep. 7 (1), 46568(2017).
- Montigny, C. C., Arnou, B., Marchal, E., Champeil, P. Use of glycerol-containing media to study the intrinsic fluorescence properties of detergent-solubilized native or expressed SERCA1a. Biochemistry. 47 (46), 12159-12174 (2008).
- Simongini, M., Puglisi, A., Genovese, F., Hochkoeppler, A. Trehalose counteracts the dissociation of tetrameric rabbit lactate dehydrogenase induced by acidic pH conditions. Arch Biochem Biophys. 740, 109584(2023).
- Shivanna, B. D., Rowe, E. S. Preservation of the native structure and function of Ca2+-ATPase from sarcoplasmic reticulum: Solubilization and reconstitution by new short-chain phospholipid detergent 1,2-diheptanoyl-sn-phosphatidylcholine. Biochem J. 325 (2), 533-542 (1997).
- Sampedro, J. G., Guerra, G., Pardo, J. P., Uribe, S. Trehalose-mediated protection of the plasma membrane H+-ATPase from Kluyveromyces lactis during freeze-drying and rehydration. Cryobiology. 37 (2), 131-138 (1998).
- Sampedro, J. G., Cortés, P., Muñoz-Clares, R. A., Fernández, A., Uribe, S. Thermal inactivation of the plasma membrane H+-ATPase from Kluyveromyces lactis. Protection by Trehalose. Biochim Biophys Acta Proteins Proteomics. 1544 (1-2), 64-73 (2001).
- Sampedro, J. G., et al. Fluorescence quenching by nucleotides of the plasma membrane H+-ATPase from Kluyveromyces lactis. Biochemistry. 46 (18), 5616-5622 (2007).
- Ruiz-Granados, Y. G., De La Cruz-Torres, V., Sampedro, J. G. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958(2019).
- Zhao, P., Zhao, C., Chen, D., Yun, C., Li, H., Bai, L. Structure and activation mechanism of the hexameric plasma membrane H+-ATPase. Nat Commun. 12 (1), 6439(2021).
- Auer, M., Scarborough, G. A., Kühlbrandt, W. Surface crystallization of the plasma membrane H+-ATPase on a carbon support film for electron crystallography. J Mol Biol. 287 (5), 961-968 (1999).
- Hennessey, J. P., Scarborough, G. A. Secondary structure of the Neurospora CRASSA plasma membrane H+-ATPase as estimated by circular dichroism. J Biol Chem. 263 (7), 3123-3130 (1988).
- Scarborough, G. A. Crystallization, structure and dynamics of the proton-translocating P-type ATPase. J Exp Biol. 203 (Pt 1), 147-154 (2000).
- Sumida, K. H., et al. Improving protein expression, stability, and function with proteinMPNN. J Am Chem Soc. 146 (3), 2054-2061 (2024).
- Eleutherio, E., Panek, A., De Mesquita, J. F., Trevisol, E., Magalhães, R. Revisiting yeast trehalose metabolism. Curr Genet. 61 (3), 263-274 (2015).
- Attfield, P. V. Trehalose accumulates in Saccharomyces cerevisiae during exposure to agents that induce heat shock response. FEBS Lett. 225 (1-2), 259-263 (1987).
- Marunde, M. R., et al. Improved tolerance to salt and water stress in Drosophila melanogaster cells conferred by late embryogenesis abundant protein. J Insect Physiol. 59 (4), 377-386 (2013).
- Crowe, J. H. Anhydrobiosis: An unsolved problem with applications in human welfare. Subcell Biochem. 71, 263-280 (2015).
- Crowe, J. H., Crowe, L. M., Chapman, D. Preservation of membranes in anhydrobiotic organisms: The role of trehalose. Science. 223 (4637), 701-703 (1984).
- Sampedro, J. G., Rivera-Moran, M. A., Uribe-Carvajal, S. Kramers' theory and the dependence of enzyme dynamics on trehalose-mediated viscosity. Catalysts. 10 (6), 1-19 (2020).
- Sampedro, J. G., Uribe, S. Trehalose-enzyme interactions result in structure stabilization and activity inhibition. The role of viscosity. Mol Cell Biochem. 256 - 257 (1-2), 319-327 (2004).
- Crowe, J. H. Trehalose as a "chemical chaperone": Fact and fantasy. Adv Exp Med Biol. 594, 143-158 (2007).
- Crowe, J. H., Carpenter, J. F., Crowe, L. M., Anchordoguy, T. J. Are freezing and dehydration similar stress vectors? A comparison of modes of interaction of stabilizing solutes with biomolecules. Cryobiology. 27 (3), 219-231 (1990).
- Rivera-Morán, M. A., Sampedro, J. G. Isolation of the sarcoplasmic reticulum Ca2+-ATPase from rabbit fast-twitch muscle. Methods Protoc. 6 (5), 102(2023).
- Páez-Pérez, E. D., De La Cruz-Torres, V., Sampedro, J. G. Nucleotide binding in an engineered recombinant Ca2+-ATPase N-domain. Biochemistry. 55 (49), 6751-6765 (2016).
- Ramírez-Alonso, J. I., Sampedro, J. G. Effect of cations on ATP binding to the N-domain of Na+, K+-ATPase. J Fluoresc. , (2024).
- De la Cruz-Torres, V., Cataño, V., Olivo-Rodríguez, M., Sampedro, J. G. ANS Interacts with the Ca2+-ATPase nucleotide binding site. J Fluoresc. 30 (3), 483-496 (2020).
- Sampedro, J. G., Nájera, H., Uribe-Carvajal, S., Ruiz-Granados, Y. G. Mapping the ATP binding site in the plasma membrane H+-ATPase from Kluyveromyces lactis. J Fluoresc. 24 (6), 1849-1859 (2014).
- Kiani, A. K., et al. Ethical considerations regarding animal experimentation. J Prev Med Hyg. 63 (2 Suppl 3), E255-E266 (2022).
- Ruiz-Granados, Y., De La Cruz-Torres, V., Sampedro, J. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958(2019).
- Wang, S., et al. Structural basis for sarcolipin's regulation of muscle thermogenesis by the sarcoplasmic reticulum Ca2+-ATPase. Sci Adv. 7 (48), eabi7154(2021).
- Champeil, P., et al. ATP regulation of sarcoplasmic reticulum Ca2+-ATPase. Metal-free ATP and 8-bromo-ATP bind with high affinity to the catalytic site of phosphorylated ATPase and accelerate dephosphorylation. J Biol Chem. 263 (25), 12288-12294 (1988).
- Møller, J. V., Olesen, C. Preparation of Ca(2+)-ATPase1a enzyme from rabbit sarcoplasmic reticulum. Methods Mol Biol. 1377, 11-17 (2016).
- Wuytack, F., De Schutter, G., Casteels, R. Partial purification of (Ca2+ + Mg2+)-dependent ATPase from pig smooth muscle and reconstitution of an ATP-dependent Ca2+-transport system. Biochem J. 198 (2), 265-271 (1981).
- Mandal, A., et al. Solubilization, purification and reconstitution of Ca2+-ATPase from bovine pulmonary artery smooth muscle microsomes by different detergents: Preservation of native structure and function of the enzyme by DHPC. Biochim Biophys Acta Gen Subj. 1760 (1), 20-31 (2006).
- Inesi, G., Ma, H., Hua, S., Toyoshima, C. Characterization of Ca2+ ATPase residues involved in substrate and cation binding. Ann NY Acad Sci. 986 (1), 63-71 (2003).
- Van Winkle, W. B., Pitts, B. J., Entman, M. L. Rapid purification of canine cardiac sarcoplasmic reticulum Ca2+-ATPase. J Biol Chem. 253 (24), 8671-8673 (1978).
- Ruiz-Granados, Y. G., De La Cruz-Torres, V., Sampedro, J. G. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958(2019).
- Teramoto, N., Sachinvala, N. D., Shibata, M. Trehalose and trehalose-based polymers for environmentally benign, biocompatible, and bioactive materials. Molecules. 13 (8), 1773-1816 (2008).
- Dao, T. T., et al. Demethoxycurcumin is a potent inhibitor of P-type ATPases from diverse kingdoms of life. PLOS ONE. 11 (9), e0163260(2016).
- Bleeker, N. P., Cornea, R. L., Thomas, D. D., Xing, C. A novel SERCA inhibitor demonstrates synergy with classic SERCA inhibitors and targets multidrug-resistant AML. Mol Pharmaceutics. 10 (11), 4358-4366 (2013).
- Michelangeli, F., East, J. M. A diversity of SERCA Ca2+ pump inhibitors. Biochem Soc Trans. 39 (3), 789-797 (2011).
- Tadini-Buoninsegni, F., Smeazzetto, S., Gualdani, R., Moncelli, M. R. Drug interactions with the Ca2+-ATPase from Sarco(Endo)Plasmic Reticulum (SERCA). Front Mol Biosci. 5, 36(2018).
- Clausen, J. D., et al. Elucidation of antimicrobial activity and mechanism of action by N-substituted carbazole derivatives. Bioorg Med Chem Lett. 27 (19), 4564-4570 (2017).
- Leary, S., et al. AVMA guidelines for the euthanasia of animals: 2020 Edition, Members of the Panel on Euthanasia AVMA Staff Consultants. , AVMA. Schaumburg, IL. (2020).
- de Aluja, A. S. Laboratory animals and official Mexican norms (NOM-062-ZOO-1999). Gac Med Mex. 138 (3), 295-298 (2002).
- Champeil, P., Buschlen-Boucly, S., Bastide, F., Gary-Bobo, C. Sarcoplasmic reticulum ATPase. Spin labeling detection of ligand-induced changes in the relative reactivities of certain sulfhydryl groups. J Biol Chem. 253 (4), 1179-1186 (1978).
- Skalec, A., Janeczek, M., Czerski, A. Anatomy and histology of the rabbit common calcanean tendon. J Vet Med Series C: Anat Histol Embryol. 48 (5), 466-475 (2019).
- Janes, L. E., Mioton, L. M., Fracol, M. E., Ko, J. H. An in vivo comparison: Novel mesh suture versus traditional suture-based repair in a rabbit tendon model. J Hand Surg Glob Online. 4 (1), 32-39 (2022).
- Bollag, D. M., Rozycki, M. D., Edelstein, S. J. Protein Methods. , Wiley. (1996).
- Gagné, F. Tissue preparation and subcellular fractionation techniques. Biochem Ecotoxicol. , 21-31 (2014).
- Lowry, O. H., Rosebrough, N. J., Farr, A. L., Randall, R. J. Protein measurement with the Folin phenol reagent. J Biol Chem. 193 (1), 265-275 (1951).
- Autry, J. M., Rubin, J. E., Svensson, B., Li, J. L., Thomas, D. D. Nucleotide activation of the Ca-ATPase. J Biol Chem. 287 (46), 39070-39082 (2012).
- Winters, D. L., Autry, J. M., Svensson, B., Thomas, D. D. Interdomain fluorescence resonance energy transfer in SERCA probed by cyan-fluorescent protein fused to the actuator domain. Biochemistry. 47 (14), 4246-4256 (2008).
- Miles, A. J., Wallace, B. A. Circular dichroism spectroscopy of membrane proteins. Chem Soc Rev. 14 (45), 1130-1135 (2016).
- Scofano, H. M., Vieyra, A., de Meis, L. Substrate regulation of the sarcoplasmic reticulum ATPase. Transient kinetic studies. J Biol Chem. 254 (20), 10227-10231 (1979).
- Yamamoto, T., Tonomura, Y. Reaction mechanism of the Ca++-dependent ATPase of sarcoplasmic reticulum from skeletal muscle: I. Kinetic studies. J Biochem. 62 (5), 558-575 (1967).
- Murphy, A. J. Affinity labeling of the active site of the Ca2+-ATPase of sarcoplasmic reticulum. Biochim Biophys Acta - Biomembr. 946 (1), 57-65 (1988).
- Csermely, P., Katopis, C., Wallace, B. A., Martonosi, A. The E1→E2 transition of Ca2+-transporting ATPase in sarcoplasmic reticulum occurs without major changes in secondary structure. A circular-dichroism study. Biochem J. 241 (3), 663-669 (1987).
- Abràmoff, M. D., Magalhães, P. J., Ram, S. J. Image processing with ImageJ. Biophotonics Int. 11 (7), 36-42 (2004).
- Scarborough, G. A. The plasma membrane proton-translocating ATPase. Cell Mol Life Sci. 57 (6), 871-883 (2000).
- Llopis, J., McCaffery, J. M., Miyawaki, A., Farquhar, M. G., Tsien, R. Y. Measurement of cytosolic, mitochondrial, and Golgi pH in single living cells with green fluorescent proteins. Proc Natl Acad Sci. 95 (12), 6803-6808 (1998).
- Kanai, R., Vilsen, B., Cornelius, F., Toyoshima, C. Crystal structures of Na+, K+-ATPase reveal the mechanism that converts the K+-bound form to Na+-bound form and opens and closes the cytoplasmic gate. FEBS Lett. 597 (15), 1957-1976 (2023).
- Palmgren, M. P-type ATPases: Many more enigmas left to solve. J Biol Chem. 299 (11), 105352(2023).
- Frauenfelder, H. Ask not what physics can do for biology-Ask what biology can do for physics. Phys Biol. 11 (5), 053004(2014).
- Frauenfelder, H. The physics of proteins. Annu Rev Biophys Biomol Struct. 36, Springer. New York. New York, NY. (2010).
- Narhi, L. O. Biophysics for Therapeutic Protein Development. , Springer Science+Business Media. (2013).
- East, J. M. Purification of a membrane protein (Ca2+/Mg2+-ATPase) and its reconstitution into lipid vesicles. Methods Mol Biol. 27, 87-94 (1994).
- Lee, E. -H., et al. Enhancement of enzyme activity and stability by poly(γ-glutamic acid). Polymer J. 42 (10), 818-822 (2010).
- Norrild, R. K., et al. Increasing protein stability by inferring substitution effects from high-throughput experiments. Cell Rep Methods. 2 (11), 100333(2022).
- Song, Z., Zhang, Q., Wu, W., Pu, Z., Yu, H. Rational design of enzyme activity and enantioselectivity. Front Bioeng Biotechnol. 11, 1-14 (2023).
- Jidenko, M., Lenoir, G., Fuentes, J. M., le Maire, M., Jaxel, C. Expression in yeast and purification of a membrane protein, SERCA1a, using a biotinylated acceptor domain. Protein Expr Purif. 48 (1), 32-42 (2006).
- Hua, S., Ma, H., Lewis, D., Inesi, G., Toyoshima, C. Functional role of "N" (nucleotide) and "P" (phosphorylation) domain interactions in the sarcoplasmic reticulum (SERCA) ATPase. Biochemistry. 41 (7), 2264-2272 (2002).
- Pang, Y. -H., Chen, J. -W. Anisodamine causes the changes of structure and function in the transmembrane domain of the Ca2+-ATPase from sarcoplasmic reticulum. Biosci Biotechnol Biochem. 68 (1), 126-131 (2004).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados