È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Purificazione del reticolo sarco-endoplasmatico Ca2+-ATPasi dal muscolo del coniglio
In questo articolo
Riepilogo
Questo protocollo descrive un metodo di purificazione SERCA migliorato, che include il trealosio disaccaride nella fase finale di centrifugazione. Questo carboidrato stabilizza le proteine in condizioni difficili. Il SERCA purificato era cataliticamente attivo e mostrava un'elevata purezza, il che lo rendeva adatto per studi strutturali e funzionali.
Abstract
Alcune ATPasi di tipo P, come la Ca2+-ATPasi del reticolo sarco/endoplasmatico (SERCA), sono proteine di membrana intrinsecamente labili che richiedono specifiche condizioni fisico-chimiche durante la purificazione per ottenerle con elevata purezza e qualità strutturale e in una forma cataliticamente attiva. Il disaccaride trealosio è un soluto compatibile che viene sintetizzato e accumulato in alte concentrazioni nel citoplasma del lievito per stabilizzare le membrane e le proteine. L'uso del trealosio come additivo nel protocollo per la purificazione della membrana plasmatica H+-ATPasi si traduce in una preparazione di alta qualità, la cui struttura esamerica è dimostrata da metodi analitici biochimici. Il trealosio può, quindi, essere utilizzato come additivo stabilizzante per la purificazione delle proteine di membrana (P-ATPasi). Questo protocollo descrive la modifica del protocollo classico per la purificazione di SERCA sottoponendo SERCA a centrifugazione su un gradiente di concentrazione di trealosio. L'inclusione di questo carboidrato ha portato alla purificazione di SERCA in una forma cataliticamente attiva con elevata purezza e, soprattutto, in una forma stabile. La parziale caratterizzazione biochimica del SERCA purificato (SDS-PAGE, cinetica enzimatica, marcatura FITC, spettroscopia di dicroismo circolare) ha dimostrato che l'enzima è adatto per studi funzionali e strutturali. Si suggerisce l'uso del trealosio nel protocollo di purificazione delle ATPasi di tipo P e di altre proteine labili di membrana (e citosoliche).
Introduzione
Le proteine/enzimi di membrana sono componenti biologici essenziali delle cellule in quanto svolgono un ruolo critico in vari processi 1,2,3. Alcune delle funzioni possono includere il trasporto di ioni e molecole dentro e fuori la cellula/compartimenti interni (sia attivi che passivi), il riconoscimento cellula-cellula, il legame intercellulare, l'ancoraggio/attaccamento e il rilevamento dell'ambiente esterno attraverso l'integrazione con il macchinario di trasduzione del segnale in condizioni fisiche e chimiche normali e difficili (alto sale, bassa acqua, alta temperatura, resistenza ai farmaci, ecc.)3 Pertanto, la determinazione della struttura tridimensionale (3D) delle proteine di membrana e/o degli enzimi è diventata di grande importanza sia per la ricerca di base che per quella applicata 4,7,6. È importante sottolineare che le proteine/enzimi di membrana sono stati ampiamente utilizzati come bersagli per la scoperta di farmaci (sia naturali che intenzionali)7,8,9. Cioè, le proteine di membrana hanno un'importanza intrinseca nella salute 9,10,11.
Il carattere idrofobico delle proteine/enzimi di membrana è la proprietà fisico-chimica tecnicamente più impegnativa per il laboratorio sperimentale 12,13,14, ancora di più quando si lavora con proteine di membrana integrali oligomeriche e/o altamente labili 15,16. L'isolamento di quantità appropriate della proteina/enzima di membrana con la massima qualità possibile per saggi sperimentali funzionali e studi strutturali è altamente auspicabile17. Le proteine di membrana, essendo intrinsecamente idrofobiche, sono molto difficili da purificare e la scelta del detergente è solitamente una delle questioni più importanti da considerare 17,18,19,20. A questo proposito, i metodi di laboratorio utilizzati per isolare le proteine di membrana causano tipicamente un certo grado di danno alla disposizione strutturale 3D della proteina21. Alcuni di questi metodi includono l'uso di (a) sonicazione (onde ultrasoniche ad alta frequenza/energia), (b) detergenti solubilizzanti le proteine (duri, medi o delicati)22, (c) pressioni relativamente elevate nella cromatografia su colonna e nell'ultracentrifugazione ad alta velocità23, (d) molecole precipitanti, (e) enzimi digestivi e altri21. Tutti questi processi possono contribuire o essere la causa principale della destabilizzazione delle proteine durante la purificazione21. A questo proposito, alcuni protocolli sembrano funzionare relativamente bene per una data proteina di membrana. Tuttavia, l'ottimizzazione è sempre benvenuta quando si utilizzano nuovi saggi moderni o metodi che richiedono una qualità superiore della preparazione delle proteine di membrana per ottenere risultati soddisfacenti24. Le fasi di ottimizzazione possono includere, a titolo esemplificativo ma non esaustivo, il miglioramento della progettazione del costrutto, la ricerca di condizioni ottimali per l'espressione delle proteine di membrana, la definizione di migliori condizioni di manipolazione (ad es. pH, temperatura, ecc.), la ricerca del miglior detergente compatibile, la regolazione delle fasi di purificazione come il tempo di sonicazione, la velocità di centrifugazione, la riformulazione di soluzioni tampone mediante l'aggiunta di agenti stabilizzanti, ecc.25,26,27. Pertanto, qualsiasi cambiamento nella metodologia di purificazione che porti ad un aumento della qualità (purezza) e dell'attività della proteina/enzima di membrana purificato è importante.
Nella famiglia delle ATPasi di tipo P, la membrana plasmatica del lievito H+-ATPasi sembra essere uno dei membri più labili28,29. La H+-ATPasi perde la sua attività idrolizzante dell'ATP in seguito a disidratazione (liofilizzazione), shock termico, ecc.28,29. L'uso del trealosio come stabilizzatore proteico è stato testato nell'isolamento della membrana plasmatica H+-ATPasi dal lievito K. lactis30,31; il preparato di H+-ATPasi ottenuto era di elevata purezza e cataliticamente attivo. È importante sottolineare che ciò ha permesso di risolvere lo stato oligomerico dell'enzima con metodi biochimici, rivelando che si trattava di un esamero e successivamente confermato dalla microscopia crioelettronica 31,32,33,34,35. Pertanto, sembra probabile che la delicata disposizione tridimensionale (3D) delle proteine di membrana possa essere persa in condizioni relativamente difficili durante la purificazione36. La cinetica di inattivazione bifasica è osservata per H+-ATPasi durante l'inattivazione mediata dal calore29. Nelle cellule di lievito, come in molti altri organismi, il trealosio disaccaride si accumula ad alte concentrazioni in condizioni di stress ambientale 37,38,39. Il trealosio mantiene l'integrità della membrana e la funzione di trasporto stabilizzando le proteine (sia incorporate nella membrana che citosoliche) e le membrane cellulari40,41. Il meccanismo stabilizzante del trealosio è stato ampiamente studiato da diversi gruppi e dal nostro laboratorio 42,43,44,45. Esperimenti con l'H+-ATPasi e altri enzimi hanno dimostrato che il trealosio è lo stabilizzatore proteico più efficace tra mono- e disaccaridi28,29. Ciò ha portato alla sua inclusione nel protocollo di purificazione H+-ATPasi30. Recentemente, il trealosio è stato utilizzato anche nella purificazione del reticolo sarcoplasmatico Ca2+-ATPasi (SERCA) dal muscolo a contrazione rapida del coniglio con buoni risultati in termini di purezza e attività proteica46. Pertanto, il trealosio sembra essere un additivo buono e appropriato per la purificazione delle ATPasi di tipo P e probabilmente di altre proteine di membrana e citosoliche.
Per le P-ATPasi, l'esistenza di domini citoplasmatici strutturali è un vantaggio sperimentale, in particolare per gli studi di interazione substrato/ligando47; Il legame con l'ATP è stato studiato in domini N ricombinanti altamente puri 47,48,49, eliminando così le considerazioni tecniche per la purificazione degli enzimi dell'intera membrana 47,48,50, tra gli altri51. Sfortunatamente, alcuni studi funzionali (conversione catalitica/energetica) e strutturali (disposizione delle subunità e interazione con altre proteine) richiedono ancora l'intera P-ATPasi52,53. A questo proposito, la purificazione di SERCA è stata raggiunta da diversi gruppi di ricerca 54,55,56,57,58. Tuttavia, i miglioramenti possono ancora essere implementati46, ad esempio, aumentando l'integrità dell'ATPasi59 purificata, evitando la denaturazione/disgregazione proteica dei complessi proteici25, aumentando la solubilizzazione senza denaturazione della proteina di membrana (cioè, evitando la formazione di aggregati macromolecolari)60, portando a una migliore compatibilità con i saggi e altri metodi analitici a valle61 Inoltre, quando nuove strategie sperimentali, additivi, inibitori enzimatici, ecc. compaiono nella letteratura scientifica 61,62,63,64,65,66, a volte devono essere testate con l'intera P-ATPasi. Questo lavoro descrive il protocollo per la purificazione di SERCA e l'uso del trealosio come additivo per stabilizzare la struttura proteica e l'attività dell'ATPasi; Cioè, oltre ad aumentare la qualità dell'enzima (strutturale), il trealosio aiuta a prevenire la perdita della struttura e dell'attività dell'enzima durante l'isolamento e lo stoccaggio dell'enzima, il che aiuta a risparmiare materiale biologico e quindi a ridurre il numero di purificazioni enzimatiche.
Access restricted. Please log in or start a trial to view this content.
Protocollo
Tutte le procedure sugli animali sono state eseguite in conformità con le linee guida internazionali e locali (NORMA Oficial Mexicana NOM-062-ZOO-1999) per la gestione degli animali nei laboratori sperimentali 51,67,68. Il tessuto muscolare è stato ottenuto da Oryctolagus cuniculus wild-type da un'unità locale di manipolazione degli animali (INE/CITES/DGVS-ZOO-E0055-SLP-98)46. Un veterinario esperto nella gestione degli animali da laboratorio ha eseguito la dissezione muscolare iniziale e il trattamento. I dettagli dei reagenti e delle attrezzature utilizzate in questo studio sono elencati nella tabella dei materiali.
1. Isolamento del reticolo sarcoplasmatico dal muscolo di coniglio a contrazione rapida (Timing, 7 - 9 h)
NOTA: Per i dettagli sulla procedura, fare riferimento a Champeil et al.69.
- Ottenere tessuto muscolare a contrazione rapida (~70 g da entrambe le zampe posteriori per animale) da Oryctolagus cuniculus wild-type.
NOTA: È richiesta una preventiva identificazione visiva dei muscoli da sezionare70,71. - Macerare il muscolo a contrazione rapida in un frullatore sospendendolo in tre volumi da 100 mM di KCl, con 1 min in acceso e 1 min in riposo ghiacciato, ripetere tre volte72.
- Rimuovere i detriti di tessuto mediante centrifugazione (1500 × g) a 4 °C per 5 min; Se necessario, viene eseguita una seconda centrifugazione (4200 × g).
- Raccogliere il surnatante e omogeneizzarlo utilizzando un trituratore per tessuti (10 colpi a bassa velocità, cioè 500-1.500 giri/min)72,73. Centrifugare l'omogeneizzato (10.000 × g) a 4 °C per 15 minuti.
NOTA: Prestare attenzione durante l'omogeneizzazione, poiché l'uso di una velocità relativamente elevata può causare danni al sistema e all'operatore. - Centrifugare il surnatante (contenente il reticolo sarcoplasmatico) (33.000 × g) a 4 °C per 120 min.
- Sospendere i pellet in 40 mL di 0,5 M di saccarosio e centrifugare (12.000 × g) a 4 °C per 15 min. Diluire il surnatante (0,6 M di KCl e 0,15 M di saccarosio) e centrifugare la sospensione (34.000 × g) a 4 °C per 165 min.
- Sospendere il pellet contenente i fogli delle vescicole del reticolo sarcoplasmatico (SRV) in 0,3 M di saccarosio, 0,1 M di KCl e 5 mM di Tris-HCl, pH 7,0. Determinare la concentrazione proteica utilizzando il saggio di Lowry74 e l'albumina sierica umana come standard proteico.
- Aliquotare gli SRV (30-32 mg/mL) in volume di 1 mL. Conservare i campioni di SRV a -72 °C per un'ulteriore elaborazione.
NOTA: La resa proteica è di 100-110 mg di SRV per animale.
2. Purificazione SERCA (Temporizzazione, 18 - 20 h)
NOTA: Per i dettagli sulla procedura, fare riferimento a Rivera-Morán et al.46.
- Diluire la sospensione di SRV a 2 mg di proteine/mL utilizzando 75 mM ghiacciati di Tris-HCl, pH 7,2, 0,6 M di KCl, 6 mM di EDTA, 1 mM di EGTA e 0,1% (p/v) desossicolato (DOC). Incubare la sospensione di SRV su ghiaccio per 10 minuti e agitare delicatamente. Centrifugare la sospensione SRV (100.000 × g) a 4 °C per 60 min.
NOTA: DOC dovrebbe essere aggiunto goccia a goccia. - Sospendi i pellet in <3 mL di 25 mM di Tris-HCl, pH 7,5, 0,3 M di KCl, glicerolo al 45% (v/v) e 2 mM EDTA. Omogeneizzare la sospensione e regolare fino a un volume finale di 10 mL. Determinare la concentrazione proteica e quindi regolarla a 6,5 mg/mL. Aggiungere azolectina a 5 mg/mL e 0,85 (p/p) Zwittergent 3-14.
NOTA: Azolectin e Zwittergent 3-14 devono essere aggiunti goccia a goccia. - Omogeneizzare la sospensione e centrifugare (100.000 × g) a 4 °C per 60 min. Raccogliere il surnatante e diluire 1:2 con 2 mM di EGTA (pH 7,2).
- Versare delicatamente la sospensione su un gradiente di concentrazione discontinuo di trealosio (45, 40, 35 e 30% p/v) in 10 mM di Tris-HCl, pH 7,0, 1 mM di EDTA, 0,1% desossicolato e 1 mg/mL di azolectina. Centrifugare (100.000 × g) a 4 °C per 14 h. Raccogliere il pellet trasparente, leggermente giallastro, formatosi sul fondo dei tubi.
NOTA: Sia la formazione del gradiente di trealosio che l'aggiunta della sospensione del campione devono essere eseguite utilizzando tecniche appropriate. Si consiglia la centrifugazione notturna. - Sospendi delicatamente il pellet in un piccolo volume (<1,5 mL) di 25 mM di Tris-HCl, pH 7,5, 0,3 di M KCl, glicerolo al 45% e 2 mM di EDTA. Determinare la concentrazione proteica. Regolare la concentrazione di proteine mediante diluizione a 2 mg/mL. Prelevare aliquote (~50 μL) della sospensione e conservare il SERCA purificato a -72 °C fino al suo utilizzo.
- Eseguire la SDS-PAGE del SERCA72 purificato e dei campioni proteici prelevati in diverse fasi di purificazione. Colorare il gel con Coomassie Blue72.
NOTA: L'uso di trealosio 0,6 M al posto del glicerolo nel tampone di sospensione aumenta la stabilità dell'enzima durante i cicli di conservazione e congelamento-scongelamento28,29.
3. Determinazione dell'attività dell'ATPasi (Timing, 1 - 2 h)
- Saggio accoppiato a un enzima: ATPasi-piruvato chinasi-lattato deidrogenasi
- Preparare la miscela di reazione (1 mL): 50 mM MOPS (pH 7,0), 1 mM EGTA, 80 mM KCl, 5 mM MgCl2, 3 mM CaCl2, 5 mM fosfoenolpiruvato, 250 μM NADH. Includere concentrazioni variabili di ATP (da 0,001 a 0,25 mM).
- Omogeneizzare accuratamente il saggio di reazione mediante vortex. Incubare il saggio di reazione a 37 °C per 10 minuti. Aggiungere 0,9 U L-lattato deidrogenasi (LDH) e 1,5 U piruvato chinasi (PK).
- Avviare la reazione dell'ATPasi con l'aggiunta di 10 μg di SERCA.
- Determinare la formazione del NADH. Seguire la variazione dell'intensità dell'assorbanza ogni secondo per 600 s a una lunghezza d'onda (λ) di 340 nm nel tempo utilizzando uno spettrofotometro con un portacella termostatato.
NOTA: L'intervallo di tempo per la misurazione dell'assorbanza può variare, ma cercare di registrare il maggior numero possibile di punti dati per tracciare la linea retta iniziale della reazione ATPasi.
- Determinare il tasso di idrolisi dell'ATP
NOTA: Per i dettagli sulla procedura, fare riferimento a Rivera-Morán et al.46.- Calcolare il valore di pendenza (Δabs/min) della porzione lineare in ogni curva formata. Utilizzare il coefficiente di estinzione molare (ε) del NADH (λ = 6.220 M−1·cm−1) per determinare l'attività dell'ATPasi (μmoli di ATP idrolizzato/min.mg prot.)
- Determinare i parametri cinetici K, m e Vmax
- Traccia i dati di velocità rispetto alla concentrazione di ATP utilizzando il software di analisi dei dati e la creazione di grafici.
- Adattare i dati mediante regressione non lineare all'equazione di Michaelis-Menten (Eq. 1)46:
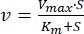
dove v è la velocità, Vmax è la velocità massima, [S] è la concentrazione di ATP e Km è la costante di Michaelis-Menten. - Determinare K, m e Vmax per iterazione.
NOTA: L'inclusione di DOC può aumentare la risposta al tasso di ATPasi; si consiglia di testare diverse concentrazioni di DOC per trovare l'ottimale.
4. Saggio di marcatura di SERCA con isotiocianato di fluoresceina (FITC) (tempistiche, 30 - 45 minuti, solo per marcatura FITC)
NOTA: Per i dettagli sulla procedura, fare riferimento a 54,75,76.
- Sospendere SERCA (20 μg) in 50 μL (volume finale) di tampone di marcatura (100 mM di KCl, 5 mM di MgCl2 e 30 mM di Tris-HCl, pH 8,9) contenente 1 mM di FITC. Mescolare a vortice.
- Incubare i campioni in momenti diversi (15 min, 10 min, 7 min, 5 min e 2 min) al buio e a temperatura ambiente.
- Arrestare la reazione di marcatura aggiungendo 1 volume di tampone di arresto ghiacciato (480 mM di saccarosio e 48 mM di MOPS, pH 7,0), contenente ATP (5 mM). Incubare su ghiaccio per 5 minuti al buio.
- Sottoporre il SERCA con etichetta FITC a SDS-PAGE72. Esporre i gel trasparenti alla luce UV (λ = 302 nm). Documento fotografico del risultato.
- Colorare il gel con il blu Coomassie. Documento fotografico del risultato.
NOTA: L'esecuzione della marcatura FITC in una stanza buia può migliorare la qualità del segnale di fluorescenza.
5. Spettri di dicroismo circolare (CD) (Temporizzazione, 30 min)
NOTA: Per i dettagli sulla procedura, fare riferimento a11,77.
- Sospendere SERCA (3 μM) in 400 μL di tampone fosfato 10 mM (pH 7,0) a 25 °C.
- Caricare il campione in una cella di 0,1 cm di lunghezza del percorso. Impostare l'intervallo λ degli spettri UV lontani tra 190 e 260 nm. Impostare la risoluzione interna e la larghezza di banda a 1 nm.
- Registrare il segnale CD a 50 nm/min a 25 °C.
Access restricted. Please log in or start a trial to view this content.
Risultati
SDS-PAGE di SERCA a diversi stadi di purificazione (Figura 1). Il gel colorato di blu di Coomassie mostra l'arricchimento della banda proteica SERCA (peso molecolare apparente >100 kDa) man mano che il protocollo di purificazione progredisce. La banda proteica corrispondente a SERCA mostra una purezza del >90% dopo centrifugazione su un gradiente di concentrazione di trealosio. Grafico del tasso di idrolisi dell'ATP rispetto alla concentrazione di A...
Access restricted. Please log in or start a trial to view this content.
Discussione
La maggior parte delle molecole e degli ioni non può attraversare liberamente le membrane cellulari, ad esempio, il protone (H+) richiede un trasportatore di membrana nella membrana plasmatica di una varietà di organismi e organelli come i mitocondri83,84. Le membrane cellulari sono selettive e le molecole e gli ioni che attraversano le membrane cellulari sono diversi, quindi nella cellula si possono trovare diversi ...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
L'autore dichiara di non avere interessi finanziari concorrenti.
Riconoscimenti
L'autore ringrazia l'aiuto di Edmundo Mata-Morales nel montaggio del video, VM, Valentín de la Cruz-Torres nella purificazione degli SRV, Miguel A. Rivera-Moran nella purificazione e analisi SERCA e Juan C. Gonzalez-Castro, Franco E. Juarez, Alejandra Nevarez, Nicolas Rocha-Vizuet e Jocelin I. Ramírez-Alonso nella produzione video. Non sono stati ricevuti fondi, sovvenzioni o altro sostegno per condurre questo studio.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| ATP | Sigma-Aldrich Corp | A2383 | |
| Azolectin from soybean | Sigma-Aldrich Corp | 44924 | |
| Benchtop UV transilluminator | Cole-Parmer | EW-97623-08 | Dual intensity High setting is ideal for analytical documentation. Low setting reduces photonicking or photobleaching of gel samples while doing preparative work. |
| CaCl2 • 2H2O | Sigma-Aldrich Corp | 223506 | |
| Coolpix B500 camera | Nikon Corp | S210 | |
| Coomassie brilliant blue G-250 | Bio-Rad | 1610406 | |
| Dodecyl maltoside | Sigma-Aldrich Corp | D4641 | |
| Ethylene glycol-bis(2-aminoethylether)-N,N,N′,N′-tetraacetic acid | Sigma-Aldrich Corp | E4378 | |
| Ethylenediaminetetraacetic acid tetrasodium salt dihydrate | Sigma-Aldrich Corp | E6511 | |
| Fluorescein isothiocyanate | Sigma-Aldrich Corp | F3651 | |
| KCl | JT Baker | 7447-40-7 | |
| MgCl2 • 6H2O | Sigma-Aldrich Corp | M9272 | |
| MOPS | Sigma-Aldrich Corp | M1254 | |
| NADH | Chem-Impex International Inc | 230 | |
| N-tetradecyl-N,N- dimethyl-3-ammonium-1-propanesulfonate | Sigma-Aldrich Corp | D0431 | |
| Phosphoenolpyruvate (PEP) | Chem-Impex International Inc | 9711 | |
| Rabbit muscle lactate dehydrogenase | Roche | 10003557103 | |
| Rabbit muscle pyruvate kinase | Sigma-Aldrich Corp | P1506 | |
| Sodium deoxycholate | Sigma-Aldrich Corp | D6750 | |
| Sodium dodecyl sulfate | Bio-Rad | 1610302 | |
| Spectropolarimeter | Jasco Corp. | Jasco J1500 | |
| Sucrose | Sigma-Aldrich Corp | 84100 | |
| Trehalose | Sigma-Aldrich Corp | T0167 | Dihydrate |
| Tris(hydroxymethyl)aminomethane hydrochloride | Sigma-Aldrich Corp | 857645 | |
| Ultracentrifuge | Beckman | Optima XPN | |
| UV/VIS spectrophotometer | Agilent Technologies | 8453 | The Agilent 8453 UV-Vis Spectrophotometer uses a photodiode array for simultaneous measurement of the complete ultra-violet to visible light spectrum |
| WiseStir HS- 30E | Daihan Scientific Co. | DH.WOS01010 | Ideal for all disperging and homogenizing applications, designed for tissue grinders. |
Riferimenti
- Schuberth, C., Wedlich-Söldner, R. Building a patchwork - The yeast plasma membrane as model to study lateral domain formation. Biochim Biophys Acta Mol Cell Res. 1853 (4), 767-774 (2015).
- Douglas, L. M., Konopka, J. B. Fungal membrane organization: The eisosome concept. Annu Rev Microbiol. 68 (1), 377-393 (2014).
- Borrell, J. H., Domènech, Ò, Keough, K. M. W. Membrane protein - Lipid interactions: Physics and Chemistry in the Bilayer. , Springer International Publishing. (2016).
- Moraes, I., Evans, G., Sanchez-Weatherby, J., Newstead, S., Stewart, P. D. The next generation in membrane protein structure determination. , Springer International Publishing. (2016).
- White, S. H. Biophysical dissection of membrane proteins. Nature. 459 (7245), 344-346 (2009).
- Lacapère, J. -J. Membrane protein structure determination. Methods Mol Biol. , Humana Press. 459(1002).
- Palmgren, M. G. Plant plasma membrane H+-ATPases: Powerhouses for nutrient uptake. Annu Rev Plant Physiol Plant Mol Biol. 52 (1), 817-845 (2001).
- Yatime, L., et al. P-type ATPases as drug targets: Tools for medicine and science. Biochim Biophys Acta Bioenerg. 1787 (4), 207-220 (2009).
- Drews, J. Tetrahydrobiopterin biosynthesis as an off-target of sulfa drugs. Science. 287 (5460), 1960-1964 (2000).
- Xu, Z., Meshcheryakov, V. A., Poce, G., Chng, S. -S. MmpL3 is the flippase for mycolic acids in mycobacteria. Proc Natl Acad Sci USA. 114 (30), 7993-7998 (2017).
- Siligardi, G., Hussain, R., Patching, S. G., Phillips-Jones, M. K. Ligand- and drug-binding studies of membrane proteins revealed through circular dichroism spectroscopy. Biochim Biophys Acta Biomembr. 1838 (1 PARTA), 34-42 (2014).
- Rawlings, A. E. Membrane proteins: Always an insoluble problem. Biochem Soc Trans. 44 (3), 790-795 (2016).
- Popot, J. -L. Membrane proteins in aqueous solutions. , Springer International Publishing. 708(2018).
- Palmgren, M. G., Sommarin, M., Ulvskov, P., Larsson, C. Effect of detergents on the H+-ATPase activity of inside-out and right-side-out plant plasma membrane vesicles. Biochim Biophys Acta Biomembr. 1021 (2), 133-140 (1990).
- McBride, Z., Chen, D., Reick, C., Xie, J., Szymanski, D. B. Global analysis of membrane-associated protein oligomerization using protein correlation profiling. Mol Cell Proteomics. 16 (11), 1972-1989 (2017).
- Yoneda, J. S., et al. Multimeric species in equilibrium in detergent-solubilized Na, K-ATPase. Int J Biol Macromol. 89, 238-245 (2016).
- Ohlendieck, K. Extraction of membrane proteins. Protein Purif Protocols. 59, 283-294 (2004).
- Champeil, P., et al. A robust method to screen detergents for membrane protein stabilization, revisited. Anal Biochem. 511, 31-35 (2016).
- Gimpl, K., Klement, J., Keller, S. Characterising protein/detergent complexes by triple-detection size-exclusion chromatography. Biol Proced Online. 18, 4(2016).
- le Maire, M., et al. Gel chromatography and analytical ultracentrifugation to determine the extent of detergent binding and aggregation, and Stokes radius of membrane proteins using sarcoplasmic reticulum Ca2+-ATPase as an example. Nat Protoc. 3 (11), 1782-1795 (2008).
- Du, M., et al. Progress, applications, challenges and prospects of protein purification technology. Front Bioeng Biotechnol. 10, 1-26 (2022).
- Slotboom, D. J., Duurkens, R. H., Olieman, K., Erkens, G. B. Static light scattering to characterize membrane proteins in detergent solution. Methods. 46 (2), 73-82 (2008).
- Harding, S. E. Analytical ultracentrifugation as a matrix-free probe for the study of kinase related cellular and bacterial membrane proteins and glycans. Molecules. 26, 6080(2021).
- Chen, Y. -C., et al. Thermal stability, storage and release of proteins with tailored fit in silica. Sci Rep. 7 (1), 46568(2017).
- Montigny, C. C., Arnou, B., Marchal, E., Champeil, P. Use of glycerol-containing media to study the intrinsic fluorescence properties of detergent-solubilized native or expressed SERCA1a. Biochemistry. 47 (46), 12159-12174 (2008).
- Simongini, M., Puglisi, A., Genovese, F., Hochkoeppler, A. Trehalose counteracts the dissociation of tetrameric rabbit lactate dehydrogenase induced by acidic pH conditions. Arch Biochem Biophys. 740, 109584(2023).
- Shivanna, B. D., Rowe, E. S. Preservation of the native structure and function of Ca2+-ATPase from sarcoplasmic reticulum: Solubilization and reconstitution by new short-chain phospholipid detergent 1,2-diheptanoyl-sn-phosphatidylcholine. Biochem J. 325 (2), 533-542 (1997).
- Sampedro, J. G., Guerra, G., Pardo, J. P., Uribe, S. Trehalose-mediated protection of the plasma membrane H+-ATPase from Kluyveromyces lactis during freeze-drying and rehydration. Cryobiology. 37 (2), 131-138 (1998).
- Sampedro, J. G., Cortés, P., Muñoz-Clares, R. A., Fernández, A., Uribe, S. Thermal inactivation of the plasma membrane H+-ATPase from Kluyveromyces lactis. Protection by Trehalose. Biochim Biophys Acta Proteins Proteomics. 1544 (1-2), 64-73 (2001).
- Sampedro, J. G., et al. Fluorescence quenching by nucleotides of the plasma membrane H+-ATPase from Kluyveromyces lactis. Biochemistry. 46 (18), 5616-5622 (2007).
- Ruiz-Granados, Y. G., De La Cruz-Torres, V., Sampedro, J. G. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958(2019).
- Zhao, P., Zhao, C., Chen, D., Yun, C., Li, H., Bai, L. Structure and activation mechanism of the hexameric plasma membrane H+-ATPase. Nat Commun. 12 (1), 6439(2021).
- Auer, M., Scarborough, G. A., Kühlbrandt, W. Surface crystallization of the plasma membrane H+-ATPase on a carbon support film for electron crystallography. J Mol Biol. 287 (5), 961-968 (1999).
- Hennessey, J. P., Scarborough, G. A. Secondary structure of the Neurospora CRASSA plasma membrane H+-ATPase as estimated by circular dichroism. J Biol Chem. 263 (7), 3123-3130 (1988).
- Scarborough, G. A. Crystallization, structure and dynamics of the proton-translocating P-type ATPase. J Exp Biol. 203 (Pt 1), 147-154 (2000).
- Sumida, K. H., et al. Improving protein expression, stability, and function with proteinMPNN. J Am Chem Soc. 146 (3), 2054-2061 (2024).
- Eleutherio, E., Panek, A., De Mesquita, J. F., Trevisol, E., Magalhães, R. Revisiting yeast trehalose metabolism. Curr Genet. 61 (3), 263-274 (2015).
- Attfield, P. V. Trehalose accumulates in Saccharomyces cerevisiae during exposure to agents that induce heat shock response. FEBS Lett. 225 (1-2), 259-263 (1987).
- Marunde, M. R., et al. Improved tolerance to salt and water stress in Drosophila melanogaster cells conferred by late embryogenesis abundant protein. J Insect Physiol. 59 (4), 377-386 (2013).
- Crowe, J. H. Anhydrobiosis: An unsolved problem with applications in human welfare. Subcell Biochem. 71, 263-280 (2015).
- Crowe, J. H., Crowe, L. M., Chapman, D. Preservation of membranes in anhydrobiotic organisms: The role of trehalose. Science. 223 (4637), 701-703 (1984).
- Sampedro, J. G., Rivera-Moran, M. A., Uribe-Carvajal, S. Kramers' theory and the dependence of enzyme dynamics on trehalose-mediated viscosity. Catalysts. 10 (6), 1-19 (2020).
- Sampedro, J. G., Uribe, S. Trehalose-enzyme interactions result in structure stabilization and activity inhibition. The role of viscosity. Mol Cell Biochem. 256 - 257 (1-2), 319-327 (2004).
- Crowe, J. H. Trehalose as a "chemical chaperone": Fact and fantasy. Adv Exp Med Biol. 594, 143-158 (2007).
- Crowe, J. H., Carpenter, J. F., Crowe, L. M., Anchordoguy, T. J. Are freezing and dehydration similar stress vectors? A comparison of modes of interaction of stabilizing solutes with biomolecules. Cryobiology. 27 (3), 219-231 (1990).
- Rivera-Morán, M. A., Sampedro, J. G. Isolation of the sarcoplasmic reticulum Ca2+-ATPase from rabbit fast-twitch muscle. Methods Protoc. 6 (5), 102(2023).
- Páez-Pérez, E. D., De La Cruz-Torres, V., Sampedro, J. G. Nucleotide binding in an engineered recombinant Ca2+-ATPase N-domain. Biochemistry. 55 (49), 6751-6765 (2016).
- Ramírez-Alonso, J. I., Sampedro, J. G. Effect of cations on ATP binding to the N-domain of Na+, K+-ATPase. J Fluoresc. , (2024).
- De la Cruz-Torres, V., Cataño, V., Olivo-Rodríguez, M., Sampedro, J. G. ANS Interacts with the Ca2+-ATPase nucleotide binding site. J Fluoresc. 30 (3), 483-496 (2020).
- Sampedro, J. G., Nájera, H., Uribe-Carvajal, S., Ruiz-Granados, Y. G. Mapping the ATP binding site in the plasma membrane H+-ATPase from Kluyveromyces lactis. J Fluoresc. 24 (6), 1849-1859 (2014).
- Kiani, A. K., et al. Ethical considerations regarding animal experimentation. J Prev Med Hyg. 63 (2 Suppl 3), E255-E266 (2022).
- Ruiz-Granados, Y., De La Cruz-Torres, V., Sampedro, J. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958(2019).
- Wang, S., et al. Structural basis for sarcolipin's regulation of muscle thermogenesis by the sarcoplasmic reticulum Ca2+-ATPase. Sci Adv. 7 (48), eabi7154(2021).
- Champeil, P., et al. ATP regulation of sarcoplasmic reticulum Ca2+-ATPase. Metal-free ATP and 8-bromo-ATP bind with high affinity to the catalytic site of phosphorylated ATPase and accelerate dephosphorylation. J Biol Chem. 263 (25), 12288-12294 (1988).
- Møller, J. V., Olesen, C. Preparation of Ca(2+)-ATPase1a enzyme from rabbit sarcoplasmic reticulum. Methods Mol Biol. 1377, 11-17 (2016).
- Wuytack, F., De Schutter, G., Casteels, R. Partial purification of (Ca2+ + Mg2+)-dependent ATPase from pig smooth muscle and reconstitution of an ATP-dependent Ca2+-transport system. Biochem J. 198 (2), 265-271 (1981).
- Mandal, A., et al. Solubilization, purification and reconstitution of Ca2+-ATPase from bovine pulmonary artery smooth muscle microsomes by different detergents: Preservation of native structure and function of the enzyme by DHPC. Biochim Biophys Acta Gen Subj. 1760 (1), 20-31 (2006).
- Inesi, G., Ma, H., Hua, S., Toyoshima, C. Characterization of Ca2+ ATPase residues involved in substrate and cation binding. Ann NY Acad Sci. 986 (1), 63-71 (2003).
- Van Winkle, W. B., Pitts, B. J., Entman, M. L. Rapid purification of canine cardiac sarcoplasmic reticulum Ca2+-ATPase. J Biol Chem. 253 (24), 8671-8673 (1978).
- Ruiz-Granados, Y. G., De La Cruz-Torres, V., Sampedro, J. G. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958(2019).
- Teramoto, N., Sachinvala, N. D., Shibata, M. Trehalose and trehalose-based polymers for environmentally benign, biocompatible, and bioactive materials. Molecules. 13 (8), 1773-1816 (2008).
- Dao, T. T., et al. Demethoxycurcumin is a potent inhibitor of P-type ATPases from diverse kingdoms of life. PLOS ONE. 11 (9), e0163260(2016).
- Bleeker, N. P., Cornea, R. L., Thomas, D. D., Xing, C. A novel SERCA inhibitor demonstrates synergy with classic SERCA inhibitors and targets multidrug-resistant AML. Mol Pharmaceutics. 10 (11), 4358-4366 (2013).
- Michelangeli, F., East, J. M. A diversity of SERCA Ca2+ pump inhibitors. Biochem Soc Trans. 39 (3), 789-797 (2011).
- Tadini-Buoninsegni, F., Smeazzetto, S., Gualdani, R., Moncelli, M. R. Drug interactions with the Ca2+-ATPase from Sarco(Endo)Plasmic Reticulum (SERCA). Front Mol Biosci. 5, 36(2018).
- Clausen, J. D., et al. Elucidation of antimicrobial activity and mechanism of action by N-substituted carbazole derivatives. Bioorg Med Chem Lett. 27 (19), 4564-4570 (2017).
- Leary, S., et al. AVMA guidelines for the euthanasia of animals: 2020 Edition, Members of the Panel on Euthanasia AVMA Staff Consultants. , AVMA. Schaumburg, IL. (2020).
- de Aluja, A. S. Laboratory animals and official Mexican norms (NOM-062-ZOO-1999). Gac Med Mex. 138 (3), 295-298 (2002).
- Champeil, P., Buschlen-Boucly, S., Bastide, F., Gary-Bobo, C. Sarcoplasmic reticulum ATPase. Spin labeling detection of ligand-induced changes in the relative reactivities of certain sulfhydryl groups. J Biol Chem. 253 (4), 1179-1186 (1978).
- Skalec, A., Janeczek, M., Czerski, A. Anatomy and histology of the rabbit common calcanean tendon. J Vet Med Series C: Anat Histol Embryol. 48 (5), 466-475 (2019).
- Janes, L. E., Mioton, L. M., Fracol, M. E., Ko, J. H. An in vivo comparison: Novel mesh suture versus traditional suture-based repair in a rabbit tendon model. J Hand Surg Glob Online. 4 (1), 32-39 (2022).
- Bollag, D. M., Rozycki, M. D., Edelstein, S. J. Protein Methods. , Wiley. (1996).
- Gagné, F. Tissue preparation and subcellular fractionation techniques. Biochem Ecotoxicol. , 21-31 (2014).
- Lowry, O. H., Rosebrough, N. J., Farr, A. L., Randall, R. J. Protein measurement with the Folin phenol reagent. J Biol Chem. 193 (1), 265-275 (1951).
- Autry, J. M., Rubin, J. E., Svensson, B., Li, J. L., Thomas, D. D. Nucleotide activation of the Ca-ATPase. J Biol Chem. 287 (46), 39070-39082 (2012).
- Winters, D. L., Autry, J. M., Svensson, B., Thomas, D. D. Interdomain fluorescence resonance energy transfer in SERCA probed by cyan-fluorescent protein fused to the actuator domain. Biochemistry. 47 (14), 4246-4256 (2008).
- Miles, A. J., Wallace, B. A. Circular dichroism spectroscopy of membrane proteins. Chem Soc Rev. 14 (45), 1130-1135 (2016).
- Scofano, H. M., Vieyra, A., de Meis, L. Substrate regulation of the sarcoplasmic reticulum ATPase. Transient kinetic studies. J Biol Chem. 254 (20), 10227-10231 (1979).
- Yamamoto, T., Tonomura, Y. Reaction mechanism of the Ca++-dependent ATPase of sarcoplasmic reticulum from skeletal muscle: I. Kinetic studies. J Biochem. 62 (5), 558-575 (1967).
- Murphy, A. J. Affinity labeling of the active site of the Ca2+-ATPase of sarcoplasmic reticulum. Biochim Biophys Acta - Biomembr. 946 (1), 57-65 (1988).
- Csermely, P., Katopis, C., Wallace, B. A., Martonosi, A. The E1→E2 transition of Ca2+-transporting ATPase in sarcoplasmic reticulum occurs without major changes in secondary structure. A circular-dichroism study. Biochem J. 241 (3), 663-669 (1987).
- Abràmoff, M. D., Magalhães, P. J., Ram, S. J. Image processing with ImageJ. Biophotonics Int. 11 (7), 36-42 (2004).
- Scarborough, G. A. The plasma membrane proton-translocating ATPase. Cell Mol Life Sci. 57 (6), 871-883 (2000).
- Llopis, J., McCaffery, J. M., Miyawaki, A., Farquhar, M. G., Tsien, R. Y. Measurement of cytosolic, mitochondrial, and Golgi pH in single living cells with green fluorescent proteins. Proc Natl Acad Sci. 95 (12), 6803-6808 (1998).
- Kanai, R., Vilsen, B., Cornelius, F., Toyoshima, C. Crystal structures of Na+, K+-ATPase reveal the mechanism that converts the K+-bound form to Na+-bound form and opens and closes the cytoplasmic gate. FEBS Lett. 597 (15), 1957-1976 (2023).
- Palmgren, M. P-type ATPases: Many more enigmas left to solve. J Biol Chem. 299 (11), 105352(2023).
- Frauenfelder, H. Ask not what physics can do for biology-Ask what biology can do for physics. Phys Biol. 11 (5), 053004(2014).
- Frauenfelder, H. The physics of proteins. Annu Rev Biophys Biomol Struct. 36, Springer. New York. New York, NY. (2010).
- Narhi, L. O. Biophysics for Therapeutic Protein Development. , Springer Science+Business Media. (2013).
- East, J. M. Purification of a membrane protein (Ca2+/Mg2+-ATPase) and its reconstitution into lipid vesicles. Methods Mol Biol. 27, 87-94 (1994).
- Lee, E. -H., et al. Enhancement of enzyme activity and stability by poly(γ-glutamic acid). Polymer J. 42 (10), 818-822 (2010).
- Norrild, R. K., et al. Increasing protein stability by inferring substitution effects from high-throughput experiments. Cell Rep Methods. 2 (11), 100333(2022).
- Song, Z., Zhang, Q., Wu, W., Pu, Z., Yu, H. Rational design of enzyme activity and enantioselectivity. Front Bioeng Biotechnol. 11, 1-14 (2023).
- Jidenko, M., Lenoir, G., Fuentes, J. M., le Maire, M., Jaxel, C. Expression in yeast and purification of a membrane protein, SERCA1a, using a biotinylated acceptor domain. Protein Expr Purif. 48 (1), 32-42 (2006).
- Hua, S., Ma, H., Lewis, D., Inesi, G., Toyoshima, C. Functional role of "N" (nucleotide) and "P" (phosphorylation) domain interactions in the sarcoplasmic reticulum (SERCA) ATPase. Biochemistry. 41 (7), 2264-2272 (2002).
- Pang, Y. -H., Chen, J. -W. Anisodamine causes the changes of structure and function in the transmembrane domain of the Ca2+-ATPase from sarcoplasmic reticulum. Biosci Biotechnol Biochem. 68 (1), 126-131 (2004).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon