È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Quantificare cronologica Span lievito Life di conseguenza di cellule invecchiate
In questo articolo
Riepilogo
Invecchiamento cronologico di lievito si riferisce alla perdita di vitalità cellulare associato con il tempo in fase stazionaria. Qui si descrivono un elevato throughput metodo per determinare quantitativamente la durata della vita cronologico lievito.
Abstract
Il lievito in erba
Protocollo
Parte 1: Preparazione delle colture invecchiamento
- Ceppi striscia di interesse da parte scorte congelate su piastre di agar YEPD (1 estratto di lievito%, 2% bacto-peptone, 2% agar, 2% di glucosio).
- Incubare le cellule a 30 ˚ C per 48 ore o fino a singole colonie appaiono.
- Scegli singole colonie e inoculare in 5 ml di liquido YEPD (1 estratto di lievito%, 2% bacto-peptone, 2% di glucosio) in provetta.
- Grow culture notte a 30 ˚ C, mantenendo costante agitazione utilizzando uno shaker o Rullo.
- Inoculare 5 ml di sintesi completa (SC), medio (Tabella 1) con 50 microlitri della cultura durante la notte. Generalmente tre culture SC sono preparati per ogni sforzo per assicurare la replica biologico triplice copia delle analisi ciclo di vita per ogni ceppo in esame.
- Mantenere le culture a 30 ˚ C con agitazione costante su un rullo di tamburo per l'intero esperimento (in genere 2 o più settimane).
Parte 2: Facendo una vitalità età-punto
Dopo due giorni di cultura in SC mezzi di comunicazione, le cellule devono essere in fase stazionaria e la prima età punto è pronto per essere preso. Successive età punti dovrebbero essere prese ogni 2-3 giorni per un minimo di due settimane. Per ogni punto di età:
- Preparare il Bioscreen piatti a nido d'ape 100-bene per l'inoculazione, compilando tutti i pozzetti con 145 ml di YEPD. Assicurarsi di lasciare almeno un pozzetto riempito solo YEPD e non le cellule per l'analisi dei dati in seguito.
- Rimuovere le culture invecchiamento dal termostato.
- Mescolare brevemente nel vortex la prima cultura ad essere inoculato nella piastra a nido d'ape, facendo attenzione a non versare della cultura.
- Prelevare 5 ml di cultura mista e pipetta in primo pozzo della piastra a nido d'ape. Fiamma la bocca di ogni provetta prima e dopo la rimozione dei 5 aliquota microlitri.
- Ripetere questa procedura per ogni cultura invecchiamento avendo cura di notare la posizione e corrispondenti a ciascuna cultura. Posizioni identiche bene dovrebbe essere utilizzato per ogni successiva età-point durante l'intero esperimento.
- Sostituire le culture nel 30 ˚ C incubatore al termine con la vaccinazioni.
Parte 3: Caricamento del Bioscreen C macchina MBR
- Esporre il vano incubatore sollevando il coperchio e rimuovere il coperchio per il vassoio campione.
- Inserire la nuova piastra inoculata a nido d'ape nel vassoio del campione (usare lo slot di sinistra se siete solo leggendo una piastra).
- Sostituire il coperchio per il vassoio campione e abbassare il coperchio del vano incubatrice.
- Verificare che il fluido termovettore è superiore al livello minimo di riempimento. Se basso, aggiungere più di 1000 con pipetta microlitri con il fluido termovettore in dotazione.
- Utilizzando il software Bioscreen "EZExperiment", impostare i seguenti parametri per ottenere curve di crescita adatto per Saccharomyces cerevisiae:
- Numero di campioni: Inserire il numero di pozzi con i media o 200
- Filtro: 420-580nm, a banda larga
- Temperatura: 30 ˚ C
- Esperimento Durata: 1 giorno, 0 ore, 0 secondi
- Intervallo di misura: 30 minuti
- Agitazione: On, scuotendo continuo, ad alta
- Fare clic su "Start" per iniziare la lettura.
Parte 4: Analisi dei dati
Solitamente, a sei anni-punti sono presi nel corso di due settimane. L'età punti sono prese a giorni 2, 4, 6, 9, 11 e 13. A seconda del disegno sperimentale e le tensioni in fase di sperimentazione, può essere opportuno tenere età punti più o meno frequentemente o per più di 2 settimane. E 'importante caricare la piastra a nido d'ape nello stesso ordine per ogni età-point in modo tale che ogni cultura invecchiamento corrisponde alla stessa posizione e ad ogni età-punto, perché in questo modo l'analisi dei dati molto più facile.
- Ottenere i file di output dalla macchina Bioscreen C MBR. Il "EZExperiment" software visualizzerà i dati Bioscreen come un file delimitato da tabulazioni che è compatibile con Microsoft Excel e altri software. La prima colonna indica a che ora la misura OD è stata presa durante l'esperimento, con le successive colonne rappresentano ciascun pozzetto nelle piastre Bioscreen a nido d'ape (Figura 1).
- Eliminare la prima lettura OD da ogni colonna. Questa lettura è "rumore".
- Normalizzare i dati sottraendo il valore OD del bene con YEPD solo da tutti i valori di OD in ogni colonna. Questo rimuove l'assorbanza di fondo da parte dei media.
- Tracciare la curva escrescenza. Le curve si sposterà in funzione dell'età (Figura 2). Per esempio, tracciando le curve escrescenza dalla colonna 1 (e 1 del piatto a nido d'ape) oltre i sei punti del tempo, c'è uno spostamento verso destra distinta delle curve nel tempo.
- Calcolare il tempo di raddoppio (δ) per ogni bene in base alla cinetica di crescita del giorno 2 età-point. L'equazione utilizzata per calcolare il tempo di raddoppio is:

dove OD OD 1 e 2 rappresentano successive misurazioni OD e t 1 e t 2 è il tempo tra le misurazioni. Calcola il raddoppio volte solo tra i valori OD di 0,2 a 0,5. La media di questi valori è il tempo di raddoppio per quel bene. Calcola il raddoppio volte solo tra i valori OD di 0,2 a 0,5. La media di questi valori è il tempo di raddoppio per quel bene. La maggior parte dei ceppi wild-type lievito dovrebbe dare un valore tempo di raddoppio tra 85 a 90 minuti. - Per ogni età-punto, calcolare il tempo di spostamento (At) nelle curve escrescenza relativi ai primi anni-punto (giorno 2). Un modo semplice per farlo è quello di determinare la differenza nella lunghezza del tempo impiegato per ogni bene per raggiungere un diametro di 0,3 tra il punto iniziale per età e ogni successiva età-point. Il tempo che un particolare così raggiunto un diametro di 0,3 può essere calcolato con l'equazione di regressione lineare corrispondente alla ln (OD) in funzione del tempo tra le due parentesi OD tempo-punti = 0,3.
- Calcolare la frazione di sopravvissuti ad ogni età-point al fine di generare una curva di sopravvivenza (Figura 3). Definire il punto iniziale per età (giorno 2) di essere al 100% vitalità. Per ogni successivo all'età punto calcolare la percentuale di sopravvivenza con l'equazione:
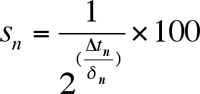
dove s n è la percentuale di sopravvivenza, Dt n è il turno di tempo, e δ n è il tempo di raddoppio. - Generare le curve di sopravvivenza (Figura 3B) come desiderato per ciascun bene (o pozzi replica) tracciando la frazione (o percentuale) di cellule vitali in funzione dell'età.
- Calcolare la sopravvivenza integrale (SI) per ogni bene. SI è definita come l'area sotto la curva di sopravvivenza e può essere stimato con la formula:

in cui l'età è l'età n-punto (per esempio 2, 4, 6, 9, 11 e 13) e s n è il valore di sopravvivenza a quell'età-point. - Determinare parametri statistici di pozzi replicare dai dati SI e la sopravvivenza. Comuni parametri statistici di interesse: media, mediana e varianza per ogni set di replica biologico. Una t-test o analisi simile può essere utilizzato per il confronto a coppie di SI a fini sperimentali e gruppi di controllo. Può anche essere auspicabile per generare le curve di sopravvivenza, come descritto in 4.8 da media biologici replica.
Parte 5: risultati Rappresentante
Al termine dell'esperimento, si dovrà tracciare la curva di sopravvivenza ed eseguito l'analisi dei dati sufficienti per determinare il potenziale di invecchiamento cronologico per diversi ceppi diversi o condizioni. Se eseguita correttamente, la crescita curve ottenute dalla macchina Bioscreen C MBR dovrebbe essere simile a quelli mostrati nella Figura 2 e le curve di sopravvivenza risultante dovrebbe essere simile a quelli mostrati nella figura 3. In generale, wild-type cellule coltivate nelle condizioni descritte qui avrà una durata di vita media per ordine cronologico di 7 giorni. Sostanziale variazione nella sopravvivenza si osserva in diversi ceppi e sotto alcune condizioni, come la crescita in media del glucosio 0,05%, la sopravvivenza mediana può superare i 30 giorni.

Figura 1. Uscita dati dal Bioscreen programma "EZExperiment". Una colonna visualizza l'ora in cui è stata presa una lettura di assorbanza. Colonne successive rappresentano la pozzetti della piastra a nido d'ape inoculati con cellule prelevate da culture invecchiamento.
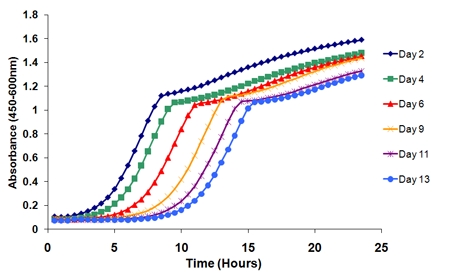
Figura 2. Curve escrescenza da un singolo replicare biologico nel corso di un esperimento. C'è uno spostamento distinto nelle curve nel tempo come le cellule nella cultura invecchiamento perdono vitalità. Il periodo di tempo tra il punto di tempo iniziale (giorno 2) e un punto di tempo successivi determina la vitalità a questa età particolare.
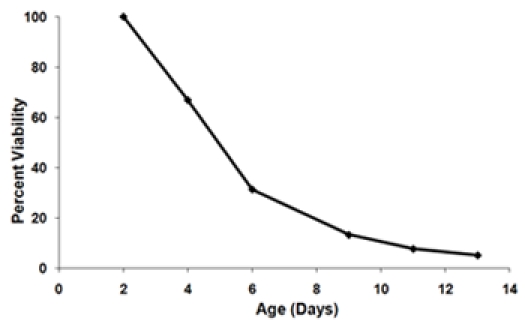
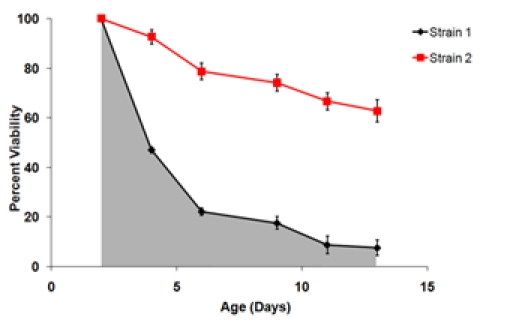
Figura 3. A) Una curva di sopravvivenza generato utilizzando i dati escrescenza dalla Figura 1. I 2 punti di giorno è impostato come punto di vitalità del 100%. B), curve di sopravvivenza finale di due ceppi testati nello stesso esperimento. Queste curve di sopravvivenza Viabilità rappresentano la media di tre biologici repliche per ogni ceppo. Le barre di errore rappresentano la deviazione standard all'interno biologici replica. L'area ombreggiata sotto la curva di sopravvivenza rappresenta la sopravvivenza integrale (SI) per il ceppo 1.
| Tabella 1. Sintetico di media definite utilizzate per cronologica Studi Invecchiamento (ceppo backgrou° BY4743) | |
| Componente | Concentrazione (g / L) |
| D-glucosio | 20 |
| Lievito di base di azoto (-AA/AS) | 1,7 |
| (NH 4) 2 SO 4 | 5.0 |
| Adenina | 0,04 |
| L-Arginina | 0,02 |
| L-acido aspartico | 0,1 |
| L-acido glutammico | 0,1 |
| L-Istidina | 0,1 |
| L-Leucina | 0,3 |
| L-lisina | 0,03 |
| L-metionina | 0,02 |
| L-Fenilalanina | 0,05 |
| L-Serina | 0,375 |
| L-treonina | 0,2 |
| L-triptofano | 0,04 |
| L-tirosina | 0,03 |
| L-Valina | 0,15 |
| Uracile | 0,1 |
Nota: Questa ricetta spiega auxotrophies diploidi BY4743 ceppo. Auxotrophies aminoacido deve essere compensata con l'aggiunta di una concentrazione 5X finale al terreno sintetico completo.
Discussione
L'high-throughput vita saggio arco cronologico qui descritto è un metodo efficace per quantificare il potenziale di invecchiamento di un gran numero di ceppi con elevata accuratezza e precisione. L'anticipo principale di questo metodo rispetto ai metodi classici per stabilire la sopravvivenza contando unità formanti colonie (ad esempio vedi 3) è l'uso di un dispositivo di lettura shaker / incubatore / piastra, come la macchina Bioscreen C MBR di ottenere alta risoluzione curve di crescita a ogn...
Riconoscimenti
Questo lavoro è stato sostenuto da NIH di Grant 1R21AG031965-01A1. MK è una Ellison Medical Foundation Scholar Nuove invecchiamento.
Materiali
| Name | Company | Catalog Number | Comments | |
| Bacto Peptone | Reagent | BD Biosciences | 211677 | |
| Bacto Yeast Extract | Reagent | BD Biosciences | 288620 | |
| Difco Agar | Reagent | BD Biosciences | 214530 | |
| Yeast Nitrogen Base w/o A.A. and A.S. | Reagent | MidSci | J630-500G | |
| Amino Acids | Reagent | Sigma-Aldrich | ||
| Ammonium Sulfate | Reagent | Spectrum | AM185 | |
| Dextrose | Reagent | Fisher Scientific | D16-10 | |
| Bioscreen C MBR machine | Tool | Growth Curves USA | 5101370 | |
| Bioscreen 100-well Honeycomb plate | Tool | Growth Curves USA | 9502550 |
Riferimenti
- Steinkraus, K. A., Kaeberlein, M., Kennedy, B. K. Replicative aging in yeast: the means to the end. Annu Rev Cell Dev Biol. 24, 29 (2008).
- Kaeberlein, M., Conn, P. M. . Handbook of models for human aging. , 109 (2006).
- Fabrizio, P., Longo, V. D. The chronological life span of Saccharomyces cerevisiae. Aging Cell. 2 (2), 73 (2003).
- Murakami, C. J., Burtner, C. R., Kennedy, B. K., Kaeberlein, M. A method for high-throughput quantitative analysis of yeast chronological life span. J Gerontol A Biol Sci Med Sci. 63 (2), 113 (2008).
- Piper, P. W., Harris, N. L., MacLean, M. Preadaptation to efficient respiratory maintenance is essential both for maximal longevity and the retention of replicative potential in chronologically ageing yeast. Mech Ageing Dev. 127 (9), 733 (2006).
- Fabrizio, P., et al. Superoxide is a mediator of an altruistic aging program in Saccharomyces cerevisiae. J Cell Biol. 166 (7), 1055 (2004).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon