È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Generazione robusta di epatociti-come le cellule da popolazioni di cellule staminali embrionali
In questo articolo
Riepilogo
Questo articolo si concentrerà sulla creazione di endoderma epatiche umane da popolazioni di cellule staminali embrionali.
Abstract
Nonostante i progressi nella modellazione tossicità dei farmaci umani, molti composti non durante gli studi clinici per effetti collaterali imprevisti. Il costo degli studi clinici sono notevoli, quindi è essenziale che più schermi tossicologia predittiva sono sviluppati e distribuiti nelle prime fasi di sviluppo dei farmaci (Greenhough et al 2010). Epatociti umani rappresentano il modello attuale gold standard per valutare la tossicità della droga, ma sono una risorsa limitata che presentano la funzione variabile. Pertanto, l'uso di linee cellulari immortalizzate e modelli tessuti animali sono normalmente impiegati per la loro abbondanza. Mentre entrambe le fonti sono informativi, essi sono limitati dalla scarsa funzionalità, la variabilità delle specie e / o instabilità nella cultura (Dalgetty et al 2009). Le cellule staminali pluripotenti (PSC) sono una fonte interessante alternativa di epatociti umani come cellule (HLCS) (Medine et al 2010). PSC sono in grado di rinnovamento del sé e la differenziazione di tutti i tipi di cellule somatiche trovato negli adulti e, quindi, rappresentanouna fonte potenzialmente inesauribile di cellule differenziate. Abbiamo sviluppato una procedura che è semplice, altamente efficiente, suscettibili di automazione e rese funzionali HLCS umano (Hay et al 2008; Fletcher et al 2008; Hannoun et al 2010; Payne et al 2011 e Hay et al 2011). Crediamo che la nostra tecnologia porterà alla produzione scalabile di HLCS per la scoperta, la modellazione della malattia, la costruzione di dispositivi di extra-corporea e, eventualmente, le terapie cellulari a base di trapianto.
Protocollo
1. Preparazione iniziale di tutti gli stock chimica e rivestimento della cultura Plasticware
Tutti i passaggi da effettuare in una cappa di coltura dei tessuti in condizioni asettiche.
- Preparazione delle risorse umane fattore di crescita fibroblastico (hbFGF)
- Preparare soluzione al 10% BSA in PBS e filtrare su un filtro di 0,22 micron.
- Dalla soluzione al 10% di BSA preparare una soluzione 0,2% BSA.
- Aggiungere 10 ml 0,2% di BSA hbFGF mcg solution/100.
- Pre-bagnato di un filtro da 0,22 micron di filtraggio 5 ml di soluzione di BSA al 10% attraverso il filtro. Eliminare i 10 ml BSA lavaggio.
- Filtrare il hbFGF attraverso il pre-lavaggio del filtro.
- Aliquota del hbFGF in eppendorfs sterile e conservare a -20 ° C.
- Preparazione del Activin umani una soluzione madre
- Aggiungere 1 ml di 0,2% di BSA in una siringa pre e bagnare il filtro.
- Diluire il Activin A 0,2% di BSA ad una concentrazione stock di 100 mg / ml.
- Filtrare l'attivitàn Una soluzione e aliquota in eppendorfs sterile, conservare a -20 ° C.
- Preparazione della soluzione del mouse archivi Wnt3a
- Aggiungere 200 ml di PBS a una fiala da 2 mg di Wnt3a ad una concentrazione stock di 10 mcg / ml.
- Aliquota in eppendorfs sterile e conservare a -20 ° C.
- Preparazione di Human soluzione stock HGF (1000X)
- Diluire il HGF in PBS ad una concentrazione stock di 10 mcg / ml.
- Filtrare la soluzione HGF e aliquota in eppendorfs sterile, conservare a -20 ° C.
- Preparazione della soluzione oncostatina stock M (1000X)
- Diluire la OSM in PBS ad una concentrazione stock di 20 mg / ml.
- Filtrare la soluzione OSM e aliquota in eppendorfs sterile, conservare a -20 ° C.
- Rivestimento di cultura Plasticware con Matrigel
- Scongelare il flacone da 10 ml di magazzino Matrigel notte a 4 ° C il ghiaccio e poi aggiungere 10 ml di DMEM-KO. Mescolare bene con pipette refrigerati e 1 negoziomL aliquote a -20 ° C.
- Scongelare una aliquota di Matrigel a 4 ° C per almeno 2 ore o durante la notte per evitare la formazione di un gel.
- Aggiungere 5 mL di freddo KO-DMEM al Matrigel, mescolare bene con una pipetta.
- Portare a 15 ml con il freddo KO-DMEM e mescolare con una pipetta.
- Aggiungi matrigel alla piastra o pallone da rivestire (tabella (i))
| Piastra / Flask | Volume / Bene o Flask |
| 12-pozzetti | 0,5 ml per pozzetto |
| 6 pozzetti | 1 ml per pozzetto |
| 25 centimetri 2 fiasco | 2 ml per bombola |
Tabella 1. Volumi consigliati di matrigel per Plasticware rivestimento tipico della cultura hESC.
- Incubare la piastra rivestita o fiasco notte a 4 ° C o cameratempature per 1 ora prima dell'uso.
- Piatti o recipienti sono stati rivestiti con matrigel possono essere conservati a 4 ° C per un massimo di 1 settimana. Essi devono essere chiaramente etichettati con la data sono stati rivestiti loro. Eliminare le piastre o fiaschi non utilizzata entro una settimana.
- Prima di utilizzare permettono il contenitore culturale rivestito di venire fino a temperatura ambiente all'interno di una cappa coltura di tessuti.
- Immediatamente prima dell'uso aspirare il matrigel e aggiungere la sospensione cellulare al bene o fiasco.
2. Manutenzione ordinaria di culture hESC e la caratterizzazione
- Rianimazione di linee di hESC
- Rimuovere hESC dallo stoccaggio di azoto liquido e scongelare rapidamente in un bagno d'acqua a 37 ° C.
- Trasferire la sospensione cellulare con cura in una provetta sterile contenente alcuni mL di medium caldo.
- Agglomerare le cellule tramite centrifugazione @ bassa velocità per 5 min (1000 RPM).
- Aspirare al largo della resus surnatante e molto delicatamentepend le cellule ES in media calda e piastra su uno strato alimentatore MEF.
- Cellule Refeed quotidianamente con media ES fresca e su subconfluence, le cellule, le cellule richiedono passaging.
- HESC manutenzione ordinaria
Le cellule staminali vengono coltivate in piastre o fiaschi rivestiti con matrigel. Le cellule devono essere esaminati e alimentato ogni giorno:- Esaminare al microscopio per la morfologia delle cellule contaminazione, e confluenza.
- Aspirare il terreno spesi.
- Aggiungere un volume appropriato di fresco embrionali di topo fibroblasto condizionata Media (MEF-CM) + umano bFGF (concentrazione finale 4 ng / ml) o altri supporti i livelli di free 6.
- Cellule Passaging con collagenasi
linee di hESC (H1, H9, RCM-1) raggiungerà confluenza ogni 5-7 giorni dopo passaging ad un rapporto di divisione 1:3. HESC passaggio iniziale in presenza di stroma crescono più lentamente e il tempo sul Matrigel può diventare un fattore importante. CES umano non dovrebbe essere lasciato più di 14giorni sul Matrigel stesso a causa della degradazione della matrice e in questo caso può essere necessario per il passaggio delle cellule con un rapporto 1:1 o 1:2 split se le cellule sono subconfluent.
Enzima Incubazione- Tutti i passaggi da effettuare in una cappa di coltura dei tessuti in condizioni asettiche.
- Assicurarsi che ci sia un nuovo piatto matrigel rivestiti o fiasco preparato conformemente al punto 1.6.
- Decidere il rapporto di divisione desiderato per le cellule. Una serie di fattori sono coinvolti nel decidere il rapporto di divisione:
- Alto livello di stroma con un basso numero di colonie: possono essere diversi passaggi tornare a una dimensione inferiore o possono essere ben diversi passaggi 01:01 che sbarazzarsi di alcuni stroma e quindi aumentare la colonia di rapporto stroma, promuovere la crescita hESC.
- Alto livello di stroma con le colonie di grandi dimensioni, a seconda del numero di colonie, possono essere diversi passaggi 1:1 o 1:2 se ci sono colonie hESC abbastanza grandi.
- Tipico hESC crescita con un po 'stroma o non stroma e / o alcuni di differenziazione possono essere diversi passaggi 1:2 o 3.
- Aspirare il supporto dal bene o fiasco.
- Lavare una volta con 2 ml di PBS (-MgCl 2,-CaCl 2).
- Aggiungere un volume adeguato di collagenasi (200 U / mL diluite in DMEM-KO) e incubare a 37 ° C per 2-5 min. Da 2 minuti in poi esaminare regolarmente al microscopio (intervalli di 1 minuto). Al punto le cellule differenziate cominciano a sollevare e le colonie cominciano a sollevarsi sul bordo le cellule sono pronte per essere diversi passaggi.
- Aspirare la collagenasi.
- Lavare una volta con 2 ml di PBS (-MgCl 2,-CaCl 2).
- Aggiungere un volume adeguato di MEF-CM a seconda del rapporto di divisione, e con un raschietto cellula rimuovere fisicamente le cellule dalla superficie del bene o del matraccio, quindi triturare generazioneTLY pipettando su e giù per 2-3 volte con una pipetta 10 ml. E 'importante che le hESC vengono tenuti in gruppi di cellule e non sono suddivisi in singole cellule.
- Replate la sospensione cellulare risultante sul nuovo matrigel fiaschi rivestiti o pozzi.
- Portare al volume del MEF-CM fino a 4 ml per un pozzo di un 6-pozzetti.
- Quando si posizionano le cellule in incubatore agitare il contenitore di coltura dei tessuti per garantire quanto più possibile una distribuzione di colonie come le colonie tendono a stabilirsi nel centro della piastra di coltura dei tessuti / fiasco interessano replating e la differenziazione cellulare.
- Caratterizzazione di routine delle popolazioni hESC mediante citometria di flusso
- popolazioni hESC vengono esaminati tramite citometria a flusso una volta al mese per i marcatori di cellule staminali come il 3 ottobre / 4, Tra 1-60 e SSE-4.
- hESCpopolazioni sono rimossi dal loro substrato come sospensioni singola cella a seguito di un trattamento di 5 minuti con tripsina / EDTA (Invitrogen).
- Risospendere le cellule singole a 1x10 6 cellule / ml in PBS integrato con 0,1% BSA e sodio azide allo 0,1%.
- Incubare preparazioni cellulari a 4 ° C con gli anticorpi adeguati per 40 minuti.
- Lavare le cellule due volte con PBS integrato con 0,1% BSA e sodio azide allo 0,1% per rimuovere l'anticorpo non legato e risospendere ad un volume finale di 100 l ed analizzare mediante citometria a flusso.
- Dati per 30000-40000 eventi "live" vengono acquisiti per ogni campione con un citometro FACS calibro dotato di un 488-nm laser e analizzati utilizzando il software CellQuest (Becton Dickinson, San Jose, CA). Le cellule non colorate sono inclusi come controlli. Le cellule morte e apoptotiche con detriti sono stati esclusi dalle analisi utilizzando un cancello elettronico in diretta su scatter in avanti e parametri di dispersione laterale.
3. Differentizione di hESC a endoderma epatica
- Preparazione di supporti per la differenziazione delle hESC a endoderma epatica. Preparazione di tutti i media dovrebbero essere effettuate in una cappa di coltura dei tessuti in condizioni asettiche.
- Preparazione di RPMI: B27 medio adescamento per la differenziazione endoderma
- Per RPMI-B27 media, mix RPMI 1640 (500 ml) e B27 (50x, 10 ml)
- Agitare componenti.
- Aggiungi tutti i componenti di una unità filtro e filtro sotto vuoto, conservare a 4 ° C.
- Preparazione di SR-DMSO medio per la differenziazione degli epatociti
- Per SR-DMSO media, mix 80% KO-DMEM, 20% KO-SR, 0,5% di L-glutamina, 1% di aminoacidi non essenziali, 0.1mm β-mercaptoetanolo e 1% di DMSO.
- Filtrare la soluzione sotto vacum, conservare a 4 ° C e aliquota e conservare a -20 ° C se necessario.
- Utilizzare 4 mL per pozzetto di un 6-pozzetti e 6 mL per T25 pallone.
- Preparazione del L15 media maturazione per hepamaturazione tocyte
- Per L-15 di media, mescolare 500 ml Leibovitz L-15 di media, triptosio fosfato brodo (concentrazione finale 8,3%), inattivato con il calore di siero fetale bovino (concentrazione finale 8,3%), 10 mM idrocortisone 21-emisuccinato, 1 mM insulina (pancreas bovino ), 1% di L-glutammina, acido ascorbico 0,2%.
- Filtrare la soluzione sotto vacum, conservare a 4 ° C e aliquota e conservare a -20 ° C se necessario.
- Preparazione della finale RPMI: B27 medio adescamento
- Erogare il volume di terreno necessaria innesco per l'esperimento (1 ml per pozzetto di un 6-pozzetti, e 2 ml per beuta T25).
- Aggiungi Activin A per una concentrazione finale di 100 ng / mL.
- Aggiungi ricombinante Wnt3a ad una concentrazione finale di 50 ng / mL.
- Mescolare bene e il supporto è pronto per l'uso.
- Questo supporto finale deve essere composta da fresco ogni giorno.
- Preparazione di finale L-15 di media maturazione
- Erogare il necessariovolume di L-15 di media per l'esperimento (4 ml per pozzetto di una piastra ben 6, e 6 ml per beuta T25).
- Aggiungi HGF ad una concentrazione finale di 10 ng / ml.
- Aggiungi OSM a una concentrazione finale di 20 ng / mL.
- Mescolare bene e il supporto è pronto per l'uso.
- Questo supporto finale deve essere composta da fresco ogni giorno.
- Preparazione di RPMI: B27 medio adescamento per la differenziazione endoderma
- HESC adescamento di endoderma definitivo
- Cultura hESC (H1, H9, RCM-1) e si propagano su Matrigel piatti rivestiti con fibroblasti embrionali di topo MEF-CM integrato con bFGF.
- Avviare la differenziazione epatica hESC quando raggiungono un livello confluenza di circa il 30% -60% (a seconda della linea hESC) sostituendo il MEF-CM con il mezzo di adescamento (RPMI 1640-B27 integrato con 100 ng / mL Activin A e 50 ng / mL Wnt3a.
- Le cellule sono coltivate in media di adescamento per 3 giorni (cambiando la media ogni 24 ore), e medie adescamento finale con Activin A e Wnt3a è costituito fresco ogni giorno.
- Dopo 72 ore in media di adescamento, il cambiamento medio del mezzo di differenziazione secondo (SR-DMSO) per 5 giorni (cambiando il media ogni 48 ore).
- Al giorno 8 coltura le cellule in media maturazione e di manutenzione (L-15) integrato con 10 ng / mL hHGF e 20 ng / mL OSM per 9 giorni (cambio medio ogni 48 ore). Media maturazione e di manutenzione con hHGF e OSM è composto da fresco ogni giorno.
- Le cellule gradualmente esibiscono cambiamenti morfologici da una forma appuntita / triangolare ad una morfologia caratteristica del fegato mostrare un aspetto poligonale (Figura 2A).
- Schematico per epato-cellulare differenziazione:

4. Caratterizzazione di endoderma epatica derivati dalle hESC
- Immunocolorazione
- Lavare HLCS derivati dalle hESC due volte con PBS, 1 minuto ogni lavaggio.
- Fissare il HLCS con 4% PFA per 20 minuti a temperatura ambiente, (la cellulas può essere conservato in PBS a 4 ° C e colorati in un secondo momento). Le cellule possono anche essere fissati con metanolo ghiacciata per 10 minuti a -20 ° C.
- Lavare le cellule due volte con PBS, a 5 minuti ogni lavaggio.
- Incubare le cellule per 2 minuti a temperatura ambiente con 100% di etanolo per la colorazione nucleare (questo passaggio non è necessario se la fissazione metanolo viene usato).
- Lavare le cellule due volte con PBS, a 5 minuti ogni lavaggio.
- Bloccare le cellule con PBS / T (0,1% tra) / 10% di BSA per 1 ora a temperatura ambiente.
- Rimuovere il tampone di bloccaggio e aggiungere l'anticorpo rispettivi primario diluito in PBS / T (0,1% tra) / 1% BSA e incubare per 2 ore a temperatura ambiente, oppure una notte a 4 ° C con agitazione.
- Lavare le cellule 3 volte con PBS / T (0,1% tra) / 1 BSA% a temperatura ambiente, a 5 minuti ogni lavaggio.
- Aggiungere la secondaria appropriata Alexa Fluor anticorpi (1:400) diluito in PBS per le cellule e incubare a temperatura ambiente per 1 ora al buio con agitazione.
- Lavare le cellule 3 volte con PBS, a 5 minuti ogni lavaggio.
- Montare ogni pozzetto con MOWIOL 4-88 e DAPI (1:1000). Coprire il bene con un vetrino, premendo il coprioggetto delicatamente per assicurare che tutte le bolle d'aria vengono rimossi e conservare a 4 ° C al buio.
- RNA isolamento e l'estrazione
- Lavare i HLCS derivati dalle hESC con PBS ed aspirare.
- Aggiungere 1 ml di reagente TRIZOL ed incubare a temperatura ambiente per 5 minuti.
- Raschiare le cellule e mettere in un eppendorf da 1,5 ml (conservare a -80 ° C per un uso successivo, se necessario).
- Aggiungere 0,5 ml di cloroformio al eppendorf e mescolare invertendo; assicurarsi che questo avviene in una cappa aspirante.
- Centrifugare la soluzione a 13.000 rpm per 15 minuti a 4 ° C.
- Raccogliere lo strato acquoso e posto in una eppendorf pulita, assicurarsi che non vi è alcuna contaminazione dall'interfaccia.
- Aggiungere 1 ml di isopropanolo e mescolare invertendo, lasciare a temperatura ambiente per 10 minuti di precipitazioneTate l'RNA.
- Centrifugare a 13.000 rpm per 10 minuti a 4 ° C.
- Aspirare il surnatante e fare attenzione a non disturbare il pellet di RNA. Lavare con 0,5 mL di etanolo al 70% e lasciare a temperatura ambiente per 5 minuti.
- Centrifugare a 8000 rpm per 5 minuti a 4 ° C.
- Aspirare l'etanolo e lasciare asciugare a temperatura ambiente per 5-10 minuti.
- Una volta che tutti l'etanolo è evaporato, risospendere il pellet in 30 ml di acqua deionizzata. Conservare l'RNA a -80 ° C per un uso successivo.
- Quantificare la concentrazione di RNA usando un NanoDrop.
- PCR di trascrizione inversa
- Impostare una reazione con precedenza isolato RNA (200 ng), esameri casuale, nucleotidi (10mm) della trascrittasi inversa e il buffer rispettivi sottile mL eppendorf parete 0,5.
- Impostare un negativo RT, la reazione sopra senza la trascrittasi inversa.
- Posizionare i tubi in un cycler termico, macchina PCR, e impostare i seguenti programma:
- 37 ° C - 5 minuti (1 ciclo)
- 42 ° C - 1 ora (1 ciclo)
- 95 ° C - 5 minuti (1 ciclo)
- Conservare il cDNA a -20 ° C per usi futuri, se necessario.
- TaqMan quantitativa della transcriptasi inversa reazione a catena della polimerasi Harvest le cellule in diversi momenti durante il protocollo di differenziazione. Estrarre l'RNA ed effettuare qPCR trascrizione inversa utilizzando i seguenti primer e di analisi su richiesta, (Applied Biosystems) protocollo:
- 4 ottobre Hs03005111_g1
- Nanog Hs02387400_g1
- Albumina Hs00910225_m1
- Alfa-fetoproteina Hs00173490_m1
Per l'isolamento dell'RNA e si prega di estrazione riferimento alla sezione 3.3.2
- Trascrizione inversa e TaqMan qPCR
- Prendere 1 microg di RNA e invertire trascrivere a cDNA utilizzando Invitrogen Apice trascrizione inversa IIIkit, come da istruzioni del produttore.
- 1 microlitri del cDNA utilizzato in una reazione di 25 microlitri TaqMan costituito dal primer appropriato da Applied Biosystems, primer di controllo 18S ribosomiale e Invitrogen 2x platino qPCR supermix UDG con rox e un volume adeguato di acqua.
- Mescolare bene e versare 10 ml di ogni campione in due pozzi di entrambi una piastra da 96 o 384 qPCR bene.
- Una volta che tutti i campioni sono stati caricati, oltre i controlli del caso), sigillare il piatto e l'analisi sulla macchina Applied Biosystems 7900HT TaqMan.
- I risultati sono espressi come espressione parente su un campione di controllo.
- Analisi funzionale di Endoderma epatica hESC Derivato citocromo P450 saggi - http://www.promega.com/tbs/tb325/tb325.pdf
- Incubare 17 giorni hESC HE derivati con il substrato specifico per 5 ore a 37 ° C (n = 3). Utilizzare terreni di coltura dei tessuti come uncontrollo negativo e incubare a 37 ° C per 5 ore.
- Raccogliere i surnatanti ed effettuare il test secondo le istruzioni del produttore.
- Misurare i livelli relativi di attività basale e normalizzare per mg di proteina per come determinato con il Test BCA (http://www.piercenet.com/products/browse.cfm?fldID=02020101).
Note
- Tutti i volumi sono basate su un 6-ben formato piatto. Regolare il volume di conseguenza per il piatto richiesto o borraccia.
- Tutti i media adescamento, la differenziazione e maturazione viene filtrato sotto vuoto prima dell'uso.
- Medio di adescamento, la differenziazione e maturazione è conservato a 4 ° C per non più di 2 settimane. Valutare la quantità media è necessario per l'esperimento e aliquota media residua e conservare a -20 ° C per uso futuro.
- Matrigel si compone come da istruzioni del produttore, 1 ml di aliquote possono essere conservati a -20 ° C fino al momento dell'uso.
- Grofattori wth una volta costituito e aliquotati possono essere conservati a -20 ° C e quando scongelato può essere conservato a 4 ° C per non più di 2 settimane.
- L'anticorpo primario di solito utilizzato per caratterizzare HLCS derivati dalle hESC è albumina (1:250, Sigma-Aldrich, Saint Louis, MO).
5. Rappresentante dei risultati:
Caratterizzazione delle hESC mantenuto prima differenziazione epatica
Al fine di caratterizzare lo stato di cellule staminali del H9 hESC utilizzati nello studio abbiamo studiato una serie di parametri. Le cellule esposte morfologia hESC, piccole celle fitto cresce in colonie definiti (Figura 1A) e ha espresso il pluripotenti marcatori genica su cellule staminali, ottobre-3 / 4 e Nanog (Figura 1B). Non abbiamo trovato differenze significative nell'espressione di questi geni rispetto a una linea di controllo hESC H7 positivo. Inoltre, 90,1% della popolazione hESC è stato positivo per il marcatore di cellule staminali SSEA-4 (Figura 1C).
luiSC differenziazione endoderma epatica
Cellule staminali embrionali umane possono essere efficacemente differenziati endoderma epatiche in vitro (Hay et al 2008). Al giorno 9 del differenziamento, le cellule sono state raccolte e la differenziazione delle hESC a HLCS è stata valutata. Come precedentemente riportato la hESC espone una serie di profondi cambiamenti morfologici, e di giorno 9, esposto all'inizio morfologia degli epatociti sviluppare un aspetto poligonale (Figura 2A). Inoltre, down-regulation di ottobre-3 / 4 nel corso 9 giorni il tempo è stato osservato (Figura 2B). Al contrario, il fegato trascrizioni AFP e albumina up-regolati (Figura 2C) dal 7 ° giorno in poi.
Maturazione in vitro di Endoderma epatica
Endoderma epatica è maturato in vitro utilizzando la procedura stabilita (Hay et al 2008). L'ultimo giorno di culture differenziazione sono stati immunostained per i marcatori epatici albumina umana, AFP e la E-caderina. La resa di HLCS utilizzando il nostro procedura è in genere il 90% (Hay et al 2008; Hanoun et al 2010; Payne et al 2011). HLCS macchiato positivo per l'albumina, alfa-fetoproteina ed E-caderina (Figura 3).
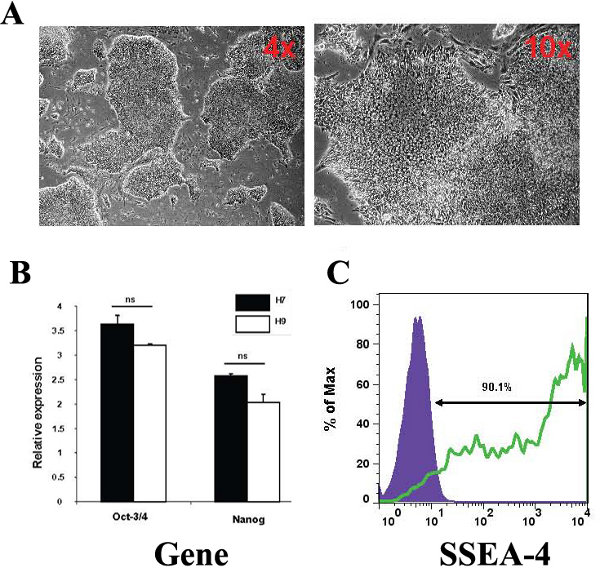
Figura 1. Caratterizzazione delle hESC utilizzato in questo studio. (A) contrasto di fase rappresentante microscopia immagini di morfologia hESC osservata nella cultura con ingrandimento 4x e 10x. Le immagini sono state catturate utilizzando un TE3000 / U Nikon microscopio invertito (B) hESC coltivate sono Octamer (ottobre-3 / 4) positivo positivo e Nanog. H7 hESC erano usare come controllo per i livelli di espressione genica pluripotenza. Espressione relativa si riferisce a pieghe di induzione rispetto al controllo gene endogeno, β-2-microglobulina. (C) appezzamenti FACS mostrano hESC superficie livelli di espressione marcatore, fra cui lo stadio embrionale antigeni specifici SSE 4.
69/2969fig2.jpg "/>
Figura 2. (A) Fase contrasto rappresentante immagini di microscopia di hESC derivati morfologia endoderma epatica al giorno 9 della differenziazione osservato nella cultura, a x4 e ingrandimento x10. (B) Caratterizzazione dei cambiamenti nell'espressione genica. RNA è stato estratto ed il cDNA è stato analizzato dalla reazione a catena della polimerasi quantitativa, mostrando downregulation progressiva di espressione genica su cellule indifferenziate (ottobre-3 / 4) e (C) sovraregolazione dell'espressione genica epatociti (albumina e α-fetoproteina). Espressione relativa si riferisce a pieghe di induzione rispetto al controllo gene endogeno, β-2-microglobulina, al giorno 0 di differenziazione. P <0,05 è denotato * e P <0,001 è denotato *** misurata dagli studenti t-test rispetto alle hESC al giorno 0. Le barre di errore rappresentano 1 deviazione standard.
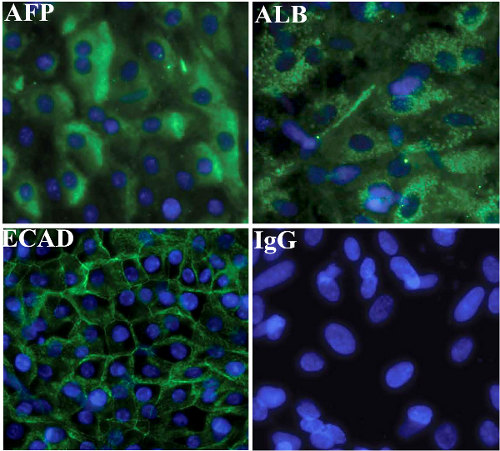
Figura 3. Characterisation di hESC di derivazione epatica endoderma
Immunocitochimica che mostra l'espressione di marcatori di epatociti, albumina, AFP ed E-caderina in hESC (H9) endoderma epatiche derivate. I controlli negativi sono stati effettuati con immunoglobuline G corrispondente (IgG) e rappresentanti sono mostrati immagini.
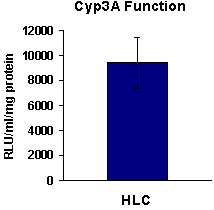
Figura 4. HESC (H9) HLCS derivati mostrano l'attività metabolica. Al giorno 17, H9 differenziate per HLCS esposto l'attività metabolica del CYP3A (n = 6).
Discussione
Abbiamo sviluppato un modello semplice, omogeneo e altamente riproducibili in vitro per generare livelli scalabili di HLCS umano. Il nostro modello è stato convalidato da una serie di laboratori esterni collaborazione. Noi abitualmente caratterizzano HLCS cellule staminali derivate usando il nostro strumento di casa nella casella dei marcatori dello sviluppo e del fegato analisi specifiche funzionali (la maggior parte dei quali sono disponibili in commercio). Le fasi critiche del nostro processo sono: il mante...
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Il dottor Hay è stato sostenuto da una borsa di RCUK, il dottor West è stato sostenuto dal Dipartimento di Chirurgia, il dottor Medine è stata sostenuta da una sovvenzione del Fondo Nucleo BHF, il Sig. Baltasar Lucendo-Villarin è stata sostenuta da un dottorato di ricerca MRC Studenship. Il dottor Zhou è stato sostenuto da una borsa di studio dal governo cinese.
Materiali
Lastre di verniciatura Matrigel e fiaschi
- Matrigel (10 ml, BD Biosciences, UK); conservare a -20 ° C.
- KO-DMEM (500 ml, Gibco, Invitrogen, UK); conservare a 4 ° C.
- Piastre di coltura tissutale (6 bene, ben 12, Corning, UK)
- Tessuto fiasco cultura (25 cm2 ventilati, Corning, UK)
hESC manutenzione
- Topo di medie fibroblasti embrionali condizionata (MEF-CM) (100 ml, R & D Systems, USA); conservare a -20 ° C.
- BSA soluzione (50 ml, Sigma Aldrich, UK); conservare a 4 ° C.
- Umano fondamentale fattore di crescita dei fibroblasti (100 mcg, Peprotech, USA); conservare a -20 ° C.
HESC Passaging con collagenasi
- Ben confluenti o fiasco di hESC.
- Matrigel pozzi rivestiti o fiaschi a seconda dei casi.
- Fosfato salina tamponata (-MgCl 2,-CaCl 2) (500 ml, Gibco, Invitrogen, UK); conservare a temperatura ambiente.
- Collagenasi IV (1 g, Gibco, InvitrOgen, UK); conservare a 4 ° C.
- Topo di medie fibroblasti embrionali condizionata (MEF-CM) (100 ml, R & D Systems, USA).
- Umano fondamentale fattore di crescita dei fibroblasti (100 mcg, Peprotech, USA).
Differenziazione delle hESC a endoderma epatica
- RPMI 1640 (500 ml, Gibco, Invitrogen, UK); conservare a 4 ° C.
- Supplemento B27 (10 ml, Gibco, Invitrogen, UK); conservare a -20 ° C.
- Activin A (2 mg, Peprotech, USA); conservare a -20 ° C.
- Ricombinante Wnt3a del mouse (2 mg, R & D Systems, USA); conservare a -20 ° C.
- KO-DMEM (500 ml, Gibco, Invitrogen, UK); conservare a 4 ° C.
- KO-SR (500 ml, Gibco, Invitrogen, UK); conservare a -20 ° C.
- Aminoacidi non essenziali (100 ml, Gibco, Invitrogen, UK); conservare a 4 ° C.
- β-mercaptoetanolo (10 ml, Gibco, Invitrogen, UK); conservare a 4 ° C.
- DMSO (Sigma Aldrich, UK); conservare a temperatura ambiente
- Leibovitz L-15 terreno di coltura (500 ml, SIGMuno Aldrich, UK); conservare a 4 ° C.
- Triptosio fosfato brodo (100 ml, Sigma Aldrich, UK); conservare a 4 ° C.
- Siero fetale bovino, inattivato con il calore (500 ml, Gibco, Invitrogen, UK); conservare a -20 ° C.
- Idrocortisone 21-emisuccinato (100 mg, Sigma Aldrich, UK); conservare a -20 ° C.
- Insulina (pancreas bovino) (100 mg, Sigma Aldrich, UK); conservare a -20 ° C.
- L-Glutammina (100 ml, Gibco, Invitrogen, UK); conservare a -20 ° C.
- Acido ascorbico (25 g, Sigma Aldrich, UK); conservare a -20 ° C.
- Umano HGF (10 mcg, Peprotech, USA); conservare a -20 ° C.
- Ricombinante umana oncostatina M (OSM) (50 mcg, R & D Systems, USA); conservare a -20 ° C.
- Siringa guidato unità filtro 0,22 micron (Millipore, UK)
Caratterizzazione di Endoderma epatica derivati dalle hESC
Immunocolorazione
- Tampone fosfato salino (-MgCl 2,-CaCl 2) (500 ml, Gibco, Invitrogen, UK); STOre a temperatura ambiente.
- PBST, PBS composto con 0,1% di Tween 20 (Sigma-Aldrich, UK).
- Paraformaldeide (PFA) (Sigma-Aldrich, UK) è costituito in PBS, conservare a -20 ° C.
- Glicerolo (Sigma-Aldrich, UK), conservare a temperatura ambiente.
- Tris base (Sigma-Aldrich, UK), conservare a temperatura ambiente.
- Etanolo
- Siero (ABD Serotech, UK), conservare a -20 ° C.
- Anticorpo secondario, Fluorofori Alexa (Molecular Probes, Invitrogen, UK).
- MOWIOL 4-88 (Polysciences Inc, USA) è realizzata in Tris HCL e glicerolo come da istruzioni del produttore. DAPI (Pierce, Thermo Fisher Scientific, UK) è aggiunto alla soluzione MOWIOL a una diluizione 1:1000.
| Name | Company | Catalog Number | Comments |
| Anticorpi primari | |||
| Antigen * | Tipo | Fornitore | Diluizione |
| ALB | Monoclonali del mouse | Sigma Aldrich | 1 / 500 |
| E-caderina | Monoclonali del mouse | Millipore | 1 / 100 |
| α-fetoproteina | Monoclonali del mouse | Sigma | 1 / 500 |
| SSE-4 FITC | Monoclonali del mouse | Biolegend | 1 / 100 |
| IgG | Monoclonali del mouse | DAKO | 1 / 500 |
| Anticorpi secondari | |||
| Anti-topo coniugato FITC | Capra monoclonali | Invitrogen | 1 / 400 |
Tabella 2. Gli anticorpi utilizzati per immunocolorazione epatiche derivate endoderma hESC, le concentrazioni utilizzate, la specie ha sviluppato nel e le società che essi siano acquistati.
Analisi funzionale di Endoderma epatica e Normalisation (per mg di proteina)
saggi citocromo P450
- p4-GLO CYP3A4, CYP1A2, Kit e luminometro (Promega, USA).
- Bianco fondo piatto piastra da 96 dosaggio bene (BD Biosciences, UK).
- BCA Assay Kit (Pierce, Thermo Fisher Scientific, UK).
- Trasparente piastra da 96 dosaggio bene (Iwaki, UK)
Riferimenti
- Asgari, S., Pournasr, B., Salekdeh, G. H., Ghodsizadeh, A., Ott, M., Baharvand, H. Induced pluripotent stem cells: a new era for hepatology. J. Hepatol. 53, 738-751 (2010).
- Hay, D. C., Pernagallo, S., Diaz-Mochon, J. J., Medine, C. N., Greenhough, S., Hannoun, Z., Schrader, J., Black, J. R., Fletcher, J., Dalgetty, D. Unbiased Screening of Polymer Libraries to Define Novel Substrates for Functional Hepatocytes with Inducible Drug Metabolism. Stem Cell Research. 6, 92-101 (2011).
- Payne, C. M., Samuel, K., Pryde, A., King, J., Brownstein, D., Schrader, J., Medine, C. N., Forbes, S. J., Iredale, J. P., Newsome, P. N. Persistence of Functional Hepatocyte Like Cells in Immune Compromised Mice. Liver International. 31, 254-262 (2011).
- Greenhough, S., Medine, C., Hay, D. C. Pluripotent Stem Cell Derived Hepatocyte Like Cells and their Potential in Toxicity Screening. Toxicology. 278, 250-255 (2010).
- Medine, C. N., Greenhough, S., Hay, D. C. The Role of Stem Cell Derived Hepatic Endoderm in Human Drug Discovery. Biochemical Society Transactions. 38, 1033-1036 (2010).
- Hannoun, Z., Fletcher, J., Greenhough, S., Medine, C. N., Samuel, K., Sharma, R., Pryde, A., Black, J. R., Ross, J. A., Wilmut, I., Iredale, J. P., Hay, D. C. The Comparison between Conditioned Media and Serum Free Media in Human Embryonic Stem Cell Culture and Differentiation. Cellular Reprogramming. 12, 133-140 (2010).
- Dalgetty, D. M., Medine, C., Iredale, J. P., Hay, D. C. Progress and Future Challenges in Stem Cell-Derived Liver Technologies. American Journal of Physiology - Gastrointestinal and Liver Physiology. 297, 241-248 (2009).
- Hay, D. C., Fletcher, J., Payne, C., Terrace, J. D., Gallagher, R. C. J., Snoeys, J., Black, J., Wojtacha, D., Samuel, K., Hannoun, Z., Pryde, A. Highly Efficient Differentiation of hESCs to Functional Hepatic Endoderm Requires ActivinA and Wnt3a Signalling. Proceedings of the National Academy of Sciences. 105, 12301-12306 (2008).
- Fletcher, J., Cui, W., Samuels, K., Black, J. R., Currie, I. S., Terrace, J. D., Payne, C., Filippi, C., Newsome, P., Forbes, S. J., Ross, J. A., Iredale, J. P., Hay, D. C. The Inhibitory Role of Stromal Cell Mesenchyme on Human Embryonic Stem Cell Hepatocyte Differentiation is Overcome by Wnt3a Treatment. Cloning and Stem Cells. 10, 331-340 (2008).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon