É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Geração robusto de Hepatócito-like Células de populações humanas células-tronco embrionárias
Neste Artigo
Resumo
Este artigo incidirá sobre a geração de endoderme hepática humana de populações humanas de células-tronco embrionárias.
Resumo
Apesar dos progressos na modelagem de toxicidade dos medicamentos humanos, muitos compostos falhar durante os ensaios clínicos devido a efeitos colaterais imprevisíveis. O custo de estudos clínicos são substanciais, por isso, é essencial que mais telas de toxicologia preditiva são desenvolvidos e implantados no início do desenvolvimento de drogas (Greenhough et al 2010). Hepatócitos humanos representam o modelo atual do ouro padrão para avaliar a toxicidade dos medicamentos, mas são um recurso limitado que exibem função variável. Portanto, o uso de linhagens de células imortalizadas e modelos de tecido animal são rotineiramente empregados devido à sua abundância. Enquanto ambas as fontes são informativos, eles são limitados pela função pobres, a variabilidade das espécies e / ou instabilidade na cultura (Dalgetty et al 2009). Células-tronco pluripotentes (PSC) são uma importante fonte alternativa de hepatócitos humanos como células (HLCS) (Medine et al 2010). PSCs são capazes de auto-renovação e diferenciação de todos os tipos de células somáticas encontrada no adulto e, portanto, representamuma fonte potencialmente inesgotável de células diferenciadas. Temos desenvolvido um procedimento que é simples, altamente eficiente, passível de automação e rendimento funcional HLCS humano (Hay et al 2008; Fletcher et al 2008; Hannoun et al 2010; Payne et al 2011 e Hay et al 2011). Acreditamos que nossa tecnologia irá levar à produção escalonável de HLCS para descoberta de drogas, modelagem de doença, a construção de extra-corpórea dispositivos e, possivelmente, terapias celulares transplante base.
Protocolo
1. Preparação inicial de todas as ações químicas e revestimento da cultura plasticware
Todos os passos a serem realizadas em uma capa de cultura de tecidos sob condições assépticas.
- Preparação de fator de crescimento básico de fibroblastos humanos (hbFGF)
- Prepare solução de 10% BSA em PBS e filtrar através de um filtro de 0,22 mM.
- A partir da solução BSA 10% preparar uma solução de 0,2% BSA.
- Adicionar 10 mL de 0,2% BSA hbFGF mg solution/100.
- Pré-molhada 0,22 mM um filtro por filtragem 5 mL de solução BSA 10% através do filtro. Descartar a 10 mL BSA lavagem.
- Filtrar o hbFGF através do filtro pré-lavado.
- HbFGF alíquota do em eppendorfs estéreis e armazenar a -20 ° C.
- Preparação de Activin Humanos uma solução estoque
- Adicionar 1 mL de BSA 0,2% para uma seringa pré-filtro e piso molhado.
- Diluir o Activin A em BSA 0,2% a uma concentração de ações de 100 mcg / mL.
- Filtrar o Activin A solução e alíquota em eppendorfs estéreis, armazenar a -20 ° C.
- Preparação de rato Solução stock Wnt3a
- Adicionar 200 mL de PBS para um frasco mg 2 de Wnt3a a uma concentração de ações de 10 mcg / mL.
- Alíquota em eppendorfs estéreis e armazenar a -20 ° C.
- Preparação da solução Humanos stock HGF (1000X)
- Diluir o HGF em PBS a uma concentração de ações de 10 mcg / mL.
- Filtrar a solução HGF e da alíquota em eppendorfs estéreis, armazenar a -20 ° C.
- Preparação de Oncostatina Solução stock M (1000X)
- Diluir o OSM em PBS a uma concentração de ações de 20 mcg / mL.
- Filtrar a solução OSM e da alíquota em eppendorfs estéreis, armazenar a -20 ° C.
- Revestimento da cultura plasticware com Matrigel
- Descongele os 10 garrafa de ações mL de Matrigel overnight a 4 ° C em gelo e em seguida, adicione 10 mL de DMEM-KO. Misture bem, usando pipetas refrigerados e armazenar umaalíquotas mL a -20 ° C.
- Descongelar uma alíquota de Matrigel a 4 ° C por pelo menos 2 horas ou durante a noite para evitar a formação de um gel.
- Adicionar 5 mL de frio KO-DMEM à Matrigel, misture bem com uma pipeta.
- Compõem a 15 mL de frio KO-DMEM e misture com uma pipeta.
- Adicionar matrigel à placa ou frasco a ser revestida (Tabela (i))
| Placa / Flask | Volume / Bem ou Flask |
| 12 bem-placa | 0,5 mL por poço |
| 6 bem-placa | 1 mL por poço |
| 25 centímetros balão 2 | 2 mL por frasco |
Tabela 1. Volumes recomendados para o revestimento de Matrigel plasticware típico para a cultura CTEh.
- Incubar a placa revestida ou frasco overnight a 4 ° C ou salatempature por 1 hora antes de usar.
- Placas ou frascos que foram revestidas com Matrigel pode ser armazenado a 4 ° C por até 1 semana. Eles devem ser claramente identificados com a data em que foram revestidos. Descartar qualquer placas ou frascos não utilizados dentro de uma semana.
- Antes de usar permitem que o container cultura revestido para chegar à temperatura ambiente dentro de uma capa de cultura de tecidos.
- Imediatamente antes da utilização aspirar o Matrigel e adicione a suspensão de células para o bem ou frasco.
2. Manutenção de rotina de culturas CTEh e caracterização
- Ressuscitação de linhas CTEh
- Remover hESCs de armazenamento de nitrogênio líquido e rapidamente descongelar em banho-maria a 37 ° C.
- Transferir a suspensão celular com cuidado para um tubo estéril contendo várias mL de meio morno.
- Agregar as células por centrifugação @ baixa velocidade por 5min (1000 RPM).
- Aspirar fora da reanimação sobrenadante e muito gentilmentepend as células em meio ES quente e placa para fora em uma camada de alimentador de MEF.
- Células Refeed diária com o meio fresco ES e sobre subconfluence, as células, as células necessitam de passaging.
- Manutenção de rotina CTEh
Células-tronco embrionárias são cultivadas em placas ou frascos revestidos com Matrigel. As células precisam ser examinados e alimentados diariamente:- Examinar sob o microscópio de contaminação a morfologia das células, e confluência.
- Aspirar o meio gasto.
- Adicionar um volume adequado de produtos frescos do rato Médio Fibroblasto-condicionado embrionárias (MEF-CM) + bFGF humano (concentração final 4 ng / mL) ou outros meios de comunicação livres de soro 6.
- Células Passaging com colagenase
linhas hESC (H1, H9 e RCM-1) chegará a confluência cada 5-7 dias após passaging em razão de separação 1:3. HESCs passagem no início da presença de estroma crescem mais lentamente eo tempo sobre o Matrigel pode se tornar um fator importante. CES humano não deve ser deixado mais de 14dias na matrigel mesmo devido à degradação da matriz e, neste caso, pode ser necessário para a passagem das células em uma razão de separação 1:1 ou 1:2, se as células são confluentes.
A incubação da enzima- Todos os passos a serem realizadas em uma capa de cultura de tecidos sob condições assépticas.
- Garantir que há um prato novo matrigel revestidos ou frasco preparado conforme a secção 1.6.
- Decidir sobre a razão de separação desejada para as células. Uma série de fatores estão envolvidos na decisão sobre a taxa de divisão:
- Alto nível de estroma com baixo número de colônias: pode ser várias passagens de volta para um tamanho menor ou bem pode ser várias passagens 01:01 que irá se livrar de algumas estroma e, assim, aumentar a colônia à relação estroma, promovendo o crescimento CTEh.
- Alto nível de estroma com grandes colônias, dependendo do número de colônias, várias passagens podem ser 1:1 ou 1:2 se existem colônias CTEh grande o suficiente.
- Típico hESCs crescendo com um pouco de stroma ou nenhum estroma e / ou alguma diferenciação podem ser várias passagens 1:2 ou 3.
- Aspirar a mídia do bem ou frasco.
- Lave uma vez com 2 ml de PBS (-MgCl 2, CaCl-2).
- Adicionar um volume adequado de colagenase (200 U / mL diluído em KO-DMEM) e incubar a 37 ° C por 2-5 min. De 2 minutos em diante examinar regularmente sob o microscópio (intervalos de 1 minuto). No ponto de as células diferenciadas começam a decolar e as colônias começam a levantar na borda as células estão prontas para serem várias passagens.
- Aspirar a colagenase.
- Lave uma vez com 2 ml de PBS (-MgCl 2, CaCl-2).
- Adicionar um volume adequado de MEF-CM, dependendo da razão de separação, e com uma espátula de células fisicamente remover as células da superfície do poço ou frasco, em seguida, triturar genTLY pipetando cima e para baixo 2-3 vezes usando uma pipeta de 10 mL. É importante que o hESCs são mantidos em grupos de células e não estão dividido em células individuais.
- Refaça a suspensão de células, resultando em novos matrigel os frascos revestidos ou poços.
- Fazer o volume de MEF-CM até 4 mL para um poço de uma placa de 6 poços.
- Ao colocar as pilhas no agitam incubadora do recipiente de cultura de tecidos para assegurar, até mesmo como uma possível distribuição de colônias como as colônias tendem a se estabelecer no centro da placa de cultura de tecidos / frasco afetando repique e diferenciação celular.
- Caracterização de rotina das populações CTEh por citometria de fluxo
- populações CTEh são examinadas através de citometria de fluxo, uma vez por mês para marcadores de células-tronco, tais como 03 de outubro / 4, 10-60 e SSEA Tra-4.
- CTEhpopulações são removidas de suas suspensões substrato como única célula após um tratamento cinco minutos com tripsina / EDTA (Invitrogen).
- Ressuspender as células individuais de 1x10 6 células / mL em PBS suplementado com BSA 0,1% e azida sódica a 0,1%.
- Incubar preparações de células a 4 ° C com os anticorpos apropriados para 40 minutos.
- Lave as células duas vezes com PBS suplementado com BSA 0,1% e 0,1% de azida de sódio para remover os anticorpos não ligados e ressuspender a um volume final de 100 mL e analisar por citometria de fluxo.
- Dados para 30.000-40.000 "ao vivo" eventos são adquiridos para cada amostra usando um citômetro Caliber FACS equipado com um laser de 488 nm e analisados usando CellQuest software (Becton Dickinson, San Jose, CA). Células não coradas são incluídos como controles. Células mortas e apoptóticas juntamente com detritos foram excluídos da análise usando um portão eletrônico ao vivo na dispersão para a frente e parâmetros de dispersão lateral.
3. Diferenciadoção de hESCs a endoderme hepática
- Preparação de meios para a diferenciação de hESCs a endoderme hepática. Toda a preparação de mídia deve ser realizada em uma capa de cultura de tecidos sob condições assépticas.
- Preparação de RPMI: B27 médio priming para a diferenciação endoderme
- Para RPMI-B27 média, misture RPMI 1640 (500 mL) e B27 (50x, 10 mL)
- Agite para misturar componentes.
- Adicione todos os componentes para uma unidade de filtro e filtro em loja de vácuo, a 4 ° C.
- Preparação de SR-DMSO médio para a diferenciação dos hepatócitos
- Para SR-DMSO média, misture 80% KO-DMEM, 20% KO-SR, 0,5% L-glutamina, 1% não-aminoácidos essenciais, 0,1 mM β-mercaptoetanol e DMSO 1%.
- Filtrar a solução em vacum, armazenar a 4 ° C e alíquotas e armazenar a -20 ° C, se necessário.
- Use 4 mL por poço de uma placa de 6-bem, e 6 ml por frasco T25.
- Preparação de L15 meio de maturação para hepamaturação tocyte
- Para L-15 médio, misture 500 mL Leibovitz L-15 médio, triptose fosfato de caldo (concentração final 8,3%), o calor de soro fetal bovino inativado (concentração final 8,3%), hidrocortisona M 10 21-hemissuccinato, 1 mM de insulina (pâncreas bovino ), 1% L-Glutamina, ácido ascórbico 0,2%.
- Filtrar a solução em vacum, armazenar a 4 ° C e alíquotas e armazenar a -20 ° C, se necessário.
- Preparação de RPMI final: B27 médio priming
- Dispense o volume necessário de médio priming para o experimento (1 mL por poço de uma placa de 6-bem, e 2 mL por frasco T25).
- Adicionar Activin A a uma concentração final de 100 ng / mL.
- Adicionar recombinante Wnt3a a uma concentração final de 50 ng / mL.
- Misture bem e os meios de comunicação já está pronto para uso.
- Esta mídia final deve ser feita até fresco todos os dias.
- Preparação de meio de maturação final, L-15
- Dispensar a exigidavolume de meio L-15 para o experimento (4 mL por poço de uma placa de 6-bem, e 6 ml por frasco T25).
- Adicionar HGF para uma concentração final de 10 ng / mL.
- Adicionar OSM para uma concentração final de 20 ng / mL.
- Misture bem e os meios de comunicação já está pronto para uso.
- Esta mídia final deve ser feita até fresco todos os dias.
- Preparação de RPMI: B27 médio priming para a diferenciação endoderme
- HESCs Preparação para endoderme definitiva
- Cultura hESCs (H1, H9 e RCM-1) e propagar em Matrigel placas revestidas com fibroblastos de rato embrionárias MEF CM-suplementado com bFGF.
- Iniciar a diferenciação hepática quando hESCs atingir um nível confluência de aproximadamente 30% -60% (dependendo da linha CTEh), substituindo o MEF CM-priming com o meio (RPMI 1640-B27 suplementada com 100 ng / mL Activin A e 50 ng / mL Wnt3a.
- As células são cultivadas em meio priming por 3 dias (mudando o meio a cada 24 horas), e média final com priming Activin A e Wnt3a é composto fresco todos os dias.
- Após 72 horas em meio de priming, mudar o meio para o meio de diferenciação segundo (SR-DMSO) durante 5 dias (mudando o meio a cada 48 horas).
- No dia 8 cultura as células em meio de maturação e manutenção (L-15) suplementado com 10 ng / mL hHGF e 20 ng / mL OSM por 9 dias (mudança média a cada 48 horas). Maturação e manutenção médio com hHGF e OSM é composto fresco todos os dias.
- Gradualmente as células apresentam alterações morfológicas de uma forma spiky / triangular para uma morfologia do fígado característica exibindo uma aparência poligonal (Figura 2A).
- Esquema de hepato-celular diferenciação:

4. Caracterização da CTEh endoderme hepática derivada
- Imunocoloração
- Lavar HLCS CTEh derivados com PBS duas vezes, um minuto cada lavagem.
- Corrigir o HLCS com PFA 4% por 20 minutos em temperatura ambiente, (a células podem ser armazenados em PBS a 4 ° C e coradas em uma data posterior). As células podem também ser fixados com metanol gelada por 10 minutos a -20 ° C.
- Lave as células duas vezes com PBS, 5 minutos cada lavagem.
- Incubar as células durante 2 minutos à temperatura ambiente com etanol a 100% para a coloração nuclear (este passo não é necessário se a fixação do metanol é usado).
- Lave as células duas vezes com PBS, 5 minutos cada lavagem.
- Impedir as células com PBS / T (0,1% tween) / BSA 10% por 1 hora em temperatura ambiente.
- Remova o tampão de bloqueio e adicionar o anticorpo primário respectivos diluído em PBS / T (0,1% tween) / BSA 1% e incubar por 2 horas em temperatura ambiente, ou durante a noite a 4 ° C com agitação.
- Lave as células 3 vezes com PBS / T (0,1% tween) / 1% BSA em temperatura ambiente, a 5 minutos cada lavagem.
- Adicione o secundário apropriado Alexa Fluor anticorpos (1:400) diluído em PBS para as células e incubar à temperatura ambiente por uma hora no escuro com agitação.
- Lave as células 3 vezes com PBS, 5 minutos cada lavagem.
- Monte cada poço com MOWIOL 4-88 e DAPI (1:1000). Cobrir o poço com uma lamínula, pressionando a lamínula com cuidado para assegurar que todas as bolhas de ar são removidos e armazenar a 4 ° C no escuro.
- RNA de isolamento e extração
- Lave o HLCS CTEh derivados com PBS e aspirado.
- Adicionar 1 mL de reagente Trizol e incubar à temperatura ambiente por 5 minutos.
- Raspe as células e coloque em um eppendorf de 1,5 mL (armazenar a -80 ° C para uso posterior se necessário).
- Adicionar 0,5 mL de clorofórmio para o eppendorf e misturar por inversão, certifique-se isto é feito em uma capela.
- Centrifugar a solução a 13.000 RPM por 15 minutos a 4 ° C.
- Recolher a fase aquosa e coloque em um eppendorf limpo, certifique-se não há contaminação da interface.
- Adicionar 1 mL de isopropanol e misturar por inversão, deixe em temperatura ambiente por 10 minutos para precipitaçãotate a RNA.
- Centrifugar a 13.000 RPM por 10 minutos a 4 ° C.
- Aspirar o sobrenadante e certificar-se de não perturbar o pellet de RNA. Lave com 0,5 mL de etanol 70% e deixar à temperatura ambiente por 5 minutos.
- Centrifugar a 8.000 RPM por 5 minutos a 4 ° C.
- Aspirar o etanol e deixar secar à temperatura ambiente por 5-10 minutos.
- Uma vez que todo o etanol tenha evaporado, ressuspender o sedimento em 30 mL de água desionizada. Armazenar o RNA a -80 ° C para uso posterior.
- Quantificar a concentração de RNA usando um nanodrop.
- Transcrição reversa PCR
- Configurar uma reação usando anteriormente RNA isolado (200 ng), hexâmeros aleatórios, os nucleotídeos (10mm) da transcriptase reversa e os respectivos tampão em uma parede fina eppendorf de 0,5 mL.
- Criar um RT negativo, a reação acima, sem a transcriptase reversa.
- Coloque os tubos em um ciclador térmico, máquina de PCR, e configurar o seguinte pROGRAMA:
- 37 ° C - 5 minutos (1 ciclo)
- 42 ° C - 1 hora (1 ciclo)
- 95 ° C - 5 minutos (1 ciclo)
- Armazenar o cDNA a -20 ° C para uso posterior se necessário.
- TaqMan reverse transcriptase polimerase quantitativa Colheita reação em cadeia das células em diferentes momentos ao longo do protocolo de diferenciação. Extrair o RNA e realizar qPCR transcrição reversa utilizando os seguintes primers e Ensaio sobre a procura, (Applied Biosystems) protocolo:
- 04 de outubro Hs03005111_g1
- Nanog Hs02387400_g1
- Hs00910225_m1 albumina
- Alfa-fetoproteína Hs00173490_m1
RNA para isolamento e extração por favor consulte a seção 3.3.2
- Transcrição reversa e TaqMan qPCR
- Tomar 1 mg de RNA e transcrição reversa para cDNA usando Sobrescrito Invitrogen da transcrição reversa IIIkit, conforme instruções do fabricante.
- 1 ml do cDNA usada em uma reação TaqMan 25 mL que consiste na primers apropriados da Applied Biosystems, primers 18S ribosomal controle e 2x Invitrogen de platina qPCR Supermix UDG com rox e um volume adequado de água.
- Misture bem e coloque 10 ml de cada amostra em dois poços de qualquer uma placa qPCR 96 ou 384 também.
- Uma vez que todas as amostras são carregados, além de os controles adequados), selar a placa e analisar sobre a Applied Biosystems 7900HT máquina TaqMan.
- Os resultados são expressos como expressão relativa sobre uma amostra de controlo.
- Análise funcional da CTEh endoderma hepática Derivado do citocromo P450 Ensaios - http://www.promega.com/tbs/tb325/tb325.pdf
- Incubar 17 dias CTEh derivados HE com o substrato específico para 5 horas a 37 ° C (n = 3). Use meios de cultura de tecidos como umcontrole negativo e incubar a 37 ° C por 5 horas.
- Recolher o sobrenadante e realizar o ensaio de acordo com as instruções do fabricante.
- Medir os níveis relativos de atividade basal e normalizar a proteína por mg, conforme determinado pelo Assay BCA (http://www.piercenet.com/products/browse.cfm?fldID=02020101).
Notas
- Todos os volumes são baseados em um formato de placa de 6 poços. Ajustar os volumes de acordo para a placa exigida ou frasco.
- Todos médio diferenciação priming, e maturação é filtrado sob vácuo antes do uso.
- Médio diferenciação priming, e maturação é armazenada a 4 ° C por não mais de duas semanas. Avaliar o quanto é necessário para o meio da experiência e da alíquota da mídia restantes e armazenar a -20 ° C para uso futuro.
- Matrigel é composta de acordo com as instruções do fabricante; 1 mL alíquotas podem ser armazenadas a -20 ° C até o uso.
- Grofatores wth uma vez constituída e alíquotas podem ser armazenadas a -20 ° C e quando descongeladas podem ser armazenadas a 4 ° C por não mais de duas semanas.
- O anticorpo primário geralmente usado para caracterizar HLCS CTEh derivados é a albumina (1:250, Sigma-Aldrich, Saint Louis, MO).
5. Resultados representativos:
Caracterização da hESCs mantida antes da diferenciação hepática
A fim de caracterizar o estado de células-tronco da H9 hESCs utilizados no estudo estudamos uma série de parâmetros. As células apresentaram morfologia CTEh, pequenas células hermeticamente embalados em crescimento em colônias definidos (Figura 1A) e expressou a célula-tronco pluripotentes de marcadores de genes, Oct-3 / 4 e Nanog (Figura 1B). Não encontramos diferenças significativas na expressão destes genes em comparação com um controle de linha CTEh H7 positivo. Além disso, 90,1% da população hESCs foi positivo para o marcador de células-tronco SSEA-4 (Figura 1C).
eleSC diferenciação para endoderme hepática
Células estaminais embrionárias humanas podem ser eficientemente diferenciados para endoderme hepática in vitro (Hay et al 2008). No dia 9 de diferenciação, as células foram colhidas e diferenciação de CTEh para HLCS foi avaliada. Como relatado anteriormente o hESCs exibiu uma série de profundas mudanças morfológicas, e de dia 9, exibiram morfologia dos hepatócitos cedo a desenvolver uma aparência poligonal (Figura 2A). Além disso, downregulation de outubro-3 / 4 ao longo do tempo 9 dias foi observada (Figura 2B). Em contraste, no fígado transcrições AFP e albumina foram up-regulada (Figura 2C) de dia 7 em diante.
Maturação in vitro de endoderma hepática
Endoderme hepática foi amadurecido in vitro utilizando o procedimento estabelecido (Hay et al 2008). No último dia de culturas diferenciação histoquímica para marcadores hepáticos da albumina humana, AFP e E-caderina. O rendimento de HLCS usando nosso procedimento é geralmente 90% (Hay et al 2008; Hanoun et al 2010; Payne et al 2011). HLCS marcação positiva para albumina, alfa-fetoproteína e caderina-E (Figura 3).
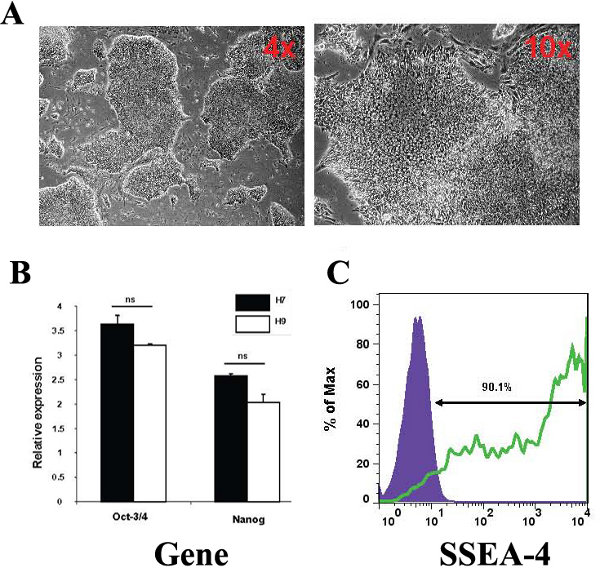
Figura 1. Caracterização da hESCs utilizados neste estudo. (A) de contraste de fase microscopia representante imagens da morfologia observadas na cultura CTEh com ampliação de 4x e 10x. As imagens foram capturadas usando um microscópio Nikon TE3000 / U invertido (B) hESCs cultivadas são octâmero (Oct-3 / 4) positiva positiva e Nanog. H7 hESCs foram usar como um controle para níveis de expressão do gene pluripotência. Expressão relativa refere-se a dobra de indução comparados com o controle do gene endógeno, β-2-microglobulina. (C) parcelas FACS mostrar CTEh superfície níveis de marcadores de expressão, incluindo estágio específico antígenos embrionários SSEA 4.
69/2969fig2.jpg "/>
Figura 2. (A) de contraste de fase microscopia de imagens representativas de CTEh derivados morfologia endoderme hepática no dia 9 de diferenciação observada na cultura, no x4 e ampliação x10. (B) Caracterização das alterações na expressão gênica. RNA foi extraído eo cDNA foi analisada pela reação em cadeia da polimerase quantitativa, mostrando downregulation progressiva de células indiferenciadas de expressão de genes (Oct-3 / 4) e (C) Upregulation da expressão gênica dos hepatócitos (albumina e α-fetoproteína). Expressão relativa refere-se a dobra de indução comparados com o controle do gene endógeno, β-2 microglobulina no dia 0 de diferenciação. P <0,05 é denotada * e P <0,001 é denotada *** medido pelos alunos t-teste em comparação com hESCs no dia 0. Barras de erro representam um desvio padrão.
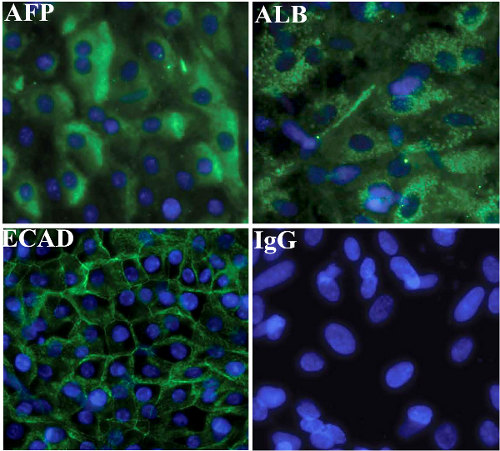
Figura 3. Characterisation de CTEh derivados endoderme hepática
Imunocitoquímica mostrando a expressão de marcadores de hepatócitos, albumina, AFP e E-caderina em hESC (H9) endoderme hepática derivadas. Controles negativos foram realizados com os correspondentes de imunoglobulina G (IgG) e representantes imagens são mostradas.
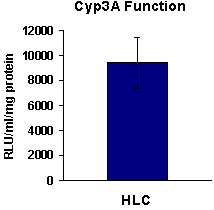
Figura 4. HESC (H9) HLCS derivados exibem atividade metabólica. No dia 17, H9 diferenciadas para HLCS exibiu CYP3A atividade metabólica (n = 6).
Discussão
Nós desenvolvemos um modelo simples, homogêneo e altamente reprodutível in vitro para gerar níveis escalável de HLCS humana. Nosso modelo foi validado por uma série de laboratórios colaboradores externos. Rotineiramente caracterizar HLCS com células-tronco derivadas usando a nossa caixa de ferramentas na casa de marcadores específicos de desenvolvimento e testes funcionais do fígado (a maioria dos quais estão disponíveis comercialmente). As fases críticas em nosso processo são: a manutenção da p...
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Dr Hay foi apoiado por uma bolsa RCUK, o Dr. West foi apoiado pelo Departamento de Cirurgia, Dr. Medine foi apoiado por uma subvenção do Fundo Núcleo BHF, o Sr. Baltasar Lucendo-Villarin foi apoiado por um PhD MRC Studenship. Dr. Zhou foi apoiado por uma bolsa do governo chinês.
Materiais
Placas de revestimento Matrigel e frascos
- Matrigel (10 mL, BD Biosciences, UK); armazenar a -20 ° C.
- KO-DMEM (500 mL, Gibco, Invitrogen, UK); armazenar a 4 ° C.
- Placas de cultura de tecido (6 assim, 12 bem, Corning, UK)
- Frasco de cultura de tecidos (25 cm2 ventiladas, Corning, UK)
Manutenção CTEh
- Rato médio de fibroblastos embrionários condicionado (MEF-CM) (100 mL, R & D Systems, EUA); armazenar a -20 ° C.
- BSA solução (50 mL, Sigma Aldrich, UK); armazenar a 4 ° C.
- Fator de crescimento básico de fibroblastos humanos (100 mg, Peprotech, EUA); armazenar a -20 ° C.
HESCs Passaging com colagenase
- Bem confluentes ou frasco de hESCs.
- Matrigel poços revestidos ou frascos conforme o caso.
- Fosfato salina tamponada (-MgCl 2, CaCl-2) (500 mL, Gibco, Invitrogen, UK); armazenar à temperatura ambiente.
- Colagenase IV (1 g, Gibco, InvitrOgen, UK); armazenar a 4 ° C.
- Rato médio de fibroblastos embrionários condicionado (MEF-CM) (100 mL, R & D Systems, EUA).
- Fator de crescimento básico de fibroblastos humanos (100 mg, Peprotech, EUA).
Diferenciação de hESCs a endoderme hepática
- RPMI 1640 (500 mL, Gibco, Invitrogen, UK); armazenar a 4 ° C.
- Suplemento B27 (10 mL, Gibco, Invitrogen, UK); armazenar a -20 ° C.
- Ativina A (2 mg, Peprotech, EUA); armazenar a -20 ° C.
- Recombinante Wnt3a mouse (2 mg, R & D Systems, EUA); armazenar a -20 ° C.
- KO-DMEM (500 mL, Gibco, Invitrogen, UK); armazenar a 4 ° C.
- KO-SR (500 mL, Gibco, Invitrogen, UK); armazenar a -20 ° C.
- Não-aminoácidos essenciais (100 mL, Gibco, Invitrogen, UK); armazenar a 4 ° C.
- β-mercaptoetanol (10 mL, Gibco, Invitrogen, UK); armazenar a 4 ° C.
- DMSO (Sigma Aldrich, UK); armazenar à temperatura ambiente
- Leibovitz L-15 meio de cultura (500 mL, Sigmuma Aldrich, UK); armazenar a 4 ° C.
- Triptose fosfato de caldo de carne (100 mL, Sigma Aldrich, UK); armazenar a 4 ° C.
- Fetal de soro bovino, inactivado pelo calor (500 mL, Gibco, Invitrogen, UK); armazenar a -20 ° C.
- Armazenar a -20 ° C.; hidrocortisona 21 hemissuccinato (100 mg, Sigma Aldrich, UK)
- Insulina (pâncreas bovino) (100 mg, Sigma Aldrich, UK); armazenar a -20 ° C.
- L-Glutamina (100 mL, Gibco, Invitrogen, UK); armazenar a -20 ° C.
- O ácido ascórbico (25 g, Sigma Aldrich, UK); armazenar a -20 ° C.
- Humana HGF (10 mg, Peprotech, EUA); armazenar a -20 ° C.
- Recombinante humana Oncostatina M (OSM) (50 mg, R & D Systems, EUA); armazenar a -20 ° C.
- Seringa de unidade orientado filtro de 0,22 mM (Millipore, UK)
Caracterização da CTEh endoderma hepática derivada
Imunocoloração
- Tampão fosfato salino (-MgCl 2, CaCl-2) (500 mL, Gibco, Invitrogen, UK); store à temperatura ambiente.
- PBST, PBS feita com 0,1% de Tween 20 (Sigma-Aldrich, UK).
- Paraformaldeído (PFA) (Sigma-Aldrich, UK) é composto, em PBS, armazenar -20 ° C.
- Glicerol (Sigma-Aldrich, UK), armazenar em temperatura ambiente.
- Tris Base (Sigma-Aldrich, UK), armazenar em temperatura ambiente.
- Etanol
- Soro (ABD Serotech, UK), armazenar a -20 ° C.
- Anticorpo secundário, fluoróforos Alexa (Molecular Probes, Invitrogen, UK).
- MOWIOL 4-88 (Polysciences Inc, EUA) é composta de Tris HCL e glicerol como por instruções dos fabricantes. DAPI (Pierce, Thermo Fisher Scientific, UK) é adicionado à solução MOWIOL na diluição de 1:1000.
| Name | Company | Catalog Number | Comments |
| Anticorpos primários | |||
| * Antígeno | Tipo | Fornecedor | Diluição |
| ALB | Monoclonal de camundongo | Sigma Aldrich | 05/0100 |
| E-caderina | Monoclonal de camundongo | Millipore | 1 / 100 |
| α-fetoproteína | Monoclonal de camundongo | Sigma | 1 / 500 |
| SSEA-4 FITC | Monoclonal de camundongo | Biolegend | 1 / 100 |
| IgG | Monoclonal de camundongo | DAKO | 1 / 500 |
| Anticorpos secundários | |||
| Anti-camundongo conjugado FITC | Cabra Monoclonal | Invitrogen | 1 / 400 |
Tabela 2. Os anticorpos utilizados para CTEh imunocoloração derivados hepática endoderme, as concentrações utilizadas, a espécie desenvolveu e as empresas que são comprados a partir de.
Análise funcional do endoderma hepática e normalização (por mg de proteína)
citocromo P450 Ensaios
- p4-GLO CYP3A4, CYP1A2, Kits e luminômetro (Promega, EUA).
- Branco flat bottom 96 placa de ensaio bem (BD Biosciences, UK).
- BCA Assay Kit (Pierce, Thermo Fisher Scientific, UK).
- 96 placa transparente de ensaio bem (IWAKI, UK)
Referências
- Asgari, S., Pournasr, B., Salekdeh, G. H., Ghodsizadeh, A., Ott, M., Baharvand, H. Induced pluripotent stem cells: a new era for hepatology. J. Hepatol. 53, 738-751 (2010).
- Hay, D. C., Pernagallo, S., Diaz-Mochon, J. J., Medine, C. N., Greenhough, S., Hannoun, Z., Schrader, J., Black, J. R., Fletcher, J., Dalgetty, D. Unbiased Screening of Polymer Libraries to Define Novel Substrates for Functional Hepatocytes with Inducible Drug Metabolism. Stem Cell Research. 6, 92-101 (2011).
- Payne, C. M., Samuel, K., Pryde, A., King, J., Brownstein, D., Schrader, J., Medine, C. N., Forbes, S. J., Iredale, J. P., Newsome, P. N. Persistence of Functional Hepatocyte Like Cells in Immune Compromised Mice. Liver International. 31, 254-262 (2011).
- Greenhough, S., Medine, C., Hay, D. C. Pluripotent Stem Cell Derived Hepatocyte Like Cells and their Potential in Toxicity Screening. Toxicology. 278, 250-255 (2010).
- Medine, C. N., Greenhough, S., Hay, D. C. The Role of Stem Cell Derived Hepatic Endoderm in Human Drug Discovery. Biochemical Society Transactions. 38, 1033-1036 (2010).
- Hannoun, Z., Fletcher, J., Greenhough, S., Medine, C. N., Samuel, K., Sharma, R., Pryde, A., Black, J. R., Ross, J. A., Wilmut, I., Iredale, J. P., Hay, D. C. The Comparison between Conditioned Media and Serum Free Media in Human Embryonic Stem Cell Culture and Differentiation. Cellular Reprogramming. 12, 133-140 (2010).
- Dalgetty, D. M., Medine, C., Iredale, J. P., Hay, D. C. Progress and Future Challenges in Stem Cell-Derived Liver Technologies. American Journal of Physiology - Gastrointestinal and Liver Physiology. 297, 241-248 (2009).
- Hay, D. C., Fletcher, J., Payne, C., Terrace, J. D., Gallagher, R. C. J., Snoeys, J., Black, J., Wojtacha, D., Samuel, K., Hannoun, Z., Pryde, A. Highly Efficient Differentiation of hESCs to Functional Hepatic Endoderm Requires ActivinA and Wnt3a Signalling. Proceedings of the National Academy of Sciences. 105, 12301-12306 (2008).
- Fletcher, J., Cui, W., Samuels, K., Black, J. R., Currie, I. S., Terrace, J. D., Payne, C., Filippi, C., Newsome, P., Forbes, S. J., Ross, J. A., Iredale, J. P., Hay, D. C. The Inhibitory Role of Stromal Cell Mesenchyme on Human Embryonic Stem Cell Hepatocyte Differentiation is Overcome by Wnt3a Treatment. Cloning and Stem Cells. 10, 331-340 (2008).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados