Method Article
Protocolli Preparazione del campione, Imaging, e d'analisi per Knife-edge Microscopia Scansione
In questo articolo
Erratum Notice
Riepilogo
L'intero processo, dalla preparazione del campione di cervello di serie di immagini utilizzando il sezionamento Knife-Edge microscopio a scansione, alla visualizzazione dei dati e l'analisi è descritta. Questa tecnica è attualmente utilizzata per acquisire dati cervello di topo, ma è applicabile ad altri organi, altre specie.
Abstract
Importanti progressi nella high-throughput ad alta risoluzione, tecniche di microscopia 3D hanno permesso l'acquisizione di grandi volumi di dati neuroanatomici a risoluzione submicrometer. Uno dei primi strumenti quali la produzione di tutto il cervello scala dei dati è il Knife-Edge microscopio a scansione (KESM) 7, 5, 9, sviluppato e ospitato nel laboratorio degli autori '. KESM è stato utilizzato per sezione e cervelli del mouse intera immagine a una risoluzione submicrometer, rivelando gli intricati dettagli delle reti neuronali (Golgi) 1, 4, 8, reti vascolari (inchiostro di china) 1, 4, e il corpo di distribuzione delle cellule (Nissl) 3 . L'uso di KESM non è limitato al mouse né il cervello. Abbiamo ripreso con successo il cervello polpo 6, polmone del mouse e cervello di ratto. Attualmente stiamo lavorando su tutto embrioni di pesce zebra. Dati come questi possono contribuire ampiamente a connectomics ricerca 10; per la microcircolazione e la ricerca emodinamica, e alla ricerca stereologia da disposizioniding un esatto terra-verità.
In questo articolo descriveremo il gasdotto, compresa la preparazione del campione (fissaggio, colorazione, and Embedding), la configurazione e setup KESM, sezionamento e di imaging con la KESM, elaborazione delle immagini, la preparazione dei dati e visualizzazione dei dati e analisi. L'accento sarà posto sulla preparazione dei campioni e la visualizzazione / analisi dei dati KESM ottenuti. Ci aspettiamo che il protocollo dettagliato presentato in questo articolo per contribuire ad ampliare l'accesso alle KESM e aumentare il suo utilizzo.
Protocollo
1. Preparazione del campione: Golgi-Cox
- Questo protocollo segue da vicino il protocollo di Mayerich et al. 7.
- Il mouse C57BL/6J è profondamente anestetizzato in anestesia isoflurano inalanti e poi decapitato.
- Il cervello è rimosso e collocato in una Golgi-Cox soluzione di fissaggio (1% cromato di potassio, bicromato di potassio 1% e 1% di cloruro di mercurio in acqua deionizzata).
- Il cervello è lasciato nel Golgi-Cox soluzione al buio, a temperatura ambiente, da 10 a 16 settimane.
- I cervelli vengono risciacquati in acqua deionizzata una notte al buio.
- Il cervello risciacquata è immerso in una soluzione di idrossido di ammonio al 5% in acqua deionizzata per 7 a 10 giorni al buio a temperatura ambiente. Questa volta una lunga preparazione è stato quello di garantire l'infiltrazione di tutto il cervello, in modo che il precipitato nero avuto la possibilità di formare completamente nel tessuto.
- Il cervello è nuovamente sciacquati con acqua deionizzata a temperatura ambiente per 4 ore e tgallina disidratati attraverso una serie graduata di alcool etilico: 50% e il 70% in frigorifero (4 ° C), e 85%, 95% (3 modifiche), e del 100% (da 4 a 5 cambi) a temperatura ambiente. Il cervello è lasciato in ogni soluzione per 24 ore.
- Il cervello disidratato viene poi messo in acetone (3 a 4 cambiamenti, ognuno per un giorno), seguita da un araldite-acetone con Araldite-to-acetone rapporto di 1:2, 1:1, 2:1, e infine in 100 Araldite% (3 modifiche, di volta in volta durante la notte), in frigorifero (4 ° C).
- Infine, il cervello trattata è incorporato nel 100% Araldite e riscaldata a 60 ° C per 3 giorni (embedding protocollo cfr. in Abbott e Sotelo 2).
Se i campioni sono incorporati in LR bianco poi gli esemplari sono trasferiti dal l'ultima soluzione di etanolo al 100% attraverso tre cambi di soluzione LR bianchi che vengono ogni conservato in frigorifero per una notte, che è una procedura standard prima di polimerizzazione. Tre cambiamenti sono fatto per garantire l'infiltrazione completa. Il campione è tgallina trasferito al fresco LR Bianca che è polimerizzato in un contenitore chiuso a 60 ° C per 24 ore, che è la quantità di tempo e temperatura corretta per la polimerizzazione. - Fig. 1 mostra una Golgi-Cox cervello colorato incorporato in Araldite.
- Il campione e curata è poi montato sul ring campione di metallo con resina epossidica, e ai lati del blocco tagliato, se necessario (vedi fig. 2).
2. Preparazione del campione: Nissl
- Il mouse è profondamente anestetizzati con ketamina e xilazina (1,7 mg di ketamina / xylazina 0,26 mg per 20 grammi di peso corporeo) iniettato per via intraperitoneale e poi perfusi transcardially con 50 ml della temperatura ambiente PBS (pH 7,4), seguita da 250 ml di stanza temperatura 10% formalina tamponata neutra (pH 7,4). e, infine, con 3,0 cc di puro inchiostro di china.
- Perfusione del corpo intero con soluzione salina e fissativo è necessaria per cancellare il sangue dal sistema cardiovascolare e per fissare il ti UESTIONI. Perfusione con inchiostro di china è necessario riempire completamente la vascolarizzazione del sistema cardiovascolare.
- Il cervello risultante viene poi disidratati attraverso una serie di graduata alcoli etilico (25% -100%) e poi incorporato in plastica araldite seguendo il protocollo di 1,8-1,9 sopra. Se LR bianco è il mezzo di inclusione poi il protocollo di 1,10 come indicato precedentemente.
4. Preparazione del campione: specie generico, organi generico
- Per il caso generico, la fissazione può essere fatta sia con il 10% neutra paraformaldeide tamponata% formalina o 4.
- Per i volumi di tessuto di piccole, la diffusione invece di perfusione è consigliato per la fissazione e colorazione. Nel caso di piccoli organismi o organi, la colorazione può anche essere fatta durante il processo di disidratazione.
- Il protocollo embedding è la stessa di sopra, anche se i tempi per l'infiltrazione soluzione può essere ridotto per organi o organismi che sono molto più piccoli di cervello di topo intero.
- Spegnere la pompa, sono avvicendati sul palco, accendere fotocamera, accendere illuminatore.
- Sollevare coltello e obiettivo.
- Installare l'anello campione.
- Misura dimensione del campione e creare un file di configurazione per l'applicazione Fase KESM Controller2.
- Inizio KESM fase Controller2 e inizializzare palco.
- Inferiore coltello / obiettivo, accendere la pompa.
- Messa a fuoco obiettivi e macchina fotografica, ruotando la manopola di messa a fuoco sul treno ottico e regolazione campo visivo della telecamera di vista rispetto al bordo coltello.
- Premere sul [Vai] per avviare l'imaging.
- Vedi fig. 3-8. Vedi 9, 7 per i dettagli tecnici.
6. Elaborazione delle immagini e la preparazione dei dati
- Copiare lo stack eseguibile KESM processore nella cartella dati nella cartella file di configurazione (00000, 00001, ecc.)
- Eseguire il processore Stack KESM per rimuovere artefatti di illuminazione e normalizzare l'intensità di fondo in tutte lele immagini. Ripetere l'operazione per tutte le sottocartelle dei dati.
- Preparare piastrelle multiscala dal set di dati e caricare e configurare i file nel server web cervello KESM Atlas.
- Vedi fig. 9.
7. Dati di visualizzazione e analisi
- Visualizzazione utilizzando l'Atlante Cervello KESM. Vai a http://kesm.cs.tamu.edu e selezionare l'atlante appropriato.
- Come navigare nelle mappe di Google.
- Zoom avanti e indietro come nelle mappe di Google.
- Per passare a una diversa profondità, selezionare la profondità di andare ad ogni passo dal menu a discesa, quindi fare clic su entrambi [+] o [-] per aumentare o diminuire la profondità z.
- Regolare il numero di sovrapposizione utilizzando il menu a discesa.
- Regolare l'intervallo di sovrapposizione utilizzando il menu a discesa.
- Vedi fig. 11.
- Visualizzazione utilizzando MeVisLab.
- Lancio MeVisLab.
- Creare un nuovo progetto.
- In seguito, nuovi moduli possono essere la creazione di Typing nel nome del modulo nella casella dei comandi.
- Creare modulo [Compose3Dfrom2DFiles], e andare alla cartella desiderata. Inserire la stringa di file e fare clic in [GetFileList]. Assicurarsi che le immagini selezionate può andare bene nella memoria. Fare clic su [Create3D] per creare volume.
- Creare modulo [SetWorldMatrix] e impostare la "Scala" sotto "Elementare Transforms" x, y, z per la dimensione appropriata voxel (0,6, 0,7, 1,0 per un obiettivo 10X 0.45NA). Link [Compose3Dfrom2DFiles] a [SetWorldMatrix].
- Impostare Matrix e trasformate in "Composizione Matrix" a "Ignora", "Ignora", "Avanti".
- Creare modulo [Arithmetic1] e impostare la "funzione" a "Inversione (Max-Img)".
Link [SetWorldMatrix] a [Arithmetic1]. - Creare modulo [View3D] e collegare [Arithmetic1].
- In View3D, mentre il pulsante destro del mouse è premuto, spostate il mouse per regolare la soglia. È possibile ritagliare le regioni, orientamento cambiamento (muovere il mouse mentre il tasto sinistro del mouse è premuto), l'illuminazione cambia (renderin il volumego MIP), accendere / spegnere sfondo, ecc
- Vedi fig. 10.
8. Rappresentante dei risultati:
Qui vi presentiamo tutto il cervello di dati e dettagli. Fichi. 12-15 intero spettacolo-cervello India Ink, Golgi, e Nissl set di dati 1, 4, 3.

Figura 1. Cervello di topo fisso, macchiato, ed embedded

Figura 2. Embedded campione cervello di topo montate sul ring campione.

Figura 3. Knife Edge Microscopio Scansione (KESM). Una foto del KESM è mostrato con i suoi principali componenti segnato: (1) linea ad alta velocità di scansione fotocamera, (2) obiettivo microscopio, (3) assemblaggio coltello di diamante e collimatore di luce, (4) specimen serbatoio (per l'imaging di immersione in acqua), (5) a tre assi di precisione aria-cuscinetto stadio, (6) a luce bianca illuminatore microscopio, (7) pompa dell'acqua (nella parte posteriore) per il prelievo di tessuti sezionati, (8) PC server per il controllo del palco e acquisizione delle immagini (9), base in granito, e (10) ponte di granito.
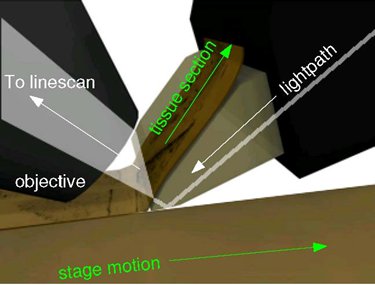
Figura 4. Imaging principi della KESM. Il principio di funzionamento di KESM è illustrata. L'obiettivo e il coltello è tenuto sul posto, mentre il campione apposto sul palco si muove il posizionamento (freccia con linea continua) alla risoluzione di 20 nm e velocità di traslazione di 1-5, ed ottiene raschiato contro il coltello di diamante (5 mm di larghezza per l'obiettivo 10X), generando una sezione sottile che scorre sopra il coltello (freccia con linea continua). Line-scansione di immagini è fatto vicino la punta del coltello.

Figura 5. KESM fotocamera e obiettivo controlli messa a fuoco.

Figura 6. Assemblaggio coltello KESM e controlli.

Figura 7. Iniziali di messa a fuoco attraverso la porta di osservazione.
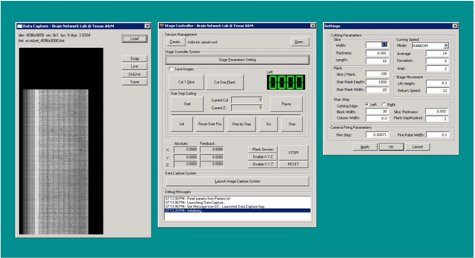
Figura 8. Controller KESM Fase 2 applicazione (screenshot).
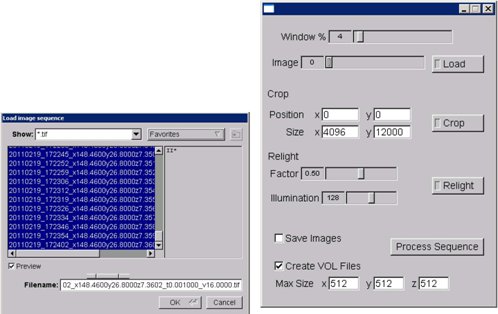
Figura 9. Stack di applicazione KESM processore (screenshot).
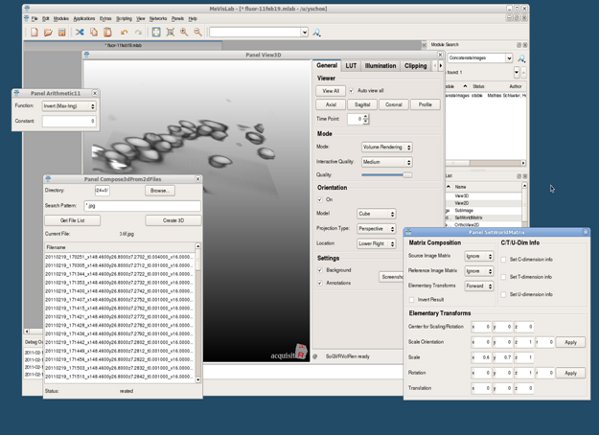
Figura 10. MeVisLab applicazione (screenshot).
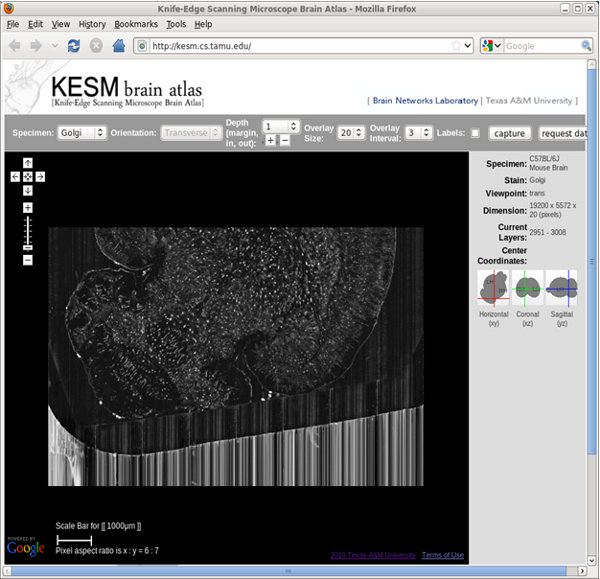
Figura 11 KESM Cervello Atlas:. Interfaccia Web (screenshot).
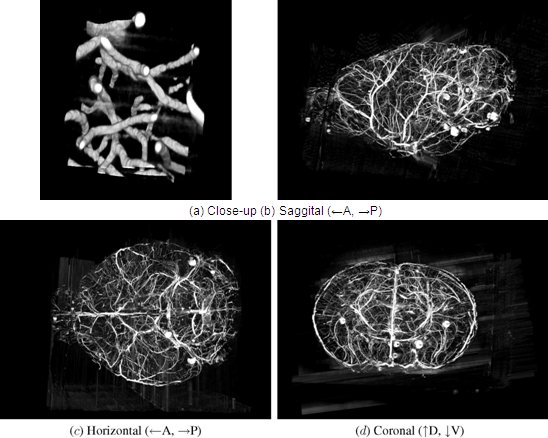
Figura 12. KESM tutto il cervello dati inchiostro di china. Visualizzazione volume di dati KESM pile sono indicati per il set di dati vascolare. (A) primo piano i dati vascolare. Larghezza ~ 100 um. (Bd) tre viste standard del cervello intero sistema vascolare del mouse (sottocampionate di dati ad alta risoluzione). Larghezza ~ 10mm.

Figura 13. KESM intero-cervello di topo dati Golgi.
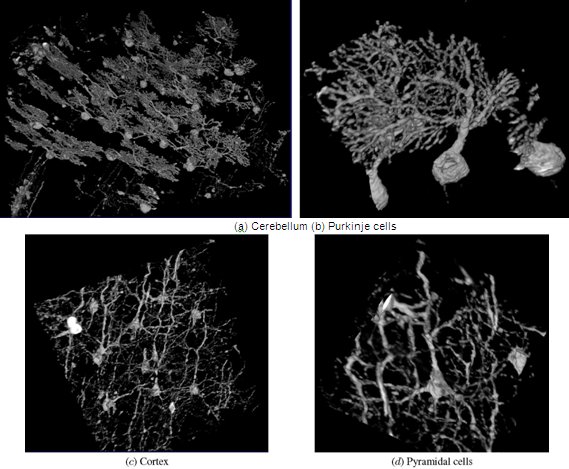
Figura 14. KESM Golgi dettagli dei dati.
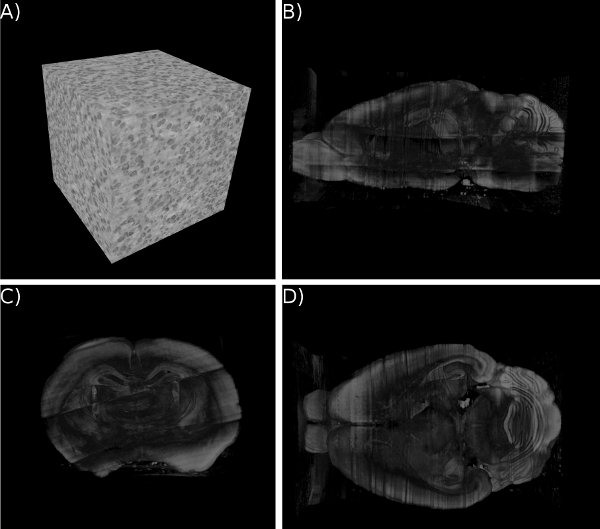
Figura 15. KESM dati Nissl tutto il cervello. (A) primo piano del Ndati ISSL. ~ 300 um 3. (Bd) tre viste standard di tutti i dati del mouse cervello Nissl (sottocampionate di dati ad alta risoluzione). Sezione mostrato per una chiara visione delle strutture interne.
Discussione
Il KESM consente una submicrometer livello sondaggio di grandi volumi di campione biologico (~ 1 cm 3). Questo tipo di volume è sufficiente a contenere interi organi per piccoli animali, come cervello, polmoni, cuori, reni, ecc acquisite le immagini da tali organi senza precedenti in grado di fornire informazioni quantitative circa l'organizzazione strutturale di questi organi, e consentire la modellazione computazionale funzionale di varie aspetti di questi organi, compreso le dinamiche di circuito e di rete, proprietà elettriche, dei fluidi e la dinamica del flusso d'aria, e la dinamica muscolare.
Il protocollo di preparazione dei campioni e post-analisi protocollo dettagliato in questo articolo sono attesi per aiutare i gruppi di ricerca esterni per avere un più facile accesso al KESM nonché i dati risultanti. Il protocollo operazione KESM aiuterà questi ricercatori esterni per apprezzare i vantaggi ei limiti di questa modalità di imaging unico.
Divulgazioni
Todd Huffman è con 3Scan, una società commerciale che produce e commercializza la tecnologia KESM (brevetto US # 6744572).
Riconoscimenti
Questo progetto è stato finanziato dal NIH / NINDS (# 1R01-NS54252), NSF (# 0905041, # 0079874), la tecnologia Texas Advanced Program (# ATP-000512-0146-2001, # ATP-000512-0261-2001), Texas A & M Fondazione per la Ricerca, Dipartimento di Informatica e Ingegneria presso la Texas A & M University, e 3Scan. Vorremmo ringraziare Bernard Mesa (Micro Technologies stella) per consulenza tecnica e supporto per la strumentazione KESM. Parti più importanti della progettazione e realizzazione del KESM è stato fatto da Bruce H. McCormick, morto nel 2007.
Riferimenti
- Abbott, L. C. High-throughput imaging of whole small animal brains with the knife-edge scanning microscope. Neuroscience Meeting Planner. Program No. 504.2, (2008).
- Abbott, L. C., Sotelo, C. Ultrastructural analysis of catecholaminergic innervation in weaver and normal mouse cerebellar cortices. Journal of Comparative Neurology. 426, 316-329 (2000).
- Choe, Y., Abbott, L. C., Miller, D. E., Han, D., Yang, H. -. F., Chung, J. R., Sung, C., Mayerich, D., Kwon, J., Micheva, K., Smith, S. J. Multiscale imaging, analysis, and integration of mouse brain networks. Neuroscience Meeting Planner. Program No. 516.3, (2010).
- Choe, Y., Han, D., Huang, P. -. S., Keyser, J., Kwon, J., Mayerich, D., Abbott, L. C. Complete submicrometer scans of mouse brain microstructure: Neurons and vasculatures. Neuroscience Meeting Planner. Program No. 389.10, (2009).
- Choe, Y., Abbott, L. C., Han, D., Huang, P. S., Keyser, J., Kwon, J., Mayerich, D., Melek, Z., McCormick, B. H., Ravi Rao, A., Cecchi, G. Knife-edge scanning microscopy: High-throughput imaging and analysis of massive volumes of biological microstructures. High-Throughput Image Reconstruction and Analysis: Intelligent Microscopy Applications. , 11-37 (2008).
- Choe, Y., Abbott, L. C., Ponte, G., Keyser, J., Kwon, J., Mayerich, D., Miller, D., Han, D., Grimaldi, A. M., Fiorito, F., Edelman, D. B., McKinstry, J. L. Charting out the octopus connectome at submicron resolution using the knife-edge scanning microscope. BMC Neuroscience. 11, 136-136 (2010).
- Mayerich, D., Abbott, L. C., McCormick, B. H. Knife-edge scanning microscopy for imaging and reconstruction of three-dimensional anatomical structures of the mouse brain. Journal of Microscopy. 231, 134-143 (2008).
- Mayerich, D., Kwon, J., Sung, C., Abbott, L. C., Keyser, J., Choe, Y. Fast macro-scale Transmission Imaging of Microvascular Networks Using KESM. Biomedical Optics Express. 2, 2888-2896 (2008).
- McCormick, B. H. Development of the Brain Tissue Scanner. Technical Report. , (2002).
- Sporns, O., Tononi, G., Kötter, R. The human connectome: A structural description of the human brain. PLoS Computational Biology. 1, 42-42 (2005).
Erratum
Formal Correction: Erratum: Specimen Preparation, Imaging, and Analysis Protocols for Knife-edge Scanning Microscopy
Posted by JoVE Editors on 4/07/2013. Citeable Link.
A correction was made to Specimen Preparation, Imaging, and Analysis Protocols for Knife-edge Scanning Microscopy. Important modifications were made to the Nissl staining protocol.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon